 ,1, 陈晓菲
,1, 陈晓菲 ,1, 王枚阁1, 郑先波1, 宋尚伟1, 焦健1, 王苗苗1, 马锋旺2, 白团辉
,1, 王枚阁1, 郑先波1, 宋尚伟1, 焦健1, 王苗苗1, 马锋旺2, 白团辉 ,1
,1Identification of Candidate Genes for Waterlogging Tolerance in Apple Rootstock by Using SLAF-seq Technique
SONG ChunHui ,1, CHEN XiaoFei
,1, CHEN XiaoFei ,1, WANG MeiGe1, ZHENG XianBo1, SONG ShangWei1, JIAO Jian1, WANG MiaoMiao1, MA FengWang2, BAI TuanHui
,1, WANG MeiGe1, ZHENG XianBo1, SONG ShangWei1, JIAO Jian1, WANG MiaoMiao1, MA FengWang2, BAI TuanHui ,1
,1通讯作者:
责任编辑: 赵伶俐
收稿日期:2021-01-28接受日期:2021-04-13
| 基金资助: |
Received:2021-01-28Accepted:2021-04-13
作者简介 About authors
宋春晖,E-mail:
陈晓菲,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2317KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
宋春晖, 陈晓菲, 王枚阁, 郑先波, 宋尚伟, 焦健, 王苗苗, 马锋旺, 白团辉. 基于SLAF-seq技术鉴定苹果砧木耐涝候选基因. 中国农业科学, 2021, 54(18): 3932-3944 doi:10.3864/j.issn.0578-1752.2021.18.012
SONG ChunHui, CHEN XiaoFei, WANG MeiGe, ZHENG XianBo, SONG ShangWei, JIAO Jian, WANG MiaoMiao, MA FengWang, BAI TuanHui.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】水分是决定植物生长的重要因子,但土壤水分过高则不利于植株生长,甚至导致涝害发生,涝害已成为影响作物生长发育,产量和品质的主要非生物胁迫之一[1,2,3]。苹果(Malus×domestica Borkh)是我国主要栽培果树树种之一,其面积和产量均居世界首位。但在实际生产栽培中,由于夏、秋季的大量集中降雨和果园排水不良等因素,使部分苹果树常处于淹水状态下,导致苹果树叶片黄化、脱落,树体生长受抑制,果实产量和品质下降,严重时甚至植株死亡,造成严重的经济损失,这已成为部分苹果产区生产栽培中亟待解决的问题。因此,深入研究苹果耐涝的遗传机制,鉴定苹果耐涝关键基因,对苹果分子标记辅助育种和优质高产栽培具有重要意义。【前人研究进展】水分过多对植物的伤害并不在于水分本身,而在于淹水导致的根系缺氧[4]。植物在低氧逆境下,有氧呼吸受到抑制,呼吸电子传递受阻或中断,内源激素代谢紊乱,细胞质酸化,乙醇、乳酸、活性氧等有害物质积累,造成生长受抑,严重时导致植物死亡[5,6,7,8,9]。随着现代分子生物学的快速发展,在水稻、玉米、小麦和黄瓜耐涝遗传机制研究方面取得了较大的突破和进展。XU等[10]通过基因定位的方法,找到2个与Sub1位点紧密连锁的标记,证实了Sub1A-1是一个乙烯响应因子,并用转基因的方法验证了Sub1A-1控制水稻的耐涝性。SEPTININGSIH等[11]利用含有敏感等位基因Sub1A-2的两个中等耐淹亲本,鉴定了3个新的耐淹QTL。MANO等[12]将玉米淹水胁迫后不定根形成的相关QTL分别定位到染色体3、4、7和8上。XU等[13]构建了黄瓜的遗传连锁图谱,用图位克隆技术分离到了一个黄瓜耐涝主效基因ARN6.1,该基因通过促进下胚轴不定根形成来适应涝害胁迫。【本研究切入点】简化基因组测序(SLAF-seq)是一种基于高通量测序的目标性状基因定位的技术,具有效率高、成本低、周期短等突出优势,已被广泛应用于作物遗传图谱构建和基因定位[14,15,16]。随着苹果全基因组测序的完成,利用SLAF-seq鉴定苹果砧木耐涝基因已可以实现。前期研究表明新疆野苹果(M. sieverii Roem.)为不耐涝砧木[17,18],G41为耐涝苹果砧木[19],也为耐涝基因的筛选提供了基础。【拟解决的关键问题】本研究以G41和新疆野苹果F1杂交群体及其亲本为材料,基于SLAF-seq技术,利用双亲本与两个极端混池,对苹果耐涝性状进行关联分析,获得目标基因的候选区域,对定位区间的基因进行功能注释,挖掘苹果耐涝的关键基因,旨在为苹果耐涝相关基因的克隆和定向遗传改良提供理论基础。1 材料与方法
试验于2015—2017年在河南农业大学和西北农林科技大学进行。1.1 供试材料
本研究以耐涝砧木G41和不耐涝砧木新疆野苹果(Malus sieversii(Ledeb.)Rome.)为亲本及其构建的495株F1代群体为材料。1.2 涝性性评价分析
将杂交种子层积处理,3月将层积好的种子播种到穴盘,当长到4片叶后,将其移栽入花盆(盆口直径23 cm,盆底直径20 cm,深度25 cm),正常管理。7月,杂交苗平均株高达到70 cm,将杂交苗进行淹水处理,花盆完全浸没于水中,水面高于杂交苗根茎3—5 cm。处理18 d后,统计幼苗的受害情况,然后进行恢复培养,恢复培养7 d后再次统计幼苗的受害情况。涝害分级标准用10级记分制评定[16]。0级:正常;1级:顶尖叶片轻微黄化,1—3片黄化叶;2级:顶尖叶片中黄化,黄化叶片占整个植株叶片数10%以下;3级:顶尖黄化,黄化叶片占整个植株叶片数20%以下;4级:顶尖黄化,叶片下垂,黄化叶片占整个植株叶片数40%以下;5级:顶尖黄化,叶片下垂,黄化叶片占整个植株叶片数50%以下;6级:顶尖萎蔫,黄化叶片(15—20片黄叶)占整个植株的70%以下;7级:顶尖萎蔫,黄化叶片(25以上黄叶)占整个植株的80%以上;8级:叶片黄化干枯脱落,占整个植株的90%以下;9级:干枯死亡。
1.3 DNA提取、混池构建及测序
根据耐涝性评价分析结果,从495个株系中选出耐涝(0级)50株和不耐涝(9级)50株,每个株系分别提取基因组DNA,经纯度和完整性检测合格后,然后将各个混池中的基因组DNA等量混合,构建两个极端DNA混池,同时提取亲本G41和新疆野苹果的DNA,使用Rsa I和Hae Ⅲ限制性内切酶酶切基因组DNA,进行简化基因组测序,测序由北京百迈客公司完成,具体步骤参照SUN等[20]的方法。1.4 SLAF标签和SNP标记的开发
将测序数据过滤掉低质量reads后,使用BWA软件将过滤后的clean reads比对到苹果‘金冠’参考基因组GDDH13(1.5 候选基因的定位及分析
采用欧式距离法(euclidean distance,ED)计算每个位点ED值,根据ED值寻找混池间存在显著差异的标记,并以此评估所获得的分子标记与目标性状紧密关联的区域[21]。通过SNP-index关联分析计算混池间的基因型频率,获得混池间存在显著差异的基因型频率,记为ΔSNP-index[22]。将欧式距离法和SNP-index方法得到的关联区域进行综合分析。1.6 ADH1表达分析
选择耐涝和不耐涝的植株,进行淹水处理,于处理第0、0.5、1、2、4和6天采样,使用改良CTAB法提取总RNA,使用PrimeScriptTM RT Reagent Kit with gDNA Eraser试剂盒(宝生物工程(大连)有限公司)对RNA进行反转录获得cDNA。使用primer3软件设计定量引物,ADH1定量引物为:F:5′-GGTCTC GCTGTACTTGTTGGT-3′和R:5′-CGAATGGGACCG AATGAGTGAT-3′。定量仪器使用7500型实时荧光定量PCR(Applied Biosystems 7500 Fast Real Time PCR)。反应体系为20 μL:10 μL ChamQ Universal SYBR qPCR Master Mix(南京诺唯赞生物科技股份有限公司),1 μL cDNA模板,上、下游引物(10 μmol∙L-1)各1 μL,7 μL无菌蒸馏水。运行程序为:95℃预变性30 s;95℃变性3 s,60℃退火30 s,40个循环;95℃ 15 s,60℃ 60 s,95℃ 15 s。Actin作为内参,使用2-∆∆CT法计算基因相对表达量。2 结果
2.1 耐涝性评价与分析
淹水胁迫后,从第3天开始,植株表现出不同程度的受害症状,主要表现为叶尖、叶缘变黄,叶片脱落,甚至死亡。杂交后代不同株系的耐涝性存在显著差异,淹水18 d,有61个株系(12.3%)死亡,349个株系(70.6%)表现出不同程度的受害症状,然而84个株系(17.0%)未表现任何受害症状(图1、2)。从图2可以看出,G41×新疆野苹果杂交后代F1分离群体耐涝性呈现连续性分布,表明苹果耐涝性状为数量性状。淹水胁迫18 d后,进行恢复培养7 d,植株受害加重,90个株系(18.2%)死亡,158个株系(31.98%)受害严重,表现为叶片脱落,然而62个株系(12.6%)未表现任何受害症状。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1淹水胁迫18 d并恢复培养7 d后植株的受害症状
A:0级,B:1级,C:5级,D:9级
Fig. 1Plant symptoms after waterlogging for 18 days and recovery for 7 days
A: 0 grade; B: 1 grade; C: 5 grade; D: 9 grade
图2
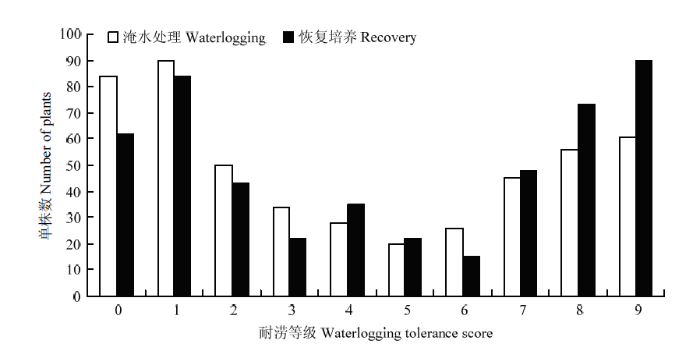 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2G41×新疆野苹果F1代植株淹水18 d并恢复培养7 d后的耐涝性评价
Fig. 2The waterlogging tolerance evaluation of the F1 progeny of G41×M. sieversii after waterlogging for 18 days and recovery for 7 days
2.2 SLAF-seq测序质量分析
根据耐涝性评价分析结果,选择50株耐涝和50株不耐涝株系,分别组成耐涝混池和不耐涝混池,分别提取基因组DNA,然后将各个混池中每个个体的基因组DNA等量混合,使用Rsa I和Hae Ⅲ限制性内切酶酶切基因组DNA后,进行简化基因组测序,同时对亲本G41和新疆野苹果进行基因组重测序。测序结果显示,G41和新疆野苹果总reads数分别为38 365 575和39 983 972,reads Q30所占比例分别为85.02%和87.82%。耐涝混池和不耐涝混池总reads数分别为10 873 511和10 279 180,reads Q30所占比例分别为89.47%和88.94%(表1)。以上数据表明测序数据充足,质量合格,可用于下一步分析。通过BWA软件将各个样本的reads比对到参考基因组上,亲本G41和新疆野苹果总比对率分别为97.55%和97.48%,基因组平均覆盖度为12X。耐涝混池和不耐涝混池reads总比对率分别为94.94%和93.01%,平均覆盖度为3X。染色体覆盖深度统计结果显示基因组被覆盖得较均匀,说明测序随机性较好。Table 1
表1
表1样品测序质量及与参考基因组的匹配情况
Table 1
| 样本 Sample | 干净reads Clean reads | 干净碱基 Clean base | Q30 (%) | GC (%) | 总比对率 Total mapped (%) | 正确比对率 Properly mapped (%) |
|---|---|---|---|---|---|---|
| G41 | 38 365 575 | 9 666 919 570 | 85.02 | 41.1 | 97.55 | 87.64 |
| 新疆野苹果M. sieversii | 39 983 972 | 10 067 312 448 | 87.82 | 41.61 | 97.48 | 85.77 |
| 耐涝混池 Tolerant pool | 10 873 511 | 2 174 702 200 | 89.47 | 39.89 | 94.94 | 82.30 |
| 不耐涝混池 Sensitive pool | 10 279 180 | 2 055 836 000 | 88.94 | 39.86 | 93.01 | 80.64 |
新窗口打开|下载CSV
2.3 SLAF标签和SNP标记的开发
将经Rsa I和Hae Ⅲ限制性内切酶酶切基因组DNA,片段长度在364—414 bp的序列定义为SLAF标签。经同一位点reads序列的相似性聚类和序列分析,共开发出119 072个SLAF标签,每个染色体SLAF标签变化范围为3 011—6 297个(表2),平均测序深度为50.44X,达到了SLAF标签预期。根据等位基因和基因序列之间的差异分析,从SLAF标签中共鉴定到多态性SLAF有11 133个。通过BWA软件将SLAF标签和多态性SLAF标记比对到参考基因组上,如图4所示,SLAF标签和多态性SLAF标记比较均匀地分布在苹果17条染色体上(图3-A、B)。Table 2
表2
表2SLAF标签和多态性SLAF标签在染色体分布
Table 2
| 染色体 Chromosome | SLAF 数目 SLAF number | 多态性 SLAF Polymorphic SLAF |
|---|---|---|
| Chr 01 | 3 809 | 354 |
| Chr 02 | 4 892 | 530 |
| Chr 03 | 4 628 | 436 |
| Chr 04 | 3 224 | 251 |
| Chr 05 | 4 304 | 495 |
| Chr 06 | 3 595 | 308 |
| Chr 07 | 3 620 | 383 |
| Chr 08 | 3 880 | 394 |
| Chr 09 | 4 790 | 455 |
| Chr 10 | 4 474 | 423 |
| Chr 11 | 4 717 | 435 |
| Chr 12 | 4 234 | 418 |
| Chr 13 | 5 244 | 486 |
| Chr 14 | 4 038 | 405 |
| Chr 15 | 6 297 | 567 |
| Chr 16 | 3 011 | 281 |
| Chr 17 | 3 214 | 337 |
| Chr 00 | 47 101 | 4175 |
| 总计Total | 119 072 | 11133 |
新窗口打开|下载CSV
图3
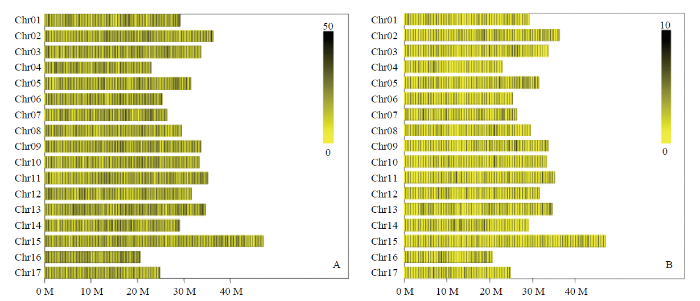 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3SLAF标签和多态性SLAF标签在苹果染色体上的分布情况
A:SLAF标签在苹果染色体上的分布情况;B:多态性SLAF标签在苹果染色体上的分布情况。每一个黄色条带代表一条染色体,图中颜色越深的区域即SLAF标签分布越集中的区域
Fig. 3Distribution of SLAF and polymorphism SLAF on the apple reference genome
A: Distribution of total SLAFs on the reference genome; B: Distribution of polymorphic SLAFs on the reference genome. Each yellow strip represents a chromosome, the darker the color area is SLAF label concentrated distribution area
图4
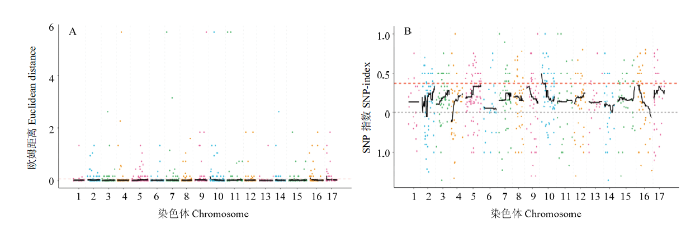 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4ED关联值和SNP-index关联值在染色体上的分布
Fig. 4Distribution of ED and SNP index association values on chromosome
使用GATK检测SNP位点,亲本间和混池间分别获得5 093 595和1 143 476个SNP(表3)。对初步鉴定到的SNP进一步过滤,过滤掉有多个基因型的SNP位点(21 715个),reads支持度小于4的SNP位点(5 950 658)、混池之间基因型一致的SNP位点(0个)以及隐性混池基因不是来自于隐性亲本的SNP位点(94 081),最终得到170 617个高质量SNP,用于后续关联分析。
Table 3
表3
表3样品SNP信息统计
Table 3
| 样本 Sample | 总SNP Total SNP | SNP数目 SNP number | 杂合率 Heterozygous ratio (%) |
|---|---|---|---|
| G41 | 5 093 595 | 3 313 306 | 35.79 |
| 新疆野苹果M. sieversii | 5 093 595 | 4 208 748 | 53.41 |
| 耐涝混池 Tolerant pool | 1 143 476 | 1 104 683 | 51.19 |
| 不耐涝混池 Sensitive pool | 1 143 476 | 1 090 476 | 49.99 |
新窗口打开|下载CSV
通过欧氏距离(ED)算法,利用11 133个多态性SLAF进行苹果砧木耐涝性状的关联分析,计算出关联阈值为0.063。ED关联值在染色体上的分布如图4-A,根据关联阈值判定,共得到2个区域,总长度为1.32 Mb,共包含124个基因,其中非同义突变SNP位点的基因共73个(表4)。
Table 4
表4
表4苹果砧木耐涝性状的关联分析
Table 4
| 方法 Method | 染色体 Chromosome | 起始位置 Start | 终止位置 End | 大小 Size (Mb) | 基因数量 Gene number |
|---|---|---|---|---|---|
| 欧式距离法 Euclidean distance | Chr10 | 1942245 | 3252362 | 1.31 | 121 |
| Chr17 | 1839746 | 1853187 | 0.01 | 3 | |
| SNP-index法 SNP-index method | Chr10 | 12955610 | 13906230 | 0.95 | 45 |
| Chr10 | 1942245 | 7959407 | 6.02 | 445 | |
| 交集 Intersection | Chr10 | 1942245 | 3252362 | 1.31 | 120 |
新窗口打开|下载CSV
利用SNP-index方法计算关联值,以‘金冠’苹果为参考基因组,用170 617个SNP位点进行苹果砧木耐涝性状的关联分析。分析每个SNP位点在每个池中的SNP-index(即SNP的频率)。为了消除假阳性位点,利用标记在基因组上的位置,对同一条染色体上标记的ΔSNP-index值进行拟合,并进一步采用DISTANCE法对ΔSNP-index进行拟合。利用拟合后ΔSNP-index的99百分位数,即0.375,共得到2个区域,总长度为6.97 Mb,共包含490个基因,其中非同义突变的基因共311个(图4-B、表4)。
将采用欧式距离法和SNP-index方法得到的关联区域进行综合分析,取其交集,将控制苹果砧木耐涝性状的基因定位在第10号染色体1.94—3.25 Mb,关联区域大小为1.31 Mb,关联区域内包含120个基因,编码区存在非同义突变的基因66个(表4、表5)。
Table 5
表5
表5定位区间基因功能预测
Table 5
| 编号No | 基因编号Gene ID | 注释信息 Function annotation |
|---|---|---|
| 1 | MD10G1014300 | 生长素输出载体家族蛋白 Auxin efflux carrier family protein |
| 2 | MD10G1014400 | 核小分子RNA71 SnoR71 |
| 3 | MD10G1014500 | 乙醇脱氢酶1 Alcohol dehydrogenase 1 |
| 4 | MD10G1014600 | 5S核糖体RNA 5S rRNA |
| 5 | MD10G1014700 | 5S核糖体RNA 5S rRNA |
| 6 | MD10G1014800 | 植物硬脂酰酰基载体蛋白去饱和酶家族蛋白 Plant stearoyl-acyl-carrier-protein desaturase family protein |
| 7 | MD10G1014900 | 管状分子分化相关6 Tracheary element differentiation-related 6 |
| 8 | MD10G1015000 | 表皮毛双折射7 TRICHOME BIREFRINGENCE-LIKE 7 |
| 9 | MD10G1015100 | 表皮毛双折射7 TRICHOME BIREFRINGENCE-LIKE 7 |
| 10 | MD10G1015200 | 植物U3 Plant U3 |
| 11 | MD10G1015300 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 12 | MD10G1015400 | 非编码RNA ncRNA |
| 13 | MD10G1015500 | 丝氨酸羧肽酶42 Serine carboxypeptidase-like 42 |
| 14 | MD10G1015600 | 三角状五肽重复序列(PPR)超家族蛋白 Pentatricopeptide repeat (PPR) superfamily protein |
| 15 | MD10G1015700 | 三角状五肽重复序列(PPR)超家族蛋白 Pentatricopeptide repeat (PPR) superfamily protein |
| 16 | MD10G1015800 | 植物糖原蛋白样淀粉起始蛋白2 Plant glycogenin-like starch initiation protein 2 |
| 17 | MD10G1015900 | 植物糖原蛋白样淀粉起始蛋白2 Plant glycogenin-like starch initiation protein 2 |
| 18 | MD10G1016000 | 液泡蛋白分选45 Vacuolar protein sorting 45 |
| 19 | MD10G1016100 | 液泡蛋白分选45 Vacuolar protein sorting 45 |
| 20 | MD10G1016200 | 5S核糖体RNA 5S rRNA |
| 21 | MD10G1016300 | 功能未知的蛋白(DUF506) Protein of unknown function (DUF506) |
| 22 | MD10G1016400 | 高尔基核苷酸糖转运蛋白1 Golgi nucleotide sugar transporter 1 |
| 23 | MD10G1016500 | ABC转运蛋白家族蛋白 ABC transporter family protein |
| 24 | MD10G1016600 | 功能未知的蛋白质(DUF300) Protein of unknown function (DUF300) |
| 25 | MD10G1016700 | snoZ221 snoR21b |
| 26 | MD10G1016800 | 发病相关家族蛋白 Pathogenesis-related family protein |
| 27 | MD10G1016900 | 核糖体RNA 5S rRNA 5S |
| 28 | MD10G1017000 | snoZ221 snoR21b |
| 29 | MD10G1017100 | 未知 Unknown |
| 30 | MD10G1017200 | 5S核糖体RNA 5S rRNA |
| 31 | MD10G1017300 | 发病相关家族蛋白 Pathogenesis-related family protein |
| 32 | MD10G1017400 | 糖脂转移蛋白2 Glycolipid transfer protein 2 |
| 33 | MD10G1017500 | 未表征的蛋白质 Uncharacterized protein |
| 34 | MD10G1017600 | 真核生物翻译起始因子6-2 Eukaryotic translation initiation factor 6-2 |
| 35 | MD10G1017700 | 功能未知的蛋白质 Protein of unknown function |
| 36 | MD10G1017800 | Dof型锌指DNA结合家族蛋白 Dof-type zinc finger DNA-binding family protein |
| 37 | MD10G1017900 | ABC转运G家族成员22 ABC transporter G family member 22-like |
| 38 | MD10G1018000 | MAP激酶底物1 MAP kinase substrate 1 |
| 39 | MD10G1018100 | 核内体靶向BRO1样结构域含蛋白 Endosomal targeting BRO1-like domain-containing protein |
| 编号No | 基因编号Gene ID | 注释信息 Function annotation |
| 40 | MD10G1018200 | RING / FYVE / PHD锌指超家族蛋白 RING/FYVE/PHD zinc finger superfamily protein |
| 41 | MD10G1018300 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 42 | MD10G1018400 | 抗病蛋白(TIR-NBS-LRR类)家族 Disease resistance protein (TIR-NBS-LRR class) family |
| 43 | MD10G1018500 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 44 | MD10G1018600 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 45 | MD10G1018700 | 嘌呤生物合成4 Purine biosynthesis 4 |
| 46 | MD10G1018800 | 未知 Unknown |
| 47 | MD10G1018900 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 48 | MD10G1019000 | 未知 Unknown |
| 49 | MD10G1019100 | 铜转运蛋白2 Copper transporter 2-like |
| 50 | MD10G1019200 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 51 | MD10G1019300 | 小核糖核蛋白家族蛋白 Small nuclear ribonucleoprotein family protein |
| 52 | MD10G1019400 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 53 | MD10G1019500 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 54 | MD10G1019600 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 55 | MD10G1019700 | 抗病蛋白(TIR-NBS-LRR类)家族 Disease resistance protein (TIR-NBS-LRR class) family |
| 56 | MD10G1019800 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 57 | MD10G1019900 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 58 | MD10G1020000 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 59 | MD10G1020100 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 60 | MD10G1020200 | 未知 Unknown |
| 61 | MD10G1020300 | 伴刀豆球蛋白A样凝集素蛋白激酶家族蛋白 Concanavalin A-like lectin protein kinase family protein |
| 62 | MD10G1020400 | 含LIM结构域的蛋白质 LIM domain-containing protein |
| 63 | MD10G1020500 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 64 | MD10G1020600 | 非编码RNA ncRNA |
| 65 | MD10G1020700 | 线粒体底物载体家族蛋白 Mitochondrial substrate carrier family protein |
| 66 | MD10G1020800 | 含LIM结构域的蛋白质 LIM domain-containing protein |
| 67 | MD10G1020900 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 68 | MD10G1021000 | 抗病蛋白(TIR-NBS-LRR类) Disease resistance protein (TIR-NBS-LRR class) |
| 69 | MD10G1021100 | 功能未知的蛋白(DUF506) Protein of unknown function (DUF506) |
| 70 | MD10G1021200 | β-酮酰基还原酶1 Beta-ketoacyl reductase 1 |
| 71 | MD10G1021300 | 具有 FYVE 锌指结构域的染色体浓缩(RCC1)家族调节因子 Regulator of chromosome condensation (RCC1) family with FYVE zinc finger domain |
| 72 | MD10G1021400 | 植物U-box 29 Plant U-box 29 |
| 73 | MD10G1021500 | RNA结合蛋白2 RNA-binding protein 2-like |
| 74 | MD10G1021600 | 核苷酸结合 Nucleotide binding |
| 75 | MD10G1021700 | 未知 Unknown |
| 76 | MD10G1021800 | 非编码RNA ncRNA |
| 77 | MD10G1021900 | ACT样蛋白酪氨酸激酶家族蛋白 ACT-like protein tyrosine kinase family protein |
| 78 | MD10G1022000 | 碳酸酐酶1 Carbonic anhydrase 1 |
| 79 | MD10G1022100 | F盒家族蛋白 F-box family protein |
| 编号No | 基因编号Gene ID | 注释信息 Function annotation |
| 80 | MD10G1022200 | 碳酸酐酶2 Carbonic anhydrase 2 |
| 81 | MD10G1022300 | 未知 Unknown |
| 82 | MD10G1022400 | 碳酸酐酶2 Carbonic anhydrase 2 |
| 83 | MD10G1022500 | RmlC样铜蛋白超家族蛋白 RmlC-like cupins superfamily protein |
| 84 | MD10G1022600 | RmlC样铜蛋白超家族蛋白 RmlC-like cupins superfamily protein |
| 85 | MD10G1022700 | 未知 Unknown |
| 86 | MD10G1022800 | 未知 Unknown |
| 87 | MD10G1022900 | RmlC样铜蛋白超家族蛋白 RmlC-like cupins superfamily protein |
| 88 | MD10G1023000 | 综合调控因子2 General regulatory factor 2 |
| 89 | MD10G1023100 | 样铜蛋白超家族蛋白 RmlC-like cupins superfamily protein RmlC |
| 90 | MD10G1023200 | TCP-1/cpn60伴侣蛋白家族蛋白 TCP-1/cpn60 chaperonin family protein |
| 91 | MD10G1023300 | 含LIM结构域的蛋白质 LIM domain-containing protein |
| 92 | MD10G1023400 | 泛素载体蛋白7 Ubiquitin carrier protein 7 |
| 93 | MD10G1023500 | 胚芽蛋白样蛋白2 Germin-like protein 2 |
| 94 | MD10G1023600 | 磷酸吡rid醛(PLP)依赖性转移酶 Pyridoxal phosphate (PLP)-dependent transferases superfamily protein |
| 95 | MD10G1023700 | 5S 核糖体RNA 5S rRNA |
| 96 | MD10G1023800 | VIRE2相互作用蛋白 1VIRE2-interacting protein 1 |
| 97 | MD10G1023900 | Tho复合亚基7 / Mft1p Tho complex subunit 7/Mft1p |
| 98 | MD10G1024000 | 液泡铁转运蛋白(VIT)家族蛋白 Vacuolar iron transporter (VIT) family protein |
| 99 | MD10G1024100 | 真核翻译起始因子2(eIF-2)家族蛋白 Eukaryotic translation initiation factor 2 (eIF-2) family protein |
| 100 | MD10G1024200 | 非编码RNA ncRNA |
| 101 | MD10G1024300 | 泛素相互作用基序蛋白 Ubiquitin interaction motif-containing protein |
| 102 | MD10G1024400 | 果糖-1,6-二磷酸酶 Fructose-1,6-bisphosphatase |
| 103 | MD10G1024500 | 小核糖核蛋白家族蛋白 Small nuclear ribonucleoprotein family protein |
| 104 | MD10G1024600 | 小核糖核蛋白家族蛋白 Small nuclear ribonucleoprotein family protein |
| 105 | MD10G1024700 | U6 |
| 106 | MD10G1024800 | 序列特异性DNA结合转录因子 Sequence-specific DNA binding transcription factors |
| 107 | MD10G1024900 | 非编码RNA ncRNA |
| 108 | MD10G1025000 | 未知 Unknown |
| 109 | MD10G1025100 | Trihelix转录因子 Trihelix transcription factor |
| 110 | MD10G1025200 | Trihelix转录因子 Trihelix transcription factor |
| 111 | MD10G1025300 | 结瘤素MtN21/EamA样转运蛋白家族蛋白 Nodulin MtN21/EamA-like transporter family protein |
| 112 | MD10G1025400 | U6 |
| 113 | MD10G1025500 | DEAD-box ATP依赖性RNA解旋酶21 DEAD-box ATP-dependent RNA helicase 21-like |
| 114 | MD10G1025600 | 桶状样F-box蛋白8(LOC103444645),mRNA Tubby-like F-box protein 8 (LOC103444645), mRNA |
| 115 | MD10G1025700 | 核小分子RNA118 snoR118 |
| 116 | MD10G1025800 | 未知 Unknown |
| 117 | MD10G1025900 | 功能未知的蛋白质(DUF793) Protein of unknown function (DUF793) |
| 118 | MD10G1026000 | 非编码RNA ncRNA |
| 119 | MD10G1026100 | HSP20样伴侣蛋白超家族蛋白 HSP20-like chaperones superfamily protein |
| 120 | MD10G1026200 | 铜氧还蛋白超家族蛋白 Cupredoxin superfamily protein |
新窗口打开|下载CSV
2.4 候选区域基因功能注释
根据关联定位区间,提取‘金冠’参考基因组(GDDH13)第10号染色体1.94—3.25 Mb基因组信息。结果显示,关联区域内共包含120个基因(表5),其中6个为非编码RNA(ncRNA),10个基因功能未知,其余为编码蛋白质的基因。在114个编码蛋白质的基因中有5个转录因子,分别为Dof(MD10G1017800)、bZIP(MD10G1023800)、Trihelix(MD10G1024800、MD10G1025100、MD10G1025200)。5个转运蛋白,分别为液泡铁转运蛋白VIT(MD10G1024000)、生长素输出载体PIN(MD10G1014300)、甘露醇运输蛋白VRG4(MD10G1016400)、铜转运蛋白SLC31A1(MD10G1019100)、线粒体S-腺苷甲硫胺酸运输蛋白SLC25A26(MD10G1020700),还有碳代谢相关基因果糖-1,6磷酸酶(MD10G1024400),乙醇脱氢酶ADH1(MD10G1014500),脂质代谢途径酰基-酰基载体蛋白质去饱和酶FAB2(MD10G1014800)、17-雌二醇17-脱氢酶/长链3-氧酰基辅酶a还原酶HSD17B12(MD10G1021200)等。2.5 ADH1表达分析
进一步对候选基因乙醇脱氢酶基因ADH1进行定量表达分析,结果显示(图5),在不耐涝植株中,ADH1在淹水处理0.5 d时显著高表达,之后迅速降低。而在耐涝植株中,ADH1表达波动较小。ADH1在淹水处理0.5 d时,在不耐涝植株中的表达量显著高于耐涝植株,而在淹水处理第1、2、4和6 d,ADH1在耐涝植株中的表达量显著高于不耐涝植株。图5
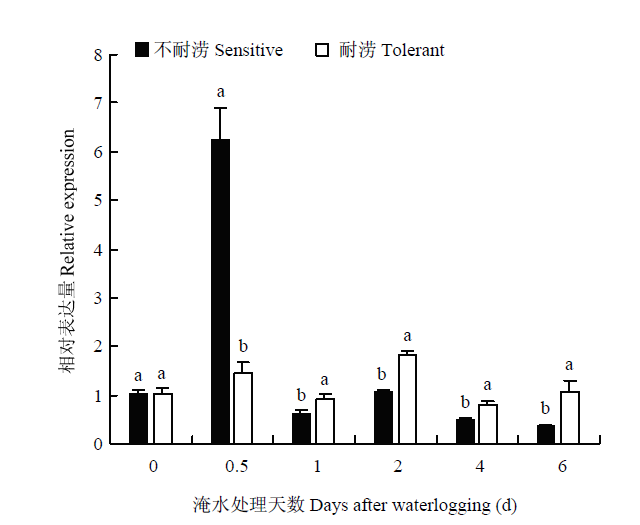 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5淹水胁迫下ADH1表达分析
不同小写字母表示差异显著(P<0.05)
Fig. 5Expression analysis of ADH1under waterlogging stress
Different lowercase letters indicate significant difference (P<0.05)
3 讨论
随着全球变暖,极端气候频繁出现,降雨量分布不均衡,经常发生大面积不同程度的涝害,对农作物、蔬菜和果树等的产量和品质造成了严重影响[3,6,8]。目前,针对猕猴桃[23]、桃[5,24]、葡萄[25,26]和苹果[9,17,19,27]等果树开展了耐涝研究,但大多数集中在表型鉴定和生理生化水平。SLAF-seq技术具有低成本且高通量开发SNP标记及基因分型的优势,国内外研究者利用此技术在多种作物上获得了与目标性状显著相关的分子标记及候选基因[13,14,15,16]。SONG等[28]用SLAF-seq技术定位了水稻耐冷性状的QTL。DONG等[14]利用SLAF-Seq技术将大豆矮化基因定位到了第19条染色体上,关联区域为80.72 kb,并预测了3个与大豆矮化的相关基因。贾秀苹等[29]通过此技术挖掘到了6个向日葵耐盐候选基因。因此,SLAF-Seq技术能广泛应用于基因定位,对相关性状候选基因的预测,可为基因的精细定位和克隆奠定基础。本研究采用SLAF-seq技术共开发了多态性SLAF有11 133个,通过序列分析和检测SNP位点,共获得了高质量SNP有170 617个。最终获得一个与耐涝性状紧密关联的候选区域,位于苹果第10号染色体1.94—3.25 Mb。水涝胁迫对植物的伤害程度因物种而异,即使同一物种不同基因型间也存在显著差异,因此,阐明植物对水涝胁迫响应的差异机理和耐涝遗传机制是研究其对植物伤害的前提和基础。深水稻采取避淹策略,通过节间伸长生长使植株顶端浮出水面与空气保持接触,以避免完全淹没[30]。浅水稻采取耐淹策略,在淹水期间表现为缓慢生长、维持存活,避免与茎秆伸长等能量消耗,当水退去后,再利用保存的能量恢复生长[10,31]。黄瓜在淹水情况下,会在根茎处形成不定根,通过不定根从空气中获取氧气[13]。苹果耐涝的表型明显不同于深水稻的“避淹策略”。本研究使用一年生实生苗为材料,试验中出现不定根的情况比较少见,但在涝害严重的成年苹果园中,一些苹果树会在地面附近形成粗短的气生根,来适应涝害。因此,推测苹果可能通过不同的策略抵抗涝害,一种是形成气生根;另一种是在淹没期间表现为缓慢生长且维持存活,以避免能量消耗。前期研究表明淹水造成低氧逆境对呼吸代谢系统及相关基因表达都产生了显著影响[32]。但这些基因是如何应答淹水胁迫、作用大小及其相互关联方式,目前尚不十分清楚。
本研究在苹果耐涝性状关联区域,共注释到120个基因,其中有5个转录因子。在这些基因中,推测ADH1是一个潜在的重要基因。本研究结果显示随着淹水处理的延长,ADH1在耐涝植株中的表达量显著高于不耐涝植株。淹水胁迫会提高乙醇脱氢酶ADH1活性,为细胞维持必要的生命活动提供能量[32]。植物在淹水条件下,降低的氧气水平通过干扰电子传递链,抑制ATP的产生,限制了线粒体的呼吸作用,进而导致植物对能量的需求增加。当淹水胁迫造成能量短缺时,植物通过糖酵解和乙醇发酵途径获得必需的能量[33]。丙酮酸可通过乳酸脱氢酶LDH转化为乳酸,或丙酮酸可通过丙酮酸脱羧酶PDC脱羧形成乙醛,再通过乙醇脱氢酶ADH还原为乙醇[6,34]。控制浅水稻耐淹性状的主效基因SUB1A可以增强乙醇脱氢酶ADH1的表达[31]。在淹水胁迫下,GmADH2转基因大豆种子萌发能力得到增强[34]。因此,推测乙醇脱氢酶基因ADH1可能在苹果砧木耐涝中起重要作用,下一步将对该基因进行功能验证。
4 结论
本研究利用耐涝苹果砧木G41和不耐涝苹果砧木新疆野苹果及其构建F1杂交群体为材料,基于SLAF-seq技术,共开发了119 072个SLAF标签,其中多态性SLAF有11 133个,共获得6 237 071个SNP,其中高质量SNP有170 617个。通过ED和SNP-index方法关联分析,获得1个与耐涝性状紧密关联的候选区域,位于苹果第10号染色体1.94—3.25 Mb,关联区域大小为1.31 Mb,关联区域内包含120个基因,对该区域内基因进行功能注释,发现1个与呼吸代谢相关的基因乙醇脱氢酶基因ADH1,可能在苹果砧木耐涝中起重要作用。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1007/s11032-005-5911-2URL [本文引用: 1]
DOI:10.1016/j.fcr.2015.05.001URL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
DOI:10.1093/aob/mci206URL [本文引用: 1]
DOI:10.1016/j.envexpbot.2016.01.009URL [本文引用: 2]
DOI:10.1016/j.plaphy.2020.01.020URL [本文引用: 3]
DOI:10.1007/s11104-009-0034-xURL [本文引用: 1]
DOI:10.1016/j.plaphy.2013.02.015URL [本文引用: 2]
[本文引用: 2]
[本文引用: 2]
DOI:10.1038/nature04920URL [本文引用: 2]
DOI:10.1007/s00122-011-1751-0URL [本文引用: 1]
DOI:10.1007/s11032-007-9132-8URL [本文引用: 1]
DOI:10.1111/tpj.2018.93.issue-5URL [本文引用: 3]
DOI:10.1007/s10681-020-02633-7URL [本文引用: 3]
DOI:10.3389/fgene.2020.00178URL [本文引用: 2]
DOI:10.1186/s12870-019-2164-5URL [本文引用: 3]
[本文引用: 2]
[本文引用: 2]
DOI:10.1111/ppl.2013.147.issue-4URL [本文引用: 1]
DOI:10.1016/j.plaphy.2016.02.006URL [本文引用: 2]
DOI:10.1371/journal.pone.0058700URL [本文引用: 1]
DOI:10.1101/gr.146936.112URL [本文引用: 1]
DOI:10.1111/tpj.2013.74.issue-1URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s00344-008-9051-xURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/j.envexpbot.2020.104111URL [本文引用: 1]
DOI:10.3389/fpls.2018.00875URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/nature08258URL [本文引用: 1]
DOI:10.1105/tpc.106.043000URL [本文引用: 2]
DOI:10.1016/j.scienta.2009.12.029URL [本文引用: 2]
DOI:10.1007/s11240-011-0098-1URL [本文引用: 1]
DOI:10.5511/plantbiotechnology.12.0301aURL [本文引用: 2]
