 ,, 马小龙, 张敏, 邓平川, 陈春环, 张宏
,, 马小龙, 张敏, 邓平川, 陈春环, 张宏 ,, 刘新伦
,, 刘新伦 ,, 吉万全
,, 吉万全 ,西北农林科技大学农学院/旱区作物逆境生物学国家重点实验室,陕西杨凌 712100
,西北农林科技大学农学院/旱区作物逆境生物学国家重点实验室,陕西杨凌 712100Post-transcriptional Regulation of TaNAC Genes by Alternative Splicing and MicroRNA in Common Wheat (Triticum aestivum L.)
LÜ ShiKai ,, MA XiaoLong, ZHANG Min, DENG PingChuan, CHEN ChunHuan, ZHANG Hong
,, MA XiaoLong, ZHANG Min, DENG PingChuan, CHEN ChunHuan, ZHANG Hong ,, LIU XinLun
,, LIU XinLun ,, JI WanQuan
,, JI WanQuan ,College of Agronomy, Northwest A&F University/State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi
,College of Agronomy, Northwest A&F University/State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi通讯作者:
责任编辑: 李莉
收稿日期:2021-02-10接受日期:2021-04-7
| 基金资助: |
Received:2021-02-10Accepted:2021-04-7
作者简介 About authors
吕士凯,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (3479KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
吕士凯, 马小龙, 张敏, 邓平川, 陈春环, 张宏, 刘新伦, 吉万全. 小麦TaNAC基因基于可变剪切和microRNA的转录后调控分析. 中国农业科学, 2021, 54(22): 4709-4727 doi:10.3864/j.issn.0578-1752.2021.22.001
LÜ ShiKai, MA XiaoLong, ZHANG Min, DENG PingChuan, CHEN ChunHuan, ZHANG Hong, LIU XinLun, JI WanQuan.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】NAC(no apical meristem,NAM;Arabidopsis transcription activation factor,ATAF;cup-shaped cotyledon,CUC)转录因子基因家族几乎在植物的所有组织器官,以及包括形态发生、生长发育、衰老、对生物和非生物逆境胁迫响应等在内的生命过程各个阶段均发挥重要作用[1,2,3,4,5]。前期研究发现,在白粉菌(Powdery mildew,P,Blumeria graminis f. sp. tritici)和条锈菌(Stripe rust,S,Puccinia striiformis Westend. f. sp. Tritici Eriks.)胁迫下,普通小麦(Triticum aestivum L.)NAC转录因子的编码基因会由可变剪切(alternative splicing,AS)等转录后调控形成大量的结构变异转录本[6]。此外,小麦NAC转录因子的表达会受到microRNA(miRNA)的调控[7]。了解小麦中TaNAC可变剪切事件,分析TaNAC的miRNA调控因子,对解析小麦通过可变剪切和miRNA转录后调控方式响应真菌胁迫的机制具有重要作用。【前人研究进展】NAC转录因子是植物特有且家族成员数量最多的一类转录因子基因家族[8]。NAC转录因子得名于其保守的一致性序列,一般包括来自矮牵牛(Petunia hybrida)的NAM以及来自拟南芥(Arabidopsis thaliana)的转录激活因子ATAF1,2和CUC2[9,10]。NAC转录因子的蛋白序列一般包括一个高度保守且具有DNA结合结构域的N末端,以及行使转录调控功能且结构多变的C末端[11,12]。据报道,模式植物拟南芥和水稻(Oryza sativa L.)的转录因子家族分别有138和170个成员[13,14],而普通小麦为异源六倍体,具有庞大的基因组,其NAC转录因子家族成员众多,中国春小麦参考基因组(IWGSC RefSeq v1.1)有460个NAC基因位点,注释的转录本数量更是多达559条[6,15]。目前,关于NAC转录因子的功能研究已有很多报道,仅在普通小麦中就有涉及叶片衰老等生长发育相关的NAM-A1、TaNAC-S等[16,17],参与低温、干旱等非生物逆境胁迫响应的TaNAC69、TaNAC47等[18,19],参与条锈病、赤霉病抗性等生物逆境胁迫响应相关的TaNAC30、TaNACL-D1等[20,21],也有基于全基因组层面的对小麦NAC转录因子家族成员的特性进行系统分析的报道[6, 15]。NAC转录因子在行使转录调控功能时,其自身一般也会受到转录调控、转录后调控、翻译后调控等多种不同层次的调控[22]。其中,NAC转录因子的转录后调控一般与胁迫响应有关[22],其转录后调控包括miRNA和可变剪切等调控方式。miRNA是一类广泛存在于真核生物、长度为20—24个核苷酸的内源非编码单链RNA,在多种生物过程中发挥重要的转录后调控作用[8]。通过与基因的ORF区结合,裂解靶标mRNA和抑制其翻译,是miRNAs分子发挥转录后调控功能的一种重要方式[23]。据报道,一些NAC转录因子会受到miR164家族成员的调控,如tae-miR164和TaNAC21/22互作,调控小麦对条锈病菌的抗性[7]。可变剪接作为一种重要的转录后调控方式,可以改变mRNA的序列结构,进而在植物抗病等压力胁迫响应过程中发挥调节作用[24]。【本研究切入点】普通小麦中含有数量众多的NAC转录因子,但TaNAC相关研究与其数量相比却相对不足[7,20,25]。植物NAC基因含有大量可变剪切事件,但是关于它们的这种转录后调控方式研究有限[6,26],在小麦中关于TaNAC基因可变剪切的研究更是少之又少[6]。可变剪切带来的序列结构变异可能会影响miRNA与靶标基因在编码区结合[27],但是小麦TaNAC基因相关的可变剪切和miRNA这两种转录后调控的综合研究鲜见报道。【拟解决的关键问题】本研究通过分析病菌胁迫后克隆得到的TaNAC结构变异转录本的特征,探究TaNAC基因基于可变剪切在小麦胁迫响应中的转录后调控规律;分析已经鉴定的所有TaNAC转录本的调控miRNA,完善调控小麦TaNAC基因的miRNA家族成员;通过分析TaNAC基因的可变剪切结构变异转录本的结构特征及其与miRNAs在编码序列区域的结合位点,探究在TaNAC基因转录后调控中可变剪切与miRNA的关系。1 材料与方法
1.1 试验材料
冬小麦优异种质N9134由西北农林科技大学通过染色体工程育种技术从阿勃5B缺体和野生二粒小麦AS846的杂交后代选育而来。N9134对中国目前鉴定的所有白粉菌和多种条锈菌表现高抗到免疫,其在1B和5BL染色体均含有1个抗性基因位点[28]。条锈菌生理小种CYR31和CYR32由西北农林科技大学植物保护学院提供,白粉菌生理小种E09由西北农林科技大学小麦远缘杂交与分子染色体工程育种实验室(本室)保存。高感白粉病的普通小麦品种陕优225和高感条锈病的普通小麦地方品种辉县红的种子由本室保存,分别作为2种病菌接种质量的对照材料。1.2 材料处理
选取N9134、陕优225和辉县红3个小麦材料的等量种子,置于培养皿中,加入没过种子的无菌蒸馏水,于23℃恒温黑暗培养箱中培养约48 h,挑选芽和根发育良好且一致的幼苗移入装有育苗基质的瓦盆,转入光照培养箱培养。在18℃光照16 h/12℃黑暗8 h条件下培养5—7 d,待幼苗长至5—10 cm,利用抖粉的方式接种。白粉菌生理小种E09和条锈菌CYR31、CYR32混合小种分别在陕优225和辉县红上提前诱发培养。3种小麦材料的培养条件、接种方式、接种量等均保持一致,通过陕优225和辉县红的发病情况判断N9134的接种质量。每个材料均设置3个生物学重复,在接种后0、12、24、36、48、72、96和120 h对分别接种白粉菌和条锈菌的N9134进行取样,每个时间点的样本由3个生物学重复的各1株的叶片混样组成,取样后,立即液氮冷冻,然后-80℃保存备用。1.3 基因克隆
2种病菌分别侵染后的8个时间点样本全部取样完毕后,利用Trizol(Invitrogen, Carlsbad, CA, USA)分别提取总计16个样本的总RNA,后续利用DNaseⅠ消化样本中的DNA并对提取的RNA进行纯化。纯化后的RNA用1%琼脂糖电泳进行质量检测,并用超微量分光光度计(Nanodrop ND-1000; Nanodrop Technologies, Wilmington, DE, USA)测定浓度。将16个样本(白粉菌和条锈菌2种胁迫各8个时间点的样本)的等量RNA混合后反转录合成cDNA,反转录试剂选用PrimescriptTM II 1st Strand cDNA Synthesis Kit(TaKaRa,大连,中国)。整合本室报道的白粉菌和条锈菌胁迫下N9134苗期的转录组测序数据[6,29]和中国春小麦参考基因组(IWGSC RefSeq v1.1)数据,并参考在NCBI中比对得到的山羊草等近缘物种的同源基因序列,利用Primer 5.0软件设计引物(表1),以上述cDNA为模板批量克隆TaNAC基因。使用PrimeSTAR HS DNA Polymerase(TaKaRa,大连,中国)进行PCR扩增,经电泳、目的片段回收、3′平末端加“A”尾、TA克隆并蓝白斑筛选、菌落PCR验证后,阳性克隆送至公司测序,最后分析测序结果。涉及的引物合成和测序均在北京奥科鼎盛生物科技有限公司进行。
Table 1
表1
表1TaNAC可变剪切结构变异转录本的克隆引物序列
Table 1
| 分组 Groups | TaNAC基因 TaNAC genes | 正向引物序列 Forward primer sequences (5′-3′) | 反向引物序列 Reversed primer sequences (5′-3′) |
|---|---|---|---|
| 1 | TaNAC_NTL5_7B | ATGACCCACCCTTCCTCGTC | TTACTTGCCGTAGATGCACAG |
| 2 | TaNAC008_3A | ATGGAAACTCTACGTGACATGGCT | CTACAAACTGATCCCACCACGGT |
| 3 | TaNAC013_3A | ATGACATGGTGCAATAGCTTC | TCAGGGACCAAAGCCCGTC |
| 4 | TaNAC017_5A | ATGGAGACCCCGCCGCCG | TCATGTAGAGGACTTCCATAAC |
| 5 | TaNAC011_7D | ATGGACGACGGTGCAACG | TCACCCATGATGATCCTGGT |
| 6 | TaNAC017_5D | ATGGAGACCCCGCCGCCG | CTTTGTGCATCATCTTGTCATG |
| 7 | TaNAC024_3D | ATGAACAGGGGTCACATCAGC | CTATCCATGATGATCTTGGTTGTC |
| 8 | TaNAC039_6B | ATGGACACTTTCTCCCATGTTC | CTATTTCCACAGATGAGCCGAG |
| 9 | TaNAC073_3B | ATGGACAAGGGTCACCCAGGGTAC | CTAGTACTGGTAAGGCCATTGCAC |
| 10 | TaNAC092_2A | TCCAGACGGCAAATTCCAG | TCAGTAGCCCCACGGCGC |
| 11 | TaNAC092_2D | ATGGAGCACAGCGAGCAGG | TCAGTAGCCCCACGGCGC |
| 12 | TaNAC024_3A | ATGAACAGGGGTCACATCAGC | CTATGAGATCTCCCGGTTCTTCTC |
| 13 | TaNAC092_2B | ATGGAGCACAGCGAGCAGG | AGCTTGCTTGGTCAGTAGCC |
新窗口打开|下载CSV
1.4 洋葱表皮细胞瞬时表达分析亚细胞定位
选取1对具有可变剪切结构变异的TaNAC转录本构建融合表达GFP载体,利用洋葱表皮细胞瞬时表达分析其亚细胞定位。构建重组pYJ-GFP载体时,由限制性内切酶SpeⅠ酶切完成载体线性化;TaNAC编码区序列由特异引物从连接有相应目的片段的T载体经过PCR扩增、电泳、回收后获得。然后利用ClonExpress II One Step Cloning Kit(诺唯赞,南京,中国)同源重组定向克隆,完成各pYJ-GFP重组载体的构建。PCR扩增使用高保真酶,特异引物由pYJ-GFP载体酶切位点同源臂的通用接头序列(F:5′-TAGCCATGGTAGATCTG-3′;R:5′-GCCTTACGTAACTAGT-3′)和各目标编码框的起始和终止内侧约20 bp的特异序列组合成,其中,反向特异引物去除终止密码子。重组pYJ-GFP载体经PCR扩增检测和测序验证后使用。
将重组pYJ-GFP载体和空白对照载体(pYJ-GFP)转化到农杆菌(GV3101),然后利用液体LB培养基培养至OD600约1.0,侵染经过预处理的洋葱表皮,然后把侵染后的洋葱表皮置于MS固体培养基中28℃暗培养16—24 h,利用激光共聚焦显微镜(Olympus,IX83-FV12003040101)观察并拍照。
1.5 转录调控活性分析
选取共计5组TaNAC可变剪切结构变异转录本(具有外显子跳跃或内含子保留的可变剪切方式)为代表,分组进行转录调控活性试验。共构建17个重组pGBKT7载体,构建重组pGBKT7载体时,由限制性内切酶EcoRⅠ和BamHⅠ双酶切完成载体线性化;pGBKT7同源臂的通用接头序列为F:5′-CAT GGAGGCCGAATTC-3′和R:5′-CTGCAGGTCGAC GGAT-3′,反向特异引物不需要去除终止密码子;其余过程和使用的试剂等同构建重组pYJ-GFP载体过程类似。以构建好的各TaNAC转录本编码序列的重组pGBKT7载体为试验组、以载体pGBKT7-53+ pGADT7-T为阳性对照组、以pGBKT7空载体为阴性对照组,同步分别转化至酵母菌株Y2H Gold中,涂布于SD/-Trp固体培养基平板,在30℃恒温条件下黑暗培养3—5 d,挑选阳性单克隆,于SD/-Trp液体培养基中扩繁,在200 r/min,30℃震荡培养至OD600达到0.6左右时,以100、10-1、10-2和10-3的相对浓度稀释菌液,各取2 μL分别点涂于SD/-Trp、SD/-Trp/X-α- Gal、SD/-Trp/AbA、SD/-Trp/-His/-Ade固体培养基,于30℃黑暗培养3—5 d,观察各培养基上菌株的生长情况,分组分析结果,每组包括同一TaNAC基因不同转录本的试验组、阳性对照和阴性对照,以此判断各重组载体携带转录本的转录调控活性。
1.6 生物信息学分析
利用多序列比对软件Cluster X和BioEdit比对分析核酸序列结构;利用SMART工具(结合从中国春小麦参考基因组(IWGSC RefSeq v1.1)鉴定得到的高可信度的460个基因位点的559条TaNAC转录本[6],利用psRNATarget(
2 结果
2.1 TaNAC结构变异转录本的克隆
RNA质量检测结果(图1)表明,提取过程中DNaseⅠ处理效果理想,所有总RNA样本均没有gDNA残留,保证了克隆得到的TaNAC转录本不存在基因组序列的污染。利用这些总RNA的混合样本进行基因克隆,图2展示了部分基因PCR扩增产物的1.5%琼脂糖凝胶电泳结果。利用小麦参考基因组(IWGSC RefSeq v1.1)比对得到的TaNAC转录本序列,并对相似度高的序列进行深入分析,根据序列差异与相似度进行分类,确定各自所属的部分同源染色体及对应的物理位置,据此排除部分同源染色体上同源基因可能带来的序列结构差异的干扰。从克隆得到的TaNAC转录本中[6],筛选出13个TaNAC基因(表2)的35条序列结构变异转录本进行转录后调控分析(转录本TaNAC017_5D.4提取自中国春参考基因组IWGSC RefSeq v1.1;其余转录本均已提交至GenBank数据库并获得了相应的登录号)。在进行基因克隆和序列分析时已经排除了其他可能的序列结构变异影响因素,因此,这些转录本中存在的序列结构变异应该都是由可变剪切形成的,具体包括外显子跳跃和内含子保留2种方式。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1条锈菌和白粉菌胁迫下N9134中提取的总RNA质量检测
M:Marker DL2000;S:条锈菌胁迫条件下的样本;P:白粉菌胁迫条件下的样本;1—8:真菌侵染后0—120 h的8个取样时间点(0、12、24、36、48、72、96和120 h)
Fig. 1The quality detection of total RNA extracted from N9134 being infected by stripe rust and powdery mildew
M: Marker DL2000; S: Samples under the infection of stripe rust; P: Samples under the infection of powdery mildew; 1-8: The infected leaves of N9134 were harvested at the eight time points of 0 h (hour), 12, 24, 36, 48, 72, 96 and 120 h after being inoculated
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2利用混合cDNA扩增TaNAC基因得到的部分产物
M:Marker DL2000;1—5分别为基因TaNAC_NTL5_7B、TaNAC008_3A、TaNAC013_3A、TaNAC092_2A和TaNAC092_2B的扩增产物;白色箭头为扩增的目的条带
Fig. 2Partial amplification product of TaNAC genes from the mixed cDNA in this study
M: Marker DL2000; 1-5 were the cDNA amplification products of TaNAC_NTL5_7B, TaNAC008_3A, TaNAC013_3A, TaNAC092_2A and TaNAC092_2B, respectively; White arrows were the target bands of the amplification products
Table 2
表2
表2克隆得到的13个基因可变剪切形成的35条TaNAC结构变异转录本
Table 2
| 分组 Groups | TaNAC转录本名称 Names of TaNAC transcripts | 序列长度 Sequence length (bp) | GenBank登录号 Accessions | 对应中国春参考基因组的基因 The corresponding genes in IWGSC RefSeq v1.1 | 是否外显子跳跃 Exon skipping or not | 是否内含子保留 Intron retention or not | 转录本编码方式 Coding mode of transcripts |
|---|---|---|---|---|---|---|---|
| 1 | TaNAC_NTL5_7B.2 | 1932 | MN747301 | TraesCS7B02G196900 | 是 Yes | 否 No | 正常 N |
| TaNAC_NTL5_7B.3 | 879 | MN747304 | TraesCS7B02G196900 | 正常 N | |||
| 2 | TaNAC008_3A.1 | 1446 | MN747252 | TraesCS3A02G176500 | 是 Yes | 否 No | 正常 N |
| TaNAC008_3A.2 | 597 | MN747253 | TraesCS3A02G176500 | 正常 N | |||
| 3 | TaNAC013_3A.1 | 1017 | MN747260 | TraesCS3A02G245900 | 是 Yes | 否 No | 正常 N |
| TaNAC013_3A.2 | 1017 | MN747262 | TraesCS3A02G245900 | 正常 N | |||
| TaNAC013_3A.3 | 974 | MN786410 | TraesCS3A02G245900 | 提前终止 P | |||
| 4 | TaNAC017_5A.2 | 2193 | MN747191 | TraesCS5A02G271500 | 是 Yes | 否 No | 正常 N |
| TaNAC017_5A.3 | 1701 | MN747193 | TraesCS5A02G271500 | 正常 N | |||
| TaNAC017_5A.4 | 1181 | MN786415 | TraesCS5A02G271500 | 分段 S | |||
| TaNAC017_5A.5 | 1237 | MN786416 | TraesCS5A02G271500 | 分段 S | |||
| TaNAC017_5A.6 | 1308 | MN747194 | TraesCS5A02G271500 | 正常 N | |||
| 5 | TaNAC011_7D.1 | 1467 | MN747256 | TraesCS7D02G365200 | 否 No | 是 Yes | 正常 N |
| TaNAC011_7D.2 | 865 | MN786437 | TraesCS7D02G365200 | 分段 S | |||
| 6 | TaNAC017_5D.2 | 2160 | MN747189 | TraesCS5D02G279100 | 否 No | 是 Yes | 正常 N |
| TaNAC017_5D.4 | 2052 | IWGSC | TraesCS5D02G279100 | 正常 N | |||
| 7 | TaNAC024_3D.1 | 1320 | MN747206 | TraesCS3D02G109400 | 否 No | 是 Yes | 正常 N |
| TaNAC024_3D.2 | 1317 | MN747210 | TraesCS3D02G109400 | 正常 N | |||
| TaNAC024_3D.3 | 1457 | MN786432 | TraesCS3D02G109400 | 分段 S | |||
| 8 | TaNAC039_6B.3 | 1056 | MN747281 | TraesCS6B02G286200 | 否 No | 是 Yes | 正常 N |
| TaNAC039_6B.5 | 1171 | MN786440 | TraesCS6B02G286200 | 分段 S | |||
| 9 | TaNAC073_3B.3 | 966 | MN747291 | TraesCS3B02G410500 | 否 No | 是 Yes | 正常 N |
| TaNAC073_3B.4 | 1134 | MN786442 | TraesCS3B02G410500 | 分段 S | |||
| TaNAC073_3B.6 | 979 | MN786412 | TraesCS3B02G410500 | 提前终止 P | |||
| 10 | TaNAC092_2A.3 | 1068 | MN747237 | TraesCS2A02G338300 | 否 No | 是 Yes | 正常 N |
| TaNAC092_2A.5 | 1325 | MN786430 | TraesCS2A02G338300 | 分段 S | |||
| 11 | TaNAC092_2D.6 | 1062 | MN747248 | TraesCS2D02G324700 | 否 No | 是 Yes | 正常 N |
| TaNAC092_2D.7 | 1403 | MN786434 | TraesCS2D02G324700 | 分段 S | |||
| TaNAC092_2D.8 | 1405 | MN786435 | TraesCS2D02G324700 | 分段 S | |||
| 12 | TaNAC024_3A.1 | 1320 | MN747211 | TraesCS3A02G107400 | 是 Yes | 是 Yes | 正常 N |
| TaNAC024_3A.3 | 1317 | MN747208 | TraesCS3A02G107400 | 正常 N | |||
| TaNAC024_3A.6 | 1612 | MN786431 | TraesCS3A02G107400 | 分段 S | |||
| TaNAC024_3A.7 | 1401 | MN786419 | TraesCS3A02G107400 | 分段 S | |||
| 13 | TaNAC092_2B.1 | 1065 | MN747239 | TraesCS2B02G343600 | 是 Yes | 是 Yes | 正常 N |
| TaNAC092_2B.3 | 708 | MN747234 | TraesCS2B02G343600 | 正常 N | |||
| TaNAC092_2B.7 | 1167 | MN786429 | TraesCS2B02G343600 | 分段 S |
新窗口打开|下载CSV
分析这些TaNAC结构变异转录本的序列特征,发现有些由外显子跳跃形成的新转录本仍可按照可变剪切前原转录本的编码顺序正常编码,如TaNAC_ NTL5_7B.3;有些新转录本的编码框会发生移码导致相对原转录本分段编码,如TaNAC017_5A.4;有些新转录本的编码框会发生移码导致开放阅读框(Open reading frame,ORF)相对原转录本提前终止,如TaNAC013_3A.3。同样,内含子保留形成的新转录本也会出现上述3种情况。同时存在外显子跳跃和内含子保留的转录本,变异情况相对复杂,但概括起来仍和上述情况一样,包括新转录本可以正常编码和不能正常编码。
2.2 TaNAC可变剪切转录本的序列结构分析
选取获得的4个TaNAC基因(外显子跳跃和内含子保留这两种可变剪切方式各2个)进行序列结构分析(图3)。外显子跳跃形成的新转录本TaNAC_NTL5_7B.3相对TaNAC_NTL5_7B.2,在其起始密码子88 bp位置后,缺失1 053 bp序列,涉及第1个外显子后端389 bp序列,第2个外显子全部210 bp序列和第3个外显子前端454 bp的序列,但是TaNAC_NTL5_7B.3的读码没有变化,只是相较TaNAC_NTL5_7B.2,其编码产物几乎缺失了NAM保守结构域的全长(图3-a)。TaNAC013_3A.3因为外显子跳跃缺失了第3个外显子中段偏前的43 bp序列(位于原转录本ORF起始密码子之后的770—812 bp的序列),其读码方式在其缺失处之后出现移码,导致在ORF的第801位即提前出现了终止密码子TGA;相应未出现外显子跳跃的2条转录本TaNAC013_3A.1和TaNAC013_3A.2,在起始密码子后的第778位有1个碱基的差异,导致其编码产物在第260位由丝氨酸(S)突变为甘氨酸(G)(S260G),而此碱基变异位点恰好位于TaNAC013_3A.3被跳跃的43 bp序列区间(图3-b)。转录本TaNAC011_7D.1相对TaNAC011_7D.2在其第2个外显子后插入了一段602 bp的内含子(具有GT-AG结构特征),保留了内含子的TaNAC011_7D.1可以正常编码,而无这一内含子的TaNAC011_7D.2则出现了分段编码的情况,其在ATG之后的第387位即出现了终止密码TAG,同时,在其第2个外显子的第159位又出现了一个可以按照TaNAC011_7D.1读码方式的起始密码子(图3-c)。转录本TaNAC024_3D.1和TaNAC024_3D.2均可顺利读码,它们仅在第3个外显子末端存在3 bp的有无差异;而TaNAC024_3D.3相对TaNAC024_3D.2在第2个外显子后保留了一段140 bp的内含子(具有GT-AG结构特征),两者的其余序列完全一样;转录本TaNAC024_3D.3在ATG之后第198位即出现了终止密码子TAG,但其在插入的内含子序列区域另有一个起始密码子,形成总长1 233 bp的编码序列,包括插入内含子序列后端的30 bp以及与TaNAC024_3D.2的ORF后端完全一样的1 203 bp序列(图3-d)。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图32种可变剪切方式(外显子跳跃或内含子保留)形成的TaNAC转录本序列结构模式
蓝色:外显子;灰色:内含子
Fig. 3The structure pattern of TaNAC transcripts with alternative splicing (Exon skipping or intron retention)
Blue: Exon; Gray: Intron
2.3 TaNAC结构变异转录本编码产物的结构特征和理化性质分析
选取的35条TaNAC结构变异转录本中有24条能够正常编码(TaNAC017_5D.4直接从中国春参考基因组IWGSC RefSeq v1.1提取),本研究对它们编码产物的保守结构域、跨膜结构域等结构特征和氨基酸的总平均疏水性(grand average of hydropathicity,GRAVY)、理论等电点等理化性质进行分析或预测,发现在结构特征方面,不同剪切方式形成的结构变异转录本,其编码产物的保守结构域和跨膜结构域均可能存在有无的差异(表3),如:TaNAC_NTL5_7B.2编码具有NAM保守结构域的肽链,而因外显子跳跃形成的新转录本TaNAC_NTL5_7B.3的编码产物缺失了NAM保守域;转录本TaNAC008_3A.1编码的多肽在C末端具有跨膜结构域,而外显子跳跃形成的新转录本TaNAC008_3A.2编码的多肽却丢失了这一结构。Table 3
表3
表3克隆得到的13组TaNAC结构变异转录本中正常编码产物的结构特征和理化性质
Table 3
| 分组 Groups | 克隆的转录本名称 Name of cloned transcripts | 肽链长度 Peptide length (aa) | NAM结构域 起止位置 NAM start_end (aa) | 跨膜结构域位置 Topology of TMHs | 脂肪系数 Aliphatic index | 亲水性总 平均值 GRAVY | 不稳定系数 Instability index | 理论等电点 pI | 预测的亚细胞定位 Predicted subcellular localization |
|---|---|---|---|---|---|---|---|---|---|
| 1 | TaNAC_NTL5_7B.2 | 643 | 23—149 | o620—642i | 71.12 | -0.424 | 36.32 | 4.63 | 细胞核 Nu |
| TaNAC_NTL5_7B.3 | 292 | 无 None | o269—291i | 70.45 | -0.114 | 40.41 | 4.61 | 胞外/细胞核 Ex / Nu | |
| 2 | TaNAC008_3A.1 | 481 | 10—136 | o448—470i | 72.45 | -0.554 | 35.92 | 5.67 | 细胞核/细胞质 Nu / Cy |
| TaNAC008_3A.2 | 198 | 10—136 | 无 None | 65.56 | -0.809 | 37.89 | 9.08 | 细胞质 Cy | |
| 3 | TaNAC013_3A.1 | 338 | 73—212 | 无 None | 63.08 | -0.656 | 35.37 | 8.83 | 细胞核/线粒体 Nu / Mi |
| TaNAC013_3A.2 | 338 | 73—212 | 无 None | 63.08 | -0.655 | 35.48 | 8.83 | 细胞核/线粒体/细胞质 Nu / Mi / Cy | |
| TaNAC013_3A.3 | 266 | 73—212 | 无 None | 67.18 | -0.676 | 36.58 | 9.15 | 细胞核/线粒体 Nu / Mi | |
| 4 | TaNAC017_5A.2 | 730 | 13—136 | o695—717i | 69.74 | -0.511 | 49.35 | 4.55 | 细胞核 Nu |
| TaNAC017_5A.3 | 566 | 无 None | o531—553i | 72.86 | -0.403 | 51.11 | 4.18 | 细胞核/胞外 Nu / Ex | |
| TaNAC017_5A.6 | 435 | 13—136 | o400—422i | 76.00 | -0.476 | 50.17 | 5.92 | 细胞核 Nu | |
| TaNAC017_5A.4 | 分段 S | — — — — — — — — — — — — | |||||||
| TaNAC017_5A.5 | 分段 S | — — — — — — — — — — — — | |||||||
| 5 | TaNAC011_7D.1 | 488 | 57—226 | 无 None | 53.79 | -0.843 | 55.96 | 7.53 | 细胞核 Nu |
| TaNAC011_7D.2 | 分段 S | — — — — — — — — — — — — | |||||||
| 6 | TaNAC017_5D.2 | 719 | 13—136 | o683—705i | 69.61 | -0.490 | 49.99 | 4.57 | 细胞核 Nu |
| TaNAC017_5D.4* | 683 | 13—136 | o647—669i | 70.26 | -0.528 | 50.36 | 4.62 | 细胞核 Nu | |
| 7 | TaNAC024_3D.1 | 439 | 45—186 | 无 None | 59.13 | -0.813 | 48.33 | 6.88 | 细胞核 Nu |
| TaNAC024_3D.2 | 438 | 44—185 | 无 None | 59.27 | -0.806 | 48.24 | 6.76 | 细胞核 Nu | |
| TaNAC024_3D.3 | 分段 S | — — — — — — — — — — — — | |||||||
| 8 | TaNAC039_6B.3 | 351 | 8—135 | 无 None | 58.97 | -0.755 | 38.99 | 6.57 | 细胞核 Nu |
| TaNAC039_6B.5 | 分段 S | — — — — — — — — — — — — | |||||||
| 9 | TaNAC073_3B.3 | 321 | 46—188 | 无 None | 54.7 | -0.902 | 37.18 | 8.6 | 细胞核 Nu |
| TaNAC073_3B.6 | 278 | 46—188 | 无 None | 59.64 | -0.828 | 33.43 | 8.83 | 细胞核 Nu | |
| TaNAC073_3B.4 | 分段 S | — — — — — — — — — — — — | |||||||
| 10 | TaNAC092_2A.3 | 355 | 14—138 | 无 None | 64.14 | -0.412 | 30.91 | 7.21 | 细胞核/叶绿体 Nu / Ch |
| TaNAC092_2A.5 | 分段 S | — — — — — — — — — — — — | |||||||
| 11 | TaNAC092_2D.6 | 353 | 14—138 | 无 None | 65.33 | -0.397 | 33.56 | 6.86 | 细胞核/叶绿体 Nu / Ch |
| TaNAC092_2D.7 | 分段 S | — — — — — — — — — — — — | |||||||
| TaNAC092_2D.8 | 分段 S | — — — — — — — — — — — — | |||||||
| 12 | TaNAC024_3A.1 | 439 | 45—186 | 无 None | 58.45 | -0.828 | 46.99 | 6.76 | 细胞核 Nu |
| TaNAC024_3A.3 | 438 | 44—185 | 无 None | 58.58 | -0.821 | 46.9 | 6.67 | 细胞核 Nu | |
| TaNAC024_3A.6 | 分段 S | — — — — — — — — — — — — | |||||||
| TaNAC024_3A.7 | 分段 S | — — — — — — — — — — — — | |||||||
| 13 | TaNAC092_2B.1 | 354 | 14—138 | 无 None | 65.14 | -0.386 | 31.18 | 6.67 | 叶绿体/细胞核 Ch / Nu |
| TaNAC092_2B.3 | 235 | 14—136 | 无 None | 57.87 | -0.506 | 31.91 | 5.27 | 胞外/细胞核 Ex / Nu | |
| TaNAC092_2B.7 | 分段 S | — — — — — — — — — — — — | |||||||
新窗口打开|下载CSV
在理化性质方面(表3),这些TaNAC转录本的编码产物均表现亲水性(总平均疏水性负值是亲水蛋白,正值为疏水蛋白),可变剪切事件没有改变这一特性,且对应的脂肪系数变化也很小;而在蛋白质的稳定性(稳定蛋白系数<40,不稳定蛋白系数>40)、理论等电点方面,TaNAC转录本却可能因可变剪切事件使其编码产物表现出性质上的改变,如TaNAC_NTL5_7B.2编码稳定蛋白而其外显子跳跃序列异构转录本TaNAC_NTL5_7B.3编码不稳定蛋白;TaNAC008_3A.1编码产物等电点为5.67 (<7)为酸性蛋白,而其外显子跳跃序列异构转录本TaNAC008_3A.2编码的多肽链等电点为9.08(>7)为碱性蛋白。另外,TaNAC可变剪切转录本的编码产物在功能域和理化性质方面的变化,可能会导致其亚细胞定位发生变化。
2.4 TaNAC可变剪切结构变异转录本编码产物的高级结构分析
选取的13组TaNAC可变剪切转录本中有7组均包含至少2条可正常编码的转录本(提前终止视为正常编码),对这7组转录本编码产物的二级和三级结构进行预测,并分组进行比较分析(表4)。结果表明,TaNAC008_3A和TaNAC092_2B可变剪切形成的结构变异转录本的编码产物,二级结构变化较大(表4,图4-a1—a4)。转录本TaNAC008_3A.2相对于TaNAC008_3A.1,其编码产物形成α螺旋(alpha helix)的氨基酸比例降低约50%(从28.48%降至14.65%),而形成延伸链(extended strand)结构的氨基酸比例则从12.47%增至21.72%;而转录本TaNAC092_2B.1和TaNAC092_2B.3之间编码肽链的二级结构的变化主要体现在α螺旋(氨基酸的比例从18.93%增加至37.02%)和无规则卷曲(random coil)(氨基酸的比例从63.84%降至42.98%)。Table 4
表4
表4部分TaNAC可变剪切结构变异转录本正常编码产物二级结构的比较
Table 4
| 分组Groups | 转录本 Transcripts | 肽链长度 Peptide length (aa) | α螺旋中氨基酸的数量 Number of aa in alpha helix | α螺旋中氨基酸的比例 Percentage of aa in alpha helix (%) | β折叠中氨基酸的数量 Number of aa in beta turn | β折叠中氨基酸的比例 Percentage of aa in beta turn (%) | 延伸链中氨基酸的数量 Number of aa in extended strand | 延伸链中氨基酸的比例 Percentage of aa in extended strand (%) | 无规则卷曲中氨基酸的数量 Number of aa in random coil | 无规则卷曲中 氨基酸的比例 Percentage of aa in random coil (%) |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | TaNAC_NTL5_7B.2 | 643 | 140 | 21.77 | 25 | 3.89 | 95 | 14.77 | 383 | 59.56 |
| TaNAC_NTL5_7B.3 | 292 | 51 | 17.47 | 13 | 4.45 | 45 | 15.41 | 183 | 62.67 | |
| 2 | TaNAC008_3A.1 | 481 | 137 | 28.48 | 25 | 5.20 | 60 | 12.47 | 259 | 53.85 |
| TaNAC008_3A.2 | 198 | 29 | 14.65 | 12 | 6.06 | 43 | 21.72 | 114 | 57.58 | |
| 3 | TaNAC013_3A.1 | 338 | 82 | 24.26 | 29 | 8.58 | 66 | 19.53 | 161 | 47.63 |
| TaNAC013_3A.2 | 338 | 89 | 26.33 | 19 | 5.62 | 64 | 18.93 | 166 | 49.11 | |
| TaNAC013_3A.3 | 266 | 61 | 22.93 | 17 | 6.39 | 57 | 21.43 | 131 | 49.25 | |
| 4 | TaNAC017_5A.2 | 730 | 213 | 29.18 | 60 | 8.22 | 106 | 14.52 | 351 | 48.08 |
| TaNAC017_5A.3 | 566 | 163 | 28.80 | 35 | 6.18 | 85 | 15.02 | 283 | 50.00 | |
| TaNAC017_5A.6 | 435 | 121 | 27.82 | 37 | 8.51 | 75 | 17.24 | 202 | 46.44 | |
| 6 | TaNAC017_5D.2 | 719 | 217 | 30.18 | 54 | 7.51 | 99 | 13.77 | 349 | 48.54 |
| TaNAC017_5D.4 | 683 | 198 | 28.99 | 50 | 7.32 | 97 | 14.20 | 338 | 49.49 | |
| 9 | TaNAC073_3B.3 | 321 | 76 | 23.68 | 24 | 7.48 | 56 | 17.45 | 165 | 51.40 |
| TaNAC073_3B.6 | 278 | 63 | 22.66 | 32 | 11.51 | 54 | 19.42 | 129 | 46.40 | |
| 13 | TaNAC092_2B.1 | 354 | 67 | 18.93 | 15 | 4.24 | 46 | 12.99 | 226 | 63.84 |
| TaNAC092_2B.3 | 235 | 87 | 37.02 | 21 | 8.94 | 26 | 11.06 | 101 | 42.98 |
新窗口打开|下载CSV
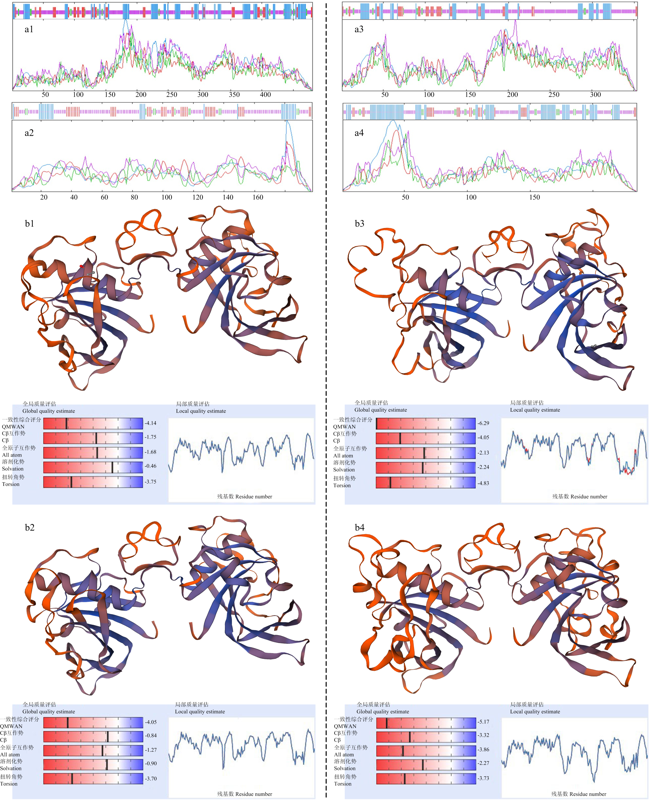
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4TaNAC008_3A和TaNAC092_2B的可变剪切编码产物的高级结构分析
a和b分别为多肽二级结构和三级结构的预测结果;1—4分别为转录本TaNAC008_3A.1、TaNAC008_3A.2、TaNAC092_2B.1和TaNAC092_2B.3的编码产物;—:延伸链,—:β折叠,—:α螺旋,—:无规则卷曲
Fig. 4Advanced structure analysis of alternatively spliced products of TaNAC008_3A and TaNAC092_2B
a and b: The predicted results of secondary and tertiary structure of peptides; 1-4: The peptides encoded by the transcripts of TaNAC008_3A.1, TaNAC008_3A.2, TaNAC092_2B.1 and TaNAC008_3A.3, respectively; —: Extended strand; —: Beta turn; —: Alpha helix; —: Random coil
基于二级结构的差异,分别对转录本TaNAC008_3A.1、TaNAC008_3A.2、TaNAC092_2B.1和TaNAC092_ 2B.3编码产物的三级结构进行了预测(图4-b1—图4-b4),发现这4条多肽序列在构建三级结构时均以水稻的压力响应转录因子NAC1为模型,相似序列区间均在其肽段N末端具有DNA结合功能的NAM保守结构域,且构建的三级结构相似,均为中间凹陷的结构,符合其功能特征。但是,预测得到的三级结构在相对于水稻NAC1的整体和局部的质量评估等参数及结构本身等方面均存在差异(图4-b1—b4),其中TaNAC008_3A.2(图4-b2)相对于TaNAC008_3A.1(图4-b1),其编码产物除了图示的三级结构差异外,还缺失了一段位于C末端的α螺旋结构;而TaNAC092_2B.1(图4-b3)和TaNAC092_2B.3(图4-b4)编码产物之间在预测的图示三级结构本身即有较明显差异。
2.5 TaNAC结构变异转录本的亚细胞定位的比对分析
根据表3中的预测结果,选择亚细胞定位信息有差异的结构变异转录本TaNAC013_3A.2和TaNAC013_ 3A.3,利用洋葱表皮细胞瞬时表达系统进行了亚细胞定位分析(图5)。结果显示,对照组(pYJ-GFP)的洋葱表皮细胞中,荧光信号在细胞核、细胞质、细胞膜中都有分布。试验组中,转化TaNAC013_3A.2- pYJ-GFP重组载体的洋葱表皮细胞中,融合绿色荧光蛋白在多个位置表达,细胞核、细胞质中均可观察到明显的绿色荧光信号;而转化TaNAC013_3A.3-pYJ- GFP载体的洋葱表皮细胞中,只在细胞核的位置能够观察到绿色荧光信号。在分别瞬时表达2个转录本的洋葱表皮细胞中,存在细胞核以外的绿色荧光信号差异,说明转录本TaNAC013_3A.2可以细胞核外(细胞质)表达,而TaNAC013_3A.3仅能在细胞核表达。结果和表3中的预测结果基本吻合。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5TaNAC013_3A.2和TaNAC013_3A.3利用洋葱表皮细胞瞬时表达系统的亚细胞定位
Fig. 5The subcellular localization of TaNAC013_3A.2 and TaNAC013_3A.3 using the transient expression system of onion epidermal cells
2.6 TaNAC结构变异转录本的转录调控活性的比较分析
通过对选取的5组TaNAC结构变异转录本进行酵母自激活检测,发现它们对于α半乳糖苷酶编码基因MEL-1的转录激活活性均有不同(图6-a和表5)。其中,TaNAC013_3A.1-pGBKT7重组载体的转化菌株有转录激活活性(B3),而代表TaNAC013_ 3A.2的菌株无转录激活活性(B4),这两条转录本仅在起始密码子之后第778位有1个碱基的差异,导致编码产物在第260位存在丝氨酸(S)和甘氨酸(G)的变异;同时,这碱基变异位点位于转录本TaNAC013_3A.3外显子跳跃事件涉及的43 bp序列区间,而代表TaNAC013_3A.3的菌株均具有转录激活活性(B1和B2);这些结果表明,这一具有单碱基变异的外显子跳跃区间(770—812 bp)序列在其转录调控中具有关键作用。在营养缺陷型培养基(SD/-Trp/-His/-Ade)和金担子素A(aureobasidin A,AbA)筛选培养基(SD/-Trp/AbA)上,仅有TaNAC008_3A的2个转录本重组pGBKT7载体转化的阳性Y2H Gold酵母菌株(A1和A2)表现出生长状态的差异,即转录本TaNAC008_3A.1在上述2种条件下均具有转录激活活性而TaNAC008_3A.2均不表现转录激活活性(图6-b和表5);而其他4组TaNAC基因共计15条结构变异转录本的重组pGBKT7载体转化的阳性Y2H Gold酵母菌株在这两种培养基上均能够正常生长,且生长情况无差异,即这些转录本在这两种条件下均具有转录激活活性(表5)。图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6部分TaNAC基因的结构变异转录本转录调控活性的比较分析
a:连接不同目的片段的重组pGBKT7载体转化至酵母菌株Y2H Gold的阳性菌株在SD/-Trp/X-α-Gal固体培养基的生长情况;b:TaNAC008_3A的2个结构变异转录本及阴阳性对照重组载体对应的酵母菌株在SD/-Trp、SD/-Trp/-His/-Ade和SD/-Trp/AbA固体培养基的生长情况;a和b中的A—E为样本的行代号,1—4为样本的列代号,行列代号组合后的“A1—E1”代表转化有连接着不同目的片段的重组pGBKT7载体的Y2H Gold阳性酵母菌株,具体见
Fig. 6Comparative analysis of transcriptional activation of some structural variant transcripts of TaNAC genes
a: Growth of yeast Y2H Gold positive strain transformed by recombinant pGBKT7 vector with different target fragments in SD/Trp/X-α-gal solid medium. b: Growth of yeast strains corresponding to the recombinant vectors of the two structural variant transcripts of TaNAC008_3A and the negative and positive control vectors in SD/-Trp, SD/-Trp/-his/-Ade and SD/-Trp/ABA solid media. In a and b, A-E are the row numbers of the samples, and 1-4 are the column numbers. After combination of the row and column numbers, A1-E1 represent Y2H Gold positive yeast strains transformed with recombinant pGBKT7 vectors with different target fragments, as detailed in
Table 5
表5
表55组TaNAC基因的17条结构变异转录本的转录调控活性分析结果
Table 5
Number in | 转录本名称 Transcripts | 插入片段的长度 Length of insert fragment (bp) | 是否具有激活功能 Activation or not | |||
|---|---|---|---|---|---|---|
| SD/-Trp | SD/-Trp/X-α-gal | SD/-Trp/AbA | SD/-Trp/-His/-Ade | |||
| A1 | TaNAC008_3A.1 | 1446 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| A2 | TaNAC008_3A.2 | 597 | 是 Yes | 否 No | 否 No | 否 No |
| A3 | TaNAC_NTL5_7B.2 | 1932 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| A4 | TaNAC_NTL5_7B.3 | 879 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| B1 | TaNAC013_3A.3 | 801/974 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| B2 | TaNAC013_3A.3 | 974 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| B3 | TaNAC013_3A.1 | 1017 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| B4 | TaNAC013_3A.2 | 1017 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| C1 | TaNAC092_2A.3 | 1068 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| C2 | TaNAC092_2A.5 | 1175 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| C3 | TaNAC092_2A.5 | 633(1—633)/1175 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| C4 | TaNAC092_2A.5 | 483(693—1175)/1175 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| D1 | TaNAC092_2B.1 | 1065 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| D2 | TaNAC092_2B.3 | 708 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
| D3 | TaNAC092_2B.7 | 1167 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| D4 | TaNAC092_2B.7 | 555(1—555)/1167 | 是 Yes | 否 No | 是 Yes | 是 Yes |
| E1 | TaNAC092_2B.7 | 483(685—1167)/1167 | 是 Yes | 是 Yes | 是 Yes | 是 Yes |
新窗口打开|下载CSV
2.7 结合于小麦TaNAC基因编码区的miRNA的预测分析
根据miRNA数据库miRBase(Table 6
表6
表6小麦参考基因组(IWGSC RefSeq v1.1)注释的TaNAC转录本与小麦tae-miRNA的靶向关系预测结果
Table 6
| miRNA名称 miRNA ID | 期望值 Expectation | 抑制方式 Inhibition | TaNAC转录本名称 TaNAC transcripts ID | 靶基因 长度 Length (bp) | 是否可 变剪切 AS or not | NAM结构域编码起止位置 NAM coding start_end (bp) | miRNA结合起止位置 Target miRNA start_end (bp) | 靶基因与miRNA匹配的序列 Target aligned fragment (5′-3′) | 匹配方式 Alignment |
|---|---|---|---|---|---|---|---|---|---|
| tae-miR164 | 1 | 裂解 Cleavage | TraesCS5A02G049100.1 | 918 | 是 Yes | 40—423 | 653—673 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: |
| TraesCS5A02G049100.2 | 921 | 是 Yes | 40—423 | 656—676 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS5B02G054200.1 | 921 | 是 Yes | 40—423 | 656—676 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS5B02G054200.2 | 924 | 是 Yes | 40—423 | 659—679 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS5D02G059700.1 | 909 | 是 Yes | 40—423 | 644—664 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS5D02G059700.2 | 912 | 是 Yes | 40—423 | 647—667 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7A02G334800.1 | 1092 | 否 No | 64—444 | 770—790 | AGCUCGUGCCCUGCUUCUCCA | :: ::::::::::::::::: | |||
| TraesCS7A02G464100.1 | 924 | 否 No | 34—435 | 653—673 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7A02G464800.1 | 927 | 否 No | 34—438 | 656—676 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7B02G246300.1 | 1089 | 否 No | 55—444 | 767—787 | AGCUCGUGCCCUGCUUCUCCA | :: ::::::::::::::::: | |||
| TraesCS7B02G364600.1 | 924 | 否 No | 34—435 | 653—673 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7B02G365300.1 | 918 | 否 No | 34—429 | 647—667 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7D02G342300.1 | 1089 | 否 No | 64—444 | 767—787 | AGCUCGUGCCCUGCUUCUCCA | :: ::::::::::::::::: | |||
| TraesCS7D02G451700.1 | 939 | 否 No | 34—438 | 656—676 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| TraesCS7D02G452500.1 | 927 | 否 No | 34—435 | 656—676 | AGCAAGUGCCCUGCUUCUCCA | ::: :::::::::::::::: | |||
| 1.5 | 裂解 Cleavage | TraesCS2A02G338300.1 | 1068 | 否 No | 40—414 | 680—700 | CGCACGUGACCUGCUUCUCCA | ::::::: :::::::::::: | |
| TraesCS2B02G343600.1 | 1065 | 否 No | 40—414 | 680—700 | CGCACGUGACCUGCUUCUCCA | ::::::: :::::::::::: | |||
| TraesCS2D02G324700.1 | 1062 | 否 No | 40—414 | 677—697 | CGCACGUGACCUGCUUCUCCA | ::::::: :::::::::::: | |||
| 2 | 裂解 Cleavage | TraesCS7A02G305200.1 | 870 | 否 No | 34—414 | 665—685 | AGCAAGUGUCCUGCUUCUCCG | ::: :::.:::::::::::. | |
| TraesCS7B02G205600.1 | 879 | 否 No | 34—414 | 674—694 | AGCAAGUGUCCUGCUUCUCCG | ::: :::.:::::::::::. | |||
| TraesCS7D02G302000.1 | 876 | 否 No | 34—420 | 671—691 | AGCAAGUGUCCUGCUUCUCCG | ::: :::.:::::::::::. | |||
| miRNA名称 miRNA ID | 期望值 Expectation | 抑制方式 Inhibition | TaNAC转录本名称 TaNAC transcripts ID | 靶基因 长度 Length (bp) | 是否可 变剪切 AS or not | NAM结构域编码起止位置 NAM coding start_end (bp) | miRNA结合起止位置 Target miRNA start_end (bp) | 靶基因与miRNA匹配的序列 Target aligned fragment (5′-3′) | 匹配方式 Alignment |
| tae-miR1128 | 3.5 | 裂解 Cleavage | TraesCS3A02G339600.1 | 945 | 是 Yes | 97—480 | 246—266 | GUUCGGGAGCAGGGAGUGGUA | :::::. : :::::::.::: |
| TraesCS3A02G339600.2 | 882 | 是 Yes | 97—651 | 371—391 | GUUCGGGAGCAGGGAGUGGUA | :::::. : :::::::.::: | |||
| TraesCS3B02G371200.1 | 939 | 否 No | 85—468 | 234—254 | GUUCGGGAGCAGGGAGUGGUA | :::::. : :::::::.::: | |||
| TraesCS3D02G333100.1 | 951 | 否 No | 97—480 | 246—266 | GUUCGGGAGCAGGGAGUGGUA | :::::. : :::::::.::: | |||
| TraesCS4B02G328800.1 | 1044 | 否 No | 34—414 | 180—200 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS4B02G328900.1 | 1059 | 否 No | 49—429 | 195—215 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS4B02G329100.1 | 1059 | 否 No | 52—432 | 198—218 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS4D02G325800.1 | 1065 | 否 No | 52—432 | 198—218 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS5A02G500400.1 | 1017 | 否 No | 22—402 | 195—215 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS5A02G500500.1 | 1110 | 否 No | 52—432 | 168—188 | AAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS5A02G500600.1 | 1071 | 否 No | 52—432 | 198—218 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| TraesCS5A02G500700.1 | 2085 | 否 No | 19—399 | 198—218 | GAUCGGGGAGAGGGAGUGGUA | ::::. ::::::::.::: | |||
| tae-miR408 | 3 | 翻译 Translation | TraesCS6A02G378500.1 | 936 | 否 No | 31—441 | 812—832 | CCCAGGGCGGCGGCAGUGCAG | ::::::.: :::::::::: |
| 3.5 | 裂解 Cleavage | TraesCS4B02G320300.1 | 1104 | 否 No | 25—408 | 622—642 | UUCAAGGAGGAGGCGGAGCAG | .:: :::.:::::.: :::: | |
| TraesCS4D02G316800.1 | 1104 | 否 No | 25—408 | 622—642 | UUCAAGGAGGAGGCGGAGCAG | .:: :::.:::::.: :::: | |||
| TraesCS5A02G491700.1 | 1047 | 否 No | 49—429 | 622—642 | UUCAAGGAGGAGGCGGAGCAG | .:: :::.:::::.: :::: | |||
| tae-miR9780 | 3 | 翻译 Translation | TraesCSU02G120000.1 | 1239 | 否 No | 22—450 | 136—156 | GACGCGUACGCCGCCGACCCG | : ::::: :. :::::::::: |
| 3.5 | 裂解 Cleavage | TraesCS6A02G065600.1 | 1227 | 否 No | 22—444 | 136—156 | GACGCGUACGGCGCAGAUCCG | : ::::: :.:::: ::.::: | |
| 翻译 Translation | TraesCSU02G119900.1 | 1284 | 否 No | 22—450 | 136—156 | GACGCGUACGCCGCCGAUCCG | : ::::: :. ::::::.::: | ||
| tae-miR9676-5p | 2.5 | 裂解 Cleavage | TraesCS5B02G480900.1 | 900 | 是 Yes | 58—351 | 755—776 | ACCUCAGCUACGAUGACAUCCA | : ::.::::::::::::: |
| TraesCS5B02G480900.2 | 984 | 是 Yes | 58—435 | 839—860 | ACCUCAGCUACGAUGACAUCCA | : ::.::::::::::::: | |||
| tae-miR171a | 3.5 | 裂解 Cleavage | TraesCS4D02G175700.1 | 1074 | 否 No | 28—408 | 747—767 | GCUGAUGGCACGGCACGAUCA | : :. ::::::::: :.:::: |
| tae-miR444a | 3.5 | 翻译 Translation | TraesCS7A02G349500.1 | 1107 | 否 No | 31—420 | 1011—1031 | GGAGCAACUUGGAGGCAGCAG | : :::::.: :::::::::. |
| tae-miR444b | 3.5 | 翻译 Translation | TraesCS7A02G349500.1 | 1107 | 否 No | 31—420 | 1011—1031 | GGAGCAACUUGGAGGCAGCAG | : :::::.: :::::::::. |
| tae-miR9677a | 3.5 | 裂解 Cleavage | TraesCS1A02G276000.1 | 774 | 否 No | 13—405 | 296—317 | UGGUCUACUCCGCCGGCGGCCA | .::::::.::..:::::: |
新窗口打开|下载CSV
分析tae-miRNA靶标TaNAC基因编码序列上的结合位点发现,tae-miR1128、tae-miR9677a和tae-miR9780与其各自对应的TaNAC转录本(总计16条)在NAM保守结构域编码区结合,调控它们的表达;其余miRNA和TaNAC转录本的结合位点靠近ORF的3′端。本研究涉及的所有能够发生可变剪切的TaNAC基因,其转录本与miRNA的结合位点均位于非可变剪切区域。这些结果表明,调控TaNAC基因表达的miRNA和可变剪切是各自独立行使转录后调控功能的。
3 讨论
3.1 TaNAC基因可以通过可变剪切的转录后调控方式参与胁迫响应
本研究通过对克隆得到的TaNAC基因的可变剪切转录本序列分析发现,可变剪切可以导致TaNAC转录本编码序列结构变异,进而改变其编码产物的序列结构,即改变其对应蛋白的保守结构域和跨膜结构域等结构特征。对应地,编码序列结构变异会影响其编码产物的高级结构、理化性质和亚细胞定位等。进一步分析发现,由可变剪切导致的TaNAC转录本编码序列结构变异也会带来相应编码产物转录调控活性的功能变化。综上所述,可变剪切事件导致TaNAC转录本编码序列变异,其编码产物的结构和理化性质等也会发生相应变化,进而可能影响其亚细胞定位,最终通过其各自编码产物对靶基因的识别、结合和转录调控等方面的变化,影响TaNAC转录因子调控其下游靶基因的表达。另外,有报道表明,在小麦抗病[30,31]、籽粒发育[32]等过程中均有可变剪切这种转录后调控方式参与,因此,推断TaNAC基因应该可以通过可变剪切这种转录后调控方式参与胁迫响应。本文选取并分析的13组TaNAC可变剪切结构变异转录本是在条锈菌和白粉菌胁迫下,从普通小麦兼抗种质N9134中克隆得到的,这些可变剪切形成的新TaNAC转录本大多没有被中国春参考基因组(IWGSC RefSeq v1.1)注释到,但是,它们到底是由于材料差异而特异存在的,还是因为小麦为了响应条锈菌和白粉菌的胁迫由可变剪切形成的,这些还需要深入研究。另外,基于克隆得到的TaNAC可变剪切结构变异转录本在小麦参考基因组注释信息中不存在的事实,推断小麦TaNAC基因在特定组织、不同生长阶段、或为了响应特定逆境,可能会通过可变剪切这种转录后调控方式,形成更多的转录本。本研究仅发现、比对了这些TaNAC可变剪切结构变异转录本,并对它们进行了初步分析,然而TaNAC基因可变剪切结构变异的影响因素、调控机制、引起的功能变化和对下游靶基因的影响等还需要进一步研究。
3.2 TaNAC基因可变剪切和miRNA耦联的转录后调控
从miRBase数据库收录的miRNA最新数据可知,在小麦、玉米(Zea mays L.)、水稻这三种最重要的粮食作物和两种常用的模式植物拟南芥和二穗短柄草(Brachypodium distachyon (L.) Beauv.)中,小麦中的miRNA前体和成熟miRNA的数量均是最少的(表7)[33]。比较这5个物种中其他功能基因家族成员数量的差异[6, 14],可以发现,基因组较小的物种所包含的基因家族成员数量一般不会比小麦、玉米等较大基因组的物种多。miRNA作为功能基因的调控因子均有对应的靶基因,据此推断,小麦、玉米等大基因组的物种中,miRNA家族的成员数量应该不会比基因组较小的模式植物少,至少不会像目前这样差距如此之大(表7)。Table 7
表7
表7小麦、玉米、水稻、二穗短柄草和拟南芥中已报道miRNAs的汇总分析
Table 7
| 植物 Plants | miRNA前体数量 Number of miRNA precursors | 成熟miRNA数量 Number of mature miRNAs | miRNA家族数量 Number of miRNA families | 基因组大小 Genome sizes (G) |
|---|---|---|---|---|
| 普通小麦 Triticum aestivum | 122 | 125 | 99 | 14.5 |
| 玉米 Zea mays | 174 | 325 | 31 | 2.4 |
| 水稻 Oryza sativa | 604 | 738 | 342 | 0.5 |
| 二穗短柄草 Brachypodium distachyon | 317 | 525 | 183 | 0.385 |
| 拟南芥 Arabidopsis thaliana | 326 | 428 | 215 | 0.13 |
新窗口打开|下载CSV
进一步对已报道的不同物种中miRNA家族成员的存在情况进行比较分析(图7),发现仅有miR156、miR159、miR160等14个miRNA家族在上述5个物种中同时存在,其余miRNA家族成员大多为单个物种独有。综合上述分析推断,基因组庞大且复杂的小麦中应该存在大量的miRNA待继续挖掘。
图7
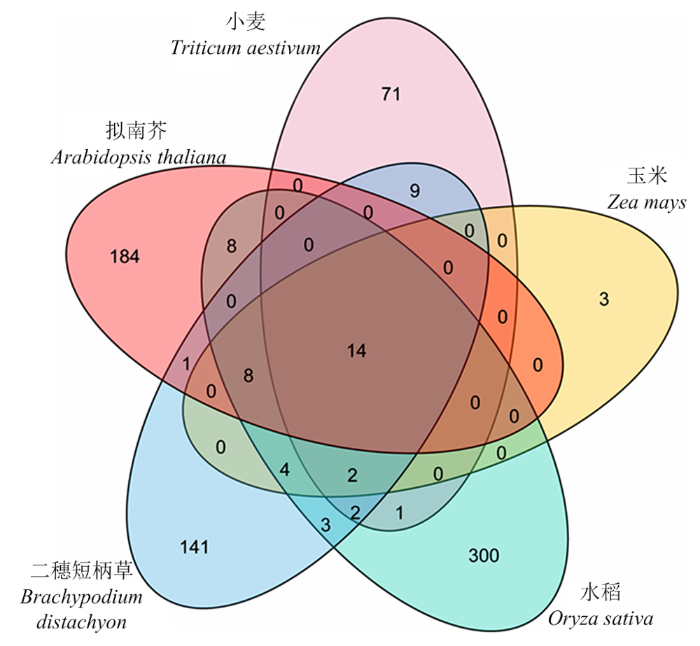 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7小麦、玉米、水稻、二穗短柄草和拟南芥中已报道miRNA家族成员的比较分析
Fig. 7Comparison of miRNA family members reported in Triticum aestivum, Zea mays, Oryza sativa, Brachypodium distachyon and Arabidopsis thaliana
目前,见诸报道的调控NAC基因的miRNA多为miR164家族成员[7, 34-35]。本研究利用已报道的小麦TaNAC基因家族和tae-miRNAs数据,预测到TaNAC基因除了tae-miR164家族同样可以受到其他miRNA家族的转录后调控,如tae-miR1128等。本研究丰富了靶向调控小麦TaNAC基因表达的tae-miRNA,但具体它们之间的调控关系、共同发挥的生物学功能等还需深入研究;基于小麦中有大量miRNA待挖掘,所以能够靶向调控TaNAC基因的tae-miRNA应该能够继续丰富。
miRNA可以通过和靶基因的ORF区结合参与转录后调控[23],本研究分析结果显示,在小麦中,tae-miRNA与TaNAC基因ORF区的结合位点均不涉及可变剪切事件,但因为小麦TaNAC基因应该存在更多的可变剪切事件[6],同时推断小麦中应该存在更多的miRNA,所以可以考虑将可变剪切和miRNA耦联的转录后调控方式作为它们各自单独对TaNAC基因进行转录后调控的扩充进行研究。
4 结论
同一编码基因可变剪切形成的结构变异转录本可以具有不同的生物学功能,据此推断,TaNAC基因应该可以通过可变剪切这种转录后调控方式参与小麦真菌胁迫响应。另外发现,TaNAC基因相关的miRNA相对于可变剪切独立行使转录后调控功能。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1111/tpj.2004.39.issue-6URL [本文引用: 1]
DOI:10.1105/tpc.104.022699URL [本文引用: 1]
DOI:10.1111/tpj.2006.46.issue-4URL [本文引用: 1]
[本文引用: 1]
DOI:10.1126/science.1248417URL [本文引用: 1]
DOI:10.3390/genes11091073URL [本文引用: 10]
DOI:10.1111/mpp.2014.15.issue-3URL [本文引用: 4]
DOI:10.1126/science.290.5499.2105URL [本文引用: 2]
DOI:10.1016/S0092-8674(00)81093-4URL [本文引用: 1]
DOI:10.1105/tpc.9.6.841URL [本文引用: 1]
DOI:10.1093/dnares/10.6.239URL [本文引用: 1]
DOI:10.1016/j.tplants.2004.12.010URL [本文引用: 1]
DOI:10.1371/journal.pone.0209769URL [本文引用: 1]
[本文引用: 2]
DOI:10.1371/journal.pone.0213390URL [本文引用: 2]
DOI:10.1111/plb.2015.17.issue-4URL [本文引用: 1]
DOI:10.1186/s12870-019-2022-5URL [本文引用: 1]
DOI:10.1071/FP05161URL [本文引用: 1]
[本文引用: 1]
DOI:10.1111/pbi.v17.10URL [本文引用: 2]
DOI:10.1111/jipb.v60.5URL [本文引用: 1]
DOI:10.1016/j.tplants.2012.02.004URL [本文引用: 2]
DOI:10.1038/nrm3611URL [本文引用: 2]
DOI:10.1016/j.plantsci.2019.05.023URL [本文引用: 1]
DOI:10.1016/j.pmpp.2018.02.002URL [本文引用: 1]
DOI:10.1093/plcell/koab046URL [本文引用: 1]
DOI:10.1105/tpc.113.117523URL [本文引用: 1]
DOI:10.1007/s00122-012-1809-7URL [本文引用: 1]
DOI:10.1186/1471-2164-15-898URL [本文引用: 1]
DOI:10.1016/j.plantsci.2019.05.023URL [本文引用: 1]
DOI:10.1038/s41477-021-00869-2URL [本文引用: 1]
DOI:10.1111/pbi.v19.8URL [本文引用: 1]
DOI:10.1093/nar/gky1141URL [本文引用: 1]
DOI:10.1093/jxb/eru072URL [本文引用: 1]
DOI:10.1126/science.1166386URL [本文引用: 1]
