 ,, 李治, 蒋庆, 陈姝霖, 欧霞, 陈永艳, 任天恒
,, 李治, 蒋庆, 陈姝霖, 欧霞, 陈永艳, 任天恒 ,四川农业大学农学院/四川省植物遗传和育种省级重点实验室,成都 611130
,四川农业大学农学院/四川省植物遗传和育种省级重点实验室,成都 611130Development and Effect Evaluation of KASP Markers Closely Linked to Major QTLs of Spike Number Per Unit Area and Grain Length in Wheat
FAN Tao ,, LI Zhi, JIANG Qing, CHEN ShuLin, OU Xia, CHEN YongYan, REN TianHeng
,, LI Zhi, JIANG Qing, CHEN ShuLin, OU Xia, CHEN YongYan, REN TianHeng ,College of Agronomy, Sichuan Agricultural University/Provincial Key Laboratory of Plant Genetics and Breeding of Sichuan Province, Chengdu 611130
,College of Agronomy, Sichuan Agricultural University/Provincial Key Laboratory of Plant Genetics and Breeding of Sichuan Province, Chengdu 611130通讯作者:
责任编辑: 李莉
收稿日期:2020-12-12接受日期:2021-02-3
| 基金资助: |
Received:2020-12-12Accepted:2021-02-3
作者简介 About authors
范涛,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (784KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
范涛, 李治, 蒋庆, 陈姝霖, 欧霞, 陈永艳, 任天恒. 小麦单位面积穗数和粒长主效QTL紧密连锁KASP标记的开发及其效应评价[J]. 中国农业科学, 2021, 54(14): 2941-2951 doi:10.3864/j.issn.0578-1752.2021.14.002
FAN Tao, LI Zhi, JIANG Qing, CHEN ShuLin, OU Xia, CHEN YongYan, REN TianHeng.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】普通小麦(Triticum aestivum L.)是世界上最重要的粮食作物之一,“养育”了世界范围内大约三分之一的人口。在所有的人类食物中,小麦提供的蛋白质和卡路里约占20%[1]。但是,根据预测,到2050年粮食的产量至少每年保持2.4%的增长率才能满足需求[2]。单位面积穗数、千粒重和每穗粒数是小麦主要的产量决定因素,因此,在育种中识别和引入相关的有利等位基因或QTL,对提高小麦产量具有重要意义。小麦的产量三要素单位面积穗数、穗粒数和粒重都是复杂的农艺性状,利用表型选择进行小麦的高产育种不仅费时费力,育种周期长从而导致育种成本提高。然而,与小麦产量单位面积穗数、穗粒数和千粒重相关的有利等位基因(或QTL)紧密连锁的分子标记的开发可以快速鉴定、识别相应的基因(或QTL),节约育种成本,提高育种效率。【前人研究进展】小麦籽粒粒重主要由籽粒形态决定,包括2个主要因素,即粒长和粒宽。目前分布在小麦所有染色体上的小麦籽粒形态以及千粒重相关的QTL已有诸多报道[3]。并且,与粒重或籽粒形态相关的TaGL3-5A、TaGW2-6A、TaTPP-6AL1、TaGW2-6B等基因也已经被克隆[4,5,6,7]。除此之外,还开发出许多与小麦粒重相关的功能基因的分子标记,并应用于育种上[8]。迄今为止,国内外研究****利用不同的研究群体,将小麦单位面积穗数的相关的QTL定位在1A、1B、1D、2A、2B、2D、3A、3B、4A、4B、5A、5B、5D、6A、7A、7B、7D等多条染色体上,并且开发了较多的分子标记[3,9-12]。但是相应的分子标记大多具有品种特异性,因此,并未应用到实际育种中。随着测序技术的发展,利用分子标记辅助育种作为一种传统育种方法的补充工具,越来越受到育种家们的广泛关注。刘子会等[13]通过简单关联分析鉴定到一个与小麦耐热相关的分子标记Xwmc44。陈泠等[14]对2个抗穗发芽的分子标记Tamyb10D和Vp1B3进行了有效性验证。LI等[15]针对小麦的穗颈长主效QTL开发了一个KASP分子标记,能够有效追踪该性状。MA等[16]开发了一个与粒长、粒宽和千粒重主效QTL紧密连锁的KASP分子标记,并且在重组自交群体中验证了它的有效性。胡洋山等[10]验证了2个分别位于3A和3B染色体上的SSR标记与小麦分蘖成穗呈现极显著正相关性,并证明它们可用于小麦分蘖成穗数的筛选和育种。【本研究切入点】利用SNP开发出的KASP标记筛选多穗和长粒小麦资源的研究仍鲜见报道。本文以分蘖成穗能力强的小麦品种川农18和具有籽粒性状优良的新品系材料T1208构建的371个RILs群体为材料,在前期研究中,通过QTL定位分析发现了一个与提高单位面积穗数的主效且稳定位于2D染色体上的QTL位点QSn.sau-2D.2,在中国春参考基因组(IWGSC RefSeq v1.0)上的物理位置是82.19—95.90 Mb,此QTL在3个环境中被稳定检测到,平均LOD值为17.20,平均解释表型变异率达到19.04%,遗传效应来源于川农18;此外,3D染色体上定位到一个提高籽粒粒长的QTL位点QKl.sau-3D.2,在中国春参考基因组(IWGSC RefSeq v1.0)上的物理位置是463.59—518.34 Mb,此QTL平均LOD值达到15.78,平均解释表型变异率为9.40%,遗传效应来源于亲本T1208,与以往研究对比,发现这两个QTL都是控制相应性状的新QTL位点[17]。【拟解决的关键问题】本研究依据QTL位点QSn.sau-2D.2和QKl.sau-3D.2区间内55K SNP基因芯片中的SNP分子标记设计KASP引物,并运用川农18×T1208杂交构建的RIL群体进行验证,评估这两个QTL对产量相关农艺性状的效应,以期为小麦分子标记辅助育种和多性状聚合育种提供理论依据。1 材料与方法
1.1 小麦田间材料及种植
供试材料一共445份,其中包括由川农18×T1208杂交构建的371个家系的重组自交系(RILs)用于KASP标记的开发,由川农17×川农11杂交构建的74个RILs群体为验证群体。川农18×T1208杂交构建的RILs群体于2015—2016、2016—2017、2017—2018、2018—2019、2019—2020年(以下简称2015、2016、2017、2018和2019)种植于四川农业大学小麦育种基地(四川省邛崃市,30°25′N,103°28′E,海拔493.3 m),川农17×川农11构建的RILs群体于2019—2020年(以下简称2019)种植于相同地点。采用随机区组设计,小区行长2 m,每行10株,行距0.25 m,每个株系种植4行,3次重复,栽培管理同大田标准化管理方法,使用除草剂和杀菌剂,以保证生长过程中田间无虫害和病害,避免对相应农艺性状的影响。在以SNP基因分型和相应性状的表型匹配中,表型选择以当年群体材料中表型平均值为标准划分,川农18×T1208的RILs群体在2015、2016和2017单位面积穗数平均值分别是291.06、280.80和258.38穗/m2,当年群体中高于此表型值的定义为多穗株系,粒长在2016、2018和2019年的平均值分别是7.27、7.22和6.82 mm,当年群体中高于此表型值的定义为长粒株系。川农17×川农11杂交构建的RILs群体中单位面积穗数和粒长平均值分别是238.50穗/m2和6.90 mm,高于此表型值的材料分别定义为多穗株系和长粒株系。1.2 小麦各农艺性状的调查
在植株成熟后,调查株高,株高的调查是从土壤表面到植株的顶端(不包含麦芒);单位面积穗数调查则是每行调查5株,每个株系3个重复,最后取平均值,再乘以4得到每平方米单位面积穗数;收获时每个株系收获10穗,脱粒称取重量后,计算每穗粒重,公式为每穗粒重=十穗粒重/10。采用万深SC-G自动考种分析及千粒重分析仪(由杭州万深检测科技有限公司生产)测定千粒重、粒长和粒宽。1.3 KASP引物设计及合成
根据构建的遗传连锁图谱上的信息[18],分别查找位于QSn.sau-2D.2内的SNP分子标记AX-109283238、AX-86163992、AX-108802182、AX-110073027和AX- 111151907,以及位于QKl.sau-3D.2内的SNP分子标记AX-109962767和AX-108923200,这些SNP标记的序列如表1所示,根据它们的SNP序列,使用在线网站平台PolyMarker(Table 1
表1
表1试验中涉及的SNP标记及序列
Table 1
| SNP标记 SNP marker | 染色体 Chromosome | 变异碱基 Base | 序列 Sequence (5′-3′) |
|---|---|---|---|
| AX-109283238 | 2D | C//T | CCCTTGTCTCTTCCATCTATTTTCCCACCATCGTA[C/T]GGTTCCTTCTTTAATATTGTGTCTCTTTCCTCGAT |
| AX-111151907 | 2D | G//T | GCCACCTACCTAGAAACCCTACCCAGCAGCCACAA[G/T]ATCCCCCAAAAATCGGATTCCTTTGCTGGACTAAC |
| AX-86163992 | 2D | A//G | TTGCTCAGCTTCTGGAAATACTGCTGCTTCTGAAC[A/G]AGCTTCTCGCGTGGGTAAAATGGTGCTTCTTCGAG |
| AX-108802182 | 2D | A//G | TGCAGGATAATCCTAGAGCCAGGGGGATGTACGGT[A/G]TTCCATTTCCGCATTTCGATGTGTTTGATGCGGTG |
| AX-110073027 | 2D | G//T | TCGATATGAAGTCTGGTGAAACCGATGGGTACGAC[G/T]CTGTTTTCTTGAGTCCTCACAAATTTGTCGGGGGA |
| AX-109962767 | 3D | A//G | AACTATCGGATTGGAAACAAATTGACTCACATACA[A/G]CAAATGATATGTGAGGCCGAGTAGCACAAGCTAGG |
| AX-108923200 | 3D | A//G | CTCGAACAAATGAGTTGAAGAAACTGCCATCATCA[A/G]AAGACTGAAGCAAAACAAGTTTGAGACAACCACTG |
新窗口打开|下载CSV
Table 2
表2
表2试验中涉及的KASP分子标记引物序列
Table 2
| QTL | 分子标记 Molecular marker | 引物序列 Primer sequence (5′-3′) |
|---|---|---|
| QSn.sau-2D.2 | KASP-AX-109283238-1 | GAAGGTGACCAAGTTCATGCTCCATCTATTTTCCCACCATCGTAC |
| KASP-AX-109283238-2 | GAAGGTCGGAGTCAACGGATTCCATCTATTTTCCCACCATCGTAT | |
| KASP-AX-109283238-3 | ACCTTTATCGAGGAAAGAGACACA | |
| KASP-AX-111151907-1 | GAAGGTGACCAAGTTCATGCTCCTACCCAGCAGCCACAAG | |
| KASP-AX-111151907-2 | GAAGGTCGGAGTCAACGGATTCCTACCCAGCAGCCACAAT | |
| KASP-AX-111151907-3 | AAGGCCAGTGTTAGTCCAGC | |
| KASP-AX-86163992-1 | GAAGGTGACCAAGTTCATGCTGGAAATACTGCTGCTTCTGAACA | |
| KASP-AX-86163992-2 | GAAGGTCGGAGTCAACGGATTGGAAATACTGCTGCTTCTGAACG | |
| KASP-AX-86163992-3 | TTGTATGCAGGATGACGCTC | |
| KASP-AX-108802182-1 | GAAGGTGACCAAGTTCATGCTCATCGAAATGCGGAAATGGAAT | |
| KASP-AX-108802182-2 | GAAGGTCGGAGTCAACGGATTCATCGAAATGCGGAAATGGAAC | |
| KASP-AX-108802182-3 | GTGTCTTTGTGTTGATTTGTTTCA | |
| KASP-AX-110073027-1 | GAAGGTGACCAAGTTCATGCTTGTGAGGACTCAAGAAAACAGC | |
| KASP-AX-110073027-2 | GAAGGTCGGAGTCAACGGATTTGTGAGGACTCAAGAAAACAGA | |
| KASP-AX-110073027-3 | ATCCTTTCTGCAGTGGACCC | |
| QKl.sau-3D.2 | KASP-AX-109962767-1 | GAAGGTGACCAAGTTCATGCTTGGAAACAAATTGACTCACATACAA |
| KASP-AX-109962767-2 | GAAGGTCGGAGTCAACGGATTTGGAAACAAATTGACTCACATACAG | |
| KASP-AX-109962767-3 | GTGCTACTCGGCCTCACATA | |
| KASP-AX-108923200-1 | GAAGGTGACCAAGTTCATGCTGTTGAAGAAACTGCCATCATCAA | |
| KASP-AX-108923200-2 | GAAGGTCGGAGTCAACGGATTGTTGAAGAAACTGCCATCATCAG | |
| KASP-AX-108923200-3 | CGTCCAGTGGTTGTCTCAAAC |
新窗口打开|下载CSV
1.4 KASP引物多态性筛选及群体中验证
对亲本川农18和T1208随机选取10粒种子,于培养皿中发芽培养至三叶期,根据李荣华等[19]方法并稍做改进提取基因组DNA,使用1%琼脂糖凝胶电泳检测DNA质量。利用上述设计引物进行PCR扩增。PCR反应体系为4.5 μL 1×KASP Master mixture(LGC Genomics, Hoddeston, UK)、2 μL(50 ng·μL-1)基因组DNA、2 μL KASP引物mix(正向引物1﹕正向引物2﹕反向引物﹕水=6﹕6﹕15﹕23)和1.5 μL ddH2O。PCR反应程序为95℃ 10 min;95℃ 20 s,61℃ 40 s,循环10次,每次循环退火延伸温度下降0.6℃;95℃ 20 s,55℃ 40 s,循环30次;25℃保温,采集荧光信号。PCR反应在荧光定量PCR仪进行(BioRad,CFX-96)。利用筛选出来具有多态性的KASP标记在川农18×T1208构建的371个株系的RILs群体进行基因分型,以及在川农17×川农11杂交构建的74个RILs群体进行验证,PCR的反应条件同上。1.5 数据统计分析
数据统计分析采用软件Microsoft Excel 2016和SPSS 22(IBM SPSS, Armonk,NY,USA)对农艺性状等数据进行方差分析、T检验、相关性分析和描述性统计分析。参照李聪等[20]方法计算各农艺性状最佳线性无偏预测(best linear ubiased prediction,BLUP)值。利用CFX96荧光定量PCR仪的配套软件BioRad CFX Manager的基因分型模块,读取各样品不同荧光信号值,并分析基因型。KASP-AX-111151907基因分型结果与川农18相同的基因型定义为多穗基因型株系,与T1208相同的基因型定义为少穗基因型株系;KASP-AX-109962767基因分型结果与T1208相同的基因型定义为长粒基因型株系,与川农18相同的基因型定义为短粒基因型株系。2 结果
2.1 川农18×T1208 RILs群体中单位面积穗数和粒长表型变异分析
在该群体中,单位面积穗数在2015、2016和2017年的平均值分别是291.06、280.80和258.38穗/m2,表型变异范围分别是185.3—442.7、179.3—387.3和120.0—442.7穗/m2;粒长在2016、2018和2019年的平均值分别是7.27、7.22和6.82 mm,表型变异范围是6.20—8.80、6.15—8.60和5.74—8.13 mm。单位面积穗数与粒长呈显著性负相关(r=-0.44,P<0.001)。2.2 KASP引物筛选结果
筛选QSn.sau-2D.2区间内引物时发现,KASP-AX- 111151907在亲本中具有多态性,该引物对亲本川农18分型结果是等位基因1类型,发出FAM荧光类型,T1208则是等位基因2类型,发出HEX荧光类型。其余设计的KASP引物不能很好地同时将2个亲本基因型识别出来(图1-a)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1双亲间多态性引物的筛选
a:KASP-AX-111151907对亲本基因分型;b:KASP-AX-109962767对亲本基因分型
Fig. 1Screening of polymorphic primers between parents
a: Genotyping of parents by KASP-AX-111151907; b: Genotyping of parents by KASP-AX-109962767
筛选QKl.sau-3D.2区间内引物时发现,KASP-AX- 109962767在亲本中具有多态性,此引物对亲本川农18分型结果是等位基因1类型,发出FAM荧光类型,T1208则是等位基因2类型,发出HEX荧光类型。其余设计的KASP引物不能很好地同时将2个亲本基因型识别出来(图1-b)。
2.3 分子标记KASP-AX-111151907和KASP-AX-109962767在RILs群体中分型结果
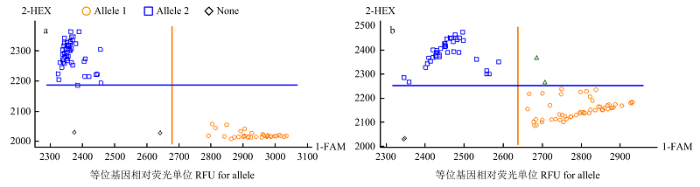
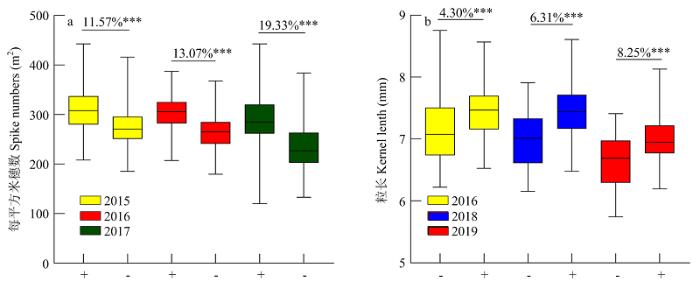
利用筛选到的多态性引物KASP-AX-111151907对上述371个川农18×T1208的RILs群体进行基因分型。该引物对群体中不同的材料能够很好的进行分型,各信号点荧光值较高,且不同基因型之间的夹角较大(图2-a),因此,分型效果较好。与亲本川农18相同的多穗基因型信号聚合在X轴,亲本T1208相同的少穗基因型信号聚合在Y轴。对371个株系的分型结果显示,有2个株系是杂合基因型,因此,在后续的分析中也剔除掉它们进行分析。引物KASP-AX- 111151907鉴定到的369个株系中有158个株系是多穗基因型,这些多穗基因型中对应的多穗表型株系在2015、2016和2017年分别有99、122和123株,对应的多穗材料选择率分别是62.66%、77.22%和77.85%;剩下的211个株系是少穗基因型,其对应的少穗表型株系在2015、2016和2017年分别有148、151和155株,对应的少穗材料选择率分别是70.01%、71.56%和73.46%。在2015、2016和2017年分别有164、183和180株多穗表现型的材料,其中多穗基因型分别有100、183和180株,因此,这3年多穗材料中多穗基因型分别占60.98%、86.89%和88.33%。159个多穗基因型株系在2015、2016和2017年的表型平均值分别是311.60、303.41和290.50穗/m2,211个少穗基因型株系在2016、2018和2019年的平均值分别是275.56、263.75和234.36穗/m2。2种基因型材料在单位面积穗数上存在显著性差异(P<0.001),表明KASP-AX-111151907与QSn.sau-2D.2紧密连锁(表3和图3-a)。图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2分子标记在RIL群体中验证
a:KASP-AX-111151907在RIL群体中部分基因分型结果;b:KASP-AX-109962767在RIL群体中部分基因分型结果
Fig. 2Validation of molecular markers in RIL population
a: Partial genotyping results of KASP-AX-111151907 in RIL population; b: Partial genotyping results of KASP-AX-109962767in RIL population
Table 3
表3
表3基于KASP分子标记验证QSn.sau-2D.2和QKl.sau-3D.2对相应性状的影响
Table 3
| QTL | 环境 Environment | 基因型A Genotype A | 基因型B Genotype B | 差值 Difference | P值 P value |
|---|---|---|---|---|---|
| QSn.sau-2D.2 | 2015 | 311.60(n=158) | 275.56(n=211) | 36.04 | <0.001 |
| 2016 | 303.41(n=158) | 263.75(n=211) | 39.66 | <0.001 | |
| 2017 | 290.50(n=158) | 234.36(n=211) | 56.14 | <0.001 | |
| QKl.sau-3D.2 | 2016 | 7.11(n=188) | 7.43(n=175) | 0.32 | <0.001 |
| 2018 | 6.98(n=188) | 7.45(n=175) | 0.47 | <0.001 | |
| 2019 | 7.01(n=188) | 7.64(n=175) | 0.63 | <0.001 |
新窗口打开|下载CSV
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3QSn.sau-2D.2对单位面积穗数的影响(a)和QKl.sau-3D.2对粒长的影响(b)
+:含有相应QTL的株系,-:不含有相应QTL的株系;***:在P<0.001水平差异极显著
Fig. 3The effect of QSn.sau-2D.2 on spike numbers per unit area (a) andQKl.sau-3D.2 effect on kernel length (b)
+: Lines containing the corresponding QTL; -: Lines without the corresponding QTL; ***: Significant at the level of P<0.001
利用筛选到的多态性引物KASP-AX-109962767对川农18×T1208的RILs群体进行基因分型,结果与KASP-AX-111151907类似,对不同基因型材料的分型结果较好(图2-b)。该引物将与亲本T1208相同的长粒基因型信号聚合在Y轴,与亲本川农18相同的短粒基因型聚合在X轴。对371个株系基因分型结果显示,有8个株系是杂合基因型,因此,在后续的分析中剔除掉它们进行分析。KASP-AX-109962767鉴定到的363个株系中,有188个株系是长粒基因型,这些长粒基因型对应的长粒表型株系在2016、2018和2019年分别有127、132和135株,对应的长粒材料选择率分别是67.55%、70.21%和71.81%。剩下的175个株系是短粒基因型,其对应的短粒表型株系在2016、2018和2019年分别有104、114和105株,对应的短粒材料的选择率分别是59.43%、65.14%和60.00%。在2016、2018和2019年分别有199、194和206株长粒表型株系,而在这3年里长粒基因型分别有128、133和136株,因此,在这3年中长粒材料中长粒基因型分别占64.32%、68.56%和66.02%。188个长粒基因型株系在2016、2018和2019年的平均值分别是7.43、7.45和7.64 mm,175个短粒基因型株系在2016、2018和2019年的平均值分别是7.11、6.98和7.01 mm。2种基因型材料在粒长上存在显著性差异(P<0.001),表明KASP-AX-109962767与QKl.sau- 3D.2紧密连锁(表3和图3-b)。
另外,在这371个群体中,只有39个株系含有多穗基因型和长粒基因型,有117个株系只含有多穗基因型而不含有长粒基因型,有149株只含有长粒基因型而不含有多穗基因型,既不含有多穗基因型又不含有长粒基因型的株系58株。
2.4 基于KASP分子标记基因分型评价这两个主效QTL对其他产量相关性状的影响
基于各农艺性状的BLUP值,针对上述2个分别QSn.sau-2D.2连锁的分子标记KASP-AX-111151907和与QKl.sau-3D.2连锁的分子标记KASP-AX-109962767分型结果,利用t检验分析这两个QTL对于其他农艺性状的影响(表4)。结果表明,QSn.sau-2D.2对于株高、千粒重、粒长、粒宽、粒径比和每穗粒重都具有显著性降低的影响(P<0.001),QKl.sau-3D.2对于株高、千粒重、粒宽、粒径比和每穗粒重有显著性提高的作用,但是对于单位面积穗数是降低的作用(P<0.001)。Table 4
表4
表4基于KASP分子标记验证QSn.sau-2D.2和QKl.sau-3D.2对产量相关性状的影响
Table 4
| QTL | 基因型 Genotype | 株高 Plant height (cm) | 千粒重 1000 kernel weight (g) | 粒长 Kernel length (mm) | 粒宽 Kernel width (mm) | 粒径比 Kernel diameter ratio | 单位面积穗数 Spike numbers per unit area/m2 | 每穗粒重 Kernel weight per spike (g) |
|---|---|---|---|---|---|---|---|---|
| QSn.sau-2D.2 | A | 78.85*** | 45.77*** | 6.89*** | 3.45*** | 1.99*** | 290.67*** | 2.26*** |
| B | 88.47 | 49.58 | 7.30 | 3.50 | 2.09 | 234.49 | 2.62 | |
| QKl.sau-3D.2 | A | 80.25 | 45.89 | 6.90 | 3.46 | 1.99 | 281.48 | 2.29 |
| B | 88.26*** | 49.95*** | 7.34*** | 3.50*** | 2.10*** | 235.13*** | 2.62*** |
新窗口打开|下载CSV
2.5 分子标记KASP-AX-111151907和KASP-AX-109962767在其他群体中的检验
为了验证本研究中在川农18×T1208的RILs群体中开发的KASP标记是否可用于其他材料中小麦单位面积穗数和粒长的筛选,利用川农17×川农11的74个RILs群体进行基因分型(图4),KASP-AX- 111151907和KASP-AX-109962767能够很好地将不同基因型的小麦材料进行区分。KASP-AX-111151907基因分型结果显示,有43个材料是多穗基因型,其中20个材料是多穗表现型,多穗材料选择率是46.51%,有31个材料为少穗基因型,其中少穗表现型材料有14个,对于少穗材料的选择率达到45.16%。KASP-AX-109962767基因分型结果显示,长粒基因型有38个,其中17个为长粒表现型,长粒的选择率达到44.74%,而短粒基因型有36个,其中短粒表现型有18个,对短粒基因型的选择率达到50.00%。表明这两个分子标记对川农17×川农11杂交群体中长粒和多穗的选择具有一定的作用。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4分子标记KASP-AX-111151907(a)和KASP-AX-109962767(b)在川农17×川农11 RIL群体中验证的部分结果
Fig. 4Partial results of molecular markers KASP-AX-111151907(a) and KASP-AX-109962767 (b) in the RIL population of Chuannong17×Chuannong 11
3 讨论
3.1 协调型小麦
提高产量一直以来是众多育种学家们的基本目标。单位面积穗数、穗粒数和粒重作为小麦的产量三要素,与小麦的产量直接相关。四川盆地属于中国的西南麦区,是典型的雨养农业区,阴天较多,光照少导致小麦的分蘖成穗能力弱,从而导致单位面积穗数减少,进而造成产量下降[21,22]。在过去普遍的看法是四川盆地由于光照少和暖冬而存在“生态穗容量”,本课题组根据西南麦区的气候特征、土壤类型、光照条件及栽培模式等选育的多穗协调型小麦,其代表品种有川农12、川农17和川农18[23]。该类型的小麦品种能在保持较高穗重的前提下具有较高穗数,穗数及穗重相互补偿能力较强,为打破中国西南小麦生态区“生态穗容量”的限制提供了新的思路[24]。在前期的研究中也发现川农18控制单位面积穗数的主效QTL位于2D染色体上[17],为阐明协调型小麦品种川农18成穗能力机制奠定了基础。本研究中开发的与川农18控制单位面积穗数的主效稳定QTL位点QSn.sau-2D.2紧密连锁的KASP标记将有助于育种人员开展多穗小麦品种的选育工作。3.2 多穗和长粒KASP标记的利用
传统的杂交育种可以将不同的优良等位基因聚合在一起,获得理想性状,但是具有很大的局限性,对相应性状分子遗传机制不了解,育种周期长,并且需要花费大量时间和精力来鉴定表型,从而导致育种成本的提高,育种效率低下[25]。分子标记辅助育种如SSR标记、CAPS标记等已经广泛应用于小麦的育种工作中,张兆萍等[26]利用4个与穗发芽抗性相关的标记在350份不同小麦种质材料进行验证,最终筛选出2个分子标记可用于穗发芽抗性筛选,为筛选抗穗发芽小麦品种提供理论指导。WANG等[27]根据粒重相关基因TaGS5-A1的多态性序列开发了相应的CAPS标记,能够有效区分不同等位基因从而达到筛选具有更高粒重的小麦品种。SNP分子标记作为植物基因组中最丰富的一类分子标记类型,目前已经广泛应用于遗传图谱构建,遗传变异分析等[28]。但是其在分子标记辅助育种应用程度仍然不够普遍化[29]。本研究中开发的2个KASP分子标记,其中单位面积穗数相关的KASP分子标记在川农18×T1208杂交构建的RILs群体中能有效地对多穗基因型材料进行选择,在多年试验条件下,对多穗基因型材料的平均选择率可达到72.58%,对低穗基因型的材料平均选择率可达71.68%,而在多穗材料中,3年试验条件下,多穗基因型分别占了60.98%、86.89%和88.33%,而在川农17×川农11杂交构建的RIL群体中该标记对于多穗基因型的选择率可达46.51%,以上结果表明本研究开发的用于筛选多穗材料的KASP分子标记可靠性较高。值得注意的是,在本研究中单位面积穗数KASP分子标记对于多穗基因型材料的选择率分析是建立在稀播的基础上,以往的研究表明小麦品种川农18在大田生产密度下相比于对照成穗数显著性增加[23],因此,在大田种植密度下该分子标记对于多穗基因型材料的选择率应该会有所增加。另外,本研究中开发的与粒长相关的KASP分子标记,在川农18×T1208杂交构建的RILs群体中对长粒基因型的平均选择率可达69.86%,对短粒基因型的平均选择率可达61.52%,在长粒材料中,3年试验条件下,长粒基因型分别占了64.32%、68.56%和66.02%,而在川农17×川农11杂交构建的RIL群体中该标记对于长粒基因型的选择率达44.74%,以上结果表明本研究开发的用于筛选长粒材料的KASP分子标记可靠性较高。本研究中多穗表型材料中多穗基因型占比和长粒表型材料中长粒基因型占比并未达到百分之百,可能的原因是因为这两个性状都属于数量性状由多基因控制,除了鉴定到的稳定主效QTL控制以外,还有许多微效基因共同控制相应的性状,而在之前的研究中也表明了单位面积穗数和粒长还有诸多微效QTL控制[17]。基因聚合育种与传统聚合育种相结合已经成为了已经成为当今作物育种的新思路,并且已经取得了一定进展,如SALAMEH等[30]通过分子标记辅助育种选择将春小麦品系CM-82036的Fhbl和Qfhs.ifa-5A这两个抗性主效基因导入到9个欧洲冬小麦品系中,结果发现当Fhbl和Qfhs.ifa-5A聚合在一起时对赤霉病的抗性相比于单独Fhb1更强。众所周知,由于营养竞争的关系,小麦的穗数增加,往往伴随着粒重的下降[31,32,33]。因此,如果将增加粒重的基因与增加穗数的基因聚合在一起,理论上小麦的产量将得到提高。在本研究中,川农18×T1208的RILs群体中,有39个株系同时含有增加穗数和粒长的基因型,但是它们具体的产量表现如何,还有待进一步的验证。
3.3 多穗和长粒KASP分子标记的其他用途
在小麦的产量相关性状中,各农艺性状之间往往具有显著的相关性,如小麦的千粒重与粒长、粒宽之间呈现显著性正相关,而与单位面积穗数、小穗数呈现出显著性负相关[34]。这往往归功于基因的“一因多效”,基因的“一因多效”在植物中广泛存在,如小麦的矮秆基因Rht-B1也多次被报道对于多个农艺性状具有影响,包括株高、千粒重、粒宽、单位面积穗数、旗叶大小等[17]。LIN等[35]定位到的2个控制小麦籽粒灌浆速度的QTL也同时对千粒重、粒长、粒宽、籽粒体积、籽粒面积、籽粒周长具有显著性影响。在本研究中,具有多穗基因型的株系在株高、千粒重、粒长、粒宽、粒径比和穗粒重方面都显著性低于少穗基因型的株系;具有长粒基因型的株系在株高、千粒重、粒宽、粒径比和穗粒重方面显著高于短粒基因型株系,在单位面积穗数上显著性低于短粒株系,这也表明本研究中开发的2个KASP分子标记对于农艺性状株高、千粒重、粒长、粒宽、粒径比、单位面积穗数和穗粒重的选择上都具有一定的作用。4 结论
开发的与单位面积穗数相关的KASP分子标记KASP-AX-111151907和粒长相关的KASP分子标记KASP-AX-109962767能够分别区分川农18×T1208构建的RILs群体中的单位面积穗数和籽粒粒长的基因型,并有效鉴定单位面积穗数和粒长表型值的相对高低,可用于小麦高产育种。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1007/s12571-013-0248-xURL [本文引用: 1]
DOI:10.1371/journal.pone.0066428URL [本文引用: 1]
DOI:10.1007/s00122-020-03562-8URL [本文引用: 2]
DOI:10.1007/s00122-019-03316-1URL [本文引用: 1]
DOI:10.1007/s00122-010-1437-zURL [本文引用: 1]
DOI:10.1007/s11032-017-0676-yURL [本文引用: 1]
DOI:10.1186/1471-2229-14-1URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.3389/fpls.2019.00187URL
DOI:10.1007/s13353-016-0349-2URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s00122-020-03675-0URL [本文引用: 1]
DOI:10.1186/s12863-019-0782-4URL [本文引用: 1]
URL [本文引用: 4]
DOI:10.3389/fpls.2018.00333URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1111/pbi.v18.6URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/s41598-017-04028-6URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s11032-010-9498-xURL [本文引用: 1]
DOI:10.1371/journal.pone.0118847URL [本文引用: 1]
DOI:10.1093/oxfordjournals.aob.a085523URL [本文引用: 1]
DOI:10.1023/A:1017597923568URL [本文引用: 1]
DOI:10.1007/s00122-017-2954-9URL [本文引用: 1]
DOI:10.1007/s00122-020-03605-0URL [本文引用: 1]
