 ,1, 张建2, 温常龙2, 樊秀彩1, 张颖1, 孙磊1, 刘崇怀1, 姜建福
,1, 张建2, 温常龙2, 樊秀彩1, 张颖1, 孙磊1, 刘崇怀1, 姜建福 ,1
,1Identification of Grape Cultivars Based on KASP Markers
WANG FuQiang ,1, ZHANG Jian2, WEN ChangLong2, FAN XiuCai1, ZHANG Ying1, SUN Lei1, LIU ChongHuai1, JIANG JianFu
,1, ZHANG Jian2, WEN ChangLong2, FAN XiuCai1, ZHANG Ying1, SUN Lei1, LIU ChongHuai1, JIANG JianFu ,1
,1通讯作者:
责任编辑: 赵伶俐
收稿日期:2020-08-26修回日期:2021-01-23网络出版日期:2021-07-01
| 基金资助: |
Received:2020-08-26Revised:2021-01-23Online:2021-07-01
作者简介 About authors
王富强,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (173KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王富强, 张建, 温常龙, 樊秀彩, 张颖, 孙磊, 刘崇怀, 姜建福. 基于KASP标记的葡萄品种鉴定[J]. 中国农业科学, 2021, 54(13): 2830-2842 doi:10.3864/j.issn.0578-1752.2021.13.012
WANG FuQiang, ZHANG Jian, WEN ChangLong, FAN XiuCai, ZHANG Ying, SUN Lei, LIU ChongHuai, JIANG JianFu.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】葡萄为葡萄科(Vitaceae)葡萄属(Vitis L.)多年生藤本植物[1],全世界至少有23 000个品种(http://www.vivc.de/),其适应性强,适栽范围广。不同地区间葡萄品种交流频繁,而部分引种或地方品种又没有确切的科学名称,难免发生同物异名或同名异物现象[2]。另外,当前葡萄苗木市场管理暂不完善,随意更改品种名称、炒作品种等不良现象时有发生,极大损害了育种家的利益,不利于葡萄产业的健康、可持续发展。因此,寻找一种经济、高效、准确的品种鉴别方法,对于提高我国葡萄种质资源管理效率和品种保护能力均具有重要意义。【前人研究进展】DNA分子标记具有周期短、受环境影响小、可实现高通量检测等优点,为葡萄品种鉴定提供了新的手段。其中SSR(simple sequence repeat)和SNP(single nucleotide polymorphism)分子标记已被国际植物新品种权保护联盟(UPOV)BMT分子测试指南[3]和国内《植物品种鉴定DNA指纹方法总则》(NY/T 2594-2016)[4]定为优先推荐的两种标记方法。目前,国内已研发出一套用于中国葡萄品种鉴定的30个SSR标记体系[5],并利用其中8个引物区分国内290份葡萄品种[6]。但因SSR标记数量和检测通量有限、检测成本偏高、数据读取费时耗力等因素[7],限制了更大范围的葡萄品种鉴定工作。SNP分子标记作为最新一代标记,具有数量多、具二态性、稳定遗传等优点[8],加上各种SNP高通量检测平台的出现,可以很好弥补SSR标记的技术缺陷[9],已在生物学、农业、医学和生物进化等领域得到了广泛应用[10]。其中,高通量SNP检测技术主要有基因芯片和LGC公司推出的KASP(kompetitive allele specific PCR)技术,适用于不同检测需求[9]。基因芯片技术主要适用于少样本多位点检测,如我国自主研发的Wheat 660K[11]、Rice SNP50[12]、Maize 50K[13]基因芯片,单次位点检测量达上万个,不足之处是当前使用成本偏高,开发难度较大;KASP技术则适于多样本、少位点的检测,具有高效、灵活、准确、低成本的特点[14],国内外研究者已利用KASP技术分别构建小麦[15,16]、玉米[17]、水稻[10]、棉花[18]、甘蓝[19]、黄瓜[20]等作物的核心标记体系,为相应作物的品种鉴定和品种保护等提供了便利。而SNP标记在葡萄品种鉴定中,主要使用成本较高的基因芯片和测序技术,如CABEZAS等[21]已构建了针对欧亚种葡萄的SNP鉴定体系,并确定了48个SNP为鉴定品种的标准位点;LAUCOU等[22]基于18K SNPs葡萄基因芯片从945份材料中筛选出14个高多态性SNP标记用于欧洲地区栽培品种鉴定。【本研究切入点】目前国际上筛选出的SNP标记仅在二倍体欧亚种中得到应用,而我国主栽的葡萄多为多倍体品种,因此有必要开发适用范围更广的SNP标记。虽然李贝贝等[23]和LIANG等[24]利用不同重测序技术获得上万个SNP位点,分别对304份和472份葡萄种质进行了遗传多样性分析,但没有筛选出用最少的SNP位点组合来鉴别葡萄品种的具体结果。在实际的葡萄品种鉴定工作中,研究者希望用最少的位点区分更多的样本,以达到高效、准确、廉价的鉴定目的,比较而言,KASP技术更适合SNP分子标记开发。【拟解决的关键问题】本研究基于前人开发的葡萄SNP标记,以国内主栽葡萄品种、新育成品种为试材,首次尝试利用KASP技术筛选出一套能够鉴别中国主栽葡萄品种的核心KASP标记,并构建葡萄品种SNP指纹图谱,为我国葡萄新品种保护、市场维权等提供技术支持。1 材料和方法
1.1 试验材料
试验材料于2020年6月采自中国农业科学院郑州果树研究所国家果树种质郑州葡萄圃。KASP标记初筛:使用23份葡萄代表性品种,其具备不同倍性、不同种性、不同用途等特点,如二倍体欧亚种酿酒品种‘赤霞珠’‘黑比诺’,二倍体欧美杂种鲜食品种‘阳光玫瑰’‘金手指’,三倍体欧美杂种鲜食品种‘夏黑’,四倍体欧美杂种鲜食品种‘巨峰’‘巨玫瑰’等(表1)。KASP标记复筛:使用76份葡萄品种,其中13份国内主栽品种、63份近年新育成品种(表2)。
Table 1
表1
表1KASP标记初筛使用的23份葡萄代表品种信息
Table 1
| 编号 Code | 品种 Cultivar | 倍性 Ploidy | 种性 Species | 用途 Purpose | 来源 Origin |
|---|---|---|---|---|---|
| 1 | 赤霞珠 Cabernet Sauvignon | 2x | 欧亚种V. vinifera | 酿酒Wine grape | 法国France |
| 2 | 霞多丽Chardonnary | 2x | 欧亚种V. vinifera | 酿酒Wine grape | 法国France |
| 3 | 黑比诺Pinot Noir | 2x | 欧亚种V. vinifera | 酿酒Wine grape | 法国France |
| 4 | 梅鹿辄Merlot | 2x | 欧亚种V. vinifera | 酿酒Wine grape | 法国France |
| 5 | 紫秋Ziqiu | 2x | 刺葡萄V. davidii | 酿酒Wine grape | 中国China |
| 6 | 北冰红 Beibinghong | 2x | 山欧杂种 V. amurensis-V. vinifera | 酿酒Wine grape | 中国China |
| 7 | 无核白鸡心 Centennial Seedless | 2x | 欧亚种V. vinifera | 制干Raisin grape | 美国America |
| 8 | 康可Concord | 2x | 美洲种V. labrusca | 制汁Juice grape | 美国America |
| 9 | 贝达Beta | 2x | 美河杂种 V. labrusca-V. riparia | 砧木Rootstock | 美国America |
| 10 | 抗砧3号 Kangzhen No.3 | 2x | 冬河杂种 V. berlandieri-V. riparia | 砧木Rootstock | 中国China |
| 11 | SO4 | 2x | 冬河杂种 V. berlandieri-V. riparia | 砧木Rootstock | 德国Germany |
| 12 | 香妃Xiangfei | 2x | 欧亚种V. vinifera | 鲜食Table grape | 中国China |
| 13 | 玫瑰香 Muscat Hamburg | 2x | 欧亚种V. vinifera | 鲜食Table grape | 英国England |
| 14 | 红地球Red Globe | 2x | 欧亚种V. vinifera | 鲜食Table grape | 美国America |
| 15 | 美人指 Manicure Finger | 2x | 欧亚种V. vinifera | 鲜食Table grape | 日本Japan |
| 16 | 金手指Gold Finger | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 日本Japan |
| 17 | 阳光玫瑰 Shine Muscat | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 日本Japan |
| 18 | 夏黑Summer Black | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 日本Japan |
| 19 | 月光无核 Yueguangwuhe | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 中国China |
| 20 | 峰光Fengguang | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 中国China |
| 21 | 京亚Jingya | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 中国China |
| 22 | 巨玫瑰Jumeigui | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 中国China |
| 23 | 巨峰Kyoho | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食Table grape | 日本Japan |
新窗口打开|下载CSV
Table 2
表2
表2KASP标记复筛使用的76份葡萄品种信息
Table 2
| 编号 Code | 品种 Cultivar | 倍性 Ploidy | 种性 Species | 用途 Purpose | 编号 Code | 品种 Cultivar | 倍性 Ploidy | 种性 Species | 用途 Purpose | |
|---|---|---|---|---|---|---|---|---|---|---|
| G1 | 惠良刺葡萄 Huiliangciputao | 2x | 刺葡萄 V. davidii | 制汁 Juice grape | G26 | 短枝玉玫瑰 Duanzhiyumeigui | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G2 | 野酿2号 Yeniang No.2 | 2x | 毛葡萄 V. heyneana | 酿酒 Wine grape | G27 | 玉波黄地球 Yubohuangdiqiu | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G3 | 媚丽 Meili | 2x | 欧亚种 V. vinifera | 酿酒 Wine grape | G28 | 瑞都红玫 Ruiduhongmei | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G4 | 泰美 Taimei | 2x | 欧亚种 V. vinifera | 酿酒 Wine grape | G29 | 瑞都脆霞 Ruiducuixia | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G5 | MCS2 | 2x | 欧亚种 V. vinifera | 酿酒 Wine grape | G30 | 瑞都无核怡 Ruiduwuheyi | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G6 | 玉玲珑 Yulinglong | 2x | 欧亚种 V. vinifera | 酿酒 Wine grape | G31 | 瑞都香玉 Ruiduxiangyu | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G7 | 碧香无核 Bixiangwuhe | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G32 | 红特沙 Hongtesha | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G8 | 早霞玫瑰 Zaoxiameigui | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G33 | 晨香 Chenxiang | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G9 | 里扎马特 Rizamat | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G34 | 红艳香 Hongyanxiang | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G10 | 脆红宝 Cuihongbao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G35 | 红艳无核 Hongyanwuhe | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G11 | 翠香宝 Cuixiangbao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G36 | 科玉无籽 Keyuwuzi | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G12 | 早康宝 Zaokangbao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G37 | 郑美 Zhengmei | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G13 | 丽红宝 Lihongbao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G38 | 华葡早玉 Huapuzaoyu | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G14 | 秋黑宝 Qiuheibao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G39 | 瑞都早红 Ruiduzaohong | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G15 | 无核翠宝 Wuhecuibao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G40 | 瑞都科美 Ruidukemei | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G16 | 云楚无核 Yuchuwuhe | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G41 | 赤霞珠 Cabernet Sauvignon | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G17 | 卓越黑香蜜 Zhuoyueheixiangmi | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G42 | 霞多丽 Chardonnary | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G18 | 卓越玫瑰 Zhuoyuemeigui | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G43 | 红地球 Red Globe | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G19 | 岳秀无核 Yuexiuwuhe | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G44 | 玫瑰香 Muscat Hamburg | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G20 | 华葡翠玉 Huapucuiyu | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G45 | 美人指 Manicure Finger | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G21 | 华葡紫峰 Huapuzifeng | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G46 | 香妃 Xiangfei | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G22 | 爱格丽 Ecolly | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G47 | 红宝石无核 Ruby Seedless | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | |
| G23 | 玉波一号 Yubo 1 hao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G48 | 无核白鸡心 Centennial Seedless | 2x | 欧亚种 V. vinifera | 制干 Raisin grape | |
| G24 | 玉波二号 Yubo 2 hao | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G49 | 北冰红 Beibinghong | 2x | 山欧杂种 V. amurensis-V. vinifera | 酿酒 Wine grape | |
| G25 | 玉珍香 Yuzhenxiang | 2x | 欧亚种 V. vinifera | 鲜食 Table grape | G50 | 凌丰红 Lingfenghong | 2x | 山欧杂种 V. amurensis-V. vinifera | 酿酒 Wine grape | |
| 编号 Code | 品种 Cultivar | 倍性 Ploidy | 种性 Species | 用途 Purpose | 编号 Code | 品种 Cultivar | 倍性 Ploidy | 种性 Species | 用途 Purpose | |
| G51 | 凌砧1号 Lingzhen No.1 | 2x | 山欧杂种 V. amurensis-V. vinifera | 砧木 Rootstock | G64 | 申烁 Shenshuo | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G52 | 庆丰 Qingfeng | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G65 | 申奕 Shenli | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G53 | 着色香 Zhuosexiang | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G66 | 华葡玫瑰 Huapumeigui | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G54 | 碧玉香 Biyuxiang | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G67 | 华葡黑峰 Huapuheifeng | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G55 | 郑艳无核 Zhengyanwuhe | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G68 | 瑞峰 Ruifeng | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G56 | 金手指 Gold Finger | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G69 | 瑞紫香 Ruizixiang | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G57 | 阳光玫瑰 Shine Muscat | 2x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G70 | 红蜜香 Hongmixiang | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G58 | 春香无核 Chunxiangwuhe | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G71 | 华葡黄玉 Huapuhuangyu | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G59 | 烟葡一号 Yanpu 1 hao | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G72 | 华葡瑰香 Huapuguixiang | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G60 | 润堡早夏 Ruibaozaoxia | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G73 | 巨峰 Kyoho | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G61 | 天工墨玉 Tiangongmoyu | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G74 | 巨玫瑰 Jumeigui | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G62 | 夏黑 Summer Black | 3x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G75 | 天工玉液 Tiangongyuye | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | |
| G63 | 申丽 Shenli | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape | G76 | 天工翠玉 Tiangongcuiyu | 4x | 欧美杂种 V. vinifera-V. labrusca | 鲜食 Table grape |
新窗口打开|下载CSV
1.2 DNA提取与检测
使用爱森生物科技有限公司的植物基因组DNA提取试剂盒,提取葡萄叶片DNA,采用NanoDrop 1000 spectrophotometer(Themo Scientific)紫外分光光度计检测DNA浓度和纯度(OD260/OD280=1.7—2.0),并用超纯水将浓度稀释至50 ng·μL-1,按编号装入96孔PCR板中,用于高通量扩增检测[8]。1.3 KASP标记转化和引物合成
根据前人研究结果,共选用60个SNP位点作为KASP标记的筛选库,分别来自CABEZAS等[21]的46个和LAUCOU等[22]的14个SNP位点。为保证筛选的SNP位点具有基因特异性,从Ensemblplants网站下载SNP位点前后各100 bp的葡萄DNA序列(1.4 PCR扩增与标记检测
PCR反应体系(10.14 µL)为KASP Master mix 5 μL、KASP Primer mix 0.14 μL和模板DNA(50 ng·μL -1)5 μL。PCR反应条件:第一轮94℃ 15 min;94℃ 20 s,61—55℃ 60 s,10个Touch Down循环(每个循环降低0.6℃);第二轮94℃ 20 s,55℃ 60 s,26个循环。扩增产物使用荧光微孔板检测仪检测,然后采用LGC公司开发的SNP viewer 2.0软件读取检测数据 [20]。1.5 数据统计与分析
根据SNP分型结果数据计算相关遗传多样性参数,包括MAF(次要等位基因频率)、PIC(多态信息含量)[20,25]、Heterozygosity(杂合度)、Missing rate(缺失率)等,均在Excel 2019中使用公式进行计算:
$PIC=1-\sum_{i=0}^{i} P_{i}^{2}-\sum_{i=1}^{i-1} \sum_{j-i+1}^{l} 2 P_{i}^{2} P_{j}^{2}$
式中,Pi和Pj是SNP的两个等位基因在所有被测品种中的发生频率,l是样本数量[20]。
依据SNP标记具有的二态性特点,将分型数据转化为二元编码数据,野生型(与葡萄基因组一致)表示为(1、0),突变体表示为(0、1),杂合基因型表示为(1、1),缺失位点记为(999、999)[8],然后利用PowerMarker V3.25软件的邻接算法(Neighbor- joining)计算76个葡萄品种的遗传距离[26],并用Figtree v1.4.4构建聚类图。
使用STRUCTURE 2.3.4进行贝叶斯聚类[27],并确定最佳的类群分组。设定等位变异频率特征数(遗传群体数)K=1—15,Burn-in周期为100 000,MCMC的重复次数为100 000次,采用混合模型和相关等位基因频率,对不同的K值进行15次重复运行,然后将后缀为“_f”的结果文件压缩,上传到“STRUCTURE HARVESTER”网站(http://taylor0.biology.ucla.edu/ struct_harvest/),根据EVANNO等[28]的方法计算得到∆K和似然值的对数函数lnp(D),分别针对基因库数(K)建模,确定最佳K值。
采用北京市农林科院蔬菜改良中心提供的Perl脚本计算出最优组合标记[20]。
2 结果
2.1 KASP标记转化结果
对60个SNP位点进行特异性分析,与葡萄参考基因组(Standard databases:RefSeq Representative genomens;Organism:wine grape (taxid: 29760))序列比对后,发现3个非特异性标记,最终57个SNP位点成功设计出51个KASP标记,转化率为89.47%(附表1)。Schedule 1
附表1
附表1成功设计的51个KASP引物序列
Schedule 1
| 名称 Name | 变异类型Alleles type | 引物序列 Sequence of primer (5′-3′) |
|---|---|---|
| VIT_1_729514 | A/G | F: GAAGGTGACCAAGTTCATGCTGCTGAAGGTTTCTTGAAAAAGTACTGAT F: GAAGGTCGGAGTCAACGGATTCTGAAGGTTTCTTGAAAAAGTACTGAC R: CACAGGTTGGACAAGCAAAGGGAATT |
| VIT_1_5948674 | A/G | F: GAAGGTGACCAAGTTCATGCTATGGTTGAGGAAAGAATATTGGAAGCT F: GAAGGTCGGAGTCAACGGATTGGTTGAGGAAAGAATATTGGAAGCC R: CCGCTAGTCTTTCTAACCATGAGCTA |
| VIT_1_22828604 | A/T | F: GAAGGTGACCAAGTTCATGCTCTACTATTATCTCTCTCGCTTATTGCTA F: GAAGGTCGGAGTCAACGGATTCTACTATTATCTCTCTCGCTTATTGCTT R: GGCAAATCCCAAAGAACTATGAATGGAA |
| VIT_2_5141894 | A/G | F: GAAGGTGACCAAGTTCATGCTTGCGGCAAGAAGGTCCCAACT F: GAAGGTCGGAGTCAACGGATTGCGGCAAGAAGGTCCCAACC R: GTAGTGAGAACTAGAGCGGAGAACTAT |
| VIT_2_6474327 | T/A | F: GAAGGTGACCAAGTTCATGCTCTCACCATTAATTGGGGTGAAGATTAA F: GAAGGTCGGAGTCAACGGATTCTCACCATTAATTGGGGTGAAGATTAT R: AAAGTAATCTCCATTTTGGTGTGATAACCTT |
| VIT_2_17198115 | G/C | F: GAAGGTGACCAAGTTCATGCTTCCTCCGCTTCTCCGCCC F: GAAGGTCGGAGTCAACGGATTTCCTCCGCTTCTCCGCCG R: AAAATCTTGCGGGCGTAGTCGAGAT |
| VIT_3_1348328 | C/T | F: GAAGGTGACCAAGTTCATGCTGATAAGACGGAGTGGCTGGAC F: GAAGGTCGGAGTCAACGGATTGGATAAGACGGAGTGGCTGGAT R: CCTGGCCGAACACCACGTGCTT |
| VIT_3_3676120 | A/C | F: GAAGGTGACCAAGTTCATGCTAATTTAGCCAATTTCTATCTTTTGCCGTTT F: GAAGGTCGGAGTCAACGGATTTTTAGCCAATTTCTATCTTTTGCCGTTG R: ATCTACAAGGCAGGAAGGAAAGAGTTAT |
| VIT_3_5724485 | T/C | F: GAAGGTGACCAAGTTCATGCTCCCTTTTTGGTGTAACATCCTTCCA F: GAAGGTCGGAGTCAACGGATTCCTTTTTGGTGTAACATCCTTCCG R: GATAGGAATAGCCTCGTAGAATTTTATGTAAAA |
| VIT_4_680574 | C/T | F: GAAGGTGACCAAGTTCATGCTGGAGCTTTGATCCTGAAACCGAG F: GAAGGTCGGAGTCAACGGATTTAGGAGCTTTGATCCTGAAACCGAA R: GCGTGCAGCTATCTCTTGAAGCAAAAA |
| VIT_4_6409234 | C/T | F: GAAGGTGACCAAGTTCATGCTATTATCCCAAAGGGAAAATAAAAACTTTCC F: GAAGGTCGGAGTCAACGGATTGATTATCCCAAAGGGAAAATAAAAACTTTCT R: GTTCTCACAGGACAAAGCATCTTGGTT |
| VIT_4_21849155 | A/G | F: GAAGGTGACCAAGTTCATGCTCACTGGTAATAGATAGAATTAAACACATCT F: GAAGGTCGGAGTCAACGGATTACTGGTAATAGATAGAATTAAACACATCC R: CTTCCTAGGTGGAAGCAGCTGTATTAA |
| VIT_5_1785979 | C/T | F: GAAGGTGACCAAGTTCATGCTGGTCCCTCACCTATTACTCCAG F: GAAGGTCGGAGTCAACGGATTCGGTCCCTCACCTATTACTCCAA R: TTTCGGACATGGAAAGCTTGAGTTTCTT |
| VIT_5_5773320 | C/T | F: GAAGGTGACCAAGTTCATGCTGGCTGTATCCTGTCTTGAAGCTC F: GAAGGTCGGAGTCAACGGATTTGGCTGTATCCTGTCTTGAAGCTT R: GATCCAAGGTCACGAGCTGTTTATGAT |
| VIT_5_6744629 | A/G | F: GAAGGTGACCAAGTTCATGCTTGAGTCAATCTCGCCGAATATGAGT F: GAAGGTCGGAGTCAACGGATTAGTCAATCTCGCCGAATATGAGC R: AAACTCAAGATTGGACAGCAATATCCATATT |
| VIT_6_327200 | A/G | F: GAAGGTGACCAAGTTCATGCTAAAAGCCTTTGGAGCATCTCCAGAA F: GAAGGTCGGAGTCAACGGATTAGCCTTTGGAGCATCTCCAGAG R: GAAGGTTTTTGAAAATGGATCAGTTGCCAA |
| VIT_6_4258638 | C/T | F: GAAGGTGACCAAGTTCATGCTTGTCCAGAGATCCTGTTTTCTCG F: GAAGGTCGGAGTCAACGGATTACTGTCCAGAGATCCTGTTTTCTCA R: CTTTCAGCAGGCAGCAATGGAAAGTT |
| VIT_6_17593092 | A/T | F: GAAGGTGACCAAGTTCATGCTCTGCATTCGTTCACCTGTCAACTTA F: GAAGGTCGGAGTCAACGGATTCTGCATTCGTTCACCTGTCAACTTT R: ATCTGCATCTTCGAGCTTGTCCTTAATT |
| VIT_7_1388822 | A/G | F: GAAGGTGACCAAGTTCATGCTCGTCCGGATGCATTGCGCCT F: GAAGGTCGGAGTCAACGGATTGTCCGGATGCATTGCGCCC R: GGCAAACGCTGATTGGCTGGAGTA |
| VIT_7_18046355 | A/G | F: GAAGGTGACCAAGTTCATGCTAAATGTGGCTGCAGTTGAGAAGACT F: GAAGGTCGGAGTCAACGGATTTGTGGCTGCAGTTGAGAAGACC R: CATACCACAGGATTAACTGATACATCCATA |
| VIT_8_3320936 | C/T | F: GAAGGTGACCAAGTTCATGCTTGGAGGGTAAAAATGAACTCAATTTGAC F: GAAGGTCGGAGTCAACGGATTATTGGAGGGTAAAAATGAACTCAATTTGAT R: CCTTTCTGATGATAGAAGCAGTGGGAA |
| VIT_8_13401437 | A/C | F: GAAGGTGACCAAGTTCATGCTCTGGTTCCAATCCCTCATCCGTA F: GAAGGTCGGAGTCAACGGATTTGGTTCCAATCCCTCATCCGTC R: AAGGAGACTAAGATAAAGGTGTAGTATACAT |
| VIT_9_3123999 | A/T | F: GAAGGTGACCAAGTTCATGCTTGTCTTGACTCATCATATTTGACAGCA F: GAAGGTCGGAGTCAACGGATTTGTCTTGACTCATCATATTTGACAGCT R: GGTGTGAAGACGATAATGGGTCCAAT |
| VIT_9_21409416 | C/T | F: GAAGGTGACCAAGTTCATGCTTCCAGAAACCAGCTAGTGTGGC F: GAAGGTCGGAGTCAACGGATTATTCCAGAAACCAGCTAGTGTGGT R: CGGAAAACTTCCATACCGCGTGAAA |
| VIT_10_5489212 | C/T | F: GAAGGTGACCAAGTTCATGCTTGCTTCTCCACGAAGGCTGTC F: GAAGGTCGGAGTCAACGGATTCTGCTTCTCCACGAAGGCTGTT R: GCAACGTCTCCTACGACACGGAA |
| VIT_11_311765 | A/C | F: GAAGGTGACCAAGTTCATGCTTGGGGGAAGTAGTTGGTTGCCA F: GAAGGTCGGAGTCAACGGATTGGGGGAAGTAGTTGGTTGCCC R: GAATTAGAGACCCAGAGAGAAGGGAAA |
| VIT_11_5406647 | A/C | F: GAAGGTGACCAAGTTCATGCTTATCAGAATGTTGGATTTTGAAATTGAATGTTA F: GAAGGTCGGAGTCAACGGATTTCAGAATGTTGGATTTTGAAATTGAATGTTC R: TGTTACGGTTTTGCTAACTCAAAATCCTTAT |
| VIT_11_19390306 | G/A | F: GAAGGTGACCAAGTTCATGCTGTTCAAGCTGGGGGAGAATATATAC F: GAAGGTCGGAGTCAACGGATTGGTTCAAGCTGGGGGAGAATATATAT R: GTCGGAGTCCATGTATCGCCGTTA |
| VIT_12_739916 | C/T | F: GAAGGTGACCAAGTTCATGCTGCTGAACACACTTTTTCCAAGTTCG F: GAAGGTCGGAGTCAACGGATTAAGCTGAACACACTTTTTCCAAGTTCA R: GGTTTGGGAGGGGACAAAGATCTAATT |
| VIT_12_22228357 | A/C | F: GAAGGTGACCAAGTTCATGCTCAACTTCACGGTGACCGAATTCATA F: GAAGGTCGGAGTCAACGGATTAACTTCACGGTGACCGAATTCATC R: GGTCGACGGCAGCCACAGGTTT |
| VIT_13_21618145 | A/T | F: GAAGGTGACCAAGTTCATGCTTCGAGTTGCTTAGATAAACACATTAATCT F: GAAGGTCGGAGTCAACGGATTTCGAGTTGCTTAGATAAACACATTAATCA R: CATCATGTGATTCTATGCAAAAATTATTAGAATTAA |
| VIT_14_4947068 | T/G | F: GAAGGTGACCAAGTTCATGCTTATTCCTACCTGCGTTTCGTCGA F: GAAGGTCGGAGTCAACGGATTATTCCTACCTGCGTTTCGTCGC R: CTGTGGCCCGGTGGGGCTT |
| VIT_14_5687725 | A/T | F: GAAGGTGACCAAGTTCATGCTATGATGCTTTGAATAGAATAAATGAGAACAAT F: GAAGGTCGGAGTCAACGGATTATGATGCTTTGAATAGAATAAATGAGAACAAA R: AACTTGCAACATGACAACCACAAACCAA |
| VIT_14_23135445 | A/T | F: GAAGGTGACCAAGTTCATGCTAATTAGCAGGGAGGGGTGGCAA F: GAAGGTCGGAGTCAACGGATTAATTAGCAGGGAGGGGTGGCAT R: GAACTTTAGCAGTAGTTCAGCTTCATGAT |
| VIT_14_29590769 | C/T | F: GAAGGTGACCAAGTTCATGCTTTCCTCCAAGTCTCCATGTGCG F: GAAGGTCGGAGTCAACGGATTGTTCCTCCAAGTCTCCATGTGCA R: CAAAGCCCTTTGACAGACAACCTTCAA |
| VIT_15_15145042 | A/C | F: GAAGGTGACCAAGTTCATGCTGTATATTTGTTCATCAGACATACTAACACT F: GAAGGTCGGAGTCAACGGATTTATATTTGTTCATCAGACATACTAACACG R: GCAGAGAGAAGCTCAGACATCTCATTA |
| VIT_15_18031506 | A/C | F: GAAGGTGACCAAGTTCATGCTTAACTGATACATAAAACTGAAAAATCGGGT F: GAAGGTCGGAGTCAACGGATTACTGATACATAAAACTGAAAAATCGGGG R: GGGAGTAGAACCTGAAAGCAAGCATA |
| VIT_15_18127737 | T/G | F: GAAGGTGACCAAGTTCATGCTGGATGTGATGTAGGGGTCCTTGT F: GAAGGTCGGAGTCAACGGATTGATGTGATGTAGGGGTCCTTGG R: CATATCACCTTTTCTAGGACTAAAACCCAA |
| VIT_15_18567587 | T/G | F: GAAGGTGACCAAGTTCATGCTCTGTATTAAAGCTCTTGGGTGAAAAACT F: GAAGGTCGGAGTCAACGGATTTGTATTAAAGCTCTTGGGTGAAAAACG R: ATTGTTGGGCACAAATACGCTCAAGATT |
| VIT_16_13454358 | A/G | F: GAAGGTGACCAAGTTCATGCTGCTTTTGACGTGAGAGAGCAAGAATT F: GAAGGTCGGAGTCAACGGATTCTTTTGACGTGAGAGAGCAAGAATC R: CTGGTGCATATAAGGTTGCAGTTGTTAA |
| VIT_16_16198599 | A/G | F: GAAGGTGACCAAGTTCATGCTGCACAACAATTTCTCCAGCTTCGTT F: GAAGGTCGGAGTCAACGGATTCACAACAATTTCTCCAGCTTCGTC R: AGAAATGGACTCACGTAGGTGTTAAGTT |
| VIT_16_17950801 | A/G | F: GAAGGTGACCAAGTTCATGCTTCTGCTTGAGGGGGAGTGACAA F: GAAGGTCGGAGTCAACGGATTCTGCTTGAGGGGGAGTGACAG R: TAGTGGAAACATCTCCCACTTGATTCATA |
| VIT_16_21202286 | A/G | F: GAAGGTGACCAAGTTCATGCTACCCAATGGAATCGATCCAATGTCA F: GAAGGTCGGAGTCAACGGATTCCAATGGAATCGATCCAATGTCG R: CTGGTTAATTTCAGACGAAGCCAAGTATA |
| VIT_17_126505 | A/G | F: GAAGGTGACCAAGTTCATGCTTCAGTATTATGAGGAATGCTATTTATGAGT F: GAAGGTCGGAGTCAACGGATTCAGTATTATGAGGAATGCTATTTATGAGC R: GGTCCTGCAAGATATCCCAGAACATAT |
| VIT_17_6000914 | C/T | F: GAAGGTGACCAAGTTCATGCTAACTGTTCCCAGCCGGATTGAG F: GAAGGTCGGAGTCAACGGATTGAACTGTTCCCAGCCGGATTGAA R: GCCGACCCCATCCTGTCACTAT |
| VIT_17_12206201 | A/G | F: GAAGGTGACCAAGTTCATGCTCGCCATCTTATGACAATTGATGAACTA F: GAAGGTCGGAGTCAACGGATTGCCATCTTATGACAATTGATGAACTG R: CCTTTTAGTGACAGGTGGAATAAGAGAAAT |
| VIT_18_3829207 | A/C | F: GAAGGTGACCAAGTTCATGCTTGCAAACTGGGAAGCAAATGTCTCT F: GAAGGTCGGAGTCAACGGATTCAAACTGGGAAGCAAATGTCTCG R: CAAGGATATTGTTGAGCTCCGAAATCAA |
| VIT_18_6487636 | A/C | F: GAAGGTGACCAAGTTCATGCTAGATTGAATATCTCCATACCTTCGTCA F: GAAGGTCGGAGTCAACGGATTGATTGAATATCTCCATACCTTCGTCC R: GGGATAAAACATTCAAAGCCCATAAATTAAATTT |
| VIT_18_11138668 | A/G | F: GAAGGTGACCAAGTTCATGCTGACATGTTTCCATACTGATCCTCCTA F: GAAGGTCGGAGTCAACGGATTACATGTTTCCATACTGATCCTCCTG R: CTGGACATTTTCAACCATGATGATGATGAT |
| VIT_19_7217380 | A/T | F: GAAGGTGACCAAGTTCATGCTGGTCTTCATCCAAGATATGTATCTCATT F: GAAGGTCGGAGTCAACGGATTGGTCTTCATCCAAGATATGTATCTCATA R: CATACGATTAAATGGATGCGGGCAGTT |
| VIT_19_17751334 | G/T | F: GAAGGTGACCAAGTTCATGCTCATGAGGCGTTTTTCATCACCAAC F: GAAGGTCGGAGTCAACGGATTGCATGAGGCGTTTTTCATCACCAAA R: GTTTGGCTCCAGACTTGCCTGAATTT |
新窗口打开|下载CSV
2.2 KASP标记的初筛
利用KASP技术对23份葡萄品种进行基因分型,51个KASP标记全部成功分型。其中,MAF值大于0.25的标记有31个,PIC值大于0.35的标记有35个,缺失率小于0.2的标记有44个,杂合率小于0.6的标记有50个,同时满足以上4个条件的标记有27个,并用于下一步的复筛(图1)。图1
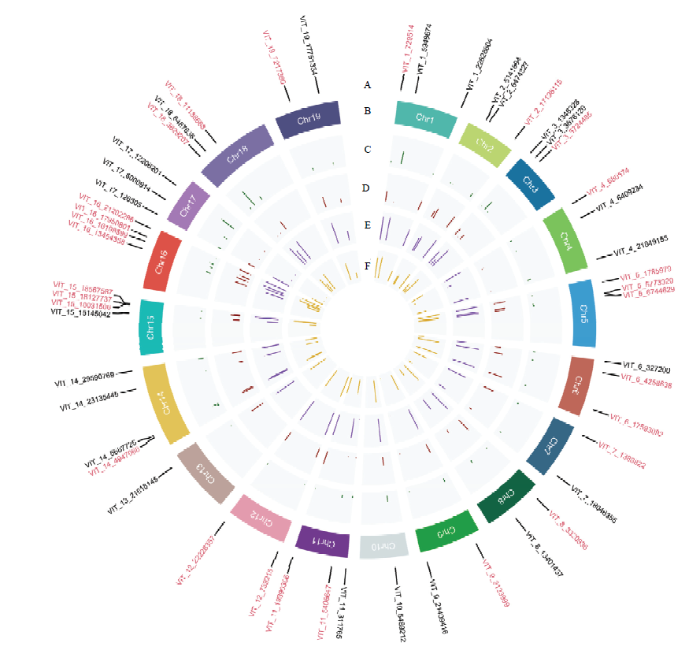 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1基于23份代表性葡萄品种对51个KASP标记进行检测获得的遗传信息
A:51个KASP标记的名称(红色字体为初筛出的27个高质量KASP标记);B:葡萄染色体号;C:缺失率;D:杂合率;E:多态性信息含量;F:次要等位基因频率
Fig. 1The genetic information of 51 KASP markers detected on 23 representative grape cultivars
A: 51 KASP markers names (The 27 high-quality KASP markers marked in red); B: Grape chromosome number; C: missing rate; D: Heterozygosity rate; E: polymorphism information content (PIC); F: minor allele frequency (MAF)
2.3 KASP标记的复筛及DNA指纹图谱构建
使用初筛出的27个KASP标记对76份葡萄品种进行基因分型。结果表明,VIT_3_5724485、VIT_4_680574、VIT_11_5406647、VIT_15_18031506、VIT_18_3829207等5个标记分型结果不具备差异性,并舍弃;其余22个KASP标记在76份样本中均分型成功(图2)。其中,22个标记的缺失率均小于0.12,杂合率在0.4—0.6的占77%,PIC值在0.4—0.5的占95%,MAF值大于0.3的占95%(图3)。此外,使用筛出的22个KASP标记对同一品种不同树体提取的23份代表性品种的DNA扩增检测,前后两次检测的分型结果一致,表明筛选的22个KASP标记具有较好的重复性和稳定性,可以初步作为一组核心标记用于葡萄的品种鉴定。将这22个标记获得的76份葡萄品种分型结果转化为二元编码数据,得到76份中国主要栽培葡萄品种的SNP指纹图谱(图4)。图2
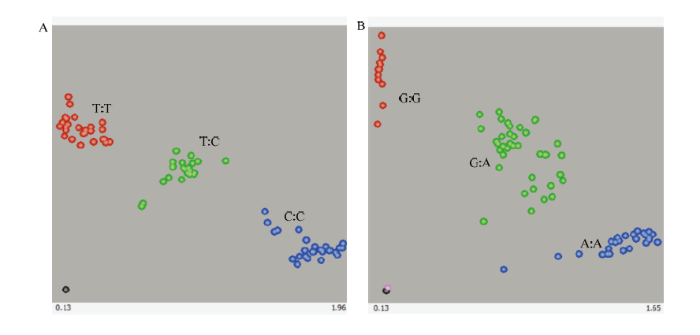 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基于76份葡萄品种,VIT_6_4258638(A)和VIT_16_21202286(B)KASP标记荧光检测结果
Fig. 2VIT_16_21202286 (A) and VIT_16_21202286 (B) KASP labeled fluorescence detection results based on 76 grape cultivars
图3
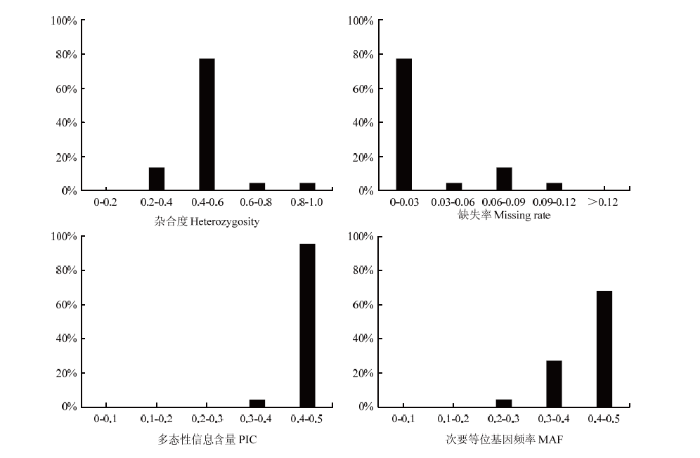 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图322个KASP标记基于76份葡萄品种获得的缺失率、杂合度、多态性信息含量、次要等位基因频率值在不同范围内所占百分比
Fig. 3The percentage of missing rate, heterozygosity, PIC and MAF values for the 22 KASP markers based on the data of 76 grape cultivars
图4
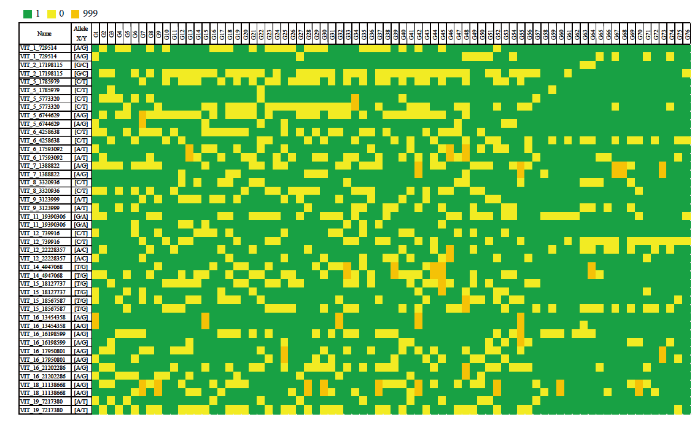 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图476份中国主要栽培葡萄品种的KASP指纹图谱数据库
第1列:KASP标记名称;第2列:KASP标记对应的突变碱基类型;其余列:依次为76个主要栽培葡萄品种的基因型
Fig. 4KASP finger-printing database of 76 main cultivated grape cultivars in China
The first column: KASP marker name; the second column: KASP marker corresponding mutation base type; the remaining columns: in turn are the genotypes of 76 main cultivated grape cultivars
2.4 76个葡萄品种的聚类分析和群体结构分析
基于22个KASP标记的分型结果,利用PowerMarker V3.25和Figtree v1.4.4软件绘制76份葡萄品种的聚类图(图5-A)。76份葡萄品种可以分为3大类:第Ⅰ大类由19份多倍体欧美杂交品种单独聚为一大类(图5-A中标红色的分枝),其中‘春香无核’‘润堡早夏’‘天工墨玉’同为‘夏黑’的芽变品种,和‘夏黑’聚在一起;亲本之一同为‘红富士’的‘天工翠玉’‘天工玉液’2份品种也在该大类中,但因另一亲本不同,没有紧密聚在一起;‘巨玫瑰’‘华葡黄玉’‘华葡瑰香’同为‘巨峰’和‘沈阳玫瑰’的杂交后代,和‘巨峰’聚在一起;其他含有‘巨峰’亲缘的多倍体品种也与‘巨峰’聚在一起。第Ⅱ大类由39份二倍体品种组成,包含35份欧亚种、3份欧美杂种、1份毛葡萄(图5-A中标绿色的分枝),其中‘早康宝’和‘丽红宝’、‘翠香宝’和‘秋黑宝’、‘郑艳无核’和‘庆丰’、‘云楚无核’和‘岳秀无核’、‘瑞都红玫’‘瑞都脆霞’‘瑞都香玉’‘瑞都早红’5组分别出自同一杂交组合后代群体的品种,各自聚类在一起;具有‘玫瑰香’亲缘关系的‘脆红宝’‘晨香’‘卓越玫瑰’‘华葡翠玉’‘华葡早玉’5份品种与‘玫瑰香’聚类在一起;同样,具有‘红地球’亲缘关系的‘玉波一号’‘玉波黄地球’‘云楚无核’‘岳秀无核’‘华葡翠玉’‘华葡紫峰’‘红艳无核’7份品种也与‘红地球’聚类在一起;‘红特沙’与亲本之一‘里扎马特’紧密聚在一起。第Ⅲ大类主要由11份欧亚种、3份欧美杂种、3份山欧杂种、1份刺葡萄,共18份品种组成,同为二倍体品种(图5-A中标蓝色的分枝),其中‘赤霞珠’‘北冰红’‘爱格丽’‘媚丽’‘泰美’‘玉玲珑’6份酿酒葡萄品种聚在该大类中;‘北冰红’‘凌丰红’2份出自同一育种单位的山欧杂交品种,和‘惠良刺葡萄’紧密聚类在一起;‘玉波二号’‘短枝玉玫瑰’同为‘达米娜’和‘紫地球’的杂交后代,紧密聚在一起;由‘金手指’和‘紫地球’杂交获得的‘玉珍香’也与‘金手指’聚在该类群中。图5
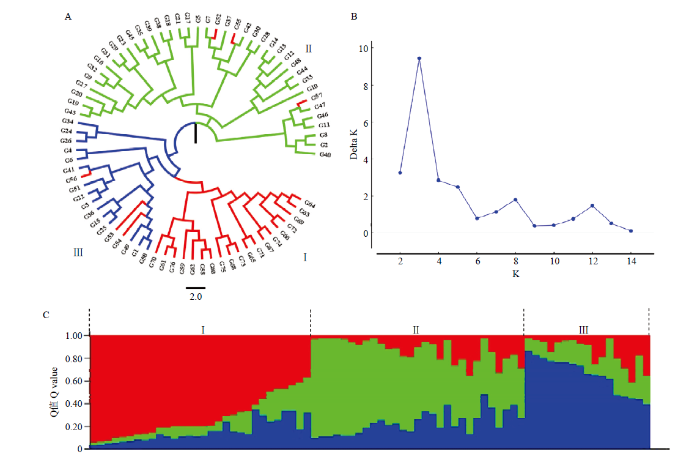 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图576份中国主要栽培葡萄品种的聚类图和群体结构图
A:邻接聚类分析;B:K值与∆K趋势变化图;C:K=3时群体遗传结构分析图
Fig. 5Cluster and population structure diagram of 76 main cultivated grape cultivars in China
A: Neighbor-joining (N-J) tree cluster analysis; B: The distribution of K-values with ∆K; C: Population structure analysis when K=3
用STRUCTURE 2.3.4进行种群遗传结构相关性分析,根据EVANNO等[28]的方法,用最大似然值∆K来确定K值。本研究中当K=3时,∆K取得最大值,推断76份葡萄品种分为3个类群(图5-B、图5-C),Ⅰ、Ⅱ、Ⅲ类分别占总群体数的39.47%、38.16%、22.37%,其中Ⅰ类群包含18个欧美杂种品种,占总欧美杂种群体数(25份)的72.00%,Ⅱ和Ⅲ群体中共包含36份欧亚种品种,占总欧亚种群数(46份)的78.26%,与聚类分析结果具有一定的一致性。
2.5 KASP标记在品种鉴定中的应用
对76份葡萄品种的指纹数据比对分析,发现3组基因型一致的品种分别为:‘夏黑’和‘润堡早夏’(‘夏黑’的芽变品种),‘天工翠玉’和‘天工墨玉’(同一家育种单位选育的品种),‘里扎马特’和‘红特沙’(亲本之一为‘里扎马特’)。采用北京市农林科学院蔬菜改良中心提供的Perl脚本计算出一组仅用10个标记就能区分70份品种的最优组合标记(图6),有54份葡萄品种达到品种鉴定的标准(差异位点数≥2)[29]。其中,中国主栽的‘红地球’‘玫瑰香’‘美人指’‘香妃’‘红宝石无核’‘碧香无核’‘金手指’‘阳光玫瑰’‘赤霞珠’9份葡萄品种含有至少2个KASP标记的基因型可区别于其他品种。新育成的品种中,‘脆红宝’‘翠香宝’‘早康宝’‘丽红宝’‘秋黑宝’‘无核翠宝’6份“宝”系列品种,‘瑞都无核怡’‘瑞都科美’‘瑞都脆霞’‘瑞都香玉’‘瑞都早红’‘瑞都红玫’6份“瑞都”系列品种,‘玉波一号’‘玉波二号’‘玉珍香’‘短枝玉玫瑰’‘玉波黄地球’5份“玉”系列品种,‘华葡紫峰’‘华葡翠玉’‘华葡早玉’‘华葡黄玉’‘华葡玫瑰’5份“华葡”系列品种,同一亲本后代的‘云楚无核’‘岳秀无核’及‘庆丰’‘郑艳无核’、单亲后代的‘MCS2’‘泰美’、山欧杂种的‘北冰红’‘凌丰红’‘凌砧1号’、刺葡萄种的‘惠良刺葡萄’以及多倍体‘天工玉液’‘烟葡一号’等品种间均有不少于2个KASP标记的差异。图6
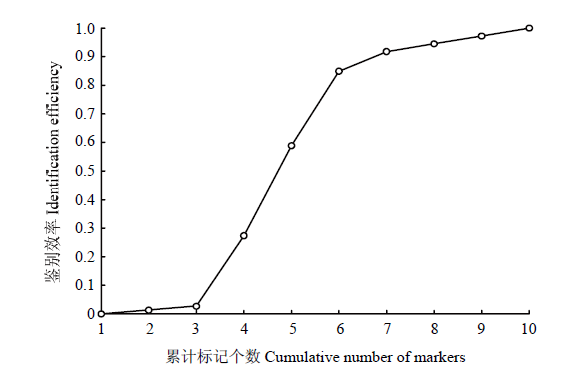 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6区分70份葡萄品种的KASP分子累计标记效率
1: VIT_15_18567587; 2: VIT_6_4258638; 3: VIT_8_3320936; 4: VIT_12_ 22228357; 5: VIT_11_19390306; 6: VIT_16_17950801; 7: VIT_18_ 11138668; 8: VIT_12_739916; 9: VIT_16_13454358; 10: VIT_16_21202286
Fig. 6Cumulative KASP molecular marker efficiency for distinguishing 70 grape cultivars
3 讨论
中国作为东亚种群的集中分布区,包含至少38个种[1],保存各类葡萄种质资源达3 000多份[30],且中国新育成的葡萄品种数量还以每年至少20份的速度增长[31]。但由于人们频繁的引种交流和长期的葡萄传播过程,同物异名或同名异物的现象时有发生[2],如云南‘水晶’、贵州‘水晶’、关口葡萄实为尼加拉的同物异名品种,盐井‘黑珍珠’和茨中‘教堂’葡萄实为‘巴柯’的同物异名品种[32]。此外,葡萄无性繁殖能力强,成活率高,育种家的成果一旦被窃取,极易被扩繁、易名、炒作,不仅侵害育种家的利益,而且加大了葡萄苗木市场的管理难度。构建DNA指纹图谱技术(DNA-Fingerprinting)对于快速准确的品种鉴定和纯度鉴定、维护育种家的利益等具有重大意义[8]。当前,研究者已在水稻[29]、玉米[17]、棉花[33]、甘蓝[34]等多种作物中进行了以SSR和SNP核心标记为主的指纹图谱构建。在葡萄作物上,李贝贝等[6]利用8对引物构建国内最大群体量的SSR指纹图谱。但SSR标记在实际应用中还存在数量和检测通量有限、位点存在一定突变率、数据准确读取费时耗力、无法有效区分芽变品种等不足[15]。与SSR相比,SNP是基于单核苷酸的突变,突变频率更低,遗传稳定性更高,批量检测时平均每个孔位的使用成本比SSR的低1/10—1/20;同时,SNP也是构建DNA指纹数据库的推荐标记之一,但目前国内还没有利用SNP标记构建葡萄指纹图谱的研究[35]。而构建SNP指纹图谱首先要选择合适的SNP分型技术。目前,至少有20种SNP分型技术,其中SEMAGN等[14]以玉米为材料,从转化率、准确性、成本、灵活性等角度对比分析了KASP和Illumina GoldenGate两种技术,认为KASP基因分型技术在少位点检测大样本群体时具有更大的优势。赵勇等[36]基于53份大豆材料,使用4个SNP比较GBS、CAPS、KASP三种分型方法,也认为KASP技术更具有灵活实用性。刘丽华等[9]同样认为KASP技术可实现高通量的少量位点检测大量群体的目的,更适合作物品种鉴定工作。马丽等[17]、李志远等[8]、刘宝平等[37]和LI等[38]基于KASP技术分别筛选出22个、59个、48个和50个核心KASP标记,用于玉米、甘蓝、番茄和白菜等作物的品种鉴定中。
本研究基于KASP技术,利用前人筛选的SNP位点,首次尝试构建我国76份主要栽培葡萄品种的SNP指纹图谱。基于KASP分型技术的检测原理,选择的SNP位点需要在群体中稳定遗传、具备高多态性。同时,为保证分型结果准确,筛选的标记应具有基因组特异性。本研究使用的SNP位点分别来自CABEZAS等[21]对1 324份葡萄材料有效分型验证的46个SNPs和LAUCOU等[22]基于18K SNPs葡萄基因芯片对945份材料筛出的14个SNPs,保证了选择的位点具有足够的区分能力。
为保证构建的76份葡萄指纹图谱的真实性,本研究基于检测获得的基因型数据进行聚类分析和群体分析,两种分析结果一致。聚类分析结果表明所有的多倍体欧美杂种起源于二倍体,但又区别于二倍体,在聚类图中单独聚为一类。其他二倍体品种分为两大枝,符合对应的亲缘关系,与王富强等[5]基于30对核心SSR引物对52份葡萄品种聚类分析结果具有一致性,首次验证了KASP分子标记可用于研究多倍体和二倍体葡萄之间的遗传进化关系,使用KASP标记构建葡萄指纹图谱具有可行性。
在品种鉴定分析中,因‘春香无核’‘润堡早夏’‘天工墨玉’同为‘夏黑’的芽变品种,以及亲本之一为‘里扎马特’的‘红特沙’,遗传相似度极高,使用22个KASP标记无法有效区分。只有‘无核白鸡心’的芽变品种‘科玉无籽’与‘无核白鸡心’含有6个纯合基因型的差异,说明芽变品种的鉴定依然是当前品种鉴定的难点,需要用更多的标记进行区分。而按照SSR分子标记的品种有效区分标准,本研究获得的10个标记(VIT_15_18567587、VIT_6_4258638、VIT_8_3320936、VIT_12_22228357、VIT_11_19390306、VIT_16_17950801、VIT_18_11138668、VIT_12_739916、VIT_16_13454358、VIT_16_21202286)可完全区分54份品种,包含了我国主栽的‘红地球’‘红宝石无核’‘金手指’‘阳光玫瑰’等品种,以及亲缘关系较近的“宝”系列、“瑞都”系列、“玉”系列、“华葡”系列品种,这与使用SSR标记区分相关品种具有一致性[5],表明使用KASP标记对葡萄进行品种鉴定的实用性,且比SSR标记具有更快更准、更易判读等优势。
4 结论
从60个葡萄SNP位点,成功转化51个KASP标记,并筛选出22个高质量KASP标记,构建了76份中国主要栽培葡萄品种的SNP指纹图谱。经邻接聚类分析和群体结果分析,可将76份葡萄品种分为3类,并能正确区分开二倍体和多倍体。仅用10个标记就能区分70份品种,其中有54份品种达到品种鉴定的标准(差异位点数≥2),首次验证了KASP技术在我国葡萄品种鉴定中的可行性。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 2]
DOI:10.21273/HORTSCI11802-17URL [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 3]
[本文引用: 2]
[本文引用: 2]
DOI:10.1016/j.molp.2017.06.008URL [本文引用: 1]
[本文引用: 5]
[本文引用: 5]
[本文引用: 3]
[本文引用: 3]
DOI:10.1186/s12284-019-0272-3URL [本文引用: 2]
DOI:10.1111/pbi.v18.6URL [本文引用: 1]
DOI:10.1093/mp/sst135URL [本文引用: 1]
DOI:10.1007/s11032-016-0586-4URL [本文引用: 1]
DOI:10.1007/s11032-013-9917-xURL [本文引用: 2]
DOI:10.1007/s00122-016-2743-xURL [本文引用: 2]
DOI:10.1111/pbi.v18.3URL [本文引用: 1]
ZL201910623802.0.
[本文引用: 3]
ZL201910623802.0
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/s41598-019-57145-9URL [本文引用: 5]
DOI:10.1186/1471-2229-11-153URL [本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/s41467-018-07983-4URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1093/genetics/164.4.1567URL [本文引用: 1]
DOI:10.1111/mec.2005.14.issue-8URL [本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
ZL201910916095.4.
[本文引用: 1]
ZL201910916095.4
[本文引用: 1]
chinensis Makino.
DOI:10.1111/pbr.2019.138.issue-3URL [本文引用: 1]
