 ,1, 朱行
,1, 朱行 ,1, 魏宁1, 龙凤1, 武建颖1, 张燕1, 董金皋2,3, 申珅
,1, 魏宁1, 龙凤1, 武建颖1, 张燕1, 董金皋2,3, 申珅 ,1, 郝志敏
,1, 郝志敏 ,1,2
,1,2The Expression Pattern and Interaction Analysis of the Homologues of Splicing Factor SC35 in Setosphaeria turcica
LI TianCong ,1, ZHU Hang
,1, ZHU Hang ,1, WEI Ning1, LONG Feng1, WU JianYing1, ZHANG Yan1, DONG JinGao2,3, SHEN Shen
,1, WEI Ning1, LONG Feng1, WU JianYing1, ZHANG Yan1, DONG JinGao2,3, SHEN Shen ,1, HAO ZhiMin
,1, HAO ZhiMin ,1,2
,1,2通讯作者:
责任编辑: 岳梅
收稿日期:2020-05-12接受日期:2020-05-28网络出版日期:2021-02-16
| 基金资助: |
Received:2020-05-12Accepted:2020-05-28Online:2021-02-16
作者简介 About authors
李天聪,E-mail:
朱行,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (953KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
李天聪, 朱行, 魏宁, 龙凤, 武建颖, 张燕, 董金皋, 申珅, 郝志敏. 玉米大斑病菌SC35同源基因表达规律与互作分析[J]. 中国农业科学, 2021, 54(4): 733-743 doi:10.3864/j.issn.0578-1752.2021.04.006
LI TianCong, ZHU Hang, WEI Ning, LONG Feng, WU JianYing, ZHANG Yan, DONG JinGao, SHEN Shen, HAO ZhiMin.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】玉米大斑病菌(Setosphaeria turcica)是一种侵染玉米叶片的丝状真菌。目前对玉米大斑病的防治主要以抗病品种利用为主,由于致病菌突变频率高、生理分化明显,给大斑病防治带来较大难度[1,2]。前体mRNA的可变剪接(alternative splicing)可通过不同调控元件与剪接因子相互作用之后产生不同的剪切、拼接结果,是真核生物中一种比较灵活的基因表达调控方式[3]。明确玉米大斑病菌致病过程中剪接因子所发挥的调节作用,将为寻找病害防控新策略提供思路。【前人研究进展】1977年,ROBERTS和SHARP首次发现基因是分裂而不连续的[4]。20世纪80年代初,MAKI等发现在真核生物小鼠编码免疫球蛋白重链的基因中存在可变剪接现象[5]。在对 RNA剪接的研究过程中,人们还发现了一类起重要作用的非snRNP剪切因子——SR蛋白[6]。随后,可变剪接现象在植物和动物中被广泛发现,但在病原真菌中鲜有报道。随着生物信息技术的发展,使得在植物病原真菌基因组和转录组范围内研究可变剪接成为可能。现在公认的SR蛋白有12种,包括SRSF1(ASF/SF2)、SRSF2(SC35)、SRSF3(SRp20)、SRSF4(SRp75)、SRSF5(SRp40)、SRSF6(SRp55)、SRSF7(9G8)、SRSF2B(SRp46)、SRSF9(SRp30c)、SRSF13A(SRp38)、SRSF11(SRp54)、SRSF13B(SRrp35)[7]。剪切因子SC35最初是FU等在哺乳动物细胞核中发现的一种非snRNP类蛋白剪切组分,分子量为35 kD,其N端主要由具有结合RNA能力的RNA识别结构域组成(RNA-recognition motif,RRM);其C端含有大量的SR/RS(S-丝氨酸、R-精氨酸)二肽重复序列,即RS结构域[8]。关于SC35在动物细胞的分布已得到了广泛和深入的研究,体外实验表明,其在剪切位点的选择及剪切复合体的组装上起至关重要的作用,并在转录延伸过程中也发挥着重要作用[9,10,11]。与正常组织相比,头颈癌细胞中SFRS2(SC35)表达水平上调,促进了肿瘤抑制基因——钙黏附蛋白E(E-cadherin)前体mRNA第11个外显子跳跃,从而造成E-cadherin前体mRNA错误剪接[12]。在乳腺癌细胞中,过表达SC35有助于细胞膜糖蛋白CD44 C5-V6-C6亚型的产生,进而促进癌细胞的侵染和转移[13]。YAN等对拟南芥(Arabidopsis thaliana)SC35编码基因进行了敲除,发现突变体的形态和生长过程均受到影响,包括出现锯齿状叶片、延迟开花、短根和异常的叶序;进行序列分析发现,SC35蛋白能够结合在RNA序列的特定位点AGAAGA上,此外,敲除SC35也影响着基因的转录,且SC35与RNA聚合酶Ⅱ的特定亚族NRPB4有相互作用[14]。目前,有关丝状真菌中SC35的研究报道极少,仅在构巢曲霉(Aspergillus nidulans)中发现SR蛋白(swoK)与菌丝的极性生长和细胞壁的正常发育有关[15]。【本研究切入点】剪切因子SC35是RNA转录的重要调节因子。前期通过全基因组的同源搜索和分析发现在玉米大斑病菌中存在丰富的可变剪接调控蛋白,转录组学的初步分析也证实了玉米大斑病菌RNA可变剪接事件的存在,在此基础上本研究对该病菌SC35家族的剪接因子进行结构、表达模式及互作关系的分析。【拟解决的关键问题】利用生物信息学手段从玉米大斑病菌全基因组数据库中获取SC35,并对其蛋白结构、系统进化进行分析,利用实时荧光定量PCR(qRT-PCR)技术对SC35在病菌不同发育时期及在侵染寄主过程中的表达模式进行系统检测,并对其互作关系进行分析,为深入探究剪切因子SC35在调控病菌致病力方面的功能提供理论依据。1 材料与方法
试验于2019年在河北农业大学真菌毒素与植物分子病理学实验室完成。1.1 供试菌株及寄主
玉米大斑病菌野生型菌株01-23、玉米自交系B73的抽雄期新鲜叶片、大肠杆菌(Escherichia coli)DH5α、酵母双杂交试验用菌株酿酒酵母(Saccharomyces cerevisiae)AH109及载体pGADT7(AD)、pGBKT7(BD)均由河北农业大学真菌毒素与植物分子病理学实验室保存。1.2 主要培养基及试剂
PDA培养基、水琼脂培养基、玻璃纸、YPD培养基、二缺培养基、三缺培养基、四缺培养基;总RNA提取试剂盒、RNA反转录试剂盒购于生工生物工程(上海)股份有限公司。PrimeScript?RT reagent Kit(Perfect Real Time)试剂盒,SYBR? Premix Ex TaqTM II(Perfect Real Time)和T4 DNA连接酶均购自TaKaRa(中国大连)公司,2×TransStart? FastPfu PCR SuperMix(-dye)购自全式金(北京)公司,BM无缝克隆试剂盒(CL116-01)购自博迈德(北京)公司,引物均由生工生物工程(上海)股份有限公司合成。1.3 玉米大斑病菌SC35同源蛋白的生物信息学分析
利用网站(1.4 不同发育时期和侵染时间的病菌材料收集
玉米大斑病菌不同发育时期的材料收集参考贾慧等[16]的方法,将野生型菌株01-23接种于PDA培养基,25℃黑暗培养10 d后,轻轻刮取菌落表面收集菌丝;将在同样培养条件下生长15 d后的菌株平皿中加入10 mL无菌水,然后轻轻刮取菌落表面,经两层纱布过滤后离心并收集分生孢子;将上述收集的分生孢子调整成浓度为104个/mL的孢子悬浮液,接种2 mL于在水琼脂培养基表面平铺的玻璃纸上,分别在接种后3 h(芽管时期)、12 h(附着胞时期)、24 h(侵入丝时期)离心收集菌体,此时80%以上的分生孢子发育到相应时期。按照上述收集方法将浓度为104个/mL的病菌孢子悬浮液以20 μL/滴的液滴接种至玉米叶片表面,分别收集侵染后1、3、6、9、12、15和18 h的菌液,再次离心富集菌体。1.5 SC35同源基因的表达规律分析
利用NCBI(Table 1
表1
表1实时荧光定量PCR所用引物序列
Table 1
| 引物Primer | 序列Sequence (5′-3′) |
|---|---|
| Tubulin-F/R | GGGAACTCCTCACGGATGTTG/TAACAACTGGGCAAAGGGTCA |
| StSC1-F/R | AGAGCCAGAAACACTGAGCC/AAGCGAGAAAAGACACGCCT |
| StSC2-F/R | GAAAATCAAGACGACCCGCC/CTTTGCCCATTGCTTTCGCT |
| StSC3-F/R | AGCGCAAGGAGAAAGAGAGG/CCGTCTTCCTTGATACGCCT |
| StSC4-F/R | CACTGGAGATGGTTGGCGAT/TTGCACACAAACAGCACGTT |
| StSC5-F/R | GAACCTCTTCGTCACAGGCA/GCATGATGTTGCACTGCTCC |
| StSC6-F/R | GCTTTGGCTTTGTCCGCTAC/ACGCGGATTCTACGTCCATC |
| StSC7-F/R | GCGACTTTGGCACCGTTAAG/TCGAGCGTGGCATACTCAAT |
| StSC8-F/R | GGCCTTGACCAAGAACGTCA/GCGGACATCCTTGATGACAC |
新窗口打开|下载CSV
1.6 玉米大班病菌SC35同源蛋白互作分析
利用BioEdit软件对各个基因序列进行酶切位点分析,并通过NEBuilder(Table 2
表2
表2酵母双杂交试验所用引物
Table 2
| 引物Primer | 序列Sequence (5′-3′) |
|---|---|
| StSC4-F/R | GGCCATGGAGGCCAGTGAATTCATGTCTGTGCTTCTAGAAACATCAC/TTCATCTGCAGCTCGAGCTCGATGGATCCTCATCGTCGACGGTCCCGGT |
| StSC6-F/R | CATATGGCCATGGAGGCCAGTGAATTCATGTCGAAACTCTTCATTG/CATCTGCAGCTCGAGCTCGATGGATCCCTACCATTGCTGCTG |
| StSC8-F/R | TATGGCCATGGAGGCCGAATTCATGCCGTCCCGTTCGC/CTAGTTATGCGGCCGCTGCAGGTCGACGGATCCTCATCTGCGCTCCCTACTTG |
| StSC3-F/R | ATATGGCCATGGAGGCCGAATTCATGAACTCGATCCGCGCCATCCAGGAG/GTTATGCGGCCGCTGCAGGTCGACGGATCCTTACACGCGAGTGTCTCTCT |
| StSC7-F/R | CATATGGCCATGGAGGCCGAATTCATGGAGGACGACGCCAAGATG/TTATGCGGCCGCTGCAGGTCGACGGATCCCTACTCGTCGTCCATATCGTC |
新窗口打开|下载CSV
2 结果
2.1 玉米大斑病菌SC35同源蛋白的获取
通过对玉米大斑病菌基因组数据库(Table 3
表3
表3玉米大斑病菌SC35的蛋白信息
Table 3
| 名称 Name | JGI标号 ID in JGI | NCBI登录号 Accession number in NCBI | 大小 Size (aa) | 基因长度 Gene length (bp) | 基因定位 Gene mapping |
|---|---|---|---|---|---|
| StSC1 | 46444 | XP_008023357.1 | 391 | 1505 | scaffold_14:633653-635474 (+) |
| StSC2 | 21139 | XP_008027986.1 | 282 | 1031 | scaffold_4:463120-464150 (-) |
| StSC3 | 171745 | XP_008027251.1 | 304 | 915 | scaffold_3:679211-680616 (-) |
| StSC4 | 113046 | XP_008020610.1 | 477 | 1555 | scaffold_1:618088-619811 (+) |
| StSC5 | 99605 | XP_008030866.1 | 111 | 336 | scaffold_8:693429-693764 (+) |
| StSC6 | 168126 | XP_008023544.1 | 161 | 835 | scaffold_14:724868-726377 (-) |
| StSC7 | 88128 | XP_008025196.1 | 159 | 537 | scaffold_2:109463-109999 (-) |
| StSC8 | 156630 | XP_008029952.1 | 343 | 1157 | scaffold_6:1923760-1924916 (+) |
新窗口打开|下载CSV
2.2 玉米大斑病菌SC35的生物信息学分析
2.2.1 SC35蛋白结构分析 玉米大斑病菌的剪接因子SC35与其他物种SR蛋白结构相似(图1),N端具有1个典型的RNA识别结构域RRM1,C端具有随机排列组成的SR/RS重复序列,此外有的蛋白内部可能还存在亚结构域RRM2。剪接因子StSC1与其他SR蛋白明显不同的是它具有两个靠近C端的RRM结构域,类似的结构特征还未有研究报道。剪接因子StSC2 N端具有eIF-3G结构域,eIF3参与细胞生长和细胞周期的调控,在哺乳动物细胞中由约13个亚基组成,分子量在550—700 kD,是一种非特异性RNA结合蛋白[18],推测StSC2可能主要在玉米大斑病菌中某些蛋白质翻译起始过程发挥作用。图1
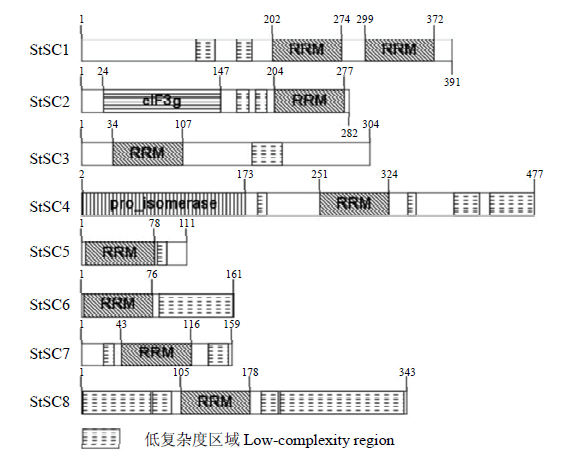 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1玉米大斑病菌SC35蛋白结构示意图
Fig. 1Schematic diagram of protein structure of SC35 in S. turcica
2.2.2 SC35进化分析 由于SR蛋白在真菌中的研究很少,通过构建系统发育树发现除StSC3与StSC4有较近亲缘关系外,其他剪接因子之间分支明显,不具有同源性(图2)。其中剪接因子StSC6与番茄匍柄霉(Stemphylium lycopersici)中富含甘氨酸的RNA结合蛋白聚于同一分支,StSC2与穿孔陷球壳(Trematosphaeria pertusa)等多种真核生物翻译起始因子eIF-3G同源,StSC8与链格孢(Alternaria alternata)中富含丝氨酸结构域(RNPS1)的RNA结合蛋白聚于同一分支;StSC1和StSC7分别与小麦核腔菌(Pyrenophora tritici-repentis)和禾谷镰孢(Fusarium graminearum)聚于同一分支,与代表RNA结合的蛋白同源;StSC5与裂殖酵母(Schizosaccharomyces pombe)中具有RRM结构域的RNA结合蛋白亲缘关系较近;StSC3与粗糙脉孢菌(Neurospora crassa)中注释为U2 snRNP功能的蛋白同源且亲缘关系较近,StSC4与裂殖酵母中假定的U1 snRNP具有同源性,说明它们可能是剪接体复合物的一部分,可能会在某个阶段影响U1或U2蛋白对mRNA的识别。
图2
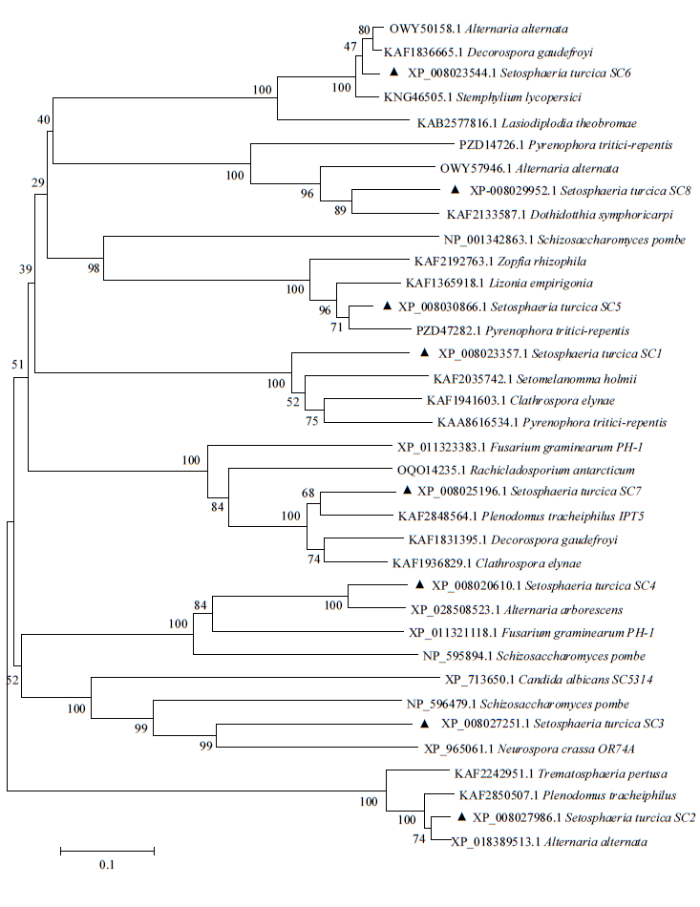 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2玉米大斑病菌SC35类可变剪接因子与其他真菌同源蛋白系统进化分析
Fig. 2Phylogenetic analysis of splicing factor SC35 of S. turcica and other fungi
2.3 病菌不同发育时期和侵染寄主不同时间SC35基因表达规律
2.3.1 不同发育时期SC35基因表达量分析 统计分析玉米大斑病菌各生长时期可变剪接因子的表达量,定义菌丝生长时期各个基因的表达量为1,发现各基因在分生孢子、芽管、附着胞和侵入丝时期均有表达(图3)。整体来说StSC2、StSC3、StSC4、StSC5、StSC6、StSC7、StSC8表达量呈下调趋势,转录活性低于菌丝时期;StSC1呈现下调-上调-下调的趋势,在附着胞时期表达量最高,是分生孢子时期的24.44倍,在侵入丝形成时期StSC1表达量极显著下调(P<0.001),但仍高于其他时期,StSC4和StSC6表达量极显著上调(P<0.001),但稍低于菌丝时期;StSC4和StSC6在侵入丝时期比较活跃,说明它们可能参与病菌侵染结构的形成,对致病性具有重要调控作用。图3
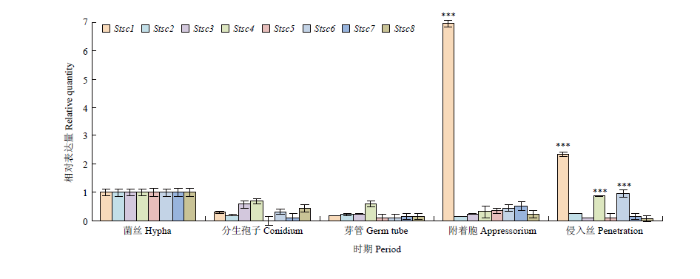 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3不同发育时期可变剪接因子的表达量
Fig. 3Expression of alternative splicing factor at different developmental stages
***: P<0.001
2.3.2 侵染寄主不同时间SC35基因表达量分析 在玉米大斑病菌的分生孢子侵染玉米叶片过程中,定义侵染1 h时的表达量为1,此时分生孢子未萌发(图4)。其中StSC1、StSC2、StSC3和StSC4表达模式一致,呈现下调-上调-下调-上调-下调-上调的趋势,在侵染后期18 h时表达最活跃(P<0.001),分别是分生孢子时期的1.72、3.90、6.32、16.00倍,此时分生孢子开始产生侵入丝,说明这4个可变剪接因子对侵入丝的形成具有重要的调控作用,虽然StSC1、StSC2和StSC3在侵染过程中表达量一直在改变但变化幅度很小;StSC5和StSC6表达变化水平和趋势一致,呈现上调-下调-上调-下调-上调的趋势,StSC5、StSC6和StSC8在侵染前期3 h时表达量最高(P<0.001),分别是分生孢子时期的13.60、13.60和2.94倍,此时分生孢子开始萌发产生芽管,说明这3个剪接因子在芽管发育初期发挥重要作用;6 h时芽管顶端开始生成附着胞,StSC5、StSC6和StSC8表达量下调,StSC4的表达量极显著升高(P<0.001)到分生孢子时期的12.46倍,同时在侵染中期9—15 h时也表现出较高水平的转录活性,说明它在病菌整个侵染过程中具有重要调控作用。StSC7在此次侵染过程中呈降低趋势,在整个侵染过程中不活跃。
图4
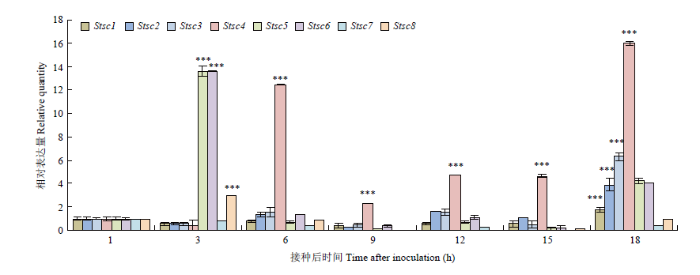 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4接种不同时间后可变剪接因子的表达量
Fig. 4Expression of alternative splicing factor after inoculation at different times
***: P<0.001
2.4 病菌可变剪接因子SC35蛋白间的互作
2.4.1 酵母双杂交载体构建 利用EcoRⅠ和BamHⅠ对各个基因重组质粒进行双酶切验证,分别得到大小为1 434、468、1 032、915、408、8 000 bp和1 434、468、1 032、915、408、7 300 bp的条带,与预期结果相符(图5),说明酵母双杂交载体构建成功。图5
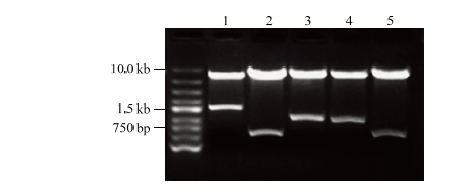 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5酵母双杂交载体的酶切验证
Fig. 5Enzyme digestion verification of two-hybrid vectors
1: StSC4-BD; 2: StSC6-BD; 3: StSC8-BD; 4: StSC3-BD; 5: StSC7-BD
2.4.2 酵母双杂交试验验证 在酵母双杂交试验中,重组质粒pGADT7/pGBKT7-StSC4与pGADT7/ pGBKT7-StSC6、pGADT7/pGBKT7-StSC3与pGADT7/ pGBKT7-StSC8、pGADT7/pGBKT7-StSC3与pGADT7/ pGBKT7-StSC4、pGADT7/pGBKT7-StSC4与pGADT7/ pGBKT7-StSC8组合的转化子在二缺板上能正常生长,而在四缺板上也都能生长,说明剪接因子StSC4与StSC6、StSC3与StSC8、StSC3与StSC4、StSC8与StSC4有明显的互作,并且这种互作是特异性的(图6),推测StSC4、StSC6、StSC3、StSC8可能通过组合成不同的复合体对前体mRNA形成不同的剪接结果,进而编码不同蛋白质。
图6
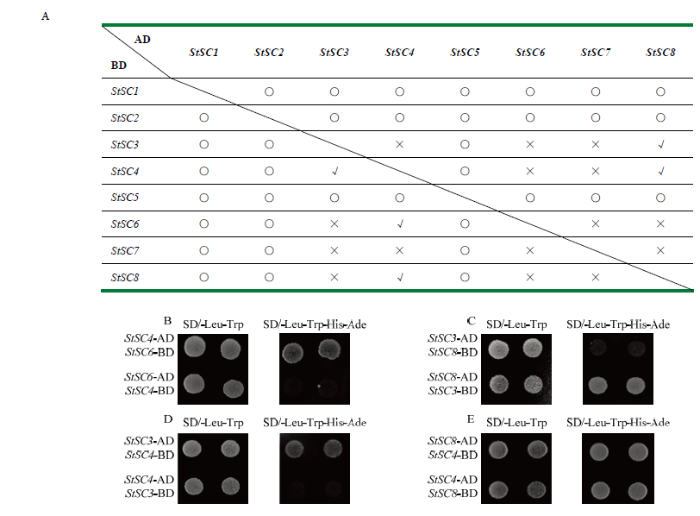 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6酵母双杂交验证玉米大斑病菌可变剪接因子间的互作
Fig. 6Verification of the alternative splicing factor interaction through yeast two-hybrid
A:8个剪接因子间相互作用关系 The interaction of 8 alternative splicing factors;B:StSC4-AD/BD与StSC6-AD/BD StSC4-AD/BD and StSC6-AD/BD; C:StSC3-AD/BD与StSC8-AD/BD StSC3-AD/BD and StSC8-AD/BD;D:StSC3-AD/BD与StSC4-AD/BD StSC3-AD/BD and StSC4-AD/BD; E:StSC8-AD/BD与StSC4-AD/BD StSC8-AD/BD and StSC4-AD/BD
○:未验证Not verified;√:互作Interacted;×:不互作Not interacted
3 讨论
SR蛋白家族数量庞大,种类多样,其中RNA识别基序(RRM)介导SR蛋白与前体mRNA上的特定RNA序列结合;RS结构域介导SR蛋白与其他剪接因子和剪接体蛋白(snRNP)的相互作用,通过调控剪接体的组装进而协同完成对前体mRNA剪接位点的选择。最早在裂殖酵母中发现两种编码SR蛋白的基因(Srp1、Srp2),Srp1是首个在裂殖酵母中发现的具有典型SR蛋白特征的酵母蛋白,它虽然不是裂殖酵母生长发育所必需的基因,但是分别过表达RBD和RS结构域,裂殖酵母均停止生长,且前体RNA大量聚集,剪接受到抑制;Srp2蛋白是N端含有两个RBD结构域的SR蛋白,其中RS结构域可以影响其在细胞核中的定位,并且两者能够相互作用构成一个蛋白复合体[19,20,21,22]。裂殖酵母中Srp1和Srp2的相互作用也受蛋白磷酸化的调节,Dsk1蛋白激酶介导的Srp1磷酸化抑制了Srp1/Srp2二聚体的形成,但是目前还不清楚Dsk1磷酸化调控Srp蛋白功能的机制[22]。酿酒酵母中虽然没有Srp1的同源蛋白,但是发现了Srp2的同源蛋白Npl3p,Dsk1的同源蛋白Sky1。1999年,YEAKLEY等发现Sky1的缺失不仅抑制了RS结构域介导的SR蛋白间的相互作用,而且SR蛋白的核转运功能也被抑制,同时细胞核中含有RS结构域的蛋白定位也出现异常[23]。大量研究表明,SR蛋白RS结构域中的丝氨酸残基广泛地被磷酸化,这种磷酸化影响SR蛋白的亚细胞定位及SR蛋白之间的相互作用,两者均可能改变SR蛋白在剪接中起作用的能力[24]。LORKOVIC等在拟南芥中鉴定出SR蛋白家族两种新蛋白CypRS64和CypRS92,其N端含有肽基-脯氨酰顺/反异构酶结构域,C端含有SR/SP二肽组成的结构域,研究发现亲环蛋白具有肽基-脯氨酰顺/反异构酶活性,其与蛋白质折叠、组装和运输有关[25]。CAVAREC等发现果蝇(Drosophila melanogaster)和人类(Homo sapiens)中含RS结构域的亲环蛋白Moca-cyp和SRcyp分别与转录调节因子p300/CBP和RNA聚合酶II(PolⅡ)的C端结构域结合,表明它们可能参与了p300/CBP对下游靶基因的转录调控和对PolⅡ前体mRNA的处理[26,27]。1997年,CHANDLER等发现SF2/ASF的非典型RRM结构域不仅具有剪接的功能,而且可以对N端RRM的剪接特异性进行调控。因此,SR蛋白中的多个RRM协同作用,实现了前体mRNA的底物特异性[28]。LEE等从酿酒酵母中克隆了一个RNA结合蛋白基因——葡萄糖抑制基因(RBP1),RBP1蛋白序列也包含两个RNA识别基序,其功能的破坏使细胞在对数早期的生长速度加快,RBPI基因产物的具体功能尚不清楚,推测RBP1可能通过改变RNA代谢过程,参与了对细胞生长的调节[29]。在对玉米大斑病菌剪接因子蛋白结构的分析中,笔者也发现在StSC1中距离N端较远处具有两个RRM结构域,但其具体作用还不清楚。2017年,张艺美克隆出禾谷镰孢中与裂殖酵母Srp1和Srp2同源的蛋白FgSrp1和FgSrp2,发现FgSrp1是生长发育所必需的,这与裂殖酵母正好相反,FgSrp1功能的丧失使得菌体生长速率、产孢量、致病性都降低,而对FgSrp1/2双缺失突变体的研究发现其他SR蛋白可以弥补这个缺陷,也证明真菌体内SR蛋白功能存在冗余[30]。总体来说,SR蛋白在植物与动物领域研究广泛,而在丝状真菌中的研究还很少。笔者通过系统进化分析发现StSC2与真核翻译起始因子eIF3有密不可分的关系,同时StSC4也与裂殖酵母的亲环蛋白Rct1具有同源性,推测StSC2参与了RNA转录后的翻译起始过程。根据病菌侵染寄主表面过程6 h的qRT-PCR结果,推测StSC4对芽管生长后期细胞内蛋白质的加工有重要调控作用。StSC6与番茄匍柄霉中富含甘氨酸的RNA结合蛋白(GRP)的序列覆盖率为57%,相似度为95.65%,已有研究发现GRP可能参与植物体细胞壁及细胞骨架的构成[31];而StSC8与多种真菌中富含丝氨酸的RNA结合蛋白具有相似性。笔者发现在病菌侵染寄主表面过程中StSC5、StSC6和StSC8在分生孢子萌发3 h即芽管形成初期表达活跃,推测StSC5、StSC6和StSC8在芽管萌发中发挥重要作用,StSC6可能同时参与调控真菌细胞壁的发育。StSC1在附着胞时期相对表达量最高,与小麦核腔菌中和核糖体翻译功能相关的蛋白聚于同一支,笔者推测StSC1可能在将mRNA运输到菌丝尖端的过程中起作用,也可能通过抑制细胞顶端核糖体翻译功能的发生,影响细胞的极性生长,导致细胞的形态发生变化,使其肿胀膨大进而形成附着胞。LORKOVIC等研究表明,拟南芥的剪接因子之间具有密切的同源性[32],但是本研究通过发育树的建立发现玉米大斑病菌中SC35剪接因子间的同源性比较低,这与动物类似。
SC35蛋白对于依赖ATP的U1 snRNP和U2 snRNP与前体mRNA之间的组合是不可缺少的[7]。20世纪末已经证明哺乳动物剪接因子SC35和SF2/ASF能够与U1 snRNP蛋白相互作用[33,34]。2002年,LOPATO等在拟南芥中发现一种新的富含RS的蛋白质AtRSZ33,同时发现SCL家族的另外两个蛋白SCL30a和AtSC35均与AtRSZ33相互作用,因为SF2/ASF和SC35蛋白的功能之一是识别5′剪接位点,并且AtRSZ和AtSCL蛋白均与U1 snRNP蛋白结合,推测AtRSZ33最有可能在5′剪接位点附近并参与剪接体装配[35]。SC35不仅可以招募其他SR蛋白在相同的剪接位点形成剪接复合体,也可能和其他SR蛋白相互作用帮助5′和3′剪接位点接触进而完成对前体mRNA的剪切和连接[33,36]。通过生物信息学分析发现,玉米大斑病菌剪接因子StSC3与粗糙脉孢菌中所明确的U2 snRNP蛋白具有51.92%的同源性,StSC4与裂殖酵母中U1 snRNP相关蛋白USP101具有26.85%的同源性,有体外研究证明过量的SR蛋白可以弥补剪接复合体中U1 snRNP的缺少[15],因此笔者推测StSC3可能在3′剪接位点附近参与剪接体装配;StSC4可能在5′剪接位点附近参与装配。通过酵母双杂交验证发现StSC4、StSC6、StSC3、StSC8之间能够相互作用,推测它们可能通过组合成不同的复合体发挥功能,参与玉米大斑病菌中某些剪接复合体对5′和3′剪接位点的选择,综合两者,笔者推测这些剪接因子有两种作用方式:一种是剪接因子StSC6和StSC8可能在5′剪接位点与StSC4共同参与剪接复合体的组装,然后StSC3通过与StSC8或StSC4的互作关系将3′和5′剪接位点连接在一起,完成对前体mRNA的剪接;另一种可能是StSC8与StSC3在3′剪接位点参与剪接复合体的组装,StSC4与StSC6在5′剪接位点参与组装,然后StSC3通过与StSC4的互作关系将3′和5′剪接位点连接在一起。但是不能排除四者共同参与更复杂的剪接复合体的构成。
4 结论
获得了玉米大斑病菌的8个可变剪接因子,发现在玉米大斑病菌的不同发育时期及侵染过程中可变剪接因子的表达模式不同。StSC1、StSC2、StSC3、StSC5、StSC6、StSC8在病菌侵染感病玉米叶片18 h时表达活跃,StSC7在整个过程中不活跃,StSC5、StSC6和StSC8在侵染前期即分生孢子萌发初期发挥重要作用,StSC4在病菌整个侵染过程中具有重要调控作用;菌丝、分生孢子、芽管、附着胞及侵入丝时期的表达规律分析表明,StSC1、StSC4和StSC6对病菌的附着胞和侵入丝形成有比较明显的调控作用;酵母双杂交试验证明剪接因子StSC4与StSC6、StSC3与StSC8、StSC3与StSC4、StSC8与StSC4有互作关系,促使剪接复合体在5′或3′剪接位点进行装配和剪接。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
