 ,1, 李春辉
,1, 李春辉 ,2
,2Phenotypic Variation and Genome-wide Association Analysis of Root Architecture at Maize Flowering Stage
ZHANG XiaoQiong1, GUO Jian2, DAI ShuTao3, REN Yuan4, LI FengYan5, LIU JingBao3, LI YongXiang2, ZHANG DengFeng2, SHI YunSu2, SONG YanChun2, LI Yu2, WANG TianYu2, ZOU HuaWen ,1, LI ChunHui
,1, LI ChunHui ,2
,2通讯作者:
责任编辑: 李莉
收稿日期:2019-03-20接受日期:2019-04-25网络出版日期:2019-07-16
| 基金资助: |
Received:2019-03-20Accepted:2019-04-25Online:2019-07-16
作者简介 About authors
张小琼,E-mail: qiong2017@foxmail.com。

摘要
关键词:
Abstract
Keywords:
PDF (5010KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
张小琼, 郭剑, 代书桃, 任元, 李凤艳, 刘京宝, 李永祥, 张登峰, 石云素, 宋燕春, 黎裕, 王天宇, 邹华文, 李春辉. 玉米花期根系结构的表型变异与全基因组关联分析[J]. 中国农业科学, 2019, 52(14): 2391-2405 doi:10.3864/j.issn.0578-1752.2019.14.002
ZHANG XiaoQiong, GUO Jian, DAI ShuTao, REN Yuan, LI FengYan, LIU JingBao, LI YongXiang, ZHANG DengFeng, SHI YunSu, SONG YanChun, LI Yu, WANG TianYu, ZOU HuaWen, LI ChunHui.
0 引言
【研究意义】玉米是世界上重要的粮食作物和饲料作物。根系作为植株与土壤直接接触的器官,对水分和养分的吸收至关重要[1,2,3,4]。因此,解析玉米根系结构形成的遗传基础,挖掘玉米根系相关性状的优异等位基因,对进一步提高玉米产量具有重要意义。【前人研究进展】已有研究表明,根系的长度和数量与小区产量显著正相关,相关系数分别为0.39和0.73[5,6];干旱条件下,根系和水平地面的角度与小区产量极显著正相关,相关系数达0.91[7],根直径、根长度以及根系在土壤中分布的变化与作物的抗旱性密切相关[8,9];玉米通过减少地下节根条数及增加根系的下扎深度提高水分利用率,进而减少干旱胁迫造成的伤害[10]。研究表明,玉米根系表型变异丰富,根系的数量、长度、直径和角度共同在土壤中形成一个复杂、动态的三维根系结构[11],玉米自交系苗期的根数量、长度、直径和干重的变异系数分别为0.43、25.2—0.48、0.25、0.30—0.36[12,13]。玉米根系结构分为胚生根系统和胚后根系统,胚生根系统主要由数条种子根和1条初生根构成,在玉米苗期发挥重要作用,但其在植株生长后期退化,不同玉米品种的种子根条数0—13条不等;胚后根系统包括节根和侧根,节根从茎节上长出,从地下茎节长出的称为地下节根,从地上茎节长出的节根又称支持根、气生根,胚后根主要起到固定支持和吸收水分、养分的作用,玉米约有6层地下节根和2—3层地上节根,约70条节根[14]。随着玉米植株的生长、发育,根系生物量在籽粒灌浆结束后达到最大[15],因此,大多研究者选择在开花期或成熟期对玉米根系的胚后根系统进行研究[5,16-17]。前人研究结果表明,不同年代的玉米品种在节根条数、长度、角度、干重等性状上存在着显著的差异[5,7,18-19]。且有研究证实,在美国玉米品种更替过程中,根系构型的变化对产量具有直接影响[4]。目前,利用连锁分析方法已经定位到一些玉米节根相关性状的QTL。CAI等[5]利用Ye478×Wu312构建的BC4F3群体,分别在拔节期、吐丝期、成熟期3个时期对地下节根条数和地下节根长度进行定位,共检测到3个与地下节根条数相关的QTL位点,8个与地下节根长度有关的QTL,其中有2个QTL在至少2个生育期共定位。蔡红光等[20]鉴定了一个BC4F3群体抽雄期和灌浆期的根干重、总根长、侧根长、地下节根长、地下节根数等根系性状,QTL分析共检测到抽雄期根系结构20个QTL和灌浆期根系结构14个QTL。ZHANG等[17]利用大刍草和W22构建的包含866份材料的BC2S3群体,在开花期分别对海南和北京的9个节根数相关性状进行QTL分析,共定位到133个QTL(海南69个、北京64个),其中62个QTL与开花期QTL相互重叠,15个QTL与株高QTL相互重叠,共检测到5个控制节根数、开花期和株高的一因多效QTL。KU等[21]以黄早四和CML288构建的RIL群体和永久F2群体为材料,对气生根总层数和有效气生根层数进行了QTL定位,在RIL群体中共检测到10个QTL,在永久F2群体中共定位到15个QTL。GU等[22]以F2:3群体作为试验材料,对气生根直径、层数、鲜重、干重等性状进行QTL分析,共检测到93个QTL。GUO等[23]收集整理了已发表的428个玉米根系相关性状QTL,采用元分析方法,获得53个Meta-QTL。全基因组关联分析(genome-wide association studies,GWAS)是研究数量性状的重要方法之一,随着测序技术的快速发展,结合高密度分子标记,利用GWAS方法解析植物重要性状的遗传机制的应用越来越多。PACE等[13]基于Ames panel中的384个自交系,调查了22个苗期根系相关性状,结合GBS测序获得的基因型数据利用GWAS方法鉴定出268个与根系性状显著相关的标记。SANCHEZ等[24]对玉米DH群体的苗期根系相关性状进行GWAS分析,共检测到17个显著关联SNP。ZAIDI等[16]利用396份热带玉米自交系开展了水分胁迫条件下成熟期根系性状的GWAS分析,共检测到67个与根深、根干重、根长、根体积、根表面积、根长密度等性状显著关联的SNP位点。ZHANG等[17]利用663份玉米自交系对节根数进行GWAS分析,结合大刍草和W22构建的BC2S3群体连锁分析结果,其中3个显著关联SNP与节根数和开花期相关的QTL共定位。【本研究切入点】尽管前人利用分离群体和关联群体检测到一些玉米根系性状相关的QTL,但利用全基因组关联分析方法挖掘开花期根系结构遗传变异的研究较少,且研究不同育种时期和不同杂种优势类群材料的根系结构变化较少。【拟解决的关键问题】本研究以111份玉米自交系为试验材料,对6个花期地下节根相关性状进行测定,对不同年代玉米自交系根系的变化及不同杂种优势类群玉米自交系根系的差异进行分析,并对玉米地下节根相关性状进行全基因组关联分析,旨在了解玉米不同育种时期根系结构的变化规律和探究玉米地下节根相关性状的遗传机制。1 材料与方法
1.1 试验材料及试验设计
于2017年在北京、陕西永寿、山西定襄及河南原阳4个试验点进行田间试验。供试材料由111份玉米自交系组成。田间设计采用完全随机区组设计,2次重复,每个材料双行区种植,行长3 m,行距0.6 m,株距0.25 m,密度67 500株/hm2,田间采用常规管理。1.2 性状测定与方法
玉米花期时,选取各小区内长势一致、花期一致的3株玉米,通过人工挖根调查根系性状。每个单株以长25 cm、宽25 cm、深度为30 cm的根系主要分布的土块为取样单位,收集完整根系,使用高压水枪将根系冲洗干净。收集的根系在4个方位进行图片采集,利用RESTroot根系分析软件对地下节根角度(belowground node root angle,RA)、地下节根面积(belowground node root shadow area,RS)进行分析取平均值,地下节根角度为地下节根张开的夹角,地下节根面积为地下节根的总投影面积。完成图片采集的根系按照玉米根系生长的层次使用工具剪刀逐一剪下,记录地下节根层数(belowground node root layer number,RLN)和地下节根总条数(total number of belowground node root,TRN),采用排水法测量地下节根体积(belowground node root volume,RV)。各项指标测定完成后,将根系装入牛皮纸袋中,105℃杀青30 min,85℃烘干至恒重,测定地下节根干重(belowground node root dry weight,RDW)。1.3 表型数据的统计分析
采用Excel2016、SPSS16.0软件对地下节根层数(RLN)、地下节根总条数(TRN)、地下节根角度(RA)、地下节根面积(RS)、地下节根体积(RV)和地下节根干重(RDW)等6个性状进行统计分析,分别取4个环境下各性状的平均值,并计算各性状的标准差和变异系数。采用QTL IciMapping V4.1软件中的ANOVA计算遗传力,利用SPSS16.0软件对数据进行单因素方差分析,利用Bonferroni方法进行多重比较,使用Minitab 16绘制根系性状的频数分布图,运用R语言进行根系性状间的相关性分析。1.4 全基因组关联分析与候选基因的挖掘
实验室前期对111份自交系进行了低深度的重测序,共获得152 352个高质量SNP标记(缺失率小于10%,微效等位基因频率(minor allele frequency,MAF)大于0.05,位点杂合度小于10%)。基于这些高质量的标记,利用TASSEL5.0软件对111份自交系进行群体结构分析,结合材料系谱,利用iTOL软件绘制Neighbor-joining tree。利用Fixed and random model Circuitous Probability Unification(FarmCPU)模型[25]对4个环境下的6个节根相关性状的平均值进行全基因组关联分析,以P=1.0×10-5为阈值,判定SNP标记与目标性状关联的显著性。使用TASSEL5.0软件计算群体的连锁不平衡(linkage disequilibrium,LD)平均衰减距离,基于显著关联的SNP标记在玉米B73基因组序列RefGen_v4上的物理位置,在其LD范围内挖掘玉米地下节根相关性状的候选基因,在NCBI(https://blast.ncbi.nlm.nih. gov)、MaizeGDB(https://www.maizegdb.org)、TAIR(https://www.arabidopsis.org)网站上对候选基因进行功能注释,使用agriGO在线分析软件(http://bioinfo. cau.edu.cn/agriGO/)对候选基因的功能进行富集分析。
2 结果
2.1 地下节根相关性状表型分析
取4个环境下的根系相关性状的平均值作为表型数据,对6个地下节根相关性状进行统计分析,111份玉米自交系的6个表型性状RLN、TRN、RA、RS、RV、RDW均呈现近似正态分布(图1),表明这些性状均为典型的数量性状,由微效多基因控制。在111份玉米自交系中,6个数量性状的表型表现出较大的变异(表1)。变异系数分析表明,RV和RDW的变异系数最大,与RLN、TRN、RA和RS的比值差异在1.99—4.00倍,且6个性状均显示较高的遗传力。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1玉米地下节根相关性状变异
A:玉米根系表型多样性;B:地下节根相关性状频数分布图;RLN:地下节根层数;TRN:地下节根总条数;RA:地下节根角度(°);RS:地下节根面积(cm2);RV:地下节根体积(cm3);RDW:地下节根干重(g)。下同
Fig. 1Phenotypic variation of belowground node root-related traits of the maize
A: Phenotypic diversity of maize root; B: Frequency distribution of belowground node root-related traits; RLN: Belowground node root layer number; TRN: Total number of belowground node root; RA: Belowground node root angle (°); RS: Belowground node root shadow area (cm2); RV: Belowground node root volume (cm3); RDW: Belowground node root dry weight (g). The same as below
Table 1
表1
表1地下节根相关性状统计分析
Table 1
| 性状 Trait | 均值 Mean | 最大值 Max | 最小值 Min | 标准差 SD | 变异系数 CV (%) | 广义遗传力 H2 |
|---|---|---|---|---|---|---|
| 地下节根层数RLN | 6.67 | 8.67 | 5.38 | 0.62 | 9.29 | 0.67 |
| 地下节根总条数TRN | 59.41 | 88.00 | 42.00 | 8.96 | 15.08 | 0.74 |
| 地下节根角度RA | 58.67 | 80.70 | 35.82 | 8.51 | 14.50 | 0.80 |
| 地下节根面积RS | 76.28 | 111.91 | 45.55 | 13.85 | 18.15 | 0.79 |
| 地下节根体积RV | 68.75 | 144.17 | 19.63 | 24.84 | 36.13 | 0.75 |
| 地下节根干重RDW | 13.77 | 32.60 | 4.09 | 5.11 | 37.12 | 0.82 |
新窗口打开|下载CSV
2.2 地下节根相关性状的相关性分析
通过对4个环境下的6个根系性状进行相关性分析(图2),地下节根干重与地下节根总条数、角度、面积和体积均呈现显著正相关关系。其中,地下节根体积与地下节根干重的相关系数达到0.88,相关性达极显著水平;地下节根角度与地下节根面积的相关系数为0.72,也达到极显著的水平;地下节根角度、面积与地下节根干重的相关系数为0.42和0.68,因此,根系所占空间越大,根系生物量越高,根系角度越大,根系的投影面积就越大。地下节根层数与地下节根总条数呈显著正相关,地下节根层数和总条数均与地下节根角度和面积呈负相关,说明地下节根层数和数量越多,根系结构可能越紧凑,根系角度越小,根系面积减小。图2
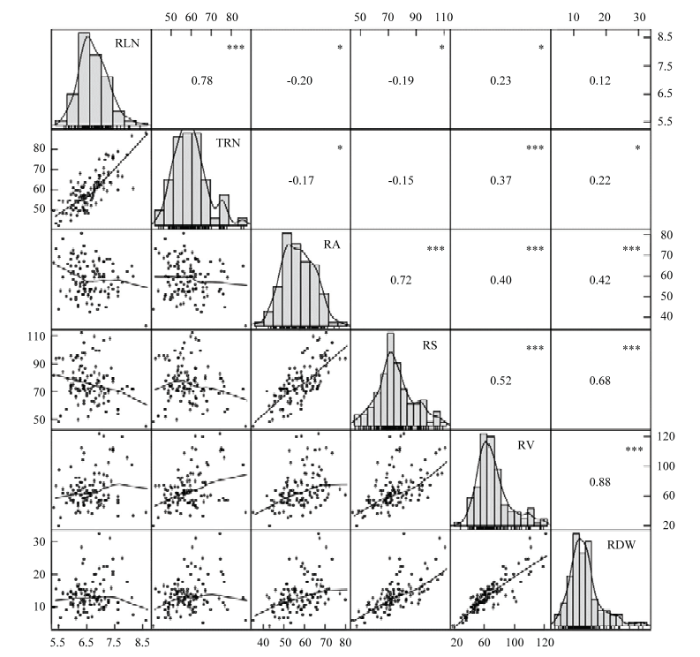 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2地下节根相关性状的相关性分析
*:在0.05水平上显著相关;***:在0.001水平上显著相关
Fig. 2Correlation analysis of belowground nodal root-related traits
*: Correlation is significant at the 0.05 level; ***: Correlation is significant at the 0.001 level
2.3 不同年代玉米自交系地下节根相关性状的表型分析
通过查阅文献对供试材料的选育时间进行标注(表2),111份自交系中有46份自交系可根据其选育时间划分成4个年代,分别为1970s(10份)、1980s(17份)、1990s(14份)及2000s(5份)。随着年代的更替,根系各性状均呈现出不同程度的变化,根干重和根体积在各年代间的差异未达到显著水平;地下节根层数、总条数、角度和面积在2000s前各年代间无显著差异,2000s自交系地下节根层数和总条数显著减少,地下节根角度和面积显著增加;地下节根层数和总条数在年代的更替间表现出下降的趋势,地下节根角度和面积在年代的更替间表现出上升的趋势(图3)。Table 2
表2
表2不同年代玉米自交系
Table 2
| 年代 Era | 材料数量 Number of materials | 材料名称 Name of materials |
|---|---|---|
| 1970s | 10 | C103、E28、H21、Mo17、丹黄02 Danhuang02、黄早四Huangzaosi、威风322 Weifeng322、掖8112 Ye8112、原武02 Yuanwu02、吉63 Ji63 |
| 1980s | 17 | 853、5003、81162、4F1、B73、K12、Mo17Ht、X178、丹340 Dan340、合344 He344、齐319 Qi319、天涯4 Tianya4、铁7922 Tie7922、掖478 Ye478、掖488 Ye488、掖52106 Ye52106、郑58 Zheng58 |
| 1990s | 14 | 444、5237、A801、C8605-2、Ki3、KL4、P138、昌7-2 Chang7-2、丹598 Dan598、多229 Duo229、黄野四3 Huangyesi3、获唐黄17 Huotanghuang17、沈137 Shen137、郑22 Zheng22 |
| 2000s | 5 | 沈3336 Shen3336、四-144 Si-144、PH6JM、京724 Jing724、京725 Jing725 |
新窗口打开|下载CSV
图3
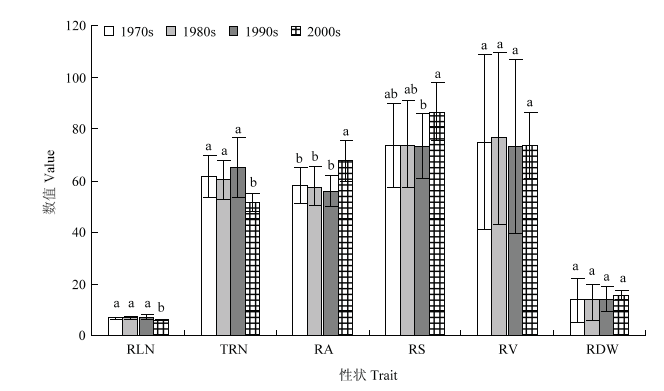 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3不同年代自交系地下节根相关性状的差异分析
a、b:在0.05水平上差异显著。下同
Fig. 3Differences of belowground nodal root-related traits of inbred lines developed in different eras
a, b: Significant at the 0.05 level respectively. The same as below
2.4 不同类群玉米自交系地下节根相关性状的表型分析
基于111份自交系基因型,利用TASSEL5.0软件对其进行聚类分析(图4),并结合刘志斋等[26]对820份玉米自交系的群体结构的分析结果,111份材料中的93(84%)份材料可划分为瑞德群(Reid)、旅大红骨群(Lüda Red Cob,LRC)、兰卡斯特群(Lancaster)、塘四平头群(Tang Si Ping Tou,TSPT)与P群(P group),其余18份(16%)材料形成了一个混合群(mixed group)。不同类群根系相关性状的统计分析表明,不同类群间的地下节根层数、总条数、角度和面积变异幅度较小,地下节根干重和地下节根体积在5个亚群中的变异幅度较大(表3)。不同类群根系性状差异分析表明,旅大红骨类群的6个地下节根性状均高于其余类群,但未达到显著水平;兰卡斯特类群的6个地下节根性状值均低于旅大红骨类群;P群的6个地下节根性状值均与旅大红骨类群无显著差异;塘四平头类群的地下节根角度、面积显著低于其余类群,地下节根层数、总条数和体积与旅大红骨类群无显著差异;瑞德类群的地下节根总条数显著低于旅大红骨类群,其余5个地下节根性状值与旅大红骨类群无显著差异(图5)。图4
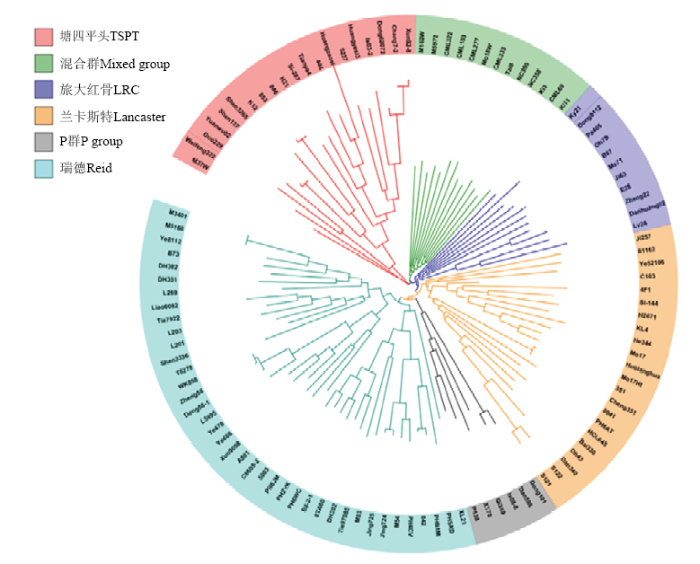 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4111份玉米自交系聚类分析
Fig. 4Cluster analysis of 111 maize inbred lines used in this study
Table 3
表3
表3不同类群地下节根相关性状的统计分析
Table 3
| 性状/类群 Trait/Group | 均值 Mean | 标准差 SD | 极小值 Min | 极大值 Max | 变异系数 CV (%) |
|---|---|---|---|---|---|
| RLN | |||||
| 瑞德Reid | 6.47ab | 0.64 | 5.38 | 8.22 | 9.94 |
| 旅大红骨LRC | 6.86a | 0.56 | 6.17 | 8.00 | 8.13 |
| 兰卡斯特Lancaster | 6.44b | 0.43 | 5.56 | 7.33 | 6.73 |
| 塘四平头TSPT | 7.02a | 0.64 | 6.17 | 8.17 | 9.12 |
| P群P group | 6.54ab | 0.52 | 5.83 | 7.21 | 7.92 |
| 混合群Mixed group | 7.08a | 0.55 | 6.33 | 8.67 | 7.76 |
| TRN | |||||
| 瑞德Reid | 56.86b | 7.33 | 43.13 | 75.83 | 12.90 |
| 旅大红骨LRC | 63.72a | 10.53 | 49.39 | 77.33 | 16.52 |
| 兰卡斯特Lancaster | 55.83b | 6.51 | 42.00 | 68.67 | 11.66 |
| 塘四平头TSPT | 63.79a | 10.47 | 48.11 | 86.67 | 16.41 |
| P群P group | 60.40ab | 9.26 | 47.92 | 74.80 | 15.33 |
| 混合群Mixed group | 63.35a | 9.77 | 51.67 | 88.00 | 15.42 |
| RA | |||||
| 瑞德Reid | 61.32a | 7.13 | 48.65 | 80.70 | 11.62 |
| 旅大红骨LRC | 62.66a | 4.81 | 53.71 | 67.65 | 7.67 |
| 兰卡斯特Lancaster | 58.43ab | 9.82 | 43.89 | 78.03 | 16.81 |
| 塘四平头TSPT | 54.82b | 6.94 | 44.87 | 66.00 | 12.66 |
| P群P group | 57.52ab | 8.23 | 43.71 | 67.99 | 14.31 |
| 混合群Mixed group | 55.76ab | 9.94 | 35.82 | 71.87 | 17.82 |
| RS | |||||
| 瑞德Reid | 82.07a | 11.62 | 60.58 | 109.61 | 14.16 |
| 旅大红骨LRC | 82.68a | 12.71 | 68.47 | 106.65 | 15.37 |
| 兰卡斯特Lancaster | 74.90a | 14.99 | 48.38 | 111.91 | 20.02 |
| 塘四平头TSPT | 65.50b | 10.22 | 51.80 | 88.01 | 15.60 |
| P群P group | 73.68ab | 13.88 | 57.50 | 97.73 | 18.84 |
| 混合群Mixed group | 74.34a | 13.85 | 45.55 | 107.94 | 18.63 |
| RV | |||||
| 瑞德Reid | 69.72ab | 23.36 | 34.21 | 140.83 | 33.51 |
| 旅大红骨LRC | 81.82a | 33.68 | 38.31 | 144.17 | 41.17 |
| 兰卡斯特Lancaster | 66.95ab | 23.85 | 19.63 | 124.52 | 35.63 |
| 塘四平头TSPT | 71.59ab | 29.86 | 33.33 | 139.17 | 41.71 |
| P群P group | 68.97ab | 24.45 | 44.37 | 107.99 | 35.45 |
| 混合群Mixed group | 60.62b | 19.36 | 38.33 | 123.11 | 31.93 |
| RDW | |||||
| 瑞德Reid | 14.25a | 4.34 | 6.49 | 24.86 | 30.44 |
| 旅大红骨LRC | 16.21a | 7.84 | 7.55 | 32.60 | 48.38 |
| 兰卡斯特Lancaster | 13.07a | 4.92 | 4.09 | 28.32 | 37.68 |
| 塘四平头TSPT | 12.26a | 4.99 | 5.21 | 25.07 | 40.66 |
| P群P group | 14.22a | 4.60 | 8.99 | 23.49 | 32.35 |
| 混合群Mixed group | 13.74a | 5.55 | 5.86 | 31.29 | 40.41 |
新窗口打开|下载CSV
图5
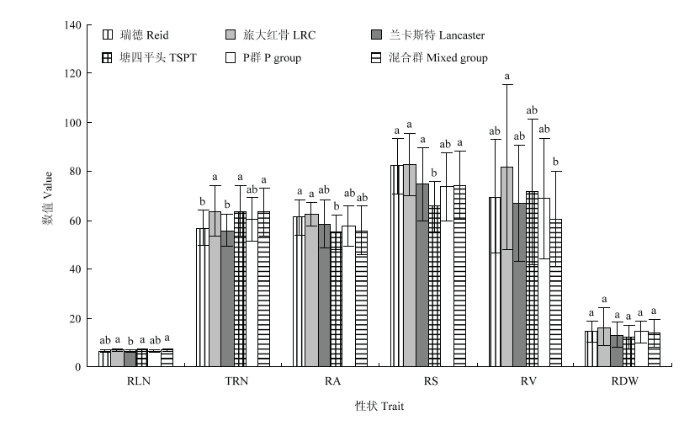 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5不同类群自交系地下节根相关性状的差异分析
Fig. 5Differences of belowground nodal root-related traits in inbred lines of different heterotic groups
2.5 地下节根相关性状全基因组关联分析
采用FarmCPU模型对4个环境下的111份玉米自交系地下节根相关性状的平均值进行全基因组关联分析,在P<0.00001水平上,判定SNP标记与研究性状关联的显著性,共检测到26个显著关联的SNP位点(图6和表4)。其中,检测到6个地下节根层数SNP位点,分别位于第1、6、8染色体上;4个地下节根总数SNP位点,分别位于第4、6染色体上;10个地下节根体积SNP位点,分别位于第3、4、6、8、9、10染色体上;6个地下节根干重SNP位点,分别位于第3、4、6、7染色体上。图6
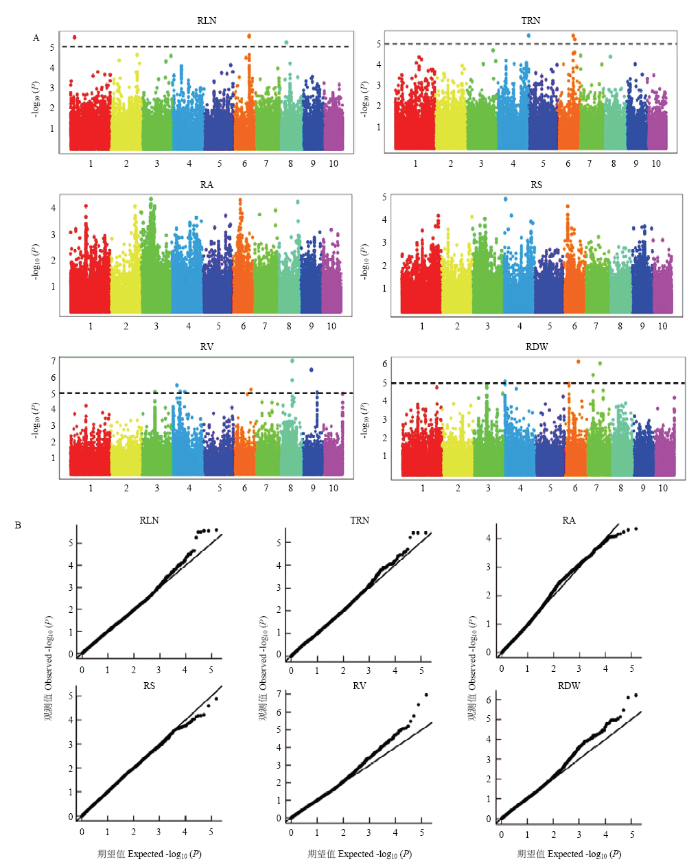 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6全基因组关联分析曼哈顿图(A)和QQ图(B)
Fig. 6(A) Manhattan plot and (B) Quantile-quantile plot for genome-wide association study of maize root traits
Table 4
表4
表4影响玉米地下节根相关性状的SNP及最可能的候选基因及其功能注释
Table 4
| 性状 Trait | SNP | Chr. | 位置a Position (bp) | P值 P value | 候选基因b Candidate gene | 基因功能注释 Gene annotation | 参考文献c Reference |
|---|---|---|---|---|---|---|---|
| RLN | Chr.1.S_31312069 | 1 | 31312069 | 3.07E-06 | Zm00001d028337 | 富含脯氨酸的受体样蛋白激酶PERK4 Proline-rich receptor-like protein kinase PERK4 | |
| RLN | Chr.1.S_31312087 | 1 | 31312087 | 3.07E-06 | Zm00001d028344 | 推定的金属耐受蛋白C3 Putative metal tolerance protein C3 | |
| RDW | Chr.3.S_103120493 | 3 | 103120493 | 1.07E-05 | Zm00001d041183 | 蛋白激酶超家族蛋白质/苏氨酸蛋白激酶prpf4B-liken Protein kinase superfamily proteiserine/threonine-protein kinase prpf4B-liken | |
| RV | Chr.3.S_104251070 | 3 | 104251070 | 8.33E-06 | Zm00001d041192 | 蔗糖转运蛋白4 Sucrose transporter4 | |
| RDW | Chr.4.S_9975093 | 4 | 9975093 | 7.24E-06 | Zm00001d048943 | 硫氧还蛋白超家族蛋白 Thioredoxin superfamily protein | [14] |
| RV | Chr.4.S_38214896 | 4 | 38214896 | 3.38E-06 | Zm00001d049623 | SNARE相关蛋白 SNARE-associated protein-related | |
| Zm00001d049627 | 蛋氨酸氨肽酶 Methionine aminopeptidase | ||||||
| RV | Chr.4.S_63797065 | 4 | 63797065 | 7.79E-06 | Zm00001d050069 | 海藻糖-6-磷酸合成酶8 Trehalose-6-phosphate synthase8 | |
| RV | Chr.4.S_100804613 | 4 | 100804613 | 8.30E-06 | Zm00001d050558 | OSJNBa0043A12.20蛋白 OSJNBa0043A12.20 protein | |
| TRN | Chr.4.S_246664102 | 4 | 246664102 | 3.96E-06 | Zm00001d054104 | E3泛素蛋白连接酶UPL3 E3 ubiquitin-protein ligase UPL3 | |
| Zm00001d054105 | 尿苷激酶样蛋白3 Uridine kinase-like protein 3 | ||||||
| RDW | Chr.6.S_34950886 | 6 | 34950886 | 9.19E-06 | Zm00001d035587 | G型凝集素S受体样丝氨酸/苏氨酸蛋白激酶At2g19130 G-type lectin S-receptor-like serine/threonine-protein kinase At2g19130 | [5,20] |
| Zm00001d035588 | G型凝集素S受体样丝氨酸/苏氨酸蛋白激酶At2g19130 G-type lectin S-receptor-like serine/threonine-protein kinase At2g19130 | ||||||
| RDW | Chr.6.S_108781517 | 6 | 108781517 | 6.09E-07 | Zm00001d037004 | Ras相关蛋白RABA1d Ras-related protein RABA1d | [17] |
| Zm00001d037010 | 毛发相关蛋白激酶iota Shaggy-related protein kinase iota | ||||||
| RLN/TRN | Chr.6.S_123079904 | 6 | 123079904 | 4.00E-06 | Zm00001d037360 | 推定的bZIP转录因子超家族蛋白 Putative bZIP transcription factor superfamily protein | [20] |
| RLN/TRN | Chr.6.S_123079927 | 6 | 123079927 | 4.00E-06 | Zm00001d037368 | 未知 Unknown | [20] |
| RLN | Chr.6.S_123845438 | 6 | 123845438 | 2.51E-06 | Zm00001d037384 | 花青素3-O-葡糖基转移酶 Anthocyanidin 3-O-glucosyltransferase | [20] |
| TRN | Chr.6.S_135051799 | 6 | 135051799 | 6.13E-06 | Zm00001d037712 | MAR结合丝状蛋白1 MAR-binding filament-like protein 1 | |
| RV | Chr.6.S_136591446 | 6 | 136591446 | 6.19E-06 | Zm00001d037751 | 晚期胚胎发育丰富(LEA)富含羟脯氨酸的糖蛋白家族 Late embryogenesis abundant (LEA) hydroxyproline-rich glycoprotein family | |
| RDW | Chr.7.S_48142656 | 7 | 48142656 | 3.42E-06 | Zm00001d019648 | 核酸结合蛋白1 Nucleic acid binding protein 1 | [14] |
| RDW | Chr.7.S_101804639 | 7 | 101804639 | 7.71E-07 | Zm00001d020242 | 锌指CCCH结构域的蛋白质64 Zinc finger CCCH domain-containing protein 64 | [14] |
| RLN | Chr.8.S_55681954 | 8 | 55681954 | 5.47E-06 | Zm00001d009324 | AMSH样泛素蛋白硫酯酶3 AMSH-like ubiquitin thioesterase 3 | |
| RV | Chr.8.S_100196991 | 8 | 100196991 | 1.68E-06 | Zm00001d010119 | 冷休克蛋白CS66 Cold shock protein CS66 | [20] |
| RV | Chr.8.S_100680141 | 8 | 100680141 | 1.07E-07 | Zm00001d010128 | 酸性核糖体蛋白P2b(rpp2b) Acidic ribosomal protein P2b (rpp2b) | [20] |
| RV | Chr.9.S_67485696 | 9 | 67485696 | 3.82E-07 | Zm00001d046152 | 推定的RING-H2指蛋白ATL71 Putative RING-H2 finger protein ATL71 | |
| RV | Chr.9.S_111860806 | 9 | 111860806 | 9.44E-06 | Zm00001d046942 | 果糖-1,6-二磷酸酶 Fructose-1,6-bisphosphatase | |
| RV | Chr.10.S_150110617 | 10 | 150110617 | 1.08E-05 | Zm00001d026687 | 生长素响应因子17 Auxin response factor 17 | [14] |
| Zm00001d026694 | 生长素响应因子13 Auxin response factor 13 |
新窗口打开|下载CSV
2.6 地下节根相关性状候选基因分析
该群体的LD分析结果显示,r2衰减到0.2时群体的连锁不平衡衰减距离为250 kb(图7)。对关联分析得到的显著关联的SNP位点进行整理后,利用MaizeGDB网站在显著关联位点的LD范围内查找玉米地下节根相关性状的候选基因,并对候选基因进行统计。26个控制节根性状的基因组区段内共鉴定出177个候选基因,其中,具有功能注释的135个候选基因的功能富集分析表明,40个候选基因主要参与植物体内的代谢调节,13个候选基因与植物应激反应中涉及的生物途径相关,57个候选基因的功能未富集到结果,其余候选基因功能涉及运输活性、催化活性、结合蛋白及细胞成分等(图8)。根据每个显著关联SNP的LD范围内所有候选基因的功能注释,选择与目标性状相关的候选基因及其注释列于表4。图7
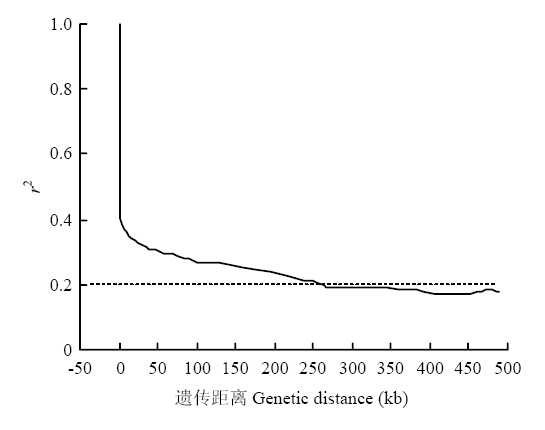 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7群体连锁不平衡衰减距离(LD)
Fig. 7Linkage disequilibrium attenuation distance of the population
图8
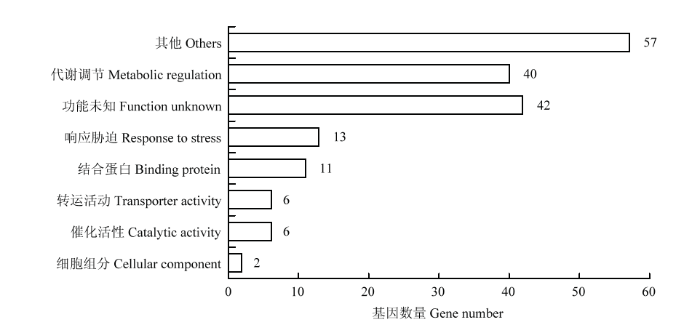 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8候选基因生物学途径注释直方图
Fig. 8Histogram of annotations for biological pathways of candidate genes
3 讨论
3.1 不同年代玉米自交系地下节根相关性状的表型分析
优异种质资源是玉米品种遗传改良和杂种优势利用的基础,在中国玉米产业发展的不同阶段,玉米自交系的选育目标不断改变。随着不同育种时期玉米地上部性状的改良,地下部根系结构可能被间接的选择,可能导致不同年代的玉米自交系根系性状间存在着差异。本研究通过对不同年代玉米自交系地下节根性状进行分析,结果表明,随着育种进程的推进,不同年代的玉米种质地下根系结构表现出了不同程度的差异,地下节根层数和总条数呈下降的趋势,地下节根角度和面积呈上升的趋势,根干重和根体积无显著差异。前人研究表明,从1980s到2000s随着品种的更替,玉米根系性状中的根长、根条数、根干重均减小,植株通过减少根系的冗余从而减少营养物质的消耗[27,28]。玉米植株对氮素的吸收利用影响根系的构型及分布[29],20世纪90年代随着氮肥施用量的增加,根系对氮肥的吸收量增加,植物根系通过降低代谢消耗的成本来提高植物光合作用利用率[30],最终根系表现为减少冗余。与本研究得到的地下节根层数和总条数随年代更替减少的结果一致。
3.2 不同类群玉米自交系地下节根相关性状的表型分析
中国玉米种质类群主要划分为塘四平头群、旅大红骨群、瑞德群、兰卡斯特群和P群等[31]。已有研究表明,不同类群的苞叶相关性状、籽粒灌浆与脱水相关性状等存在显著差异[32,33]。然而,对不同类群的玉米自交系根系结构差异的研究鲜有报道。本研究发现不同类群的地下节根相关性状存在差异,旅大红骨类群地下节根层数和条数多且根系生物量大,属于“大根系”;瑞德类群地下节根条数较少;兰卡斯特类群地下节根数量少,角度、体积和生物量均较小;塘四平头类群地下节根角度和面积较小,分布紧凑;P群地下节根数量、体积、干重等均处于中等水平。不同类群地下节根性状的差异,可为根系结构遗传改良提供参考信息。本研究中玉米开花期地下节根的6个性状之间均存在显著正相关或者负相关,说明各性状的发育存在着相互协同促进作用。因此,对玉米根系的研究不应局限于简单的表型分析,在今后的研究中应将玉米根系结构的遗传变异与产量形成之间的关系放在首要位置,解析玉米根系结构的遗传机制。3.3 地下节根相关性状的全基因组关联分析
玉米是异花授粉作物,其LD衰减较快[34],有利于利用关联分析方法进行复杂数量性状的遗传解析。本研究采用FarmCPU模型进行全基因组关联分析,共检测到26个与玉米地下节根相关性状(地下节根层数、总条数、体积和干重)显著关联的SNP位点,地下节根角度和地下节根面积没有检测到显著关联的SNP。在这些显著关联的标记中,存在一因多效现象,位于第6染色体上的Chr.6.S_123079904和Chr.6.S_123079927标记同时与地下节根层数和总条数相关联。本研究共检测到26个显著关联SNP,其中11个位于已定位的QTL置信区间内(表4)。标记Chr.6.S_108781517与地下节根干重显著关联,位于蔡红光等[20]检测到的控制根干重和根条数的QTL区间umc1257-umc1857内。标记Chr.6.S_34950886与地下节根干重相关联,位于CAI等[5]定位到的控制成熟期地下节根长度的区间bins 6.00—6.01内。位于第4、7、10染色体上显著关联标记Chr.4.S_9975093、Chr.7.S_48142656、Chr.7.S_101804639和Chr.10.S_ 150110617均定位于ZHANG等[17]利用BC2S3群体检测到的节根数相关性状QTL区间内。本研究中共有6个显著关联SNP落在GUO等[23]通过整合玉米根系相关性状QTL获得的Meta-QTL区段内。本研究获得的26个显著关联SNP共涉及177个候选基因,其中135个有功能注释的基因涉及到转导蛋白、金属离子转运蛋白、调节蛋白、蛋白激酶等,功能涉及能量代谢、物质运输、生物调控等生物学途径。候选基因Zm00001d037004为Ras相关蛋白RABA1d,与根毛生长有关;Zm00001d037010为与毛发相关的蛋白激酶;将候选基因与通过MaizeGDB查找到的已知与玉米根系相关的基因进行比对,发现Zm00001d037751的功能与NCBI网站上收录的一些玉米根系相关基因的功能相关,已知根系相关的基因Zm00001d003807为LEA相关蛋白质,Zm00001d037751属于LEA富含羟脯氨酸的糖蛋白家族;值得注意的是,与地下节根总条数和层数相关的显著关联位点Chr.6.S_123079904和Chr.6.S_123079927均落在Zm00001d037368的启动子区域,Zm00001d037368的功能未知;ZmSUT2(Zm00001d041192)促进玉米植株的生长发育及产量的形成,zmsut2突变体植株较野生型植株生长缓慢、雄穗和雌穗较小、产量低[35]。因此,ZmSUT2可能与根系发育相关。
4 结论
不同年代的玉米自交系地下根系存在不同程度的差异,地下节根层数和总条数在年代的更替间表现出下降的趋势,地下节根角度和面积在年代的更替间表现出上升的趋势,根干重和根体积在各年代间无显著差异。玉米自交系的根系结构在不同类群间存在差异,旅大红骨群的地下节根层数、条数、角度、面积、体积和干重均高于其余类群。利用高密度SNP标记对玉米根系的6个性状进行GWAS分析,共检测到26个显著关联的SNP位点,涉及177个候选基因,其中135个有功能注释。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1093/aob/mcs293URL [本文引用: 1]
DOI:10.1016/j.tplants.2014.02.002URL [本文引用: 1]
[本文引用: 1]
DOI:10.2135/cropsci2008.03.0152URL [本文引用: 2]
[本文引用: 5]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s11104-015-2554-xURL [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
DOI:10.1016/S2095-3119(13)60243-9URL [本文引用: 1]
[本文引用: 2]
DOI:10.1111/tpj.2018.93.issue-6URL [本文引用: 4]
DOI:10.1007/s10681-015-1533-xURL [本文引用: 1]
[本文引用: 1]
DOI:10.11674/zwyf.2011.0179URLMagsci [本文引用: 2]

在田间原位条件下,利用根系形态差异显著的自交系掖478和武312为亲本构建的BC4F3群体,采用改进的PLABQTL软件中的复合区间作图法对抽雄期(开花前10 d)和灌浆初期(开花后15 d)玉米根系性状的变化和地上部生物量进行QTL定位。并分析其遗传机制。结果表明,花前花后对根干重、总根长、侧根长、轴根长、轴根数等根系性状共检测出27个QTL,单个QTL贡献率为52%157%,其中在染色体臂602和1004区域同时检测到控制着地上部生物量、总根长、侧根长和轴根数等性状的QTLs,两个不同生育时期检测到的共同QTL共有8个。玉米花前花后控制根系生长的QTL因生长发育阶段不同而存在着特异性,而且对地上部生物量形成有重要贡献,这为了解田间条件下根系的生长发育和进一步进行遗传改良奠定了遗传基础。
DOI:10.11674/zwyf.2011.0179URLMagsci [本文引用: 2]

在田间原位条件下,利用根系形态差异显著的自交系掖478和武312为亲本构建的BC4F3群体,采用改进的PLABQTL软件中的复合区间作图法对抽雄期(开花前10 d)和灌浆初期(开花后15 d)玉米根系性状的变化和地上部生物量进行QTL定位。并分析其遗传机制。结果表明,花前花后对根干重、总根长、侧根长、轴根长、轴根数等根系性状共检测出27个QTL,单个QTL贡献率为52%157%,其中在染色体臂602和1004区域同时检测到控制着地上部生物量、总根长、侧根长和轴根数等性状的QTLs,两个不同生育时期检测到的共同QTL共有8个。玉米花前花后控制根系生长的QTL因生长发育阶段不同而存在着特异性,而且对地上部生物量形成有重要贡献,这为了解田间条件下根系的生长发育和进一步进行遗传改良奠定了遗传基础。
DOI:10.1007/s11032-011-9655-xURL [本文引用: 1]
DOI:10.2135/cropsci2016.01.0031URL [本文引用: 1]
DOI:10.1007/s10681-018-2283-3 [本文引用: 2]
[本文引用: 1]
DOI:10.1371/journal.pgen.1005767URL [本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2012.11.001URLMagsci [本文引用: 1]

【目的】选择有代表性的材料进行遗传多样性与群体结构的解析是进行等位基因发掘、复杂性状关联 分析、研究,及作物育种实践的重要基础。【方法】利用覆盖玉米全基因组的40 个核心SSR 标记,采用基于测序 的基因型鉴定技术对820 份代表中国玉米育种资源种质基础的自交系进行全基因组扫描,通过PowerMarker V3.25 与Structure V2.3.3 等软件揭示其基因多样性与群体结构。【结果】在820 份自交系中,40 个SSR 标记所检测到 的等位变异为10—72 个,平均36.87 个/位点;基因多样性为0.46—0.9458,平均0.8430;PIC 为0.43—0.94, 平均0.83。聚类分析表明,K=5 时,△K 值最大,即这些自交系可以划分成5 个类群,依次为兰卡斯特、旅大红 骨、塘四平头、瑞德与P 群,各类群内平均等位变异与遗传多样性分别为24.23 个/位点与0.8145、22 个/位点与 0.8398、11.8 个/位点与0.7054、17.45 个/位点与0.7686 以及14.65 个/位点与0.7495。【结论】中国玉米自交 系在育种实践中形成了相对独立的优势类群,蕴含了比较丰富的遗传变异,显示出了较高水平的基因多样性。不 同类群之间的遗传多样性水平存在一定的差异,在划分的5 个类群中,兰卡斯特与旅大红骨类群的遗传变异相对 较丰富,基因多样性相对较高,其次为瑞德与P 群,塘四平头相对较低。
DOI:10.3864/j.issn.0578-1752.2012.11.001URLMagsci [本文引用: 1]

【目的】选择有代表性的材料进行遗传多样性与群体结构的解析是进行等位基因发掘、复杂性状关联 分析、研究,及作物育种实践的重要基础。【方法】利用覆盖玉米全基因组的40 个核心SSR 标记,采用基于测序 的基因型鉴定技术对820 份代表中国玉米育种资源种质基础的自交系进行全基因组扫描,通过PowerMarker V3.25 与Structure V2.3.3 等软件揭示其基因多样性与群体结构。【结果】在820 份自交系中,40 个SSR 标记所检测到 的等位变异为10—72 个,平均36.87 个/位点;基因多样性为0.46—0.9458,平均0.8430;PIC 为0.43—0.94, 平均0.83。聚类分析表明,K=5 时,△K 值最大,即这些自交系可以划分成5 个类群,依次为兰卡斯特、旅大红 骨、塘四平头、瑞德与P 群,各类群内平均等位变异与遗传多样性分别为24.23 个/位点与0.8145、22 个/位点与 0.8398、11.8 个/位点与0.7054、17.45 个/位点与0.7686 以及14.65 个/位点与0.7495。【结论】中国玉米自交 系在育种实践中形成了相对独立的优势类群,蕴含了比较丰富的遗传变异,显示出了较高水平的基因多样性。不 同类群之间的遗传多样性水平存在一定的差异,在划分的5 个类群中,兰卡斯特与旅大红骨类群的遗传变异相对 较丰富,基因多样性相对较高,其次为瑞德与P 群,塘四平头相对较低。
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1104/pp.114.241711URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
