 ,1,2, 刘兴3, 辛正琦1,2, 谢海艳1,2, 辛余凤1,2, 吴能表
,1,2, 刘兴3, 辛正琦1,2, 谢海艳1,2, 辛余凤1,2, 吴能表 ,1,2,*
,1,2,*Molecular cloning, expression, and enzyme kinetic analysis of a phenylalanine ammonia-lyase gene in Pinellia ternate
HE Xiao ,1,2, LIU Xing3, XIN Zheng-Qi1,2, XIE Hai-Yan1,2, XIN Yu-Feng1,2, WU Neng-Biao
,1,2, LIU Xing3, XIN Zheng-Qi1,2, XIE Hai-Yan1,2, XIN Yu-Feng1,2, WU Neng-Biao ,1,2,*
,1,2,*通讯作者: *吴能表, E-mail:wunb@swu.edu.cn
收稿日期:2020-10-14接受日期:2021-03-19网络出版日期:2021-04-01
| 基金资助: |
Corresponding authors: *E-mail:wunb@swu.edu.cn
Received:2020-10-14Accepted:2021-03-19Published online:2021-04-01
| Fund supported: |
作者简介 About authors
E-mail:992651097@qq.com

摘要
本研究以半夏为研究对象, 基于其转录组数据克隆获得1个苯丙氨酸解氨酶基因, 命名为PtPAL, 通过DNAMAN、MEGA等生物信息学软件进行序列结构与系统进化分析表明, 该基因核酸序列长度为2289 bp, 编码762个氨基酸, 终止密码子为TAA, PtPAL与单子叶植物百合PAL相似度达78%, 具有PAL-HAL、PLN02457、phe_am_lyase、Lyase_aromatic及HutH结构域, 属于苯丙氨酸解氨酶家族成员(Lyase_I_like Superfamily)。半夏PtPAL与单子叶植物百合、菠萝和油棕亲缘关系较近, 归于单子叶植物。采用实时荧光定量PCR分析PtPAL在不同组织间的表达情况表明, PtPAL基因在叶中表达量最高, 其次是块茎和根, 花中表达量最低。通过对半夏PtPAL基因进行功能表达分析发现, PtPAL重组蛋白可以高效催化L-Phe生成t-CA, 且催化反应最适pH与温度分别为9.0、70℃; PtPAL的Km、Vmax、Kcat和Kcat/Km分别为0.89 mmol L-1、63.96 nKat mg-1、6.56 s-1和7.37×103 s-1 M-1。进一步探究金属离子对PtPAL酶活性的影响表明, Ba2+显著增强了PtPAL酶活性, Mn2+、Co2+、Cu2+及Zn2+抑制了PtPAL酶活性。本研究为进一步研究PtPAL的功能特点及半夏苯丙胺类生物碱代谢途径奠定基础。
关键词:
Abstract
In this study, based on the transcriptome data, a phenylalanine ammonia lyase gene was cloned and named PtPAL using Pinellia ternate as the experimental material. The analysis of sequence structure and systematic evolution revealed that the length of the gene was 2289 bp, encoding 762 amino acids, and the termination codon was TAA. PtPAL was the similarity of 78% compared with monocot Lilium regale PAL, with PAL-HAL, PLN02457, phe_am_lyase, Lyase_aromatic, and HutH domains that belonging to the Lyase_I_like superfamily. Phylogenetic tree analysis showed that PtPAL was closely related to the monocotyledon Lilium regale, Ananas comosusm, and Elaeis guineensis, and thus belonging to Moncotyledons. Real-time fluorescent qPCR indicated that the relative expression level of PtPAL gene was the highest in leaves, followed by tubers and roots, and the lowest in flowers. The functional expression analysis of PtPAL gene revealed that the PtPAL recombinant protein could efficiently catalyze L-Phe to t-CA, and the optimal pH and temperature of the reaction were 9.0℃ and 70℃, respectively. Km, Vmax, Kcat, and Kcat/Km of PtPAL were 0.89 mmol L -1, 63.96 nKat mg-1, 6.56 s-1, and 7.37×103 s-1 M-1. Further exploration of the effect of metal ions on PtPAL enzyme activity showed that Ba2+ could significantly enhance PtPAL enzyme activity, and Mn2+, Co2+, Cu2+, and Zn2+ inhibit the activity of PtPAL enzyme. This study lays the foundation for further research of the functional characteristics of PtPAL and the metabolic pathways of amphetamine alkaloids in Pinellia ternata.
Keywords:
PDF (9580KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
何潇, 刘兴, 辛正琦, 谢海艳, 辛余凤, 吴能表. 半夏PtPAL基因的克隆、表达与酶动力学分析[J]. 作物学报, 2021, 47(10): 1941-1952 DOI:10.3724/SP.J.1006.2021.04227
HE Xiao, LIU Xing, XIN Zheng-Qi, XIE Hai-Yan, XIN Yu-Feng, WU Neng-Biao.
半夏源于天南星科植物半夏(Pinellia ternate)的干燥块茎, 属于多年生草本植物, 是我国传统中药材, 其药用功效为燥湿化痰、降逆止呕、消痞散结。现代研究发现半夏含有众多化学成分, 主要包括生物碱、黄酮类、甾醇类、挥发油、芳香族成分、有机酸类、半夏蛋白、鞣质以及多种微量元素等, 其中, 生物碱为半夏药理作用的主要有效成分之一[1]。麻黄碱是半夏生物碱的主要活性成分, 属于苯丙胺类生物碱, 因具有止喘、增加心输出量、兴奋中枢神经系统的作用及升血压缓慢、温和的特点, 而具有较高的医学应用价值[2,3]。从代谢合成途径看, 植物麻黄碱生物合成属于苯丙氨酸代谢途径, 其前体物质为L-苯丙氨酸(L-Phe), 但其生物合成机制并未完全研究清楚, 因此, 研究苯丙烷类代谢途径, 对研究半夏麻黄碱合成代谢机制具有重要意义, 而研究代谢途径上的关键酶基因又是重要步骤之一。
苯丙氨酸解氨酶(phenylalanine ammonia-lyase, PAL)是催化苯丙烷类代谢第一步反应的酶, 催化L-苯丙氨酸(L-Phe)生成反式-肉桂酸(t-CA)和氨, 再进一步代谢转化为一系列苯丙素类化合物, 如黄酮体、木质素、生物碱等; 此外, 也是联系初级代谢和次级代谢的限速酶和关键酶[4]。自1961年Koukol和Conn在高等植物、1966年在微生物首次分离纯化PAL以来, 该酶引起了人们广泛研究, 至今已从裸子、被子、苔藓、蕨类等植物和真菌中分离得到大量的PAL家族基因[5]。目前针对PAL家族基因的结构、编码信息及功能已得到广泛研究。研究表明, PAL作为一个基因家族, 其数量因物种不同而存在差异[6,7]; PALDNA序列也相对保守, 在多数植物中仅由2个外显子和1个内含子组成; 其编码的蛋白酶分子量大小约为275~330 kD, 亚基分子量大小为55~88 kD[8], 多数植物PAL上包含PAL-HAL、PLN02457、phe_am_lyase和Lyase_aromatic保守结构域, 少部分植物PAL包含HutH结构域, 但PAL 酶活性位点主要位于PAL-HAL结构域上。PAL催化L-Phe脱氨生成反式肉桂酸(t-CA)属于非氧化还原反应。在大多数双子叶植物中, L-phe是PAL的最适底物, 而在一些单子叶植物和酵母中PAL还可以催化L-酪氨酸(L-Tyr)形成对香豆酸[9]。Yu等[10]在大肠杆菌中异源表达附生苔草(liverwort Plagiochasma appendiculatum) PAL重组蛋白表明, PaPAL催化L-Phe转化为t-CA具有较高的酶活性, 而在催化L-Tyr形成对香豆酸时表现出的酶活性较低。Ma等在对五倍子RcPAL的最适底物研究中发现, 将Phe126突变为His126后, 导致RcPAL酶活性降低了75%, 但TAL酶活性却增加了22倍, 这进一步为L-Phe是PAL的最适底物提供了直接证据。PAL家族基因的表达一方面受植物体内自身代谢产物的反馈调节, 一方面受外界因子如病原菌、温度、重金属、机械损伤、及外源植物激素等生物和非生物胁迫的诱导。高浓度t-CA抑制了银杏[11]和山药(Dioscoreae rhizoma)[12]PAL活性, 一定浓度的阿魏酸抑制了山药PAL活性。玉米丝黑穗病菌可以导致玉米PAL活性升高[13], 美洲南瓜(Cucurbita pepo) PAL受灰霉病菌诱导表达[14]; PAL在黄芩中参与胁迫诱导黄酮类化合物的合成[15]。外源喷施植物激素茉莉酸甲酯(methyl jasmonate, MeJA)、脱落酸(abscisic acid, ABA)及水杨酸(salicylic acid, SA)后, 植物PAL表达量也增加[16,17]。尽管目前PAL家族基因已经在多类植物中得到克隆分析, 但半夏中尚未见报道。本研究以半夏为试验材料, 根据半夏转录组测序, 通过RT-PCR方法首次成功克隆得到半夏PtPAL基因, 经过生物学信息分析、组织表达分析以及酶动力学分析, 旨在为后续深入研究PtPAL基因功能以及研究半夏麻黄碱代谢途径奠定基础。
1 材料与方法
1.1 植物材料
供试半夏块茎购自湖北荆州, 经西南大学生命科学学院吴能表教授鉴定为半夏(P. ternate)。选取生长健壮且大小一致的半夏块茎, 经自来水流水冲洗干净后, 种植于盛有河沙的培养盆里进行萌芽, 期间保持河沙湿润。待萌芽后, 挑选长势基本一致的幼苗移栽至盛有沙质土壤(土壤∶河沙=3∶1)的营养钵中, 每钵3株, 培养至60 d用于试验分析。培养条件: 温度25℃, 光照强度为80~220 μmol m-2 s-1, 置于光照时间16 h/8 h (光/暗)人工气候室进行培养。取60 d龄半夏幼苗的根、块茎、叶和花于液氮中迅速冷冻, 置于-80℃冰箱冻存备用, 用于半夏组织特异性分析。1.2 试剂
Biospin多糖多酚植物总RNA提取试剂盒、Dnase I酶购于北京贝洛生物科技有限公司; RNA PCR Kit (AMV) ver 3.0逆转录试剂盒、Ex Taq聚合酶、PrimeSTAR Max高保真酶、pMD19-T Vector载体均购于TaKaRa (大连)有限公司; DNA琼脂糖凝胶回收试剂盒、质粒提取试剂盒均购于Axygen公司; Hiafair II 1st Strand cDNA Synthesis Super Mix for qPCR逆转录试剂盒、Hieff TM qPCR SYBR Green Master Mix试剂盒均购于上海翊圣生物科技有限公司; 由Invitrogen公司合成PCR引物; 由擎科生物技术有限公司完成DNA测序。1.3 半夏总RNA提取和cDNA合成
取适量半夏组织样品于液氮中研磨成细粉, 按照Biospin多糖多酚植物总RNA提取试剂盒(Cat#BSC65S1)的使用说明提取半夏总RNA, 用1.0%琼脂糖凝胶电泳检测RNA完整性, 并用Thermo Nanodrop 2000C超微量分光光度计检测RNA的浓度和纯度, 合格RNA置于-80℃保存备用。参照TaKaRa RNA PCR Kit (AMV) 3.0 (RR019A)反转录试剂盒说明书, 以提取的总RNA为模板, 反转录合成第1条互补链cDNA。1.4 半夏PtPAL基因扩增和克隆
在半夏转录组数据库中本地BLASTP PtPAL基因中, 以拟南芥模式植物的PAL基因家族保守序列为参考, 筛选出1个含完整读码框的候选unigene基因并命名为PtPAL。利用Primer Premier 5.0软件设计PtPAL 基因特异性引物(表1)。以半夏cDNA为模板, 进行PCR扩增。PCR产物经1.0%琼脂糖凝胶电泳检测后, 切胶回收与pMD19-T vector连接, 转化大肠杆菌DH5α感受态细胞中, 菌落经PCR检测为阳性后, 送擎科生物科技有限公司测序。Table 1
表1
表1本研究所用引物
Table 1
| 引物用途 Primer function | 引物名称 Primer name | 引物序列 Primer sequence (5′-3′) |
|---|---|---|
| 基因克隆 Gene cloning | PtPAL-F PtPAL-R | CACCTAAACCTTCTTCCGCAG GATCTTACTTGTTACTGGTTCCAAT |
| 荧光定量PCR Fluorescence quantitative PCR | PtPAL-qF PtPAL-qR PtGAPDH-qF PtGAPDH-qR | CCAACATCCTGGCTCTGCTCTC GTCCTGCTTCGGCTTCGTCA TCGTCTTGAGAAATCGGCGAC GTCAAAGATGCTCGACCGCT |
| 原核表达载体构建 Construction of prokaryotic expression vector | PtPAL-Hind III-F PtPAL-Xho I-R | CGCAAGCTTTTATGGCGGCCAAGTCGAACGGTCT CGCCTCGAGTTACTGGTTCCAATAACCCCTT |
新窗口打开|下载CSV
1.5 半夏PtPAL基因生物学信息分析
利用DNAMAN 8.0软件将测序所得的编码区序列翻译成氨基酸序列, 在InterProScan软件中进行结构域分析和ORF Finder查找PtPAL基因的完整开放阅读框(open reading frame, ORF)。采用ExPASy提供的在线工具ProtParam预测PtPAL蛋白的理化性质。在SOPMA软件上进行二级结构预测。采用SWISS-MODEL进行蛋白质三级结构和四级结构同源建模。根据NCBI BLASTP的比对结果, 找出同源性较高的其他物种的氨基酸序列, 在DNAMAN 8.0软件上进行氨基酸多重序列比对, 并用MEGA 5.05软件采用邻接法(neighbor joining method, NJ) (bootstrap 1000)构建系统进化树。1.6 实时荧光定量PCR (qRFPCR)
同上方法提取半夏叶、块茎、根和花的总RNA, 参照Hiafair II 1st Strand cDNA Synthesis Super Mix for qPCR反转录试剂盒说明书反转录合成cDNA, 同时进行qPCR反应。根据PtPAL基因序列设计荧光定量PCR引物(表1), 以半夏PtGAPDH为内参基因, 使用Bio-Rad IQ5实时荧光定量PCR仪进行反应检测。所有处理均设3个生物学重复, 根据2-ΔΔCt法分析基因的相对表达量, 使用SPSS 22.0软件分析显著性, 采用GraphPad Prism 7.0软件绘图。1.7 半夏PtPAL基因原核表达
通过对PtPAL基因编码区序列的限制酶切位点和pET32a(+)原核表达载体上的多克隆酶切位点进行SnapGene软件分析, 选择Hind III和Xho I作为酶切位点进行载体构建, 引物序列见表1。将pET32a(+)-PtPAL重组载体转化大肠杆菌BL21 (DE3)进行诱导表达, 经SDS-PAGE凝胶电泳检测分析蛋白表达情况; 根据Proteinlso Ni-NTA Resin (北京全式金生物技术有限公司)说明书进行半夏PtPAL重组蛋白Ni柱纯化, 采用考马斯亮蓝法(Bradford法), 按照Easy Protein Quantitative Kit (北京全式金生物技术有限公司)的操作说明绘制标准曲线并测定蛋白浓度。1.8 半夏PtPAL重组蛋白酶活性鉴定
配置不同pH值的0.1 mol L-1硼酸缓冲液和100 μL 0.01 mol L-1 L-Phe (溶于0.1 mol L-1硼酸缓冲液, pH 9.0), 对照组为1 mL 0.1 mol L-1硼酸缓冲液(pH 9.0)进行PtPAL重组蛋白催化反应最适pH测定, 于紫外可见分光光度计290 nm处测定其吸光值; 在上述最适pH值条件下, 测定PtPAL重组蛋白在不同温度下的OD290值。每个样品重复3次, 分别制作酶活性随pH、温度变化曲线, 将最高酶活定义为100%,计算不同pH与温度下PtPAL的相对活力。在0.1 mol L-1硼酸缓冲液(pH 9.0)中, 通过加入不同浓度的L-Phe为底物, 加入100 μL (约10 μg) PtPAL重组蛋白, 总体积为1 mL, 在70℃和37℃下反应5 min, 测定PtPAL酶活力。2 结果与分析
2.1 半夏PtPAL基因克隆
根据拟南芥、玉米及烟草等植物基因编码的氨基酸序列, 利用BioEdit软件进行本地BLASTP检索, 从半夏转录组数据库中筛选出1个含完整读码框的候选unigene基因, 再利用NCBI中的BLASTX进行序列比对分析, 确定其为半夏PtPAL基因。根据PtPAL基因序列设计出特异性引物, 以半夏叶和块茎的cDNA为模板, 进行扩增, 得到一条主带, 大小为2289 bp, 与预期目的条带大小一致。2.2 半夏PtPAL基因生物信息学分析
2.2.1 半夏PtPAL核酸序列分析 经分析测序获得半夏PtPAL基因的核酸序列(图1)。PtPAL基因大小为2289 bp, 编码762个氨基酸, 终止密码子为TAA, 预测蛋白分子量为83.44 kD, 蛋白理论等电点(pI)为6.42。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1半夏PtPAL 基因全长CDS核酸及氨基酸序列
编码区核酸序列和翻译的氨基酸序列用大写字母表示。终止密码子用*表示, 起始密码子和终止密码子用横线表示。
Fig. 1Full length CDS nucleotide sequence and amino acids sequence of PtPAL in Pinellia ternate
The coding sequence and the deduced amino acid sequence are shown in uppercase letters in bold. Termination code is denoted by the asterisk; initiation code and termination code are denoted by underlines.
2.2.2 半夏PtPAL氨基酸序列多重比对与系统进化树分析 通过NCBI的BLASTR对PtPAL氨基酸序列进行在线同源比对分析发现, PtPAL与大多数植物的PAL氨基酸序列具有较高的相似性, 相似度达58%~78%。从中选择相似度较高的百合、葡萄和银杏等植物的PAL氨基酸序列与PtPAL氨基酸序列进行多重比对(图2)发现, 半夏PtPAL与单子叶植物百合PAL氨基酸序列相似度最高, 为78%; 其次, 与双子叶植物葡萄PAL氨基酸序列相似度也较高, 为75%; 与裸子植物银杏、蕨类植物问荆以及苔藓植物背苔的相似度分别为58%、66%、59%。然后, 对PtPAL做进一步的保守结构域和活性位点分析表明, PtPAL具有PAL-HAL、PLN02457、phe_am_lyase、Lyase_aromatic及HutH结构域, 属于解氨酶类家族(Lyase_I_like Superfamily)。研究发现, PtPA具有保守的酶活性中心序列(GTITASGDLVPLSYIAG, 209~ 225 aa), 其中包含高度保守的Ala-Ser-Gly氨基酸三联体(213~215 aa), 除与百合存在1个氨基酸残基差异外, 与拟南芥、葡萄、银杏和白及等植物的酶活性中心序列一致[17]。此外, PtPAL还含有保守活性位点(Y124、F151、L152、N270、Q358、Y361、R364、F410、Q497), 与背苔和欧芹报道中的一致[18], 说明PtPAL是苯丙氨酸解氨酶家族成员。
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2不同植物PAL氨基酸序列多重比对
一致氨基酸残基用白色字体黑色背景表示, 保守氨基酸残基用黑色字体红色背景表示。保守的Ala-Ser-Gly催化三联体用表示实心三角, 其他保守活性位点用空心三角表示, 保守活性基序用红色方框表示。AtPAL: 拟南芥(NP_187645.1); VvPAL: 葡萄(RVW44079.1); LrPAL: 百合(ASV46344.1); GbPAL: 银杏(ABU49842.1); EaPAL: 问荆(AAW80639.1); PaPAL: 背苔(AIU99853.1)。
Fig. 2Multiple alignment of amino acid sequences of PAL in different plants
The identical amino acids are shown in white with black background, the conserved amino acids are shown in black with red background. The conserved active site motif (Ala-Ser-Gly) is indicated by filled triangles under the sequences, the other active site residues are indicated by open triangle asterisk, the conserved active site motif is denoted by red box. AtPAL: Arabidopsis thaliana (NP_187645.1); VvPAL: Vitis vinifera (RVW44079.1); LrPAL: Lilium regale (ASV46344.1); GbPAL: Ginkgo biloba (ABU49842.1); EaPAL: Equisetum arvense (AAW80639.1); PaPAL: Plagiochasma appendiculatum (AIU99853.1).
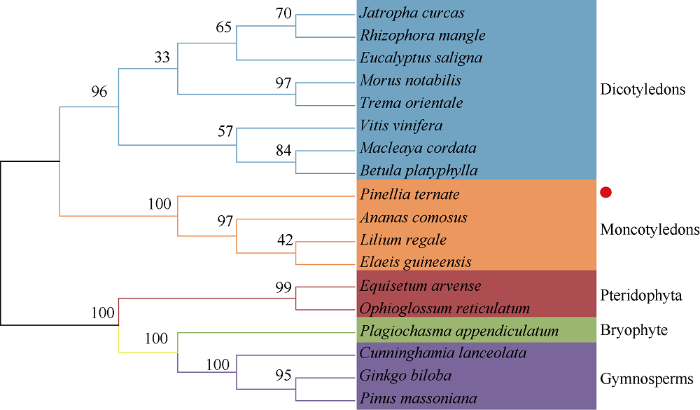
本研究从NCBI中收集到17条完整的PAL氨基酸序列, 其中包括麻风树、油棕、马尾松及白桦等植物, 通过MEGA 5.05软件中Clustal W进行氨基酸序列比对, 采用Neighbor-Joining法构建系统进化树, 对半夏进行聚类分析。结果发现, 可大致将来源于18种不同物种的PAL氨基酸序列分为5类(图3), 分别为双子叶植物、单子叶植物、蕨类植物、苔藓植物以及裸子植物; 本研究中克隆的半夏PtPAL归于单子叶植物一类, 且与单子叶植物菠萝、百合和油棕的PAL氨基酸序列遗传距离最短。
图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3半夏PtPAL和其他植物PAL系统进化树
各节点处数值表示Bootstrap值百分比(重复1000次)。半夏PtPAL用红色点表示。
Fig. 3Phylogenetic tree analysis of PtPAL and PAL from other plants
The number of each interior Branch is the percentage of bootstraps value (1000 replicates). PtPAL is denoted by red dot.
2.2.3 半夏PtPAL蛋白结构预测 运用SOPMA对半夏PtPAL蛋白的二级结构进行预测发现, PtPAL包含53.41% α-螺旋(alpha helix)、6.43% β-转角(beta turn)、8.53%延伸链(extended strand)和31.63%无规则卷曲(random coil), 表明PtPAL蛋白主要以α-螺旋为形式的二级结构。
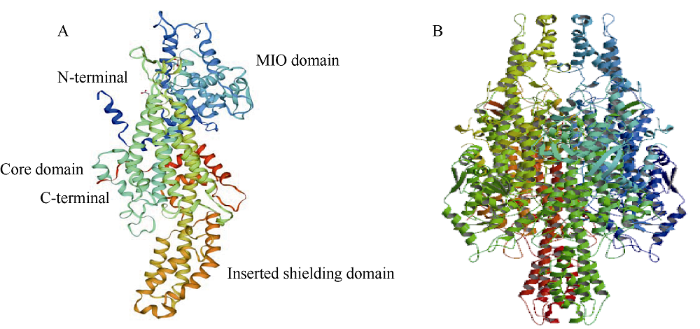
采用同源建模的方法, 在SWISS-MODEL在线软件中进行PtPAL蛋白的三维结构建模, PtPAL蛋白以双子叶植物欧芹PcPAL (PDB:1w27)[19]的晶体结构为模版, 如图4所示。建模覆盖范围为70~713位氨基酸残基, PtPAL与PcPAL氨基酸序列同源性为61.79%。PtPAL的单个亚基呈“海马”状, 由MIO域(MIO domain)、中心区域(core domain)及可插入屏蔽区域(inserted shielding domain) 3个部分组成(图4-A)。已有研究表明, PAL是由4个相同亚基构成的同源四聚体蛋白; 而本研究中同源建模分析结果显示, PtPAL与欧芹PcPAL均是由4个相同亚基构成的同源四聚体, 且每个亚基与其他2个亚单位以头尾相连的方式结合, 从而增加相邻亚基间的相互作用, 产生紧密结合地同源四聚体(图4-B)。另外, PtPAL含有植物中高度保守的Ala-Ser-Gly (ASG)氨基酸三联体, ASG氨基酸三联体可以通过自身环化脱水而自动转化为MIO作为PAL的重要辅助因子发挥作用, 进一步说明PtPAL属于苯丙氨酸解氨酶家族。
图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4PtPAL蛋白结构预测
A: 单体; B: 同源四聚体。MIO: 3,5-二氢-5-亚甲基-4H-咪唑-4-酮。
Fig. 4Deduced protein structure of PtPAL
A: monomer; B: homotetramer. MIO: 3,5-dihydro-5-methylene-4H-imidazol-4-one.
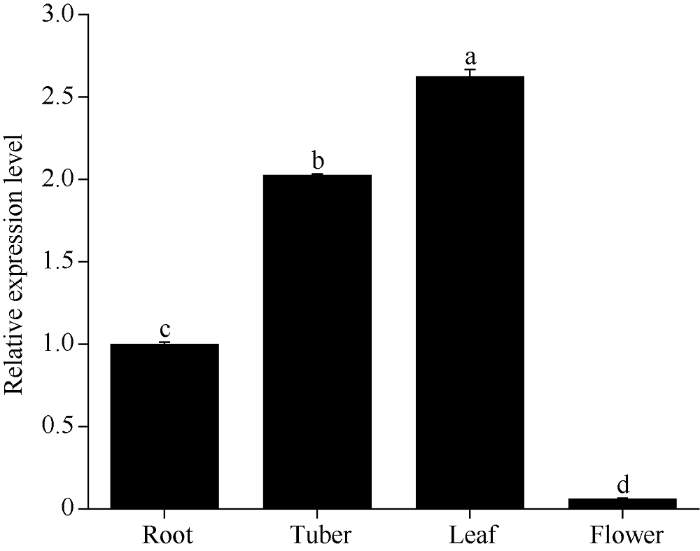
2.3 半夏PtPAL基因组织表达分析
利用实时荧光PCR技术(qRTPCR), 以半夏PtGAPDH作为内参基因, 对基因PtPAL进行组织表达谱分析(图5): 在转录水平上, PtPAL基因在叶中大量表达, 其次在块茎、根中有少量表达, 在花中表达量相对较微弱。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5不同组织中半夏PtPAL基因表达分析
不同小写字母表示不同组织间在0.05水平差异显著。
Fig. 5Relative expression levels of PtPAL in different tissues
Values with different lowercase letters show significant difference among different tissues at the 0.05 probability level.
2.4 半夏PtPAL基因原核表达和分离纯化
以pET32a(+)载体作为原核表达载体, 通过在大肠杆菌BL21 (DE3)中进行异源表达PtPAL重组蛋白, 获得分子量大小(MW)为102.3 kD的融合His标签的PtPAL重组蛋白。将BL21-pET32a(+)-PtPAL工程菌诱导, 经SDS-PAGE凝胶电泳检测, 结果如图6-A所示: 在1 h时, PtPAL在102 kD大小位置上有明显加粗的条带; 随着诱导时间的延长, 条带逐渐加粗。说明有目的蛋白产生且随诱导时间逐渐增加, 表明PtPAL重组蛋白可以被诱导。采用Ni-NTA亲和层析柱纯化, 用不同咪唑浓度的PBS平衡缓冲液进行洗脱, 收集蛋白, 经SDS-PAGE凝胶电泳检测发现, PtPAL重组蛋白在150 mmol L-1咪唑浓度洗脱时, 目的蛋白较多而杂蛋白较少且纯化的PtPAL重组蛋白约为102.3 kD (图6-B)。图6
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6PtPAL重组蛋白时间梯度诱导表达和融合His标签PtPAL重组蛋白纯化
M为蛋白marker。A: PtPAL重组蛋白时间梯度诱导表达; B: 融合His标签PtPAL重组蛋白纯化。
Fig. 6Time gradient induced of the recombinant PtPAL and its purified His-tagged PtPAL
M: protein molecular weight standards. A: time gradient induced of reconstructive PtPAL; B: the purified His-tagged PtPAL.
2.5 半夏PtPAL重组蛋白活性鉴定
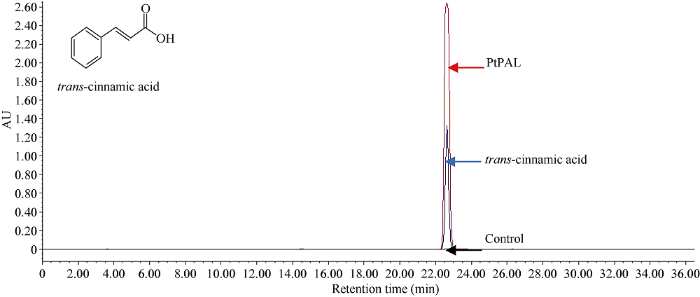
2.5.1 半夏PtPAL重组蛋白功能鉴定 已知PAL催化L-Phe脱氨形成反式肉桂酸(t-CA)。通过LC-MS检测PtPAL反应对照样品、反应样品和反式肉桂酸的分析标准品(图7)发现, 蓝色峰线为反式肉桂酸标品, 保留时间为22.638 min; 红色峰线为反应样品, 保留时间为22.630 min, 与标准品保留时间基本一致; 黑色蜂线为反应对照样品, 无明显波峰, 即无反式肉桂酸生成。表明PtPA催化L-Phe生成了t-CA。图7
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7PtPAL重组蛋白功能鉴定
蓝色、红色和黑色峰图分别表示反式肉桂酸标品、反应样品、反应对照样品。
Fig. 7Functional identification of the recombinant PtPAL
The blue, red, and black peak maps are trans-cinnamic acid standard, reaction, and control reaction sample, respectively.
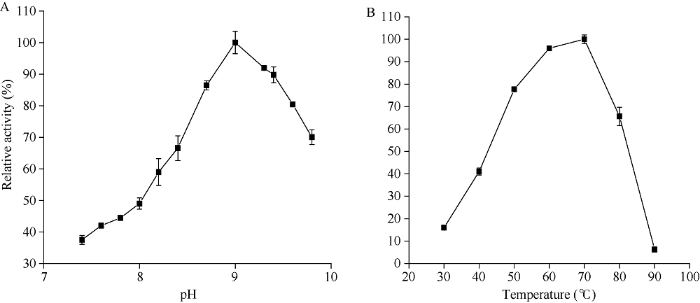
2.5.2 半夏PtPAL重组蛋白催化反应最适条件和酶动力学分析 为探究半夏PtPAL重组蛋白催化反应的最适条件, 本研究在不同pH值与不同温度条件下分别测定PtPAL催化活性。PtPAL催化活性在pH为7.4~9.8范围内先升高后下降, 且当pH为9时, 催化活性最大, 即为最适pH (图8-A)。当温度在30~70℃之间时, PtPAL活性呈逐渐升高的趋势; 当温度大于70℃时, PtPAL活性逐渐降低; 而PtPAL在温度为90℃时, 基本完全失活(图8-B)。
图8
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8PtPAL催化反应最适pH和温度
A: 不同pH下PtPAL催化活性测定; B: 不同温度下PtPAL催化活性测定。误差线表示生物学重复的标准误差(n = 3)。
Fig. 8Optimum pH and temperature of PtPAL for the reaction
A: the activity of PtPAL under changing pH levels; B: the activity of PtPAL across a range of temperatures. Bars indicate the standard errors of the means (n = 3).
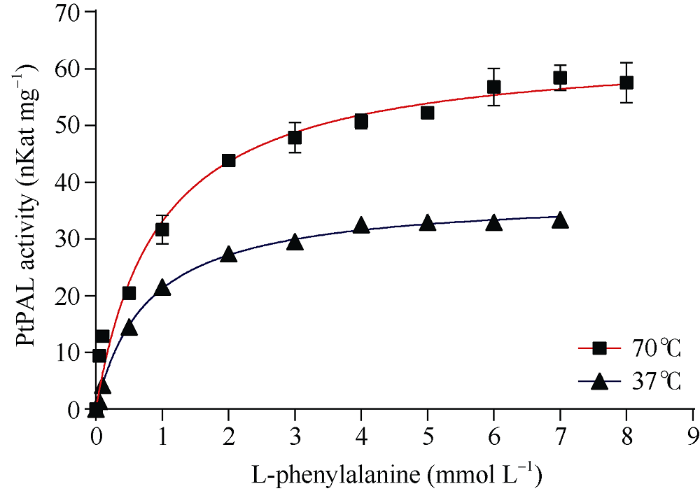
在pH为9.0和温度为70℃、37℃的条件下, 通过测定PtPAL对底物L-Phe酶动力学参数, 拟合PtPAL的米曼氏动力学曲线(图9), 再计算得到酶动力学参数, 最后与其他物种的PAL酶动力学参数进行比对分析(表2)发现, 最适温度70℃下, PtPAL Km为0.89 mmol L-1, Vmax为63.96 nKat mg-1, Kcat为6.56 s-1, Kcat/Km为7.37×103 s-1 M-1; 当反应温度为37℃时, PtPAL Km为0.80 mmol L-1, Vmax为37.67 nkat mg-1, Kcat为3.75 s-1, Kcat/Km为4.69×103 s-1 M-1; 且较于其他物种, PtPAL的Km较大, 但从Kcat/Km参数来看, PtPAL对L-Phe的催化效率较高。
图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9PtPAL米曼氏动力学曲线
误差线表示生物学重复的标准误差(n = 3)。
Fig. 9Michaelis-Menten curves for the velocity of PtPAL
Bars indicate the standard errors of the means (n = 3).
Table 2
表2
表2不同植物PAL酶动力学参数比较
Table 2
| 酶 Enzyme | 温度 Temperature (℃) | 米氏常数 Km (mmol L-1) | 最大酶促反应速率 Vmax (nkat mg-1) | 催化常数 Kcat (s-1) | 催化效率 Kcat/Km (s-1 M-1) | 参考文献 References |
|---|---|---|---|---|---|---|
| PtPAL | 70 | 0.89 | 63.96 | 6.56 | 7.37×103 | 本研究 This study |
| 37 | 0.80 | 37.67 | 3.75 | 4.69×103 | ||
| EsPAL1 | 37 | 0.144 | 7.45×10-3 | 0.588 | 3.87×103 | Taketo et al. (2008) [20] |
| EsPAL2 | 37 | 0.152 | 5.77×10-3 | 0.457 | 3.18×103 | |
| BoPAL1 | 50 | 1.01 | — | 10.11 | 1.0 ×104 | Hsieh et al. (2011) [21] |
| BoPAL2 | 50 | 0.33 | — | 16.04 | 4.86×104 | |
| CdPAL | 55 | 0.101 | — | 3.36 | 3.32×103 | Hu et al. (2011) [22] |
| RgPAL | 50 | 1.3 | — | 25×103 | 19.20 | Zhu et al. (2013) [23] |
| RcPAL | 45 | 7.90 | — | 52.31 | 6.62 ×103 | Ma et al. (2013) [24] |
新窗口打开|下载CSV
2.5.3 金属离子对PtPAL重组蛋白酶活性影响
本研究通过添加不同外源金属盐离子, 探究金属离子对催化活性的影响(图10)发现, Na+、K+、Ca2+ 对PtPAL催化活性的没有显著影响; Ba2+对PtPAL催化活性具有显著的激活作用, 相对酶活性达到110.20%; Mg2+、Mn2+、Co2+、Zn2+及Cu2+金属离子对PtPAL催化活性具有显著的抑制作用。
图10
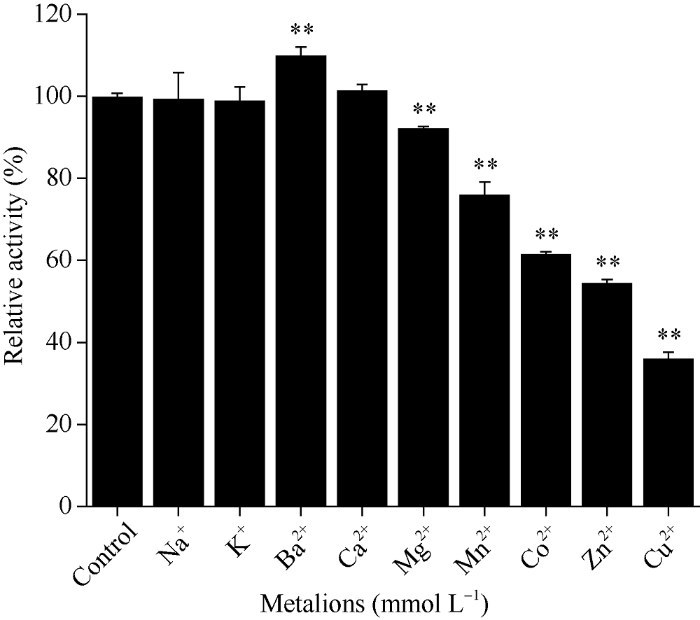 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图10金属离子对PtPAL重组蛋白活性的影响
**表示在0.01水平差异显著。
Fig. 10Effects of metalions on the recombinant PtPAL
** means significant difference at the 0.01 probability level.
3 讨论
随着人们对野生半夏的不断挖掘、开发利用, 导致野生半夏资源几近枯竭, 又因为野生半夏中生物碱含量较低, 而市场需求有增无减[25], 麻黄碱是半夏生物碱的主要活性成分, 含量低, 化学合成复杂且成本高昂, 而苯丙氨酸解氨酶(PAL)是植物苯丙胺类生物碱生物合成途径的第1个关键酶, 因此研究苯丙氨酸解氨酶(PAL)对进一步探究麻黄碱生物合成途径中的具有重要价值与意义。本研究基于半夏转录组测序结果, 从半夏中成功克隆获得1个PtPAL基因, 编码762个氨基酸, 具有PAL-HAL、PLN02457、phe_am_lyase、Lyase_ aromatic及HutH结构域, 属于苯丙氨酸解氨酶家族成员。研究表明, PAL是多基因家族, 一个染色体组中往往含有2~6个PAL基因, 其基因个数因不同物种而不同; 但对已报道的不同物种中PAL家族基因进行序列比对发现, 其具有较高的同源性[26]。通过氨酸序列比对和系统进化树等分析表明, 半夏PtPAL与多种单子叶植物PAL聚为一类, 不与双子叶植物、蕨类植物、苔藓植物以及裸子植物PAL聚集在一起, 这符合植物分类学研究结果。基于同源建模法构建的PtPAL蛋白结构含有在苯丙氨酸解氨酶家族成员中高度保守的Ala-Ser-Gly (ASG)氨基酸三联体及保守的脱氨位点(L、V、L、A)和催化活性位点(N、G、H、NDN及HNDQV), 与已报道的欧芹一样, 表明PtPAL是苯丙氨酸解氨酶家族成员且可能具有催化苯丙氨酸(L-Phe)生成反式肉桂酸(t-CA)的功能。
为进一步鉴定PtPAL的功能, 在大肠杆菌BL21 (DE3)中进行异源表达发现, PtPAL重组蛋白能够高效地催化L-Phe生成t-CA。在大肠杆菌中异源表达附生苔草PaPAL重组蛋白发现, PaPAL催化L-Phe转化为t-CA具有较高的酶活性, 而在催化L-Tyr形成对香豆酸时表现出较低的酶活性[10]。Chen等[27]研究发现, 在拟南芥中, AtPAL的最适底物是L-Phe, 当底物换成L-Tyr时, AtPAL1、AtPAL2和AtPAL4催化效率极低, 然而AtPAL1对L-Tyr没有酶活性。本研究中PtPAL并不能催化L-Tyr生成对香豆酸, 这说明半夏体内, PtPAL只具有催化L-Phe生成t-CA的活性。对PtPAL进行最适酶促反应条件探究发现, PtPAL催化反应最适pH值为9.0, 介于研究已报道的PAL最适pH范围内[28,29]; PtPAL催化反应最适温度为70℃, 与已报道的油茶PAL最适温度一致[30], 略高于其他已报道植物中PAL的最适温度, 如拟南芥AtPAL2、AtPAL3最适温度分别为48℃、31℃, 附生苔草PaPAL最适温度为60℃, 表明物种不同, PAL的最适温度也会不同, 在同一物种中, PAL家族不同成员的最适温度也存在一定差异。酶动力学分析表明, 在最适条件下, PtPAL催化L-Phe反应Km为0.89 mmol L-1, Vmax为63.96 nKat mg-1, Kcat为6.56 s-1, Kcat/Km为7.37×103 s-1 M-1。Km参数显示, PtPAL对底物L-Phe的亲和力较高, 但低于已报道的绿竹(BoPAL)[30]和酵母(RgPAL、RaPAL)[21]; 从Kcat/Km来看, PtPAL对L-Phe的催化效率较高, 且高于草麻黄(EsPAL1、EsPAL2)、肉苁蓉(CdPAL)。探究重金属盐离子对PtPAL酶活影响, 结果表明Ba2+显著增强半夏PtPAL酶活。
PAL家族基因在不同植物组织中的表达模式已得到广泛研究。胡永胜等[31]研究发现, 丹参SmPAL在根茎叶中都有表达, 但在根中表达量最多, 其可能参与木质素的合成; 刘俊频等[32]从膜荚黄芪中克隆获得AmPAL1、AmPAL2、AmPAL3基因, 其中AmPAL2在根中表达量最高, 与毛蕊异黄酮葡萄糖苷积累一致, AmPAL2也可能与毛蕊异黄酮葡萄糖苷的合成有关; 李炳国等[33]研究发现, RgPAL在地黄叶中表达量明显高于花和根中; 而PAL基因在水稻中却主要在茎中得以表达[34]。这说明PAL基因在不同植物中具有表达部位特异性。在本研究中, PtPAL基因在半夏叶中的表达量最高, 其次在块茎与根中, 而一直以来, 半夏以块茎用药, 半夏块茎也被认为是生物碱合成主要器官, 这可能是因为麻黄碱合成所需的前体物质主要位于半夏叶片, 然后运输至块茎中合成麻黄碱。综上所述, PtPAL可能在半夏叶中参与麻黄碱的生物合成, 为开展半夏麻黄碱工程提供候选基因。
4 结论
从半夏中克隆得到PtPAL基因, 编码762个氨基酸, 含有高度保守的Ala-Ser-Gly氨基酸三联体, 与其他已报道的植物PAL具有较高的相似度。PtPAL基因在半夏叶中表达量较高, 块茎、根中次之, 在花中表达量较少。纯化的PtPAL重组蛋白具有催化L-Phe生成t-CA的功能, 且具有较高的催化效率, PtPAL Km为0.89 mmol L-1, Vmax为63.96 nkat mg-1, Kcat为6.56 s-1, Kcat/Km为7.37×103 s-1 M-1; PtPAL催化反应最适pH为9.0, 最适温度为70℃。但在相同催化条件下, PtPAL不能催化L-酪氨酸生成对香豆酸。探究重金属离子对PtPAL酶活性的影响表明, Ba2+增强了PtPAL酶活性, Mn2+、Co2+、Cu2+及Zn2+抑制了PtPAL酶活性。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOIURL [本文引用: 1]
DOIPMID [本文引用: 1]

Phenylalanine ammonia-lyase (PAL) is one of the branch point enzymes between primary and secondary metabolism. It plays an important role during plant development and defense. A PAL gene designated as SmPAL1 was cloned from Salvia miltiorrhiza using genome walking technology. The full-length SmPAL1 was 2,827 bp in size and consisted of an intron and two extrons encoding a 711-amino-acid polypeptide. Sequence alignment revealed that SmPAL1 shared more than 80% identity with the PAL sequences reported in Arabidopsis thaliana and other plants. The 5' flanking sequence of SmPAL1 was also cloned, and a group of putative cis-acting elements such as TATA box, CAAT box, G box and TC-rich repeats were identified. Transcription pattern analysis indicated that SmPAL1 expressed in all tissues examined, but more highly in leaf. Besides, expression of SmPAL1 was found to be induced by various treatments including ABA, wounding, and dehydration. To further confirm its function, SmPAL1 was expressed in Escherichia coli strain M15 with pQE-30 vector. The recombinant protein exhibited high PAL activity and could catalyze the conversion of L: -Phe to trans-cinnamic acid. This study will enable us to further understand the role SmPAL1 plays in the synthesis of active pharmaceutical compounds in S. miltiorrhiza at molecular level.
DOIURL [本文引用: 1]
DOIURL [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOIPMID [本文引用: 1]

To investigate the putative crosstalk between JA and ABA in Solanum lycopersicum plants in response to drought, suppressor of prosystemin-mediated responses2 (spr2 JA-deficient) and flacca (flc, ABA-deficient) mutants together with the naphthalene/salicylate hydroxylase (NahG) transgenic (SA deficient) line were used. Hormone profiling and gene expression of key enzymes in ABA, JA and SA biosynthesis were analyzed during early stages of drought. ABA accumulation was comparable in spr2 and wild type (WT) plants whereas expression of 9-cis-epoxycarotenoid dioxygenase 1 (NCED1) and NCED2 was different, implying a compensation mechanism between NCED genes and an organ-specific regulation of NCED1 expression. JA levels and 12-oxo-phytodienoic acid reductase 3 (OPR3) expression in flc plants suggest that ABA regulates the induction of the OPR3 gene in roots. By contrast, ABA treatment to flc plants leads to a reduction of JA and SA contents. Furthermore, different pattern of SA accumulation (and expression of isochorismate synthase and phenylalanine ammonia lyase 1) was observed between WT seedlings and mutants, suggesting that SA plays an important role on the early response of tomato plants to drought and also that JA and ABA modulate its biosynthesis. Finally, hormone profiling in spr2 and NahG plants indicate a crosstalk between JA and SA that could enhance tolerance of tomato to water stress.
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 2]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOIURL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
DOIURL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
PMID [本文引用: 1]

Phenylalanine ammonia-lyase (PAL) genomic sequences were isolated from a rice (Oryza sativa L.) genomic library using a PCR-amplified rice PAL DNA fragment as a probe. There is a small family of PAL genes in the rice genome. The nucleotide sequence of one PAL gene, ZB8, was determined. The ZB8 gene is 4660 bp in length and consists of two exons and one intron. It encodes a polypeptide of 710 amino acids. The transcription start site was 137 bp upstream from the translation initiation site. Rice PAL transcripts accumulated to a high level in stems, with lower levels in roots and leaves. Wounding of leaf tissues induced ZB8 PAL transcripts to a high level. In rice suspension-cultured cells treated with fungal cell wall elicitors, the ZB8 PAL transcript increased within 30 min and reached maximum levels in 1-2 h. The transcription of the ZB8 gene was investigated by fusing its promoter to the reporter gene beta-glucuronidase (GUS) and transforming the construct into rice and tobacco plants, as well as rice suspension-cultured cells. High levels of GUS activity were observed in stems, moderate levels in roots and low levels in leaves of transgenic rice and tobacco plants. Histochemical analysis indicated that in transgenic rice the promoter was active in root apical tips, lateral root initiation sites, and vascular and epidermal tissues of stems and roots. In rice flowers, high GUS activity was observed in floral shoots, receptacles, anthers and filaments, occasionally GUS activity was also detected in lemma and awn tissues. In tobacco flowers, high GUS activity was detected in the pink part of petals. Consistent with the activity of endogenous PAL transcripts, wounding of rice and tobacco leaf tissues induced GUS activity from low basal levels. Tobacco mosaic virus (TMV) infection of tobacco leaves induced GUS activity to a high level. Fungal cell wall elicitors strongly induced GUS activity and GUS transcripts to high levels in transgenic rice suspension-cultured cells. We demonstrated that the promoter of ZB8 gene is both developmentally regulated and stress-inducible.
