 ,1,*
,1,*Exploration of specific gene(s) for ear rot resistance to Fusarium verticilloides in maize
WEN Jing1, SHEN Yan-Qi1, HAN Si-Ping1, XING Yue-Xian2, ZHANG Ye1, WANG Zi-Yu1, LI Shi-Jie1, YANG Xiao-Hong3, HAO Dong-Yun1, ZHANG Yan ,1,*
,1,*通讯作者:
收稿日期:2020-01-18接受日期:2020-04-15网络出版日期:2020-09-12
| 基金资助: |
Received:2020-01-18Accepted:2020-04-15Online:2020-09-12
| Fund supported: |

摘要
关键词:
Abstract
Keywords:
PDF (3010KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
闻竞, 沈彦岐, 韩四平, 邢跃先, 张叶, 王梓钰, 李世界, 杨小红, 郝东云, 张艳. 玉米拟轮枝镰孢菌穗腐病抗性基因的挖掘[J]. 作物学报, 2020, 46(9): 1303-1311. doi:10.3724/SP.J.1006.2020.03004
WEN Jing, SHEN Yan-Qi, HAN Si-Ping, XING Yue-Xian, ZHANG Ye, WANG Zi-Yu, LI Shi-Jie, YANG Xiao-Hong, HAO Dong-Yun, ZHANG Yan.
玉米是世界第一大粮食作物, 2018年我国的玉米种植面积达到4200万公顷, 而玉米病害一直是影响玉米产量提高和品质改善的限制因素。玉米穗腐病(ear rot)是一类由真菌引发的病害, 普遍发生在全球各个玉米种植区[1,2,3,4]。自20世纪80年代以来, 我国相继报道了玉米穗腐病的发生, 主要发生在东北、黄淮海和西南三大玉米种植区, 且有日渐加剧的趋势, 已成为我国玉米生产的一个重要限制因素。1987年潘惠康等首次报道了由拟轮枝镰孢菌引起的玉米穗腐病[5]。在我国, 一般年份玉米穗腐病的发病率为10%~20%, 严重年份可达30%~40%, 感病品种的发病率高达50%左右[6]。玉米穗腐病不仅会引起果穗腐烂而导致直接减产, 而且有些病原菌还会产生大量的有害毒素, 严重威胁人类和动物的健康和生命安全[7]。玉米穗腐病的发生导致玉米霉粒的增加, 当霉粒含量超过国家标准(≤2%)时, 玉米将无法销售, 导致大量带菌玉米在农户储藏环节积压, 进而引发玉米的收购安全、流通安全、储藏安全和质量安全等一系列问题。据调查, 2013年和2014年吉林省玉米因穗腐病引起的霉粒含量分别为3.4%和2.9%, 给售粮农户带来了巨大的经济损失[8]。
玉米穗腐病的致病菌十分复杂, 研究发现拟轮枝镰孢菌(Fusarium verticilloides)、禾谷镰刀菌(Fusarium graminearum)、赤霉菌(Gibberella fujikuroi)、蠕孢菌(Helm inthosporium sp.)、粉红聚端孢菌(Trichothecium roseum)、曲霉菌(Aspergilllus spp.)、青霉菌(Penicillium spp.)等20多种病原菌都能引起病害发生[9]。但不同区域环境下, 穗腐病的主要致病菌存在差异。据前期研究结果表明, 国内玉米穗腐病的病原菌主要以拟轮枝镰孢菌和禾谷镰刀菌为优势种[10,11]。
到目前为止尚未找到对玉米穗腐病有效的防治方法(如化学防治、生物防治等), 而且目前玉米生产上主推品种基本缺乏对穗腐病的抗性, 亟须培育高抗的玉米品种。已有大量的研究表明玉米穗腐病的抗性主要受遗传因素控制, 其中以加性效应为主, 显性效应较小[12,13,14,15,16,17,18,19,20]。本课题针对玉米穗腐病的主要致病菌拟轮枝镰孢菌进行了全基因组关联分析和转录组测序分析, 获得了玉米拟轮枝镰孢菌穗腐病抗性相关的候选基因。其结果不仅能为玉米和病原菌的相互作用机理的解析奠定基础, 而且能为玉米的抗病分子育种提供一定的理论依据和重要的基因资源。
1 材料与方法
1.1 试验材料
1.1.1 植物材料 527份关联群体, 由中国农业大学杨小红教授提供, 已完成重测序并已开发1,250,000个SNPs位点; 用于转录组测序的玉米穗腐病抗性自交系法A和感病自交系掖81162, 由吉林省农业科学院玉米研究所邢跃先研究员提供[21]。1.1.2 病原菌 本研究中使用的拟轮枝镰孢菌由中国农业大学徐明良教授实验室提供。
1.2 试验方法
1.2.1 田间设计 于吉林省公主岭市吉林省农业科学院院区试验地种植关联群体的527份自交系, 并进行拟轮枝镰孢菌的抗性鉴定。试验设2次重复, 每个重复的每个材料种植1行, 行长5 m, 行距0.6 m, 株距0.25 m。将已鉴定出的玉米穗腐病的抗病自交系法A和高感自交系掖81162各种植6行, 行长5 m, 行距0.6 m, 株距0.25 m。1.2.2 病原菌的培养 将拟轮枝镰孢菌接种到PDA培养基平板上, 28℃培养大约1周, 待菌丝长满平板, 4℃保存待用。在接种前10~15 d取适量健康饱满的玉米籽粒在开水中煮30 min, 装入三角瓶, 121℃高压灭菌30 min。将平板培养基上长好的菌种接种到三角瓶内, 封好瓶口, 室温培养15 d。在接种前一天, 用无菌蒸馏水配制成浓度为5×106个 mL-1的孢子悬浮液。在孢子悬浮液中加入表面活性剂吐温-80, 吐温-80在孢子悬浮液中浓度为2 μL mL-1, 混匀。
1.2.3 人工接种 根据往年的试验经验, 玉米吐丝后12 d为最佳接种时期, 用注射器将制备的孢子悬浮液注射到玉米果穗的中部。每个果穗注射接菌量为2 mL, 接菌后用手指轻捏伤口处, 防止菌液外流。
1.2.4 抗性鉴定 在玉米成熟期(吉林省公主岭9月下旬)进行田间发病调查。将各接种处理的玉米果穗剥去苞叶, 逐个调查记载果穗发病情况, 计算平均发病级别。将抗性分为5级: 1、3、5、7、9, 其中1级为高抗, 9级为高感(参照《玉米抗病虫性鉴定技术规范NY/T 1248.8-2106》, 表1) [21,22]。
Table 1
表1
表1玉米穗腐病病情分级标准
Table 1
| 病情评级Disease severity | 发病百分比Percentage of symptom |
|---|---|
| 1 | 发病面积占雌穗总面积0~1% Percentages of lesion area: 0-1% |
| 3 | 发病面积占雌穗总面积2%~10% Percentages of lesion area: 2%-10% |
| 5 | 发病面积占雌穗总面积11%~25% Percentages of lesion area: 11%-25% |
| 7 | 发病面积占雌穗总面积26%~50% Percentages of lesion area: 26%-50% |
| 9 | 发病面积占雌穗总面积51%~100% Percentages of lesion area: 51%-100% |
新窗口打开|下载CSV
1.2.5 玉米穗腐病抗性的转录组测序和CCCH域蛋白19的表达分析 分别对玉米穗腐病的抗病自交系法A和高感自交系掖81162人工接种拟轮枝镰孢菌, 在接种前和接种后1 d、2 d、3 d、4 d及7 d的同一时间点田间取样(上午9:00), 在果穗中部注射孢子悬浮液的位置周围取30粒玉米籽粒混成一个样品, 共设置3个生物学重复, 用于转录组测序。在接种前和接种后1 d、2 d、3 d、4 d、5 d、6 d、7 d、8 d和9 d的相同时间取样, 用于CCCH域蛋白19的表达分析。
采用EasyPure Plant RNA Kit (北京全式金生物技术有限公司)提取玉米籽粒总RNA, 随后立即反转录成cDNA, 具体操作流程参考试剂盒附带的说明书。
以cDNA为模板, 根据玉米中的内参基因actin和真菌中的管家基因β-tubulin2设计相应的引物(表2), 进行RT-PCR实验。设计产物大小为150~ 200 bp的特异性引物, 以反转录获得的cDNA为模板, 做标准曲线进行PCR扩增效率的验证。随后采用非特异性SYBR Green I染料法, 在ABI Prism 7900的Real-time PCR仪器上进行试验, 利用配套的分析软件分析数据, 采用2-ΔΔCt方法处理Real-time PCR数据。
Table 2
表2
表2引物序列
Table 2
| 基因名称 Gene name | 上游引物序列 Forward primer sequence (5'-3') | 下游引物序列 Reverse primer sequence (5'-3') | 产物长度 Product length (bp) |
|---|---|---|---|
| actin | CCTTCATTGGCATGGAATCT | GCAACCACCTTCACCTTCAT | 128 |
| β-tubulin2 | AGACCGGTCAGTGCGGTAAC | CGTGCTCGCCAGAGAGATGGT | 70 |
| CCCH domain protein 19 | CAGGTGAGTTGTGGACATGG | CTCCAAGCACAAGCAAACAA | 197 |
新窗口打开|下载CSV
转录组测序和分析所提取RNA一部分送至北京通州国际种业科技有限公司进行测序文库构建和转录组测序, 另留一部分进行反转录和后续Real- time PCR验证试验。
公司提交的测序结果已经进行了测序接头和低质量序列的过滤。我们得到测序结果后, 首先使用fastqc软件进行质量评估, 并进行转录本的比对、拼接、转录本表达水平的计算和DEGs的筛选等, 具体流程是: 采用Tophat (v2.0.9)将reads比对到玉米参考基因组B73 RefGen_v3上, 包含Working Gene Set (WGS)基因的GFF文件作为转录本注释的参考文件。使用Cufflink软件拼接转录本, 采用Samtools工具实现reads的拼接情况可视化及拼接后序列的提取, Cuffdiff (v2.0.2)用来分析基因的差异表达情况, 参数设置包括偏差修正(-b)和分位数标准化(-n), 使用的GFF文件为过滤后的基因集(Filtered Gene Set, FGS)。用FPKM (Fragments Per Kilobase exon per Million reads mapped)计算基因的表达水平, 根据每个基因在不同生物学重复间的表达量确定其最终的FPKM值。
1.2.6 玉米穗腐病抗性的全基因组关联分析 采用全基因组关联分析和预测整合工具GAPIT (
2 结果与分析
2.1 玉米穗腐病抗性的转录组测序
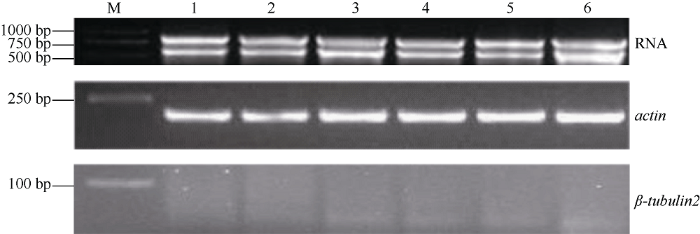
2.1.1 RNAseq材料的总RNA提取和β-tubulin2基因的RT-PCR分析 从电泳结果可知, 提取的RNA完整性较好, 28S和18S两条带清晰, 无降解, 可以用于后续试验。感病自交系掖81162的RT-PCR的结果表明, 接菌前的籽粒样品中没有β-tubulin2基因的表达(图1), 接菌后的籽粒样品中有β-tubulin2 基因的表达, 而且接菌后7 d的样品中的表达量较高, 说明可通过β-tubulin2基因的表达量检测病原菌在植物体内的侵染程度。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1掖81162样品中β-tubulin2基因的RT-PCR分析
M: DL2000 marker; 1~6: 接菌前和接菌后1 d、2 d、3 d、4 d、7 d的玉米籽粒样品。
Fig. 1RT-PCR analysis of β-tubulin2 of Ye 81162
M: DL2000 marker; 1-6: Grain samples from ears before inoculation and at 1 d, 2 d, 3 d, 4 d, 7 d after inoculation.
2.1.2 Real-time PCR检测抗感自交系样品中β-tubulin2的表达量 在人工接种拟轮枝镰孢菌之前, 抗病自交系法A和感病自交系掖81162的籽粒样品中β-tubulin2基因的表达量为0。在人工接种拟轮枝镰孢菌之后, 法A和掖81162的籽粒样品中均有β-tubulin2基因的表达, 随时间的推移, 表达量大小也在发生变化, 而且在相同的时间点, 抗病自交系法A的籽粒样品中β-tubulin2基因的表达量均低于感病自交系掖81162。另外, 在人工接种拟轮枝镰孢菌后7 d的时间点抗病自交系法A和感病自交系掖81162的样品中β-tubulin2基因的表达量差异最大(图2)。
图2
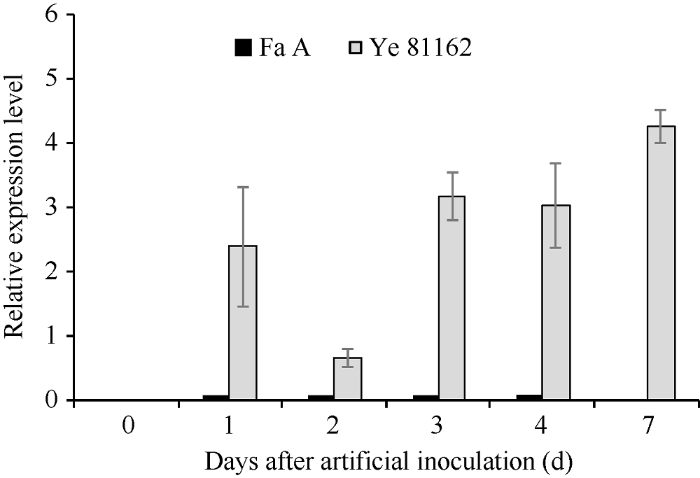 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2人工接种拟轮枝镰孢菌后β-tubulin2基因的相对表达量
Fig. 2Relative expression of β-tubulin2 gene after artificial inoculation with Fusarium verticillium
2.1.3 人工接种拟轮枝镰孢菌后的转录组测序分析
通过Real-time PCR结果得知, 在人工接种拟轮枝镰孢菌后7 d时, 玉米抗病自交系法A和感病自交系掖81162之间β-tubulin2基因的相对表达量的差异最大。所以分别选取接菌前和人工接种拟轮枝镰孢菌7 d的抗感自交系样品进行转录组测序, 每个样品设置3个生物学重复。
抗病自交系法A在人工接种拟轮枝镰孢菌前和接菌后7 d的差异表达基因数量为374个; 感病自交系掖81162在人工接种拟轮枝镰孢菌前和接菌后7 d的差异表达基因数量为9055个; 人工接种拟轮枝镰孢菌之前, 抗病自交系法A和感病自交系掖81162的差异表达基因数量为7522个; 人工接种拟轮枝镰孢菌后7 d, 抗病自交系法A和感病自交系掖81162的差异表达基因数量为10,761个(图3)。
图3
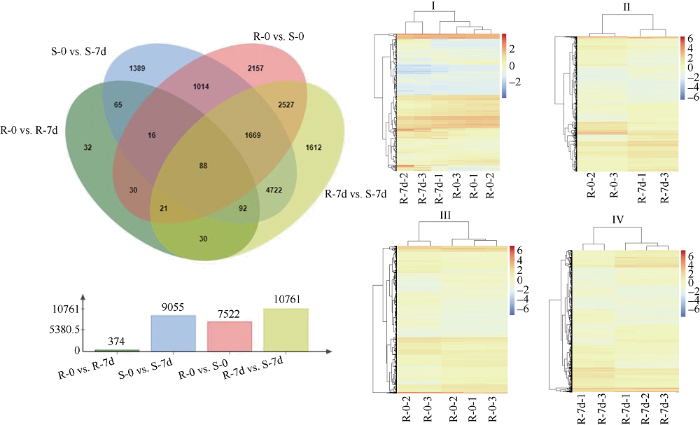 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3人工接种拟轮枝镰孢菌后法A和掖81162的差异表达基因分析
R: 抗病; S: 感病; R-0: 接菌前的法A; R-7d: 接菌后7 d的法A; S-0: 接菌前的掖81162; S-7d: 接菌后7 d的掖81162。
Fig. 3Analysis of differentially expressed genes of Fa A and Ye 81162 after inoculation with Fusarium verticillium
R: resistant; S: susceptible; R-0: Fa A before inoculation; R-7d: Fa A at seven days after inoculation; S-0: Ye 81162 before inoculation; S-7d: Ye 81162 at seven days after inoculation.
2.2 玉米拟轮枝镰孢菌穗腐病的全基因组关联分析
利用不同来源的527份材料为关联群体, 通过人工接种拟轮枝镰孢菌的方法, 对玉米穗腐病抗性进行全基因组关联分析, 共检测到5个与穗腐病抗性显著相关的SNP (图4), 这些SNP分布在第1和第9染色体上。通过比对B73 RefGen_v3并注释, 发现其中有SNP位点附近涉及的基因包括酰基激活酶1过氧化物酶体、蛋白磷酸酶2C 48、镁转运蛋白、受体蛋白激酶 CRINKLY4和锌指CCCH域蛋白19 (表4)。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4玉米拟轮枝镰孢菌穗腐病抗性全基因组关联分析的Manhattan图和QQ-plot图
Fig. 4Manhattan plot and QQ plot for genome-wide association analysis of resistance to ear rot caused by Fusarium verticillium in maize
Table 3
表3
表3人工接种拟轮枝镰孢菌后β-tubulin2基因的相对表达量
Table 3
| 自交系 Inbred line | β-tubulin2基因的相对表达量 Relative expression of β-tubulin2 gene | |||||
|---|---|---|---|---|---|---|
| 接菌前 Before inoculation | 1 d | 2 d | 3 d | 4 d | 7 d | |
| 法A Fa A | 0±0.00 | 0.0004±0.00 | 0.0002±0.00 | 0.0003±0.00 | 0.0002±0.00 | 1.006E-07±0.00 |
| 掖81162 Ye 81162 | 0±0.00 | 2.3894±0.93 | 0.6537±0.14 | 3.1675±0.38 | 3.0244±0.65 | 4.2477±0.26 |
新窗口打开|下载CSV
Table 4
表4
表4拟轮枝镰孢菌穗腐病抗性的相关SNP位点涉及的基因信息
Table 4
| 标记 Marker | 注释 Annotation | 染色体 Chromosome | 描述 Description |
|---|---|---|---|
| chr1.S_7569254 | GRMZM2G110616 | 1 | 酰基激活酶1过氧化物酶体 Probable acyl-activating enzyme 1 peroxisomal |
| chr1.S_8288770 | GRMZM5G891266 | 1 | 蛋白磷酸酶2C 48 Probable protein phosphatase 2C 48 |
| chr1.S_8353697 | GRMZM2G018706 | 1 | 镁转运蛋白10 Magnesium transporter 10 |
| chr9.S_154383692 | GRMZM2G092776 | 9 | 受体蛋白激酶CRINKLY4 Receptor protein kinase CRINKLY4 |
| chr9.S_154384544 | GRMZM2G393471 | 9 | 锌指CCCH域蛋白19 Zinc finger CCCH domain-containing protein 19 |
新窗口打开|下载CSV
2.3 全基因组关联分析和转录组测序的联合分析
将在转录组测序中获得的差异表达基因和全基因组选择中关联到的基因比对发现, 在拟轮枝镰孢菌穗腐病的全基因组关联分析中关联到的基因锌指CCCH域蛋白19是人工接种拟轮枝镰孢菌后7 d在抗病自交系法A和感病自交系掖81162之间的差异表达基因。为了进一步分析锌指CCCH域蛋白19在抗病自交系法A和感病自交系掖81162之间的表达情况, 分别对二者在接种前和接种后1~9 d取样, 进行表达量分析(图5)。结果表明在人工接种拟轮枝镰孢菌之前, 锌指CCCH域蛋白19在感病自交系掖81162中的表达量高于抗病自交系法A; 在人工接种拟轮枝镰孢菌后的1 d, 锌指CCCH域蛋白19在感病自交系掖81162中的表达量较接菌前下降, 但是在抗病自交系法A中的表达量较接菌前上升; 在人工接种拟轮枝镰孢菌后的2 d、3 d、4 d和5 d, 锌指CCCH域蛋白19在感病自交系掖81162中的表达量与接菌后1 d的表达量相当而且表达量相对稳定, 但是在抗病自交系法A中的表达量和接菌后1 d的表达量相比有所下降而且表达量相对稳定, 而且在以上时间点抗感自交系之间的表达量相当; 在人工接种拟轮枝镰孢菌后的6 d, 锌指CCCH域蛋白19在抗病自交系法A中的表达量上升, 而在感病自交系掖81162中的表达量保持不变; 在人工接种拟轮枝镰孢菌后的7 d, 锌指CCCH域蛋白19在抗感自交系中的表达量均处于最高水平, 而且在抗病自交系法A中的表达量显著高于感病自交系掖81162。随后在人工接种拟轮枝镰孢菌后的8 d和9 d, 锌指CCCH域蛋白19在抗感自交系中的表达量均下降而且抗病自交系法A中的表达量显著高于感病自交系掖81162。图5
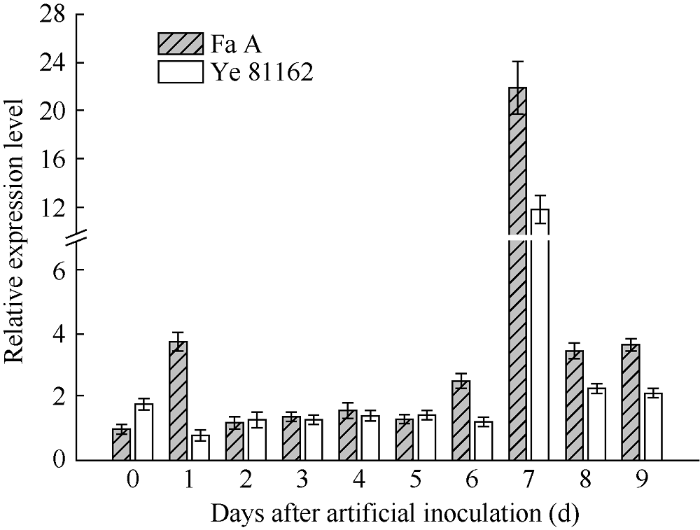 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5人工接种拟轮枝镰孢菌后锌指CCCH域蛋白19基因的相对表达量
Fig. 5Relative expression of zinc finger CCCH domain-containing protein 19 gene after artificial inoculation with Fusarium verticillium
3 讨论
在转录组测序中, 样品选取的时间点非常重要。在本研究中, 观察到感病自交系掖81162在接种后7 d开始表现出发病症状, 7 d之前几乎不能通过肉眼观察到发病症状, 而抗病自交系法A在接种后7 d仍观察不到发病症状。接菌后7 d之前不能通过肉眼观察确定抗感自交系的发病差异最大的时间点, 所以很难确定转录组测序的样品的最佳时间点。为了比较准确地确定转录组测序的最佳时间点, 本研究中分别在接菌前和接种后1 d、2 d、3 d、4 d和7 d对抗感自交系样品中来自真菌管家基因βtubulin2的表达量进行分析, 结果表明在人工接种拟轮枝镰孢菌后7 d抗感自交系样品中β-tubulin2的表达差异最大, 进而对接菌前和人工接种拟轮枝镰孢菌后7 d的样品进行转录组测序。所以, 在玉米穗腐病的抗性研究中, 可以通过真菌的管家基因β-tubulin2的表达量的分析在一定程度上确定病原菌在玉米籽粒中的侵染程度。根据抗病自交系法A和感病自交系掖81162接种后1 d和2 d的β-tubulin2基因的相对表达量结果可以看出, 两个自交系在接菌后2 d的样品中较接菌后1 d的样品中β-tubulin2基因的相对表达量不但没有提高, 反而降低。这可能是由于病原菌在初始侵染玉米籽粒后, 在抗感自交系中均能在一定程度上引发玉米中的基础防御体系, 比如植物的系统性抗性, 所以β-tubulin2基因的表达量会受到一定程度的抑制, 但是随着时间推移, 植物中的基础防御体系被病原菌攻克, 植物的感病性增加, 从而样品中β-tubulin2基因的表达量也会提高。但是由于抗病自交系中可能存在其他的抗性防御机制, 所以随着时间的推移, 抗感自交系中β-tubulin2基因的表达量差异也会随之增加。
全基因组关联分析可用于对数量性状位点的定位, 该方法的优点是以富含基因资源的自然群体为目标性状的基因资源库, 结合表型数据, 搜索目标基因, 不需要构建专门的作图群体, 这可以大大缩短研究年限。但是该方法也有缺点, 因其以自然群体作为基因资源库, 存在遗传漂变、群体结构和自然选择等不可控因素, 会引起假阳性或伪关联。因此, 定位相关位点还需通过其他分析方法进一步验证。在本研究中, 通过全基因组关联分析关联到了5个基因, 为了验证该位点的准确性, 将在转录组测序中获得的差异表达基因和全基因组选择中关联到的基因进行比对, 发现全基因组关联分析中关联到的锌指CCCH域蛋白19同时也是转录组测序中获得的差异表达基因。另外, 通过对法A和掖81162在接种前和接种后不同时间的锌指CCCH域蛋白19表达量的差异性检测, 进一步验证了锌指CCCH域蛋白19与玉米穗腐病的抗性有关。CCCH型锌指蛋白是一类生物体中广泛存在的转录因子, 有研究表明, CCCH型锌指蛋白通过与DNA、RNA相互作用, 在生物与非生物胁迫及生长发育中行使重要功能[23,24]。在拟南芥中, CCCH型锌指蛋白ZFAR1的表达受霉灰菌的诱导, 而且其突变体zfar1对ABA处理高度敏感[25]。另外, CCCH型锌指蛋白AtSZF1和AtSZF2参与调控拟南芥对盐胁迫的耐性[26]。此外, 也有研究表明大多数与植物防御相关的差异表达基因在抗病材料中上调表达, 而与植物生长发育调节相关的基因在感病材料中上调表达。推测当病原菌侵染玉米籽粒时, 抗病材料会重新调节其生长和抗性的平衡, 并合成更多的次生防御代谢产物, 进而形成对穗腐病的早期防御反应[27]。因此, 锌指CCCH域蛋白19的大量合成很可能就是玉米植株抗拟轮枝镰孢菌侵染的防御反应。
本研究中1个关联位点的基因为受体蛋白激酶 CRINKLY4。植物蛋白激酶中的受体蛋白激酶在植物抗病反应中参与较多, 它由胞外受体结构域、跨膜结构域和胞内激酶结构域3个结构域组成。胞外受体结构域的功能是识别受体感受外界信号, 跨膜结构域将被胞外受体识别的信号传递给胞内激酶结构域, 而胞内激酶结构域能够通过磷酸化作用将信号传递给下一级信号传递体。当病原物侵染植物后, 植物受体蛋白激酶能识别和接受病原信号物, 激活受体蛋白激酶胞内激酶域发生磷酸化反应, 将胞外信号转导到胞内, 诱导防御反应, 保护植物免受病原菌的进一步侵染。植物的数量抗性一般参与植物免疫系统的PAMP激活的防御反应(PAMP-triggered immunity, PTI), 病原菌的一些保守结构可以被植物体内穿膜类型的受体所识别, 引发下游的防御反应, 抑制病原菌的侵染。所以该基因可作为一个玉米穗腐病抗性相关的候选基因进行进一步的研究和功能验证。
由于该研究中进行转录组测序的材料为玉米自交系, 而不是近等基因系, 获得的差异基因数目非常多, 筛选到候选基因非常困难。所以, 需要结合其他方法共同进行, 比如全基因组关联分析, 连锁分析等等。将转录组测序结果和全基因组关联分析的结果比对后发现, 在全基因组关联分析中关联到的基因与转录组测序中获得差异表达基因是共同的, 即锌指CCCH域蛋白19, 而且进一步的Real-time表达分析初步表明该基因与玉米拟轮枝镰孢菌穗腐病抗性具有一定的相关性, 证明了不同手段相结合挖掘抗性基因的有效性。
4 结论
全基因组关联分析检测到5个与拟轮枝镰孢菌穗腐病抗性显著相关的SNP位点, 关联位点所在的基因涉及酰基激活酶1过氧化物酶体、蛋白磷酸酶2C 48、镁转运蛋白和受体蛋白激酶 CRINKLY4。同时结合玉米自交系法A和掖81162的转录组测序, 获得一个玉米拟轮枝镰孢菌抗性的候选基因锌指CCCH域蛋白19。本研究结果为玉米穗腐病抗病基因的克隆和玉米的抗病分子育种提供了一定的理论依据和遗传资源。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.3969/j.issn.1004-3268.2009.04.022URL [本文引用: 1]

采用室内平皿生长速率法,比较了4种杀菌剂对穗粒腐病3个主要病原菌,即串珠镰刀菌、禾谷镰刀菌和聚端孢菌的抑制作用。结果表明:甲基托布津、多菌灵、烯唑醇、三唑酮4种杀菌剂对3种主要致病菌有明显抑制作用,药剂浓度越大抑制效果越明显。其中多菌灵对3种病原菌的室内抑制作用最强,其次为烯唑醇,甲基托布津和三唑酮较差。在杀菌剂浓度为0.002mg/mL时,多菌灵、烯唑醇与甲基托布津、三唑酮相比,其差异达到了极显著水平。
[本文引用: 1]
DOI:10.3969/j.issn.1004-3268.2005.11.019URL [本文引用: 1]

对河南省玉米杂交种穗粒腐病原菌分离鉴定的结果表明,玉米杂交种籽粒(或病粒)中串珠镰刀菌的带菌率最高,分离频率为59.61%,其次为木霉菌,分离频率为14.97%。从玉米杂交种中分离到的穗粒腐病菌对玉米幼苗致病性的测定结果表明,串珠镰刀菌、蠕孢菌和禾谷镰刀菌对玉米幼苗的致病力较强,只是发病时间、病害蔓延速度有所差异。病原菌分离频次的高低与病原菌的致病性强弱无相关性。
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1146/annurev.phyto.34.1.479URLPMID:15012553 [本文引用: 1]

Quantitative trait locus (QTL) mapping is a highly effective approach for studying genetically complex forms of plant disease resistance. With QTL mapping, the roles of specific resistance loci can be described, race-specificity of partial resistance genes can be assessed, and interactions between resistance genes, plant development, and the environment can be analyzed. Outstanding examples include: quantitative resistance to the rice blast fungus, late blight of potato, gray leaf spot of maize, bacterial wilt of tomato, and the soybean cyst nematode. These studies provide insights into the number of quantitative resistance loci involved in complex disease resistance, epistatic and environmental interactions, race-specificity of partial resistance loci, interactions between pathogen biology, plant development and biochemistry, and the relationship between qualitative and quantitative loci. QTL mapping also provides a framework for marker-assisted selection of complex disease resistance characters and the positional cloning of partial resistance genes.
DOI:10.1007/s11032-008-9184-4URL [本文引用: 1]
[本文引用: 1]
DOI:10.1186/s12870-014-0372-6URLPMID:25547028 [本文引用: 1]

BACKGROUND: Resistance to Fusarium ear rot of maize is a quantitative and complex trait. Marker-trait associations to date have had small additive effects and were inconsistent between previous studies, likely due to the combined effects of genetic heterogeneity and low power of detection of many small effect variants. The complexity of inheritance of resistance hinders the use marker-assisted selection for ear rot resistance. RESULTS: We conducted a genome-wide association study (GWAS) for Fusarium ear rot resistance in a panel of 1687 diverse inbred lines from the USDA maize gene bank with 200,978 SNPs while controlling for background genetic relationships with a mixed model and identified seven single nucleotide polymorphisms (SNPs) in six genes associated with disease resistance in either the complete inbred panel (1687 lines with highly unbalanced phenotype data) or in a filtered inbred panel (734 lines with balanced phenotype data). Different sets of SNPs were detected as associated in the two different data sets. The alleles conferring greater disease resistance at all seven SNPs were rare overall (below 16%) and always higher in allele frequency in tropical maize than in temperate dent maize. Resampling analysis of the complete data set identified one robust SNP association detected as significant at a stringent p-value in 94% of data sets, each representing a random sample of 80% of the lines. All associated SNPs were in exons, but none of the genes had predicted functions with an obvious relationship to resistance to fungal infection. CONCLUSIONS: GWAS in a very diverse maize collection identified seven SNP variants each associated with between 1% and 3% of trait variation. Because of their small effects, the value of selection on these SNPs for improving resistance to Fusarium ear rot is limited. Selection to combine these resistance alleles combined with genomic selection to improve the polygenic background resistance might be fruitful. The genes associated with resistance provide candidate gene targets for further study of the biological pathways involved in this complex disease resistance.
[本文引用: 1]
DOI:10.1007/s00122-015-2600-3URLPMID:26643764 [本文引用: 1]

[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:16925600 [本文引用: 1]
DOI:10.1093/pcp/pcm088URLPMID:17609218 [本文引用: 1]

The molecular mechanism governing the response of plants to salinity stress, one of the most significant limiting factors for agriculture worldwide, has just started to be revealed. Here, we report AtSZF1 and AtSZF2, two closely related CCCH-type zinc finger proteins, involved in salt stress responses in Arabidopsis. The expression of AtSZF1 and AtSZF2 is quickly and transiently induced by NaCl treatment. Mutants disrupted in the expression of AtSZF1 or AtSZF2 exhibit increased expression of a group of salt stress-responsive genes in response to high salt. Significantly, the atszf1-1/atszf2-1 double mutant displays more sensitive responses to salt stress than the atszf1-1 or atszf2-1 single mutants and wild-type plants. On the other hand, transgenic plants overexpressing AtSZF1 show reduced induction of salt stress-responsive genes and are more tolerant to salt stress. We also showed that AtSZF1 is localized in the nucleus. Taken together, these results demonstrated that AtSZF1 and AtSZF2 negatively regulate the expression of salt-responsive genes and play important roles in modulating the tolerance of Arabidopsis plants to salt stress.
[本文引用: 1]
