 ,2,*, 黎裕
,2,*, 黎裕 ,2,*
,2,*Genome-wide association studies of leaf orientation value in maize
PENG Bo1,**, ZHAO Xiao-Lei1,**, WANG Yi1,**, YUAN Wen-Ya1, LI Chun-Hui2, LI Yong-Xiang2, ZHANG Deng-Feng2, SHI Yun-Su2, SONG Yan-Chun2, WANG Tian-Yu ,2,*, LI Yu
,2,*, LI Yu ,2,*
,2,*通讯作者:
第一联系人:
收稿日期:2019-12-3接受日期:2020-01-15网络出版日期:2020-06-12
| 基金资助: |
Received:2019-12-3Accepted:2020-01-15Online:2020-06-12
| Fund supported: |
作者简介 About authors
彭勃,E-mail:snbopeng@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (2510KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
彭勃, 赵晓雷, 王奕, 袁文娅, 李春辉, 李永祥, 张登峰, 石云素, 宋燕春, 王天宇, 黎裕. 玉米叶向值的全基因组关联分析[J]. 作物学报, 2020, 46(6): 819-831. doi:10.3724/SP.J.1006.2020.93063
PENG Bo, ZHAO Xiao-Lei, WANG Yi, YUAN Wen-Ya, LI Chun-Hui, LI Yong-Xiang, ZHANG Deng-Feng, SHI Yun-Su, SONG Yan-Chun, WANG Tian-Yu, LI Yu.
过去的几十年中, 玉米株型结构的改良, 尤其是直立叶型的品种对玉米产量的遗传增益起了重要的作用[1,2]。直立叶型品种比平展叶型品种通常更耐密植, 通过较高的叶面积指数来增加群体光能截获, 促进群体光合作用, 进而获得高产[1,3-4]。叶向值是可以同时反映叶片“直”和“立”两个特性的综合指标。穗上叶向值高, 上部叶片直而不弯, 叶夹角小而上冲, 是理想株型育种的重要选择目标[3]。因此, 阐明玉米叶向值的遗传基础对玉米理想株型育种具有重要的意义。
玉米叶向值是典型的数量性状, 受多基因控制, 遗传基础复杂[5,6]。前人以不同杂交组合构建的F2、F2:3、四交F1单株、RILs群体为材料, 利用SSR分子标记和连锁作图方法, 在染色体的很多区段都检测到了控制叶向值的QTL[5,6,7,8,9,10,11]。其中, 主效QTL(解释表型变异率大于10.00%的QTL)遗传效应大, 受环境影响小, 历来是分子育种和QTL图位克隆优先关注的位点[2,12-13]。在1.01 bin、1.02 bin、1.10 bin、2.02 bin、2.04 bin、3.06 bin、3.08 bin、3.09 bin、4.01~4.02 bin、7.00 bin和7.04 bin上先后发现了叶向值的主效QTL[5,6,7,8,9,10,11]。随着技术的发展, 近年来, 利用第3代分子标记SNP标记通过连锁作图方法, 在3.05 bin、7.05 bin上也发现了叶向值的主效QTL[14,15]。Zhao等[6]整合了前人关于叶夹角、叶向值、叶长、叶宽、叶面积和叶长/叶宽6个叶型性状的QTL定位结果, 通过元分析方法, 发现22个meta-QTL (mQTL), 并预测了其候选基因。
综上, 连锁作图为玉米叶向值遗传基础的解析提供了重要的参考, 但是由于受到群体重组率、标记密度等限制, QTL定位区间较大, 而且仅仅能分析少数几个等位基因。与连锁分析互补, 全基因组关联分析(genome-wide association study, GWAS)可以高效挖掘多个多样性玉米种质资源的优异等位基因。关联分析已经被用于叶夹角遗传基础的剖析[12,16-17], 而且ZmCLA4[12]、UPA1和UPA2[2]、ZmILI1[13] 4个控制叶夹角的QTL已经实现了图位克隆。在玉米叶向值上, 仅Lu等[18]以80份玉米骨干自交系为材料, 利用1.49×106个SNP结合2年的表型鉴定, 通过全基因组关联分析(GWAS)方法检测到33个与叶夹角和叶向值显著相关的SNP (P<1.00E-6), 并在显著的SNP位点上下游9.7 kb的LD衰减距离内, 挖掘到22个叶夹角候选基因和7个叶向值候选基因。相比于叶夹角, 叶向值全基因组关联分析的报道较少。本研究以285份遗传多样性丰富的玉米自交系为关联群体, 结合覆盖玉米整个基因组的56,110个SNP标记, 通过全基因组关联分析的方法(GWAS)挖掘玉米种质资源中调控叶向值的基因资源, 旨在为耐密植的玉米理想株型分子设计育种提供理论参考。
1 材料与方法
1.1 材料
所用材料由中国农业科学院作物科学研究所提供, 是从890份包含玉米自交系核心种质[19]和近年来我国玉米育种常用优良自交系, 以及近1000份其他国家的自交系[20]中, 选择出适应天津地区种植, 且遗传多样性较高的285份玉米自交系, 其中271份来源于我国北方、黄淮海、西南主要玉米生产区域, 12份来源于美国, 1份来源于非洲, 1份来源于墨西哥。1.2 方法
1.2.1 田间试验设计 全部材料于2017年和2018年连续2年种植在天津市农业科学院武清创新基地, 采用完全随机区组试验设计, 单行区, 2次重复, 行长3 m, 行距0.6 m, 密度67,500株 hm-2。施肥、灌溉、防虫和除草同当地大田管理。在玉米散粉后10 d, 从第4株开始, 每行连续测量5株穗上3片叶的叶夹角(θ)、叶长(L)和高点长(叶片基部至叶片最高点的距离, Lf), 并采用Pepper等[21]提出的方法计算叶向值(leaf orientation value, LOV), LOV = 1/n Σ(90 - θ) × (Lf/L), n表示测定叶片数。
1.2.2 表型数据分析 利用SPSS 22.0统计软件对2个环境下叶向值(LOV)的平均值、标准差、频率分布等进行描述性统计分析。利用R语言(
1.2.3 基因型鉴定与SNP质量控制 285份材料利用MaizeSNP50 BeadChip芯片基因分型, 该芯片包括56,110个SNP标记, 基因型数据由中国农业科学院作物科学研究所提供。利用PLINK1.09软件[22]过滤掉最小等位基因频率(maf)<0.05, 基因型缺失率(geno)>0.2的SNP标记。最终39,827个SNP通过软件质量控制。
1.2.4 连锁不平衡(linkage disequilibrium, LD)分析
利用Haploview 4.2软件计算SNP之间的LD衰减距离, 同一染色体上两个位点之间的距离以25 kb的基本分类单元分类, 并计算不同分类单元的平均LD[23]。标记间相关系数(r2)小于0.1, 认为没有相关性[24]。
1.2.5 全基因组关联分析 (1)阈值确定: 由于很多SNP之间可能存在高度连锁不平衡, 因此采用GEC软件(
2 结果与分析
2.1 叶向值表型数据分析
不同年份叶向值的基本统计量分析(表1和图1)显示, 285份多样性自交系关联群体的叶向值在不同年份呈现广泛的连续变异。2018年叶向值群体均值高于2017年, 表明关联群体总体上在2017年更趋于平展, 2018年更趋于紧凑上冲, 而2年叶向值BLUP居中。不同年份叶向值的峰度和偏度的绝对值都小于1 (表1), 表明叶向值在不同年份均服从正态分布, 说明叶向值为典型的数量性状, 受多基因控制。Table 1
表1
表1不同年份叶向值的基本统计量分析及相关分析
Table 1
| 年份 Year | 均值 Mean | 变异范围 Range | 标准差 SD | 偏度 Skewness | 峰度 Kurtosis | 相关系数Correlation coefficient | |
|---|---|---|---|---|---|---|---|
| 2017 | 2018 | ||||||
| 2017 | 59.45 | 12.99-86.28 | 13.77 | -0.58 | 0.13 | ||
| 2018 | 63.86 | 17.05-90.00 | 13.11 | -0.79 | 0.59 | 0.75** | |
| BLUP | 61.58 | 23.46-82.65 | 11.14 | -0.63 | 0.30 | 0.95** | 0.90** |
新窗口打开|下载CSV
图1
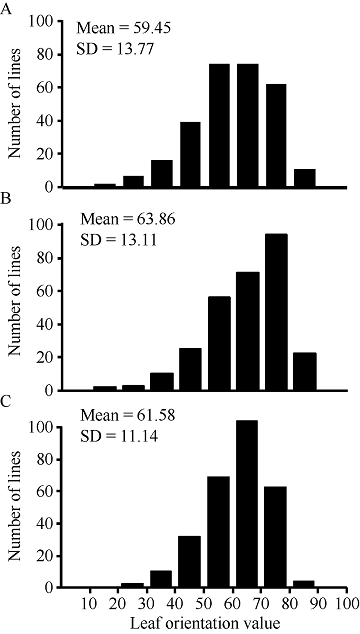 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1不同年份玉米叶向值的频率分布图
A: 2017年; B: 2018年; C: 2年联合的BLUP值。
Fig. 1Frequency distribution of leaf orientation value in two years
A: Year of 2017; B: Year of 2018; C: BLUP for combined 2 years.
方差分析结果表明(表2), 不同年份关联群体叶向值的基因型效应、年份效应和基因型×年份互作效应达到极显著水平(P<0.01), 而重复间方差不显著(P>0.05)。因此, 各自交系间的叶向值存在极显著差异, 且年份对叶向值也有显著的影响。叶向值在不同年份间的遗传力达到87.86%, 提示该性状主要受遗传因素控制, 不同年份的环境效应影响较小。相关分析结果表明不同年份之间叶向值的相关达到极显著水平(P<0.01)。最佳线性无偏预测(Best Linear Unbiased Prediction, 简称BLUP)可以对多环境数据整合, 消除环境效应, 得到材料稳定遗传的表型。BLUP与不同年份叶向值相关系数高达0.90以上, 进一步验证了叶向值具有较高的遗传力, 受环境影响较小。
Table 2
表2
表2不同年份叶向值的方差分析
Table 2
| 变异来源 Variation source | 方差分量 Variance | 遗传力 H2 (%) |
|---|---|---|
| 基因型 Genotype | 141.46** | 87.86 |
| 年份Year | 8.97** | |
| 基因型×年份 Genotype×year | 21.41** | |
| 重复Replication | 14.63 | |
| 残差Residual | 35.32 |
新窗口打开|下载CSV
2.2 LD分析
285份材料10条染色体平均LD衰减距离为457 kb (图2), 其中1号到10号染色体LD衰减距离分别为332、299、432、500、382、421、445、500、631和694 kb (图2)。图2
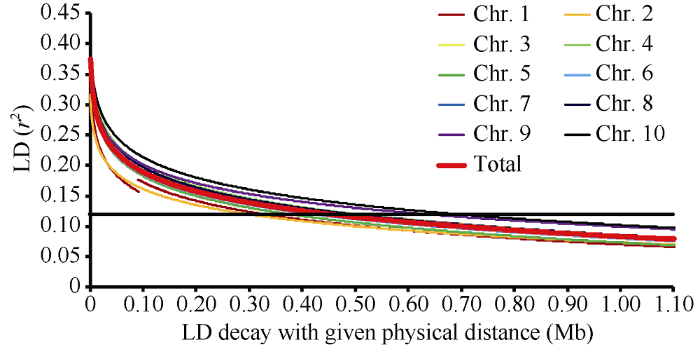 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2285份自交系全基因组LD衰减图
Fig. 2Picture of whole-genome LD in the panel of 285 lines
2.3 叶向值的全基因组关联分析
2.3.1 阈值确定 利用GEC软件分析39,827个高质量SNP的有效标记数表明, 有效标记数Ne为24,703个, 建议P值为4.05E-5, 即-lg P为4.39。因此, 该值将作为叶向值全基因组关联分析中标记-性状显著关联的阈值。2.3.2 模型选择 利用3种模型(Q、K和Q+K)分别对2个年份叶向值及其BLUP值进行了GWAS分析。QQ图显示(图3), Q模型(图中绿色点)和K模型(图中蓝色点)对假阳性的控制效果较差, 而Q+K模型(图中紫色点)对假阳性的控制较好。因此, 全基因组关联分析利用基于Q+K的混合线性模型来检测与叶向值显著相关的SNP。
图3
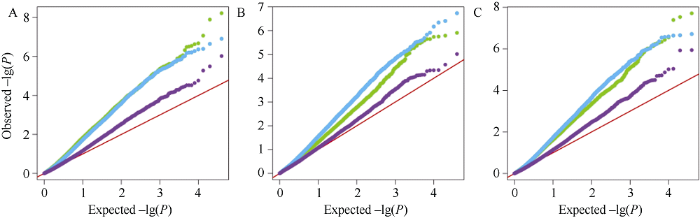 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3不同年份玉米叶向值3种GWAS模型的QQ图
A: 2017年; B: 2018年; C: 2年联合的BLUP值。绿色点: Q模型; 蓝色点: K模型; 紫色点: Q+K模型。
Fig. 3Quantile-quantile (QQ) plots resulting from GWAS results using three methods for leaf orientation value in two years
A: Year of 2017; B: Year of 2018; C: BLUP for combined two years. Green spot: Q model; blue spot: K model; purple spot: Q+K model.
2.3.3 全基因组关联分析 利用覆盖基因组的39,827个SNP标记结合285份多样性自交系连续2年的叶向值表现及其BLUP值进行了全基因组关联分析。基于Q+K模型, 一共检测到27个与叶向值显著关联的SNP (P<4.05E-5)(图4和表3)。
图4
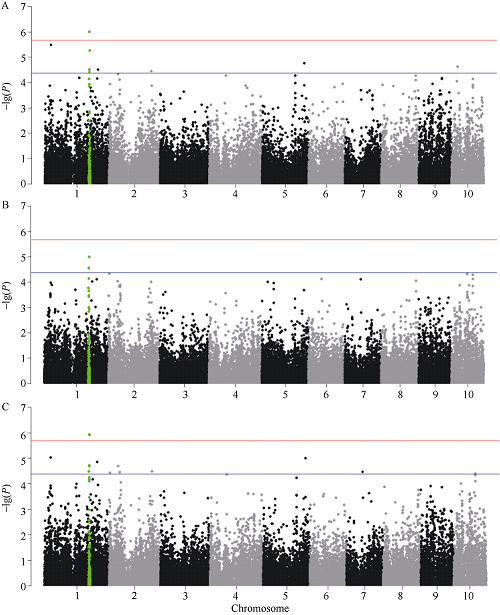 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4不同环境下玉米叶向值GWAS (Q+K模型)的曼哈顿图
A: 2017年; B: 2018年; C: 2年联合的BLUP值。绿色点: 位点2峰值SNP上下游各50个SNP在不同年份的曼哈顿图。
Fig. 4Manhattan plots resulting from GWAS results using Q+K methods for leaf orientation value in two years
A: Year of 2017; B: Year of 2018; C: BLUP for combined two years. Green spots: the Manhattan plots of 50 SNPs in the upstream and downstream of lead SNP at site 2 in two years.
Table 3
表3
表3不同年份玉米叶向值全基因组关联分析检测的显著的SNP位点及其候选基因
Table 3
| 位点a Locusa | Bin | 年份 Year | SNP | 基因型 Genotype | 物理位置b Physical position | P值 P-value | 贡献率 R2 (%) | 峰值SNP 物理位置b Lead SNP physical position | QTL区间b QTL region b | 候选基因 Candidate gene | 基因功能注释 Gene annotation | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| -LD | +LD | |||||||||||
| 1 | 1.02 | BLUP | PUT-163a-149085801-877 | [A/G] | 28,638,770 | 9.25E-06 | 7.95 | 28,638,770 | 28,306,770 | 28,638,770 | Zm00001d028265 | 细胞分裂素响应因子 Cytokinin response regulator |
| 1 | 1.02 | 2017 | PUT-163a-149085801-877 | [A/G] | 28,638,770 | 3.21E-06 | 8.73 | |||||
| 2 | 1.07 | BLUP | PZE-101164453 | [T/C] | 210,517,983 | 3.22E-05 | 6.94 | 211,113,165 | 210,781,165 | 211,445,165 | Zm00001d032050 | 细胞周期蛋白依赖性蛋白激酶 Regulation of cyclin-dependent protein serine/threonine kinase activity |
| 2 | 1.07 | BLUP | PZE-101164815 | [A/G] | 210,772,541 | 1.16E-06 | 8.15 | |||||
| 2 | 1.07 | 2017 | PZE-101164815 | [A/G] | 210,772,541 | 9.67E-07 | 8.49 | |||||
| 2 | 1.07 | 2018 | PZE-101164815 | [A/G] | 210,772,541 | 2.71E-05 | 6.24 | |||||
| 2 | 1.07 | 2017 | SYN25384 | [A/G] | 211,113,165 | 3.97E-05 | 6.99 | |||||
| 2 | 1.07 | BLUP | SYN25375 | [T/C] | 211,116,324 | 1.90E-05 | 7.32 | |||||
| 2 | 1.07 | 2017 | SYN25375 | [T/C] | 211,116,324 | 3.78E-05 | 6.93 | |||||
| 3 | 1.07 | BLUP | PZE-101165699 | [A/G] | 211,785,378 | 2.04E-05 | 7.55 | 211,785,378 | 211,453,378 | 212,117,378 | Zm00001d032078 | 细胞分裂周期相关蛋白激酶 Cell division cycle 2-related protein kinase 7 |
| 3 | 1.07 | 2017 | PZE-101165699 | [A/G] | 211,785,378 | 3.02E-05 | 7.30 | |||||
| 4 | 1.07 | BLUP | PZE-101166565 | [A/G] | 212,687,705 | 1.20E-06 | 7.65 | 212,687,705 | 212,355,705 | 213,019,705 | Zm00001d032100 | 果胶酶/果胶酶抑制剂 Pectinesterase/pectinesterase inhibitor 13 |
| 4 | 1.07 | 2017 | PZE-101166565 | [A/G] | 212,687,705 | 5.31E-06 | 6.93 | |||||
| 4 | 1.07 | 2018 | PZE-101166565 | [A/G] | 212,687,705 | 9.69E-06 | 6.67 | |||||
| 5 | 1.08 | BLUP | PZE-101196520 | [A/G] | 248,302,322 | 1.38E-05 | 7.78 | 248,302,322 | 247,970,322 | 248,634,322 | Zm00001d033047 | 含SPX结构域蛋白 SPX domain-containing protein 3 |
| 6 | 1.08 | 2017 | PZE-101199858 | [T/C] | 252,942,320 | 2.99E-05 | 6.73 | 252,942,320 | 252,610,320 | 253,274,320 | Zm00001d033180 | 油菜素内酯缺乏性矮秆基因 Brassinosteroid-deficient dwarf1,brd1 |
| 6 | 1.08 | 2017 | PZE-101199859 | [A/G] | 252,942,792 | 2.99E-05 | 6.73 | |||||
| 7 | 2.01 | BLUP | SYN9223 | [A/G] | 3,825,340 | 3.71E-05 | 7.11 | 3,825,340 | 3,526,340 | 4,124,340 | Zm00001d001968 | 细胞周期相关蛋白 Cyclin-related |
| 8 | 2.04 | BLUP | PZE-102062747 | [A/C] | 43,019,177 | 1.98E-05 | 7.91 | 43,019,177 | 42,720,177 | 43,318,177 | Zm00001d003401 | 类似14-3-3蛋白 14-3-3-like protein GF14-6 |
| 9 | 2.04 | BLUP | SYN29642 | [T/C] | 50,700,996 | 3.50E-05 | 6.15 | 50,700,996 | 50,401,996 | 50,999,996 | Zm00001d003626 | NAC-转录因子 NAC-transcription factor 76 |
| 10 | 2.07 | 2017 | PZE-102150850 | [T/G] | 203,858,592 | 3.50E-05 | 6.82 | 203,858,592 | 203,559,592 | 204,157,592 | Zm00001d006293 | NLP转录因子 NLP-transcription factor 17 |
| 11 | 2.07 | BLUP | PZE-102152182 | [A/G] | 205,338,006 | 3.24E-05 | 5.54 | 205,338,006 | 205,039,006 | 205,637,006 | Zm00001d006348 | 生长调节因子 Growth-regulating factor 9 |
| 12 | 5.06 | BLUP | PZE-105142621 | [A/G] | 201,997,615 | 9.76E-06 | 7.70 | 201,997,615 | 201,615,615 | 202,379,615 | Zm00001d017618 | ABI3-VP1-转录因子 ABI3-VP1-transcription factor 16 |
| 12 | 5.06 | 2017 | PZE-105142621 | [A/G] | 201,997,615 | 1.72E-05 | 7.52 | |||||
| 13 | 7.02 | BLUP | PZE-107043106 | [A/G] | 87,913,892 | 3.32E-05 | 8.24 | 87,913,892 | 87,468,892 | 88,358,892 | Zm00001d020041 | 参与细胞壁果胶代谢蛋白 Cell wall pectin metabolic process |
| 14 | 10.03 | 2017 | PZE-110020884 | [T/C] | 28,391,702 | 2.32E-05 | 7.65 | 28,391,702 | 27,697,702 | 29,085,702 | Zm00001d023927 | 锌指CCCH结构域蛋白 Zinc finger CCCH domain-containing protein 30 |
| 15 | 10.04 | BLUP | PZE-110053328 | [A/G] | 100,750,723 | 4.02E-05 | 6.73 | 100,750,723 | 100,056,723 | 101,444,723 | Zm00001d025033 | TCP转录因子 TCP-transcription factor 40 |
新窗口打开|下载CSV
基于每条染色体的LD衰减距离, 27个与叶向值显著关联的SNP分布于基因组的15个位点, 分别位于1号、2号、5号、7号和10号5条染色体上(图5), 其中2017年、2018年和BLUP下分别检测到8个、2个和12个位点。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5本研究检测的与叶向值显著关联的位点在染色体上的分布及与前人研究结果的比较
染色体上粗体的bin代表本研究检测的与叶向值显著关联的位点。
Fig. 5Distribution of the loci significantly association with leaf orientation value detected in this study on chromosomes and the comparison with the results of previous studies
Bin with bold on the chromosomes represents the site detected in this study that is significantly association with leaf orientation value.
位点1位于1.02 bin上, 能够在2017年和2个年份联合分析(BLUP)下检测到, 可以解释8.73%和7.95%的表型变异。位点2、3和4都位于1.07 bin上, 其中位点2能够在2017年和2018年2个环境以及2环境联合(BLUP)均被检测到, 且发现7个与叶向值显著关联的SNP, 可以解释6.24%~8.49%的表型变异; 位点3能够在2017年和2个年份联合分析(BLUP)下检测到, 可以解释7.30%和7.55%的表型变异; 位点4能够在2017年、2018年和2环境联合(BLUP)检测到, 与叶向值显著关联的SNP都为PZE-101166565, 可以解释6.67%~7.65%的表型变异。位点5、6、7、8、9、10、11、13、14和15, 分布在1.08 bin、2.01 bin、2.04 bin、2.07 bin、7.02 bin、10.03 bin和10.04 bin上, 仅在1个环境或者2环境联合(BLUP)下检测到, 可解释5.54%~8.24%的表型变异。位点12位于5.06 bin上, 能够在2017年和2个年份联合分析(BLUP)下检测到, 可以解释7.52%和7.70%的表型变异。
2.3.4 叶向值候选基因挖掘 基于该群体每条染色体的LD衰减距离, 在每个位点内-log10(P)峰值的SNP上下游各LD衰减距离的区间范围内挖掘候选基因。在与叶向值显著关联的15个位点内共计挖掘15个候选基因(表3)。位点1至位点15的候选基因依次为Zm00001d028265、Zm00001d032050、Zm00001d032078、Zm00001d032100、Zm00001d033047、Zm00001d033180、Zm00001d001968、Zm00001d003401、Zm00001d003626、Zm00001d006293、Zm00001d006348、Zm00001d017618、Zm00001d020041、Zm00001d023927和Zm00001d025033。这些基因主要是与叶枕部位细胞分裂和细胞伸长、植物激素的生物合成和信号转导、细胞壁的生物合成或降解或者一些转录因子有关的基因。
3 讨论
3.1 叶向值全基因组关联分析结果分析与比较
根据叶向值的计算公式[21]可知, 叶向值是反映叶片“直”和“立”两个特性的综合指标, 叶夹角越小(“立”), 叶片越直而不弯(“直”), 叶向值越大。前人研究也表明, 叶向值与叶夹角极显著负相关(相关系数为-0.72至-0.81), 但是与叶长和叶宽相关性不显著[9,14]。Zhao等[6]发现14个具有叶向值QTL效应的mQTL中, 约78.60%具有叶夹角QTL效应。因此, 作为叶向值构成因素之一的叶夹角, 其发育调控的关键基因也可能对叶向值的形态建成起重要作用[27]。本研究共检测到15个与叶向值显著关联的位点, 这些位点可能是玉米叶向值发育调控的重点区域。其中, 位点2能够在2017年和2018年2个年份环境, 以及2年份联合的BLUP中均被检测到(图4), 且包含最多显著关联的SNP, 效应最大的SNP解释表型变异超过8.00%, 表明这个位点遗传效应较大, 受环境影响较小, 是叶向值分子设计育种或者叶向值QTL图位克隆工作重点关注的位点。
由于前人检测的叶向值QTL主要是采用低密度的SSR分子标记和基因重组次数有限的连锁作图群体(如F2:3或RILs群体), QTL定位的精度较低。因此, 在与前人研究结果相比较时, 若QTL位于相同bin, 则认为不同研究检测的QTL是同一位点(图5)[28]。
位于1.02 bin上的位点1, 在2017年解释叶向值表型变异超过8.00%, 遗传效应较大。前人利用不同群体通过连锁分析在该位点检测到控制叶向值的QTL[10-11,14]和叶夹角的QTL[9,17,29-32]。其中, 张姿丽等[11]同时在3个环境在该位点都检测到叶向值主效QTL, 可解释10.10%~19.90%的表型变异。于永涛等[29]、刘正等[30]、Chen等[31]在该位点检测到叶夹角主效QTL, 解释14.00%~26.99%的表型变异。Ku等[9]利用豫82和沈137构建的229个F2:3家系群体, 通过3个环境下联合连锁分析, 在1.02 bin位点上同时检测到控制叶夹角和叶向值的主效QTL, LOD值分别为10.8和11.0, 分别解释20.40%和23.20%的表型变异。Ku等[5]和Zhao等[6]整合前人研究也发现该位点同时存在叶向值和叶夹角的QTL。因此, 该位点可能存在1个或几个不同环境不同遗传背景稳定表达的、同时控制叶夹角和叶向值的主效基因, 是玉米叶片形态建成的重要位点, 可用于玉米株型分子育种的前景标记选择。
位点2、3、4均位于1.07 bin上, 其中位点2是本研究重点发现的位点。利用不同遗传群体, Zhao等[6]和Ding等[32]分别都在2个环境下在该位点检测到叶夹角QTL, 解释4.88%~6.97%表型变异。然而, 可能由于群体类型、标记密度或是叶向值QTL定位研究相对较少, 目前还未发现该位点在调控叶向值中的重要作用。本研究发现了3个位点共计12个与叶向值显著相关的SNP标记都位于1.07 bin上, 提示该区段可能至少有3个重要基因参与叶向值的发育调控。
位点5和位点6都位于1.08 bin上, 包含3个与叶向值显著关联的SNP标记, 在该位点发现的叶向值和叶夹角QTL较少。Zhao等[6]在不同水分处理条件下, 在该位点发现了稳定遗传的叶夹角QTL, 可解释5.30%~20.58%的表型变异。Ding等[32]也在该位点发现叶夹角QTL。因此, 该位点的遗传效应, 还需要更多研究来证实。
位于2.01 bin上的位点7, 在2年联合的BLUP下被检测到, 可解释7.11%的叶向值表型变异。前人利用不同群体在不同环境下在该位点都检测到了叶向值QTL[5,7]和叶夹角QTL[7,14,32]。其中, Ku等[5]和Zhao等[6]分别整合前人关于叶夹角和叶向值QTL定位结果, 通过元分析发现2.01 bin位点存在同时调控叶向值和叶夹角的mQTL。
位点8和位点9都位于2.04 bin上, 都是在2年联合的BLUP下发现的, 可解释6.15%~7.91%的叶向值表型变异。路明等[7]、徐德林等[33]、刘鹏飞等[10]和张姿丽等[11]在该位点均检测到叶向值QTL, 分别解释10.92%、5.67%、10.60%和8.90%的表型变异。Ding等[32]还在该位点检测到叶夹角QTL, 解释4.54%表型变异。因此, 该位点可能存在调控玉米叶向值的主效基因。
2.07 bin上挖掘到位点10和位点11共2个控制叶向值的位点, 可解释5.54%~6.82%表型变异。刘鹏飞等[10]和张姿丽等[11]分别在该位点检测到叶向值QTL, 解释6.90%和5.40%的表型变异。总体来看, QTL解释的表型变异都较低。因此, 该位点可能存在效应较小的叶向值调控基因。
位于5.06 bin的位点12附近检测到多个叶向值和叶夹角QTL[5-7,11,14,34], 其中, 张姿丽等[11]在5.05~ 5.07 bin上检测到的叶向值QTL可解释7.80%~7.90%表型变异, 叶夹角QTL解释9.30%~9.40%表型变异。Ku等[5]在5.06检测到1个mQTL, 同时包含叶夹角QTL和叶向值QTL。Zhao等[6]在5.05~5.06 bin上同样检测到环境稳定遗传的叶向值QTL和叶夹角QTL, 可解释3.38%~8.99%的表型变异。该位点还需进一步精细定位, 挖掘其潜在的候选基因。
位点13位于7.02 bin上, 只在2年联合的BLUP下检测到, 解释叶向值表型变异较高(8.24%)。在该位点上, 张姿丽等[11]检测到1个不同环境稳定遗传的叶向值主效QTL, 可解释16.40%~20.10%的表型变异; Lu等[18]通过GWAS方法分别检测到1个叶向值和1个叶夹角QTL。此外, 一些研究[5,32,34]也在该位点检测到叶夹角QTL。因此, 该基因组区段可能是玉米叶型性状重要的调控区段。
位点14和位点15分别位于10.03 bin和10.04 bin, 分别在2017年和2年联合的BLUP中被检测到。前人尚未在这两个染色体区段检测到调控叶向值和叶夹角的QTL。
3.2 叶向值候选基因挖掘
叶的发育是植物形态建成的一个重要部分, 其调控机理十分复杂。叶原基形成后, 在一系列基因精确调控下, 叶原基开始建立近-远轴(面向茎的为近轴面, 背向茎的为远轴面)、基-顶轴(由叶的基部指向尖部)和中-边轴(从叶的主脉指向边缘)三维轴向上的极性[35], 引导原基细胞朝着特定的方向分裂和分化, 最终发育成一定形态和大小的叶片。叶向值的形成与基-顶轴和近-远轴极性的建立有关。根据与叶向值显著关联的峰值SNP物理位置及LD衰减距离, 从15个位点共计挖掘最有可能的候选基因15个(表3)。与前人探索的叶夹角分子调控机制类似, 叶向值候选基因主要与叶枕部位细胞分裂和细胞伸长、植物激素的生物合成和信号转导有关[2,12-13,16,36-37]。
植物激素的生物合成和信号转导途径是叶片发育调控重要分子机制之一。位点1的候选基因Zm00001d028265 (cytokinin response regulator8, crr8), 其功能注释与细胞分裂素信号转导有关。细胞分裂素是植物生长所必需的激素, 主要作用是调节细胞增殖[38]。因此, 推测Zm00001d028265基因可能通过影响细胞分裂素的信号转导来调控叶枕部位细胞分裂或者叶片近-远轴面厚壁组织细胞不对称发育等途径实现对叶向值的调控。此外, 1.02 bin上的Zm00001d028216基因(indeterminate floral apex1, ifa1)编码C2C2-YABBY类型转录因子, 突变体叶片不能形成正常的中脉, 叶夹角减小, 叶片稍短, 叶片较窄, 叶鞘较长, 表现披叶的特性[39]。ifa1与本研究位点1峰值SNP距离1.86 Mb。因此, 1.02 bin上可能存在多个控制叶向值的主效基因。位点6的候选基因Zm00001d033180是参与油菜素内酯(BR)合成途径的brd1 (brassinosteroid-deficient dwarf1)基因, 距离本研究峰值SNP仅220 kb, 该QTL已被Tian等[2]图位克隆。brd1通过影响叶枕内源BR水平控制叶枕细胞增殖, 最终影响叶夹角的大小。进一步证实了本研究所用关联群体通过GWAS手段可有效挖掘调控叶向值的基因资源。位点5候选基因Zm00001d033047编码一种SPX结构域蛋白, 参与细胞对磷胁迫和冷胁迫响应的生物过程。Ruan等[40]研究表明, 水稻磷饥饿诱导蛋白SPX1(Syg1/Pho81/XPR1)和SPX2负调控叶片倾斜角度。磷缺乏诱导产生SPX1蛋白, 后者与叶倾角调节因子1(RLI1)直接相互作用, 阻止RLI1与下游基因BRASSINOSTEROID UPREGULATED1 (BU1)和BU1-LIKE 1 COMPLEX1启动子结合, 从而抑制水稻叶枕细胞伸长, 限制叶枕的大小, 诱导叶片直立。位点8候选基因Zm00001d003401编码类似14-3-3蛋白, 是植物初级代谢和细胞信号转导的主要调节因子, 参与油菜素内酯的信号转导[41]。位点11候选基因Zm00001d006348, 编码生长调节因子(GRF-transcription factor 2, GRF2), 是一种转录激活因子, 通过控制细胞增殖来调节分生组织功能[42]。在拟南芥叶片形态建成中, 由miR396介导的生长调节因子(AtGRFs)调控叶片近-远轴向的细胞极性分化[42]。位点12候选基因Zm00001d017618编码一种B3转录因子, 调节BR合成途径和信号转导途径基因的表达[2,43]。最近Tian等[2]发现玉米2号染色体上的B3转录因子Zm00001d002562 (ABI3-VP1-transcription factor 12)基因(即ZmRAVL1)激活下游基因brd1的表达, 从而提高叶枕内源BR水平, 影响叶枕细胞的增殖, 导致叶夹角增大。位点14候选基因Zm00001 d023927编码一种含锌指CCCH结构域的蛋白质。Wang等[44]研究表明, 水稻中OsLIC基因编码一种锌指CCCH结构域蛋白, 负调控BR信号。抑制内源OsLIC基因表达, 会导致水稻叶片和分蘖角显著增加, 株高降低, 从而降低产量。
叶枕部位近轴面和远轴面细胞分裂的不平衡也可调控叶向值。位点2是本研究发现的最重要位点, 其候选基因Zm00001d032050功能注释具有细胞周期蛋白依赖性蛋白激酶(cyclin-dependent protein kinase, CDK)活性。该蛋白激酶是细胞周期调控机制的核心, 细胞周期蛋白(cyclin)依次激活相应的细胞周期蛋白依赖性激酶(CDK), 推动细胞跨越细胞周期各时相转换的限制点, 是推动细胞周期运行的动力因素[45]。其水稻同源基因CycD2 (Os06g0217900)是一种与Cyclin-D3-1类似的细胞周期蛋白。水稻细胞周期蛋白可通过控制远轴面厚壁组织细胞增殖, 正向调控叶片的直立性, 已被前人证实[46]。因此, 有必要进一步精细定位并图位克隆该位点的功能基因。位点3的候选基因Zm00001d032078, 同样具有调节细胞分裂和细胞周期的蛋白酶活性。其水稻同源基因CDKC (Os08g0453800)也是一种细胞周期蛋白依赖性蛋白激酶。位点7候选基因为Zm00001 d001968编码一种细胞周期类似蛋白。位点10候选基因Zm00001d006293编码NLP转录因子。有研究表明, 在缺氮条件下类似NIN蛋白转录因子(NLP转录因子)与Teostine branched1/cycloidea/proliferating cell factor (TCP20)相互作用, 调控细胞周期蛋白基因CYCB1;1[47]。位点15候选基因Zm00001d025033编码一种TCP转录因子。TCP蛋白是植物特有的bHLH转录因子。在玉米中有两个TCP家族的基因被较深入研究。一个是BAD1 (branch angle deffective 1)编码TCPⅡ类基因, 通过促进细胞增殖影响侧枝角度的出现, 并影响花序结构[48]。另一个是玉米驯化基因TB1 (teosinte branched 1), 可抑制侧枝的生长和发育, TB1基因突变体导致玉米侧枝的增加[49]。
叶枕部位机械支撑也是调控叶向值的重要机制。位点4的候选基因Zm00001d032100编码果胶酯酶, 参与果胶分解代谢过程和细胞壁修饰等生物过程。果胶是植物细胞壁的重要组成之一, 与植物组织结构形成等生物过程密切相关。果胶酯酶对细胞壁的降解[50]和果实软化[51]具有重要作用。已有研究发现, 叶枕部位细胞壁机械强度降低, 叶夹角增大[52]。位点13候选基因Zm00001d020041参与细胞壁果胶代谢生物过程, 可能影响机械强度或者细胞扩展[53]来调控叶向值。
位点9候选基因Zm00001d003626编码NAC转录因子, 该类型转录因子对番茄叶发育具有重要作用[54], 其调控玉米叶向值的机理还需进一步研究。
4 结论
发现15个与叶向值显著关联(P<4.05E-5)的位点, 发掘出15个候选基因, 其中1.07 bin是最重要位点, 其候选基因Zm00001d032050有待进一步利用图位克隆技术和CRISPR/Cas9基因敲除等技术验证。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 2]
[本文引用: 7]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 9]
.
[本文引用: 10]
[本文引用: 6]
[本文引用: 6]
[本文引用: 2]
[本文引用: 2]
[本文引用: 5]
[本文引用: 5]
[本文引用: 5]
[本文引用: 9]
[本文引用: 9]
[本文引用: 4]
[本文引用: 3]
[本文引用: 5]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
DOI:10.1186/s12870-015-0646-7URL [本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
DOI:10.1371/journal.pone.0141619URL [本文引用: 6]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1016/S1360-1385(01)02045-3URL [本文引用: 1]
DOI:10.1093/jxb/erh227URL [本文引用: 1]
DOI:10.1105/tpc.111.093419URL [本文引用: 1]

Mitogen-activated protein kinase kinase kinases (MAPKKKs), which function at the top level of mitogen-activated protein kinase cascades, are clustered into three groups. However, no Group C Raf-like MAPKKKs have yet been functionally identified. We report here the characterization of a rice (Oryza sativa) mutant, increased leaf angle1 (ila1), resulting from a T-DNA insertion in a Group C MAPKKK gene. The increased leaf angle in ila1 is caused by abnormal vascular bundle formation and cell wall composition in the leaf lamina joint, as distinct from the mechanism observed in brassinosteroid-related mutants. Phosphorylation assays revealed that ILA1 is a functional kinase with Ser/Thr kinase activity. ILA1 is predominantly resident in the nucleus and expressed in the vascular bundles of leaf lamina joints. Yeast two-hybrid screening identified six closely related ILA1 interacting proteins (IIPs) of unknown function. Using representative IIPs, the interaction of ILA1 and IIPs was confirmed in vivo. IIPs were localized in the nucleus and showed transactivation activity. Furthermore, ILA1 could phosphorylate IIP4, indicating that IIPs may be the downstream substrates of ILA1. Microarray analyses of leaf lamina joints provided additional evidence for alterations in mechanical strength in ila1. ILA1 is thus a key factor regulating mechanical tissue formation at the leaf lamina joint.
DOI:10.1007/s00709-011-0371-5URL [本文引用: 1]
[本文引用: 1]
