 ,*江苏省农业科学院 / 江苏省农业生物学重点实验室 / 江苏省现代作物生产协同创新中心, 江苏南京 210014
,*江苏省农业科学院 / 江苏省农业生物学重点实验室 / 江苏省现代作物生产协同创新中心, 江苏南京 210014Genetic analysis of plant height and its components for wheat (Triticum aestivum L.) cultivars Ningmai 9 and Yangmai 158
JIANG Peng, HE Yi, ZHANG Xu, WU Lei, ZHANG Ping-Ping, MA Hong-Xiang ,*Jiangsu Academy of Agricultural Sciences / Jiangsu Provincial Key Laboratory for Agrobiology / Jiangsu Collaborative Innovation Center for Modern Crop Production, Nanjing 210014, Jiangsu, China
,*Jiangsu Academy of Agricultural Sciences / Jiangsu Provincial Key Laboratory for Agrobiology / Jiangsu Collaborative Innovation Center for Modern Crop Production, Nanjing 210014, Jiangsu, China通讯作者:
收稿日期:2019-10-15接受日期:2020-01-15网络出版日期:2020-06-12
| 基金资助: |
Received:2019-10-15Accepted:2020-01-15Online:2020-06-12
| Fund supported: |
作者简介 About authors
E-mail:hmjp2005@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (1853KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
姜朋, 何漪, 张旭, 吴磊, 张平平, 马鸿翔. 宁麦9号与扬麦158株高及其构成因素的遗传解析[J]. 作物学报, 2020, 46(6): 858-868. doi:10.3724/SP.J.1006.2020.91063
JIANG Peng, HE Yi, ZHANG Xu, WU Lei, ZHANG Ping-Ping, MA Hong-Xiang.
株高是小麦重要的农艺性状, 影响植株的形态结构, 并与田间群体产量密切相关。小麦矮秆基因的利用是绿色革命的重要组成部分, 对现代小麦育种具有深远影响[1]。经典遗传学研究表明, 小麦株高是一个复杂性状, 由多个基因控制, 存在主效基因, 也有微效位点。迄今为止, 已有25个Rht基因被命名[2,3,4]。位于 4B 和 4D 染色体上的Rht-B1和Rht-D1基因, 以及2D染色体上的Rht8基因在世界范围内广为应用, 其相关分子标记已成功开发[5,6]。此外, 在小麦21条染色体上均检测到影响株高的QTL[7,8,9,10,11]。McCartney等[12]利用RL4452בAC’ Domain构建的DH群体对包括株高在内的多个农艺性状进行QTL分析, 在 2D、 4B、4D、5B、7A和7B染色体检测到6个株高QTL, 其中QHt.crc-4B和QHt.crc-4D的定位区间与Rht-B1和Rht-D1位置重合。Griffiths等[13]利用4个DH群体进行了株高meta-QTL分析, 在除3D、4A、5D及第7同源群外的15条染色体上找到16个meta-QTL。Liu等[9]在5个生长阶段对小麦株高进行了跟踪调查, 利用条件与非条件QTL作图方法进行分析, 检测到8个条件QTL与9个非条件QTL。其中QHt-4B-2在2个时期被重复检测到, 表型贡献率达13.42%~16.13%, 此研究表明株高基因的表达具有一定的时空特异性。小麦株高是主穗长与各节间长的总和, 但控制主穗长及各茎节间长的遗传位点对小麦株高的遗传贡献并不一致。Cui等[14]利用条件QTL与非条件QTL相结合的分析方法在单个QTL水平揭示了株高与穗长及各节间长之间的关系, 其中倒三节对株高影响最大。Zhang等[15]测定了不同时期的各个节间长度, 明确了5个稳定的株高QTL的表达模式及贡献率。
遗传连锁图为遗传定位、标记开发及候选基因发掘等提供了强有力的工具。近年来发展起来的SNP标记具有遗传稳定、数量多、分布广等特点, 并且适于高通量检测, 基于其开发的9k、90k、660k等基因型芯片集合了成千上万个SNP标记, 不仅加快了小麦遗传图谱构建速度, 也大大提高了小麦遗传图谱的密度[16,17,18]。中国春小麦参考基因组序列的公布, 有助于小麦遗传图谱质量的提高, 并可直接基于目标区段进行候选基因预测[19]。
长江中下游麦区是我国第二大麦区, 同时也是我国最大的弱筋小麦产区, 是我国小麦生产的重要组成部分。宁麦9号与扬麦158分别是由江苏省农业科学院与江苏里下河地区农业科学研究所育成的高产优质抗病小麦品种, 都曾经是生产上的主栽品种, 具有较大的推广面积, 而且目前仍是重要的骨干亲本, 以此为基础育成了数十个小麦品种[20,21]。宁麦9号的衍生品种宁麦13、扬辐麦4号等, 扬麦158的衍生品种扬麦20等均已成为当前生产中的主栽品种及常用亲本。在生产中, 宁麦9号株高一般在80~ 85 cm, 扬麦158约为90~95 cm, 呈现较大差异, 明确其株高遗传机制对小麦育种工作具有重要的指导意义。本研究以来源于宁麦9号/扬麦158的282份重组自交系为材料, 结合高密度遗传图谱, 对株高及其构成开展QTL定位研究, 以期为长江中下游麦区育种工作中的株高选择提供帮助。
1 材料与方法
1.1 试验材料
以宁麦9号×扬麦158构建重组自交系(RIL)群体(F2:8), 包括282个家系。宁麦9号是扬麦6号与日本西风小麦的杂交后代, 前者具有骨干亲本南大2419与江东门的遗传背景; 扬麦158来源于St1472/506与扬麦4号杂交组合, St1472/506是黄淮麦区的常用亲本, 扬麦4号含有南大2419、胜利麦与阿夫等多个骨干亲本的遗传背景。宁麦9号与扬麦158株高及各节间长度、穗长等性状差异显著(图1)。通过Ellis等[5]和Asplund等[6]报道的分子标记确定宁麦9号与扬麦158均为Rht-B1b/ Rht-D1a变异类型, 均不含Rht8位点(图2), 其株高差异可能来源于其他位点。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1宁麦9号与扬麦158的田间表现
Fig. 1Field phenotype for Ningmai 9 and Yangmai 158
图2
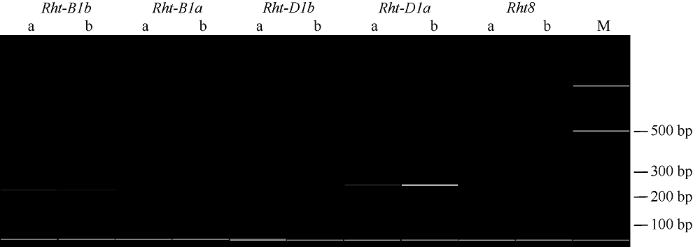 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2宁麦9号与扬麦158株高相关分子标记检测
a: 宁麦9号; b: 扬麦158; M: marker。
Fig. 2Molecular marker test of plant height for Ningmai 9 and Yangmai 158
a: Ningmai 9; b: Yangmai 158; M: marker.
1.2 田间试验及数据处理
2016—2017、2017—2018和2018—2019连续3个生长季将RIL群体及其亲本种植于江苏省农业科学院六合基地, 为方便描述, 以收获年份2017、2018和2019分别表示3个环境。采用随机区组设计, 单行种植, 每行60粒, 行长1.6 m, 行距0.25 m, 2次重复, 常规田间管理。于小麦乳熟期从每个家系中取10个单茎, 从茎基部开始向上利用直尺测量每个节间-倒五节(The fifth internode from the top, D5)、倒四节(The fourth internode from the top, D4)、倒三节(The third internode from the top, D3)、倒二节(The second internode from the top, D2)、倒一节(The first internode from the top, D1)长度及穗长, 株高为各节间与穗长之和, 并计算平均值用于进一步统计分析。另同时测量101份2018国家及省区域试验材料的株高, 用于后续验证。
采用Microsoft Excel 2016进行表型初步统计与相关分析, 采用SPSS 19.0进行方差分析及t检测。依据He等[22]的方法, 按h2 = δg2/(δg2 +δg×y2/y +δe2/ry)计算遗传力, 其中δg2表示基因型方差, δg×y2表示基因型与环境互作方差, δe2为误差, y代表环境数目, r代表重复数。
1.3 遗传图谱与QTL分析
采用Illumina 90k芯片获取基因型。遗传图谱覆盖21条染色体, 包含41个连锁群, 2285个bin标记, 总长为3022 cM[23]。采用QTL IciMapping 4.1软件[24]的完备区间作图法(inclusive composite interval mapping, ICIM)进行QTL定位[25], 设walking step为0.1 cM, LOD阈值为2.5。1.4 KASP标记的开发
根据SNP位点和侧翼序列设计PCR扩增引物, 开发KASP分子标记。设计每个标记2条SNP特异性引物(F1/F2)和一条通用引物(R), F1尾部添加能够与FAM荧光结合的特异性序列, F2尾部添加能够与HEX荧光结合的特异性序列。利用Polymarker (http://www.polymarker.info/)设计KASP引物, 由生工生物工程(上海)股份有限公司合成。从RILs群体中随机挑选46份材料进行幼嫩叶片取样, 采用CTAB法[25]提取基因组DNA。KASP反应总体系为5 μL, 包含2×KASP Master Mix 2.5 μL、KASP Assay Mix (引物混合工作液) 0.07 μL、浓度为20 ng μL-1的模板DNA 2.43 μL。KASP反应程序第一步为94℃, 15 min; 第二步为94℃, 20 s, 61~55℃, 1 min, 每个循环降低0.6℃, 共进行10个循环; 第三步为94℃, 20 s, 55℃, 1 min, 共进行26个循环, 在LGC公司为Hydrocycler-16水浴PCR仪中进行PCR。通过KASP荧光分析仪(LGC公司型号为PHERAstar plus)扫描分析PCR结果。
2 结果与分析
2.1 株高及其构成因素的表型分析
在连续3年的调查中(表1), 扬麦158的各节间长度、穗长及株高均高于宁麦9号, 其中D5与D4差异较小, 一般为1~2 cm, D3、D2与D1差异较大, 约为2~3 cm, 穗长相差约1 cm, 最终造成株高相差约10 cm。在RIL群体中, 各节间长度、穗长及株高均呈现较大变异, D5变异系数最大, 约20%, 其余节间、穗长及株高变异系数在10%左右, 最大株高与最小株高差异均超过30 cm, 节间差距从D5的5 cm左右递增至D1的近20 cm。从D5至D1, 性状遗传力从0.43逐步增大至0.83, 穗长与株高遗传力均超过0.7。Table 1
表1
表1株高及其构成因素的表型统计
Table 1
| 性状 Trait | 环境 Environment | 宁麦9号 Ningmai 9 (cm) | 扬麦158 Yangmai 158 (cm) | 重组自交系群体 RIL population | 遗传力 Heritability | ||||
|---|---|---|---|---|---|---|---|---|---|
| 最大值 Max (cm) | 最小值 Min (cm) | 平均值 Mean (cm) | 标准差 SD | 变异系数 CV(%) | |||||
| 倒五节 D5 | 2017 | 4.71 | 5.64 | 8.72 | 2.92 | 5.83 | 1.06 | 18.14 | 0.43 |
| 2018 | 4.39 | 5.66 | 10.15 | 3.49 | 5.84 | 1.06 | 18.17 | ||
| 2019 | 3.72 | 3.76 | 6.36 | 1.88 | 3.86 | 0.87 | 22.58 | ||
| 倒四节 D4 | 2017 | 7.38 | 8.05 | 12.59 | 5.58 | 8.16 | 1.04 | 12.79 | 0.51 |
| 2018 | 6.89 | 7.76 | 12.75 | 5.50 | 8.47 | 1.01 | 11.96 | ||
| 2019 | 5.28 | 6.78 | 10.63 | 4.93 | 7.04 | 0.92 | 13.10 | ||
| 倒三节 D3 | 2017 | 8.43 | 10.08 | 15.84 | 8.50 | 11.28 | 1.31 | 11.62 | 0.61 |
| 2018 | 11.56 | 13.80 | 18.87 | 9.09 | 13.05 | 1.56 | 11.92 | ||
| 2019 | 9.11 | 11.16 | 15.73 | 7.61 | 11.17 | 1.30 | 11.63 | ||
| 倒二节 D2 | 2017 | 15.65 | 19.24 | 25.58 | 12.33 | 19.62 | 1.91 | 9.74 | 0.76 |
| 2018 | 18.66 | 20.63 | 27.27 | 16.83 | 22.17 | 1.88 | 8.47 | ||
| 2019 | 17.40 | 21.34 | 26.21 | 15.66 | 21.11 | 1.82 | 8.60 | ||
| 倒一节 D1 | 2017 | 26.28 | 28.99 | 38.16 | 20.04 | 29.40 | 2.97 | 10.11 | 0.83 |
| 2018 | 29.83 | 32.31 | 42.63 | 23.87 | 32.43 | 2.82 | 8.70 | ||
| 2019 | 27.53 | 31.26 | 41.41 | 23.02 | 32.07 | 3.05 | 9.50 | ||
| 穗长 Spike length | 2017 | 9.85 | 10.43 | 12.37 | 6.50 | 9.49 | 0.89 | 9.40 | 0.72 |
| 2018 | 8.14 | 9.11 | 10.78 | 5.97 | 8.73 | 0.88 | 10.03 | ||
| 2019 | 7.31 | 8.21 | 10.33 | 5.44 | 7.82 | 0.87 | 11.13 | ||
| 株高 Plant height | 2017 | 72.29 | 82.43 | 101.93 | 59.54 | 83.84 | 6.45 | 7.69 | 0.78 |
| 2018 | 79.48 | 89.26 | 108.17 | 72.53 | 90.78 | 6.20 | 6.83 | ||
| 2019 | 70.35 | 82.51 | 102.62 | 64.25 | 83.07 | 6.11 | 7.36 | ||
新窗口打开|下载CSV
株高、穗长及各节间长度在不同基因型与不同年份间差异极显著, 同时基因型与年份的互作对其也有极显著影响(表2)。各节间长度间均呈极显著正相关, 而与穗长相关系数普遍较低。株高与各节间长度、穗长均呈极显著正相关, 而同一节间在不同环境间亦呈现出良好的相关性(表3)。
Table 2
表2
表2株高及其构成因素的方差分析(F值)
Table 2
| 项目 Item | 倒五节 D5 | 倒四节 D4 | 倒三节 D3 | 倒二节 D2 | 倒一节 D1 | 穗长 Spike length | 株高 Plant height |
|---|---|---|---|---|---|---|---|
| 基因型 Genotype | 4.15** | 7.08** | 8.37** | 18.23** | 17.98 ** | 13.91** | 40.51** |
| 环境 Environment | 1009.08** | 719.59** | 783.47** | 1205.13** | 693.65** | 1859.74** | 2491.39** |
| 基因型×环境 Genotype×Environment | 2.35** | 3.45** | 3.25** | 4.30** | 3.06** | 3.87** | 8.81** |
新窗口打开|下载CSV
Table 3
表3
表3株高及其构成因素的相关分析
Table 3
| 性状 Trait | 环境 Environment | 倒五节D5 | 倒四节D4 | 倒三节D3 | 倒二节D2 | 倒一节D1 | 穗长 Spike length | 株高 Plant height |
|---|---|---|---|---|---|---|---|---|
| 倒五节 D5 | 2017 | (0.266**) | ||||||
| 2018 | (0.294**) | |||||||
| 2019 | (0.049) | |||||||
| 倒四节 D4 | 2017 | 0.632** | (0.370**) | |||||
| 2018 | 0.551** | (0.400**) | ||||||
| 2019 | 0.714** | (0.070) | ||||||
| 倒三节 D3 | 2017 | 0.546** | 0.631** | (0.347**) | ||||
| 2018 | 0.466** | 0.697** | (0.500**) | |||||
| 2019 | 0.531** | 0.803** | (0.268**) | |||||
| 倒二节 D2 | 2017 | 0.388** | 0.510** | 0.637** | (0.506**) | |||
| 2018 | 0.285** | 0.487** | 0.549** | (0.639**) | ||||
| 2019 | 0.219** | 0.452** | 0.570** | (0.459**) | ||||
| 倒一节 D1 | 2017 | 0.242** | 0.277** | 0.309** | 0.471** | (0.616**) | ||
| 2018 | 0.163** | 0.320** | 0.184** | 0.524** | (0.733**) | |||
| 2019 | 0.057 | 0.190** | 0.198** | 0.618** | (0.512**) | |||
| 穗长 Spike length | 2017 | 0.026 | 0.055 | 0.059 | 0.169** | 0.157** | (0.467**) | |
| 2018 | -0.058 | -0.042 | -0.171** | 0.105 | 0.187** | (0.524**) | ||
| 2019 | 0.222** | 0.125* | -0.015 | 0.067 | 0.118* | (0.326**) | ||
| 株高 Plant height | 2017 | 0.610** | 0.683** | 0.737** | 0.815** | 0.771** | 0.287** | (0.533**) |
| 2018 | 0.531** | 0.719** | 0.670** | 0.821** | 0.765** | 0.199** | (0.689**) | |
| 2019 | 0.490** | 0.673** | 0.678** | 0.840** | 0.784** | 0.267** | (0.456**) |
新窗口打开|下载CSV
2.2 株高及其构成因素的QTL分析
综合3个环境表型数据, 利用完备区间作图法共鉴定到96个QTL, 分布在18条染色体上, 表型贡献率为1.72%~12.32%。其中, D5至D1分别检测到7、14、17、13和13个QTL, 与穗长相关的QTL有11个, 控制最终株高的QTL有21个。进一步对这些QTL比较分析, 最终在2A、2D、5A、5D、7A等5条染色体上获得14个多环境表达的稳定QTL (图2和表4)。其中, 控制D5、D4及D3长度的QTL均为1个, 控制D2与D1均为3个, 而控制株高的为5个, 没有检测到与穗长相关的稳定QTL。Table 4
表4
表4株高及其构成因素的QTL定位
Table 4
| 序号 No. | QTL | 遗传位置 Genetic position | 区间 Interval | 2017 | 2018 | 2019 | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LOD | 表型贡献率 PVE (%) | 加性效应 AEa | LOD | 表型贡献率 PVE (%) | 加性效应 AE | LOD | 表型贡献率 PVE (%) | 加性效应 AE | ||||||
| 1 | Qd5-5A | 52.35?55.75 | wsnp_Ex_rep_c104539_89224552-RFL_Contig4162_1285 | 5.90 | 8.89 | 0.31 | 3.11 | 4.69 | 0.24 | |||||
| 2 | Qd4-5A | 52.35?56.55 | wsnp_Ex_rep_c104539_89224552-RAC875_c103967_76 | 4.22 | 7.66 | 0.26 | 4.40 | 2.73 | 0.24 | |||||
| 3 | Qd3-5A | 52.45?59.75 | wsnp_Ex_c49211_53875575-BS00000365_51 | 6.90 | 7.89 | 0.39 | 4.23 | 5.18 | 0.38 | 4.73 | 5.25 | 0.32 | ||
| 4 | Qd2-2A | 109.15?112.05 | Tdurum_contig61938_424-wsnp_Ex_c3695_6740339 | 3.97 | 5.93 | ?0.46 | 3.34 | 4.26 | ?0.39 | |||||
| 5 | Qd2-5A | 55.85?59.25 | BS00096758_51-BS00098207_51 | 6.58 | 9.97 | 0.58 | 7.50 | 10.18 | 0.60 | |||||
| 6 | Qd2-7A | 49.55?51.95 | RAC875_c16624_970-wsnp_Ex_c14009_21899923 | 4.10 | 6.13 | ?0.48 | 8.13 | 11.05 | ?0.62 | 5.30 | 6.13 | ?0.49 | ||
| 7 | Qd1-2A | 109.35?110.25 | Tdurum_contig61938_424-IAAV880 | 4.74 | 6.53 | ?0.79 | 3.65 | 5.51 | ?0.63 | 6.22 | 9.29 | ?0.89 | ||
| 8 | Qd1-5A | 56.95?58.55 | RAC875_c103967_76-BS00098207_51 | 3.46 | 5.20 | 0.60 | 3.51 | 5.38 | 0.66 | |||||
| 9 | Qd1-5D | 9.35?13.85 | Kukri_c14878_97-Kukri_c7786_81 | 3.57 | 4.87 | 0.67 | 3.92 | 5.90 | 0.64 | 2.96 | 4.30 | 0.59 | ||
| 10 | Qph-2A | 109.35?110.55 | Tdurum_contig61938_424-IAAV880 | 4.65 | 5.79 | ?1.56 | 3.03 | 3.45 | ?1.26 | |||||
| 11 | Qph-2D | 15.55?29.65 | BS00022211_51-RAC875_c173_905 | 3.16 | 3.99 | ?1.25 | 4.48 | 4.88 | ?1.45 | |||||
| 12 | Qph-5A.1 | 18.85?26.55 | wsnp_Ex_c12440_19836844-Ex_c6161_335 | 3.07 | 3.76 | 1.21 | 2.99 | 2.84 | 1.08 | |||||
| 13 | Qph-5A.2 | 52.65?59.75 | wsnp_Ex_c49211_53875575-BS00000365_51 | 6.49 | 8.24 | 1.79 | 7.34 | 9.53 | 2.03 | 6.35 | 6.38 | 1.67 | ||
| 14 | Qph-7A | 43.45?52.05 | Ex_c6196_971-wsnp_Ex_c14009_21899923 | 7.09 | 8.94 | ?2.02 | 5.45 | 5.57 | ?1.62 | |||||
新窗口打开|下载CSV
通过位置比对发现, Qd5-5A、Qd4-5A、Qd3-5A、Qd2-5A、Qd1-5A和Qph-5A.2遗传位置接近或重合, 很可能为1个一因多效QTL, 同时控制多个节间的长度。Qd2-2A、Qd1-2A与Qph-2A, Qd2-7A与Qph-7A, 均是相同的情况。为方便描述, 将Qd5-5A/Qd4-5A/ Qd3-5A/Qd2-5A/Qd1-5A/Qph-5A.2、Qd2-2A/Qd1-2A/ Qph-2A与Qd2-7A/Qph-7A分别简写为Q5A、Q2A与Q7A。除此3个QTL外, Qph-2D对D1具有调节作用, Qph-5A.1同时对D4与D3产生影响。以株高及各节间长度减少定义其加性效应, 这3个位点对应多个性状的加性效应表现一致, Q5A的加性效应来源于扬麦158, 而Q2A与Q7A的加性效应来源于宁麦9号。此外, Qph-2D加性效应来源于宁麦9号, Qph-5A.1与Qd1-5D加性效应来源于扬麦158。
2.3 KASP标记开发
根据定位结果, 我们最终获得6个株高及其构成因素控制区段(图3), 每个区段内含有多个SNP标记, 选择其中序列同源性较低的标记进行KASP标记开发, 最终将IAAV880 (Q2A)、IACX9152 (Qph-2D)、BobWhite_c1796_701 (Qph-5A.1)、BS00098207_51 (Q5A)、Kukri_c7786_81 (Qd1-5D)与wsnp_JD_c6050_ 7214383 (Q7A)等成功转化(表5和图4)。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3株高及其构成因素的QTL定位
图中数字与
Fig. 3QTL mapping for plant height and its components
The numbers in the figure are consistent with the sequence numbers of QTL in
Table 5
表5
表5株高及其构成因素的KASP标记序列
Table 5
| 标记 Marker | QTL | SNP | F1 | F2 | R |
|---|---|---|---|---|---|
| IAAV880 | Q2A | T/C | ccAgaacgcagtggagagtT | ccAgaacgcagtggagagtC | tgcagacgggagcttagG |
| IACX9152 | Qph-2D | T/C | aacagagtctcaGtctctccA | aacagagtctcaGtctctccG | gcctgtCattctcttttcctAtCtT |
| BobWhite_c1796_701 | Qph-5A.1 | T/G | ggccccggatccaaaatcT | ggccccggatccaaaatcG | acggagcaaagtggtcttgt |
| BS00098207_51 | Q5A | T/C | ccaagcttcccgtgacaT | ccaagcttcccgtgacaC | gccatgatgttTCtgaacttctcT |
| Kukri_c7786_81 | Qd1-5D | T/C | catcaccttgtatccttcctcA | catcaccttgtatccttcctcG | ggttctttgttttgagaagagagG |
| wsnp_JD_c6050_7214383 | Q7A | A/G | GtCgttccacctaacagtacT | GtCgttccacctaacagtacC | gCtgaatcagaagaatgcacagA |
新窗口打开|下载CSV
图4
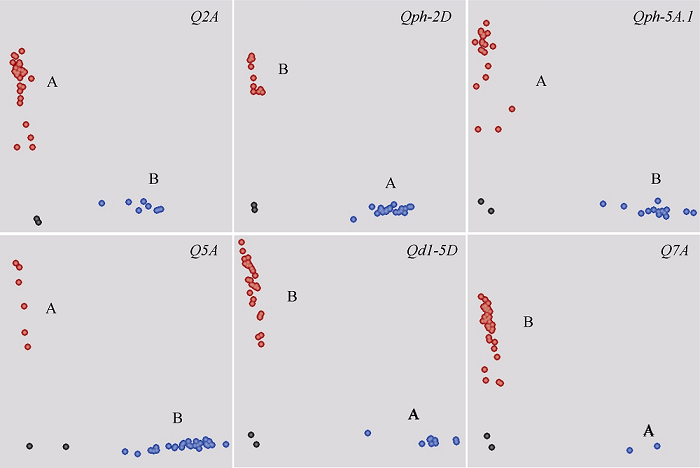 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4株高及其构成因素的KASP标记开发
A表示宁麦9号等位变异类型, B表示扬麦158等位变异类型。
Fig. 4Development of KASP markers for plant height and its components
A indicates the allele of Ningmai 9 and B indicates the allele of Yangmai 158.
2.4 KASP标记的验证
为检测已开发KASP标记的功效, 利用101份2018国家及省区域试验材料进行验证。结果显示, 除Q5A与Qd1-5D外, 其他4个标记的加性效应与定位结果一致(表6)。对于Q5A与Q7A位点, 仅有不到10%的检测材料中含有宁麦9号等位变异, 这可能影响统计检测结果的准确性; 而Qd1-5D主要调控D1长度, 对最终株高影响不显著。在其他3个位点中, Qph-2D对株高有显著影响, 可产生近2 cm的株高差异, Q2A、Qph-5A.1分别产生0.59 cm、1.32 cm的差异, 但未达到显著水平。Table 6
表6
表6区域试验材料中不同等位变异的t-检测
Table 6
| Q2A | Qph-2D | Qph-5A.1 | Q5A | Qd1-5D | Q7A | |
|---|---|---|---|---|---|---|
| 宁麦9号等位变异Ningmai 9 allele (cm) | 87.82 (79) | 87.08 (53) | 88.53 (57) | 86.44 (9) | 87.75 (40) | 86.50 (2) |
| 扬麦158等位变异Yangmai 158 allele (cm) | 88.41 (22) | 88.92 (48) | 87.20 (44) | 88.10 (92) | 88.08 (61) | 87.98 (99) |
| 差值 Difference (cm) | 0.59 | 1.84 | 1.32 | 1.65 | 0.33 | 1.48 |
| t值 t-value | -0.54 | 2.08 | 1.47 | -1.05 | 0.36 | 0.46 |
| P值 P-value | 0.59 | 0.04* | 0.15 | 0.30 | 0.72 | 0.65 |
新窗口打开|下载CSV
按照差异大小逐步分析几个位点的聚合效应发现, 当聚合Qph-2D与Qph-5A.1位点时, 株高可相差4 cm, 进一步聚合Q2A后, 差异超过10 cm, 但材料数量急剧减少, 从降低株高的角度出发, 结合Qph-2D与Qph-5A.1较单一分子标记选择更加有效, 当聚合更多标记后, 株高降低不显著, 而入选材料有所减少, 选择效率降低。
Table 7
表7
表7QTL聚合的效应分析
Table 7
| Qph-2D+Qph-5A.1 | Qph-2D+Qph-5A.1+Q2A | |
|---|---|---|
| 株高+ Plant height+ (cm) | 89.64 (22) | 95.50 (2) |
| 株高? Plant height? (cm) | 85.61 (18) | 84.85 (14) |
| 差值 Difference (cm) | 4.03 | 10.65 |
| t值 t-value | 2.45 | 2.21 |
| P值 P-value | 0.02* | 0.04* |
新窗口打开|下载CSV
3 讨论
宁麦9号与扬麦158分别于1997年和1993年通过品种审定, 育成后均在生产上大面积种植, 为长江中下游麦区乃至全国具有重大影响力的品种。扬麦158适应性广, 且具有良好的农艺性状和较好的赤霉病抗性[27], 宁麦9号具有突出的弱筋小麦品质, 且多花多粒、结实性高、丰产性好, 还是我国小麦栽培品种赤霉病抗性QTL-Fhb1的主要供体[28]。这2个品种已经育成超过20年, 在生产中已被后代品种替代, 但它们目前仍被作为杂交亲本应用于小麦育种。当前小麦生产中大面积推广的宁麦13、扬辐麦4号、扬麦20等均为宁麦9号或扬麦158的衍生后代。据统计, 近3年江苏省淮南麦区与国家长江中下游麦区通过审定的小麦品种中, 超过80%是宁麦9号或扬麦158的衍生后代(宁麦9号与扬麦158株高差异较大。本研究显示扬麦158各节间长度与穗长均大于宁麦9号, 同时遗传因素在株高及其构成因素中占主导作用, 与前人研究结果一致[30,31]。我们通过分子标记检测确定宁麦9号与扬麦158均为Rht-B1b/Rht-D1a变异类型, 均不含Rht8位点。对遗传定位结果比对分析, 最终将影响株高的QTL聚焦到6个染色体区段。通过物理位置比对, Qph-2D位于2D染色体短臂中部, 与位于2D染色体短臂末端的Rht8[32]相距较远, 而与Wang等[33]利用条件与非条件QTL作图检测到的QPh.cgb-2D.1接近。梁子英等[34]在5A染色体barc165-barc180区段检测到株高、D4与D5的控制位点, 其位置与Qph-5A.1基本重合, 而Qph-5A.1在2019环境中与D4、D3也表现相关。Q5A位于5A染色体长臂, 与Rht9的关联标记barc151位置接近, 并且Rht9来源于日本小麦品种, 而Q5A亦来源于含有日本西风小麦血统的宁麦9号, 推测可能为同一位点[32]。Rht23位于5D染色体, 其连锁标记Xgdm63、Xbarc110均与Qd1-5D相近, 但由于5D染色体标记较少, 覆盖度不足, 是否为同一位点有待进一步验证[32]。前人在2A染色体上鉴定到多个株高控制位点[10,33-34], 但与Q2A均相距较远。Peng等[35]在7A染色体短臂末端Xgwm471-Xgwm350区间定位到Rht22, 而Q7A位于7A染色体短臂中部, 可能为不同的位点。
在小麦育种中, 一般将株高较低的基因型作为优势等位变异。Q5A与Q7A在101份区域试验材料中呈现较强的选择效应, 多数材料在Q5A位点处呈现来源于扬麦158的优势等位变异, Q5A与所有节间长度都有关, 可能为一个非常重要的选择位点, 并且很可能经历过早代选择, 使其在高代材料中分布频率较低; 针对此位点, 今后可在低代材料中进行验证, 并同时构建次级分离群体, 为进一步精细定位奠定基础。通过比较基因组学分析发现, Q7A附近区域为许多小麦重要农艺、抗性、品质性状控制位点的热点聚集区, 包括开花期、叶枯病、黄色素含量等[36,37,38,39], 对其中某些性状的定向选择促使某种基因型被大量保留, 最终导致Q7A呈现大量的非优势等位变异。
各节间长度与不同的农艺性状具有一定的相关性[40,41], 特别是植株抗倒性, 研究表明矮秆、D5较短、D1较长的植株有利于协调高产与抗倒性[42]。本研究显示Q2A同时调控D1与株高, Q5A同时调控D5、D1与株高, 且其对应各性状的加性效应来源一致, 无法通过这2个位点实现D1与D5、株高的差异化选择。前述Q5A在多数区域试验材料中呈现来源于扬麦158的降低株高、缩短各节间长度的等位变异, Q2A也是多数保留了来源于宁麦9号的降低株高的等位变异, 表明育种家在株高选择中仍以整株株高为主要选择目标, 未考虑各节间的差异性选择, 在选择方向上对这2个一因多效位点的选择以降低株高的等位变异为主。Qd1-5D仅控制D1长度, 且对整体株高影响不大(表4和表6), 可作为D1的选择标记。
4 结论
共定位到6个株高控制区段, 并成功转化为适用于大规模筛选的KASP标记, 经过初步验证, 聚合Qph-2D、Qph-5A.1标记对整体株高具有较高的选择效率; 继续聚合Q2A后, 中选材料显著减少, 可能降低选择效率, 而对Q5A需要在早代育种材料进一步验证, 并且对Q2A与Q5A一因多效位点的选择建议以降低株高的等位变异为主; Qd1-5D可作为D1的选择标记对株高进行优化选择。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
. Accessed 17 Feb
URL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
DOI:10.1007/s00122-002-0994-1URL [本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 2]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
