 , 陈建荣
, 陈建荣 , 昌洪涛
, 昌洪涛* 通讯作者(Corresponding author): 郭清泉, E-mail:qingqg@yahoo.com 第一作者联系方式: E-mail:462994081@qq.com **同等贡献(Contributed equally to this work)
收稿日期:2014-12-26 接受日期:2015-05-04网络出版日期:2015-06-03基金:
摘要
关键词:苎麻; 肉桂酰辅酶A还原酶基因; 木质素; 组织表达
cDNA Cloning and Analysis of Cinnamoyl-CoA Reductase Gene from Boehmeria nivea
TANG Ying-Hong
 , CHEN Jian-Rong
, CHEN Jian-Rong , CHANG Hong-Tao
, CHANG Hong-TaoAbstract
Keyword: Boehmeria nivea; Cinnamoyl-CoA reductase gene; Lignin; Tissue expression
Show Figures
Show Figures



















近年来, 肉桂酰辅酶A还原酶(cinnamoyl-CoA reductase, CCR)受到了许多研究者的关注, 早在1972年就有****发现其与木质素生物合成相关, 随即CCR基因在拟南芥、象草、杉木、白桦、丹参、桉树等[1, 2, 3, 4, 5, 6]植物中被成功分离纯化, 并利用分子生物学手段对CCR基因进行研究, 证实CCR基因是木质素合成途径上的关键酶基因, 对木质素代谢途径乃至次生代谢均有重要的调节作用。目前CCR基因的研究主要集中在细胞壁的生成、抗菌和抗逆等方面。CCR作为催化木质素特异合成途径的第一步反应酶, 被认为是调节碳素流向木质素潜在的控制点, 可能是木质素合成的限速反应, 控制着碳素进入木质素合成途径[7]。研究苎麻CCR基因对苎麻木质素代谢途径及代谢调控具有重要意义。
苎麻[Boehmeria nivea (L.) Gaud.]是中国传统的优质纤维植物, 其纤维与木质素、果胶、半纤维素等伴生, 降低伴生物的含量有利于纤维的多用途利用。植物木质纤维的生物合成已成为研究热点, 对其进行基因调控已有成功案例[8, 9]。研究苎麻木质纤维生物合成机制有利于改良其品质。陈建荣等[10]研究了苎麻木质素生物合成关键酶CCoAOMT基因, 田志坚等[11]研究了苎麻纤维素合成酶, 近年来人们陆续研究了苎麻的CesA[12, 13, 14]、FB27[15]、COMT[16]、CAD[17]等基因, 而有关苎麻CCR基因的研究报道几乎没有。
本试验通过克隆苎麻木质素生物代谢途径中的CCR基因, 分析该基因的相关生物信息学信息、预测其部分蛋白功能、基因表达特异性等。为通过干扰CCR基因的表达来减少苎麻木质素的合成, 以增加纤维素含量, 提高苎麻纤维品质。
1 材料与方法1.1 试验材料苎麻品种湘苎3号(湘品审第67号)由湖南农业大学郭清泉等选育, 取成熟期的苎麻茎段提取总RNA。SV Total RNA Isolation Kit购自Promega; M-MLV First Strand Kit购自Invitrogen; 3° 和5° -Full RACE Kit、Ex Taq DNA聚合酶、pMD18-T Vector、2× SYBR Premix Ex Taq购自TaKaRa; DNA Marker购自Fermentas; Gel Extraction Kit购自OMEGA; Stratagene Mx3005 System购自Agilent。
1.2 苎麻总RNA的提取按SV Total RNA Isolation试剂盒说明书, 提取苎麻茎段的混合总RNA, 并用1%琼脂糖凝胶电泳检测总RNA的完整性, 紫外分光光度计检测总RNA的浓度和质量。
1.3 PCR产物纯化、连接、转化及测序方法按Gel Extraction Kit说明书回收纯化PCR产物; 将PCR产物与pMD18-T载体连接, 转化大肠杆菌DH5α , 以目标引物验证阳性克隆, 送铂尚生物技术有限公司测序, 并比对分析测序结果, 确认含目的基因cDNA序列片段的克隆。
1.4 CCR基因核心cDNA序列克隆根据湘苎3号转录组测序数据[18]中2个CCR基因的序列信息, 分别记为CCR1、CCR2, 采用Primer Premier 5.0软件分别设计1对特异引物, CCR1上游引物为5′ -CTGCTGGAGCGATGAATCACTATGC-3′ , CCR1下游引物为5′ -CCAATGAACCCGTGCCACA AAT-3′ , CCR2上游引物为5′ -CCACTGGAGTTGACC GACA-3′ , CCR2下游引物为5′ -CCCTTGAGCGACA GGTTTAT-3′ 。按照反转录试剂盒M-MLV First Strand Kit说明书合成cDNA第1链, 以cDNA为模板进行PCR, 经多因素试验将反应体系和反应程序优化, 反应体系为20 μ L, 包含cDNA 1 μ L、上下游引物(10 μ mol L-1)各1.04 μ L、10× Ex Taq缓冲液2.0 μ L、dNTP 2.0 μ L、Ex Taq酶0.16 μ L、ddH2O 12.76 μ L。反应程序为94℃ 5 min, 94℃ 30 s、57℃ 30 s (CCR1 57℃、CCR2 52℃)、72℃ 1 min、30个循环, 72℃ 10 min, 反应终止于4℃。PCR产物经纯化、连接、转化和测序获得目的片段。
1.5 CCR基因全长cDNA序列的克隆在CCR基因核心序列靠近3° 末端和5° 末端位置分别设计3° 和5° -RACE巢式PCR引物, CCR1 3° RACE IN Primer为5° -CTAAGATTTGTGGCACGGGTTCAT TGG-3′ , CCR1 3° RACE OUT Primer为5′ -ATAGAGCA GAGAAGGCAGCGT-3′ , CCR1 5° RACE IN Primer为5′ -CCTGCTCATTGCCTCAAACAC-3′ , CCR1 5° RACE OUT Primer为5′ -TGTGTCGCCCAATGAACCCGTG- 3′ , CCR2 3° RACE IN Primer为5′ -CCGAGGCTTTGA ACATTTTGCGACAG-3′ , CCR2 3° RACE OUT Primer为5′ -GCAACCAACCGTTAATCTCACT-3′ , CCR2 5° RA CE IN Primer为5′ -GCTGTCGCAAAATGTTCAAAG CCTCG-3′ , CCR2 5° RACE OUT Primer为5′ -CCCTTG AGCGACAGGTTTAT-3′ 。然后按照3° 和5° -Full RACE Kit说明书, 将提取的总RNA反转录合成3° 和5° RACE cDNA第1链, 并以这2种cDNA为模板, 分别完成3° 端和5° 端序列的RACE扩增。PCR产物经纯化、连接、转化后测序比对分析获得目的片段。
1.6 BnCCR基因的生物信息学分析将RACE测序结果和核心序列用DNAStar软件拼接得到BnCCR基因全长cDNA序列; 然后利用在线分析网站(http://www.ncbi.nlm.nih.gov/gorf/gorf. html)预测BnCCR基因的开放读码框, 并通过NCBI网站对BnCCR基因核苷酸和氨基酸序列进行Blast比对分析; 最后利用ExPASy Protparam预测与分析BnCCR基因编码蛋白的理化性质、ExPASy SMART预测与分析蛋白功能结构域、Swiss-Model和PyMOL预测与分析蛋白三维结构。
1.7 BnCCR基因的表达分析分别取湘苎3号快速生长期、成熟期和成熟后期茎秆木质部与韧皮部的材料, 参照1.2方法提取总RNA, 以苎麻肌动蛋白(Actin 1)基因为内参, 利用Stratagene Mx3005 System进行实时荧光定量PCR (表1), 研究不同时期、不同部位BnCCR基因的表达情况。按照2× SYBR Premix Ex Taq说明书, 反应体系为12.5 μ L, 包含2× SYBR Premix Ex Taq 6.25 μ L、cDNA 1 μ L、上下游引物(10 μ mol L-1)各0.5 μ L, ddH2O 4.25 μ L; 反应程序为95℃ 30 s, 95℃ 5 s、55℃ 30 s (CCR1 55℃、CCR2 55.9℃)、40个循环, 每个样品设4个重复, 利用2-Δ Δ CT法计算目的基因的表达量。
表1
Table 1
表1(Table 1)
| 表1 实时荧光定量PCR检测BnCCR基因表达所用引物 Table 1 List of primers used in BnCCR expression with qPCR |
2 结果与分析2.1 苎麻茎段总RNA的提取从电泳图谱上看到, RNA条带清晰, 可见28S、18S两条带, 且28S条带的亮度是18S的2倍(图1), 表明RNA基本无降解且没有蛋白质的污染。经紫外分光光度计检测表明, RNA浓度为2084 μ g mL-1, OD260/280=1.95, 可见RNA提取质量满足分子克隆实验需求。
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 苎麻茎段总RNA电泳图Fig. 1 Electrophoresis analysis of total RNA of ramie’ s stem |
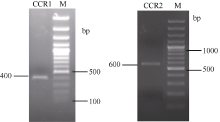
2.2CCR基因全长cDNA序列的克隆 以苎麻cDNA为模板, 经PCR扩增、电泳检测 (图2), 获得CCR1基因核心序列约400 bp, 测序后实际长度为404 bp; CCR2基因核心序列约600 bp, 测序后实际长度为593 bp。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 苎麻CCR1基因和CCR2基因核心序列电泳图Fig. 2 Electrophoresis profile of core sequence of CCR1gene and CCR2gene in ramie |
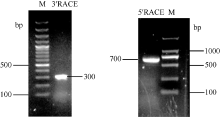
CCR1基因3° RACE和5° RACE PCR产物经凝胶电泳检测(图3)长度大约分别为300 bp和700 bp, 测序得到实际片段长度分别为280 bp和687 bp。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 苎麻CCR1基因3° RACE和5° RACE电泳图Fig. 3 Electrophoresis profile of 3° RACE and 5° RACE of CCR1 gene in ramie |
CCR2基因3° RACE和5° RACE PCR产物经凝胶电泳检测(图4)长度大约分别为450 bp和1000 bp, 测序得到实际片段长度分别为402 bp和914 bp。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 苎麻CCR2基因3° RACE和5° RACE电泳图Fig. 4 Electrophoresis profile of 3° RACE and 5° RACE of CCR2 gene in ramie |
2.3 CCR基因序列分析通过DNAStar软件拼接CCR1基因序列, 得到全长为1057 bp的核苷酸序列, 其中开放阅读框长831 bp, 5° 非翻译区78 bp, 3° 非翻译区144 bp, 其编码的蛋白含277个氨基酸(图5), 命名为BnCCR1 (GenBank登录号为KP001344。将BnCCR1基因序列进行BlastN比对分析, 发现其核苷酸序列与白桦(Betula platyphyllaJF732912)、蓖麻(Ricinus communis XM_002524605)CCR基因核苷酸序列相似性均为70%。将BnCCR1基因编码氨基酸序列进行BlastP比对分析, 发现其氨基酸序列与蓖麻(Ricinus communisXP_002524651)、蒺藜状苜蓿(Medicago truncatula KEH24813)、白桦(Betula platyphyllaAEO45117) CCR基因氨基酸序列相似性分别为77%、75%、74%。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 BnCCR1基因cDNA序列及推测的氨基酸序列Fig. 5 cDNA sequence and its putative amino acid sequences of BnCCR1 gene |
通过DNAStar软件拼接CCR2基因序列, 得到全长为1291 bp的核苷酸序列, 其中开放阅读框长747 bp, 5° 非翻译区341 bp, 3° 非翻译区203 bp, 其编码的蛋白含248个氨基酸(图6), 命名为BnCCR2 (GenBank登录号为KP001342。BnCCR2基因核苷酸序列的BlastN比对表明, 其与毛果杨(Populus trichocarpaXM002314017)、蓖麻(Ricinus communisXM_002524605) CCR基因核苷酸序列相似性分别为74%、72%。BnCCR2基因编码氨基酸序列的BlastP 比对表明, 其与蓖麻(Ricinus communisXP_002517 775.1)、毛果杨(Populus trichocarpaXP_002314052.1)氨基酸序列相似性均为81%。
图6
Fig. 6
| Figure OptionViewDownloadNew Window | |
 | 图6 BnCCR2基因cDNA序列及推测的氨基酸序列Fig. 6 cDNA sequence and its putative amino acid sequences of BnCCR2 gene |
2.4 BnCCR基因编码蛋白理化性质和结构的预测与分析2.4.1 BnCCR1基因编码蛋白理化性质和结构的预测与分析 理化性质分析结果表明, BnCCR1基因所编码蛋白中含量较高的氨基酸有Ala (9.4%)、Val (9.0%)、Glu (9.0%)、Thr (8.3%)、Ser (7.9%)、Arg (6.5%)、Leu (6.5%)、Gly (6.5%); 其中Asp和Glu为碱性氨基酸, Arg和Lys为酸性氨基酸; 该蛋白分子量为30.6667 kD、理论等电点为5.84; 总平均疏水指数为-0.299, 脂肪系数为69.35; 蛋白质不稳定指数为35.30, 这一分类的蛋白稳定。
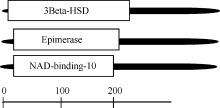
NCBI中Conserved Domains数据库中分析显示, BnCCR1基因编码蛋白具有其他植物肉桂酰辅酶A还原酶共有的FR-SDR-e [flavonoid reductase (FR), Short-chain dehydrogenases/reductases (SDR), extended (e), 类黄酮还原酶-短链脱氢酶/还原酶]保守结构域模型, 在相同的蛋白质序列中生成的重叠为SDR superfamily。蛋白功能结构域分析结果表明, 在BnCCR1基因编码蛋白的N端存在一个脱氢酶/差向异构酶/NAD结合蛋白的结构域, 即与3 Beta-HSD/ Epimerase/NAD-binding-10等保守域具有很高的同源性(图7)。
图7
Fig. 7
| Figure OptionViewDownloadNew Window | |
 | 图7 BnCCR1蛋白结构域的预测Fig. 7 Predicted domain sites of BnCCR1 |
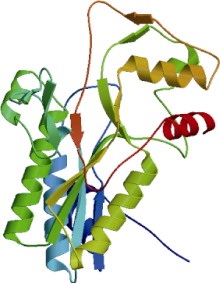
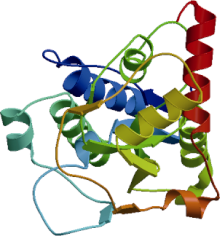
采用Swiss-Model在线软件对BnCCR1基因编码蛋白质的三维结构进行同源建模, 选择矮牵牛[19]CCR蛋白为建模模板, 并用PyMOL对建模结果进行处理。结果表明, BnCCR1蛋白的三维结构与矮牵牛CCR蛋白相似性约为30.66% (图8)。
图8
Fig. 8
| Figure OptionViewDownloadNew Window | |
 | 图8 BnCCR1蛋白三维结构模型的预测Fig. 8 Three-dimensional structure prediction of BnCCR1 protein |
2.4.2 BnCCR2基因编码蛋白理化性质和结构的预测与分析 理化性质分析结果显示, BnCCR2基因所编码蛋白中含量较高的氨基酸有Leu (11.3%)、Val (9.7%)、Ser (8.1%)、Glu (6.9%)、Ile (6.9%)、Gly (6.0%)和Lys (6.0%); 其中Asp和Glu为碱性氨基酸, Arg和Lys为酸性氨基酸; 该蛋白分子量为27.3345 kD、理论等电点为5.64; 总平均疏水指数为0.045, 脂肪系数为104.88; 蛋白质不稳定指数为31.70, 这一分类的蛋白稳定。
NCBI中Conserved Domains数据库中分析显示, 其具有FR-SDR-e的保守结构域模型, 在相同的蛋白质序列中生成的重叠为SDR superfamily。功能结构域的分析结果表明, 在BnCCR2基因编码蛋白的N端存在一个脱氢酶/差向异构酶/NAD结合蛋白的结构域, 即3 Beta-HSD/Epimerase/NAD-binding-4等保守域具有很高的同源性(图9)。
图9
Fig. 9
| Figure OptionViewDownloadNew Window | |
 | 图9 BnCCR2蛋白结构域的预测Fig. 9 Predicted domain sites ofBnCCR2 |
BnCCR2基因编码蛋白质三维结构的同源建模模板选择矮牵牛[19]CCR蛋白, 结果表明, BnCCR2蛋白的三维结构与矮牵牛CCR蛋白相似性约为44.77% (图10)。
图10
Fig. 10
| Figure OptionViewDownloadNew Window | |
 | 图10 BnCCR2蛋白三维结构模型的预测Fig. 10 Three-dimensional structure prediction of BnCCR2 protein |
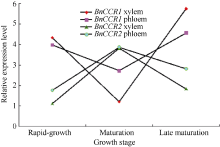
2.5 BnCCR基因的表达特征分析BnCCR1基因的扩增曲线趋势正常、基线平整、熔解曲线峰值单一(图11-A); 从不同时期来看, BnCCR1基因在木质部和韧皮部的相对表达量成熟后期较高、快速生长期居中、成熟期较低; 从不同部位来看, BnCCR1基因于快速生长期和成熟后期时在木质部的相对表达量均高于韧皮部, 于成熟期时在韧皮部的相对表达量高于木质部(图12)。
图11
Fig. 11
| Figure OptionViewDownloadNew Window | |
 | 图11 BnCCR1基因和BnCCR2基因表达定量RT-PCR溶解曲线图(Actin1为内标分子)Fig. 11 Melting curve in quantitative RT-PCR of BnCCR1 and BnCCR2(Actin1 is internal reference) |
图12
Fig. 12
| Figure OptionViewDownloadNew Window | |
 | 图12 BnCCR基因在苎麻木质部和韧皮部的表达分析Fig. 12 Differential expression of BnCCR1 and BnCCR2 in xylem and phloem of the ramie |
BnCCR2基因的扩增曲线趋势正常、基线平整、熔解曲线峰值单一(图11-B); 从不同时期来看, BnCCR2基因在木质部和韧皮部的相对表达量成熟期较高、成熟后期居中、快速生长期较低; 从不同部位来看, 3个时期BnCCR2基因在韧皮部的相对表达量均高于木质部(图12)。
因此可知, BnCCR1基因和BnCCR2基因在木质部与韧皮部中均有表达, 说明两基因均不具有组织表达特异性, 但不同组织中表达量具有差异; BnCCR1和BnCCR2基因在不同时期表达规律不一致, 说明两基因在不同时期具有表达差异, 可能在不同部位的表达时间不同, 其相互关系尚不明确, 正在进一步研究中。
3 讨论根据相关报道, CCR基因存在多基因家族现象, 如小麦的基因组中至少存在两类CCR基因[20]。基于此, CCR基因在自然界中具有多样性的序列, 比如在蓝桉中就发现了9种在保守区中发生变异的CCR基因序列[21]。在苎麻转录组测序数据信息中也发现了多种核心序列不同的CCR基因序列信息, 本试验克隆出其中2个CCR基因序列, 其他序列已取得阶段性研究进展, 将稍后发布。通过Blast比对, 发现2个BnCCR基因与其他物种的CCR基因核苷酸相似性和氨基酸序列相似性均在70%~81%左右, 2个BnCCR基因的保守序列均存在于编码区域中, 这与马尾松中CCR基因相似[22], 且2个BnCCR基因编码蛋白与NCBI数据库中其他物种CCR基因均具有相同的FR-SDR-e保守结构域模型, 在相同的蛋白质序列中生成的重叠均为SDR superfamily。另外, BnCCR蛋白结构和功能的预测结果显示BnCCR1和BnCCR2蛋白的N端存在一个脱氢酶/差向异构酶/NAD结合蛋白结构域, 这与象草、拟南芥、亚麻和苜蓿等[2, 23]植物中CCR蛋白结构一致, 推测可能是其催化还原反应的主要部位; 苎麻BnCCR1和BnCCR2基因与水稻、拟南芥等其他双子叶植物CCR基因[7]中含量较高的氨基酸都是Val、Glu和Leu; BnCCR1和BnCCR2基因编码蛋白的三维结构均与矮牵牛CCR蛋白结构相似性在30%~50%之间, 说明建模结果可靠, 进一步证明克隆的两个CCR基因是苎麻肉桂酰辅酶A还原酶。从表达分析结果看, BnCCR1基因在木质部和韧皮部的表达规律是先降后升, BnCCR2则是先升后降, 这可能是因为BnCCR1和BnCCR2基因对苎麻木质素生物合成的作用保持一种平衡的状态, 快速生长期时BnCCR1起主要作用, 成熟期时2个基因共同作用, 成熟后期时BnCCR2基因发挥主要作用, 2个基因的具体作用还有待进一步研究; 2个基因均表现出在韧皮部中相对表达量的变化幅度小于木质部, 这可能是因为CCR基因是木质素生物合成途径中的关键酶基因, 其对韧皮部木质化的作用低于对木质部木质化的作用。综上所述可预测, 苎麻中至少存在2个CCR基因, 这与小麦中至少存在2种CCR基因分别在不同组织中表达[20]一致, 且这2个BnCCR基因蛋白具有催化活性, 对进一步研究蛋白质结构和功能之间的关系具有至关重要的作用, 本试验结果为阐明苎麻木质素生物合成途径机理奠定了基础。
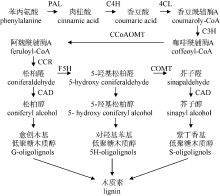
木质素(lignin)是一种具芳香族特性的三维高分子化合物, 是地球上含量仅次于纤维素的天然有机物。木质素的生物合成是十分复杂的, 目前, 研究最多的是苯丙烷途径和木质素合成特异途径。以拟南芥木质素代谢为例[1], 由苯丙氨酸到形成单体木质醇, 由十余种酶来催化, 依次为苯丙氨酸裂解酶(PAL)、肉桂酸-4-羟基化酶(C4H)、4-香豆酸辅酶A连接酶(4CL)、香豆酸-3-羟化酶(C3H)、羟基肉桂酰辅酶A: 奎尼酸羟基肉桂酰转移酶 (CQT)、咖啡酰辅酶-A-O-甲基转移酶(CCoAOMT)、肉桂酰CoA还原酶(CCR)、阿魏酸-5-羟基化酶(F5H)、咖啡酸/5-羟基阿魏酸-O-甲基转移酶(COMT)、肉桂醇脱氢酶(CAD)等。从木质素生物合成简图(图13)可知, 阿魏酰辅酶A由CCR催化生成松柏醛后; 松柏醛经两条路径(CAD和F5H分别催化)分别生成3种木质素单体, 因此CCR是木质素形成的重要关键点。黑麦草[8]中CCR干扰表达遗传转化证实可显著降低木质素含量, 平均降低了34.67%; 宋恩慧等[9]对南林95杨CCR基因进行干扰表达发现其木质素含量与对照相比平均降低了9.86%, 纤维素含量与对照相比平均增加了3.17%, 纤维长宽比明显增加, 均表明其转基因植株更有利于造纸; Dauwe等[24]抑制烟草CAD基因表达发现苯丙烷合成降低、细胞壁相关基因有较高表达。目前, 苎麻木质素代谢途径中CAD、COMT、CCoAOMT等基因都已被分离验证, 因此, 苎麻CCR基因的克隆与分析, 为进一步研究木质素生物合成机制提供了研究数据, 为通过基因工程手段调控木质素代谢途径创造环保型苎麻品种奠定了基础。
图13
Fig. 13
| Figure OptionViewDownloadNew Window | |
 | 图13 木质素生物合成简图Fig. 13 Diagram of lignin biosynthesis |
4 结论基于苎麻转录组数据库克隆的2个CCR基因属于苎麻肉桂酰CoA还原酶家族基因。BnCCR1基因在木质部和韧皮部的表达规律是先降后升, BnCCR2则是先升后降, 2个基因均表现出在韧皮部中相对表达量的变化幅度小于木质部的趋势, 推测这2个基因对苎麻木质素生物合成的作用保持一种平衡状态, 对韧皮部木质化的作用低于对木质部木质化的作用。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] |
