 , 孙杰
, 孙杰*通讯作者(Corresponding author): 刘永昌, E-mail: liuyongchang2003@126.com, Tel: 18799292449 收稿日期:2015-02-06 基金:
摘要
关键词:棉花; 纤维; 驱动蛋白; 表达分析
Cloning and Expression Analysis of Ghkinesin13 Subfamily Genes in Gossypium hirsutum
ZHAO Lan-Jie
 , SUN Jie
, SUN JieAbstract
Keyword:Cotton; Fiber; Kinesin; Expression analysis
Show Figures
Show Figures








我国是世界上最大的棉花生产和消费国, 棉纤维是纺织工业的主要原料, 其品质优劣直接决定纺织品的质量和效益。因此, 提高棉纤维的品质一直是棉花育种的主要目标。棉纤维是由胚珠表皮细胞分化、发育而形成的单细胞, 其形成和发育过程可分为纤维原始细胞分化和突起、纤维伸长、次生壁增厚和脱水成熟4个时期[1]。在纤维发育的各个时期都有大量基因对其发育起调控作用, 了解棉纤维发育的调控机制对提高棉纤维品质具有十分重要的理论意义。到目前为止, 已从棉纤维中筛选并克隆了多个棉纤维发育相关基因, 这些基因在棉纤维发育过程中特异或优势表达, 其中少数基因已被证实对纤维发育起一定的调控作用, 如GhRGP1、GhBIN2、E6、KATANIN、WRINKLED1[2, 3, 4, 5]等。但是, 更多的调控纤维发育的关键基因有待分离鉴定。驱动蛋白(Kinesin)是一类蛋白质超级家族, 属于分子马达的一种。驱动蛋白能够利用ATP水解所释放的能量驱动自身所携带的分子, 将微管作为运动轨道定向移动, 并能对微管的动态变化产生影响[6]。它们在胞内运输、有丝分裂、信号转导、细胞形成等方面起着至关重要的作用[7, 8, 9, 10]。驱动蛋白家族包含14个亚家族, 不同种类的驱动蛋白对应着不同的工作机制、结构和物质束缚力[11]。对植物Kinesin基因的功能研究大多来自模式植物, 水稻和拟南芥基因组中分别发现41个和61个Kinesin基因[12, 13, 14, 15]。目前, 棉花中仅克隆了4个Kinesin基因。GhKCBP呈串珠状模式点缀于周质微管, 可能在棉纤维发育过程中微管陈列动态重组时发挥作用[16]; GhKCH1含有与微丝结合的结构域, 在体外能与微丝结合, 在棉纤维细胞中表现为与微管共定位, 在某些部位也与微丝共分布, 可能在微丝、微管的动态分布中起重要调节作用[17]; GhKCH2具有与微管和微丝的双重结合能力, 可能作为动态桥梁在2种细胞骨架系统同时参与的细胞活动中发挥重要作用[18]; Ghkinesin-13A定位于高尔基体上[19], 没有进一步的研究报道。近期的功能研究发现Kinesin13亚家族成员在细胞形成过程中发挥着重要作用。邓祝云等[20]通过诱变筛选获得一个籽粒变圆的水稻突变体sar1, 进一步的缺失和互补分析显示Kinesin13亚家族成员SAR1基因的变异是导致sar1表型的直接原因。水稻OsSRS3基因编码一个Kinesin13驱动蛋白, 其突变体srs3籽粒长度变短。电镜扫描发现突变体srs3种子细胞的数目与野生型无明显差异, 但细胞的轴向长度明显短于野生型[21]。拟南芥Atkinesin-13A功能缺失突变导致其叶表皮毛分叉增多, 由原来的三分支变成四分支[19]。拟南芥表皮毛和棉纤维发育机制相似, 因此Kinesin-13A在2种植物中可能具有相同的生物学功能。棉花纤维细胞形状为直线形、无分支, 棉花中的Kinesin13基因在纤维发育中又会有什么样的作用有待研究。目前棉花中仅分离出1个Kinesin13亚家族基因
(Ghkinesin13), 为了获得更多该亚家族基因, 本研究利用拟南芥Atkinesin-13A蛋白为查询序列, 查询雷蒙德氏棉基因组数据库, 找到棉花中的Kinesin13亚家族基因, 并对其序列及在棉纤维中的表达模式进行分析, 为进一步探索Ghkinesin13亚家族基因在棉纤维发育中的作用机制奠定基础。
1 材料与方法1.1 材料及处理陆地棉(Gossypium hirsutumL.)品种新陆早33由石河子大学棉花研究所提供。PCR反应中所用高保真Pfu DNA聚合酶购自天根生化科技(北京)有限公司; pGEM-T Easy Vector试剂盒购自普洛麦格(北京)生物技术有限公司; 各种限制性内切酶、DNase I、Reverse Transcriptase M-MLV (RNase H-)反转录酶等购自宝生物工程(大连)有限公司; SYBR Premix Ex Taq试剂盒购于TaKaRa公司, 其他试剂均为国产分析纯。大肠杆菌(Escherichia coli)菌株TOP10为本实验室保存。
将棉花种植在石河子大学试验站, 在盛花期挂牌标记当日花, 随后摘取发育3、6、9、12、15、18、21、24、27和30 DPA (days post anthesis)棉铃, 室内剥取纤维; 取新陆早33种子, 剥去种皮后用0.1% HgCl2浸泡10 min, 灭菌水冲洗3~4次, 播种于1/2 MS培养基上, 28℃暗培养4 d, 水培2周后取幼苗的根、茎、叶; 从田间植株摘取花。将上述材料液氮速冻后保存于-80℃冰箱, 用于RNA的提取。
1.2 棉花总RNA的提取及cDNA第1链的合成采用CTAB法提取棉花样品总RNA[22], 用DNase I处理后, 利用ND1000紫外分光光度计测定OD260和OD280值, 计算RNA的浓度与纯度。用1.2%琼脂糖凝胶电泳检测RNA的完整性, 保存于-80℃备用。cDNA第1链按照大连宝生物工程有限公司Reverse Transcriptase M-MLV(RNase H-)的说明书合成。
1.3 棉花Ghkinesin13亚家族基因的克隆 利用http://www.phytozome.net/网站, 以拟南芥Atkinesin13A蛋白序列(GenBank登录号为AAL07208)作为探针序列, 与二倍体雷蒙德氏棉的基因组序列进行Blast比对, 获得7个具有较高同源关系的基因(其序列号分别为Gorai.006G076100.1、Gorai.006G262200.1、Gorai.011G190700.1、Gorai. 002G059900.1、Gorai.011G075200.1、Gorai.009G 024600.1、Gorai.004G280000.1)。根据7个基因与
Atkinesin-13A和Atkinesin-13B的同源性高低, 将它们分别命名为GrKIS13A1、GrKIS13A2、GrKIS13A3、GrKIS13B1、GrKIS13B2、GrKIS13B3和GrKIS13B4。根据7个基因的电子序列, 设计扩增开放阅读框的引物(表1), 以陆地棉新陆早33棉纤维cDNA为模板进行PCR扩增, 反应体系含: 10× buffer 2 μ L、2.5 mmol L-1 dNTPs 1.6 μ L、5 U μ L-1 Pfu Taq DNA聚合酶0.2 μ L、10 μ mol L-1上下游引物各1 μ L, 补ddH2O至20 μ L。反应程序为 94℃ 5 min; 94℃ 45 s, 52~56℃ 45 s, 72℃ 2.5 min; 重复30个循环; 72℃延伸10 min。PCR产物经1%琼脂糖电泳检测, 回收目的片段, 连接至pGEM-T Easy载体, 转化大肠杆菌Top10感受态细胞, 挑取单菌落, 提取质粒进行PCR和酶切鉴定, 鉴定正确的质粒送至北京华大基因公司测序。根据测序结果, 将从陆地棉中克隆得到的7个基因与雷蒙德氏棉中的命名相对应, 分别为GhKIS13A1、GhKIS13A2、GhKIS13A3、GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4。将这些基因的序列登录至GenBank, GenBank序列号为KP036626至KP036632。
1.4 棉花Ghkinesin13亚家族基因生物信息学分析 采用SMART (http://smart.embl-heidelberg.de/)和NCBI中BlastP对氨基酸序列进行蛋白质保守区分析; 采用WOLF PSORT (http://psort.hgc.jp/)进行信号肽及亚细胞定位分析; 采用DOG 2.0软件绘制蛋白结构域图; 采用Clustal X和TreeView软件比对序列和构建进化树。
1.5 棉花Ghkinesin13亚家族基因的实时荧光定量PCR分析(quantitative real-time PCR, qPCR) 为了研究棉花Ghkinesin13亚家族基因在棉花不同组织中的表达模式, 根据7个基因的cDNA序列, 分别设计7对特异性实时荧光定量引物(表1), 以棉花根、茎、叶、花和不同发育时期的棉纤维cDNA为模板进行qPCR扩增。在罗氏LightCycler480实时荧光定量PCR仪上进行qPCR, 反应程序为: 94℃ 1 min; 95℃ 15 s, 52~56℃ 20 s, 72℃ 30 s, 45个循环。3次重复, 每次实验设置3个技术重复, 按照2-∆ ∆ Ct法分析结果。
表1
Table 1
表1(Table 1)
| 表1 实验中所用的引物 Table 1 The primers used in the experiment | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
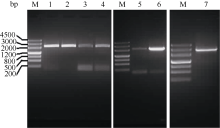
2 结果与分析2.1 Ghkinesin13亚家族基因全长cDNA序列的克隆利用拟南芥Atkinesin-13A蛋白序列作为查询序列, 从雷蒙德氏棉中得到7个Kinesin13亚家族基因。雷蒙德氏棉基因组中7个基因的基因组序列长度为5328~7192 bp, 编码区(coding sequence, CDS)长度为2115~2727 bp, 编码蛋白序列长度为704~ 908氨基酸(表2)。根据7个基因的电子序列, 设计扩增ORF的引物, 以陆地棉纤维cDNA为模板, 分别进行PCR扩增, 克隆获得7个基因的cDNA全长序列(图1)。测序后发现7个基因cDNA序列与雷蒙德氏棉基因组数据库中的序列存在一些碱基差异, 这可能是由于这7个基因是以四倍体陆地棉纤维cDNA为模板扩增的, 表明雷蒙德氏棉和陆地棉2个不同的种间核酸序列存在一些差异。将从陆地棉中克隆得到的7个基因分别命名为GhKIS13A1、GhKIS13A2、GhKIS13A3、GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4。
表2
Table 2
表2(Table 2)
| 表2 Grkinesin13亚家族基因在雷蒙德氏棉基因组数据库中的序列信息 Table 2 Sequence information of Ghkinesin13 subfamily genes in genome database of Gossypium raimondii |
图1
Fig. 1
| Figure OptionViewDownloadNew Window | |
 | 图1 Ghkinesin13亚家族基因PCR扩增电泳图 M: marker III; 1~7: GhKIS13B1、GhKIS13B4、GhKIS13B3、GhKIS13B2、GhKIS13A2、GhKIS13A3和GhKIS13A1的PCR扩增结果。Fig. 1 PCR results of Ghkinesin13 subfamily genesM: marker III; 1-7: PCR results of GhKIS13B1, GhKIS13B4, GhKIS13B3, GhKIS13B2, GhKIS13A2, GhKIS13A3, and GhKIS13A1, respectively. |
2.2 Ghkinesin13亚家族基因编码蛋白的生物信息学分析为了进一步确定7个基因是否属于Kinesin基因家族, 利用SMART在线分析, 结合NCBI中BlastP比对, 对7个Ghkinesin13亚家族基因编码产物的氨基酸序列进行蛋白质保守区分析, 发现7个蛋白均具有Kinesin家族典型的KISC结构域(马达区), 且均位于蛋白的中央部位, 为中央马达蛋白, 属于Kinesin13亚家族成员; 7个蛋白中GhKIS13A1、GhKIS13A2、GhKIS13A3、GhKIS13B1、GhKIS13B2具有coiled coil, 其位置如图2所示。由WOLF PSORT软件在线预测7个Ghkinesin13亚家族蛋白的信号肽及亚细胞定位情况, 表明GhKIS13A2和GhKIS13B1定位在质膜上, GhKIS13A1、GhKIS13A3、GhKIS13B2、GhKIS13B3和GhKIS13B4定位在内质网上。
2.3 Ghkinesin13蛋白多重序列比对和蛋白进化树分析利用http://www.phytozome.net/网站进行Blast比对发现GhKIS13A1、GhKIS13A2和GhKIS13A3与Atkinesin-13A蛋白序列一致性较高, 分别为88%、87.1%、86.9%; GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4与Atkinesin-13B蛋白序列一致性较高, 分别为90.6%、90.4%、86.1%和84.3% (表3)。分析结果表明GhKIS13A1、GhKIS13A2和GhKIS13A3可能属于Kinesin13A亚家族基因, 因此命名中含有“ A” 字母, 而GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4可能属于Kinesin13B亚家族基因, 因此命名中含有“ B” 字母。
利用Clustal X软件将Ghkinesin13亚家族蛋白的KISC结构域与拟南芥Atkinesin-13A/B的KISC结构域进行多重序列比较。如图3所示, 7个Ghkinesin13蛋白的KISC结构域中均具有微管结合位点(三角符号所示)和ATP结合域(星号所示)。
为了明确7个Ghkinesin13蛋白与Kinesin家族其他蛋白的进化关系, 选取发现的部分植物kinesin蛋白, 利用Tree View软件构建系统进化树。分析发现7个蛋白均与已报道的Kinesin13亚家族蛋白聚为一类。Kinesin13亚家族的KISC属于中央马达, 包括Kinesin13A和Kinesin13B两类, GhKIS13A1、GhKIS13A2和GhKIS13A3与Atkinesin-13A蛋白关系较近, 聚为一类, 而GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4与Atkinesin-13B蛋白关系较近, 聚为一类, 进一步明确了7个蛋白在Kinesin家族中的类别。
图2
Fig. 2
| Figure OptionViewDownloadNew Window | |
 | 图2 Ghkinesin13亚家族蛋白的结构 黑色和斜线区域分别表示蛋白的KISC结构域和coiled coil结构。Fig. 2 Structure of Ghkinesin13 subfamily proteins Black areas and that filled with oblique lines respectively represent the KISC and coiled coil domain of protein. |
表3
Table 3
表3(Table 3)
| 表3 Ghkinesin13与拟南芥Atkinesin-13A/B氨基酸序列一致性分析 Table 3 Identity analysis of amino acid sequence of Ghkinesin13 and Atkinesin-13A/B | ||||||||||||||||||||||||||||||||||||||||||||||||||||
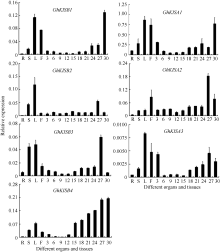
2.4 Ghkinesin13亚家族基因在棉花不同组织中的表达特征分析为研究7个Ghkinesin13亚家族基因在棉花中表达模式, 对棉花根、茎、叶、花以及不同发育时期纤维中的表达模式进行了实时荧光定量PCR分析, 如图5所示, 7个GhKinesin13亚家族基因在根、茎、叶、花和纤维中均有表达, 但表达模式各不相同。GhKIS13A1、GhKIS13A3、GhKIS13B1、GhKIS13B2和GhKIS13B3在叶中优势表达, 其中GhKIS13A1、GhKIS13A3、GhKIS13B1在花中表达量也较高, GhKIS13B3在茎中表达量也较高。GhKIS13A2、GhKIS13A3、GhKIS13B2和GhKIS13B3在纤维中的表达量呈现先升高再降低的趋势, GhKIS13A1、GhKIS13B1和GhKIS13B4在纤维中的表达呈现上升趋势。GhKIS13A2、GhKIS13B3在27 DPA纤维中表达量明显较高, GhKIS13A1和GhKIS13B1在30 DPA纤维中表达量明显较高, 但在其他时期表达量均较低; GhKIS13A3和GhKIS13B2在纤维中的表达量明显低于在其他器官中, 因此GhKIS13A1、GhKIS13A2、GhKIS13A3、GhKIS13B1、GhKIS13B2和 GhKIS13B3在纤维中均没有表达优势。7个基因中仅有GhKIS13B4在纤维中优势表达, 在纤维中的表达量明显高于在其他器官中, 其表达量随着纤维的发育逐渐升高, 暗示其在纤维发育过程中可能发挥着重要作用。
图3
Fig. 3
| Figure OptionViewDownloadNew Window | |
 | 图3 Ghkinesin13与拟南芥Atkinesin-13A/B蛋白的KISC结构域多重序列比对 星号表示ATP结合位点; 三角符号表示微管结合位点, 所用序列包括Atkinesin-13A (拟南芥, AAL07208)和Atkinesin-13B (拟南芥, AAK96543)。Fig. 3 Amino acid sequences alignment of Ghkinesin13 and Atkinesin-13A/B protein The letters marked with asterisks represent the ATP-binding sites; the letters marked with triangle represent the microtubule binding sites. The sequences used are Atkinesin-13A (Arabidopsis thaliana, AAL07208) and Atkinesin-13B (Arabidopsis thaliana, AAK96543). |
3 讨论Kinesin是一类蛋白质超级家族, 是与微管结合而起运输作用的马达蛋白。根据结构特征及氨基酸序列同源性, 将Kinesin分为14大类, 即Kinesin1至Kinesin14亚家族[23, 24]。14个Kinesin亚家族蛋白中, Kinesin1至Kinesin12亚家族的马达区位于肽链的N-末端, 为N-末端马达蛋白; Kinesin14亚家族的马达区位于肽链的C-末端, 为C-末端马达蛋白; Kinesin13亚家族的马达区位于肽链的中央, 为中央马达蛋白[23]。本研究利用拟南芥Atkinesin-13A蛋白序列作为探针序列, 从二倍体雷蒙德氏棉的基因组数据库中发现7个Kinesin13基因, 并从陆地棉中分离了相应的7个基因。序列分析发现7个基因编码的蛋白长度各异, 均具有Kinesin家族典型的KISC结构域(马达区), 且均位于蛋白的中央部位, 本研究使我们掌握了棉花中Kinesin13亚家族基因的存在情况和结构特征。
qPCR分析发现, 7个Kinesin基因在棉花各组织中均有表达, 但表达模式各不相同。7个基因中仅有GhKIS13B4在纤维中优势表达, 可选作目标基因转化棉花改良棉纤维的品质。研究中还发现7个Kinesin13基因中GhKIS13A2、GhKIS13A3、GhKIS13B2和GhKIS13B3在27 DPA纤维中表达量最高, GhKIS13A1、GhKIS13B1和GhKIS13B4在30 DPA纤维中表达量最高。27、30 DPA为纤维发育次生壁合成期, 7个基因均在该时期表达量高, 表明Kinesin13亚家族基因可能在纤维发育的次生壁合成期发挥重要作用。
图4
Fig. 4
| Figure OptionViewDownloadNew Window | |
 | 图4 Ghkinesin13与其他Kinesin蛋白的进化树分析 所用序列包括: AtKCBP/Kinesin-14 (拟南芥, AAC37475), GhKCBP/Ghkinesin-14 (陆地棉, AAP41107), Ghkinesin-13A (陆地棉, AAQ18797), Atkinesin-13A (拟南芥, AAL07208), Atkinesin-13B (拟南芥, AAK96543), Atkinesin-12A (拟南芥, AAF78893), Atkinesin-12B (拟南芥, AEE76799), AtK1/Kinesin-1 (拟南芥, BAA01972), AtNACK1/Kinesin-7 (拟南芥, BAB88748), AtNACK2/Kinesin-7 (拟南芥, BAC03248), OsNACK1/Kinesin-7 (水稻, BAB86283), GhKCH1/Kinesin-14 (陆地棉, AAW03152), GhKCH2/Kinesin-14 (陆地棉, ABO28522), OsSRS3-13A (水稻, EEE62380), Zmkinesin-1 (玉米, DAA35856), OsKCH1/Kinesin-14 (水稻, NP_001066967), AtFRA1/Kinesin-4 (拟南芥, AAN86114)。Fig. 4 Phylogenic relationship of Ghkinesin13 and other Kinesin proteins The sequences used are AtKCBP/Kinesin-14 (Arabidopsis thaliana, AAC37475), GhKCBP/Ghkinesin-14 (Gossypium hirsutum, AAP41107); Ghkinesin-13A (Gossypium hirsutum, AAQ18797), Atkinesin-13A (Arabidopsis thaliana, AAL07208), Atkinesin-13B (Arabidopsis thaliana, AAK96543), Atkinesin-12A (Arabidopsis thaliana, AAF78893), Atkinesin-12B (Arabidopsis thaliana, AEE76799), AtK1/Kinesin-1 (Arabidopsis thaliana, BAA01972), AtNACK1/Kinesin-7 (Arabidopsis thaliana, BAB88748), AtNACK2/Kinesin-7 (Arabidopsis thaliana, BAC03248), OsNACK1/Kinesin-7 (Oryza sativa, BAB86283), GhKCH1/Kinesin-14 (Gossypium hirsutum, AAW03152), GhKCH2/Kinesin-14 (Gossypium hirsutum, ABO28522), OsSRS3-13A (Oryza sativa, EEE62380), Zmkinesin-1 (Zea mays, DAA35856), OsKCH1/Kinesin-14 (Oryza sativa, NP_001066967), AtFRA1/Kinesin-4 (Arabidopsis thaliana, AAN86114). |
棉花中已报道4个Kinesin基因, 其中GhKCBP、GhKCH1和GhKCH2属于Kinesin14亚家族成员, Ghkinesin-13属于Kinesin13亚家族成员。Kinesin14亚家族基因常与微管或微丝共定位, 棉纤维中的Kinesin14蛋白可能通过调控微管和微丝的排向和动态分布, 进而影响纤维的伸长和次生壁的合成, 改变纤维的品质[16, 17, 18]。与Kinesin14的工作机制不同, Kinesin13亚家族蛋白常与高尔基体共定位, 可能参与调控高尔基体的结构、功能和分布。免疫荧光和免疫金标实验发现烟草Ntkinesin-13定位于高尔基体的囊泡膜上[25]。Atkinesin-13A在拟南芥的各细胞中与高尔基体共定位。Atkinesin-13A缺失突变后, 拟南芥根冠分泌活动旺盛的细胞中高尔基体的囊泡变得小而少[26]。Atkinesin-13A突变体表皮毛中高尔基体呈现聚集现象, 而高尔基体的分布对表皮毛的形态发生具有调控作用[19]。因此, Atkinesin-13A可能是一个参与高尔基体结构、分布和功能调控的驱动蛋白。棉花Ghkinesin-13也定位于相应的高尔基体上[19], 它可能影响纤维中高尔基体的结构、功能或分布, 进而调控纤维的发育, 但该基因在纤维发育过程中的功能有待进一步研究。本研究从纤维中分离了7个Ghkinesin13亚家族基因, 为今后进一步研究Ghkinesin13亚家族基因在棉纤维发育过程中的功能奠定了基础。
图5
Fig. 5
| Figure OptionViewDownloadNew Window | |
 | 图5 Ghkinesin13亚家族基因的实时荧光定量PCR分析 R: 根, S: 茎; L: 叶; F: 花; 3~30: 3、6、9、12、15、18、21、24、27、30 DPA纤维细胞。Fig. 5 Relative expression ofGhkinesin13 subfamily genes R: root; S: stem; L: leaf; F: flower; 3-30: fiber cells at 3, 6, 9, 12, 15, 18, 21, 24, 27, and 30 DPA, respectively. |
4 结论从陆地棉中克隆到7个Kinesin13亚家族基因, 分别命名为GhKIS13A1、GhKIS13A2、GhKIS13A3、GhKIS13B1、GhKIS13B2、GhKIS13B3和GhKIS13B4。它们编码的蛋白在704~908氨基酸之间, 均含有典型的KISC中央马达区域、ATP结合位点和微管结合位点, 分为Kinesin13A和Kinesin13B两个亚类。它们均在棉花各组织中表达, 表达模式各不相同, 其中GhKIS13B4在纤维中优势表达, 在纤维发育过程中可能发挥着重要作用。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献View Option
原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | |
| [3] | |
| [4] | |
| [5] | |
| [6] | |
| [7] | |
| [8] | |
| [9] | |
| [10] | |
| [11] | |
| [12] | |
| [13] | |
| [14] | |
| [15] | |
| [16] | |
| [17] | |
| [18] | |
| [19] | |
| [20] | |
| [21] | |
| [22] | |
| [23] | |
| [24] | |
| [25] | |
| [26] |
