彭文仪1,2, 吴芷莹1,2, 罗晓萱3, 陈思杰3, 周诗妍3, 邓素萍1, 罗湘1, 封少龙4,5

 , 柏琴琴1,2
, 柏琴琴1,2

1. 南华大学公共卫生学院, 湖南 衡阳 421001;
2. 衡阳市健康危害因子检验检疫新技术研究重点实验室, 湖南 衡阳 421001;
3. 南华大学衡阳医学院, 湖南 衡阳 421001;
4. 桂林医学院公共卫生学院, 预防医学研究所, 广西 桂林 541004;
5. 中国科学院广州地球化学研究所, 有机地球化学国家重点实验室, 广东省环境资源利用与保护重点实验室, 广东 广州 510640
收稿日期:2021-02-10;修回日期:2021-04-21;网络出版日期:2021-05-10
基金项目:湖南省自然科学基金(2019JJ50494);湖南省教育厅科学研究项目(16C1390);湖南省重点实验室资助项目(2019TP1027);南华大学大学生创新项目(X202010555385)
*通信作者:封少龙, Tel: +86-773-3662175;E-mail: slfeng@glmc.edu.cn;
柏琴琴, Tel: +86-734-8281393;E-mail: baiqinqin1213@126.com.
摘要:[目的] 预测并分析82株全基因组测序的鲍曼不动杆菌前噬菌体的携带情况,鉴定前噬菌体编码的抗生素耐药基因和毒力因子。[方法] 利用PHASTER(Phage Search Tool Enhanced Release)软件预测鲍曼不动杆菌携带的前噬菌体,采用CARD(The Comprehensive Antibiotic Research Database)和VFDB(Virulence Factors Database)在线分析软件预测前噬菌体编码的抗生素耐药基因和毒力因子。[结果] 预测到472条鲍曼不动杆菌前噬菌体,其中完整型前噬菌体201条,疑似型前噬菌体91条,缺陷型前噬菌体180条。平均每株鲍曼不动杆菌基因组中可携带至少2条完整型前噬菌体。每株鲍曼不动杆菌所携带的全部前噬菌体占其基因组比例约为4%-6%。29条前噬菌体携带77个耐药基因,耐药表型共有14种,分别来自15个不同的家族,涵盖6种抗生素耐药的作用机制。132条前噬菌体编码毒力基因,归类为38种毒力基因和34种毒力因子。不同类型的前噬菌体普遍携带1-2种毒力因子,少数前噬菌体携带3种及以上毒力因子。分析毒力因子可能的宿主来源构成比发现,除鲍曼不动杆菌外,脑膜炎奈瑟菌、痢疾志贺氏菌、嗜肺军团菌及其亚种等也有较高的结构比例,是可能的宿主来源。[结论] 鲍曼不动杆菌普遍携带前噬菌体,但前噬菌体基因在鲍曼不动杆菌基因组中所占比例不高。部分前噬菌体携带抗生素耐药基因,以氨基糖苷类、磺胺类及β-内酰胺类耐药为主。约30%的前噬菌体携带毒力基因。前噬菌体可能在鲍曼不动杆菌抗生素耐药性的获得、传播及致病性演变中发挥重要作用。
关键词:鲍曼不动杆菌前噬菌体基因组学耐药基因毒力因子
Bioinformatic analysis of prophages carried by Acinetobacter baumannii
Wenyi Peng1,2, Zhiying Wu1,2, Xiaoxuan Luo3, Sijie Chen3, Shiyan Zhou3, Suping Deng1, Xiang Luo1, Shaolong Feng4,5

 , Qinqin Bai1,2
, Qinqin Bai1,2

1. School of Public Health, University of South China, Hengyang 421001, Hunan Province, China;
2. Key Laboratory of Hengyang for Health Hazard Factors Inspection and Quarantine, Hengyang 421001, Hunan Province, China;
3. Hengyang Medical College, University of South China, Hengyang 421001, Hunan Province, China;
4. The Institute of Preventive Medicine, School of Public Health, Guilin Medical University, Guilin 541004, Guangxi Zhuang Autonomous Region, China;
5. Guangdong Provincial Key Laboratory of Environmental Protection and Resources Utilization, The State Key Laboratory of Organic Geochemistry, Guangzhou Institute of Geochemistry, Chinese Academy of Sciences, Guangzhou 510640, Guangdong Province, China
Received: 10 February 2021; Revised: 21 April 2021; Published online: 10 May 2021
*Corresponding author: Shaolong Feng, Tel: +86-773-3662175;E-mail: slfeng@glmc.edu.cn;
Qinqin Bai, Tel: +86-734-8281393;E-mail: baiqinqin1213@126.com.
Foundation item: Supported by the Natural Science Foundation of Hunan Province (2019JJ50494), by the Scientific Research Fund of Hunan Provincial Education Department (16C1390), by the Fund of Hunan Key Laboratory (2019TP1027) and by the University of South China Student Innovation Project (X202010555385)
Abstract: [Objective] To analyze the prophages carried by 82 strains of Acinetobacter baumannii with completely sequenced genomes, then identify antibiotic resistance genes and virulence factors encoded by prophages. [Methods] Phage Search Tool Enhanced Release (PHASTER) software was used to predict the prophages carried by A. baumannii. The Comprehensive Antibiotic Research Database (CARD) and Virulence Factors Database (VFDB) online analysis software were used to predict the antibiotic resistance genes and virulence factors encoded by the prophages, respectively. [Results] A total 472 prophages were found, of which 201 were "intact", 91 were "questionable", and 180 were "incomplete". On average, at least two intact prophages were carried per A. baumannii genome. The percentage of the prophage genome ranged between 4% and 6% in each A. baumannii genome. Twenty-nine out of the 472 prophages carried 77 resistance genes, which belong to 14 different antimicrobial types, could be grouped into 15 different drug families, and could be classified into six antibiotic resistance mechanisms. One hundred and thirty-two out of the 472 prophages encode putative virulence genes, grouped into 38 virulence gene and 34 virulence factor classes. Most of these prophages encode one or two virulence factors, and a few of them encode three or more virulence factors. Analysis of component ratios of possible virulence factor host sources indicated Neisseria meningitidis, Shigella dysenteriae, and Legionella pneumophila, as well as A. baumannii, as host sources. [Conclusion] A. baumannii generally carries prophages, but the proportion of prophage genes in the genome of A. baumannii is not high. Some prophages carry antibiotic resistance genes, mainly aminoglycosides, sulfonamides, and beta-lactams. Nearly 30% of the prophages carry virulence genes. Prophages may play an important role in the acquisition and spread of antibiotic resistance, and evolution of pathogenicity in A. baumannii.
Keywords: Acinetobacter baumanniiprophagegenomicsantibiotics resistance genesvirulence factor
鲍曼不动杆菌(Acinetobacter baumannii)为不动杆菌属细菌,是一种常见条件致病菌,广泛生存在医院环境和自然界之中,可引起医院获得性肺炎、血流感染、腹腔感染、中枢神经系统感染、泌尿系统感染和皮肤软组织感染等[1]。近年来,由于抗生素的不合理和大量使用,鲍曼不动杆菌耐药性不断增强。全国细菌耐药监测网2014–2019年细菌耐药性监测报告显示鲍曼不动杆菌对常用抗菌药物的耐药率高于50%,即使是被视为最后的选择——多黏菌素,其耐药率也有2.0%–3.8%[2]。多重耐药、广泛耐药甚至全耐药鲍曼不动杆菌在全球范围内流行,使其成为了全球抗感染领域的挑战[1, 3–4]。
前噬菌体(prophage)是整合到细菌DNA上或以质粒的形式存在的噬菌体基因,是细菌基因水平转移的载体,与细菌毒力、耐药性和进化等密切相关[5]。已有大量文献报道前噬菌体可编码毒力因子(virulence factors,VFs)影响细菌的致病性。除志贺毒素和白喉毒素等毒素外,前噬菌体还编码一些VFs参与细菌的粘附、侵袭和胞内存活等[6–7]。近年来,前噬菌体在抗生素耐药性的获得与传播方面的作用也受到了研究人员的关注[8]。文献报道前噬菌体可通过转导[9]、“自转导”[10]等方式介导抗生素耐药基因(antibiotics resistance genes,ARGs)的水平传播。此外,前噬菌体亦能编码ARGs,如Wang等[11]报道132株肠毒素型大肠埃希菌中鉴定出1206个ARGs,其中2.65%(32/1206)由前噬菌体编码。
鲍曼不动杆菌前噬菌体的研究资料较少。Costa等[12]报道鲍曼不动杆菌的109条完整型前噬菌体中编码毒力基因的比例高达78%,但未对缺陷型及疑似型前噬菌体编码毒力基因的情况进行分析。Lopez-Leal等[13]报道鲍曼不动杆菌的前噬菌体普遍编码耐药基因,但未针对ARGs的耐药表型和可能的耐药机制进行分析。本研究利用前噬菌体预测软件PHASTER(Phage Search Tool Enhanced Release)[14]分析了82株全基因组测序的鲍曼不动杆菌携带的前噬菌体基因组的基本特征,对不同类型前噬菌体编码的ARGs和毒力基因进行预测,并对ARGs的耐药表型和可能的耐药机制及毒力基因可能的宿主来源进行了分析。
1 材料和方法 1.1 鲍曼不动杆菌基因组数据 82株鲍曼不动杆菌的全基因组数据,均下载自美国国立生物技术信息中心(NCBI)官网(https://www.ncbi.nlm.nih.gov/genome/browse/),最后访问时间为2020年11月08日。
1.2 菌株的分布概况 将82株鲍曼不动杆菌的所在地区、样本来源、所致疾病、宿主来源等分布信息用Excel整理,采用Originp2016软件作图。
1.3 前噬菌体预测 收集细菌全基因组重叠群信息(即细菌染色体及其质粒GenBank编号),使用PHASTER在线预测工具(http://phaster.ca/)[14]预测82株鲍曼不动杆菌的全基因组中前噬菌体的位点,定位前噬菌体序列并显示基因组特征,将预测结果下载保存以便后续分析。PHASTER根据DNA序列编码CDS的数量以及是否存在噬菌体相关基因的标准,将预测得到的前噬菌体根据得分情况判断其基因组完整程度,并将其分为完整型前噬菌体(intact prophage,score > 90)、疑似型前噬菌体(questionable prophage,score 70-90)和缺陷型前噬菌体(incomplete prophage,score < 70)三大类。将预测得到的前噬菌体基因组数据进行整理,并对前噬菌体基因组大小、GC含量等进行分析。
1.4 前噬菌体携带ARGs的预测 收集PHASTER预测得到的前噬菌体基因组序列文件(fna格式文件),然后整理成单个前噬菌体基因组的fasta格式文件,并对这些前噬菌体统一以“phi+Strain-Contig-Region”的格式进行命名。使用抗生素耐药基因数据库(The Comprehensive Antibiotic Research Database,CARD) (https://card.mcmaster.ca/)[15]预测鲍曼不动杆菌前噬菌体基因组中可能携带的ARGs。
1.5 前噬菌体携带VFs的预测 使用毒力因子数据库(Virulence Factors Database,VFDB)(http://www.mgc.ac.cn/VFs/)[16]中的“Search”工具,在其SetA和SetB两个核心数据下提交前噬菌体基因组的fasta格式序列进行blastn,以预测其可能携带的VFs。
2 结果和分析 2.1 菌株的分布概况 从图 1-A可知,收集的82株鲍曼不动杆菌主要来源于印度、中国、墨西哥和美国,分别占35.37%、18.29%、10.98%和9.76%。图 1-B显示宿主来源包括人类、犬、羊及银胶菊,其中人类为主要宿主来源。由图 1-C可知鲍曼不动杆菌主要来源于血液和痰液样本中,两者占比均为25.61%,此外,支气管肺泡灌洗和组织样本分别占9.76%和8.54%。图 1-D显示82株鲍曼不动杆菌引起的疾病类型主要以肺炎和败血症为主,均占14.63%(附表 1)。
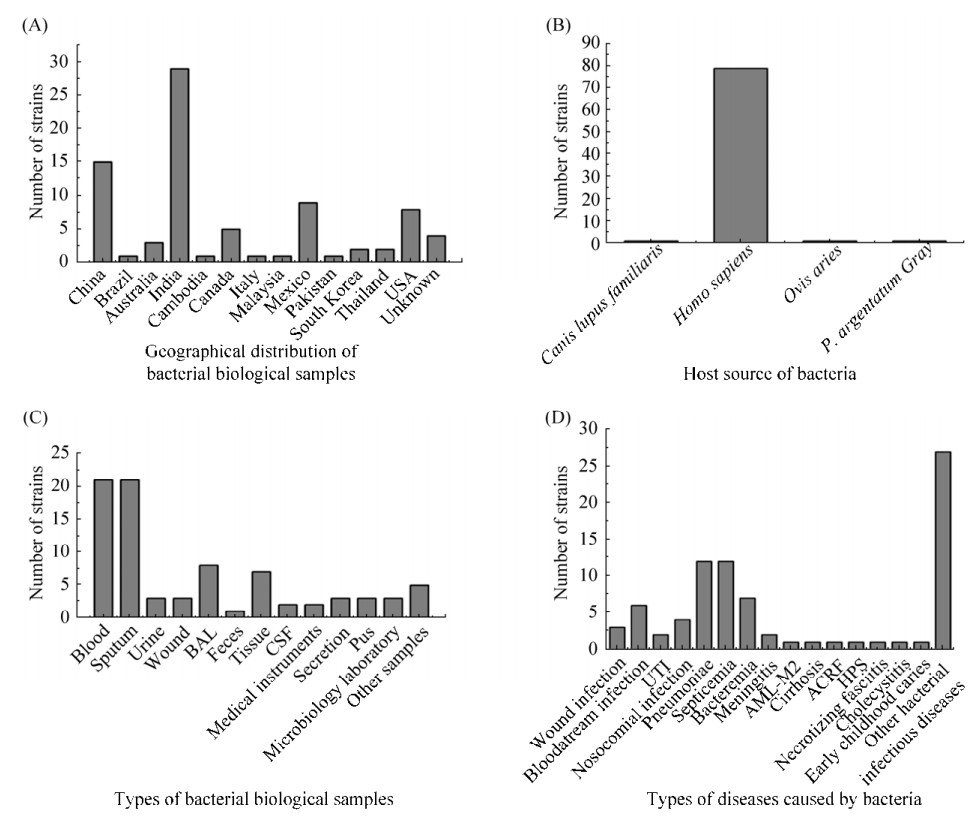 |
| 图 1 82株鲍曼不动杆菌的分布 Figure 1 Distribution of 82 strains of A. baumannii. A: Geographical distribution; B: Host distribution. C: Sample source distribution; D: Disease type distribution. |
| 图选项 |
2.2 鲍曼不动杆菌基因组特征 从图 2可知,鲍曼不动杆菌的基因组较小,82株鲍曼不动杆菌的基因组大小约3.8–4.4 Mb,多数菌株的基因组长4.0–4.1 Mb。GC含量约38.8%–39.2%,多数菌株基因组GC含量为38.95%–39.00%。
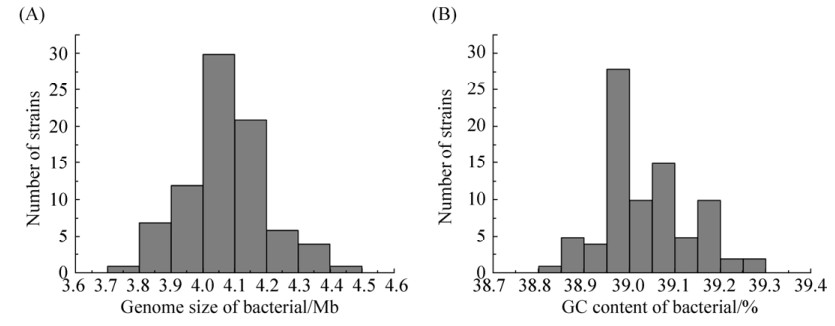 |
| 图 2 82株鲍曼不动杆菌基因组信息 Figure 2 Genomic information of 82 strains of A. baumannii. A: Distribution of bacterial genome size; B: Distribution of GC content in bacterial genome. |
| 图选项 |
2.3 前噬菌体的预测 通过PHASTER在线预测工具,对82株鲍曼不动杆菌全基因组进行前噬菌体预测。本研究共预测到472条前噬菌体,其中完整型前噬菌体201条,疑似型前噬菌体91条,缺陷型前噬菌体180条(图 3-A,附表 1)。由图 3-B可知,平均每个鲍曼不动杆菌基因组中可携带至少5条前噬菌体,包括至少2条完整型前噬菌体、1条疑似型前噬菌体、2条缺陷型前菌体。
 |
| 图 3 前噬菌体预测结果 Figure 3 The prediction results of prophages. A: The number of different types of prophages (intact, questionable, incomplete, and total); B: The average number of different types of prophage carried in per bacterial genome. |
| 图选项 |
2.4 前噬菌体基因组特征分析 对预测的前噬菌体基因组数据进行分析,结果如图 3所示,鲍曼不动杆菌前噬菌体的基因组长度为10–60 kb,GC含量主要为36%–42.5% (图 4-A,4-B)。其中完整型前噬菌体的基因组大小主要为30–60 kb,GC含量主要为38%–42% (图 4-C,4-D);缺陷型前噬菌体的基因组长度相对较小,主要位于10–30 kb范围内,其GC含量则略有偏低,主要分布在34%–42% (图 4-E,4-F)。疑似型前噬菌体的基因组长度主要位于20–60 kb,GC含量主要在36%–42% (图 4-G、H)。值得注意的是,每株鲍曼不动杆菌所携带的全部前噬菌体占其基因组比例约为4%–6%(图 5)。
 |
| 图 4 不同类型前噬菌体的基因组信息 Figure 4 Prophage genome information of different types. A, C, E, G: The size of total prophage, intact, incomplete and questionable prophage genome respectively; B, D, F, H: The GC content of total prophage, intact, incomplete and questionable prophage genomes. |
| 图选项 |
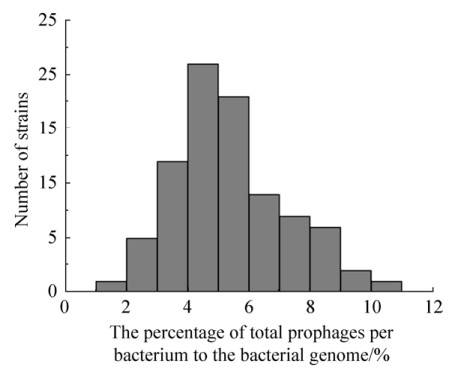 |
| 图 5 前噬菌体占鲍曼不动杆菌基因组的比例 Figure 5 Proportional distribution of prophages carried in A. baumannii genome. |
| 图选项 |
2.5 前噬菌体携带ARGs的预测 本研究通过CARD对前噬菌体的ARGs进行预测,以探讨这些ARGs在鲍曼不动杆菌前噬菌体的分布情况。预测结果显示,29条前噬菌体携带77个耐药基因(附表 2)。耐药表型共有14种,其中主要以氨基糖苷类抗生素(18/29)、磺胺类抗生素(14/29)以及β-内酰胺类抗生素(单环β-内酰胺类、头孢菌素类、青霉烷类和青霉素类抗生素)(13/29)为主。值得注意的是,有3株前噬菌体携带7种ARGs,此外有未明确抗生素类型的占(11/29)(图 6-A)。图 6-B表示的是抗生素耐药基因的家族来源,它们分别来自15个不同的家族,其中以氨基糖苷乙酰转移酶(aminoglycoside acetyltransferases,AAC)、氨基糖苷核苷转移酶(aminoglycoside nucleotidyltransferases,ANT)、MFS抗生素外排泵、耐磺胺类抗生素和TEM β-内酰胺酶5个家族为主。图 6-C显示了这些抗性基因的可能作用机制,包括抗生素灭活(58.44%)、抗生素靶标替代(18.18%)、抗生素靶标保护(3.9%)、抗生素靶点改变 & 抗生素外排(1.3%)、抗生素靶点改变(1.3%)和抗生素外排(16.88%) 6大类。
 |
| 图 6 29株前噬菌体携带ARGs预测结果 Figure 6 Prediction results of 29 prophages carrying ARGs. A: Distribution of drug resistance; B: Source of antibiotic resistance gene family; C: Drug resistance mechanism. |
| 图选项 |
2.6 前噬菌体携带VFs的预测 使用VFDB预测发现132条前噬菌体携带VFs (附表 3),其中完整型前噬菌体73条、疑似型前噬菌体15条、缺陷型前噬菌体44条,分别占总前噬菌体28%、完整型前噬菌体36%、疑似型前噬菌体16%和缺陷型噬菌体24%(图 7-A)。132条前噬菌体共编码503个毒力基因,归类为38种毒力基因(附表 4)和34种VFs(附表 5)。在这34种VFs中,以生物膜相关蛋白Bap(17/132)、荚膜Capsule(35/132)、细菌全局性调控因子CsrA (17/132)和Ⅲ型分泌系统组分Mxi-Spa——CVF465(37/132)为主(图 8)。不同类型的前噬菌体普遍携带1–2种VFs,少数前噬菌体携带3种及以上VFs (图 7-C)。图 7-B显示每个不同类别前噬菌体携带VFs的数量,其中完整型和缺陷型前噬菌体平均约携带1.5个VFs,而疑似型前噬菌体平均约携带1.3个VFs。此外,本研究结果显示不同类型鲍曼不动杆菌前噬菌体所携带VFs的宿主菌来源所占结构比例具有较大差异,除鲍曼不动杆菌外,脑膜炎奈瑟菌(Neisseria meningitidis)、痢疾志贺氏菌(Shigella dysenteriae)、嗜肺军团菌(Legionella pneumophila)及其亚种等也有较高的结构比例,是前噬菌体中VFs的主要来源菌株(图 9)。这提示前噬菌体在不同细菌间毒力基因的水平转移中可能发挥重要作用。
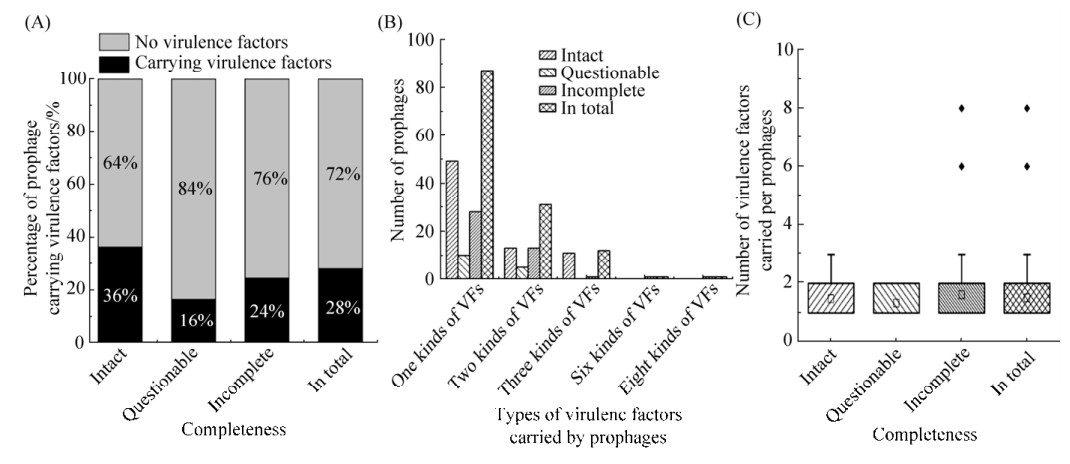 |
| 图 7 毒力因子预测结果 Figure 7 Predicted results of VFs. A: Percentage of virulence factors carried by different types of prophages; B: The average number of VFs carried in each different type of prophages; C: The average number of virulence factors carried by each different type of prophage. |
| 图选项 |
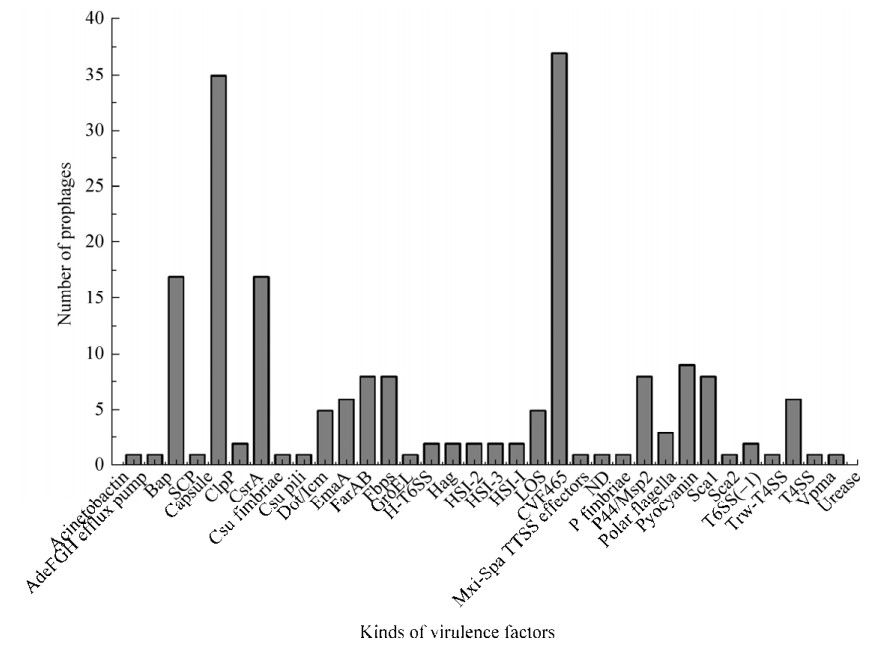 |
| 图 8 不同毒力因子在132条前噬菌体中的分布 Figure 8 The distribution of different VFs in 132 prophages. |
| 图选项 |
 |
| 图 9 毒力因子的宿主来源 Figure 9 The host source of VFs carried by prophages. |
| 图选项 |
3 讨论 不同种细菌之间前噬菌体的携带情况差异较大,有些细菌没有携带前噬菌体,而有些细菌则携带多个前噬菌体,甚至可以携带十几个前噬菌体[17]。本研究利用PHASTER软件从82株鲍曼不动杆菌基因组中预测出472条前噬菌体序列,所有菌株均携带前噬菌体,平均每株菌至少携带5条前噬菌体,其中包括2条完整型前噬菌体,表明鲍曼不动杆菌携带前噬菌体的普遍性。完整型前噬菌体占42.59%,这与Costa等[12]报道的22.88%存在较大差异,这可能与选取的鲍曼不动杆菌基因组的完整性及样本数量有关。前噬菌体在许多细菌中含量很高,最多可占宿主基因组的10%–20%[18]。本研究中鲍曼不动杆菌所携带的前噬菌体占其基因组的比例约4%–6%,这低于埃希菌属、链球菌属和螺旋体属(10%–20%)[18–19],与沙门氏菌属(4.9%)接近,高于洋葱伯克霍尔德氏菌(3.67%)、假伯克霍尔德氏菌(2.34%)和越南伯克霍尔德氏菌(占0.1%–3.4%)[20]。
众所周知噬菌体或前噬菌体可以介导ARGs在细菌间转移[21]。文献报道前噬菌体可通过普遍性转导的方式介导β-内酰胺类、四环素类和萘啶酸类等ARGs在鲍曼不动杆菌中的转移[22]。但有关前噬菌体编码抗生素耐药基因的研究资料较少。Lopez-Leal等[13]报道鲍曼不动杆菌中许多前噬菌体编码ARGs,分属于9个不同的抗生素耐药家族。本研究中29条前噬菌体携带77个耐药基因,分属于14种不同的耐药表型主要以氨基糖苷类抗生素、磺胺类抗生素以及β-内酰胺类抗生素耐药为主,这些结果与Clark等[23]报道的鲍曼不动杆菌抗生素耐药的表型基本相符。
鲍曼不动杆菌的抗生素耐药机制主要有下列几种:(1) 通过抗菌药物灭活酶致使抗菌药物失去效果出现对抗菌药物的耐药,一般是由质粒介导[24–25]。(2) 通过改变药物作用靶点来实现喹诺酮类药物的耐药性[26]。(3) 质粒qnr基因的表达产物对氟喹诺酮类抗菌药物靶位有保护作用也可介导A. baumannii耐药[27]。(4) 外排泵对抗生素药物外排作用,减少了细胞内药物的聚集并提高了最低抑菌浓度(minimum inhibitory concentration,MIC),使菌株产生耐药性[28–29]。本研究中,我们对前噬菌体携带的ARGs可能的耐药机制进行了分析,结果显示耐药机制涵盖6大类,其中主要以抗生素灭活、抗生素靶标替代和抗生素外排为主。这与鲍曼不动杆菌抗生素耐药的主要机制相符,提示前噬菌体在鲍曼不动杆菌ARGs的获得及传播中可能发挥重要作用。
Costa等[12]报道鲍曼不动杆菌前噬菌体可编码多种潜在VFs的潜力,109条完整型前噬菌体中有78%携带有VFs,平均每条前噬菌体携带1.75个VFs。本研究中132条前噬菌体共编码503个毒力基因,归类为38种毒力基因和34种VFs。除73条完整型前噬菌体外,另有44条缺陷型和15条疑似型前噬菌体亦编码毒力基因。在这34种VFs中,以Bap、Capsule、CsrA和CVF465为主。目前,鲍曼不动杆菌中已被报道的VFs主要包括外膜蛋白A(OmpA)、脂多糖(LPS)、荚膜多糖、磷脂酶D(PLD)、青霉素结合蛋白(PBP)和外膜囊泡(OMV)等[30],涉及运动性、黏附性、生物膜的形成和铁的获取[31]。而这些VFs大多数可以在前噬菌体中被找到。此外,我们发现VFs-CsrA和CVF465普遍存在于鲍曼不动杆菌前噬菌体中。值得注意的是,分析这些毒力基因的可能宿主来源发现除鲍曼不动杆菌外,N. meningitidis、S. dysenteriae、L. pneumophila及其亚种菌等具有较高的结构比例。前噬菌体编码的毒力基因在大肠埃希菌、化脓链球菌和金黄色葡萄球菌等细菌致病过程中的作用均已被证实[7]。尽管鲍曼不动杆菌前噬菌体编码了多种毒力基因,但其致病作用还有待进一步证实。
4 结论 鲍曼不动杆菌普遍携带前噬菌体,但前噬菌体基因在鲍曼不动杆菌基因组中所占比例不高,约4%–6%。部分前噬菌体携带ARGs,以氨基糖苷类、磺胺类及β-内酰胺类耐药为主。近30%的前噬菌体携带毒力基因,共鉴定出34种毒力因子。前噬菌体可能在鲍曼不动杆菌抗生素耐药性的获得、传播及致病性演变中发挥重要作用。
补充材料附表 1. 鲍曼不动杆菌及其前噬菌体的基因信息
附表 2. 鲍曼不动杆菌前噬菌体携带抗生素耐药基因的预测
附表 3. 鲍曼不动杆菌前噬菌体携带毒力因子的预测
附表 4. 132 株鲍曼不动杆菌前噬菌体毒力基因的分布情况
附表 5. 132 株鲍曼不动杆菌前噬菌体毒力因子的分布情况
本文补充材料见网络版 http://journals.im.ac.cn/actamicrocn。补充材料为作者提供的原始数据,作者对其学术质量和内容负责。
References
| [1] | Zhou H, Zhou JY, Yu YS. The interpretation of Chinese expert consensus for the diagnosis, treatment, prevention and control of Acinetobacter baumannii infection. Chinese Journal of Evidence-Based Medicine, 2016, 16(1): 26-29. (in Chinese) 周华, 周建英, 俞云松. 中国鲍曼不动杆菌感染诊治与防控专家共识解读. 中国循证医学杂志, 2016, 16(1): 26-29. |
| [2] | System CARS. Antimicrobial resistance of bacteria: surveillance report from China Antimicrobial Resistance Surveillance System in 2014-2019. Chinese Journal of Infection Control, 2021, 20(1): 15-31. (in Chinese) 全国细菌耐药监测网. 全国细菌耐药监测网2014—2019年细菌耐药性监测报告. 中国感染控制杂志, 2021, 20(1): 15-31. |
| [3] | Chen BY, He LX, Hu BJ, Ni YX, Qiu HB, Shi Y, Shi Y, Wang H, Wang GM, Yang Y, Zhang J, Yu YS. Chinese expert consensus for the diagnosis, treatment, prevention and control of Acinetobacter baumannii infection. China Medicine and Pharmacy, 2012, 2(8): 3-8. (in Chinese) 陈佰义, 何礼贤, 胡必杰, 倪语星, 邱海波, 石岩, 施毅, 王辉, 王明贵, 杨毅, 张菁, 俞云松. 中国鲍曼不动杆菌感染诊治与防控专家共识. 中国医药科学, 2012, 2(8): 3-8. |
| [4] | Gales AC, Seifert H, Gur D, Castanheira M, Jones RN, Sader HS. Antimicrobial susceptibility of Acinetobacter calcoaceticus-Acinetobacter baumannii complex and Stenotrophomonas maltophilia clinical isolates: results from the SENTRY antimicrobial surveillance program (1997-2016). Open Forum Infectious Diseases, 2019, 6(Supplement_1): S34-S46. DOI:10.1093/ofid/ofy293 |
| [5] | Harrison E, Brockhurst MA. Ecological and evolutionary benefits of temperate phage: what does or doesn't kill you makes you stronger. BioEssays, 2017, 39(12): 1700112. DOI:10.1002/bies.201700112 |
| [6] | Boyd EF, Brüssow H. Common themes among bacteriophage-encoded virulence factors and diversity among the bacteriophages involved. Trends in Microbiology, 2002, 10(11): 521-529. DOI:10.1016/S0966-842X(02)02459-9 |
| [7] | Taylor VL, Fitzpatrick AD, Islam Z, Maxwell KL. The diverse impacts of phage morons on bacterial fitness and virulence. Advances in Virus Research, 2019, 103: 1-31. |
| [8] | Balcázar JL. Implications of bacteriophages on the acquisition and spread of antibiotic resistance in the environment. International Microbiology, 2020, 23(4): 475-479. DOI:10.1007/s10123-020-00121-5 |
| [9] | Wendling CC, Refardt D, Hall AR. Fitness benefits to bacteria of carrying prophages and prophage-encoded antibiotic-resistance genes peak in different environments. Evolution, 2021, 75(2): 515-528. DOI:10.1111/evo.14153 |
| [10] | Haaber J, Leisner JJ, Cohn MT, Catalan-Moreno A, Nielsen JB, Westh H, Penadés JR, Ingmer H. Bacterial viruses enable their host to acquire antibiotic resistance genes from neighbouring cells. Nature Communications, 2016, 7: 13333. DOI:10.1038/ncomms13333 |
| [11] | Wang MZ, Zeng ZL, Jiang FW, Zheng Y, Shen HG, Macedo N, Sun YX, Sahin O, Li GW. Role of enterotoxigenic Escherichia coli prophage in spreading antibiotic resistance in a porcine-derived environment. Environmental Microbiology, 2020, 22(12): 4974-4984. DOI:10.1111/1462-2920.15084 |
| [12] | Costa AR, Monteiro R, Azeredo J. Genomic analysis of Acinetobacter baumannii prophages reveals remarkable diversity and suggests profound impact on bacterial virulence and fitness. Scientific Reports, 2018, 8(1): 15346. DOI:10.1038/s41598-018-33800-5 |
| [13] | López-Leal G, Santamaria RI, Cevallos Má, Gonzalez V, Castillo-Ramírez S. Letter to the editor: prophages encode antibiotic resistance genes in Acinetobacter baumannii. Microbial Drug Resistance, 2020, 26: 1275-1277. DOI:10.1089/mdr.2019.0362 |
| [14] | Arndt D, Grant JR, Marcu A, Sajed T, Pon A, Liang YJ, Wishart DS. PHASTER: a better, faster version of the PHAST phage search tool. Nucleic Acids Research, 2016, 44(W1): W16-W21. DOI:10.1093/nar/gkw387 |
| [15] | Alcock BP, Raphenya AR, Lau TTY, Tsang KK, Bouchard M, Edalatmand A, Huynh W, Nguyen ALV, Cheng AA, Liu SH, Min SY, Miroshnichenko A, Tran HK, Werfalli RE, Nasir JA, Oloni M, Speicher DJ, Florescu A, Singh B, Faltyn M, Hernandez-Koutoucheva A, Sharma AN, Bordeleau E, Pawlowski AC, Zubyk HL, Dooley D, Griffiths E, Maguire F, Winsor GL, Beiko RG, Brinkman FSL, Hsiao WWL, Domselaar GV, Mcarthur AG. CARD 2020: antibiotic resistome surveillance with the comprehensive antibiotic resistance database. Nucleic Acids Research, 2020, 48: D517-D525. DOI:10.1093/nar/gkz1136 |
| [16] | Liu B, Zheng DD, Jin Q, Chen LH, Yang J. VFDB 2019: a comparative pathogenomic platform with an interactive web interface. Nucleic Acids Research, 2019, 47(D1): D687-D692. DOI:10.1093/nar/gky1080 |
| [17] | Touchon M, Bernheim A, Rocha EP. Genetic and life-history traits associated with the distribution of prophages in bacteria. The ISME Journal, 2016, 10(11): 2744-2754. DOI:10.1038/ismej.2016.47 |
| [18] | Casjens S. Prophages and bacterial genomics: what have we learned so far?. Molecular Microbiology, 2003, 49(2): 277-300. DOI:10.1046/j.1365-2958.2003.03580.x |
| [19] | Bobay LM, Rocha EPC, Touchon M. The adaptation of temperate bacteriophages to their host genomes. Molecular Biology and Evolution, 2013, 30(4): 737-751. DOI:10.1093/molbev/mss279 |
| [20] | Weiser R, Yap ZL, Otter A, Jones BV, Salvage J, Parkhill J, Mahenthiralingam E. A novel inducible prophage from Burkholderia vietnamiensis G4 is widely distributed across the species and has lytic activity against pathogenic Burkholderia. Viruses, 2020, 12(6): 601. DOI:10.3390/v12060601 |
| [21] | Torres-Barceló C. The disparate effects of bacteriophages on antibiotic-resistant bacteria. Emerging Microbes & Infections, 2018, 7(1): 168. |
| [22] | Wachino JI, Jin WC, Kimura K, Arakawa Y. Intercellular transfer of chromosomal antimicrobial resistance genes between Acinetobacter baumannii strains mediated by prophages. Antimicrobial Agents and Chemotherapy, 2019, 63: e00334-19. DOI:10.1128/AAC.00334-19 |
| [23] | Clark NM, Zhanel GG, Lynch JP. Emergence of antimicrobial resistance among Acinetobacter species: a global threat. Current Opinion in Critical Care, 2016, 22(5): 491-499. DOI:10.1097/MCC.0000000000000337 |
| [24] | Cecchini T, Yoon EJ, Charretier Y, Bardet C, Beaulieu C, Lacoux X, Docquier JD, Lemoine J, Courvalin P, Grillot-Courvalin C, Charrier JP. Deciphering multifactorial resistance phenotypes in Acinetobacter baumannii by genomics and targeted label-free proteomics. Molecular & Cellular Proteomics, 2018, 17(3): 442-456. |
| [25] | Thomson GK, Ayaz M, Lutes K, Thomson KS. An improved extended-spectrum-β-lactamase detection test utilizing aztreonam plus clavulanate. Journal of Clinical Microbiology, 2018, 56(1): e01309-17. DOI:10.1128/JCM.01309-17 |
| [26] | Gu DX, Hu YJ, Zhou HW, Zhang R, Chen GX. Substitutions of Ser83Leu in GyrA and Ser80Leu in ParC associated with quinolone resistance in Acinetobacter pittii. Microbial Drug Resistance: Larchmont, NY, 2015, 21(3): 345-351. DOI:10.1089/mdr.2014.0057 |
| [27] | Higuchi S, Shikata M, Chiba M, Hoshino K, Gotoh N. Characteristics of antibiotic resistance and sequence type of Acinetobacter baumannii clinical isolates in Japan and the antibacterial activity of DS-8587. Journal of Infection and Chemotherapy, 2014, 20(4): 256-261. DOI:10.1016/j.jiac.2013.12.001 |
| [28] | Reens AL, Crooks AL, Su CC, Nagy TA, Reens DL, Podoll JD, Edwards ME, Yu EW, Detweiler CS. A cell-based infection assay identifies efflux pump modulators that reduce bacterial intracellular load. PLoS Pathogens, 2018, 14(6): e1007115. DOI:10.1371/journal.ppat.1007115 |
| [29] | Abdi SN, Ghotaslou R, Ganbarov K, Mobed A, Tanomand A, Yousefi M, Asgharzadeh M, Kafil HS. Acinetobacter baumannii efflux pumps and antibiotic resistance. Infection and Drug Resistance, 2020, 13: 423-434. DOI:10.2147/IDR.S228089 |
| [30] | Doi Y, Murray GL, Peleg AY. Acinetobacter baumannii: evolution of antimicrobial resistance-treatment options. Seminars in Respiratory and Critical Care Medicine, 2015, 36(1): 85-98. DOI:10.1055/s-0034-1398388 |
| [31] | Garcia-Quintanilla M, Pulido MR, McConnell MJ. First steps towards a vaccine against Acinetobacter baumannii. Current Pharmaceutical Biotechnology, 2013, 14(10): 897-902. |
