陈璐, 王嘉良, 梁晶丹, 邓子新, 汪志军


上海交通大学生命科学技术学院微生物代谢国家重点实验室, 上海 200030
收稿日期:2021-03-03;修回日期:2021-04-21;网络出版日期:2021-09-28
基金项目:国家重点研发计划(2018YFA0900700, 2019YFA0905400)
*通信作者:汪志军, E-mail: wangzhijun@sjtu.edu.cn.
摘要:[目的] 利用酿酒酵母表达系统,通过乙醇脱氢酶启动子异源表达细菌源的铁载体合成蛋白PchE,并与来源于枯草芽孢杆菌的泛酰化酶Sfp同宿主共表达,探索真核表达体系表达具有生化活性的细菌源蛋白。[方法] 从大肠杆菌BAP 1染色体上扩增sfp基因,将pchE基因及串联的pchE与sfp基因分别构建到酵母-大肠杆菌穿梭质粒pXW55中,各自转化酿酒酵母BJ5464-npgA表达,经过亲和层析和离子交换层析纯化蛋白,利用HPLC检测细菌源与酵母源表达的PchE在体外重构生化反应中的催化活性。[结果] 利用酿酒酵母表达系统可以获得高纯度的原核蛋白PchE。真菌源的泛酰化基因NpgA和细菌源的Sfp,均可泛酰化修饰PchE,合成中间产物HPT-Cys。[结论] 在酿酒酵母Saccharomyces cerevisiae BJ5464-npgA表达系统中,首次证明真菌源的泛酰化基因NpgA和细菌源的Sfp,均可泛酰化修饰细菌源的非核糖体肽合酶。比较酵母和细菌宿主的目标蛋白表达,证明酵母表达的巨大蛋白PchE的纯度更高,非特异性条带减少,推测酵母宿主可能更适合表达纯化功能性的巨型蛋白质。
关键词:铁载体合成蛋白PchE酿酒酵母异源表达与纯化生化活性PPTase
Heterologous expression and purification of the activated siderophore synthetase PchE in Saccharomyces cerevisiae
Lu Chen, Jialiang Wang, Jingdan Liang, Zixin Deng, Zhijun Wang


State Key Laboratory of Microbial Metabolism and School of Life Science & Biotechnology, Shanghai Jiao Tong University, Shanghai 200030, China
Received: 3 March 2021; Revised: 21 April 2021; Published online: 28 September 2021
*Corresponding author: Zhijun Wang, E-mail: wangzhijun@sjtu.edu.cn.
Foundation item: Supported by the National Key Research and Development Program of China (2018YFA0900700, 2019YFA0905400)
Abstract: [Objective] Expressing the bacterial siderophore synthetase PchE under the ADH2 promoter in Saccharomyces cerevisiae system with the PPTase Sfp from Bacillus subtilis, to explore the heterologous expression of activated bacterial protein in eukaryotic system. [Methods] The sfp gene was amplified from Escherichia coli BAP 1. Both pchE gene and pchE tandem with sfp were cloned to the yeast-E. coli shuttle vector pXW55, and transformed into the Saccharomyces cerevisiae BJ5464-npgA strain. After the purification steps including affinity chromatography and ion-exchange chromatography, HPLC was used to detect whether the PchE from E. coli and Saccharomyces cerevisiae maintain catalytic activity in the biochemical reaction in vitro. [Results] In the Saccharomyces cerevisiae expression system, prokaryotic protein PchE was obtained with high purity. And no matter assisted by the bacterial or fungal PPTase, PchE can be modified and synthesize intermediate product HPT-Cys. [Conclusion] It was firstly demonstrated that both fungal gene npgA and bacterial gene sfp can modify bacterial nonribosomal peptide synthase. With the comparison of protein expression between yeast and bacterial host, the giant protein PchE expressed by yeast has higher purity and fewer nonspecific bands. This suggested that yeast host might be more suitable for expressing and purifying functional giant protein.
Keywords: siderophore synthetase PchESaccharomyces cerevisiaeheterologous expression and purificationbiochemical activityPPTase
重组蛋白是通过基因重组技术获得的具有一定活性和功能的蛋白质,近年来重组蛋白在药物挖掘领域中发挥越来越重要的作用[1]。获得重组蛋白可以通过体内和体外两种途径,目前常用的体外表达体系包括:原核表达体系,如大肠杆菌(Escherichia coli);以及真核表达体系,如酿酒酵母(Saccharomyces cerevisiae)、昆虫细胞和哺乳动物细胞等。
大肠杆菌凭借遗传操作简单、繁殖时间短和培养成本低的优势,通常作为重组蛋白表达的首选宿主,用于表达原核及一些真核来源的蛋白质[2]。然而,由于原核宿主往往缺乏成熟的翻译后修饰体系,很多情况下,外源蛋白容易形成胞内聚集体,丧失酶活性,缺少正确折叠构象[3];尤其对于一些大分子量的巨型蛋白[4],难以在大肠杆菌中实现高效、稳定表达。障碍蛋白表达的主要因素可能是原核表达体系中转录和翻译元件少,分子伴侣不足,蛋白溶解度低,或外源蛋白存在潜在毒性等。
酵母细胞作为真核表达体系,在异源表达巨型蛋白方面具备多重优势[5]。首先,酵母细胞含有线粒体、内质网等细胞器,利于表达有生物活性的外源蛋白;其次,酵母细胞具有丰富的翻译后修饰酶,蛋白更易折叠形成天然构象;此外,酵母表达体系无内毒素,无需添加抗生素,环境胁迫小;相比于昆虫和哺乳动物细胞,酵母细胞能实现高密度发酵,更适应工业化生产条件。酵母-大肠杆菌穿梭质粒pXW55是实现聚酮合酶(polyketide synthase,PKS)和非核糖体肽合酶(nonribosomal peptide,NRPS)异源表达的有力工具[6]。pXW55在外源基因插入位点的上下游分别携带乙醇脱氢酶(alcohol dehydrogenase)ADH2启动子和终止子,在葡萄糖耗尽时,通过乙醇起始转录并诱导巨型蛋白的过表达。同时,载体带有ura3基因,可作为营养缺陷型筛选标记。此外,pXW55还携带来自大肠杆菌的复制起点、β-内酰胺酶基因以及氨苄青霉素抗性基因,可以在大肠杆菌中进行遗传操作。
铁离子作为多种酶的辅因子,是多数生物不可或缺的[7]。铁载体是微生物在缺铁环境中合成的一种小分子化合物,能够高度亲和地螯合宿主内不可溶的Fe3+并运输到细胞内,是病原菌对抗和侵染宿主的重要手段[8]。铜绿假单胞菌(Pseudomonas aeruginosa)是一种机会致病菌,它能合成两种重要的铁载体,铜绿菌素(pyochelin)[9]和荧光嗜铁素(pyoverdine)[10]。两者是铜绿假单胞菌高致病性的重要因素[11],有研究表明铁载体的缺失会显著影响毒株的生长和毒力[12]。铁载体Pyochelin是由1分子水杨酸和2分子半胱氨酸合成的双杂环结构,相关生物合成途径由Walsh等揭示[13–14]。它的合成基因簇包括pchEF和pchDCBA,其中pchE基因编码的PchE蛋白是pyochelin合成的关键延伸模块。PchE的编码基因长4.4 kb,蛋白单体大小160 kDa,负责催化水杨酸和第一个半胱氨酸的环化,形成羟苯基噻唑-半胱氨酸(hydroxyphenyl-thiazolinyl-cysteine,HPT-Cys)。PchE属于NRPS,在结构上包括芳香基载体蛋白(aryl carrier protein,ArCP)、环化结构域(heterocyclization domain,Cy)、腺苷化结构域(adenylation domain,A)、表异构酶结构域(epimerase domain,E)和肽酰基载体蛋白(peptidyl carrier protein,PCP)。目前开展的研究中,PchE蛋白表达宿主通常为大肠杆菌和铜绿假单胞菌,均在原核表达系统中实现[15–17],使用真核宿主高效表达PchE尚未见报道。
在Pyochelin的生物合成途径中(图 1),PchD和PchE共同参与HPT-Cys的合成。PchD的A1结构域负责活化底物水杨酸,并连接到PchE ArCP结构域的4′-磷酸泛酰巯基乙胺(4′-phosphopantetheine,PPant)手臂上。PchE的A2结构域负责活化底物L-半胱氨酸,并连接到PCP的PPant手臂上,Cy1结构域催化水杨酸和半胱氨酸发生环化,E结构域催化L-半胱氨酸的异构化,最后硫解释放HPT-Cys[14, 18]。
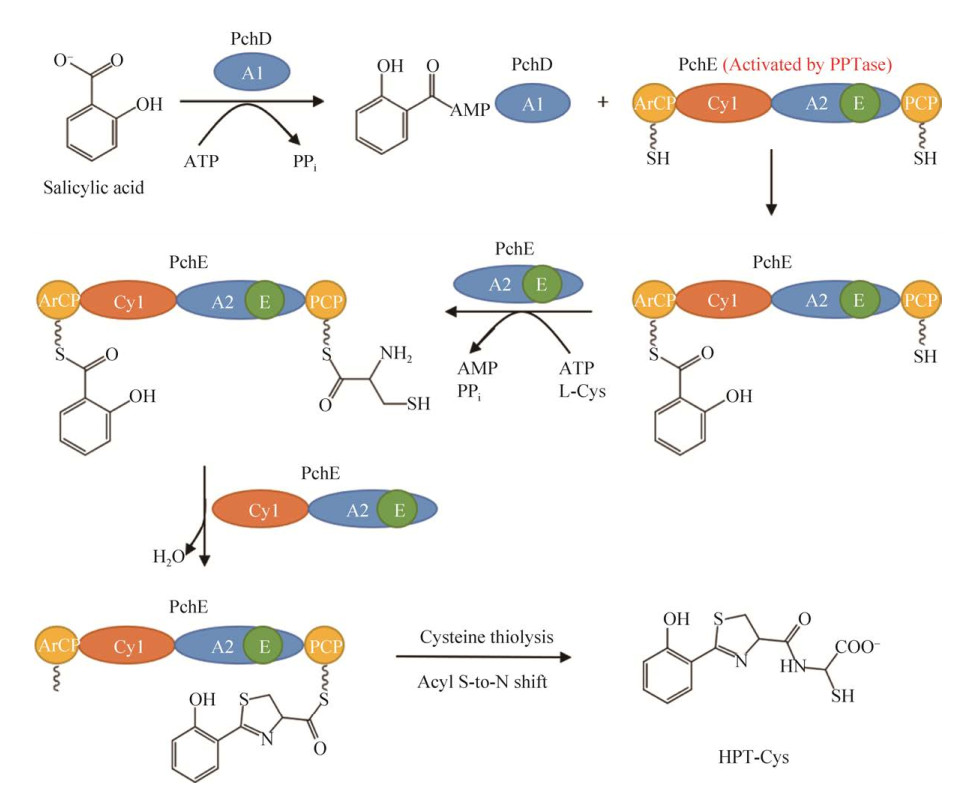 |
| 图 1 PchD与PchE合成HPT-Cys的生化途径 Figure 1 Biosynthesis pathway of HPT-Cys catalyzed by PchD and PchE. |
| 图选项 |
磷酸泛酰巯基乙胺基转移酶(phosphopantetheinyl transferase,PPTase)用于激活PKS和NRPS的载体蛋白结构域,在蛋白翻译后修饰中不可或缺。PPTase将载体蛋白的保守丝氨酸位点与来源于辅酶A的PPant手臂共价连接,使其从无活性的holo-载体蛋白转变为apo-载体蛋白,进而启动PKS和NRPS合成[19]。细菌源的PPTase Sfp蛋白最早由Grossman等在枯草芽孢杆菌中发现并鉴定[20],它的编码基因长675 bp,对应蛋白分子量为26 kDa,由225个氨基酸组成[21]。在大肠杆菌中,Sfp能高效表达,表现出广泛的催化活性和较好的修饰底物宽泛性[19]。真菌源的NpgA蛋白是在构巢曲霉(Aspergillus nidulans)中发现的一种PPTase[22],能激活初级和次级代谢。有研究表明,在酿酒酵母的赖氨酸合成途径中,Lys5蛋白执行PPTase的功能,而当lys5突变后,回补sfp和npgA基因可以在酵母细胞中发挥替代作用[23]。
本研究以铁载体合成蛋白PchE为研究对象,首次在真核表达体系酿酒酵母细胞中实现了PchE的异源表达,优化了巨型蛋白表达不完整的情况,蛋白纯度显著提高,并通过重构体外生化反应体系验证了PchE的功能活性,表明真核宿主在巨型蛋白表达上更具优势。此外,实验证明真菌源的PPTase NpgA具有较好的底物宽泛性,可以在酵母宿主中对原核蛋白PchE进行泛酰化修饰。而当共表达细菌源的PPTase Sfp蛋白后,PchE催化活性没有明显变化,两者可能并非协同发挥作用。
1 材料和方法 1.1 材料
1.1.1 质粒与菌株: 实验所用质粒pXW55-cH、pET28a-pchE-cH和pXW55-pchE-cH,菌株大肠杆菌DH10B、BAP 1(BL21菌株整合sfp基因)和酿酒酵母BJ5464-npgA为上海交通大学微生物代谢国家重点实验室保存。PchD蛋白表达纯化参照先前研究[14]。
1.1.2 培养基: LB培养基(g/L):酵母提取物5,胰蛋白胨10,氯化钠10,纯水定容至1 L,调节pH值7.0–7.4;LA培养基(g/L):酵母提取物5,胰蛋白胨10,氯化钠10,1.5%–2%琼脂,纯水定容至1 L,调节pH值7.0–7.4;YPD (yeast extract peptone dextrose)液体培养基(g/L):酵母提取物10,蛋白胨20,纯水定容至950 mL灭菌备用;母液D-葡萄糖单独115 ℃灭菌,使用时补加终浓度2%的D-葡萄糖;UDM (uracil dropout media)固体培养基(g/L):酪蛋白水解物5,无氨基酵母氮源6.7,D-葡萄糖20,腺嘌呤半硫酸盐0.2,色氨酸0.2,琼脂20。酪蛋白水解物灭菌备用,其他单独配制母液过滤除菌。UDM平板用于质粒pXW55转化酵母的营养缺陷型筛选。UDM液体培养基不添加琼脂。
1.1.3 试剂: 蛋白胨、胰蛋白胨、酵母提取物购自OXOID,琼脂购自海燕,乙酸乙酯、乙腈、甲酸、三乙胺、L-半胱氨酸购自Sigma,ATP、辅酶A、水杨酸、NADPH、异丙基-β-D-硫代半乳糖苷(IPTG)购自生工生物,SAM购自HARVEYBIO,酵母转化试剂盒购自ZYMO RESEARCH,Quick-Fusion无缝克隆试剂盒购自Biotool,Xho I限制性内切酶购自Thermo,葡萄糖、氯化钠、盐酸等常规试剂购自国药。蛋白纯化使用的Ni-NTA树脂、阴离子交换层析柱Mono Q和体积排阻色谱柱Superdex 200 10/300均购自GE。
1.2 大肠杆菌总DNA提取 在LB培养基中37 ℃过夜培养大肠杆菌BAP 1,取1.5 mL菌液收集到离心管中,12000 r/min离心2 min,弃上清。加入365 μL Tris-EDTA溶液重悬菌体,再加入10 μL 50 mg/mL溶菌酶,置于37 ℃水浴锅内温浴30 min。反应后加入100 μL 10%十二烷基硫酸钠(sodium dodecyl sulfate,SDS)溶液,继续37 ℃水浴直至溶液澄清。往裂解后的溶液中加入0.8倍体积的酚: 氯仿: 异戊醇(25:24:1)溶液,并充分摇晃混合均匀,12000 r/min离心15 min,小心移取上层部分到新离心管中,再加入0.8倍体积的氯仿并充分混匀,12000 r/min离心15 min,保留上清。加入等体积75%乙醇充分混匀后12000 r/min离心10 min。弃上清,离心管自然干燥后加入40 μL TE溶液溶解DNA,并电泳检验DNA质量。
1.3 重组质粒构建 将PCR扩增得到的载体与片段胶回收后,用Quick-Fusion无缝克隆试剂盒进行连接。本实验所用引物详见表 1。在PCR引物设计时,将酶连载体和片段的引物末端各设计18 bp的同源序列,反应时在离心管中加入如下酶连体系:载体20 ng,片段60 ng,Fusion Enzyme 1 μL,5× Fusion Buffer 2 μL,补加dd H2O至10 μL。将反应体系在37 ℃水浴锅中孵育30 min,即可用于大肠杆菌转化。
表 1. 引物名称与序列 Table 1. Primers used in this study
| Primer name | Sequence (5′→3′) |
| Sfp-18P1-38-SP | TCGTAATACCATATGGCTATGAAGATTTACGGAATTTA |
| Sfp-18P1-38-AP | TTTAAATTAGTGATGGTGTTATAAAAGCTCTTCGTACG |
| V-XW55-3H-SP | CACCATCACTAATTTAAATGA |
| V-XW55-2aa-AP | AGCCATATGGTATTACGATA |
| ADH2p-Ter-SP | GGAGCTCGGATCCATTTAGC |
| ADH2p-Ter-AP | CAGTGAATTCGAGCTCGGTA |
| V-XWgeneP1-S | GCCAAGCTCGAAATTAACCCTCA |
| V-XWgeneP1-A | GGCATGCAAGCTTGGCGTAA |
表选项
1.4 大肠杆菌转化 转化时将大肠杆菌DH10B感受态细胞与5 μL酶连体系混合,冰上静置30 min后,42 ℃热击1 min,再加入1 mL LB培养基于37 ℃摇床预培养40 min,吸取适量菌液涂布氨苄青霉素抗性LB平板,置于37 ℃培养箱培养约16 h,观察到大小合适的菌落。挑取单克隆转化子,提取质粒并进行琼脂糖凝胶电泳,选取大小正确的质粒进行测序验证,得到阳性克隆。
1.5 酵母细胞转化 按照ZYMO RESEARCH酵母转化试剂盒(货号T2001)方法,培养酿酒酵母细胞到对数生长期,离心后弃上清,用试剂盒内的EZ 1溶液清洗菌体,再用EZ 2溶液重悬并分装酵母感受态细胞。转化时吸取方法1.5中测序正确的重组质粒DNA 0.2–1.0 μg (体积小于5 μL),与50 μL感受态细胞混合,再加入500 μL EZ 3溶液并小心混匀,于28 ℃静置孵育2 h,最后将溶液全部涂布到UDM固体培养基上,28 ℃培养2–3 d,直至出现大小合适的酵母菌落。转化筛选到的转化子进行测序验证。
1.6 重组蛋白诱导表达 大肠杆菌诱导表达:将冻存的pET28a-pchE- cH/BAP 1菌液划单克隆,接种于10 mL带有卡那霉素抗性的LB培养基中,37 ℃过夜培养作为种子液。接种时按1%接种量,转接1 L带有卡那霉素抗性的LB培养基,37 ℃培养约3 h至对数生长期,将培养基降温至16 ℃,加入终浓度0.1 mmol/L的IPTG,继续16 ℃诱导表达24 h,4500 r/min离心15 min收集菌体,并将菌体转移到50 mL离心管中,–80 ℃冻存备用。
酵母细胞诱导表达:分别挑取pXW55-pchE- cH/BJ5464-npgA和pXW55-pchEcH-sfp/BJ5464- npgA的酵母细胞单克隆,接种于20 mL UDM液体培养基中(用10层纱布封口),28 ℃、220 r/min培养48 h作为种子液。按2%接种量,将种子液转接至1 L YPD培养基中,28 ℃、220 r/min培养72 h。4500 r/min离心10 min收集菌体,并将菌体转移到50 mL离心管中,–80 ℃冻存备用。
1.7 蛋白纯化 用缓冲液A (50 mmol/L Tris-HCl, 100 mmol/L氯化钠, 40 mmol/L咪唑, pH 8.0)重悬菌体,细胞破碎仪高压破碎,其中大肠杆菌8.0×104 Pa破碎3 min,酵母细胞1.2×105 Pa破碎5 min。破碎后菌液12000 r/min离心1 h,保留上清液,由于酵母细胞破碎离心后上清液不完全澄清,在亲和层析前先使用0.45 μm滤膜过滤。使用Ni-NTA作为第一步亲和层析,将上清液与2 mL Ni-NTA树脂混合,4 ℃孵育2 h,然后用5个床体积含500 mmol/L氯化钠的高盐缓冲液清洗,再加5个床体积含50 mmol/L氯化钠的低盐缓冲液平衡盐浓度,最终洗脱液的咪唑浓度为400 mmol/L。
分别收集亲和层析洗脱的蛋白质,全部上样Mono Q离子交换层析柱进行纯化和浓缩,其中A相为50 mmol/L Tris-HCl;B相为50 mmol/L Tris-HCl、1000 mmol/L氯化钠,设定程序为5个柱体积的10% B,5个柱体积的10%–20% B,20个柱体积的20%–40% B,5个柱体积的40%–100% B,5个柱体积的100%–10% B。根据出峰位置和SDS-聚丙烯酰胺凝胶电泳(SDS-PAGE)结果,收集对应的浓缩后蛋白,加终浓度10%甘油,–80 ℃冻存备用。
1.8 体外生化活性验证 体外生化反应体系为200 μL,具体体系成分见表 2。将除了蛋白外所有组分在冰上充分混合后,测定pH值在7.6左右,再往体系中加入蛋白PchD、PchE,置于25 ℃水浴2.5 h,加入20 μL 1 mol/L盐酸使酶失活终止反应。用1 mL乙酸乙酯萃取反应体系后离心取上清,37 ℃真空旋干,加40 μL 10%乙腈溶解干片,用于高效液相色谱(HPLC)测定。HPLC流动相A组分为2.3 μmol/L甲酸和1.44 μmol/L三乙胺的水溶液,B组分为2.3 μmol/L甲酸和1.44 μmol/L三乙胺的乙腈溶液,设定流速为0.8 mL/min,设定最大吸收波长254 nm,使用C18反相柱对产物进行分离和分析。
表 2. PchD与PchE体外生化反应体系成分 Table 2. In vitro biochemical reaction system of PchD and PchE
| Components | Final concentration | Stock concentration/(mmol/L) | Added volume/(μL) |
| Tris-HCl (pH 8.2) | 75 mmol/L | 1000 | 15 |
| MgCl2 | 10 mmol/L | 200 | 10 |
| ATP (Present) | 5 mmol/L | 100 | 10 |
| CoA | 0.1 mmol/L | 100 | 0.2 |
| Salicylic acid | 1 mmol/L | 100 | 2 |
| L-Cysteine (Present) | 5 mmol/L | 100 | 10 |
| NADPH | 1 mmol/L | 50 | 4 |
| SAM | 1 mmol/L | 50 | 4 |
| PchD | 2.8 μmol/L | Calculated according to actual protein concentration | |
| PchE | 1.4 μmol/L | ||
| H2O | Add to 200 μL | ||
表选项
2 结果和分析 2.1 pchE与sfp基因串联酵母表达载体构建 从大肠杆菌BAP 1总DNA中扩增sfp基因,并克隆到酵母-大肠杆菌穿梭质粒pXW55-cH的ADH2启动子下,经测序验证得到阳性克隆pXW55-sfp,并以此为PCR模板,获得载体骨架V-XW55-sfp (图 2-A,泳道1)。从团队已构建质粒pXW55- pchE-cH上扩增带有启动子和终止子的目的基因序列F-pchE-cH,pchE基因C端带有6×His标签(图 2-A,泳道2),与载体骨架无缝酶连后得到重组质粒,DNA片段大小为12.2 kb (图 2-B)。转化大肠杆菌DH10B后挑取重组子提取质粒,并用Xho I酶切验证,将酶切得到的片段长度与预计的酶切结果相符(图 2-C)且测序结果正确的质粒命名为pXW55-pchEcH-sfp。pchE基因与sfp基因分别带有独立的酵母乙醇脱氢酶启动子和终止子,可以在酿酒酵母中进行稳定的复制和表达。
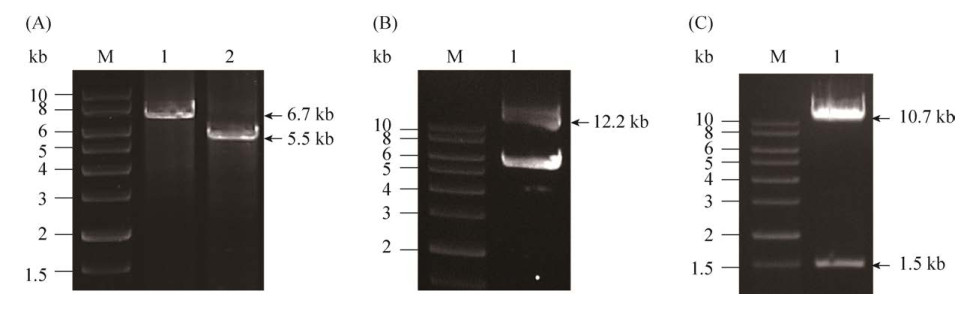 |
| 图 2 重组质粒pXW55-pchEcH-sfp克隆构建及酶切验证 Figure 2 Plasmid construction and verification of pXW55-pchEcH-sfp. A: Agarose electrophoresis of DNA segments amplified by PCR. Lane 1: The vector segment V-XW-sfp; lane 2: The fragment F-pchE-cH. B: Agarose electrophoresis of the recombinant plasmid pXW55-pchEcH-sfp. C: Verification of the recombinant plasmid by restriction enzyme Xho I. |
| 图选项 |
2.2 大肠杆菌sfp宿主表达纯化PchE与活性分析 用His-Tag纯化大肠杆菌宿主表达的PchE蛋白,SDS-PAGE结果显示(图 3-A,泳道2),PchE蛋白大小为160 kDa,洗脱液中蛋白浓度较高,但目的条带下方存在许多非特异性条带,蛋白纯度差。初步推测杂带是由于单步骤纯化效率不高造成的杂蛋白污染,进而将亲和层析洗脱的PchE蛋白穿过阴离子交换色谱柱Mono Q和体积排阻色谱柱Superdex 200,希望进一步提纯蛋白。然而,离子交换层析只对PchE进行了浓缩,并没有改善非特异性条带的现象(图 3-A,泳道3和4)。体积排阻色谱图表明(图 3-B),PchE蛋白出峰位置约在11 mL处,呈现较好的单一对称峰形,而对应出峰体积洗脱液的电泳结果却显示,杂带依然未被消除(图 3-A,泳道5和6)。经过三步纯化策略均无法优化获得高纯度PchE蛋白,并且出现的杂带尺寸与目的蛋白大小接近,且条带大小有序递减,因此推测可能是由于PchE在大肠杆菌中表达不完整导致的断带现象,或者是PchE折叠不充分导致蛋白疏水结构域暴露,与其他杂蛋白粘连,所以无法通过多步纯化步骤去除。
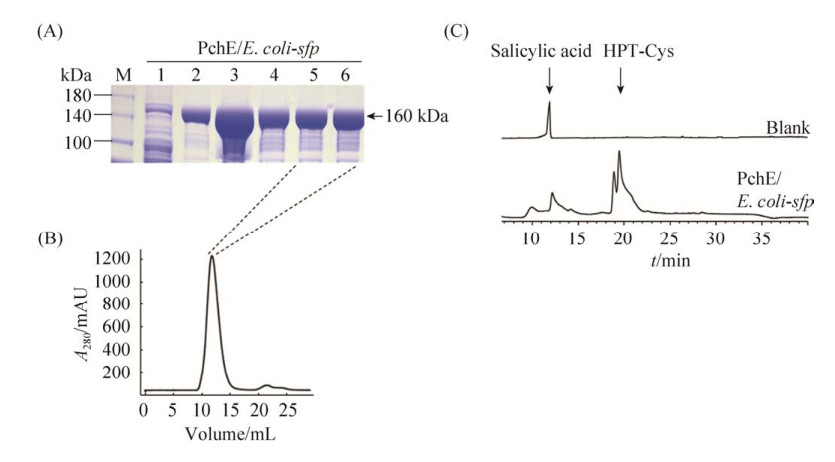 |
| 图 3 大肠杆菌表达PchE蛋白的纯化与活性验证 Figure 3 The purification and activity identification of PchE expressed by E. coli-sfp. A: SDS-PAGE of PchE purification at each step. Lane 1: Flowthrough of Ni-NTA affinity chromatography; lane 2: Elution of Ni-NTA affinity chromatography; lane 3, 4: Elution of Mono Q; lane 5, 6: Elution of size exclusion chromatography. B: Size exclusion chromatography of PchE with Superdex 200 10/300 column. C: In vitro biochemical identification of PchE using HPLC. |
| 图选项 |
在整合泛酰化酶Sfp的大肠杆菌中表达的PchE,证明可以被翻译后修饰,获得了有活性的蛋白,并在体外生化反应中催化水杨酸和半胱氨酸的环化脱水,生成中间产物HPT-Cys (图 3-C)。然而在HPT-Cys左侧存在另一个小峰,认为是受蛋白纯度和活性影响,生成了其他副产物。
2.3 酿酒酵母BJ5464-npgA表达纯化PchE与活性分析 将质粒pXW55-pchE-cH转化到酿酒酵母BJ5464-npgA中进行异源表达,并进行Ni-NTA亲和纯化。SDS-PAGE结果显示,酵母宿主表达的PchE蛋白并未出现上述非特异性条带(图 4-A,泳道4),仅经过一步纯化,蛋白纯度明显提高。通过离子交换层析后,PchE蛋白被浓缩,蛋白纯度很高(图 4-A,泳道5)。观察到酵母宿主表达的PchE蛋白纯度明显优于大肠杆菌宿主,推测是由于酵母细胞中转录和翻译系统更加成熟,能够帮助蛋白完整表达,同时酵母细胞具有丰富的酶协助翻译后蛋白的正确加工、修饰和折叠,从而形成正确的结构构象,在一定程度上有利于蛋白纯化。
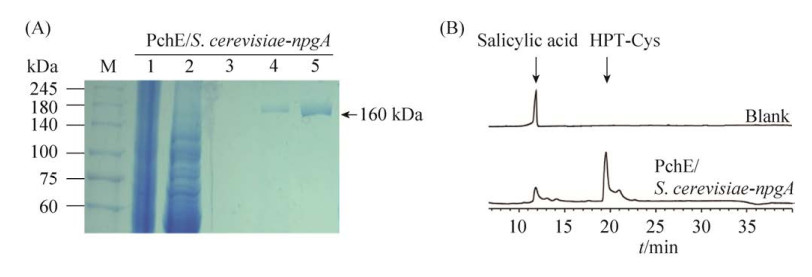 |
| 图 4 酿酒酵母-npgA表达PchE蛋白的纯化与活性验证 Figure 4 The purification and activity identification of PchE expressed by S. cerevisiae-npgA. A: SDS-PAGE of PchE purification at each step. Lane 1: Total protein; lane 2: Supernatant; lane 3: Wash of Ni-NTA; lane 4: Elution of Ni-NTA; lane 5: Elution of Mono Q. B: In vitro biochemical identification of PchE using HPLC. |
| 图选项 |
基于已经报道的研究,泛酰化酶NpgA具有修饰底物宽泛性,通过激活NRPSs参与赖氨酸、青霉素和铁载体的合成[24–25]。在体外重构生化体系中加入酿酒酵母-npgA表达的PchE蛋白,能够检测到产物HPT-Cys的生成(图 4-B),并且对比大肠杆菌表达的PchE,产物峰左侧的小峰消失,峰形更锐,推测酵母表达的PchE可能在催化中更具有专一性。在前人研究中,NpgA主要用于修饰真核蛋白,本实验表明在酵母宿主中,NpgA可以催化原核蛋白PchE的泛酰化,得到功能性PchE,证明NpgA具有修饰底物的宽泛性。
2.4 酿酒酵母BJ5464-npgA共表达PchE与Sfp的纯化与活性分析 为了确保PchE蛋白在酵母宿主中的功能性表达,本实验设计了PchE与细菌源PPTase Sfp的共表达质粒pXW55-pchEcH-sfp。将该质粒转化酿酒酵母后,进行蛋白表达与Ni-NTA亲和纯化。亲和层析和离子交换层析的SDS-PAGE结果都显示(图 5-A,泳道4和5),蛋白纯度相比大肠杆菌中有显著提高,非特异性蛋白条带消失,再次证实了酵母宿主表达巨型蛋白的优势。而相比于未引入泛酰化酶Sfp的情况,共表达条件下的PchE在两步纯化后得到的蛋白浓度有所提高,推测Sfp也在酵母宿主中发挥了一定作用。
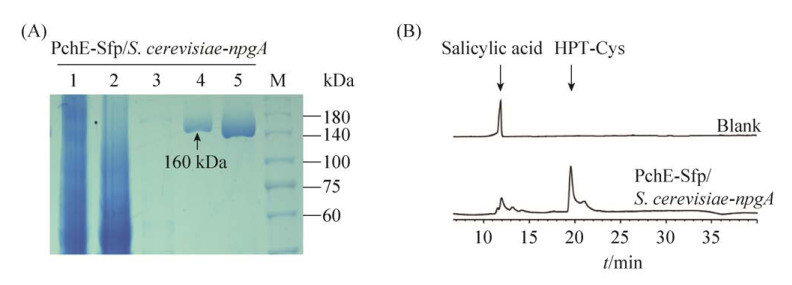 |
| 图 5 酿酒酵母-npgA表达PchE-Sfp蛋白的纯化与活性验证 Figure 5 The purification and activity identification of PchE-Sfp expressed by S. cerevisiae-npgA. A: SDS-PAGE of PchE purification at each step. Lane 1: Total protein; lane 2: Supernatant; lane 3: Wash of Ni-NTA; lane 4: Elution of Ni-NTA; lane 5: Elution of Mono Q. B: In vitro biochemical identification of PchE-Sfp using HPLC. |
| 图选项 |
在验证与Sfp共表达的PchE体外活性时发现,HPLC的结果与酵母宿主非共表达Sfp的情况非常类似。PchE作为铁载体合成过程中的延伸模块,合成了中间产物HPT-Cys (图 5-B),而在相同底物、酶浓度和相同反应时间的条件下,Sfp共表达的PchE对于底物的催化转化率并没有明显提高,推测在对PchE的泛酰化修饰作用中,NpgA与Sfp并没有显著的协同效应。这一结果也为后续将其他需要泛酰化修饰的原核蛋白单独构建到酵母宿主中表达提供了可行的方案。
3 讨论 在研究蛋白功能时,无论真核源还是原核源的蛋白,研究者一般首选大肠杆菌作为异源宿主进行表达,不乏很多成功案例[26]。然而,很多蛋白在大肠杆菌中异源表达时出现不表达、包涵体、表达量低、无活性等问题,往往会阻碍研究进展。所以,人们根据研究目的不同,尝试在更高等的宿主中实现高产、高纯度、高活性的目的蛋白表达。
前期,本课题组在研究巨型PKSs和NRPSs时,利用大肠杆菌作为异源表达宿主,多个蛋白都出现了如表达不完整、易沉淀、难纯化、无功能等问题。本研究以一个巨型NRPS的亚基PchE为研究对象,尝试在真核表达系统酿酒酵母细胞中表达PchE,成功获得了功能蛋白。在对比了大肠杆菌宿主和酿酒酵母宿主表达PchE的纯度后,证明酿酒酵母宿主表达的PchE纯度明显提高,并且消除了大肠杆菌表达产生的非特异性条带。据此推测,高等宿主可能比低等宿主更容易完整表达巨型蛋白,并且具备更丰富的翻译后修饰酶,有利于折叠形成稳定构象,这对于后续课题组开展巨型蛋白结构研究至关重要。除此以外,本研究选择的酿酒酵母表达质粒pXW55在发酵时,不存在外源抗生素的筛选压力,而是利用酵母自身代谢通路,在葡萄糖消耗完成后启动乙醇发酵途径,激活乙醇脱氢酶启动子,诱导目的蛋白过表达。这能大大减少大肠杆菌发酵过程带来的抗生素环境胁迫,是选择酵母表达宿主的另外一个优势。
同时,本研究首次论证了真菌源泛酰化酶NpgA对原核蛋白PchE的泛酰化底物位点具有宽泛的识别能力,并且修饰得到的蛋白可能具有更好的专一性。推测芽孢杆菌来源的泛酰化酶Sfp利用乙醇诱导型启动子ADH2,也可以在酿酒酵母宿主中进行有效的泛酰化修饰。但两种泛酰化酶同时存在于酵母细胞时,究竟是协同修饰或是选择性发挥作用,仍需要进一步探究。
总体而言,本研究为次生代谢领域的巨型PKSs和NRPSs在酵母表达体系中实现功能性表达提供了可行且有效的方法,也为下游进行蛋白结构和功能机理研究提供了有力支持。
References
| [1] | Pham PV. Medical biotechnology. Omics Technologies and Bio-Engineering. Amsterdam: Elsevier, 2018: 449-469. |
| [2] | Rosano GL, Ceccarelli EA. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in Microbiology, 2014, 5: 172. |
| [3] | Mattanovich D, Branduardi P, Dato L, Gasser B, Sauer M, Porro D. Recombinant protein production in yeasts. Recombinant Gene Expression. Totowa, NJ: Humana Press, 2011: 329-358. |
| [4] | Reuven NB, Koonin EV, Rudd KE, Deutscher MP. The gene for the longest known Escherichia coli protein is a member of helicase superfamily Ⅱ. Journal of Bacteriology, 1995, 177(19): 5393-5400. DOI:10.1128/jb.177.19.5393-5400.1995 |
| [5] | Nielsen J. Production of biopharmaceutical proteins by yeast: advances through metabolic engineering. Bioengineered, 2013, 4(4): 207-211. DOI:10.4161/bioe.22856 |
| [6] | Cacho RA, Tang Y. Reconstitution of fungal nonribosomal peptide synthetases in yeast and in vitro. Methods in Molecular Biology. New York, NY: Springer New York, 2016: 103-119. |
| [7] | Youard ZA, Wenner N, Reimmann C. Iron acquisition with the natural siderophore enantiomers pyochelin and enantio-pyochelin in Pseudomonas species. BioMetals, 2011, 24(3): 513-522. DOI:10.1007/s10534-010-9399-9 |
| [8] | Hood MI, Skaar EP. Nutritional immunity: transition metals at the pathogen-host interface. Nature Reviews Microbiology, 2012, 10(8): 525-537. DOI:10.1038/nrmicro2836 |
| [9] | Cox CD, Rinehart KL, Moore ML, Cook JC. Pyochelin: novel structure of an iron-chelating growth promoter for Pseudomonas aeruginosa. Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(7): 4256-4260. DOI:10.1073/pnas.78.7.4256 |
| [10] | Meyer JM, Hornsperger JM. Role of PyoverdinePf, the iron-binding fluorescent pigment of Pseudomonas fluorescens, in iron transport. Journal of General Microbiology, 1978, 107(2): 329-331. DOI:10.1099/00221287-107-2-329 |
| [11] | Braud A, Geoffroy V, Hoegy F, Mislin GL A, Schalk IJ. Presence of the siderophores pyoverdine and pyochelin in the extracellular medium reduces toxic metal accumulation in Pseudomonas aeruginosa and increases bacterial metal tolerance. Environmental Microbiology Reports, 2010, 2(3): 419-425. DOI:10.1111/j.1758-2229.2009.00126.x |
| [12] | Takase H, Nitanai H, Hoshino K, Otani T. Impact of siderophore production on Pseudomonas aeruginosa infections in immunosuppressed mice. Infection and Immunity, 2000, 68(4): 1834-1839. DOI:10.1128/IAI.68.4.1834-1839.2000 |
| [13] | Reimmann C, Serino L, Beyeler M, Haa D. Dihydroaeruginoic acid synthetase and pyochelin synthetase, products of the pchEF genes, are induced by extracellular pyochelin in Pseudomonas aeruginosa. Microbiology: Reading, England, 1998, 144(Pt 11): 3135-3148. |
| [14] | Quadri LEN, Keating TA, Patel HM, Walsh CT. Assembly of the Pseudomonas aeruginosa nonribosomal peptide siderophore pyochelin: in vitro reconstitution of aryl-4, 2-bisthiazoline synthetase activity from PchD, PchE, and PchF. Biochemistry, 1999, 38(45): 14941-14954. DOI:10.1021/bi991787c |
| [15] | Patel HM, Walsh CT. In vitro reconstitution of the Pseudomonas aeruginosa nonribosomal peptide synthesis of pyochelin: ? characterization of backbone tailoring thiazoline reductase and N-methyltransferase activities. Biochemistry, 2001, 40(30): 9023-9031. DOI:10.1021/bi010519n |
| [16] | Patel HM, Tao JH, Walsh CT. Epimerization of an l-cysteinyl to a d-cysteinyl residue during thiazoline ring formation in siderophore chain elongation by pyochelin synthetase from Pseudomonas aeruginosa. Biochemistry, 2003, 42(35): 10514-10527. DOI:10.1021/bi034840c |
| [17] | Cunrath O, Gasser V, Hoegy F, Reimmann C, Guillon L, Schalk IJ. A cell biological view of the siderophore pyochelin iron uptake pathway in Pseudomonas aeruginosa. Environmental Microbiology, 2015, 17(1): 171-185. DOI:10.1111/1462-2920.12544 |
| [18] | Ronnebaum TA, Lamb AL. Nonribosomal peptides for iron acquisition: pyochelin biosynthesis as a case study. Current Opinion in Structural Biology, 2018, 53: 1-11. DOI:10.1016/j.sbi.2018.01.015 |
| [19] | Beld J, Sonnenschein EC, Vickery CR, Noel JP, Burkart MD. The phosphopantetheinyl transferases: catalysis of a post-translational modification crucial for life. Natural Product Reports, 2014, 31(1): 61-108. DOI:10.1039/C3NP70054B |
| [20] | Nakano MM, Corbell N, Besson J, Zuber P. Isolation and characterization of sfp: a gene that functions in the production of the lipopeptide biosurfactant, surfactin, in Bacillus subtilis. Molecular and General Genetics MGG, 1992, 232(2): 313-321. DOI:10.1007/BF00280011 |
| [21] | Lambalot RH, Gehring AM, Flugel RS, Zuber P, LaCelle M, Marahiel MA, Reid R, Khosla C, Walsh CT. A new enzyme superfamily—the phosphopantetheinyl transferases. Chemistry & Biology, 1996, 3(11): 923-936. |
| [22] | Márquez-Fernández O, Trigos A, Ramos-Balderas JL, Viniegra-González G, Deising HB, Aguirre J. Phosphopantetheinyl transferase CfwA/NpgA is required for Aspergillus nidulans secondary metabolism and asexual development. Eukaryotic Cell, 2007, 6(4): 710-720. DOI:10.1128/EC.00362-06 |
| [23] | Mootz HD, Sch?rgendorfer K, Marahiel MA. Functional characterization of 4'-phosphopantetheinyl transferase genes of bacterial and fungal origin by complementation of Saccharomyces cerevisiae Lys5. FEMS Microbiology Letters, 2002, 213(1): 51-57. |
| [24] | Oberegger H, Eisendle M, Schrettl M, Graessle S, Haas H. 4'-Phosphopantetheinyl transferase-encoding npgA is essential for siderophore biosynthesis in Aspergillus nidulans. Current Genetics, 2003, 44(4): 211-215. DOI:10.1007/s00294-003-0434-z |
| [25] | Kim JM, Song HY, Choi HJ, So KK, Kim DH, Chae KS, Han DM, Jahng KY. Characterization of NpgA, a 4'-phosphopantetheinyl transferase of Aspergillus nidulans, and evidence of its involvement in fungal growth and formation of conidia and cleistothecia for development. Journal of Microbiology, 2015, 53(1): 21-31. DOI:10.1007/s12275-015-4657-8 |
| [26] | Kaur J, Kumar A, Kaur J. Strategies for optimization of heterologous protein expression in E. coli: Roadblocks and reinforcements. International Journal of Biological Macromolecules, 2018, 106: 803-822. DOI:10.1016/j.ijbiomac.2017.08.080 |
