陈加臻1,2,3#, 农添植1,2,3#, 邢方婷1,2,3, 汤华妹1,2,3, 杨文霄1,2,3, 张丽珊1,2,3, 林向民1,2,3

 , 谢小芳1,2,3
, 谢小芳1,2,3

1. 福建农林大学福建省农业生态过程与安全监控重点实验室, 福建 福州 350002;
2. 福建农林大学作物生态与分子生理重点实验室, 福建 福州 350002;
3. 福建农林大学生命科学学院, 福建 福州 350002
收稿日期:2021-03-15;修回日期:2021-06-02;网络出版日期:2021-07-27
基金项目:福建农林大学本科生科研训练项目(111ZC0020);国家自然科学基金(32171435)
*通信作者:林向民, Tel: +86-591-83789494;E-mail: xiangmin@fafu.edu.cn;
谢小芳, E-mail: xxf317@fafu.edu.cn.
#并列第一作者。
摘要:[目的] NtrC是一种与DNA结合的转录调控因子,在激活氮同化基因的转录和维持氮源供应中具有重要作用,本研究拟探究其对嗜水气单胞菌生理功能的影响及其作用机理。[方法] 本研究采用同源重组方法构建了嗜水气单胞菌ATCC 7966 ntrC的缺失株,并以野生株为对照,对缺失株的生理表型进行测定和分析,利用定量蛋白质组学技术比较野生株和ntrC缺失株的蛋白表达差异。[结果] 发现敲除ntrC基因后,嗜水气单胞菌在缺氮、渗透压、重金属离子、氧化以及不同抗生素胁迫下的耐受性都发生显著变化,且这些表型在其补救菌株中均能得到恢复。定量蛋白质组学分析发现,野生株和ntrC缺失株的差异表达蛋白可能参与氨基酸生物合成、抗坏血酸和醛糖酸盐等代谢通路的调控。[结论] 本研究阐明了ntrC在嗜水气单胞菌中的重要作用及其对细菌生物学功能的影响,探讨了ntrC直接或间接调控的蛋白与生理表型之间的联系,研究结果可为未来水产致病菌的防治提供理论支持。
关键词:嗜水气单胞菌转录调控因子NtrC同源重组
Physiological function and mechanism mediated by ntrC in Aeromonas hydrophila
Jiazhen Chen1,2,3#, Tianzhi Nong1,2,3#, Fangting Xing1,2,3, Huamei Tang1,2,3, Wenxiao Yang1,2,3, Lishan Zhang1,2,3, Xiangmin Lin1,2,3

 , Xiaofang Xie1,2,3
, Xiaofang Xie1,2,3

1. Fujian Provincial Key Laboratory of Agroecological Processing and Safety Monitoring, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
2. Key Laboratory of Crop Ecology and Molecular Physiology, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
3. School of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China
Received: 15 March 2021; Revised: 2 June 2021; Published online: 27 July 2021
*Corresponding author: Xiangmin Lin, Tel: +86-591-83789494;E-mail: xiangmin@fafu.edu.cn;
Xiaofang Xie, E-mail: xxf317@fafu.edu.cn.
Foundation item: Supported by the Program for Undergrduate Scientific Research Training (111ZC0020) and by the National Natrual Science Foundation of China (32171435)
# These authors contributed equally to this work.
Abstract: [Objective] NtrC is a DNA-binding transcriptional regulator which plays an important role in activating the transcription of nitrogen assimilation genes and maintaining nitrogen supply. The objectives of this study were to explore its physiological function and mechanism in Aeromonas hydrophila. [Methods] In this study, the homologous recombination method was used to construct the ntrC deletion strain of Aeromonas hydrophila, and the physiological phenotype of the deletion strain was measured and analyzed with the wild strain as the control. A data independent acquisition-based quantitative proteomics method was performed to compare the protein expression differences between the wild-type and ntrC deletion strains. [Results] After the ntrC gene was knocked out, the tolerance of Aeromonas hydrophila to nitrogen deficiency, osmotic pressure, heavy metal ions, oxidation and different antibiotic stress was significantly changed, and these phenotypes could be recovered in its rescued strains. Quantitative proteomics was used to compare the protein expression between the WT and the ntrC deletion strain. [Results] showed that ntrC may be involved in the regulation of biosynthesis of amino acids, ascorbate and aldose metabolism pathways. [Conclusion] This study elucidated the important role of NtrC in Aeromonas hydrophila and its impact on the biological function of the bacteria. Moreover, the relationship between the proteins directly or indirectly regulated by NtrC and the physiological phenotype was discussed, the results provide valuable information for the prevention and control of aquatic pathogens in the future.
Keywords: Aeromonas hydrophilatranscriptional regulatory factorsNtrChomologous recombination
微生物对生存环境的变化十分敏感,渗透压胁迫、重金属离子胁迫、氧化胁迫、营养缺乏等都会不同程度地影响微生物的生长繁殖。为了适应复杂多变的动态环境,微生物进化出了多种以双组分调控系统(two-component regulatory system,TCS)为主的应答机制,其中组氨酸激酶感受外界环境变化,感应调控蛋白负责调控功能基因的转录表达,从而使细菌能够迅速适应环境的变化[1]。氮素是自然界环境中分布最广泛的营养元素之一[2],是构成蛋白质、核酸等许多重要化合物的重要组成部分,也是细胞生长不可或缺的一部分,对微生物的生长繁殖起到举足轻重的作用。为了适应环境中氮含量的变化,微生物进化出了复杂的一般氮代谢调控(general nitrogen regulation,Ntr)系统,Ntr系统主要由4种蛋白构成:GlnD、NtrB、NtrC和PII。NtrB和NtrC组成一个双组分调控系统,组氨酸激酶NtrB感受外界环境变化,催化调控蛋白NtrC磷酸化或去磷酸化[3-4]。GlnD调控PII的活性,PII与NtrB互作共同调节NtrC的活性。
NtrC是一种可以结合DNA的转录调控因子。编码NtrC的ntrC基因又称glnG[4-5]。在缺氮条件下,ntrC会被强烈诱导并大量表达,而被NtrB磷酸化的NtrC (NtrC-P)具有转录激活活性[6],可以与σ54(NtrA)因子结合并激活各种σ54依赖型启动子的转录[7],进而提高氮同化基因的表达水平,保证自身的氮源供应。但也有研究发现,NtrC不仅可以作为转录激活因子,在面对不同启动子时也可以作为抑制因子,如ntrBC启动子和glnA启动子等[5]。此外,NtrC还可以参与生物降解、逆境胁迫、生物膜合成等多个生物学过程[8],这表明NtrC可能是一种全局调控因子,对细菌整个机体的生命活动都起着非常重要的调控作用。
嗜水气单胞菌(Aeromonas hydrophila)是一种异养型革兰氏阴性细菌,广泛分布于自然界,在各种水体中均可生长[9]。该菌主要通过感染淡水鱼生长繁殖,引发出血性败血症,对水产养殖业造成巨大经济损失[10],同时人类和其他动物被感染后可引发肠胃炎,严重可导致败血症等其他疾病,对人类的生命健康产生威胁[11]。为了研究嗜水气单胞菌转录调控因子NtrC在细菌中的生理功能及其参与的调控机制,本研究利用同源重组的方法构建ntrC敲除菌株及构建补救株?ntrC: : ntrC,以野生株WT (wild type,WT)和空载菌株?ntrC: : vector为对照,对其生理功能进行测定,利用定量蛋白质组学技术比较野生株和ntrC缺失株的蛋白表达差异,结合生物信息学和生理表型分析该转录调控因子参与调控的细菌生理功能和代谢途径,以期为完善嗜水气单胞菌的生理机制提供理论基础。
1 材料和方法 1.1 实验材料 本研究使用的嗜水气单胞菌ATCC 7966 (WT)、大肠杆菌(E. coli MC1061-λpir,E. coli S17-λpir)及质粒(pRE112)均保存在本实验室中。LB (Luria-Bertani,LB)培养基使用的酵母粉和胰蛋白胨购于北京拜尔迪生物技术有限公司,氯化钠购于国药集团化学试剂公司,限制性内切酶、连接酶、Taq酶(Thermus Aquaticus,Taq)均购于南京诺唯赞生物科技有限公司(Vazyme)。抗生素浓度分别为氨苄青霉素(Ampicillin,Amp) 100 μg/mL,氯霉素(Chloramphenicol,Chl) 30 μg/mL,均购买于上海翊圣生物科技有限公司(YEASEN)。质粒提取试剂盒、胶回收试剂盒、总DNA提取试剂盒均购买于美基美生物(Magen)公司。
1.2 缺失菌株及补救株的构建 利用同源重组的方法,在ntrC基因上下游各500 bp长度的序列分别设计P1/P2、P3/P4引物,P5/P6分别设计在ntrC基因上下游,P7/P8分别设计于P1上游100 bp、P4下游100 bp处(表 1)。以嗜水气单胞菌ATCC 7966的基因组DNA为模板,利用P1/P2、P3/P4引物进行扩增并连接至pRE112质粒上(酶切位点Xba I和Sac I),构建ntrC的基因敲除质粒,继而转化到E. coli MC1061-λpir感受态细胞中,用引物P1/P4进行菌液PCR验证。提取验证正确的阳性克隆质粒,转入E. coli S17-λpir感受态细胞中,再经菌液PCR验证正确后,将携带pRE112载体的E. coli S17-λpir与野生菌株(WT)按4:1接合,进行第一次同源重组。经100 μg/mL Amp和30 μg/mL Chl抗性筛选,获得第1次同源重组菌株。随后在富含20%蔗糖的LB培养基中进行第2次同源重组,经过Chl抗性平板筛选、菌液PCR和DNA测序确认嗜水气单胞菌ntrC基因缺失,通过稳定遗传,将测序正确的缺失菌株保存在-80 ℃中。
表 1. ntrC基因敲除与回复株构建引物列表 Table 1. Primers for the construction of ntrC deletion strain and rescued strain
| Primer sequence (5′→3′) | Purpose |
| P1: CGATCCCAAGCTTCTTCTAGACACCGGCATCATCATCGAA | Gene deletion |
| P2: CAAGGACGACGTTCCCTTATTTGCGAAGC | Gene deletion |
| P3: ATAAGGGAACGTCGTCCTTGGGAAAAACGATTCC | Gene deletion |
| P4: CATGAATTCCCGGGAGAGCTCATGATGGCTTCGACCACGC | Gene deletion |
| P5: GCCATGGCTGATATCGGATCCATGACAGCAAAAGTGTGGATCG | Gene deletion |
| P6: CTCGAGTGCGGCCGCAAGCTTTCAGGAGAGGTCGAGTTCTTTCA | Gene deletion |
| P7: GGATCTTCCAGAGATGGCCTGGCCCACGAGATC | Gene deletion |
| P8: CTGCCGTTCGACGATGTCGCCGGCCAGATCGAT | Gene deletion |
| ntrC-F: GTCGACGGTATCGATAAGCTTGGATCATCGACAACGGCCCC | Complementary |
| ntrC-R: CGCTCTAGAACTAGTGGATCCCTAATGGTGATGGTGATGATGGGAGAGG | Complementary |
| TCGAGTTCTTTCAGCT | |
| F: GCGCGCAATTAACCCTCACT | Complementary |
| R: GGCGGCCGCTCTAGAACTAG | Complementary |
表选项
利用CE Design V1.03软件设计嗜水气单胞菌ntrC基因的克隆引物,以pBBR1-MCS1质粒为载体(酶切位点为Hind III和BamH I),导入目的基因启动子序列(确保ntrC基因正常表达)和目的基因全序列,并在终止子前加入6×His tag (ATGGTGATGGTGATGATG),利用同源重组的方法生成引物(命名为ntrC-F和ntrC-R,如表 1),并进行PCR扩增及片段回收。将载体酶切回收后与目的片段连接后转化到DH5α感受态细胞中。第二天在平板中挑取单克隆进行菌液PCR验证,再提质粒进行测序验证。将验证结果完全正确的质粒利用电转化法转化到ΔntrC感受态细胞中。培养2 d后挑单克隆,进行PCR验证并测序比对。质粒成功转入ΔntrC缺失菌株中(pBBR1-MCS1通用引物:F和R,表 1),获得回复和空载菌株。
1.3 胁迫耐受性检测 对ntrC基因敲除株在缺氮、高盐和氧化、重金属离子、氧化以及不同抗生素胁迫下的耐受性进行研究。将铵盐作为细菌主要氮源,利用铵缺乏(含3 mmol/L NH4Cl)的Gutnick培养基进行缺氮胁迫检测;利用4%的NaCl和H2O2进行高盐(高渗透压)胁迫检测和氧化胁迫检测;利用Mn2+、Co2+等金属离子胁迫检测,即在配制含有1.5%琼脂粉的M9培养基(不添加MgSO4·7H2O和CaCl2)中添加适当浓度的KMnO4、CoCl2,制成含不同金属离子浓度的平板;利用琼脂稀释法测定最低杀菌浓度(minimum bactericidal concentrations,MBCs),即将抗生素母液进行2倍梯度稀释后加入到融化好的LB固体培养基中,制成含不同浓度抗生素的平板。分别将培养好的菌液样品以10-2-10-7进行梯度稀释,各取2 μL点板,30 ℃培养箱中倒放培养16 h,后取出拍照并记录结果。
1.4 蛋白样品制备 将过夜培养16 h的菌株以1%转接到30 mL LB培养基中,在30 ℃摇床中培养,直至OD600=1.0,5000×g离心10 min,用PBS缓冲液洗涤2次并加入1 mL的Lysis buffer混匀,超声破碎(功率为30%,工作9 s,暂停6 s)后,以18000×g离心15 min取上清,取上清到新的EP管中,并利用BCA (bicinchoninic acid,BCA)法测定蛋白质浓度[12]。
1.5 胰蛋白酶消化和LC-MS/MS鉴定 采用FASP(filter-aided sample preparation)法进行蛋白样品的酶解[13]。并利用C18除盐小柱进行除盐。通过数据依赖性采集模式(data-dependent acquisition,DDA)进行建库,利用的是LC-20AB HPLC Pump system (Shimadzu,Kyoto,Japan)色谱仪。再用反相液质联用(RPLC-MS)进行样品分析。质谱采集到的DDA原始数据导入到Spectronout Pulsar X (Biognosys,Schlieren,Switzerland)建立DDA谱图库,默认最优参数“BGS factory setting”进行建库。然后将DIA(data independent acquisition,DIA)原始数据导入到Spectronout Pulsar X进行蛋白质定性定量分析,搜索数据库设定为嗜水气单胞菌ATCC 7966。
1.6 生物信息学分析 根据质谱鉴定结果,选取错误发生率(false discovery rate,FDR) < 1%、肽段匹配数≥2以及P-value < 0.05的蛋白进行生物信息学分析,蛋白差异比值大于2倍为差异表达蛋白。借助David (https://david.ncifcrf.gov/)在线软件对差异蛋白进行KEGG富集分析,利用R studio软件进行可视化分析[14]。
1.7 免疫印迹验证 全菌蛋白样品通过SDS-PAGE分离后,利用半干转仪将蛋白转移至PVDF膜上,在添加了5% (W/V)脱脂牛奶的PBST溶液中室温封闭1 h后,先后加入一抗(1:1000)和二抗(1:5000),分别在室温中孵育1 h,在每次孵育结束后用PBST洗涤5次,将PVDF膜进行ECL曝光显色。
2 结果和分析 2.1 基因缺失突变菌株的构建 利用表 1中用于基因敲除的P1/P2、P3/P4引物,以野生型嗜水气单胞菌为模板,根据同源重组原理构建ntrC基因敲除突变株。以野生型嗜水气单胞菌为阳性对照,分别对敲除株和野生株全基因组DNA进行PCR扩增验证,结果如图 1-A所示:以P5/P6为引物进行PCR验证,发现敲除株没有扩增出任何产物(泳道1),而野生株在1000-2000 bp处有目的条带(泳道2),表明敲除株基因组中不含目的基因;以P7/P8为引物扩增,发现野生株(泳道4)和敲除株(泳道3) PCR产物片段长度差值约为1419 bp,与预计值相符。DNA测序结果与预期相符。以上结果表明成功敲除ntrC。以野生株基因组DNA为模板扩增回复片段,并构建回复载体。将回复质粒和pBBR1-MCS1分别电转入ΔntrC感受态细胞中,菌液PCR验证结果如图 1-B所示,扩增出的条带长度差值约为1500 bp的片段(泳道1-2),与预期相符,表明回复和空载菌株构建成功。
 |
| 图 1 ntrC敲除菌株和补救株的构建 Figure 1 Construction of ntrC gene deletion and rescued strains. A: validation of gene deletion. M: 2000 bp DNA marker; lane 1: ntrC-P5P6; lane 2: WT control (1419 bp); lane 3: ntrC-P7P8 (1200 bp); lane 4: WT control (2619 bp). B: validation of gene complementary. M: 2000 bp DNA marker; lane 1: reverting strain (1686 bp); lane 2: empty carrier (110 bp). |
| 图选项 |
2.2 ΔntrC的胁迫耐受性 本研究采用梯度稀释点板法测定了ntrC缺失、回复各系列菌株在缺氮胁迫下的生长状况。结果表明(图 2-A),ΔntrC应对缺氮胁迫的耐受性显著下降,对缺失菌进行补救后耐受性又得到了回复,说明ntrC参与调控细菌响应缺氮胁迫的耐受过程。ΔntrC在高渗透压、过氧化物等胁迫条件下的应激能力的测定结果表明(图 2-A),与野生型相比,ΔntrC显著耐受高盐胁迫,对H2O2更敏感,其补救菌株的功能得到恢复,表明ntrC参与调控细菌响应高渗透压与氧化胁迫环境。通过琼脂平板梯度稀释的方法测定重金属离子的耐受性,结果发现(图 2-B) ntrC缺失后显著增强了细菌对Co2+和Mn2+的耐受性,提示在嗜水气单胞菌中ntrC可能也参与金属离子胁迫相关基因的调控。以野生型嗜水气单胞菌(WT)为对照,研究嗜水气单胞菌野生型和缺失菌株对33种抗生素的MBCs,根据抗生素的类型对ΔntrC、ΔntrC: : ntrC、ΔntrC: : vector的MBCs进行汇总(图 2-C)。结果显示,ΔntrC对妥布霉素(TOB)、卡那霉素(KAN)、庆大霉素(GEN)、新霉素(NEO)、阿米卡星(AMK)、左氧氟沙星(LVX)、恩诺沙星(ENR)和利福平(RIF)等8种抗生素的MBCs升高了2倍,对莫西沙星(MFX)的MBC升高了4倍,这说明ntrC对细菌耐药的调控作用较为明显。
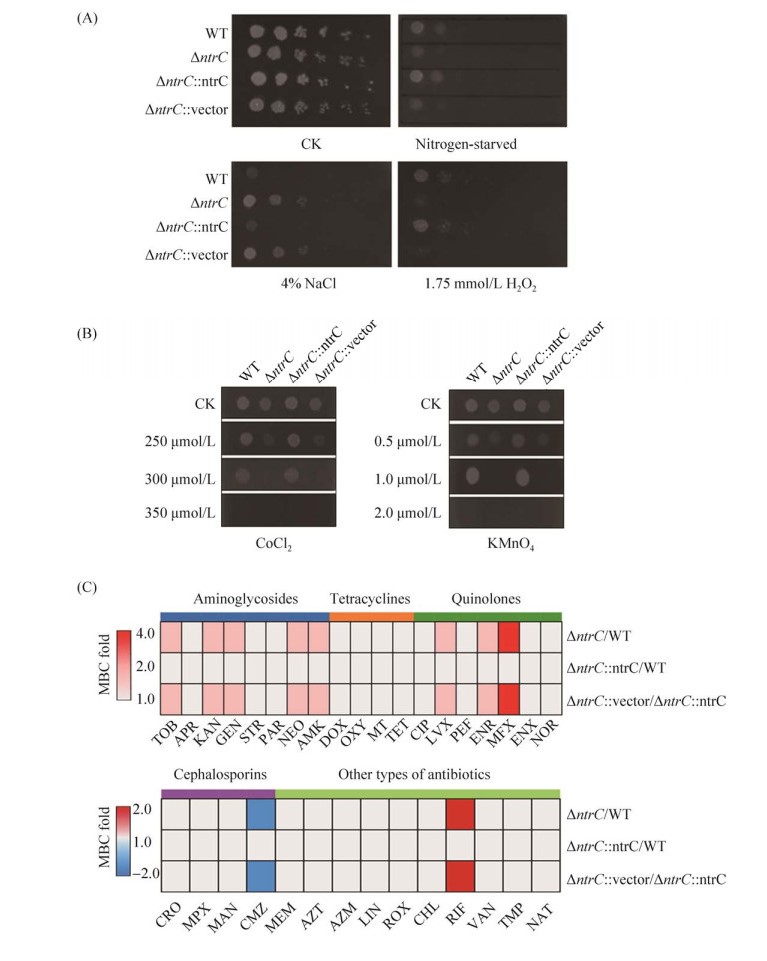 |
| 图 2 野生型WT与ΔntrC敲除菌及ΔntrC: : ntrC补救株与ΔntrC: : vector的生理表型 Figure 2 Physiological phenotypes of WT, ΔntrC, ΔntrC: : ntrC and ΔntrC: : vector strains. A: the tolerance assays under the conditions of nitrogen deficiency, high osmotic pressure and peroxide stress; B: the tolerance assays for heavy metal stress; C: antibiotic resistance assays. |
| 图选项 |
2.3 定量蛋白质组学比较分析 为了进一步研究ntrC对嗜水气单胞菌蛋白表达丰度的影响,本论文提取ntrC基因缺失株和野生株的全蛋白,经胰蛋白酶消化成多肽后进行质谱检测,共鉴定到2655个蛋白。两组样本各3次生物学重复的相关性系数R均大于0.98,说明蛋白质组学数据的重复性是可靠的(图 3-A)。ntrC基因缺失株和对照野生株的差异表达蛋白分析共鉴定到165个差异蛋白,其中105个蛋白表达上调,60个蛋白表达下调(图 3-B),部分差异蛋白(金属离子、氮调节白和氧化相关蛋白)如表 2所示。
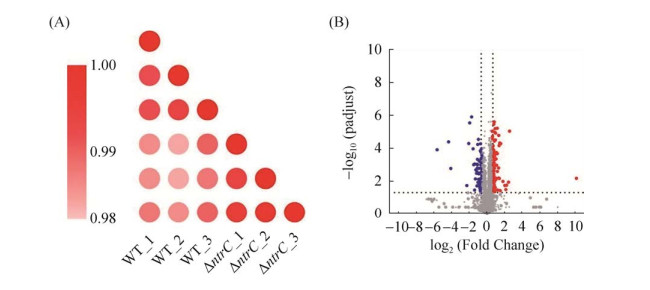 |
| 图 3 定量蛋白质组学数据分析 Figure 3 Quantitative proteomics data analysis. A: heat map of the correlation of protein intensities in three biological repeats; B: volcano map of significantly differentially expressed proteins. |
| 图选项 |
表 2. ΔntrC与野生株的部分差异表达蛋白 Table 2. Selected differential expression proteins between ΔntrC and WT
| Accession | Gene | Description | Matched peptides | P-value | log2(ΔntrC/WT) |
| A0KEY7 | glnL | Nitrogen regulation protein NR(II) | 4 | 9.12E-06 | 2.559065182 |
| A0KQX0 | glmS | Glutamine-fructose-6-phosphate aminotransferase [isomerizing] | 6 | 6.84E-05 | 1.370704359 |
| A0KGI2 | rpoS | RNA polymerase sigma factor RpoS | 4 | 1.56E-03 | 1.305425624 |
| A0KMR3 | AHA_3062 | Formate dehydrogenase iron-sulfur subunit | 4 | 6.09E-06 | 1.1632809 |
| A0KMR7 | AHA_3066 | Iron-sulfur cluster-binding protein | 5 | 3.15E-05 | 0.959377859 |
| A0KG17 | AHA_0660 | Sodium-type flagellar motor component | 4 | 6.62E-03 | 0.847340836 |
| A0KR04 | trpS-1 | Tryptophanyl-tRNA synthetase | 4 | 2.48E-06 | 0.820110187 |
| A0KM11 | AHA_2807 | Homocysteine S-methyltransferase | 4 | 9.71E-03 | 0.811892365 |
| A0KGK1 | AHA_0850 | Ribosomal-protein-serine acetyltransferase | 3 | 2.39E-02 | 0.801914481 |
| A0KMJ0 | AHA_2987 | Probable manganese-dependent inorganic pyrophosphatase | 3 | 3.66E-03 | 0.780842967 |
| A0KEA8 | AHA_0009 | Valine--pyruvate aminotransferase | 4 | 2.68E-04 | 0.707139687 |
| A0KHE9 | AHA_1159 | Zn-ribbon-containing protein | 5 | 8.61E-04 | 0.677451877 |
| A0KQA1 | AHA_4024 | Intracellular sulfur oxidation protein DsrE | 4 | 4.17E-04 | 0.635812406 |
| A0KG92 | xenB | Xenobiotic reductase B | 6 | 8.71E-05 | 0.59936223 |
| A0KQW7 | AHA_4251 | TonB system biopolymer transport component | 3 | 1.32E-03 | -0.597836752 |
| A0KJ51 | purL | Phosphoribosylformylglycinamidine synthase | 4 | 3.73E-04 | -0.607548407 |
| A0KLS3 | AHA_2716 | Spermidine N(1)-acetyltransferase | 6 | 1.74E-03 | -0.652599908 |
| A0KK14 | AHA_2085 | Arginine/ornithine antiporter | 1 | 9.84E-03 | -0.653426854 |
| A0KNS2 | AHA_3434 | TonB system biopolymer transport component | 1 | 1.61E-04 | -0.710384704 |
| A0KML1 | AHA_3008 | Extradiol ring-cleavage dioxygenase, class III enzyme, subunit B | 4 | 1.05E-02 | -0.878188549 |
| A0KME3 | AHA_2940 | Secretory tripeptidyl aminopeptidase, putative | 4 | 1.73E-02 | -0.937290305 |
| A0KFD8 | AHA_0431 | NhaP-type Na+/H+ and K+/H+ antiporters | 3 | 3.53E-03 | -0.938167566 |
| A0KHR1 | agcS-1 | Amino acid carrier protein | 2 | 3.30E-03 | -1.179282181 |
| A0KJC5 | AHA_1842 | Sigma-54 dependent transcriptional regulator | 5 | 1.25E-06 | -1.74371806 |
表选项
2.4 差异蛋白KEGG注释分析 利用DAVID在线网站将差异表达蛋白进行KEGG富集分析,通过R语言数据包GOplot进行可视化分析。结果如图 4所示,经P-value < 0.05的显著性标准筛选,发现差异蛋白富集到氨基酸生物合成、组氨酸代谢、代谢途径、次生代谢物的生物合成、抗坏血酸和醛糖酸盐代谢通路等5个代谢通路中。该结果表明差异蛋白主要涉及代谢途径和次生代谢物的生物合成通路。除抗坏血酸和醛糖酸盐代谢通路蛋白以及少数蛋白下调外,大部分相关蛋白都是上调表达。
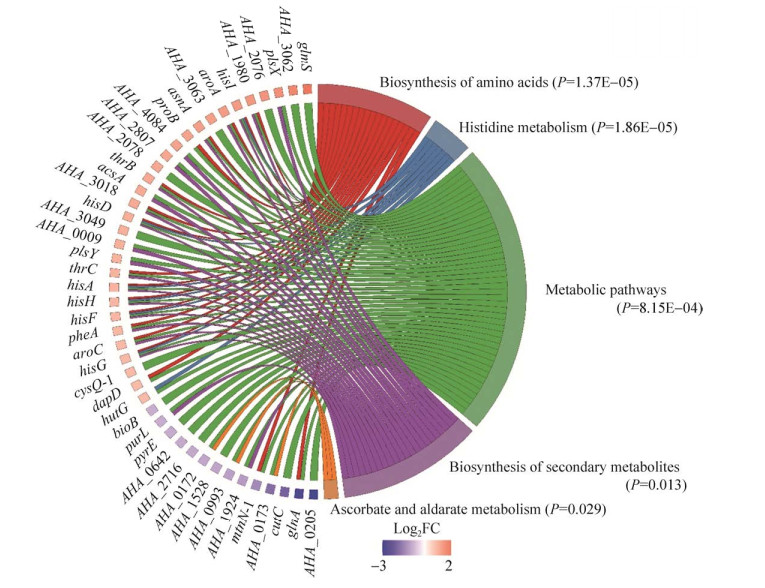 |
| 图 4 ΔntrC差异表达蛋白KEGG代谢通路富集分析 Figure 4 KEGG enrichment analysis of the differentially expressed proteins between ΔntrC and WT strains. |
| 图选项 |
2.5 Western blotting验证 为验证定量蛋白质组学数据的可靠性,利用实验室已有的3个差异蛋白特异性抗体进行Western blotting验证,结果如图 5所示。与WT比较,ΔntrC中A0KH89、A0KHA8蛋白表达减少,P55870蛋白表达增加。该验证结果与定量蛋白质组学的结果基本一致,证明质谱数据的可靠性。
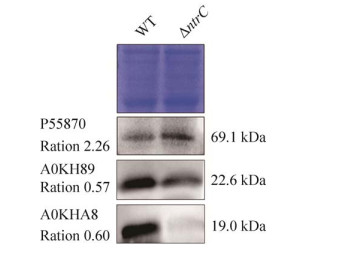 |
| 图 5 ntrC基因缺失株与野生株中3个差异表达蛋白的Western blotting比较结果 Figure 5 Western blotting of three differentially expressed proteins between ΔntrC and WT strains. Ratio represents the difference multiple of differentially expressed proteins between ΔntrC and WT strains. |
| 图选项 |
3 讨论 原核生物的转录调控因子以各种方式参与并调控细菌的各种代谢过程,从而适应复杂的外界环境[15-16]。以往的一些研究也说明NtrC在调控许多细菌的多种生理功能方面都发挥着重要作用。在巴西固氮螺菌(Azospirillum brasilense)中,Ntr调控系统控制着氮代谢,其中的NtrC有助于适应高氮和低氮环境。磷酸化的NtrC是参与替代氮源代谢基因的转录激活因子,可以激活谷胱甘肽S-转移酶,催化亲电化合物结合成还原型谷胱甘肽(glutathione),也可以促进有机抗过氧化氢蛋白Ohr的表达,Ohr属于OsmC超家族,对有机过氧化氢有解毒作用。这些都表明NtrC有利于适应由缺氮引起的氧化胁迫[3]。在新洋葱伯克霍尔德菌(Burkholderia cenocepacia)中,NtrC是缺氮条件下的主要调控因子,是σ54依赖性基因转录的激活剂,可以调控胞外聚合物(extracellular polymeric substances,EPS)的产生,对NtrC调控子的研究表明,有两组基因(bceI和bceII)参与cepacian的生产,cepacian是B. cenocepacia中主要的EPS。但该研究表明,在B. cenocepacia中,NtrC和抗生素耐药性和抗氧化能力关系不显著[17]。在霍乱弧菌(Vibrio cholerae)中,NtrC还负调控生物膜的形成[18]。
本研究采用同源重组方法构建了嗜水气单胞菌ntrC的缺失株,检测和分析了ntrC缺失株的生理表型,并进一步利用DIA定量蛋白质组学技术对差异蛋白进行比较分析。研究发现,敲除ntrC基因后,嗜水气单胞菌对缺氮胁迫的耐受性显著下降,说明嗜水气单胞菌中ntrC也参与调控细菌响应缺氮胁迫的耐受过程,这与以往的研究结果相一致[19-21]。此外,研究还发现缺失株在渗透压胁迫、重金属离子胁迫、氧化胁迫、不同抗生素胁迫等条件下的耐受性发生变化,表明嗜水气单胞菌中的ntrC除了参与氮调节外,可能还会影响细菌其他的生理功能。通过进一步差异蛋白鉴定以及KEGG代谢通路分析,发现18个氨基酸生物合成通路的相关蛋白(例如A0KJR0、A0KMM1等)表达上调或下调、3个抗坏血酸和醛糖酸盐代谢通路相关蛋白(A0KEP0、A0KGY9、A0KEP1)均表达下调,而氨基酸生物合成通路及抗坏血酸和醛糖酸盐代谢通路与细菌体内C素和N素代谢密切相关,因此ntrC缺失可能会对嗜水气单胞菌体内碳素代谢以及碳︰氮平衡产生影响[22]。当ntrC缺失后,NhaP型Na+/H+和K+/H+反转运蛋白A0KFD8表达下调,表明ntrC可能利用Na+/H+和K+/H+反转运蛋白来应对环境中高渗透压的胁迫[23];氮调节蛋白GlnL (即NtrB)表达上调,可促进NtrC磷酸化以应对缺氮胁迫条件[24];异源生物还原酶XenB表达上调、A0KML1双加氧酶表达下调可能与应对氧化胁迫有关;TonB依赖受体蛋白(例如A0KNS2、A0KQW7)表达下调,而TonB依赖受体蛋白与细菌中铁源获取密切相关[25],可能导致细菌减少对铁离子的吸收从而增强铁胁迫下的抗性;锰依赖性无机焦磷酸酶A0KMJ0表达上调,可能影响细菌应对锰离子的胁迫;RpoS表达上调,该蛋白是RNA聚合酶的σ因子之一,RpoS依赖的基因表达导致细胞产生普遍的抗逆性[26],如抗生素耐药、金属胁迫、氧化胁迫等。以上结果表明,嗜水气单胞菌的ntrC可能是一种全局调控因子,对整个细菌机体的代谢过程都起到重要的作用。
4 结论 本研究通过测定ntrC的生物学功能,阐明了嗜水气单胞菌ntrC在响应缺氮、高渗透压、氧化、重金属离子以及抗生素胁迫中发挥的重要作用。这些结果表明,ntrC基因可能参与嗜水气单胞菌生命活动中的多个调控机制,或许是一种全局调控因子。本研究为今后探讨防治嗜水气单胞菌的药物提供了理论依据。
References
| [1] | Capra EJ, Laub MT. Evolution of two-component signal transduction systems. Annual Review of Microbiology, 2012, 66: 325-347. DOI:10.1146/annurev-micro-092611-150039 |
| [2] | Mengel K, Kirkby EA, Kosegarten H, Appel T. Nitrogen. Principles of Plant Nutrition. Dordrecht: Springer Netherlands, 2001: 397-434. |
| [3] | Kukolj C, Pedrosa FO, de Souza GA, Sumner LW, Lei ZT, Sumner B, do Amaral FP, Juexin W, Trupti J, Huergo LF, Monteiro RA, Valdameri G, Stacey G, de Souza EM. Proteomic and metabolomic analysis of Azospirillum brasilensentrC mutant under high and low nitrogen conditions. Journal of Proteome Research, 2020, 19(1): 92-105. DOI:10.1021/acs.jproteome.9b00397 |
| [4] | Nixon BT, Ronson CW, Ausubel FM. Two-component regulatory systems responsive to environmental stimuli share strongly conserved domains with the nitrogen assimilation regulatory genes ntrB and ntrC. Proceedings of the National Academy of Sciences of the United States of America, 1986, 83(20): 7850-7854. DOI:10.1073/pnas.83.20.7850 |
| [5] | Hirschman J, Wong PK, Sei K, Keener J, Kustu S. Products of nitrogen regulatory genes ntrA and ntrC of enteric bacteria activate glnA transcription in vitro : evidence that the ntrA product is a Sigma factor. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(22): 7525-7529. DOI:10.1073/pnas.82.22.7525 |
| [6] | Weiss DS, Batut J, Klose KE, Keener J, Kustu S. The phosphorylated form of the enhancer-binding protein NTRC has an ATPase activity that is essential for activation of transcription. Cell, 1991, 67(1): 155-167. DOI:10.1016/0092-8674(91)90579-N |
| [7] | Klose KE, Weiss DS, Kustu S. Glutamate at the site of phosphorylation of nitrogen-regulatory protein NTRC mimics aspartyl-phosphate and activates the protein. Journal of Molecular Biology, 1993, 232(1): 67-78. DOI:10.1006/jmbi.1993.1370 |
| [8] | Hervás AB, Canosa I, Santero E. Transcriptome analysis of Pseudomonas putida in response to nitrogen availability. Journal of Bacteriology, 2008, 190(1): 416-420. DOI:10.1128/JB.01230-07 |
| [9] | Austin B, Allen-Austin D. A review. Journal of Applied Bacteriology, 1985, 58(5): 483-506. DOI:10.1111/j.1365-2672.1985.tb01490.x |
| [10] | Abdelhamed H, Banes M, Karsi A, Lawrence ML. Recombinant ATPase of virulent Aeromonas hydrophila protects channel catfish against motile Aeromonas septicemia. Frontiers in Immunology, 2019, 10: 1641. DOI:10.3389/fimmu.2019.01641 |
| [11] | Minnaganti VR, Patel PJ, Iancu D, Schoch PE, Cunha BA. Necrotizing fasciitis caused by Aeromonas hydrophila. Heart & Lung, 2000, 29(4): 306-308. |
| [12] | Walker JM. The bicinchoninic acid (BCA) assay for protein quantitation. The Protein Protocols Handbook, 2002. DOI:10.1385/1-59259-169-8:11 |
| [13] | Wi?niewski JR. Quantitative evaluation of filter aided sample preparation (FASP) and multienzyme digestion FASP protocols. Analytical Chemistry, 2016, 88(10): 5438-5443. DOI:10.1021/acs.analchem.6b00859 |
| [14] | Walter W, Sánchez-Cabo F, Ricote M. GOplot: an R package for visually combining expression data with functional analysis. Bioinformatics, 2015, 31(17): 2912-2914. DOI:10.1093/bioinformatics/btv300 |
| [15] | Fang X, Sastry A, Mih N, Kim D, Tan J, Yurkovich JT, Lloyd CJ, Gao Y, Yang L, Palsson BO. Global transcriptional regulatory network for Escherichia coli robustly connects gene expression to transcription factor activities. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(38): 10286-10291. DOI:10.1073/pnas.1702581114 |
| [16] | Hibbitts A, Lucía A, Serrano-Sevilla I, De Matteis L, McArthur M, de la Fuente JM, Aínsa JA, Navarro F. Co-delivery of free vancomycin and transcription factor decoy-nanostructured lipid carriers can enhance inhibition of methicillin resistant Staphylococcus aureus (MRSA). PLoS ONE, 2019, 14(9): e0220684. DOI:10.1371/journal.pone.0220684 |
| [17] | Liu YL, Lardi M, Pedrioli A, Eberl L, Pessi G. NtrC-dependent control of exopolysaccharide synthesis and motility in Burkholderia cenocepacia H111. PLoS ONE, 2017, 12(6): e0180362. DOI:10.1371/journal.pone.0180362 |
| [18] | Cheng AT, Zamorano-Sánchez D, Teschler JK, Wu D, Yildiz FH. NtrC adds a new node to the complex regulatory network of biofilm formation andvpsExpression in Vibrio cholerae. Journal of Bacteriology, 2018, 200(15): e00025-18. DOI:10.1128/jb.00025-18 |
| [19] | Schumacher J, Behrends V, Pan ZS, Brown DR, Heydenreich F, Lewis MR, Bennett MH, Razzaghi B, Komorowski M, Barahona M, Stumpf MPH, Wigneshweraraj S, Bundy JG, Buck M. Nitrogen and carbon status are integrated at the transcriptional level by the nitrogen regulator NtrC in vivo. mBio, 2013, 4(6): e00881-e00813. |
| [20] | McFarland N, McCarter L, Artz S, Kustu S. Nitrogen regulatory locus "glnR" of enteric bacteria is composed of cistrons ntrB and ntrC: identification of their protein products. Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(4): 2135-2139. DOI:10.1073/pnas.78.4.2135 |
| [21] | Ferro-Luzzi Ames G, Nikaido K. Nitrogen regulation in Salmonella typhimurium. Identification of an ntrC protein-binding site and definition of a consensus binding sequence. The EMBO Journal, 1985, 4(2): 539-547. DOI:10.1002/j.1460-2075.1985.tb03662.x |
| [22] | Nishijyo T, Haas D, Itoh Y. The CbrA-CbrB two-component regulatory system controls the utilization of multiple carbon and nitrogen sources in Pseudomonas aeruginosa. Molecular Microbiology, 2001, 40(4): 917-931. DOI:10.1046/j.1365-2958.2001.02435.x |
| [23] | Cheng B, Meng YW, Cui YB, Li CF, Tao F, Yin HJ, Yang CY, Xu P. Alkaline response of a halotolerant alkaliphilic Halomonas strain and functional diversity of its Na+(K+)/H+ antiporters. Journal of Biological Chemistry, 2016, 291(50): 26056-26065. DOI:10.1074/jbc.M116.751016 |
| [24] | Characterization of a gene, glnL, the product of which is involved in the regulation of nitrogen utilization in Escherichia coli. Journal of Bacteriology, 1982, 151(2): 1067. |
| [25] | Wexler M, Yeoman KH, Stevens JB, de Luca NG, Sawers G, Johnston AWB. The Rhizobium leguminosarum tonB gene is required for the uptake of siderophore and haem as sources of iron. Molecular Microbiology, 2001, 41(4): 801-816. |
| [26] | Battesti A, Majdalani N, Gottesman S. The RpoS-mediated general stress response in Escherichia coli. Annual Review of Microbiology, 2011, 65: 189-213. DOI:10.1146/annurev-micro-090110-102946 |
