李玲, 王明钰

 , 徐海
, 徐海

山东大学微生物技术研究院, 微生物技术国家重点实验室, 山东 青岛 266237
收稿日期:2021-03-22;修回日期:2021-06-22;网络出版日期:2021-10-11
基金项目:国家自然科学基金(31770042,31770043),山东省重点研发计划(2020CXGC011305)
*通信作者:王明钰, E-mail: wangmingyu@sdu.edu.cn;
徐海, E-mail: Tel/Fax: +86-532-58630856;haixu@sdu.edu.cn.
摘要:[目的] 分析新型甲氧苄啶获得性耐药蛋白DfrB7的生化性质,探究其与B家族代表性的二氢叶酸还原酶(DHFR)对甲氧苄啶获得性耐药的生化基础。[方法] 构建系统进化树分析B家族DHFRs与新型DfrB7的进化关系。将dfr基因分别构建到pACYC184和pET15b(+)载体并转化到相应大肠杆菌中。使用微量肉汤稀释法确定克隆菌株对甲氧苄啶的耐药性。测定DHFRs使用NADPH作为质子供体催化二氢叶酸还原的酶活反应参数。通过等温滴定量热法测定甲氧苄啶的解离常数。[结果] 克隆到大肠杆菌中的新型dfrB7基因表现出对甲氧苄啶的高耐药表型。系统进化树确定了dfrB7编码B家族的DHFRs。检测并比较DfrB7和代表性DHFRs的酶活性质、抑制剂的亲和力,与染色体上DHFR相比,DfrB7与DfrB1均表现出显著降低的催化活性,通过生化实验证实B家族二氢叶酸还原酶对甲氧苄啶结合力极差。[结论] 新型dfrB7基因编码的DfrB7具有B家族二氢叶酸还原酶的普遍特征。B家族二氢叶酸还原酶赋予宿主菌对甲氧苄啶的获得性耐药与该酶对甲氧苄啶的低亲和力有关。
关键词:二氢叶酸还原酶甲氧苄啶耐药性生化分析
Characterization and biochemistry analysis of a novel trimethoprim resistant DfrB7 and the mechanism of trimethoprim resistance
Ling Li, Mingyu Wang

 , Hai Xu
, Hai Xu

State Key Laboratory of Microbial Technology, Microbial Technology Institute, Shandong University, Qingdao 266237, Shandong Province, China
Received: 22 March 2021; Revised: 22 June 2021; Published online: 11 October 2021
*Corresponding author: Mingyu Wang, E-mail: wangmingyu@sdu.edu.cn;
Hai Xu, E-mail: Tel/Fax: +86-532-58630856;haixu@sdu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31770042, 31770043) and by the Shandong Province Key Research and Development Program (2020CXGC011305)
Abstract: [Objective] To characterize novel acquired trimethoprim resistance gene dfrB7, and to determine the biochemical basis of acquired trimethoprim resistance for DfrB7 coded by novel dfrB7 and previously known representative Family B DHFRs. [Methods] Phylogenetic analysis of previously reported DfrB proteins and the novel DfrB7 was performed. PCR-amplified dfr genes were cloned into pACYC184 and pET15b(+) vectors, followed by transformation into Escherichia coli. The dfr-pACYC184 plasmid containing E. coli strains were tested for trimethoprim susceptibility by microdilution broth method. Enzymatic catalysis parameters were determined by analyzing the NADPH: dihydrofolate oxidoreductase activities. Isothermal titration calorimetry was performed to measure the dissociation constants between TMP and DHFRs. [Results] Novel dfrB7 gene conferred trimethoprim resistance (MIC ≥ 1024 mg/L) when it was cloned into E. coli. Phylogenetic analysis showed that dfrB7 encodes a Family B DHFR. Novel dfrB7 and previously known representative dfr genes were overexpressed and purified for the analysis of enzymatic parameters and TMP affinity. Comparing with chromosomal DHFR, both DfrB1 and DfrB7 showed significantly lower activities, and Family B DHFRs have drastic lower affinities to trimethoprim. [Conclusion] DfrB7 encoded by a novel dfrB7 gene has common characteristics of Family B DHFRs. The acquired resistance to trimethoprim is caused by the low affinities of Family B DHFRs to trimethoprim.
Keywords: dihydrofolate reductasetrimethoprim resistancebiochemistry analysis
二氢叶酸还原酶(DHFR,EC 1.5.1.3) 是一种广泛存在于真核和原核生物的氧化还原酶,能够在辅因子NADPH的递氢下,将底物二氢叶酸(DHF)还原为四氢叶酸(THF),同时释放NADP+。四氢叶酸作为细胞中重要的前体物质,参与DNA、RNA以及某些氨基酸的合成[1]。因此二氢叶酸还原酶也是临床治疗和新药开发中经典的靶向酶,在抗癌、抗病原菌感染和抗疟疾方面都有研究。
甲氧苄啶(trimethoprim,TMP)是细菌二氢叶酸还原酶的竞争性抑制剂,于20世纪60年代早期合成并用于临床治疗[2]。因其结构简单、药效好,尤其与磺胺类药物联合使用效能的开发,TMP被广泛应用于临床和养殖业,迄今已有几十年的使用历史[3-4]。直到今天,TMP仍是治疗由大肠杆菌以及某些奇异变形杆菌、克雷伯菌属、小部分肠杆菌科细菌和腐生葡萄球菌[5-7]引起的非复杂性尿路感染的一线药物。同时TMP也是治疗由金黄色葡萄球菌引起的皮肤和软组织感染的有效药物[5]。TMP与磺胺类药物联用的复方新诺明(co-trimoxazole,SXT),已被证明对引起呼吸道、肠道和尿路感染的广泛病原菌具有协同杀灭作用,也是治疗卡氏肺孢子虫感染的低廉、有效药物[8]。然而,TMP的过度和广泛使用,例如当做常规预防性的药物,加剧了世界范围内TMP耐药菌的流行。随着对TMP耐药基因研究的深入,对TMP耐药基因的分类和耐药机制的研究也逐渐得以阐明。
一般认为细菌表现TMP耐药性的一个重要原因是通过质粒、转化子、转座子等可移动遗传元件获得了外源的二氢叶酸还原酶基因,即dfr基因。自从1972年报道了第一个质粒介导的dfr基因[9]以来,迄今已有40余种导致TMP高耐药性的dfr基因在革兰氏阴性菌中被发现[10]。这些dfr基因编码的二氢叶酸还原酶主要分为DfrA和DfrB两个家族。与细菌染色体上DHFR或DfrA家族的其他DHFR完全不同,dfrB基因编码的B家族DHFR仅有8个成员,编码非常相似且极短的低分子量蛋白(78-82个氨基酸),氨基酸序列相似性高达75%以上,使宿主菌表现出对TMP极强的耐药性[11]。DfrB1 (又名R67 DHFR,PDB ID:2RK1)的晶体结构已被解析[12],DfrA、B家族蛋白结构的比较如图 1所示[13]。研究发现除了基因上的不同外,B家族DHFR还具有不同寻常的结构,由4个相同亚基组成的同源四聚体形成一个对称的活性位点隧道,在任何给定时间只允许一个底物结合/催化事件发生[14]。由于活性位点缺乏结合特异性的催化策略,推测B家族DHFR是一种进化不完全的原始酶[15]。
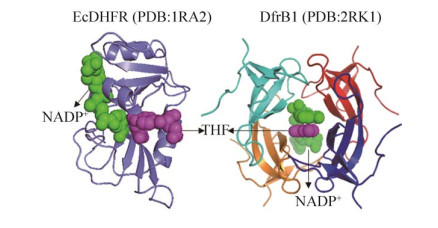 |
| 图 1 A、B家族DHFR的蛋白结构比较[13] Figure 1 Structural comparison of Family A and Family B DHFR. Green represents NADPH; magenta represents folate to EcDHFR or DHF to DfrB1. |
| 图选项 |
在此前的工作中,已确定dfrB7存在于Aeromonads hydrophila Sam7-TMC1的1型整合子的dfrB7-aadA2基因阵列中[16]。本文主要从蛋白的结构和功能角度,设计了一系列实验探究新型dfrB7编码蛋白的理化性质以及B家族二氢叶酸还原酶赋予菌体的TMP耐药策略。
1 材料和方法 1.1 菌株和药物敏感性实验 使用抗生素筛选,在自然水体中分离到耐TMP的菌株A. hydrophila Sam7-TMC1,通过对该菌株的1型整合子基因盒阵列的检测发现这一新型dfrB7基因[16]。用于对照的dfrB1基因合成于金唯智公司。根据美国药敏标准(CLSI)指示的肉汤稀释法检测TMP的最小抑菌浓度(MIC)。接种物终浓度为5×105 CFU/mL,接种到新鲜配制的药物梯度的细胞培养皿中,37 ℃培养12 h以上。为了证明菌体对TMP的高耐药性确实由dfrB7基因造成,将dfrB7基因以及用作对照的dfrB1、大肠杆菌染色体上的folA基因(编码大肠杆菌染色体上敏感型二氢叶酸还原酶EcDHFR) 分别克隆到低拷贝数的pACYC184质粒和高拷贝数的pET15b(+)质粒,并转化到E. coli DH5α感受态细胞中进行MIC的检测。含有pACYC184、pET15b(+)空载体的DH5α为实验的阴性对照。
1.2 序列比对和进化树分析 从GenBank数据库搜集所有已知的B家族DHFR的序列和A家族代表性的DHFR序列,使用MEGA (version 7.0.18) 进行序列比对,然后通过最大似然法(JTT model, 1500 bootstraps)对多序列比对后的数据进行进化树分析。
1.3 DHFR的克隆、异源表达和纯化 设计带有同源臂的引物通过PCR扩增将新型dfrB7基因、B家族代表性的dfrB1基因和作为对照的folA基因分别从A. hydrophila Sam7-TMC1基因组、合成的dfrB1-pUC57质粒、E. coli DH5α基因组中克隆出来。同时通过PCR扩增将pET15b(+)质粒线性化,使之带有相应的50 bp同源臂。使用GB/dir方法将PCR产物与线性化质粒连接,完成重组质粒构建。将重组质粒分别转化入表达菌株E. coli BL21(DE3)感受态细胞。重组菌株于37 ℃培养至OD600为0.6-0.8时加入IPTG使终浓度为0.5 mmol/L,16 ℃过夜诱导。离心收集菌体,超声破碎细胞,高速离心收集上清。细胞破碎上清液使用镍离子亲和层析柱(GE Healthcare) 进行亲和层析。纯化后的蛋白洗脱液使用PD-10脱盐柱去除咪唑,A280法测定浓度后保存于-80 ℃冰箱。蛋白纯化和贮存缓冲液体系为:20 mmol/L Tris,150 mmol/L NaCl,pH 8.0。
1.4 二氢叶酸还原酶的酶活实验 根据米氏方程v=(vmax×S)/(Km+S),酶活常数Km和vmax可以通过测量一系列底物浓度下酶催化反应的初速度,通过图像的非线性拟合进行计算[17]。酶活反应缓冲液成分为:20 mmol/L TES (pH 7.5),50 mmol/L KCl,0.5 mmol/L EDTA,10 mmol/L β-巯基乙醇,1 mg/mL BSA。每次反应(总反应体积为500 μL),稀释的5 μL酶液与5 μL NADPH溶液(反应终浓度100 μmol/L)加入酶活缓冲液后在室温下孵育5 min。在加入2.5-30.0 μL DHF溶液(反应终浓度5-60 μmol/L,溶于50 mmol/L TES,pH 7.0)后立即测量1 min内A340吸光度的变化。每个底物浓度的反应测量3次,使用Origin 9.0作图,拟合一系列底物浓度下单位时间内吸光度的变化,以获得带有标准偏差的Km值[18]。340 nm波长下DHFR还原DHF反应的摩尔消光系数为12300 (mol/L)-1·cm-1。
1.5 等温滴定量热法实验 使用MicroCal PEAQ-ITC量热仪测定298K条件下抑制剂TMP与DHFR结合产生的微量热。由于TMP溶解于DMSO,需要通过透析蛋白溶液统一样品池和滴定针中的缓冲液成分,95% (V/V) Tris-Cl buffer (20 mmol/L Tris,150 mmol/L NaCl,pH 8.0)和5% (V/V) DMSO。样品池中是与10倍摩尔量NADPH孵育的DHFR溶液。设置滴定实验中蛋白与TMP的摩尔浓度比为1:10。第一滴设置为0.4 μL,其余18滴体积均为2 μL。滴定间隔为300 s,搅拌速度为750 r/min。ITC的滴定数据根据非交互单点结合模型使用最小二乘法进行非线性回归拟合。
2 结果和讨论 2.1 新型甲氧苄啶耐药基因dfrB7的鉴定 此前已报道dfrB7基因以dfrB7-aadA2基因盒阵列的形式存在于嗜水气单胞菌A. hydrophila Sam7-TMC1的1型整合子上[16]。本文对1型整合子结构及进化关系的分析如图 2所示,通过对dfrB7基因编码的蛋白序列以及已知的其他7个B家族DHFR进行进化树分析,DfrB7与其他B家族DHFR有聚类关系,证实了此前的注释。
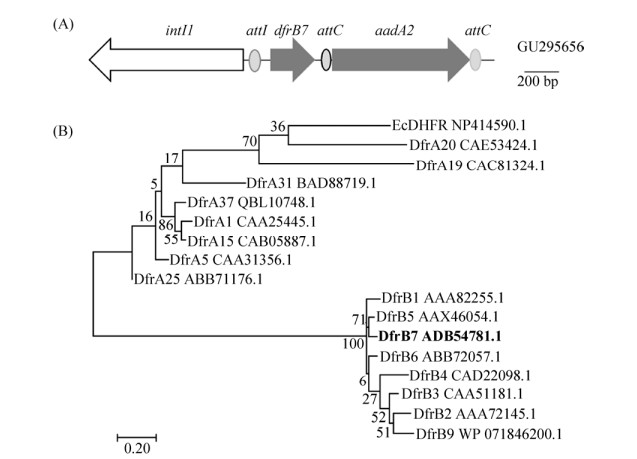 |
| 图 2 dfrB7的基因结构与B家族二氢叶酸还原酶的进化树分析 Figure 2 Genetic structure of dfrB7 and phylogenetic analysis of Family B DHFRs. A: Genetic structure of gene cassette in class 1 integron. Antibiotic resistance genes are indicated by gray arrows. The integrase gene is shown in white arrow. Integron-associated recombination sites are shown in different ellipses. GenBank number is GU295656. B: Phylogenetic analysis of Family B DHFRs and partial Family A DHFRs. GenBank numbers are listed alongside protein names. Numbers at each node are bootstrap values. Bar: evolutionary distance. |
| 图选项 |
多序列比对显示(图 3),DfrB7与其余7个B家族DHFR具有较高的序列相似性。相比B家族其他DHFR,DfrB7在C端多出GVQG四个氨基酸残基,整体看来B家族DHFR的C端序列较为保守,可能参与蛋白结构和功能的稳定,而N端氨基酸序列变化较大,有研究表明N端并非功能区而是作为四聚体域间连接的连接区[14]。新型DfrB7从一级结构上符合B家族DHFR的特征,结合图 1所示EcDHFR和DfrB1的蛋白结构,DfrB7同样缺乏底物的特异性结合位点,对底物类似物TMP的亲和力也会低于EcDHFR。
 |
| 图 3 B家族二氢叶酸还原酶多序列比对 Figure 3 Multiple sequence alignment of Family B DHFRs. DfrB1 is the reference protein; only mutated residues are shown for the other sequences with conserved residues represented by dots. It is clear that DfrB7 has a longer C-terminal end. |
| 图选项 |
2.2 dfrB7导致菌株表现甲氧苄啶耐药性 通过药敏实验确定携带dfrB7的原始菌株表现出TMP的高耐药性(表 1)。对存在dfrB7、dfrB1和folA基因的E. coli DH5α克隆菌株以及相应的转入空质粒的对照菌株测定其TMP和SXT的MIC值。从表 1可看出,dfrB7所在的2个克隆菌株对TMP的最小抑菌浓度高达1024 mg/L和2048 mg/L,远高出作为对照的空载体菌株(2 mg/L) 和敏感型folA基因所在的克隆菌株(4 mg/L),与原始菌株A. hydrophila 7T1的结果相近。类似地,dfrB7的克隆菌株对SXT的最小抑菌浓度比E. coli DH5α (pACYC184)对照菌株高了至少128倍,因此dfrB7的克隆菌株与原始菌株同样表现出较高的TMP和SXT的耐药性。此外,B家族代表性的dfrB1克隆菌株也表现出TMP的耐药性,但染色体上folA基因的克隆菌株仍对TMP表现敏感。在统一了遗传背景和启动子调控水平后,克隆菌株的药敏实验表明耐药性是由dfrB1和dfrB7基因引起,排除了TMP耐药性是由于原始菌株中启动子过表达的可能,证明新型dfrB7基因是导致菌株表现TMP高耐药性的原因。
表 1. dfr相关菌株与质控菌株对甲氧苄啶和复方新诺明的最小抑菌浓度 Table 1. MICs for TMP and co-trimoxazole in dfr-containing and control strains
| Strain (containing plasmid) | MIC/(mg/L) | |
| Trimethoprim | Co-trimoxazole | |
| E. coli ATCC25922 | S (4) | S (1/19) |
| A. hydrophila 7T1 (dfrB7) | R (2048) | R (≥8/152) |
| E. coli DH5α | S (4) | S (0.0625/1.1875) |
| E. coli DH5α (pACYC184) | S (2) | S (0.25/4.75) |
| E. coli DH5α (folA-pACYC184) | S (4) | S (1/19) |
| E. coli DH5α (dfrB1-pACYC184) | R (2048) | R (≥64/1216) |
| E. coli DH5α (dfrB7-pACYC184) | R (1024) | R (32/608) |
| E. coli DH5α (pET15b) | S (1) | S (0.5/9.5) |
| E. coli DH5α (dfrB1-pET15b) | R (2048) | R (128/2432) |
| E. coli DH5α (dfrB7-pET15b) | R (2048) | R (128/2432) |
| R: resistant; S: susceptible. | ||
表选项
2.3 DfrB7和对照DHFR的酶活参数及TMP亲和力检测 为确定新型DfrB7催化效率和酶活性质,并探究菌体对TMP耐药性与酶活效率的关系,将dfrB7、作为对照的dfrB1和folA基因分别构建到诱导型质粒pET15b(+)和大肠杆菌表达菌株BL21(DE3)中,纯化出DHFR以进行体外酶活和抑制剂结合力实验。通过亲和层析和凝胶过滤层析纯化出DfrB1 (8.4 kDa)、DfrB7 (8.7 kDa)和EcDHFR (18 kDa),SDS-PAGE结果如图 4所示。使用质谱检测分析,使这3个蛋白得到进一步确认。
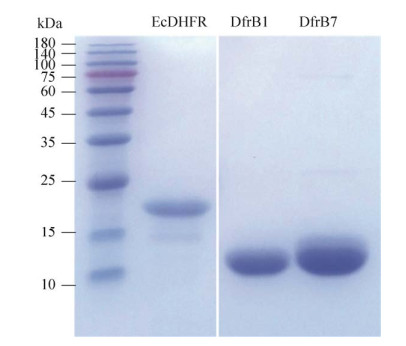 |
| 图 4 二氢叶酸还原酶的SDS-聚丙烯酰胺凝胶图 Figure 4 SDS-PAGE of purified dihydrofolate reductases. |
| 图选项 |
测定不同底物(DHF)浓度下,二氢叶酸还原酶催化NADPH∶DHF氧化还原反应在1 min内340 nm处吸光度的变化速率,所得数据进行非线性回归分析,反应中NADPH的摩尔消光系数ε为12300 (mol/L)-1·cm-1,计算得到相应的Km、kcat等酶活参数(表 2)。从表 2可看出,相比EcDHFR,B家族DfrB1和DfrB7的底物亲和力(Km)相近,但酶活效率(kcat)显著降低了200-400倍,使得催化效率(kcat/Km)也显著降低。由此可以说明,包括新型DfrB7在内的B家族DHFR催化NADPH∶DHF氧化还原反应的酶活效率显著低于细菌染色体上EcDHFR。使用等温滴定量热法(ITC)测定并比较了对TMP敏感的EcDHFR以及TMP耐受的DfrB1、DfrB7对TMP的平衡解离常数(图 5,表 2)。结果表明,EcDHFR与TMP的平衡解离常数为10.02 nmol/L,与之前文章报道的9.1 nmol/L相近[19],而ITC实验未能检测到DfrB1或DfrB7与TMP的结合。ITC实验表明,相比细菌染色体上EcDHFR,B家族DHFR对TMP的亲和力极低。综合数据说明DfrB介导的TMP高耐药性不是因为更高的酶活催化效率,更可能与抗生素的亲和力低相关。
表 2. 纯化DHFR的NADPH︰二氢叶酸氧化还原酶动力学参数 Table 2. NADPH: dihydrofolate oxidoreductase enzymatic parameters of purified DHFRs
| Enzyme | Enzymatic parameters | ITC measurements | ||||||
| Km/(μmol/L) | kcat/(s-1) | kcat/Km/[L/ (μmol·s)] | KD/(nmol/L) | ΔG/(kcal/mol) | ΔH/(kcal/mol) | -TΔS/(kcal/mol) | ||
| EcDHFR | 27.33±5.63 | 17.52±168.00 | 0.6410 | 10.02±3.31 | -10.940±0.207 | -13.260±0.986 | 2.30±1.13 | |
| DfrB1 | 25.19±5.00 | 0.08000±0.00725 | 0.0032 | ND | ND | ND | ND | |
| DfrB7 | 21.90±4.21 | 0.04500±0.00425 | 0.0021 | ND | ND | ND | ND | |
| Numbers are in mean±standard deviation. ND: not determined. | ||||||||
表选项
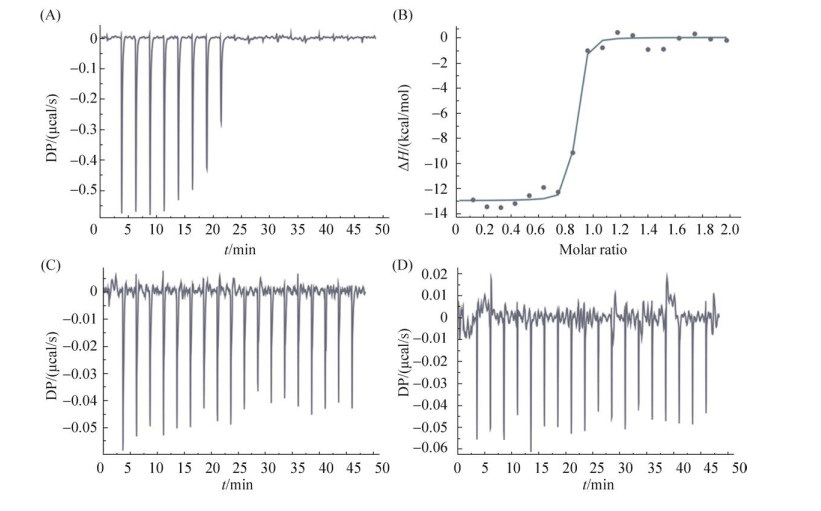 |
| 图 5 等温滴定量热法分析TMP与DHFR和NADPH二元配合物的结合 Figure 5 Analysis of TMP binding with the binary complex of DHFR and NADPH using isothermal titration calorimetry. A: Binding of TMP to EcDHFR: NADPH complex; B: Binding of TMP to DfrB1:NADPH complex; C: Binding of TMP to DfrB7:NADPH complex. Curves represent non-linear least-squares fit to a 1:1 binding model. |
| 图选项 |
3 讨论 现有研究认为,常存在于质粒上的B家族dfr基因能引起菌株对TMP的高耐药性。但解析DfrB1晶体结构后推测B家族DHFR由于缺乏专一高效的催化位点,在进化角度上并不是一个进化完全的酶,如何导致抗生素的不敏感可能需要从理化性质方面解释。B家族DHFR是一种非常独特的酶,从结构到进化关系都完全不同于A家族典型的DHFR。尤其四聚体结构形成一个单一具有222对称的活性位点孔,呈松散的沙漏状,底物DHF和辅因子NADPH都需要水分子等调节与蛋白的结合[20-21]。本文基于已发现的dfrB7基因,通过序列比对和进化树分析确定了新型dfrB7基因的家族分类。dfrB7与其余7个B家族dfr基因编码的蛋白保持88%的序列相似性,但C端多了4个氨基酸,对理化性质的影响未知。基于此,我们通过一系列生化实验和分析,探究了DfrB7的具体酶动力学等生化特性并与B家族代表性的DfrB1以及染色体上DHFR相比较,尝试解析B家族DHFR对TMP的耐受机制。此前对TMP获得性耐药的研究表明,dfr基因启动子的过表达、一些关键位点氨基酸突变使得抗生素分子结合受限等都可能导致耐药性的出现。原始菌株与克隆菌株的药敏实验表明dfrB7基因编码的功能性蛋白不能与TMP结合,是菌株出现TMP耐药表型的原因。与本文对DfrB1和DfrB7酶活性质的研究结论类似,最新的文献报道了B家族DHFRs间的各项酶活动力学参数具有很高的相似性[22]。B家族DHFR中只有DfrB1同时具有结构和动力学参数表征[12],此前以DfrB1为例,研究表明B家族DHFRs的一个特性是对TMP等抗叶酸药物具有极低的结合力[20],从结构上分析,一方面是相比底物DHF,TMP没有酰胺基团与B家族DHFRs骨架上的Ile68形成氢键[23],另一方面B家族DHFRs的疏水活性中心缺乏羧酸基团,导致其不能像典型的DHFR那样与质子化的TMP形成静电相互作用[19, 22]。异源表达和纯化出DfrB7蛋白后,我们通过具体的分子和生化实验验证了DfrB7对TMP的低亲和力特性。
本文从蛋白功能角度,考虑到酶活效率可能也是菌株躲避抗生素“攻击”的因素,设计了体外酶活实验,检测TMP与DHFR亲和力的ITC实验,尝试从生化角度解释新型DfrB7对TMP耐药的具体策略。结果表明,相比细菌染色体上进化完全的DHFR,包括DfrB7在内的B家族DHFR对TMP分子显著低的结合力是甲氧苄啶耐药性的原因,这一结果也与之前对DfrB1晶体结构的解析和推论相一致。
References
| [1] | Watson M, Liu JW, Ollis D. Directed evolution of trimethoprim resistance in Escherichia coli. The FEBS Journal, 2007, 274(10): 2661-2671. DOI:10.1111/j.1742-4658.2007.05801.x |
| [2] | Then RL. Antimicrobial dihydrofolate reductase inhibitors - achievements and future options: review. Journal of Chemotherapy, 2004, 16(1): 3-12. DOI:10.1179/joc.2004.16.1.3 |
| [3] | Huovinen P, Sundstr?m L, Swedberg G, Sk?ld O. Trimethoprim and sulfonamide resistance. Antimicrobial Agents and Chemotherapy, 1995, 39(2): 279-289. DOI:10.1128/AAC.39.2.279 |
| [4] | Sk?ld O. Resistance to trimethoprim and sulfonamides. Veterinary Research, 2001, 32(3/4): 261-273. DOI:10.1051/vetres:2001123 |
| [5] | Wróbel A, Arciszewska K, Maliszewski D, Drozdowska D. Trimethoprim and other nonclassical antifolates an excellent template for searching modifications of dihydrofolate reductase enzyme inhibitors. The Journal of Antibiotics, 2020, 73(1): 5-27. DOI:10.1038/s41429-019-0240-6 |
| [6] | Kahlmeter G. An international survey of the antimicrobial susceptibility of pathogens from uncomplicated urinary tract infections: the ECO·SENS Project. Journal of Antimicrobial Chemotherapy, 2003, 51(1): 69-76. DOI:10.1093/jac/dkg028 |
| [7] | Kahlmeter G, Poulsen HO. Antimicrobial susceptibility of Escherichia coli from community-acquired urinary tract infections in Europe: the ECO·SENS study revisited. International Journal of Antimicrobial Agents, 2012, 39(1): 45-51. DOI:10.1016/j.ijantimicag.2011.09.013 |
| [8] | Allegra CJ, Kovacs JA, Drake JC, Swan JC, Chabner BA, Masur H. Activity of antifolates against Pneumocystis carinii dihydrofolate reductase and identification of a potent new agent. The Journal of Experimental Medicine, 1987, 165(3): 926-931. DOI:10.1084/jem.165.3.926 |
| [9] | Fleming MP, Datta N, Grüneberg RN. Trimethoprim resistance determined by R factors. British Medical Journal, 1972, 1(5802): 726-728. DOI:10.1136/bmj.1.5802.726 |
| [10] | Sánchez-Osuna M, Cortés P, Llagostera M, Barbé J, Erill I. Exploration into the origins and mobilization of di-hydrofolate reductase genes and the emergence of clinical resistance to trimethoprim. Microbial Genomics, 2020, 6(11): mgen000440. |
| [11] | Alonso H, Gready JE. Integron-sequestered dihydrofolate reductase: a recently redeployed enzyme. Trends in Microbiology, 2006, 14(5): 236-242. DOI:10.1016/j.tim.2006.03.003 |
| [12] | Toulouse J, Yachnin BJ, Ruediger EH, Deon D, Gagnon M, Saint-Jacques K, Ebert MCC, Forge D, Bastien D, Colin DY, Vanden Eynde JJ, Marinier A, Berghuis AM, Pelletier JN. Structure-based design of dimeric bisbenzimidazole inhibitors to an emergent trimethoprim-resistant type Ⅱ dihydrofolate reductase guides the design of monomeric analogues. ACS Omega, 2019, 4(6): 10056-10069. DOI:10.1021/acsomega.9b00640 |
| [13] | Duff M, Desai N, Craig MA, Agarwal PK, Howell EE. Crowders steal dihydrofolate reductase ligands through quinary interactions. Biochemistry, 2019, 58(9): 1198-1213. DOI:10.1021/acs.biochem.8b01110 |
| [14] | Ebert MCCJC, Morley KL, Volpato JP, Schmitzer AR, Pelletier JN. Asymmetric mutations in the tetrameric R67 dihydrofolate reductase reveal high tolerance to active-site substitutions. Protein Science, 2015, 24(4): 495-507. DOI:10.1002/pro.2602 |
| [15] | Strader MB, Chopra S, Jackson M, Smiley RD, Stinnett L, Wu J, Howell EE. Defining the binding site of homotetrameric R67 dihydrofolate reductase and correlating binding enthalpy with catalysis. Biochemistry, 2004, 43(23): 7403-7412. DOI:10.1021/bi049646k |
| [16] | Xu H, Broersma K, Miao V, Davies J. Class 1 and class 2 integrons in multidrug-resistant gram-negative bacteria isolated from the Salmon River, British Columbia. Canadian Journal of Microbiology, 2011, 57(6): 460-467. DOI:10.1139/w11-029 |
| [17] | Reeve SM, Si D, Krucinska J, Yan YZ, Viswanathan K, Wang SY, Holt GT, Frenkel MS, Ojewole AA, Estrada A, Agabiti SS, Alverson JB, Gibson ND, Priestley ND, Wiemer AJ, Donald BR, Wright DL. Toward broad spectrum dihydrofolate reductase inhibitors targeting trimethoprim resistant enzymes identified in clinical isolates of methicillin resistant Staphylococcus aureus. ACS Infectious Diseases, 2019, 5(11): 1896-1906. DOI:10.1021/acsinfecdis.9b00222 |
| [18] | Li L, Zhang MG, Wang WJ, Xia RR, Ma YN, Wei X, Wang XH, Sun XM, Xie XH, Xie SL, Wang MY, Xu H. Identification and characterization of two novel ISCR1-associated genes dfrA42 and dfrA43 encoding trimethoprim resistant dihydrofolate reductases. Antimicrobial Agents and Chemotherapy, 2021: AAC.02010-AAC.02020. |
| [19] | Abdizadeh H, Tamer YT, Acar O, Toprak E, Atilgan AR, Atilgan C. Increased substrate affinity in the Escherichia coli L28R dihydrofolate reductase mutant causes trimethoprim resistance. Physical Chemistry Chemical Physics, 2017, 19(18): 11416-11428. DOI:10.1039/C7CP01458A |
| [20] | Krahn JM, Jackson MR, DeRose EF, Howell EE, London RE. Crystal structure of a type Ⅱ dihydrofolate reductase catalytic ternary complex. Biochemistry, 2007, 46(51): 14878-14888. DOI:10.1021/bi701532r |
| [21] | Chopra S, Dooling RM, Horner CG, Howell EE. A balancing act between net uptake of water during dihydrofolate binding and net release of water upon NADPH binding in R67 dihydrofolate reductase. Journal of Biological Chemistry, 2008, 283(8): 4690-4698. DOI:10.1074/jbc.M709443200 |
| [22] | Toulouse JL, Shi GB, Lemay-St-denis C, Ebert MCCJC, Deon D, Gagnon M, Ruediger E, Saint-Jacques K, Forge D, Vanden Eynde JJ, Marinier A, Ji XH, Pelletier JN. Dual-target inhibitors of the folate pathway inhibit intrinsically trimethoprim-resistant DfrB dihydrofolate reductases. ACS Medicinal Chemistry Letters, 2020, 11(11): 2261-2267. DOI:10.1021/acsmedchemlett.0c00393 |
| [23] | Bastien D, Ebert MCCJC, Forge D, Toulouse J, Kadnikova N, Perron F, Mayence A, Huang TL, Vanden Eynde JJ, Pelletier JN. Fragment-based design of symmetrical bis-benzimidazoles as selective inhibitors of the trimethoprim-resistant, type Ⅱ R67 dihydrofolate reductase. Journal of Medicinal Chemistry, 2012, 55(7): 3182-3192. DOI:10.1021/jm201645r |
