周秀娟, 崔妍, 何逸尘, 张利达, 史贤明


上海交通大学农业与生物学院, 中美食品安全联合研究中心, 微生物代谢国家重点实验室, 上海 200240
收稿日期:2020-08-19;修回日期:2020-11-17;网络出版日期:2021-07-13
基金项目:国家“十三五”重点研发计划(2017YFC1601200)
*通信作者:史贤明, Tel/Fax: +86-21-34206616;E-mail: xmshi@sjtu.edu.cn.
摘要:[目的] 分析沙门氏菌的泛耐药基因组特征。[方法] 以EnteroBase数据库中16365株沙门氏菌为对象,利用课题组自主研发的泛耐药基因组分析软件(PRAP),进行泛耐药基因组结构的鉴定,通过曼-惠特尼秩和检验和皮尔逊检验,来分析耐药基因与血清型、序列型(sequence type,ST)及分离株样本来源信息间的相关性。[结果] 沙门氏菌共有104种耐药基因,其中核心耐药基因18种,附属耐药基因86种,且沙门氏菌拥有一个开放型的泛耐药基因组;相同的血清型(或ST型)有相似的耐药基因谱,不同血清型(或ST型)间耐药基因的分布差异显著(P < 0.05),耐药基因与样本来源、分离国家及年份间也存在一定的相关性;在测试的23种获得性耐药基因中,43.48%(10/23)的占比逐年升高,73.91%(17/23)以单一亚型为优势。[结论] 利用PRAP软件分析获得的这些结果揭示了近年来沙门氏菌耐药基因的时空分布规律,为沙门氏菌等食源性致病菌耐药性的研究提供了新思路。
关键词:沙门氏菌泛耐药基因组耐药基因泛耐药基因组分析软件
Characteristics analysis of pan-resistant genome of Salmonella
Xiujuan Zhou, Yan Cui, Yichen He, Lida Zhang, Xianming Shi


MOST-USDA Joint Research Center for Food Safety, School of Agriculture & Biology, and State Key Lab of Microbial Metabolism, Shanghai Jiao Tong University, Shanghai 200240, China
Received: 19 August 2020; Revised: 17 November 2020; Published online: 13 July 2021
*Corresponding author: Xianming Shi, Tel/Fax: +86-21-34206616; E-mail: xmshi@sjtu.edu.cn.
Foundation item: Supported by the National Key R&D Program of China (2017YFC1601200)
Abstract: [Objective] Analyze the characteristics of the pan-resistant genome of Salmonella. [Methods] In this study, the pan-resistome analysis pipeline developed in our previous researches was used to identify the pan-resistant genomic structure and to analyze the correlation between drug-resistant genes and serotype, sequence type and isolation information, etc. based on the sequencing of 16, 365 Salmonella strains downloaded from the EnteroBase database. [Results] All drug-resistant genes were categorized into 104 alleles, among which 18 alleles belonged to the core resistome and 86 alleles belonged to the accessory resistome. It had an open pan-resistant genome. The same serotype (or sequence type) had similar drug-resistant genes profile, however, the distribution of drug-resistant genes in different serotypes (or sequence types) is significantly different (P < 0.05). Moreover, there was a certain correlation between drug-resistant genes and sample sources, separation countries and isolated years. The proportion of 23 acquired drug-resistant genes and their subtypes revealed that 10 (43.48%) of them increased yearly, and 10 (73.91%) of them had only one dominant type. [Conclusion] All the results obtained in this study have revealed the distribution and trend of Salmonella drug-resistant genes in recent years, in further to provide new ideas to study the drug-resistance of pathogens.
Keywords: Salmonellapan-resistant genomedrug-resistant genespan-resistome analysis pipeline
沙门氏菌(Salmonella)是一种重要的食源性致病菌,主要以肉、蛋、奶等动物性食品为传播载体[1-2],引发肠胃炎、败血症和伤寒等人类疾病,严重时甚至可导致死亡[3]。抗生素是其最常用的控制手段,然而,由于耐药基因的产生与转移,许多抗生素的作用效果逐渐减弱,沙门氏菌的耐药问题也日益严重[4],给食品安全和人类健康带来了巨大威胁。其中,快速精确地检测耐药基因不仅是耐药菌识别的重要方法,也是流行病学调查的重要组成部分。
目前主要采用基于PCR的传统分子生物学方法来检测耐药基因[5-6],然而,这些方法不仅较为费时费力,而且难以涵盖所有的耐药基因并精确到基因亚型。伴随着全基因测序技术的发展及其成本的降低,通过生物信息学方法从基因组数据中快速筛查耐药基因已成为研究热点[7-8]。目前,常用的耐药数据库有CARD[9]、PATRIC[10]和ResFinder[11]等,包含24305株沙门氏菌基因组信息及124977条耐药表型信息(数据更新于2020年5月)。耐药沙门氏菌数据库的积累与沙门氏菌基因组信息的丰富,为耐药基因识别软件(包含耐药基因的全面快速筛查和基因亚型的精确鉴定)的开发创造了必要条件。然而,现有的网站式的耐药基因识别软件仅支持单个基因组的分析,而无法完成大批量菌株中耐药基因的高效筛查和基于筛查结果的下游比对分析,因此,需要引入泛基因组学(pan-genomics)的方法来寻找多个菌株个体间的遗传差异[12]。泛基因组曲线在一定程度上反映了不同物种获得新基因的能力[13]。
泛耐药基因组(pan-resistome)是泛基因组概念在耐药研究中的延伸,是一个细菌物种内所有菌株耐药基因的集合。其中,所有菌株共有的耐药基因属于核心耐药基因组(core-resistome),如喹诺酮类抗生素的作用靶点基因(gyrA和parC等),它们与细菌的生理代谢直接相关,且基因的SNP(single nucleotide polymorphism)突变位点是耐药的关键[14];而耐药菌株特有的耐药基因属于附属耐药基因组(accessory-resistome),如超广谱β-内酰胺酶的编码基因(blaTEM和blaCTX-M等),耐药基因的有无直接决定细菌是否耐药[11]。鉴于核心耐药基因与附属耐药基因作用机理的差异,在研究批量基因组时,需要引入泛基因组学的方法来进行不同类型耐药基因(核心或附属)贡献和特征的区别分析。
课题组前期将耐药基因信息学鉴定与泛基因组分析相结合,建立了平台通用、用户友好且运行快速的细菌泛耐药基因组分析软件PRAP[15],可用于耐药基因的快速筛查和批量基因组的特征分析。本研究将选取EnteroBase数据库中获得的16365株沙门氏菌基因组为对象,利用PRAP软件,来系统描述沙门氏菌的泛耐药基因组特征,并分析耐药基因的时空分布规律及与菌株表型特征的相关性。
1 材料和方法 1.1 沙门氏菌基因组序列与菌株信息数据的收集 从EnteroBase数据库(http://enterobase.warwick.ac.uk)中下载获取沙门氏菌基因组序列。为了保障基因组数据质量和相关元数据(描述数据的数据,metadata)的完整性,在“Search all Strains of Salmonella”条目下选择“Ignorce Legacy Data”,将“Serovar”、“Isolation Year”、“Country”等字段(field)设置为“Not equals”,在“Experiment Data”中对“Assembly Status”、“Achtman 7 gene MLST”等字段进行过滤,来筛选数据库中的血清型、分离地区、分离年份(1994-2018年)等信息及基因组序列质量等条目,共得到16365株沙门氏菌的基因组序列及其菌株信息的描述数据。菌株的详细数据及其在EnteroBase数据库的Accession No.可从GitHub(https://github.com/syyrjx-hyc/masterSJTU)获得。
1.2 泛耐药基因组结构鉴定 依托课题组自主研发的泛耐药基因组分析软件PRAP[15](https://github.com/syyrjx-hyc/PRAP)中的ArBlastn和PanResistome模块来完成泛耐药基因组的结构鉴定。序列比对参数为一致性≥70%和覆盖率≥70%[16],将≥98%菌株中存在的耐药基因归入核心耐药基因组,在 < 98%菌株中存在的耐药基因归入附属耐药基因组。针对核心耐药基因,通过MAFFT软件(https://mafft.cbrc.jp/alignment/software/)进行多序列比对,并提取基因的SNP位点,以1、2、3、4分别代表缺失位点A、C、G、T,以0、5分别表示耐药基因的有无,对每个菌株生成其独立的核心耐药基因SNP位点谱。针对附属耐药基因,不进行序列亚型和SNP位点的区别,而仅表示等位基因的有无。由于基因组质量不一,某些SNP位点仅存在于少数几个基因组中,为了去除掉无意义的SNP位点,引入了质量参数Q,计算方法如公式(1)。
| $Q = \frac{{\sum {{N_{other}}} }}{{{N_{\max }}}}$ | 公式(1) |
若Q≥1‰,则将该SNP位点作为可靠变异位点用于后续分析。根据核心耐药基因组和泛耐药基因组分别构建高质量核心耐药基因谱和泛耐药基因谱。再以邻接法(neighbor joining,NJ)计算不同谱之间的遗传距离,并使用GrapeTree和iTOL对进化树进行可视化(https://itol.embl.de/saved_views.cgi)。
1.3 耐药基因与菌株表型特征的关联分析 对血清型、序列型(sequence type,ST)、样本来源、分离国家及时间等菌株信息类别中(菌株数目≥200的类别)耐药基因分布情况进行单独统计。在每个信息类别下,遍历
2 结果和分析 2.1 沙门氏菌泛耐药基因组的结构特征 通过PRAP软件中ArBlastn模块的分析,在16365株沙门氏菌中共鉴定到471737个耐药基因,经过序列比对聚类,分属于104种耐药基因。PanResistome模块分析结果如图 1所示,其中18种属于核心耐药基因,主要包括基因位点突变介导耐药的基因,如介导磷霉素(fosfomycin)耐药的GlpT基因、介导氟喹诺酮(fluoroquinolone)类抗生素耐药的gyrA、gyrB、parC和parE基因,及外排泵相关的mdtK、emrR、soxR和soxS基因。其余86种属于附属耐药基因,占比较高的耐药基因编码抗生素的水解酶或修饰酶,以及编码抗生素的外排泵(如oqxA、oqxB等基因);此外,与靶点保护相关的耐药基因主要属于qnr基因家族(qnrB和qnrS占比较高,qnrA、qnrD和qnrE占比较少)。特别需要指出的是,编码修饰酶、水解酶、靶点修饰和保护蛋白的耐药基因只介导1种抗生素耐药,而编码外排泵的耐药基因常与多种抗生素的耐药相关。
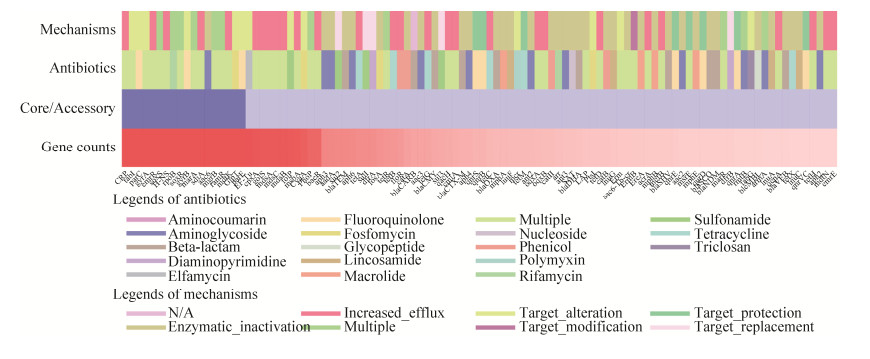 |
| 图 1 16365株沙门氏菌的耐药基因鉴定结果 Figure 1 Identification results of antibiotic resistance genes of 16365 Salmonella. The four rows of the heat map (from bottom to top): the counts of resistance genes (the number of occurrences of each gene in 16365 genomes, the darker the red, the more the counts), whether or not the core resistance genes (dark blue represents that this gene is a core drug resistance gene, light blue represents that this gene is an accessory drug resistance gene), gene-related antibiotics (different colors represent different antibiotics that the gene confers resistance to, and "multiple" represents that there are multiple antibiotic resistance related) and the drug resistance mechanism corresponding to the gene. different colors represent different resistance mechanisms, "multiple" represents that there are multiple mechanisms. |
| 图选项 |
沙门氏菌泛耐药基因组特征曲线如图 2所示,随着菌株数目的增加,泛耐药基因组的大小(即耐药基因的数目)逐渐增加,即沙门氏菌拥有一个开放型的泛耐药基因组,表明它具有较强的获得外源耐药基因的能力。核心耐药基因组的大小在菌株数目很少时(< 1000株)就稳定在18,说明附属耐药基因在不同的菌株间差别较大[12]。
 |
| 图 2 16365株沙门氏菌泛耐药基因组特征曲线 Figure 2 Pan-resistome feature curves of 16365 Salmonella. The fitting model refer to the change curves of pan-drug resistance genome accompanied with the increase in the number of genome was P=39.0722x0.1008 (R2=0.9831). |
| 图选项 |
2.2 沙门氏菌泛耐药基因组谱型分析 通过MAFFT软件的多序列比对,在18种核心耐药基因中,共得到992个SNP位点,其中SNP位点数目较多分别为gyrA(163)、gyrB(144)、parC(131)、parE(83)(基因名(SNP数目))等,SNP位点较少的分别为mgrB(2)、marA(4)、CRP(6)和H-NS(6)等。将所有SNP位点均一化命名为同一个核心耐药基因组谱型(core-resistome profile type,cRPT),结果显示16365株沙门氏菌cRPT型共1327种,菌株数目≥200的cRPT型共17种,其中有1188株菌属于cRPT10型,这是菌株数目最多的一个核心耐药基因组谱型。同样,对泛耐药基因组谱型(pan-resistome profile type,pRPT)进行分析,共有2907种pRPT型,其中菌株数目最多的为pRPT17型,共928株菌,菌株数目≥200的共11种pRPT型。基于NJ法分别构建了的16365株沙门氏菌cPRT和pRPT的系统发育树,如图 3所示。
 |
| 图 3 耐药基因谱聚类及相关性分析 Figure 3 Clustering and correlation analysis of drug resistance gene profiles. A: phylogenetic tree of cRPT and the distribution of serovar; B: phylogenetic tree of pRPT and distribution of related metadata. From inside to outside in figure 3B were the evolutionary tree, serotype, ST type, isolation source, isolation country, isolation time, and number of accessory drug resistance genes. If the number of categories was more than eight, only the first eight categories were showed in color, while the other categories were white. |
| 图选项 |
对于cPRT系统发育树(图 3-A),同一进化分支或亲缘关系较近的分支中的cPRT型与血清型(或ST型)保持高度一致,说明血清型(或ST型)和核心耐药基因的SNP位点具有较高的相关性。对于pRPT系统发育树(图 3-B),整体上进化分支与血清型仍呈现出较高的一致性,且属于同一血清型的不同ST型几乎都聚类于不同的子分支中。如:肠炎沙门氏菌(S. Enteritidis)血清型大部分都聚集在Ⅱ(a)区域的分支中,主要为ST11型,且附属耐药基因数目较其他区域的数目低,说明多数肠炎沙门氏菌获得外源耐药基因的能力可能较其他血清型弱。伤寒沙门氏菌(S. Typhi)主要分布于Ⅱ(b)区域中,又进一步分为2个分支,菌株数目较多的分支为ST1型,另一分支属于ST2型,ST2型几乎没有附属耐药基因,而ST1型中附属耐药基因数目有多有少。鼠伤寒沙门氏菌(S. Typhimurium)主要聚集在Ⅰ(a)区域内,分为ST19和ST34型的2个分支,ST34型菌株的平均分离年份比ST19型要新,ST34型菌株附属耐药基因数目也多于ST19型。综上,沙门氏菌泛耐药基因组谱型分析表明,无论是核心耐药基因的SNP位点还是附属耐药基因的分布情况均与血清型(ST型)具有较强的相关性。
2.3 耐药基因与菌株表型特征的关联性检验
2.3.1 曼-惠特尼秩和检验: 表 1展示了不同菌株信息类别中耐药基因数目的曼-惠特尼秩和检验结果,对于每种菌株信息类别仅显示显著性最高与最低的前3个组合,以及对所有组合数目的显著性统计结果,包括极显著(Ex-sig,P < 0.001),显著(Sig,P < 0.05)和不显著(Non-sig,P > 0.05)的数目和占比。
表 1. 耐药基因数目的曼-惠特尼秩和检验 Table 1. Results of Mann-Whitney U test of number of antibiotic resistance genes
| Metadata | Rank | Type1 | Type2 | P-value | CN2 | Ex-Sig (P < 0.001) | Sig (P < 0.05) | Non-sig (P > 0.05) |
| Serovar | 1 | Typhimurium | Enteritidis | 0.0 | 66 | 65 98.48% | 66 100% | 0 0% |
| 2 | Enteritidis | Infantis | 1.87e–259 | |||||
| 3 | Enteritidis | Heidelberg | 2.02e–207 | |||||
| –3 | Senftenberg | Anatum | 6.22e–05 | |||||
| –2 | Agona | Senftenberg | 6.99e–05 | |||||
| –1 | Typhimurium | Heidelberg | 0.0433 | |||||
| STs | 1 | ST19 | ST11 | 0.0 | 55 | 54 98.18% | 54 98.18% | 1 1.82% |
| 2 | ST32 | ST11 | 3.10e–245 | |||||
| 3 | ST11 | ST15 | 1.15e–197 | |||||
| –3 | ST365 | ST11 | 6.26e–11 | |||||
| –2 | ST13 | ST64 | 2.27e–09 | |||||
| –1 | ST40 | ST15 | 0.4167 | |||||
| Source | 1 | Swine | Plant | 1.90e–124 | 66 | 50 75.76% | 59 89.39% | 7 10.61% |
| 2 | Swine | Fish | 3.01e–90 | |||||
| 3 | Swine | Water | 1.08e–67 | |||||
| –3 | Fish | Water | 0.1817 | |||||
| –2 | Bovine | Environment | 0.2953 | |||||
| –1 | Plant | Laboratory | 0.3419 | |||||
| Country | 1 | Australia | Ireland | 7.49e–114 | 190 | 139 73.16% | 167 87.89% | 23 12.11% |
| 2 | China | Indonesia | 8.18e–80 | |||||
| 3 | Indonesia | France | 3.53e–70 | |||||
| –3 | Ethiopia | Ireland | 0.4339 | |||||
| –2 | Nigeria | Mexico | 0.4360 | |||||
| –1 | Australia | Italy | 0.4809 | |||||
| Year | 1 | 2013 | 2017 | 4.33e–32 | 153 | 84 54.90% | 109 71.24% | 44 28.76% |
| 2 | 2013 | 2016 | 1.92e–27 | |||||
| 3 | 2010 | 2005 | 2.91e–23 | |||||
| –3 | 2007 | 2014 | 0.4359 | |||||
| –2 | 2012 | 2009 | 0.4528 | |||||
| –1 | 2006 | 2018 | 0.4908 |
表选项
① 血清型??在66个两两组合的检验结果中,差异极显著的有65个(98.48%),另一组合差异显著(P=0.0433),说明不同的血清型间的耐药基因携带数目均存在明显的差异。其中P值最小的为S. Typhimurium和S. Enteritidis,说明该两种血清型间耐药基因数目差异最显著。
② ST型??与血清型的检测结果较一致,在55个两两组合中,差异极显著有54个(98.18%),仅ST15与ST40差异不显著(P=0.4167),分别属于S. Heidelberg和S. Derby,而ST19与ST11差异最显著,分别属于S. Typhimurium和S. Enteritidis,与血清型的检验结果相符。
③ 样本来源??在66个组合中,差异显著的共有59个(89.39%),其中差异最显著的主要集中在猪源和其他分离来源,说明从猪源分离的沙门氏菌可能携带有更多的耐药基因,而差异不显著的有7个(10.61%),主要是鱼源和水源、牛源和环境源、植物源和实验室源等。
④ 分离国家??不同国家间的菌株携带的耐药基因数目也存在差异,在190个国家组合中,差异极显著的有139个(73.16%),其中差异最显著的是澳大利亚和爱尔兰,其次是印度尼西亚和中国、法国。需要特别说明的是,来源于澳大利亚和印度尼西亚的沙门氏菌所携带的耐药基因数目均显著低于其他国家。
⑤ 分离年份??耐药基因数目均值存在显著差异的占71.42%,最低的P值为4.33e-32。然而,年份间的显著性差异与年份的时间轴无显著相关性,如:2005年和2010年差异显著,而2006年和2018年差异不显著,从整体上看,年份间的显著性差异不存在规律性,整体的差异与其他的信息类别相比是最小的。
2.3.2 皮尔逊检验: 表 2展示了经皮尔逊检验过滤得到的23种获得性耐药基因的皮尔逊相关系数(Pearson’s correlation coefficient,列表中用R表示)和检验的P值,根据相关性的大小和是否显著可以分为三类:第一类是存在显著正相关的基因,即耐药基因的占比逐年增长,包括氨基糖苷类耐药的AAC(3)、APH(4)基因,β-内酰胺类耐药的blaCMY、blaCTX-M和blaTEM基因,及氟喹诺酮类耐药的qnrB、qnrS基因等;第二类是存在显著负相关的基因,即这些耐药基因占比正在不断地下降,共有2种,即blaCARB和tetG基因:第三类的基因占比和分离年份间无显著相关性。
表 2. 获得性耐药基因占比与年份的皮尔逊检验结果 Table 2. Pearson test of proportion of acquired antibiotic resistance genes and years
| Alleles (R > 0) | R | P-value (P < 0.05) | Alleles (R < 0) | R | P-value (P < 0.05) | Alleles (R≈0) | R | P-value (P > 0.05) |
| AAC(3) | 0.5774 | 0.0121 | blaCARB | –0.6616 | 0.0028 | aadA | –0.2329 | 0.3524 |
| APH(4) | 0.5620 | 0.0152 | tetG | –0.6673 | 0.0025 | APH(3’) | 0.3223 | 0.1921 |
| blaCMY | 0.6542 | 0.0032 | APH(6) | 0.1184 | 0.6400 | |||
| blaCTX-M | 0.6306 | 0.0050 | catA | –0.3898 | 0.1099 | |||
| blaTEM | 0.6173 | 0.0063 | cmlA | 0.1956 | 0.4367 | |||
| fosA | 0.4980 | 0.0354 | dfrA | –0.0475 | 0.8516 | |||
| qnrB | 0.7381 | 0.0005 | floR | –0.0211 | 0.9337 | |||
| qnrS | 0.6772 | 0.0020 | sul1 | 0.1794 | 0.4762 | |||
| sul2 | 0.5651 | 0.0145 | sul3 | 0.3868 | 0.1128 | |||
| tetA | 0.7042 | 0.0011 | tetB | 0.2084 | 0.4066 | |||
| tetR | 0.2149 | 0.3917 |
表选项
每种获得性耐药基因亚型的占比随年份变化情况的统计结果如图 4所示。在本研究的16365株沙门氏菌中,亚型数目较多(> 10种)的获得性耐药基因为aadA(25种)、dfrA(20种)、blaTEM(20种)和blaCTX-M(15种)。73.91%(17/23)的获得性耐药基因都是以某一种亚型占据优势的。如APH(6)基因中主要是APH(6)-Id-1亚型占主要优势,而APH(6)-Id-2、APH(6)-Id-4、APH(6)-Id-5等亚型的占比都很低,同样blaCMY和blaTEM基因主要是blaCMY-2和blaTME-1B占据主要优势,其他亚型数目都很少。对于AAC(3)、blaCTX-M和tet(A)基因,主要是两种亚型占主要优势,且占比的变化趋势大致相同;而对于floR基因,虽然总体占比随年份无明显变化,但是在不同亚型间有明显的替代趋势,即floR-2型占比逐渐下降,而floR亚型占比逐渐升高;同样对于sul2基因,虽然整体呈上升趋势,但sul2-3亚型占比逐渐下降,sul2-2亚型占比逐渐上升。此外,还存在基因(如dfrA)虽然亚型很多,但不同亚型的占比优势及变化趋势都无明显相关性。
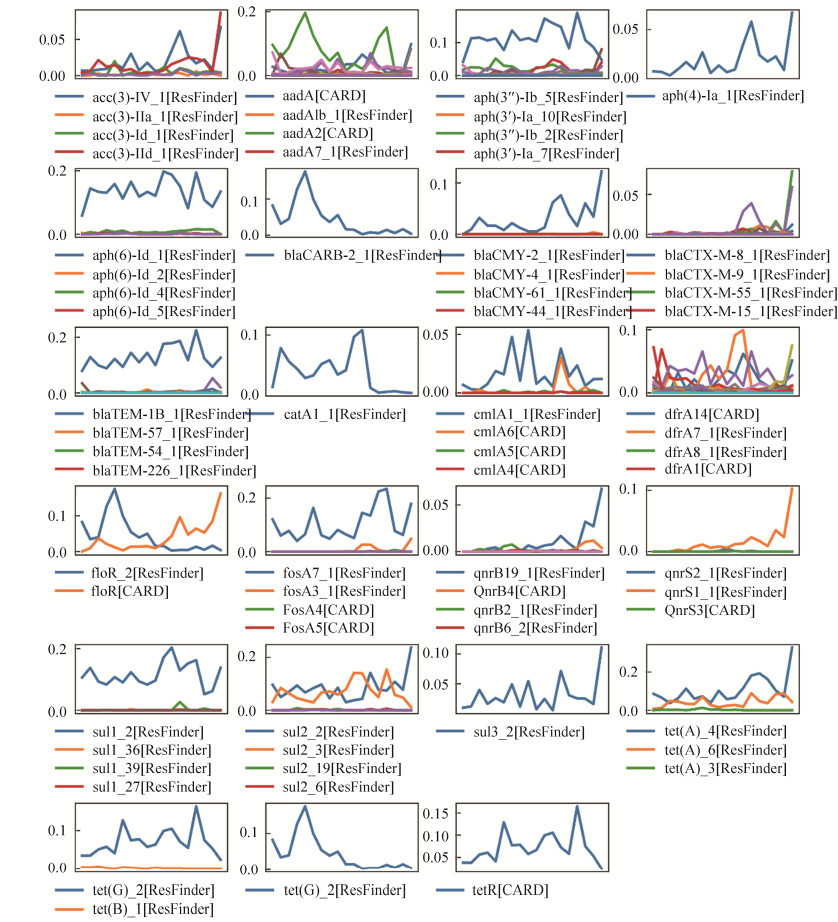 |
| 图 4 不同年份的获得性耐药基因亚型的占比变化 Figure 4 Changes in the proportion of acquired antibiotic resistance gene subtypes in different years. The subtype legend was shown below each sub-picture, and only the first four subtypes were displayed if the number of subtypes is more than 4. From left to right were in increasing order of year during 1994 to 2018. |
| 图选项 |
3 讨论和结论 泛耐药基因组是在泛基因组概念基础上提出的一个延伸性概念,研究对象集中在全部耐药基因上,核心耐药基因和附属耐药基因分别代表了不同类型的耐药机制,且与耐药基因的转播方式密切相关。本研究通过对16365株沙门氏菌菌株的泛耐药基因组特征分析,我们发现:在沙门氏菌中,核心耐药基因与靶位点突变和外排作用相关,常通过基因垂直传播的方式获得;附属耐药基因与酶水解、酶修饰和靶位点保护等相关,可通过基因水平转移的方式获得。这些结论与已报道的耐药基因的研究数据一致[17-19]。已有大量研究都表明沙门氏菌具有开放型的泛基因组[12, 20-21],本研究以耐药基因为对象创新性地表明了沙门氏菌具有开放型的泛耐药基因组,即沙门氏菌具有较强的获得外源耐药基因的能力。
本研究的另一重要内容是基于数据库中大样本(16365株)的菌株数据和信息,结合图谱分析与统计方法,从多个维度(血清型、基因型、分离来源和年份等)来对沙门氏菌耐药基因时空分布特征进行系统分析。其中,沙门氏菌的耐药基因谱聚类及假设检验分析结果表明,不同的沙门氏菌血清型(或ST型)间获得性耐药基因的类型与分布具有显著差异,这与沙门氏菌泛基因组学研究的结果一致[22],可能是由于血清型(或ST型)与沙门氏菌的生理特征存在的差异相关[23]。此外,猪源的沙门氏菌耐药基因数目显著高于其他样本来源,表明在猪的养殖过程中可能存在不同类型抗生素频繁使用的问题[24],从而导致了多重耐药菌的出现[25]。就国家间的差异分析我们发现,来源于澳大利亚与印度尼西亚的沙门氏菌携带耐药基因数目显著低于其他国家,我们推测可能有两个原因:①由于澳大利亚与印度尼西亚地区上传的菌株数据量较少,且不是针对耐药菌进行测序的,导致耐药基因数目偏少;②由于澳大利亚与印度尼西亚都是岛国,这种地域原因导致了外源菌株的耐药基因(主要是以活菌形式传播)难以传播到这两个国家中。
获得性耐药基因年度占比变化统计结果表明,有10种耐药基因(AAC(3)、APH(4)、blaCMY、blaCTX-M、blaTEM、fosA、qnrB、qnrS、sul2、tetA)在沙门氏菌中愈发流行,它们可能是导致近年来沙门氏菌对氨基糖苷类、β内酰胺类、磷霉素类、喹诺酮类、磺胺类以及四环素类抗生素的耐药性呈逐渐增强趋势的重要因素[26-27],并为后续耐药防控提供重要的研究靶标和检测对象。对耐药基因亚型的分析表明,73.91%的获得性耐药基因都仅有一种优势亚型,而floR和sul2耐药基因的优势亚型出现了交替的情况,这些现象可能与近年来氯霉素类和磺胺类抗生素的交替使用变化存在相关性。
References
| [1] | Jackson BR, Griffin PM, Cole D, Walsh KA, Chai SJ. Outbreak-associated Salmonella enterica serotypes and food commodities, United States, 1998-2008. Emerging Infectious Diseases, 2013, 19(8): 1239-1244. DOI:10.3201/eid1908.121511 |
| [2] | Yang XJ, Huang JH, Wu QP, Zhang JM, Liu SR, Guo WP, Cai SZ, Yu SB. Prevalence, antimicrobial resistance and genetic diversity of Salmonella isolated from retail ready-to-eat foods in China. Food Control, 2016, 60: 50-56. DOI:10.1016/j.foodcont.2015.07.019 |
| [3] | Coburn B, Grassl GA, Finlay BB. Salmonella, the host and disease: a brief review. Immunology & Cell Biology, 2007, 85(2): 112-118. |
| [4] | Hu YJ, He YY, Wang YR, Fanning S, Cui SH, Chen Q, Liu GH, Chen QX, Zhou G, Yang BW, Huang JL, Li FQ. Serovar diversity and antimicrobial resistance of non-typhoidal Salmonella enterica recovered from retail chicken carcasses for sale in different regions of China. Food Control, 2017, 81: 46-54. DOI:10.1016/j.foodcont.2017.05.031 |
| [5] | Ma XQ, Chen LA. Advances in detection of bacterial drug resistance. Academic Journal of Chinese PLA Medical School, 2015, 36(4): 404-407. (in Chinese) 马秀清, 陈良安. 细菌耐药性检测方法的研究进展. 解放军医学院学报, 2015, 36(4): 404-407. DOI:10.3969/j.issn.2095-5227.2015.04.027 |
| [6] | Liu JJ, Zhang W, Wang YL, Wang XC, Wei JZ, Sun P, Li Y. Establishment of multiplex PCR detective methods for the resistance genes of Salmonella. Chinese Journal of Antibiotics, 2013, 38(11): 871-875. (in Chinese) 刘军军, 张玮, 王元兰, 王星晨, 魏建忠, 孙裴, 李郁. 沙门菌耐药基因多重PCR检测方法的建立. 中国抗生素杂志, 2013, 38(11): 871-875. DOI:10.3969/j.issn.1001-8689.2013.11.016 |
| [7] | Gordon NC, Price JR, Cole K, Everitt R, Morgan M, Finney J, Kearns AM, Pichon B, Young B, Wilson DJ, Llewelyn MJ, Paul J, Peto TEA, Crook DW, Walker AS, Golubchik T. Prediction of Staphylococcus aureus antimicrobial resistance by whole-genome sequencing. Journal of Clinical Microbiology, 2014, 52(4): 1182-1191. DOI:10.1128/JCM.03117-13 |
| [8] | Moradigaravand D, Palm M, Farewell A, Mustonen V, Warringer J, Parts L. Prediction of antibiotic resistance in Escherichia coli from large-scale pan-genome data. PLoS Computational Biology, 2018, 14(12): e1006258. DOI:10.1371/journal.pcbi.1006258 |
| [9] | McArthur AG, Waglechner N, Nizam F, Yan A, Azad MA, Baylay AJ, Bhullar K, Canova MJ, De Pascale G, Ejim L, Kalan L, King AM, Koteva K, Morar M, Mulvey MR, O'Brien JS, Pawlowski AC, Piddock LJV, Spanogiannopoulos P, Sutherland AD, Tang I, Taylor PL, Thaker M, Wang WL, Yan M, Yu T, Wright GD. The comprehensive antibiotic resistance database. Antimicrobial Agents and Chemotherapy, 2013, 57(7): 3348-3357. DOI:10.1128/AAC.00419-13 |
| [10] | Wattam AR, Davis JJ, Assaf R, Boisvert S, Brettin T, Bun C, Conrad N, Dietrich EM, Disz T, Gabbard JL, Gerdes S, Henry CS, Kenyon RW, Machi D, Mao CH, Nordberg EK, Olsen GJ, Murphy-Olson DE, Olson R, Overbeek R, Parrello B, Pusch GD, Shukla M, Vonstein V, Warren A, Xia FF, Yoo H, Stevens RL. Improvements to PATRIC, the all-bacterial bioinformatics database and analysis resource center. Nucleic Acids Research, 2016, 45(D1): D535-D542. |
| [11] | Zankari E, Hasman H, Cosentino S, Vestergaard M, Rasmussen S, Lund O, Aarestrup FM, Larsen MV. Identification of acquired antimicrobial resistance genes. Journal of Antimicrobial Chemotherapy, 2012, 67(11): 2640-2644. DOI:10.1093/jac/dks261 |
| [12] | Jacobsen A, Hendriksen RS, Aaresturp FM, Ussery DW, Friis C. The Salmonella enterica pan-genome. Microbial Ecology, 2011, 62(3): 487. DOI:10.1007/s00248-011-9880-1 |
| [13] | Liu Y, Zhang DF, Zhou XJ, Xu L, Zhang LD, Shi XM. Comprehensive analysis reveals two distinct evolution patterns of Salmonella flagellin gene clusters. Frontiers in Microbiology, 2017, 8: 2604. DOI:10.3389/fmicb.2017.02604 |
| [14] | Seminati C, Mejia W, Mateu E, Martin M. Mutations in the quinolone-resistance determining region (QRDR) of Salmonella strains isolated from pigs in Spain. Veterinary Microbiology, 2005, 106(3/4): 297-301. |
| [15] | He YC, Zhou XJ, Chen ZY, Deng XY, Gehring A, Ou HY, Zhang LD, Shi XM. PRAP: pan resistome analysis pipeline. BMC Bioinformatics, 2020, 21(1): 20. DOI:10.1186/s12859-019-3335-y |
| [16] | Vernikos G, Medini D, Riley DR, Tettelin H. Ten years of pan-genome analyses. Current Opinion in Microbiology, 2015, 23: 148-154. DOI:10.1016/j.mib.2014.11.016 |
| [17] | Michael GB, Butaye P, Cloeckaert A, Schwarz S. Genes and mutations conferring antimicrobial resistance in Salmonella: an update. Microbes and Infection, 2006, 8(7): 1898-1914. DOI:10.1016/j.micinf.2005.12.019 |
| [18] | Ramirez MS, Tolmasky ME. Aminoglycoside modifying enzymes. Drug Resistance Updates, 2010, 13(6): 151-171. DOI:10.1016/j.drup.2010.08.003 |
| [19] | Kawasaki K. Complexity of lipopolysaccharide modifications in Salmonella enterica: its effects on endotoxin activity, membrane permeability, and resistance to antimicrobial peptides. Food Research International, 2012, 45(2): 493-501. DOI:10.1016/j.foodres.2011.01.026 |
| [20] | Delmar JA, Su CC, Yu EW. Bacterial multidrug efflux transporters. Annual Review of Biophysics, 2014, 43: 93-117. DOI:10.1146/annurev-biophys-051013-022855 |
| [21] | Lima NCB, Tanmoy AM, Westeel E, de Almeida LGP, Rajoharison A, Islam M, Endtz HP, Saha SK, de Vasconcelos ATR, Komurian-Pradel F. Analysis of isolates from Bangladesh highlights multiple ways to carry resistance genes in Salmonella Typhi. BMC Genomics, 2019, 20(1): 530. |
| [22] | Laing CR, Whiteside MD, Gannon VPJ. Pan-genome analyses of the species Salmonella enterica, and identification of genomic markers predictive for species, subspecies, and serovar. Frontiers in Microbiology, 2017, 8: 1345. DOI:10.3389/fmicb.2017.01345 |
| [23] | Liao JQ, Orsi RH, Carroll LM, Kovac J, Ou HY, Zhang HL, Wiedmann M. Serotype-specific evolutionary patterns of antimicrobial-resistant Salmonella enterica. BMC Evolutionary Biology, 2019, 19(1): 132. DOI:10.1186/s12862-019-1457-5 |
| [24] | Wang XD. Research on the harm of veterinary drug residues in pork and the prevention and control countermeasures. The Chinese Livestock and Poultry Breeding, 2020, 16(3): 128-129. (in Chinese) 王晓丹. 猪肉中兽药残留的危害及防控对策探究. 中国畜禽种业, 2020, 16(3): 128-129. |
| [25] | Dong XJ. Analysis of the application of antibiotics in the prevention and treatment of swine diseases. Friends of the Farmers to Get Rich, 2019(11): 152. (in Chinese) 董晓驹. 抗生素药物在猪病防治中的应用分析. 农民致富之友, 2019(11): 152. |
| [26] | Denagamage TN, Wallner-Pendleton E, Jayarao BM, Xiaoli LZ, Dudley EG, Wolfgang D, Kariyawasam S. Detection of CTX-M-1 extended-spectrum beta-lactamase among ceftiofur-resistant Salmonella enterica clinical isolates of poultry. Journal of Veterinary Diagnostic Investigation, 2019, 31(5): 681-687. |
| [27] | Fang LX, Jiang Q, Deng GH, He B, Sun RY, Zhang JF, Cen DJ, Miao YY, Wang D, Guo WY, Sun J, Liu YH, Liao XP. Diverse and flexible transmission of fosA3 associated with heterogeneous multidrug resistance regions in Salmonella enterica serovar typhimurium and Indiana isolates. Antimicrobial Agents and Chemotherapy, 2020, 64(2): e02001-19. |
