肖建龙1, 张斯童1, 孙晓仲2, 陈光1


1. 吉林农业大学生命科学学院, 吉林 长春 130118;
2. 吉林省产品质量监督检验院, 吉林 长春 130022
收稿日期:2019-08-14;修回日期:2019-11-03;网络出版日期:2020-01-21
基金项目:国家重点研发计划(2017YFD0501005)
*通信作者:陈光, E-mail:chg61@163.com.
摘要:[目的] 为了获得高产木素过氧化物酶酿酒酵母工程菌。[方法] 本研究从黄孢原毛平革菌中克隆了木素过氧化物酶(lignin peroxidases,LiP)基因,全长2193 bp,编码371个氨基酸,并与来源于酿酒酵母的PGK启动子序列、来源于pPIC9K质粒的α-信号肽序列以及来源于pSH65质粒的CYC1终止子序列通过重叠延伸PCR构建完整表达盒(PαLiC),利用rDNA整合法构建木素过氧化物酶酿酒酵母表达载体,实现木素过氧化物酶在酿酒酵母中的多拷贝表达。利用数字微滴PCR技术对拷贝数进行鉴定,探究拷贝数与蛋白表达量之间的关系。[结果] 通过rDNA整合法得到拷贝数为1、2、4、5、6、7、8、9、10、11、12和13的木素过氧化物酶酿酒酵母工程菌,通过对其酶活测定,表明当拷贝数为7时,酶活力最高,为367 U/L。[结论] 本研究在酿酒酵母中表达了木素过氧化物酶,研究了其基因拷贝数与酶活性的关系,对木质素降解技术的发展具有重要意义。
关键词:酿酒酵母木素过氧化物酶rDNA整合法微滴数字PCR技术
Construction of an engineered Saccharomyces cerevisiae for lignin peroxidase production
Jianlong Xiao1, Sitong Zhang1, Xiaozhong Sun2, Guang Chen1


1. College of Life Sciences, Jilin Agricultural University, Changchun 130118, Jilin Province, China;
2. Jilin Province Product Quality Supervision and Inspection Institute, Changchun 130022, Jilin Province, China
Received: 14 August 2019; Revised: 3 November 2019; Published online: 21 January 2020
*Corresponding author: Guang Chen, E-mail:chg61@163.com.
Foundation item: Supported by the National Key R & D Program of China (2017YFD0501005)
Abstract: [Objective] To obtain engineered Saccharomyces cerevisiae with high-yield of lignin peroxidase. [Methods] We cloned a constitutive promoter PGK, an exogenous protein secretion signal peptide α-factor, lignin peroxidases (LiP) genes and a terminator CYC1. We constructed complete expression box (PαLC) by overlap extension PCR method. Then, we established expression vector of lignin peroxidase S. cerevisiae via rDNA integration method, to achieve multi-copy expression of lignin peroxidase in S. cerevisiae. Then, we identified the copy number via droplet digital PCR technology, to explore the relationship between the copy number and protein expression amount. [Results] We got the engineered strain of S. cerevisiae to produce lignin peroxidase with the copy number of 1, 2, 4, 5, 6, 7, 8, 9, 10, 11, 12 and 13 via rDNA integration method, through enzymatic activity determination, it shows that the enzymatic activity is as highest as 367 U/L when the copy number is 7. [Conclusion] In this study, lignin peroxidase was expressed in S. cerevisiae, and the relationship between gene copy number and enzyme activity was studied, which is of great significance to the development of lignin degradation technology.
Keywords: Saccharomyces cerevisiaelignin peroxidaserDNA integration methoddroplet digital PCR technology
木素过氧化物酶(lignin peroxidases,LiP,EC1.11.1.14),是一种过氧化物酶,在木质素降解中起关键作用[1],其主要优点是无底物特异性,因此LiP在生物制浆、木质纤维素降解、治理环境污染等方面有很大的应用潜力[2]。Xin等[2]将Phanerochaete chrysosporium发酵液中的LiP使用化学方法进行了提取,并研究了最佳提取工艺。但是由于自然界中Phanerochaete chrysosporium等菌中的LiP酶活一般较低,化学提取的方法并不能解决LiP产量低下的问题,而且Phanerochaete chrysosporium本身生理代谢的特殊性以及对培养条件苛刻的要求严重制约了木素过氧化物酶(LiP)的工业化生产,所以对LiP进行高效异源表达是解决这一问题的有效途径。
近年来对于LiP的研究越来越多,但是LiP在真菌中异源表达的研究却非常少,2009年Wang等[3]将来源于Phanerochaete chrysosporium的木质素过氧化物酶基因(LiP)在毕赤酵母X-33中进行了异源表达,诱导12 h后LiP酶活力最高达到15 U/L。2015年刘金雷等[4]将来源于Phanerochaete chrysosporium的木质素过氧化物酶基因(LiP)在毕赤酵母中成功表达,但是没有对LiP的酶活力进行计算,而是以转化子对木质素降解效果为指标进行了筛选。
但是毕赤酵母表达体系缺陷在于需要不断添加甲醇诱导表达,与之相比,酿酒酵母属于食品微生物[5],不仅在改造时易于操作,表达时也不需要有毒物质进行诱导,更不会产生有毒害的副产物。目前还没有以酿酒酵母S. Cerevisiae INVSc1为表达宿主对木质素过氧化物酶基因(LiP)进行异源表达的研究,尤其可将酿酒酵母中特有的rDNA序列作为酿酒酵母整合型表达载体的同源重组位点,可以有效提高外源基因在酿酒酵母基因组中的拷贝数,外源基因在酿酒酵母细胞内的表达会由于剂量效应而实现高效表达[6]。
本研究为了构建高产木素过氧化物酶酿酒酵母工程菌,将组成型启动子PGK、分泌信号肽α-factor、木素过氧化物酶基因和终止子CYC1利用重叠延伸PCR技术组合成表达盒(PαLiC),并利用rDNA整合法构建整合型酿酒酵母表达载体,实现木素过氧化物酶在酿酒酵母中的多拷贝表达,利用ddPCR技术测定成功转化的酿酒酵母工程菌拷贝数,并探究基因拷贝数与蛋白表达量之间的关系。期望通过启动子的改造和信号肽的引入以及基因剂量的提高实现木素过氧化物酶在酿酒酵母中的高效表达。
1 材料和方法 1.1 材料与试剂 本研究用到的菌种和质粒如表 1所示。
表 1. 所用菌株和质粒 Table 1. Strains and plasmids used in this study
| Strains and plasmids | Features | Sources |
| Escherichia coli DH5α | Gene cloning host | TaKaRa corporation of Japan |
| Phanerochaete chrysosporium | Cloning of LiP Gene | China industrial microbial species preservation center |
| S. cerevisiae INVSc1 | MATa, his3, leu2, trp1, ura3, expressing host | American invitrogen company |
| S. cerevisiae INVSc1-pYES2-LiP | Free single-copy lignin peroxidase transformant from Saccharomyces cerevisiae | Pre-construction of laboratory |
| pYES2 | ura3 Saccharomyces cerevisiae expression vector | American invitrogen company |
| ppic9k | Amplification of α-factor signal peptide sequence | China plasmid strain preservation center (NTCC) |
| pMD19-T | Gene cloning vector | TaKaRa corporation of Japan |
| pSH65 | Amplification of CYC1 terminator sequences | China plasmid strain preservation center (NTCC) |
| pYES2-PαLiC | Constitutive single-copy expression vector of lignin peroxidase | This study |
| pYES2-PαLiC-rDNA | Constitutive multi-copy lignin peroxidase expression vector | This study |
表选项
去磷酸化试剂盒、反转录试剂盒、限制性内切酶(EcoR I、Not I、Xba I、SnaB I等)、T4连接酶、SD-Ura培养基等购自日本TaKaRa公司;Salmon Sperm DNA上海源叶生物科技有限公司,质粒提取试剂盒、聚合酶链式反应(polymerase chain reaction,PCR)产物纯化试剂盒购自生工生物工程(上海)股份有限公司;酵母基因组提取试剂盒购自美国OMEGA公司;ddPCR Eva Green Supermix、Droplet reader oil购自美国BIO-RAD公司;总RNA提取试剂盒购自美国AXYGEN公司;其他试剂均为国产分析纯。
1.2 引物和探针 本文所用到的引物均由生工生物工程(上海)股份有限公司合成,具体列表如表 2所示。
表 2. 引物和探针 Table 2. Primers and probes
| Primer | Sequence (5′→3′) | Annotation |
| LiP-F | CCCGGGATGGTGCTCATCCAGCG | Amplification of LiP gene |
| LiP-R | AAGCTTGATTCGGCCCGGCCGAG | Amplification of LiP gene |
| PGK-F | CCGGAATTCAGCTTTCTAACTGATCTATCCAAAAC | EcoR I, for amplification of PGK sequences |
| PGK-R | GCTGCCTTGATCTGAACCATTGTTTTATATTTGTTGTAA AAAGTAGA | Amplification of PGK sequences |
| α-F | TCTACTTTTTACAACAAATATAAAACAATGAGATTTCCT TCAATTTTTACTGC | Amplification of α-factor sequences |
| α-R | TGTCGATGCTCACTGAAGCCTGTCTTTTCTCGAGAGATA CCCCTT | Amplification of α-factor sequences |
| CYC1-F | ACACTGCTATCGCAAATGCTCTCTAGGTCATGTAATTAG TTATGTCACGC | Amplification of CYC1 sequence |
| CYC1-R | TGCTCTAGACGGCCGCAAATTAAAGCCTT | Xba I for amplification of CYC1 sequence |
| rDNA-F | CCTACGTACAACGAACGAGACCTTAACCT | SnaB I for amplification of rDNA sequences |
| rDNA-R | GGTACGTACGGAACCTCTAATCATTCGCT | SnaB I for amplification of rDNA sequences |
| ACT1-F | TATCCCCTGCATCCCTATCA | ACT1 core amplification sequence (in ddPCR) |
| ACT1-R | CAGGCTTCGTTGCAGATACA | ACT1 core amplification sequence (in ddPCR) |
| ACT1-P | CATCTTCGTTAGCTTCATCCGACGCTA | ACT1 Probe sequence |
| LiP-F | TGCGGCATGCATTTCCT | LiP core amplification sequence (in ddPCR) |
| LiP-R | TTAACATCACAGCCGATTCTG | LiP core amplification sequence (in ddPCR) |
| LiP-P | ACGAGCTCTCTCTGCAGCCCCCC | LiP Probe sequence |
表选项
1.3 PGK启动子序列、α-factor序列、LiP基因、CYC1终止子序列克隆
1.3.1 PGK启动子序列克隆:: 将酿酒酵母INVSc1菌种经YPD固体培养基活化后,用OMEGA酵母基因组提取试剂盒提取酿酒酵母基因组[7]。以提取的酿酒酵母基因组为模板,PGK-F、PGK-R为引物扩增PGK启动子序列,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 7 min。琼脂糖凝胶电泳后,切胶回收并纯化目的条带。
1.3.2 α-factor序列克隆:: 将含有pPIC9K质粒的大肠杆菌经LB平板活化后,挑取单菌落至LB液体培养基中,12 h后用质粒提取试剂盒提取pPIC9K质粒。以pPIC9K质粒为模板,α-F、α-R为引物扩增α-factor序列,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 7 min。琼脂糖凝胶电泳后,切胶回收并纯化目的条带。
1.3.3 LiP基因克隆:: 将黄孢原毛平革菌菌种经平板划线后,挑取单菌落于发酵培养基,30 ℃、150 r/min培养72 h后,收集菌体,用AXYGEN总RNA提取试剂盒提取RNA。以提取的黄孢原毛平革菌总RNA为模板,用一步法反转录试剂盒合成cDNA,以cDNA为模板,LiP-F、LiP-R为引物扩增LiP基因,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 130 s,30个循环;72 ℃ 7 min。琼脂糖凝胶电泳后,切胶回收并纯化目的条带。
1.3.4 CYC1终止子序列克隆:: 将含有pSH65质粒的大肠杆菌经LB平板活化后,挑取单菌落至LB液体培养基中,12 h后用质粒提取试剂盒提取pSH65质粒。以pSH65质粒为模板,CYC1-F、CYC1-R为引物扩增CYC1序列,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 7 min。琼脂糖凝胶电泳后,切胶回收并纯化目的条带。
1.4 克隆载体的连接、转化、鉴定 将1.3.1–1.3.4中的回收产物分别于pMD19-T载体进行连接[8],转化感受态大肠杆菌DH5α,接种到含有Amp的LB抗性培养基上进行筛选。12 h后挑取单克隆进行菌落PCR鉴定,挑取阳性克隆于含有Amp抗性的LB液体培养基37 ℃、150 r/min培养12 h后离心收集菌体,提取质粒并送至苏州金唯智有限公司测序。
1.5 重叠延伸PCR构建PαLiC完整表达盒 将1.3.1–1.3.4中回收得到的PGK、α-factor、LiP及CYC1片段,等比例混合后进行片段融合预实验[9],确定其最佳融合条件中退火温度为59 ℃,延伸时间为50 s,循环数为35,各个片段稀释8倍后使用,扩增条件为95 ℃ 5 min;94 ℃ 30 s,59 ℃ 30 s,72 ℃ 50 s,35个循环;72 ℃ 7 min。
以此融合实验的产物为模板,以PGK-F和CYC1-R为引物,扩增PGK、α-factor、LiP及CYC1全长的融合片段,称为PGK-α-factor-LiP-CYC1 (PαLiC)表达盒,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 210 s,30个循环;72 ℃ 7 min。琼脂糖电泳凝胶回收目的序列,连接pMD19-T载体,命名为pMD19-T-PαLiC,转化感受态大肠杆菌DH5α后,经菌液PCR检测,提取质粒送至苏州金唯智有限公司测序。
1.6 rDNA序列扩增 以1.3.1中提取的酿酒酵母基因组为模板,以rDNA-F、rDNA-R为引物扩增rDNA单元中的核心元件序列,扩增条件为95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 150 s,30个循环;72 ℃ 7 min。将回收产物与pMD19-T载体连接,命名为pMD19-T-rDNA,转化大肠杆菌DH5α,接种到含有Amp抗性的LB平板上筛选。10–12 h后挑取单菌落进行菌落PCR鉴定,选取阳性克隆于含有Amp抗性的LB液体培养基37 ℃、150 r/min培养12 h后离心收集菌体,提取质粒并送至苏州金唯智有限公司测序。
1.7 pYES2-PαLiC载体的构建 在上述构建PαLiC完整表达盒时,在上下游引物中引入了EcoR I和Xba I酶切位点,所以用EcoR I和Xba I分别对pYES2载体和pMD19-T- PαLiC质粒进行双酶切,酶切后回收目的条带,经T4连接酶16 ℃过夜连接,连接体系全量转化感受态大肠杆菌DH5α,经菌液PCR检测,将阳性转化子接种于含Amp的LB液体培养基中,摇瓶过夜,提取质粒,送至苏州金唯智有限公司测序。
1.8 pYES2-PαLiC-rDNA载体的构建 克隆rDNA片段时上下游引物引入了SnaB I酶切位点,对载体pYES2-PαLiC和pMD19-T-rDNA分别用SnaB I进行单酶切,对回收的pYES2- PαLiC片段进行去磷酸化处理后,经T4连接酶16 ℃过夜连接,连接体系全部用于转化感受态大肠杆菌DH5α,经菌液PCR检测,将阳性转化子接种于含Amp的LB液体培养基中,摇瓶过夜,提取质粒酶切鉴定,命名为pYES2-PαLiC-rDNA。
1.9 线性化pYES2-PαLiC-rDNA载体转化酿酒酵母 用BstE Ⅱ在rDNA位置对pYES2-PαLiC-rDNA进行酶切,使质粒iC-rDNA载体转线性化。经琼脂糖凝胶回收后,用Li Ac/SS carrier DNA/PEG法转化酿酒酵母[10-12]。转化体系如表 3所示。
表 3. 转化体系 Table 3. Conversion system
| Reagent | Sample addition/μL |
| Sensitive Saccharomyces cerevisiae | 50 |
| 50% PEG3350 | 240 |
| 1 mol/L LiAc | 36 |
| 2 mg/mL boiled carrier DNA | 50 |
| Plasmid DNA | 34 |
表选项
经42 ℃孵育后,将细胞重悬于1 mL YPD液体培养基中,30 ℃、150 r/min培养1 h,收集菌体,用100 μL ddH2O重悬菌体后,将其涂布到SD-Ura平板,培养箱30 ℃培养直至出现单菌落。对挑取的单菌落进行LiP的菌液PCR验证。
1.10 微滴数字PCR鉴定阳性转化子拷贝数 将得到的阳性转化子接种于YPD培养基中,30 ℃、150 r/min培养48 h,收集菌体使用美国OMEGA公司的酵母基因组提取试剂盒对各转化子基因组进行提取。以基因组为模板,ATC1基因为内参,利用BIO-RAD QX200数字液滴PCR进行LiP基因拷贝数鉴定[13-14]。引物探针序列如表 2所示。内参基因荧光标记基团为HEX,目的基因为FAM。
微滴数字PCR反应包括配制体系、生成微滴、扩增循环和信号读取4个步骤。微滴数字PCR体系为20 μL,包括10 μL 2×dd PCR Master Mix,10 μmol/L内参基因上游和下游引物各1.8 μL,10 μmol/L目的基因上游和下游引物各1.8 μL,探针各0.5 μL,基因组模板2 μL。微滴的生成需要专用的微滴生成卡和微滴生成仪。将20 μL PCR体系加入微滴生成卡中,并加入专用的微滴生成油后覆盖专用胶垫置入微滴生成仪,启动程序,生成微滴。微滴数字PCR扩增使用两步法,设置程序如下:94 ℃ 10 min;94 ℃ 30 s,55 ℃ 60 s,45个循环,扩增结束后进行98 ℃、10 min的热失活。每个模板重复3个平行检测。扩增结束后,将96孔板置入微滴读取仪中读取荧光信号,并使用软件Quanta Soft V1.3.2分析实验数据,获得绝对定量结果[15-16]。
1.11 转化子产酶能力测定 将1.10中微滴数字PCR鉴定得到的不同拷贝数的阳性转化子接种至YPD培养基中,30 ℃、150 r/min培养72 h后,取上清,测定木素过氧化物酶的酶活力,并与实验室前期构建的游离型单拷贝木素过氧化物酶酿酒酵母转化子S. cerevisiae INVSc1-pYES2-LiP(SPL)酶活力进行比较。酶活力测定具体方法见文献[17]。
1.12 高酶活转化子产酶稳定性实验 将1.11中得到的产酶活力最高的阳性转化子接种于YPD培养基中,连续传代10次。每次传代均在30 ℃、150 r/min培养72 h后,取上清测定木素过氧化物酶的酶活力。
2 结果和分析 2.1 PGK启动子序列、α-factor序列克隆、LiP基因序列、CYC1终止子序列克隆及PαLiC表达盒构建 以提取的酿酒酵母基因组DNA为模板,PGK-F、PGK-R为引物扩增,得到预期大小的目的片段,大小为850 bp (GenBank登记号AH001380)。以pPIC9K质粒为模板,α-F、α-R为引物扩增,得到预期大小的目的片段,大小为254 bp (GenBank登记号KM032189)。以提取的黄孢原毛平革菌总RNA为模板,用一步法反转录试剂盒合成cDNA,以cDNA为模板,以LiP-F、LiP-R为引物扩增得到LiP目的片段,大小为2193 bp (GenBank登记号M92644.1)。以pSH65质粒为模板,CYC1-F、CYC1-R为引物扩增,得到预期大小的目的片段,大小为260 bp (GenBank登记号AF298780.1)。扩增产物大小均与预期相符。将上述基因片段分别于pMD19-T载体连接,转化感受态大肠杆菌DH5α,菌液PCR验证后提取质粒送至公司测序,将测序结果与GenBank对比,相似度100%,表明序列未发生碱基突变。
在进行PGK、α-factor、LiP和CYC1序列扩增时,设计的引物是重叠延伸PCR的引物,每个序列下游均带有与下一序列的融合位点,利用重叠延伸PCR技术将组成型启动子PGK、分泌信号肽α-factor、LiP基因、终止子CYC1片段融合构建PαLiC表达盒,得到预期大小的目的片段,大小为3557 bp。如图 1所示。
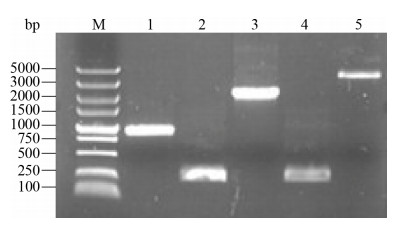 |
| 图 1 PGK启动子、α-factor信号肽、LiP基因、CYC1终止子及PαLiC表达盒PCR扩增 Figure 1 PGK promoter, α-factor signal peptide, LiP gene, CYC1 terminator and PαLiC expression cassette PCR amplification. M: DL5000 marker; lane 1: PGK promoter; lane 2: α-factor signal peptide; lane 3: LiP gene; lane 4: CYC1 terminator; lane 5: PαLiC expression cassette |
| 图选项 |
2.2 表达载体pYES2-PαLiC的构建 用EcoR I和Xba I分别对大小为5864 bp的pYES2载体和pMD19-T-PαLiC质粒进行双酶切, 回收目的片段后,T4连接酶16 ℃过夜连接后转化感受态大肠杆菌DH5α,经LiP菌液PCR鉴定得到质粒pYES2- PαLiC,如图 2所示。
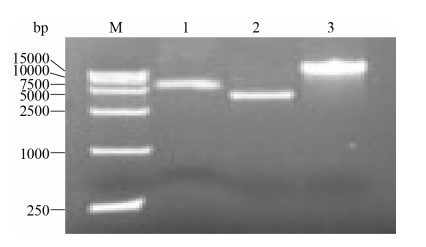 |
| 图 2 质粒pYES2-PαLiC的构建 Figure 2 Construction of plasmid pYES2-PαLiC. M: DL15000 marker; lane 1: 5864 bp pYES2 expression vector; lane 2: PαLiC expression cassette; lane 3: pYES2-PαLiC vector |
| 图选项 |
2.3 表达载体pYES2-PαLiC-rDNA的构建 以提取的酿酒酵母基因组DNA为模板,利用特异性引物rDNA-F、rDNA-R扩增,得到预期大小的目的片段,大小为2300 bp (GenBank登录号:BK006945.2)。将rDNA基因片段与pMD19-T载体连接,转化感受态大肠杆菌DH5α,菌液PCR验证后提取质粒送至公司测序。将测序结果与GenBank进行对比,相似度100%,表明序列未发生碱基突变。
用SnaB Ⅰ分别酶切质粒pYES2-PαLiC和质粒pMD19-T-rDNA,回收目的片段,T4连接酶16 ℃过夜连接后转化感受态大肠杆菌DH5α,菌液PCR验证后,提取质粒酶切验证,结果如图 3所示,rDNA片段大小为2300 bp;pYES2-PαLiC载体大小为9421 bp;pYES2-PαLiC-rDNA载体大小为11721 bp。
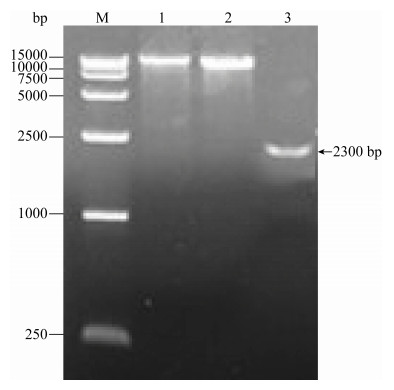 |
| 图 3 质粒pYES2-PαLiC-rDNA的构建 Figure 3 Construction of plasmid pYES2-PαLiC- rDNA. M: DL15000 marker; lane 1: pYES2-PαLiC- rDNA vector; lane 2: pYES2-PαLiC vector; lane 3: rDNA |
| 图选项 |
2.4 转化子拷贝数的dd PCR检测结果 使用BstE Ⅱ在rDNA位置对pYES2-PαLiC- rDNA线性化后,转化酿酒酵母INVSc1,对经SD-Ura培养基筛选得到的转化子提取基因组后,使用dd PCR检测转化子拷贝数。图 4显示在2个检测通道下,仪器检测到的阳性微滴数和阴性微滴数的分布图,图中阳性和阴性微滴分布区分明显,仪器可准确判断出阳性和阴性微滴数[18]。
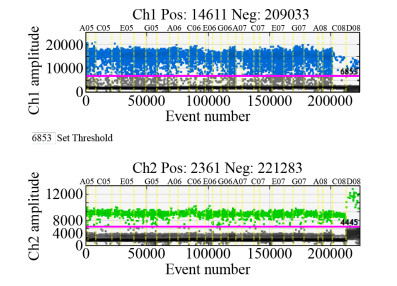 |
| 图 4 dd PCR鉴定转化子拷贝数 Figure 4 Detection of copy number of transformants by dd PCR |
| 图选项 |
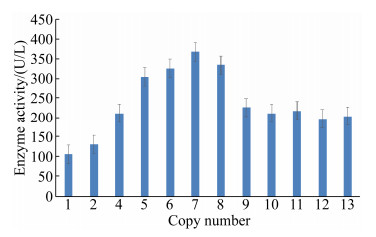
根据软件读取的各转化子LiP基因和ACT1基因的浓度,得出每个转化子中目的基因LiP的拷贝数,如图 5。dd PCR分析显示,利用rDNA整合法,成功获得了1、2、4、5、6、7、8、9、10、11、12和13共12种不同拷贝数的转化子。
 |
| 图 5 dd PCR测定转化子拷贝数结果 Figure 5 Identification of the copy number of converters by digital microdrop polymerase chain reaction |
| 图选项 |
2.5 不同拷贝数转化子产木素过氧化物酶活力分析 将12株不同拷贝数的转化子菌株,通过摇瓶发酵培养,每隔12 h取发酵液测定木素过氧化物酶活力,其不同拷贝数转化子最高酶活力与游离型单拷贝木素过氧化物酶酿酒酵母转化子(SPL,将质粒pYES2-PαLiC以游离的形式转化酿酒酵母S. cerevisiae INVSc1后得到的阳性转化子)进行比较,利用rDNA整合法得到的1拷贝木素过氧化物酶酿酒酵母转化子酶活力是SPL的2倍,证明整合到酿酒酵母基因组上的质粒与游离型相比,具有更高的表达效率。
如图 6所示,在拷贝数与酶活力关系方面,当LiP拷贝数小于7时,酶活力随拷贝数增加而提高。当拷贝数为7时,酶活力最高为367 U/L,是1拷贝的3.46倍。当拷贝数大于7时,酶活力随拷贝数的增加而下降。当拷贝数大于10时,酶活力基本趋于稳定。
 |
| 图 6 拷贝数与酶活力之间的关系 Figure 6 The relationship between copy number and enzyme activity |
| 图选项 |
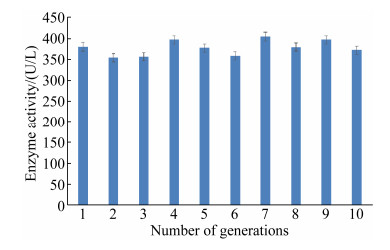
2.6 转化子产酶稳定性实验 对得到的酶活力最高的7拷贝转化子,进行遗传稳定性验证,结果如图 7,经过10次传代,每代酶活力与初代相比差距均在5%以内,所以利用rDNA整合法获得的木素过氧化物酶转化子产酶稳定性良好,经多次传代后仍能保持稳定遗传。
 |
| 图 7 传代稳定性验证 Figure 7 Progeny stability verification |
| 图选项 |
3 讨论和结论 提高外源蛋白表达量的措施有很多,例如选择具有较高活性的组织特异性启动子、适合外源基因表达系统的信号肽以及实现基因的剂量表达。
启动子是表达载体中一个重要元件,它的结构能影响它和RNA聚合酶的亲和力,从而影响外源基因表达的效率。Monfort等通过表达脂肪酶的量对酿酒酵母中各个启动子的相对强度进行比较,发现组成型PGK启动子的相对强度较高[19]。选择一个合适的启动子对外源基因的表达具有重要意义[20]。本研究在构建木素过氧化物酶表达载体时添加了组成型PGK启动子,使原本需在半乳糖培养基中诱导表达的转化子可以直接在YPD培养基中实现木素过氧化物酶的表达,且表达效率大幅提高。
上世纪70年代,Gunter Blobel等提出了信号肽假说,并在后续研究中被证明[21]。信号肽能引导一段肽链转移到内质网上折叠加工,随后信号肽被肽酶切除,蛋白质被释放到内质网腔中,最后由转运小泡运输到胞外,对于外源基因在宿主菌中的分泌表达具有重要意义。另外Ferrarese等研究表明,以酵母为表达宿主进行外源蛋白表达时,如果利用酵母蛋白信号肽引导,表达成功的外源蛋白都具有天然蛋白的活性,且与天然蛋白的分子量、等电点相同[22]。本研究在构建木素过氧化物酶表达载体时添加了酿酒酵母表达系统中适用的α-factor信号肽,成功实现木素过氧化物酶的胞外分泌表达。
限制外源基因表达水平的一个重要因素是外源基因拷贝数,基因的拷贝数的提高在一定范围内能有效提高外源蛋白的表达水平[12],超过这个范围,蛋白表达水平反而会随拷贝数的增加而降低[23-24],这是由于随着拷贝数的提高细胞内蛋白的翻译、内质网的折叠与加工压力不断增大,直到达到临界点后,一些肽链无法被正确折叠,从而不能得到更多有活性的蛋白,而且还会对菌体的生长产生影响[25],所以基因的拷贝数不是越高越好,需要通过筛选得到最适合的拷贝数。本研究构建的酿酒酵母木素过氧化物酶工程菌,利用微滴数字PCR检测转化子拷贝数,发现当其基因拷贝数为7时,其酶活力最高,就是由于拷贝数为7时,细胞内蛋白的翻译、折叠、加工压力达到最大效率,此时被正确折叠的肽链即有活力的蛋白表达量达到最高,所以酶活力最高。而当拷贝数进一步增加时,增加的肽链不能被正确折叠,堆积在细胞中,抑制菌体的生长,导致当基因拷贝数超过7时,酶活力反而下降。当拷贝数继续增大,未正确折叠的肽链对菌体生长的抑制达到最大,导致拷贝数大于10时,酶活力在较低的水平保持稳定,这也与文献[12]报道基本相同。
微滴式数字PCR (droplet digital PCR,dd PCR)是近年来兴起的一种新的基因绝对定量技术,它能将PCR的反应体系分割成上万个微滴,使每个微滴中含有一个独立的PCR体系[26],经PCR扩增后,逐个对每个微滴进行检测,以荧光信号判断每个微滴中是否发生PCR反应,根据泊松分布原理及阳性微滴的个数与所占总微滴的百分比即可得出靶分子的起始拷贝数或浓度[27],实现对基因的绝对定量。本研究利用微滴式数字PCR技术,以ATC1基因为内参,成功检测了酿酒酵母木素过氧化物酶阳性转化子的拷贝数。
本研究成功将PGK启动子、α-factor信号肽、LiP目的基因、CYC1终止子构成完整表达盒,构建酿酒酵母表达载体,利用rDNA整合法得到了酿酒酵母木素过氧化物酶工程菌,并通过微滴数字PCR检测其拷贝数,测定了不同拷贝数工程菌的酶活力,并与实验室前期构建的S. cerevisiae INVSc1-pYES2-LiP对比,证明整合到酿酒酵母基因组上的质粒与游离型相比,具有更高的表达效率。
且发现当LiP拷贝数小于7时,酶活力随拷贝数增加而提高。当拷贝数为7时,酶活力最高,为367 U/L,是1拷贝的3.46倍。当拷贝数大于7时,酶活力随拷贝数的增加而下降。当拷贝数大于10时,酶活力基本趋于稳定。
References
| [1] | Pham LTM, Eom MH, Kim YH. Inactivating effect of phenolic unit structures on the biodegradation of lignin by lignin peroxidase from Phanerochaete chrysosporium. Enzyme and Microbial Technology, 2014, 61-62: 48-54. |
| [2] | Peng X, Yuan XZ, Zeng GM, Huang HJ, Wang H, Liu H, Bao S, Ma YJ, Cui KL, Leng LJ, Xiao ZH. Synchronous extraction of lignin peroxidase and manganese peroxidase from Phanerochaete chrysosporium fermentation broth. Separation and Purification Technology, 2014, 123: 164-170. |
| [3] | Wang W, Wen XH. Expression of lignin peroxidase H2 from Phanerochaete chrysosporium by multi-copy recombinant Pichia strain. Journal of Environmental Sciences, 2009, 21(2): 218-222. |
| [4] | Liu JL, Liu TJ, Yu Y, Li TM, Yi H. Expression of lignin degrading enzyme system in Pichia pastoris and the activity of degrading lignin. Jiangsu Agricultural Sciences, 2015, 43(11): 58-62. (in Chinese) 刘金雷, 刘天佳, 于莹, 李天明, 仪宏. 木质素降解酶系在毕赤酵母中的表达及降解木质素的活性. 江苏农业科学, 2015, 43(11): 58-62. |
| [5] | Lu Y, Zhong H, Tang Q, Huang ZJ, Jing NN, Smith J, Miao RJ, Li YP, Yuan H. Construction and verification of CYP3A5 gene polymorphisms using a Saccharomyces cerevisiae expression system to predict drug metabolism. Molecular Medicine Reports, 2017, 15(4): 1593-1600. |
| [6] | James SA, West C, Davey RP, Dicks J, Roberts IN. Prevalence and dynamics of ribosomal DNA micro-heterogeneity are linked to population history in two contrasting yeast species. Scientific Reports, 2016, 6: 28555. |
| [7] | Qiao LX, Zhang SJ, Shi Y, Chen DX. A novel method for constructing recombinant plasmid of CCL3L1 tandem repeats via isocaudamer technology. Progress in Modern Biomedicine, 2012, 12(2): 239-241, 234. (in Chinese) 乔录新, 张世杰, 石英, 陈德喜. 基于同尾酶技术构建CCL3L1基因串联重组质粒的方法. 现代生物医学进展, 2012, 12(2): 239-241, 234. |
| [8] | Gao MZ, Zhu RY, Chen Y, Jin J. Construction and expression of eukaryotic plasmid with dual-promoters and dual-reporter genes. Chinese Journal of Biologicals, 2012, 25(9): 1130-1134. (in Chinese) 高明珠, 朱瑞宇, 陈蕴, 金坚. 双启动子双报告基因真核表达质粒的构建及表达. 中国生物制品学杂志, 2012, 25(9): 1130-1134. |
| [9] | 孙恒一.多拷贝人/鲑嵌合降钙素在酿酒酵母中表达优化及安全性评价研究.中国海洋大学博士学位论文, 2015. |
| [10] | Gietz RD, Woods RA. Yeast transformation by the LiAc/SS Carrier DNA/PEG method//Xiao W. Yeast Protocol. Totowa, NJ: Humana Press, 2006: 107-120. |
| [11] | Gao LL, Wang QH, Liang HC, Gong T, Yang JL, Zhu P. Construction of Saccharomyces cerevisiae haploid mutant deficient in lanosterol synthase gene. Acta Pharmaceutica Sinica, 2014, 49(5): 742-746. (in Chinese) 高丽丽, 王庆华, 梁会超, 巩婷, 杨金玲, 朱平. 酿酒酵母羊毛甾醇合酶基因单倍体缺陷型突变株的构建. 药学学报, 2014, 49(5): 742-746. |
| [12] | Lan X, Zhang ST, Li Z, Chang H, Sun Y, Wang G, Chen H, Wang CF, Chen G. Application of droplet digital PCR in screening of genetically modified Saccharomyces cerevisiae for multicopy expression of xylanase. Food Science, 2018, 39(10): 179-184. (in Chinese) 兰雪, 张斯童, 李哲, 常浩, 孙旸, 王刚, 陈欢, 王春凤, 陈光. 微滴数字PCR技术在多拷贝木聚糖酶酿酒酵母工程菌筛选中的应用. 食品科学, 2018, 39(10): 179-184. |
| [13] | Cavalli M, de Novi LA, Della Starza I, Cappelli LV, Nunes V, Pulsoni A, del Giudice I, Guarini A, Foà R. Comparative analysis between RQ-PCR and digital droplet PCR of BCL2/IGH gene rearrangement in the peripheral blood and bone marrow of early stage follicular lymphoma. British Journal of Haematology, 2017, 177(4): 588-596. |
| [14] | Srisutham S, Saralamba N, Malleret B, Rénia L, Dondorp AM, Imwong M. Four human Plasmodium species quantification using droplet digital PCR. PLoS One, 2017, 12(4): e0175771. |
| [15] | Arvia R, Sollai M, Pierucci F, Urso C, Massi D, Zakrzewska K. Droplet digital PCR (ddPCR) vs quantitative real-time PCR (qPCR) approach for detection and quantification of Merkel cell polyomavirus (MCPyV) DNA in formalin fixed paraffin embedded (FFPE) cutaneous biopsies. Journal of Virological Methods, 2017, 246: 15-20. |
| [16] | Suzawa K, Yamamoto H, Ohashi K, Hashida S, Tomida S, Kubo T, Maki Y, Soh J, Tsukuda K, Kiura K, Miyoshi S, Toyooka S. Optimal method for quantitative detection of plasma EGFR T790M mutation using droplet digital PCR system. Oncology Reports, 2017, 37(5): 3100-3106. |
| [17] | Tien M, Kirk TK. Lignin peroxidase of Phanerochaete chrysosporium. Methods in Enzymology, 1988, 161: 238-249. |
| [18] | Kline MC, Duewer DL. Evaluating droplet digital polymerase chain reaction for the quantification of human genomic DNA:lifting the traceability fog. Analytical Chemistry, 2017, 89(8): 4648-4654. |
| [19] | Monfort A, Finger S, Sanz P, Prieto JA. Evaluation of different promoters for the efficient production of heterologous proteins in baker's yeast. Biotechnology Letters, 1999, 21(3): 225-229. |
| [20] | Madhavan A, Pandey A, Sukumaran RK. Expression system for heterologous protein expression in the filamentous fungus Aspergillus unguis. Bioresource Technology, 2017, 245: 1334-1342. |
| [21] | Herrmann GF, Krezdorn C, Malissard M, Kleene R, Paschold H, Weusterbotz D, Kragl U, Berger EG, Wandrey C. Large-scale production of a soluble human β-1, 4-galactosyltransferase using a Saccharomyces cerevisiae expression system. Protein Expression and Purification, 1995, 6(1): 72-78. |
| [22] | Ferrarese L, Trainotti L, Gattolin S, Casadoro G. Secretion, purification and activity of two recombinant pepper endo-β-1, 4-glucanases expressed in the yeast Pichia pastoris. FEBS Letters, 1998, 422(1): 23-26. |
| [23] | Wu ZQ, Jia NB, Li N, Wang RJ, Ma BC, Wang CY, Li MG. Construction and functional analysis of yeast integrated vector. Bulletin of Biology, 2008, 43(5): 47-50. (in Chinese) 武志强, 贾耐兵, 李娜, 王瑞菊, 马百成, 王翠艳, 李明刚. 酵母整合型载体的构建及其功能分析. 生物学通报, 2008, 43(5): 47-50. |
| [24] | Cheng C, Xiong L, Li YH, Xu YH, Meng QS, Liu CG, Zhao XQ, Bai FW. Construction of mixed-sugar fermenting recombinant Saccharomyces cerevisiae and ethanol production from Jerusalem artichoke stalk by simultaneous saccharification and fermentation. Microbiology China, 2016, 43(7): 1411-1418. (in Chinese) 程诚, 熊亮, 李勇昊, 徐友海, 孟庆山, 刘晨光, 赵心清, 白凤武. 混合糖发酵重组酿酒酵母的菌株构建和菊芋秸秆同步糖化发酵研究. 微生物学通报, 2016, 43(7): 1411-1418. |
| [25] | Emmerstorfer A, Wimmer-Teubenbacher M, Wriessnegger T, Leitner E, Müller M, Kaluzna I, Schürmann M, Mink D, Zellnig G, Schwab H, Pichler H. Over-expression of ICE2 stabilizes cytochrome P450 reductase in Saccharomyces cerevisiae and Pichia pastoris. Biotechnology Journal, 2015, 10(4): 623-635. |
| [26] | Gerdes L, Iwobi A, Busch U, Pecoraro S. Optimization of digital droplet polymerase chain reaction for quantification of genetically modified organisms. Biomolecular Detection and Quantification, 2016, 7: 9-20. |
| [27] | Wang C, Ding Q, Plant P, Basheer M, Yang CC, Tawedrous E, Krizova A, Boulos C, Farag M, Cheng YF, Yousef GM. Droplet digital PCR improves urinary exosomal miRNA detection compared to real-time PCR. Clinical Biochemistry, 2019, 67: 54-59. |
