陈华枝#, 付中民#, 王杰, 祝智威, 范小雪, 蒋海宾, 范元婵, 周丁丁, 李汶东, 熊翠玲, 郑燕珍, 徐国钧, 陈大福, 郭睿


福建农林大学动物科学学院(蜂学学院), 福建福州 350002
收稿日期:2019-12-19;修回日期:2020-01-15;网络出版日期:2020-05-26
基金项目:国家自然科学基金(31702190);现代农业产业技术体系建设专项资金(CARS-44-KXJ7);福建省自然科学基金(2018J05042);福建省教育厅中青年教师教育科研项目(JAT170158);福建农林大学****科研人才计划(xjq201814);福建农林大学科技创新专项基金(CXZX2017342, CXZX2017343)
*通信作者:郭睿, Tel/Fax:+86-591-87640197;E-mail:ruiguo@fafu.edu.cn.
#并列第一作者。
摘要:[目的] 蜜蜂球囊菌(Ascosphaera apis,简称球囊菌)专性侵染蜜蜂幼虫而导致白垩病,危害蜜蜂健康和养蜂生产。本研究旨在探究中华蜜蜂(Apis cerana cerana,简称中蜂)6日龄幼虫响应球囊菌胁迫的环状RNA(circular RNA,circRNA)差异表达谱及差异表达circRNA(differentially expressed circRNA,DEcircRNA)在宿主胁迫应答中的潜在功能。[方法] 利用去除线性RNA的circRNA-seq技术对正常和球囊菌侵染的中蜂6日龄幼虫肠道(AcCK和AcT)进行测序。利用find_circ软件鉴定circRNA,统计circRNA的长度和环化类型。根据|log2(Fold change)|≥1和P≤0.05的标准筛选DEcircRNA。将DEcircRNA的来源基因比对Gene ontology(GO)数据库和Kyoto Encyclopedia of Genes and Genomes(KEGG)数据库,从而获得功能及通路(pathway)注释。随机挑选3个DEcircRNA进行RT-qPCR验证。[结果] AcCK和AcT的circRNA-seq分别得到76342570和68269362条原始读段(raw reads),经严格质控得到74524108和66974392条有效读段(clean reads),Q30分别为92.75%和94%,GC含量分别为54.31%和54.90%。比对上东方蜜蜂(Apis cerana)参考基因组的短序列读段(anchor reads)共计23648400条。AcCK和AcT中分别鉴定到805和702个circRNA,长度均介于201-1000 nt,数量最多的环化类型均为已注释外显子circRNA,但分布在不同长度、不同环化类型的circRNA数量存在差异。AcCK vs AcT比较组共有494个DEcircRNA,包括257个上调circRNA和237个下调circRNA;上调和下调幅度最大的circRNA分别为novel_circ_000123和novel_circ_000726。上述DEcircRNA的来源基因可注释到11条生物学进程相关条目,9条分子功能相关条目,9条细胞组分相关条目,以及73条通路。进一步分析发现,部分DEcircRNA的来源基因注释到7条细胞免疫通路和3条体液免疫通路。[结论] 中蜂6日龄幼虫响应球囊菌胁迫的过程中可能通过改变分布在不同长度和环化类型的circRNA数量,以及特异性表达一些circRNA和调节部分circRNA的表达量对病原产生应答;novel_circ_000027、novel_circ_000127、novel_circ_000312等DEcircRNA在宿主的胁迫应答过程中可能通过调控氧化磷酸化、细胞和体液免疫等通路发挥特殊作用。研究结果为深入理解中蜂幼虫对球囊菌的胁迫应答机制及二者的相互作用机制提供了新见解。
关键词:中华蜜蜂幼虫环状RNA非编码RNA蜜蜂球囊菌免疫应答
Circular RNA response of Apis cerana cerana 6-day-old larvae to Ascosphaera apis stress
Huazhi Chen#, Zhongmin Fu#, Jie Wang, Zhiwei Zhu, Xiaoxue Fan, Haibin Jiang, Yuanchan Fan, Dingding Zhou, Wendong Li, Cuiling Xiong, Yanzhen Zheng, Guojun Xu, Dafu Chen, Rui Guo


College of Animal Sciences(College of Bee Science), Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China
Received: 19 December 2019; Revised: 15 January 2020; Published online: 26 May 2020
*Corresponding author: Rui Guo. Tel/Fax: +86-591-87640197; E-mail: ruiguo@fafu.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31702190), by the Earmarked Fund for China Agriculture Research System (CARS-44-KXJ7), by the Fujian Natural Science Foundation Project (2018J05042), by the Education and Scientific Research Project for Young and Middle-Aged Teachers of Fujian Provincial Department of Education (JAT170158), by the Outstanding Young Research Talents Program of Fujian Agriculture and Forestry University (xjq201814) and by the Scientific and Technical Innovation Fund of Fujian Agriculture and Forestry University (CXZX2017342, CXZX2017343)
#Those authors contributed equally to this work.
Abstract: [Objective] Ascosphaera apis exclusively infects honeybee larvae, leading to chalkbrood, a fungal disease damaging honeybee health and beekeeping industry. The objective of this study was to investigate the differential expression pattern of circular RNAs (circRNAs) and differentially expressed circRNAs (DEcircRNAs) involved in Apis cerana cerana 6-day-old larval response to Ascosphaera apis stress, and the putative role of DEcircRNAs in host A. apis-response. [Methods] Normal and A. apis-infected 6-day-old larval guts of A. c. cerana (AcCK and AcT) were sequenced using linear RNA-removed circRNA-seq technology. CircRNAs were identified using find_circ software, followed by summary of length and circulization type of circRNAs. DEcircRNAs were screened out following the standard of |log2(Fold change)|≥1 and P≤0.05. Source genes of DEcircRNAs were annotated to Gene Ontology (Go) and Kyoto Encyclopedia of Genes and Genomes (KEGG) databases to gain function and pathway annotations. RT-qPCR was conducted to validate three randomly selected DEcircRNAs. [Results] Based on circRNA-seq, 76342570 and 68269362 raw reads were produced from AcCK and AcT, and 74524108 and 66974392 clean reads were obtained after strict quality control, with Q30 of 92.75% and 94% and GC content of 54.31% and 54.90%. In total, 23648400 anchor reads were mapped to the reference genome of Apis cerana. In AcCK and AcT, 805 and 702 circRNAs were identified; the lengths of these circRNAs were distributed among 201-1000 nt, and the most abundant circulization type was annotated exonic circRNA; however, the numbers of circRNAs distributed in various lengths and circulization types were different. There were 494 DEcircRNAs in AcCK vs AcT comparison group, including 257 up-regulated circRNAs and 237 down-regulated circRNAs, with novel_circ_000123 and novel_circ_000726 having the highest up-regulation and down-regulation levels. Source genes of the aforementioned DEcircRNAs were annotated to 11 biological process-associated terms, nine molecular function-associated terms, nine cellular component-associated terms, and 73 pathways. Further analysis demonstrated partial source genes were engaged in seven cellular immune pathways and three humoral immune pathways. [Conclusion] A. c. cerana 6-day-old larvae may response to A. apis stress by altering the numbers of circRNAs distributed in various lengths and circulization types, specifically expressing some circRNAs, and regulating the expression of partial circRNAs; several DEcircRNAs such as novel_circ_000027, novel_circ_000127 and novel_circ_000312 may play special roles in host A. apis-response via regulation of oxidative phosphorylation, cellular and humoral immune pathways. Our findings offered novel insights into further understanding molecular mechanisms underlying A. c. cerana larval response to A. apis and host-pathogen interaction.
Keywords: Apis cerana ceranalarvacircular RNAnon-coding RNAAscosphaera apisimmune response
蜜蜂白垩病是长期困扰养蜂生产的一大顽疾,由蜜蜂球囊菌(Ascosphaera apis,简称球囊菌)特异性侵染蜜蜂幼虫而引起[1]。球囊菌孢子随食物进入蜜蜂幼虫中肠,在4日龄到6日龄的幼虫期,中肠和后肠由一层隔膜阻断,至预蛹期隔膜消失,中肠和后肠连通,孢子随食物残渣进入后肠,孢子在O2的刺激下萌发,菌丝剧烈生长,先后刺穿围食膜和肠壁,继而从尾部穿出表皮,逐渐蔓延包裹整个虫尸,异宗菌丝接触后发生配合,从而产生大量子代孢子[2-3]。一般认为球囊菌仅能感染西方蜜蜂(Apis mellifera)幼虫,但近期的研究结果表明球囊菌对中华蜜蜂(Apis cerana cerana,简称中蜂)雄蜂幼虫和工蜂幼虫以及成年熊蜂同样具有侵染性[4-5]。白垩病常发生在春季和初夏,可导致幼虫大量死亡,从而造成成年蜜蜂数量和蜂群群势的急剧下降,以及造成其他病原微生物的入侵几率大增[6]。随着转地放蜂和蜂产品贸易全球化,白垩病已扩散至全球各主要养蜂国家[7]。该病现为我国蜂产品进出口检疫的对象之一[8]。目前对于白垩病尚缺乏有效的治疗药物,蜜蜂饲养管理过程中常采用改进遗传品系选育良种和科学的饲养管理方法等方法对该病进行防治[9],但成效并不显著。Qin等于2006年组装了球囊菌ARSEF 7405菌株的基因组序列,但当时并未同时公布基因功能注释信息[10],导致球囊菌的分子生物学和组学研究进展缓慢。2016年,Shang等利用二代测序技术重新对球囊菌ARSEF 7405菌株进行了基因组测序、组装、注释并公布了球囊菌的参考基因组[11],为球囊菌的分子生物学及组学研究奠定了重要基础。
环状RNA(circular RNA, circRNA)是一类非编码RNA(non-coding RNA, ncRNA),由来源基因的3′端和5′端共价闭合形成[12]。相比于线性RNA,circRNA更耐RNase R酶消化,优良的稳定性使其具备可作为内源生物标志物的条件[13]。circRNA可以通过与蛋白质、RNA聚合酶及基因启动子相互作用调节基因转录和可变剪切等生命活动[14-16]。较多的研究表明circRNA在人类肿瘤发生、生长、转移和抑制等方面发挥重要调控功能[17-19]。随着组学技术的飞速发展,越来越多的真核生物circRNA被鉴定出来[20-24]。但较之人类和少数动植物模式物种,昆虫的circRNA研究较为滞后,仅在果蝇(Drosophila)[25-28]、家蚕(Bombycis mori)[29-31]和蜜蜂[32-36]等极少数昆虫物种中见诸报道。Westholm等基于数据库中大量的果蝇总RNA数据筛选出2500个circRNA;通过分析circRNA在胚胎时期、幼虫期、蛹期和成虫期神经组织中的表达量,发现circRNA的表达水平随着果蝇的发育不断累积,在成虫期的神经组织中达到峰值,暗示着circRNA可作为果蝇衰老的潜在内源标记物[25]。Gan等利用RNA-seq技术对家蚕中部丝腺和后部丝腺进行测序,利用find_circ软件鉴定到了3916个circRNA,通过比较分析中部丝腺和后部丝腺筛选出880个上调circRNA和2532个下调circRNA,进一步分析发现中部丝腺circRNA和后部丝腺的circRNA可能在丝胶和蚕丝蛋白的合成中发挥不同的作用[29]。Chen等利用去除线性RNA的circRNA-seq技术对意大利蜜蜂(Apis mellifera ligustica,简称意蜂)处女王卵巢、产卵蜂王卵巢、产卵抑制和恢复的卵巢进行测序分析,共鉴定到12211个circRNA,进而构建了circRNA/lncRNA- miRNA-mRNA调控网络,发现ame_circ_0005197和ame_circ_0016640能够靶向调控与生物学调控和增殖相关的miRNA,从而影响蜂王产卵[32]。随后,Th?lken等利用去除线性RNA的circRNA-seq技术对西方蜜蜂哺育蜂和采集蜂的大脑进行测序和分析,共鉴定到3384个circRNA,分析结果显示许多外显子环化的circRNA与侧翼的内含子DNA甲基化相关;此外,作者还鉴定到1个调控大脑记忆的circRNA (circAmrsmep2)和1个调控劳动分工的circRNA (circAmrad)[33]。笔者前期在全基因组水平鉴定和分析了意蜂工蜂肠道的circRNA数量、种类、结构特征和表达谱[34];进而对中肠发育过程的circRNA差异表达谱和调控网络进行了分析和探讨[35]。近期,笔者首次对中蜂工蜂中肠的circRNA进行了全基因组鉴定、分析和验证,并系统解析了中肠发育过程的DEcircRNA表达谱、调控网络及潜在功能[36]。已有研究报道circRNA参与宿主和病原的相互作用[30-31, 37-38]。但对于circRNA在蜜蜂及其病原互作中的作用,相关研究仍然缺失。笔者团队前期围绕中蜂幼虫-球囊菌互作开展了一系列的组学和分子生物学研究,在mRNA组学层面解析了中蜂幼虫对球囊菌的胁迫应答[39],以及球囊菌在侵染中蜂幼虫过程中的转录组动态[40];在miRNA组学层面揭示了中蜂6日龄幼虫对球囊菌胁迫的miRNA应答[41],以及球囊菌在侵染中蜂6日龄幼虫过程中的miRNA差异表达谱及调控网络[42]。本研究利用去除线性RNA的circRNA-seq技术对正常和球囊菌胁迫的中蜂6日龄幼虫肠道进行测序,通过生物信息学方法对差异表达circRNA (differentially expressed circRNA, DEcircRNA)表达谱、调控网络及潜在功能进行分析和探讨,以期揭示DEcircRNA在宿主响应球囊菌胁迫中的作用,为阐明circRNA介导中蜂幼虫对球囊菌的胁迫应答机制提供必要基础,也为深入理解中蜂幼虫-球囊菌互作提供新的见解。
1 材料和方法 1.1 供试昆虫和真菌 中蜂幼虫取自福建农林大学动物科学学院(蜂学学院)教学蜂场。球囊菌菌株由福建农林大学动物科学学院(蜂学学院)蜜蜂保护实验室分离和保存[41-42]。
1.2 球囊菌培养、孢子纯化及中蜂幼虫的饲喂接种 按照笔者所在课题组已建立的实验方法[39],进行球囊菌的活化、培养以及孢子纯化,方法简述如下:(1)将实验室保存的球囊菌孢子接种至马铃薯葡萄糖琼脂(Potato Dextrose Agar, PDA)固体培养基进行活化培养;(2)置于33 ℃生化培养箱中培养10 d,培养基表面充满黑色孢子;(3)刮取黑色孢子至干净的EP管中,用研磨棒充分研磨,加入无菌水悬浮孢子;(4)吸取悬浮的纯化孢子至干净的EP管,梯度稀释后用血球计数板计数备用。
参照笔者课题组前期建立的方法[40-43],从外观健康、群势较强且无白垩病症状的中蜂蜂群提脾至实验室,置于35 ℃和70%相对湿度(RH)的恒温恒湿培养箱。取出一张脾,在35 ℃的室温条件下移取2日龄幼虫至48孔培养板(1只/孔)。中蜂幼虫的饲料配方参照王倩等的方法[44]并进行改良,将D-果糖和D-葡萄糖换为新鲜蜂蜜。配制含球囊菌孢子的饲料(1×107个孢子/mL)。处理组中,中蜂3日龄幼虫饲喂含球囊菌孢子的饲料50 μL,待饲料食尽,24 h后换成不含孢子的饲料,饲养至6日龄;对照组中蜂3日龄幼虫饲喂不含球囊菌孢子的饲料50 μL,饲养至6日龄;每24 h更换一次饲料。
1.3 测序样品的制备及深度测序 由于肠道是中蜂幼虫与球囊菌直接互作的主要场所,中蜂幼虫的幼虫期为1–6日龄,7–8日龄为预蛹期,球囊菌感染的中蜂幼虫在7–8日龄爆发白垩病,因此本研究选取中蜂6日龄幼虫肠道作为测序样品。分别剖取对照组中蜂6日龄幼虫肠道(AcCK)和处理组中蜂6日龄幼虫肠道(AcT),对照组集齐7只幼虫肠道置于1个RNA-Free EP管,处理组集齐7只幼虫肠道置于另1个RNA-Free EP管。经液氮速冻后立即转移到–80 ℃超低温冰箱保存备用。
利用Trizol法分别抽提AcCK和AcT肠道样品的总RNA,利用Ribo-Zerogold (Epicentre公司,美国)试剂盒去除核糖体RNA;参照Hu等[30-31]的方法,利用3 U/mg RNase R酶消除线性RNA;将富集的circRNA加入fragmentation buffer使其断成200 nt的短片段,作为模板利用六碱基随机引物合成cDNA第一链,并加入缓冲液、dNTPs、RNase H和DNA polymerase I合成cDNA第二链;经过QiaQuick PCR试剂盒纯化并加EB缓冲液洗脱经末端修复、加碱基A,加测序接头,再经琼脂糖凝胶电泳回收目的大小片段,并进行PCR扩增,最终获得cDNA文库;委托广州基迪奥生物技术有限公司用Illumina HiSeqTM 2500对上述cDNA文库进行双端测序。测序数据已上传NCBI Sequence Read Archive (SRA)数据库,SRA号为SRR9994498 (AcCK)和SRR9994497 (AcT)。
1.4 测序数据的质控及circRNA的预测与分析 将测序得到的原始读段(raw reads),过滤掉含接头(adapter)的读段、含N比例大于10%的读段和质量值Q≤10的碱基数占比50%以上的读段,得到的有效读段(clean reads)用于后续的数据分析。利用TopHat软件[45]将AcCK和AcT的有效读段比对到东方蜜蜂(Apis cerana)参考基因组(ACSNU-2.0, www.ncbi.nlm.nih.gov/genome/12051?genome_assembly_id=258367),将未比对上的有效读段两端截取20 nt,得到短序列读段(anchor reads),然后再次比对A. cerana参考基因组(ACSNU-2.0),最后把比对上的结果导入find_circ软件[20]预测circRNA (采用默认参数):(1)仅1个清晰断裂点(breakpoint)的circRNA;(2)每条读段的两条短序列读段(anchor reads)比对到参考基因组上的位置重叠部分不超过2 bp;(3)只允许2 nt错配;(4)唯一比对上的读段(unique reads)数大于2条;(5)每条读段的其中一个短序列读段比对到参考基因组上第一位的映射(mapping)结果要比第二位的映射结果的分值高35分以上;(6)支持该circRNA的唯一比对上的读段数要大于支持它的样品数;(7)该circRNA的唯一比对上的读段要大于总样品数的二分之一;(8) circRNA的长度小于100 kb。
参照Chen等的方法[36]对circRNA的长度和环化类型进行统计。环化类型包括:(1)已注释的外显子,即circRNA的断裂点位于已知基因的一个外显子的起始和另一个外显子的终止,其序列由断裂点之间的外显子的碱基构成;(2)单一外显子,即circRNA位于已知基因的某一个外显子内,其序列由断裂点之间的所有碱基构成;(3)基因反义链,即circRNA位于已知基因的反义链,其序列由反义链上断裂点之间的所有碱基构成;(4)内含子,即circRNA位于已知基因的某一个内含子内,其序列由断裂点之间的所有碱基构成;(5)外显子和内含子,即circRNA的断裂点之间有已知基因的一个或多个完整外显子但断裂点不位于已知外显子的起始或终止,其序列由上断裂点起始位点到第一个外显子起始位点、断裂点之间的外显子和最后一个外显子到断裂点终止位点的碱基构成;(6)基因间区,即circRNA位于已知基因之间,其序列由断裂点之间的所有碱基构成。
1.5 DEcircRNA的筛选及分析 采用RPM (mapped back-splicing junction reads per million mapped reads)法,计算circRNA的表达量。按照|log2(Fold change)|≥1,P≤0.05标准筛选DEcircRNA。利用Bowtie软件[46]将来源于内含子或外显子和内含子环化的circRNA两侧的短序列读段分别比对A. cerana参考基因组(ACSNU-2.0),两侧能够比对上同一基因则认为是该circRNA的来源基因,最后利用BLAST工具将各来源基因向Gene ontology (GO)数据库和Kyoto Encyclopedia of Genes and Genomes (KEGG)数据库进行映射,从而获得相应的功能和通路(pathway)注释信息,进一步统计注释上的功能条目(term)或通路,以及来源基因的数量。
1.6 DEcircRNA的RT-qPCR验证 为验证circRNA-seq数据的可靠性,随机选取3个DEcircRNA (novel_circ_000645、novel_circ_ 001097和novel_circ_000339)进行RT-qPCR。参照Guo等[23]的方法,根据所选circRNA的碱基序列,利用DNAMAN软件设计相应的特异性反向引物,扩增区域跨反向剪切位点,委托生工生物工程(上海)股份有限公司合成引物。选择actin基因(LOC108004261)作为内参。相关引物信息详见表 1。利用RNA抽提试剂盒(TaKaRa公司,中国)分别提取AcCK和AcT样品的总RNA,分为2份,其中1份总RNA用3 U/mg RNase R (吉赛生物,中国)进行消化,以去除线性RNA,37 ℃处理15 min,70 ℃处理10 min,后以随机引物进行反转录,得到cDNA作为circRNA的qPCR模板;另一份总RNA直接作为模板,以Oligo (dT)23作为引物进行反转录,得到对应的cDNA,作为actin的qPCR模板。反应体系(20 μL)包含:正、反向引物(10.0 μmol/L)各1 μL,cDNA模板DNA 1 μL,SYBR Green Dye 10 μL,DEPC水7 μL。反应在ABI QuanStudio 3荧光定量PCR仪(ThermoFisher公司,美国)上进行,程序如下:95 ℃ 1 min,95 ℃ 15 s,60 ℃ 30 s,45个循环。每个反应进行3次技术重复和生物学重复。采用2–△△Ct法[47]计算上述DEcircRNA的相对表达量。Graphpad Prism 7软件进行Student’s t test及相关绘图。
表 1. 本研究使用的引物 Table 1. Primers used in this study
| Primer name | Primer sequence (5′→3′) |
| Novel_circ_000645-F | GGCTTCTTGACTTTCAGCG |
| Novel_circ_000645-R | TTATTGTTCACCTGGACGC |
| Novel_circ_001097-F | GGTATGGTTGTCACATTTGC |
| Novel_circ_001097-R | GGAAGAATAGCATCAAGCG |
| Novel_circ_000339-F | GTCTCAAGCACTCCAGTTCTC |
| Novel_circ_000339-R | TTCCTTCTCCATCTGCGT |
| actin-F | GCAGTTAGGCACTTTCTCTGA |
| actin-R | CGTTTCCCACTTCTTCGTA |
表选项
2 结果和分析 2.1 circRNA-seq数据的质控及评估 AcCK与AcT样品测序分别得到76342570和68269362条原始读段,经过严格质控分别得到74524108和66974392条有效读段,GC含量分别为54.31%和54.90%,Q30分别达到92.75%和94.00%。上述结果说明本研究的circRNA-seq数据质量良好,有效读段可用于后续分析。从AcCK与AcT分别鉴定到91798082和85166448条短序列读段,比对上参考基因组的数量分别为14457097和9191303条。
2.2 AcCK和AcT中circRNA的结构特征及表达水平 本研究共鉴定到1101个novel circRNA。AcCK和AcT中分别鉴定到805和702个circRNA,二者共有circRNA的数量为406 (36.2%),特有circRNA的数量分别为399 (36.9%)和296 (26.9%) (图 1-A),它们的长度主要介于201–1000 nt (图 1-B)。AcCK和AcT中circRNA的环化类型最丰富的均为已注释外显子circRNA (520和460个),其次是单一外显子circRNA (114和95个)、外显子和内含子circRNA (81和85个)、反义链circRNA (45和32个)、内含子circRNA (25和17个)以及基因间区circRNA (20和13个) (图 1-C)。除了外显子和内含子circRNA,其余四种环化类型在AcCK中的数量皆多于AcT中的circRNA数量(图 1-C)。
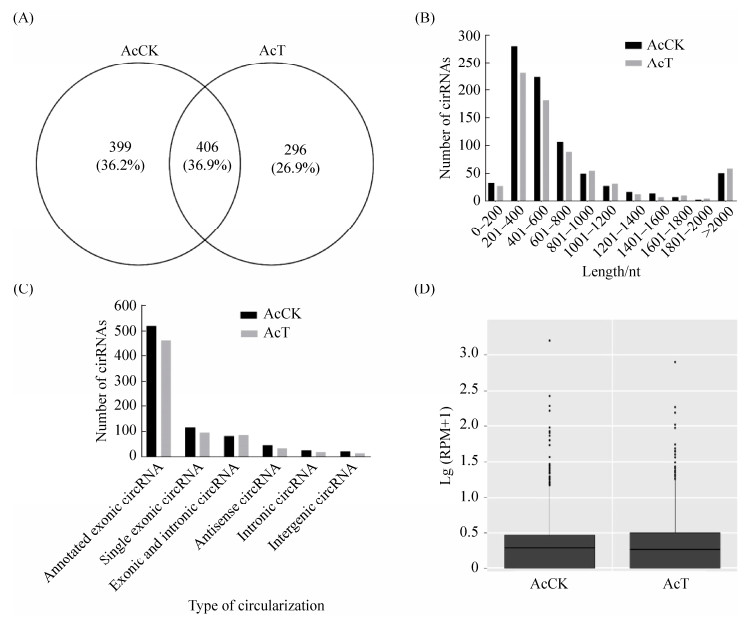 |
| 图 1 AcCK和AcT中circRNA的结构特征及总体表达量比较 Figure 1 Comparison of structural properties and total expression levels of circRNAs between AcCK group and AcT group. A: Venn analysis of circRNAs; B: length of circRNAs; C: circularization type of circRNAs; D: total expression level of circRNAs. |
| 图选项 |
此外,AcCK的circRNA整体表达水平略高于AcT的circRNA整体表达水平(图 1-D)。AcCK中表达量最高的circRNA是novel_circ_000210、novel_circ_000114和novel_circ_000805 (表 2);AcT中表达量最高的circRNA是novel_circ_000210、novel_circ_000805和novel_circ_000114 (表 3)。
表 2. AcCK中表达量最高的前10位circRNA Table 2. Top 10 circRNAs with the highest expression levels in AcCK group
| circRNA ID | RPM value | Length/nt | Type | Source gene ID |
| Novel_circ_000210 | 1574.036613 | 552 | Annotated exonic circRNA | 2288 |
| Novel_circ_000114 | 260.4948974 | 528 | Annotated exonic circRNA | 1413 |
| Novel_circ_000805 | 189.1112718 | 3776 | Exonic and intronic circRNA | 8864 |
| Novel_circ_000259 | 161.5815402 | 483 | Annotated exonic circRNA | 2706 |
| Novel_circ_000254 | 93.93310427 | 576 | Exonic and intronic circRNA | 2706 |
| Novel_circ_000727 | 83.14255621 | 394 | Annotated exonic circRNA | 7989 |
| Novel_circ_000260 | 78.85400506 | 633 | Annotated exonic circRNA | 2706 |
| Novel_circ_000457 | 71.93698707 | 1474 | Intronic circRNA | 4878 |
| Novel_circ_000508 | 62.25316189 | 559 | Single exonic circRNA | 5524 |
| Novel_circ_000555 | 61.69980045 | 640 | Annotated exonic circRNA | 5927 |
表选项
表 3. AcT中表达量最高的前10位circRNA Table 3. Top 10 circRNAs with the highest expression levels in AcT group
| circRNA ID | RPM value | Length/nt | Type | Source gene ID |
| Novel_circ_000210 | 792.9234843 | 552 | Annotated exonic circRNA | 2288 |
| Novel_circ_000805 | 181.9110957 | 3776 | Exonic and intron circRNA | 8864 |
| Novel_circ_000114 | 152.1003061 | 528 | Annotated exonic circRNA | 1413 |
| Novel_circ_000727 | 102.9233831 | 394 | Annotated exonic circRNA | 7989 |
| Novel_circ_000259 | 92.91392091 | 483 | Annotated exonic circRNA | 2706 |
| Novel_circ_000260 | 54.18165411 | 633 | Annotated exonic circRNA | 2706 |
| Novel_circ_000254 | 47.43614698 | 576 | Exonic and intron circRNA | 2706 |
| Novel_circ_000508 | 43.7369979 | 559 | Single exonic circRNA | 5524 |
| Novel_circ_000457 | 42.86660988 | 1474 | Intronic circRNA | 4878 |
| Novel_circ_000048 | 39.16746081 | 386 | Annotated exonic circRNA | 576 |
表选项
2.3 circRNA的差异表达谱分析 差异分析结果显示,AcCK vs AcT比较组中共有494个DEcircRNA,包含257个上调circRNA和237个下调circRNA,上调表达倍数介于2.04–7615.90,下调倍数介于0.000071–0.4。上调幅度最大的是novel_circ_000123 [log2(Fold change)=12.89, P=4.75E-13]、novel_circ_000585 [log2 (Fold change)=12.35, P=3.80E-09]和novel_circ_000751 [log2 (Fold change)=12.29, P=7.59E-09] (表 4);下调幅度最大的是novel_ circ_000726 [log2 (Fold change)= –13.78, P= 5.94E–18]、novel_circ_000119 [log2 (Fold change)= –13.04, P=1.68E–11]和novel_circ_000265 [log2 (Fold change)=–13.02, P=3.21E–11] (表 5)。
表 4. AcCK vs AcT比较组中前10位上调circRNA Table 4. Top 10 up-regulated circRNAs in AcCK vs AcT comparison group
| circRNA ID | RPM in AcCK | RPM in AcT | Log2 (Fold change) | P value |
| Novel_circ_000123 | 0.001 | 7.615895157 | 12.89 | 4.75E–13 |
| Novel_circ_000585 | 0.001 | 5.222328107 | 12.35 | 3.80E–09 |
| Novel_circ_000751 | 0.001 | 5.004731103 | 12.29 | 7.59E–09 |
| Novel_circ_000936 | 0.001 | 4.351940089 | 12.09 | 1.21E–07 |
| Novel_circ_000206 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
| Novel_circ_000425 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
| Novel_circ_000574 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
| Novel_circ_000629 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
| Novel_circ_000688 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
| Novel_circ_001062 | 0.001 | 3.916746081 | 11.94 | 4.82E–07 |
表选项
表 5. AcCK vs AcT比较组中前10位下调circRNA Table 5. Top 10 down-regulated circRNAs in AcCK vs AcT comparison group
| circRNA ID | RPM in AcCK | RPM in AcT | Log2 (Fold change) | P value |
| Novel_circ_000726 | 14.1107167 | 0.001 | –13.78 | 5.94E–18 |
| Novel_circ_000119 | 8.438761945 | 0.001 | –13.04 | 1.68E–11 |
| Novel_circ_000265 | 8.300421585 | 0.001 | –13.02 | 3.21E–11 |
| Novel_circ_000208 | 5.948635469 | 0.001 | –12.54 | 1.34E–07 |
| Novel_circ_000399 | 5.53361439 | 0.001 | –12.43 | 4.71E–07 |
| Novel_circ_000207 | 5.39527403 | 0.001 | –12.40 | 4.71E–07 |
| Novel_circ_000852 | 5.39527403 | 0.001 | –12.40 | 4.71E–07 |
| Novel_circ_001065 | 5.118593311 | 0.001 | –12.32 | 2.20E–07 |
| Novel_circ_000875 | 4.703572232 | 0.001 | –12.20 | 8.16E–07 |
| Novel_circ_000788 | 4.565231872 | 0.001 | –12.16 | 1.57E–06 |
表选项
2.4 DEcircRNA来源基因的功能及通路注释 GO数据库注释结果显示,DEcircRNA的来源基因可注释到29条功能条目,涉及细胞进程(94个)、单组织进程(78个)、代谢进程(78个)、生物学调控(62个)和定位(30个)等11条生物学进程相关条目(图 2);结合(120个)、催化活性(83个)、转运活性(20个)、核酸结合转录因子活性(13个)和分子转导活性(12个)等9条分子功能相关条目(图 2);细胞膜组件(64个)、细胞膜(64个)、细胞(49个)、细胞组件(48个)和细胞器(43个)等9条细胞组分相关条目(图 2)。
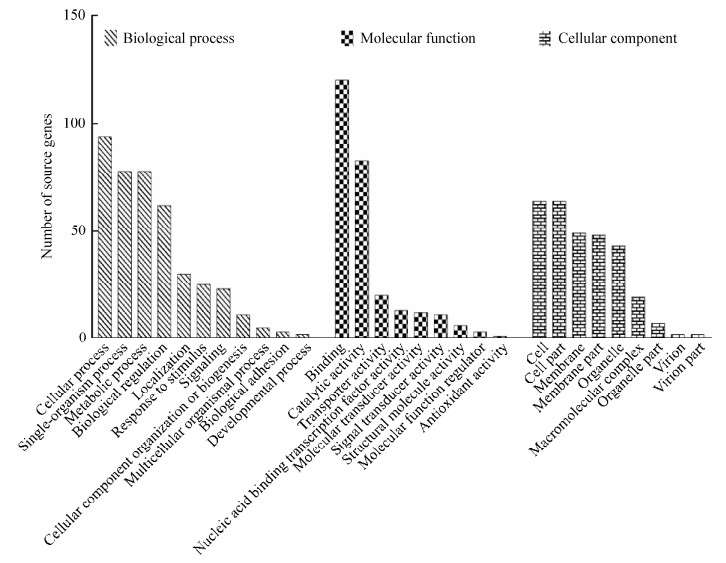 |
| 图 2 DEcircRNA来源基因的GO数据库注释 Figure 2 GO dataset annotation of source genes of DEcircRNAs. |
| 图选项 |
KEGG代谢通路注释结果显示,DEcircRNA来源基因可注释到73条通路,基因数最多的前5条分别是内质网蛋白加工(8个)、剪切体(8个)、Hippo信号通路(7个)、核糖体(7个)和内吞作用(6个) (图 3)。此外,novel_circ_000357、novel_circ_000764、novel_circ_000045、novel_circ_ 000954的来源基因可注释到氧化磷酸化通路(图 3)。
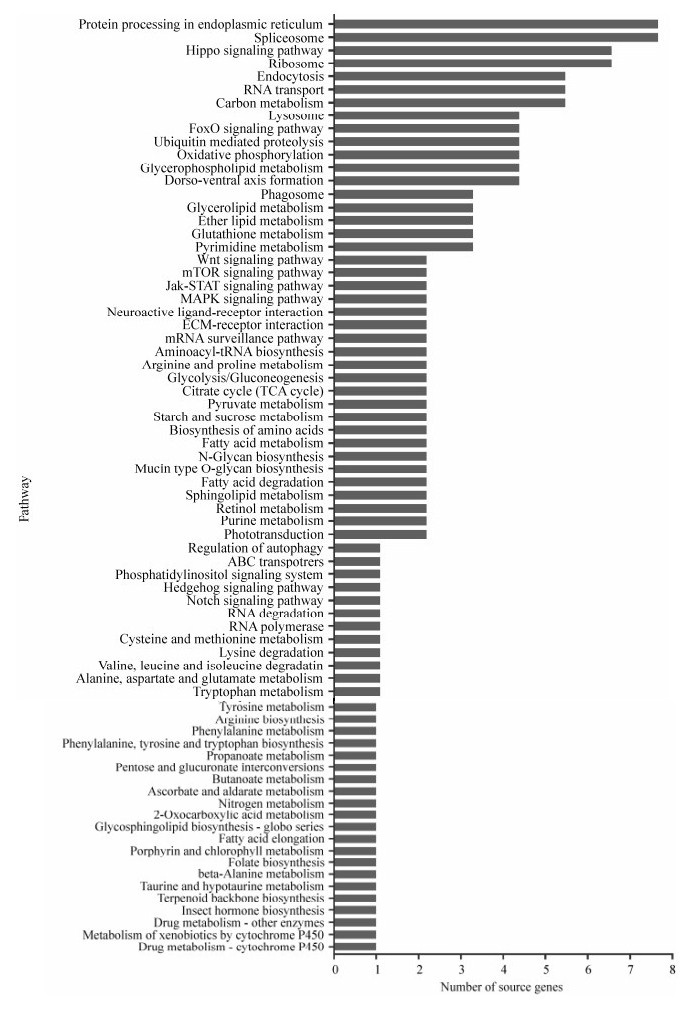 |
| 图 3 DEcircRNA来源基因的KEGG数据库注释 Figure 3 KEGG dataset annotation of source genes of DEcircRNAs. |
| 图选项 |
进一步对涉及免疫应答的通路进行分析,8、4、5、3、1、1、1个DEcircRNA的5、4、4、3、1、1、1个来源基因可分别注释到内吞作用、泛素介导的蛋白水解、溶酶体、吞噬体、细胞色素P450对外源物质代谢、昆虫激素生物合成和自噬调控等细胞免疫相关通路(表 6);5、3、2个DEcircRNA的4、2、2个来源基因可分别注释到FoxO信号通路、MAPK信号通路和Jak-STAT信号通路等体液免疫相关通路(表 7)。
表 6. 涉及细胞免疫的来源基因及相应DEcircRNA Table 6. Source genes and corresponding DEcircRNAs involved in cellular immune pathways
| Pathway | Ko number | Num of source genes | Num of DEcircRNAs | DEcircRNA ID |
| Endocytosis | ko04144 | 5 | 8 | Novel_circ_000027 Novel_circ_000127 Novel_circ_000312 Novel_circ_000313 Novel_circ_000403 Novel_circ_000405 Novel_circ_000408 Novel_circ_000749 |
| Ubiquitin mediated proteolysis | ko04120 | 4 | 4 | Novel_circ_000073 Novel_circ_000495 Novel_circ_000550 Novel_circ_000949 |
| Lysosome | ko04142 | 4 | 5 | Novel_circ_000069 Novel_circ_000357 Novel_circ_000735 Novel_circ_000736 Novel_circ_000764 |
| Phagosome | ko04145 | 3 | 3 | Novel_circ_000357 Novel_circ_000764 Novel_circ_000427 |
| Metabolism of xenobiotics by cytochrome P450 | ko00980 | 1 | 1 | Novel_circ_000087 |
| Insect hormone biosynthesis | ko00981 | 1 | 1 | Novel_circ_000015 |
| Regulation of autophagy | ko04140 | 1 | 1 | Novel_circ_000146 |
表选项
表 7. 涉及体液免疫通路的来源基因及相应DEcircRNA Table 7. Source genes and corresponding DEcircRNAs involved in humoral immune pathways
| Pathway | Ko number | Num of source genes | Num of DEcircRNAs | DEcircRNA ID |
| FoxO signaling pathway | ko04068 | 4 | 5 | Novel_circ_000312 Novel_circ_000313 Novel_circ_000304 Novel_circ_000323 Novel_circ_000606 |
| MAPK signaling pathway | ko04013 | 2 | 3 | Novel_circ_000312 Novel_circ_000313 Novel_circ_000777 |
| Jak-STAT signaling pathway | ko04630 | 2 | 2 | Novel_circ_000550 Novel_circ_000323 |
表选项
2.5 DEcircRNA的RT-qPCR验证 RT-qPCR结果显示3个DEcircRNA的表达趋势与测序结果一致(图 4),证实了测序数据和circRNA差异表达的可靠性。
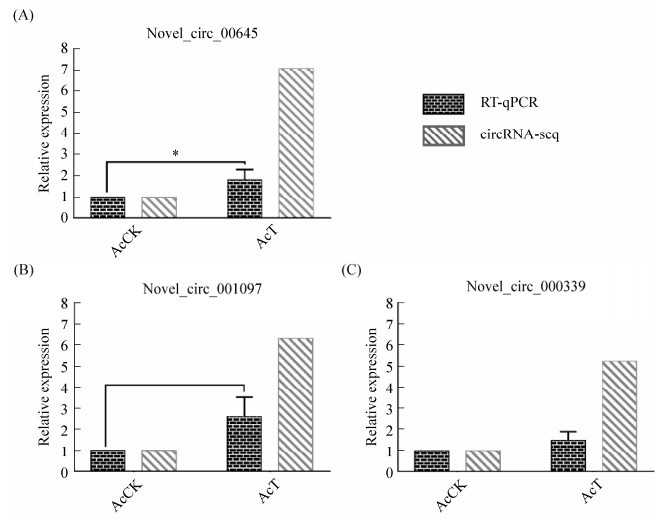 |
| 图 4 DEcircRNA的RT-qPCR验证 Figure 4 RT-qPCR validation of DEcircRNA. A–C: RT-qPCR validation of novel_circ_000645, novel_circ_ 001097 and novel_circ_000339. In RT-qPCR group, *: P≤0.05. |
| 图选项 |
3 讨论 CircRNA已成为近年来国内外的研究热点。但相关研究多集中在人类等少数哺乳动物[20-21],昆虫的circRNA研究起步较晚且进度缓慢。目前,全球关于蜜蜂circRNA的研究报道仅5例[32-36],涉及卵巢产卵[32]、大脑功能[33]及肠道发育[34-36]。CircRNA已被研究证实可参与介导动物对病原微生物的胁迫响应[30-31, 37-38]。Hu等利用RNA-seq技术对正常家蚕中肠和家蚕核质多角体病毒(Bombycis mori cytoplasmic polyhedrosis virus, BmCPV)感染的家蚕中肠进行测序,通过差异分析筛选出400个DEcircRNA,进而通过来源基因和竞争性内源RNA(competing endogenous RNA, ceRNA)调控网络等分析揭示了DEcircRNA在家蚕中肠应答BmCPV感染中的潜在作用[30]。然而,迄今有关circRNA介导蜜蜂及其病原互作的研究未见报道。
以RNA-seq为代表的高通量测序技术为circRNA的大规模挖掘提供了有力工具[16]。目前,circRNA的主流建库方法有两种,一种是先利用RNase R消化样品总RNA中的线性RNA,从而富集circRNA,再利用二代测序技术进行深度测序[30-31];另一种是先去除rRNA,再构建链特异性cDNA文库,进而利用RNA-seq技术进行深度测序[34, 36];前者能够检测到低表达丰度的circRNA,因而鉴定到的circRNA数量更多且准确性更高[48],但因为不包含mRNA和lncRNA信息,故无法通过共表达关系构建不同RNA之间的调控网络[30-31];第二种建库方法能够一次性获得测序样品的mRNA、lncRNA和circRNA信息,并同时计算三者的表达量以及构建三者之间的共表达关系[34, 36]。为最大限度鉴定circRNA,本研究利用去除线性RNA的circRNA-seq技术对正常及球囊菌侵染的中蜂6日龄幼虫肠道进行测序,基于AcCK和AcT高质量测序数据分别预测出805和702个circRNA,二者共有的circRNA为406个,特异性表达的circRNA分别有399和296个。本研究鉴定出的1101个高可信度circRNA可为东方蜜蜂的circRNA信息提供重要补充,也为其他蜂种的circRNA研究提供有益的参考信息。
AcCK和AcT中circRNA的长度均主要分布在201–1000 nt,但分布在不同长度范围的circRNA数量存在差异;AcCK和AcT中,最丰富的环化类型均为已注释的外显子circRNA (64.60%,65.53%),其次均为单一外显子circRNA、外显子和内含子circRNA、反义链circRNA、内含子circRNA以及基因间区circRNA,但每一种环化类型的circRNA数量同样存在差异;此外,AcCK中circRNA整体表达水平略高于处理组的circRNA整体表达水平。上述结果表明,中蜂6日龄幼虫响应球囊菌胁迫的过程中可能通过调整circRNA的结构对病原产生应答;不同于mRNA[43],circRNA的应答水平较低。前期研究发现,中蜂工蜂中肠circRNA的长度主要分布在201–800 nt且最丰富的环化类型为已注释的外显子circRNA (47.5%)[36],意蜂工蜂中肠circRNA的长度主要分布在15–1000 nt且最丰富的环化类型也为已注释的外显子circRNA (48.56%)[34]。果蝇circRNA的主要环化类型为已注释的外显子circRNA[25],而家蚕circRNA的主要环化类型为外显子和内含子的circRNA[29]。上述结果表明,同一蜂种的不同虫态以及不同蜂种的circRNA结构特征相似,但不同昆虫的circRNA的结构特征存在差异。
本研究中,在AcCK vs AcT比较组中鉴定出257个上调circRNA和237个下调circRNA,上调表达倍数介于2.04–7615.90,下调倍数介于0.000071–0.4,说明球囊菌胁迫导致中蜂6日龄幼虫肠道的circRNA发生差异表达,这些DEcircRNA参与宿主的胁迫应答。CircRNA可以通过与RNA聚合酶II、U1小核核糖核蛋白及基因启动子相互作用促进来源基因的转录[14-15]。本研究中,对于上述DEcircRNA,分别有94、83、78、25、2个来源基因注释到细胞进程、催化活性、代谢进程、应激反应和发育进程;此外,部分来源基因可注释到丙酮酸代谢(2个)和三羧酸循环(2个)等8条碳水化合物代谢通路;甘油磷脂代谢(4个)和醚脂类代谢(3个)等6条脂质代谢通路;酪氨酸代谢(1个)和色氨酸代谢(1个)等10条氨基酸代谢通路;嘧啶代谢(3个)和嘌呤代谢(2个)等2条核酸代谢通路;内吞作用(5个)和泛素介导的蛋白水解(4个)等10条免疫相关通路。上述结果表明,中蜂6日龄幼虫肠道响应球囊菌胁迫的过程中,DEcircRNA通过调控来源基因的表达调节宿主的新陈代谢和免疫防御,从而介导宿主对球囊菌的胁迫应答。
昆虫的先天性免疫包括细胞免疫和体液免疫,通过内吞作用、包囊作用、吞噬作用、黑化作用、酶解作用以及抗菌肽的合成与分泌来抵御病原微生物的侵染[9]。内吞作用和吞噬作用是蜜蜂的两种主要细胞免疫途径[9]。溶酶体具有抗真菌和抗病毒活性,可溶解内吞作用和吞噬作用运送来的致病蛋白[49-50]。本研究中,分别有5、4和4个来源基因注释到内吞作用、吞噬作用和溶酶体,表明相应的DEcircRNA (novel_circ_000027、novel_circ_000069、novel_circ_000357等)可能通过调控上述3种细胞免疫通路降解球囊菌侵染过程产生的毒力因子并限制病原增殖。泛素蛋白酶体降解系统被用于清理受损或不需要的细胞,在细胞周期、分裂、分化、发育和免疫等细胞进程扮演关键角色[51]。自噬在调节细胞生长、发育、凋亡和防御病原等方面中起重要作用[52]。本研究发现,分别有4和1个来源基因注释到泛素介导蛋白水解和自噬调控,暗示相应的DEcircRNA (novel_circ_000073、novel_circ_000495、novel_ circ_000550、novel_circ_000949、novel_circ_000146)可能调控泛素介导的蛋白水解和自噬通路清理被球囊菌侵染而受损的细胞,从而减轻病原侵染对宿主造成的伤害。昆虫激素是免疫系统的重要调节因子,其中蜕皮激素和保幼激素等可以促进昆虫的免疫反应[53-54]。昆虫细胞色素P450可通过催化内源物质(蜕皮激素和保幼激素等)的生物合成与降解参与昆虫的免疫反应[55]。本研究中,分别有1个来源基因注释到昆虫激素的生物合成和药物代谢-细胞色素P450;进一步分析发现有1个下调circRNA (novel_circ_000015, log2 (Fold change)=–10.43, P= 0.01564141)的来源基因可注释到昆虫激素的生物合成通路的类保幼激素环氧化物酶基因(juvenile hormone epoxide hydrolase 1-like),另有1个上调circRNA (novel_circ_000087, log2 (Fold change)=11.22, P=0.000245094)的来源基因可注释到外源物质代谢-细胞色素P450通路的类UDP-葡糖苷转移酶基因(UDP- glucuronosyltransferase 1-3-like)。上述结果表明novel_circ_000015和novel_circ_000087可能通过调节类保幼激素环氧化物酶基因和类UDP-葡糖苷转移酶基因的表达水平影响昆虫激素和细胞色素P450通路,从而参与宿主对球囊菌的免疫应答。FoxO信号通路中,FoxO蛋白可以通过促进一系列靶基因上调加快细胞凋亡[56]。MAPK信号通路能够通过产生免疫效应因子刺激宿主淋巴细胞活性,对外界入侵的病原微生物进行免疫防御[57]。Jak-STAT信号通路在果蝇中首次被发现,后被证实能诱导抗菌肽的表达[58]。本研究中,分别有5、3和2个来源基因注释到FoxO信号通路、MAPK信号通路和Jak-STAT信号通路;进一步分析发现分别有5个上调circRNA {包括novel_circ_000312 [log2 (Fold change)=3.11, P=0.000980413]、novel_circ_000304 [log2(Fold change)=11.47, P=6.14E-05]、novel_circ_000323[log2(Fold change) =1.98, P=0.007406097]、novel_circ_000606 [log2 (Fold change)=11.22, P=0.000245094]和novel_circ_000777 [log2 (Fold change)=2.24, P=8.88E–05]}和2个下调CircRNA {novel_circ_ 000313 [log2(Fold change)=–10.09, P=0.03127344]和novel_circ_000550 [log2(Fold change)=–10.81, P=0.021515144]}参与CircRNA涉及对FoxO信号通路、MAPK信号通路和Jak-STAT信号通路相关来源基因的调控,表明上述7个上调circRNA可能通过促进体液免疫通路相关来源基因的转录对球囊菌胁迫进行体液免疫应答;球囊菌通过与宿主的相互作用下调宿主novel_circ_000313和novel_circ_000550的表达水平,从而一定程度抑制宿主的体液免疫,体现了二者互作的复杂性。
氧化磷酸化是昆虫合成ATP的主要方式[59]。前期研究中,笔者团队对中蜂幼虫和意蜂幼虫进行了比较转录组学分析,并探讨了两种蜜蜂幼虫的球囊菌抗性差异产生的原因,发现受到强烈正向选择的中蜂单拷贝基因仅富集在氧化磷酸化,推测中蜂可能通过提高能量利用限制球囊菌增殖[60]。本研究发现有4个来源基因注释到氧化磷酸化通路,1个上调circRNA (novel_circ_000357, log2(Fold change)=10.77, P=0.001957522)调控的来源基因为氧化磷酸化通路相关的ATP合成酶相关基因(vacuolar H+ATP synthase 16 kDa proteolipid subunit),说明中蜂幼虫响应球囊菌胁迫的过程中可通过调节novel_circ_000357的表达量对宿主细胞的氧化磷酸化通路相关来源基因的转录水平进行调控,进而通过增加ATP合成提高能量利用水平,从而限制球囊菌从宿主获得能量。笔者团队前期研究还发现中蜂6日龄幼虫肠道响应球囊菌胁迫的过程中,差异表达miRNA (differentially expressed miRNA, DEmiRNA)可靶向调控氧化磷酸化通路中相关的38个靶mRNA[41]。上述结果表明中蜂幼虫同时利用DEcircRNA和DEmiRNA调控氧化磷酸化通路,通过调节能量利用水平影响球囊菌增殖。长链非编码RNA (long non-coding RNA, lncRNA)是另一种广泛存在且十分重要的ncRNA,已被证实能够介导真核生物对病原微生物的免疫应答[61]。中蜂幼虫对球囊菌的免疫应答必然伴随着非常复杂的分子事件,背后的分子机制需要从多种角度全方位阐释。笔者目前正在探究中蜂6日龄幼虫肠道响应球囊菌胁迫的lncRNA差异表达谱、调控网络及潜在作用,结合前期在miRNA方面的研究结果以及本研究中circRNA方面的研究结果,可以更全面和深入地解析中蜂幼虫对球囊菌的胁迫应答机制。
本研究通过对正常及球囊菌胁迫的中蜂6日龄幼虫肠道circRNA数量、长度和类型等结构特征进行比较分析,以及对宿主响应胁迫的DEcircRNA表达谱及来源基因进行全面解析,揭示了中蜂响应球囊菌胁迫的过程中可能调节不同长度、类型的circRNA数量对病原产生应答,DEcircRNA可能通过调节来源基因的转录影响宿主的内吞作用、吞噬体、溶酶体、泛素介导的蛋白水解、自噬、昆虫激素的生物合成、外源物质代谢-细胞色素P450等细胞免疫通路,FoxO信号通路、MAPK信号通路和Jak-STAT信号通路等体液免疫通路,以及氧化磷酸化通路,从而介导宿主对球囊菌的胁迫应答。涉及调控上述通路的部分DEcircRNA如novel_circ_000027和novel_ circ_000127等在中蜂幼虫的胁迫应答中扮演关键角色,可作为下一步功能研究的候选靶标。
References
| [1] | Liang Q, Chen DF, Wang JD. Effects of temperature, relative humidity and pH on germination of chalkbrood fungus, Ascosphaera apis spore. Chinese Journal of Applied Ecology, 2000, 11(6): 869-872. (in Chinese) 梁勤, 陈大福, 王建鼎. 温度、相对湿度和pH对蜜蜂球囊菌孢子萌发的影响. 应用生态学报, 2000, 11(6): 869-872. |
| [2] | Heath LAF, Gaze BM. Carbon dioxide activation of spores of the chalkbrood fungus Ascosphaera apis. Journal of Apicultural Research, 1987, 26(4): 243-246. DOI:10.1080/00218839.1987.11100768 |
| [3] | Evison SEF. Chalkbrood:epidemiological perspectives from the host-parasite relationship. Current Opinion in Insect Science, 2015, 10: 65-70. DOI:10.1016/j.cois.2015.04.015 |
| [4] | Maxfield-Taylor SA, Mujic AB, Rao S. First detection of the larval chalkbrood disease pathogen Ascosphaera apis (Ascomycota:Eurotiomycetes:Ascosphaerales) in adult bumble bees. PLoS One, 2015, 10(4): e0124868. DOI:10.1371/journal.pone.0124868 |
| [5] | Chen DF, Guo R, Xiong CL, Zheng YZ, Hou CS, Fu ZM. Morphological and molecular identification of chalkbrood disease pathogen Ascosphaera apis in Apis cerana cerana. Journal of Apicultural Research, 2018, 57(4): 516-521. DOI:10.1080/00218839.2018.1475943 |
| [6] | Zhao HX, Liang Q, Luo YX, Li JH, Zhang XF, Zeng XN. Chalkbrood disease in honey bees. Journal of Environmental Entomology, 2014, 36(2): 233-239. (in Chinese) 赵红霞, 梁勤, 罗岳雄, 李江红, 张学锋, 曾鑫年. 蜜蜂白垩病的研究进展. 环境昆虫学报, 2014, 36(2): 233-239. |
| [7] | Hitchcock JD, Christensen M. Occurrence of chalk brood (Ascosphaera apis) in honey bees in the united states. Mycologia, 1972, 64(5): 1193-1198. DOI:10.1080/00275514.1972.12019370 |
| [8] | Aizen MA, Garibaldi LA, Cunningham SA, Klein AM. How much does agriculture depend on pollinators? Lessons from long-term trends in crop production. Annals of Botany, 2009, 103(9): 1579-1588. DOI:10.1093/aob/mcp076 |
| [9] | Aronstein KA, Murray KD. Chalkbrood disease in honey bees. Journal of Invertebrate Pathology, 2010, 103(Suppl 1): S20-S29. |
| [10] | Qin X, Evans JD, Aronstein KA, Murray KD, Weinstock GM. Genome sequences of the honey bee pathogens Paenibacillus larvae and Ascosphaera apis. Insect Molecular Biology, 2006, 15(5): 715-718. DOI:10.1111/j.1365-2583.2006.00694.x |
| [11] | Shang YF, Xiao GH, Zheng P, Cen K, Zhan S, Wang CS. Divergent and convergent evolution of fungal pathogenicity. Genome Biology and Evolution, 2016, 8(5): 1374-1387. DOI:10.1093/gbe/evw082 |
| [12] | Meng SJ, Zhou HC, Feng ZY, Xu ZH, Tang Y, Li PY, Wu MH. CircRNA:functions and properties of a novel potential biomarker for cancer. Molecular Cancer, 2017, 16(1): 94-94. |
| [13] | Perkel JM. Assume nothing:the tale of circular RNA. BioTechniques, 2013, 55(2): 55-57. |
| [14] | Zhang Y, Zhang XO, Chen T, Xiang JF, Yin QF, Xing YH, Zhu SS, Yang L, Chen LL. Circular intronic long noncoding RNAs. Molecular Cell, 2013, 51(6): 792-806. DOI:10.1016/j.molcel.2013.08.017 |
| [15] | Li ZY, Huang C, Bao C, Chen L, Lin M, Wang XL, Zhong GL, Yu B, Hu WC, Dai LM, Zhu PF, Chang ZX, Wu QF, Zhao Y, Jia Y, Xu P, Liu HJ, Shan G. Exon-intron circular RNAs regulate transcription in the nucleus. Nature Structural & Molecular Biology, 2015, 22(3): 256-264. |
| [16] | Kristensen LS, Andersen MS, Stagsted LVW, Ebbesen KK, Hansen TB, Kjems J. The biogenesis, biology and characterization of circular RNAs. Nature reviews Genetics, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7 |
| [17] | Zhang PF, Wei CY, Huang XY, Peng R, Yang X, Lu JC, Zhang C, Gao C, Cai JB, Gao PT, Gao DM, Shi GM, Ke AW, Fan J. Circular RNA circTRIM33-12 acts as the sponge of MicroRNA-191 to suppress hepatocellular carcinoma progression. Molecular Cancer, 2019, 18(1): 105. DOI:10.1186/s12943-019-1031-1 |
| [18] | Liu L, Liu FB, Huang M, Xie K, Xie QS, Liu CH, Shen MJ, Huang Q. Circular RNA ciRS-7 promotes the proliferation and metastasis of pancreatic cancer by regulating miR-7-mediated EGFR/STAT3 signaling pathway. Hepatobiliary & Pancreatic Diseases International, 2019, 18(6): 580-586. |
| [19] | Cheng ZA, Yu CT, Cui SH, Wang H, Jin HJ, Wang C, Li BT, Qin ML, Yang C, He J, Zuo QZ, Wang SY, Liu J, Ye WD, Lv YY, Zhao FY, Yao M, Jiang LY, Qin WX. circTP63 functions as a ceRNA to promote lung squamous cell carcinoma progression by upregulating FOXM1. Nature Communications, 2019, 10: 3200. DOI:10.1038/s41467-019-11162-4 |
| [20] | Memczak S, Jens M, Elefsinioti A, Torti F, Krueger J, Rybak A, Maier L, Mackowiak SD, Gregersen LH, Munschauer M, Loewer A, Ziebold U, Landthaler M, Kocks C, le Noble F, Rajewsky N. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [21] | Zhang CL, Wu H, Wang YH, Zhu SQ, Liu JQ, Fang XT, Chen H. Circular RNA of cattle casein genes are highly expressed in bovine mammary gland. Journal of Dairy Science, 2016, 99(6): 4750-4760. DOI:10.3168/jds.2015-10381 |
| [22] | He LB, Zhang AD, Xiong L, Li YM, Huang R, Liao LJ, Zhu ZY, Wang YP. Deep circular RNA sequencing provides insights into the mechanism underlying grass carp reovirus infection. International Journal of Molecular Sciences, 2017, 18(9): 1977. DOI:10.3390/ijms18091977 |
| [23] | Guo R, Chen DF, Chen HZ, Fu ZM, Xiong CL, Hou CS, Zheng YZ, Guo YL, Wang HP, Du Y, Diao QY. Systematic investigation of circular RNAs in Ascosphaera apis, a fungal pathogen of honeybee larvae. Gene, 2018, 678: 17-22. DOI:10.1016/j.gene.2018.07.076 |
| [24] | Guo R, Chen DF, Chen HZ, Xiong CL, Zheng YZ, Hou CS, Du Y, Geng SH, Wang HP, Zhou DD, Guo YL. Genome-wide identification of circular RNAs in fungal parasite Nosema ceranae. Current Microbiology, 2018, 75(12): 1655-1660. DOI:10.1007/s00284-018-1576-z |
| [25] | Westholm JO, Miura P, Olson S, Shenker S, Joseph B, Sanfilippo P, Celniker SE, Graveley BR, Lai EC. Genome-wide analysis of Drosophila circular RNAs reveals their structural and sequence properties and age-dependent neural accumulation. Cell Reports, 2014, 9(5): 1966-1980. DOI:10.1016/j.celrep.2014.10.062 |
| [26] | Ashwal-Fluss R, Meyer M, Pamudurti NR, Ivanov A, Bartok O, Hanan M, Evantal N, Memczak S, Rajewsky N, Kadener S. circRNA biogenesis competes with pre-mRNA splicing. Molecular Cell, 2014, 56(1): 55-66. DOI:10.1016/j.molcel.2014.08.019 |
| [27] | Kramer MC, Liang DM, Tatomer DC, Gold B, March ZM, Cherry S, Wilusz JE. Combinatorial control of Drosophila circular RNA expression by intronic repeats, hnRNPs, and SR proteins. Genes & Development, 2015, 29(20): 2168-2182. |
| [28] | Pamudurti NR, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perez-Hernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N, Kadener S. Translation of circRNAs. Molecular Cell, 2017, 66(1): 9-21.e7. |
| [29] | Gan HY, Feng TS, Wu YQ, Liu C, Xia QY, Cheng TC. Identification of circular RNA in the Bombyx mori silk gland. Insect Biochemistry and Molecular Biology, 2017, 89: 97-106. DOI:10.1016/j.ibmb.2017.09.003 |
| [30] | Hu XL, Zhu M, Zhang X, Liu B, Liang Z, Huang LX, Xu J, Yu L, Li K, Zar MS, Xue RY, Cao GL, Gong CL. Identification and characterization of circular RNAs in the silkworm midgut following Bombyx mori cytoplasmic polyhedrosis virus infection. RNA Biology, 2018, 15(2): 292-301. DOI:10.1080/15476286.2017.1411461 |
| [31] | Hu XL, Zhu M, Liu B, Liang Z, Huang LX, Xu J, Yu L, Li K, Jiang MS, Xue RY, Cao GL, Gong CL. Circular RNA alterations in the Bombyx mori midgut following B. mori nucleopolyhedrovirus infection. Molecular Immunology, 2018, 101: 461-470. DOI:10.1016/j.molimm.2018.08.008 |
| [32] | Chen X, Shi W, Chen C. Differential circular RNAs expression in ovary during oviposition in honey bees. Genomics, 2019, 111(4): 598-606. DOI:10.1016/j.ygeno.2018.03.015 |
| [33] | Th?lken C, Thamm M, Erbacher C, Lechner M. Sequence and structural properties of circular RNAs in the brain of nurse and forager honeybees (Apis mellifera). BMC Genomics, 2019, 20: 88. DOI:10.1186/s12864-018-5402-6 |
| [34] | Xiong CL, Chen HZ, Chen DF, Zheng YZ, Fu ZM, Xu GJ, Du Y, Wang HP, Geng SH, Zhou DD, Liu SY, Guo R. Analysis of circular RNAs and their regulatory networks in the midgut of Apis mellifera ligustica workers. Acta Entomologica Sinica, 2018, 61(12): 1363-1375. (in Chinese) 熊翠玲, 陈华枝, 陈大福, 郑燕珍, 付中民, 徐国钧, 杜宇, 王海朋, 耿四海, 周丁丁, 刘思亚, 郭睿. 意大利蜜蜂工蜂中肠的环状RNA及其调控网络分析. 昆虫学报, 2018, 61(12): 1363-1375. |
| [35] | Guo R, Chen HZ, Xiong CL, Zheng YZ, Fu ZM, Xu GJ, Du Y, Wang HP, Geng SH, Zhou DD, Liu SY, Chen DF. Analysis of differentially expressed circular RNAs and their regulation networks during the developmental process of Apis mellifera ligustica worker's midgut. Scientia Agricultura Sinica, 2018, 51(23): 4575-4590. (in Chinese) 郭睿, 陈华枝, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 杜宇, 王海朋, 耿四海, 周丁丁, 刘思亚, 陈大福. 意大利蜜蜂工蜂中肠发育过程中的差异表达环状RNA及其调控网络分析. 中国农业科学, 2018, 51(21): 4575-4590. |
| [36] | Chen DF, Chen HZ, Du Y, Zhu ZW, Wang J, Geng SH, Xiong CL, Zheng YZ, Hou CS, Diao QY, Guo R. Systematic identification of circular RNAs and corresponding regulatory networks unveil their potential roles in the midguts of eastern honeybee workers. Applied Microbiology and Biotechnology, 2020, 104(1): 257-276. |
| [37] | Shi J, Hu N, Mo L, Zeng Z, Sun J, Hu Y. Deep RNA sequencing reveals a repertoire of human fibroblast circular RNAs associated with cellular responses to herpes simplex virus 1 infection. Cellular Physiology and Biochemistry, 2018, 47(5): 2031-2045. DOI:10.1159/000491471 |
| [38] | Zhang Y, Wang LD, Qiu LL, Pan R, Bai H, Jiang Y, Wang ZX, Bi YL, Chen GH, Chang GB. Expression patterns of novel circular RNAs in chicken cells after avian leukosis virus subgroup J infection. Gene, 2019, 701: 72-81. DOI:10.1016/j.gene.2019.03.030 |
| [39] | Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Zhang ZN, Huang ZJ, Zhang L, Wang HQ, Xie YL, Tong XY. Transcriptome of Apis cerana cerana larval gut under the stress of Ascosphaera apis. Scientia Agricultura Sinica, 2017, 50(13): 2614-2623. (in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 张曌楠, 黄枳腱, 张璐, 王鸿权, 解彦玲, 童新宇. 中华蜜蜂幼虫肠道响应球囊菌早期胁迫的转录组学. 中国农业科学, 2017, 50(13): 2614-2623. |
| [40] | Guo R, Chen DF, Huang ZJ, Liang Q, Xiong CL, Xu XJ, Zheng YZ, Zhang ZN, Xie YL, Tong XY, Hou ZX, Jiang LL, Dao C. Transcriptome analysis of Ascosphaera apis stressing larval gut of Apis cerana cerana. Acta Microbiologica Sinica, 2017, 57(12): 1865-1878. (in Chinese) 郭睿, 陈大福, 黄枳腱, 梁勤, 熊翠玲, 徐细建, 郑燕珍, 张曌楠, 解彦玲, 童新宇, 侯志贤, 江亮亮, 刀晨. 球囊菌胁迫中华蜜蜂幼虫肠道过程中病原的转录组学研究. 微生物学报, 2017, 57(12): 1865-1878. |
| [41] | Du Y, Tong XY, Zhou DD, Chen DF, Xiong CL, Zheng YZ, Xu GJ, Wang HP, Chen HZ, Guo YL, Long Q, Guo R. MicroRNA responses in the larval gut of Apis cerana cerana to Ascosphaera apis stress. Acta Microbiologica Sinica, 2019, 59(9): 1747-1764. (in Chinese) 杜宇, 童新宇, 周丁丁, 陈大福, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 陈华枝, 郭意龙, 隆琦, 郭睿. 中华蜜蜂幼虫肠道响应球囊菌胁迫的microRNA应答分析. 微生物学报, 2019, 59(9): 1747-1764. |
| [42] | Guo R, Wang HP, Chen HZ, Xiong CL, Zheng YZ, Fu ZM, Zhao HX, Chen DF. Identification of Ascosphaera apis microRNAs and investigation of their regulation networks. Acta Microbiologica Sinica, 2018, 58(6): 1077-1089. (in Chinese) 郭睿, 王海朋, 陈华枝, 熊翠玲, 郑燕珍, 付中民, 赵红霞, 陈大福. 蜜蜂球囊菌的microRNA鉴定及其调控网络分析. 微生物学报, 2018, 58(6): 1077-1089. |
| [43] | Guo R, Chen DF, Diao QY, Xiong CL, Zheng YZ, Hou CS. Transcriptomic investigation of immune responses of the Apis cerana cerana larval gut infected by Ascosphaera apis. Journal of Invertebrate Pathology, 2019, 166: 107210. DOI:10.1016/j.jip.2019.107210 |
| [44] | Wang Q, Sun LX, Xiao PX, Liu F, Kang MJ, Xu BH. Study on technology for indoor artificial feeding of Apis cerana cerana larvae. Shandong Agricultural Sciences, 2009(11): 113-116. (in Chinese) 王倩, 孙亮先, 肖培新, 刘锋, 康明江, 胥保华. 室内人工培育中华蜜蜂幼虫技术研究. 山东农业科学, 2009(11): 113-116. |
| [45] | Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology, 2013, 14(4): R36. DOI:10.1186/gb-2013-14-4-r36 |
| [46] | Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology, 2009, 10(3): R25. |
| [47] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT Method. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [48] | Wang JZ, Liu K, Liu Y, Lv Q, Zhang F, Wang HY. Evaluating the bias of circRNA predictions from total RNA-Seq data. Oncotarget, 2017, 8(67): 110914-110921. DOI:10.18632/oncotarget.22972 |
| [49] | Samaranayake YH, Samaranayake LP, Pow EHN, Beena VT, Yeung KWS. Antifungal effects of lysozyme and lactoferrin against genetically similar, sequential Candida albicans isolates from a human immunodeficiency virus-infected southern Chinese cohort. Journal of Clinical Microbiology, 2001, 39(9): 3296-3302. DOI:10.1128/JCM.39.9.3296-3302.2001 |
| [50] | Sardiello M, Palmieri M, di Ronza A, Medina DL, Valenza M, Gennarino VA, Di Malta C, Donaudy F, Embrione V, Polishchuk RS, Banfi S, Parenti G, Cattaneo E, Ballabio A. A gene network regulating lysosomal biogenesis and function. Science, 2009, 325(5939): 473-477. DOI:10.1126/science.1174447 |
| [51] | McBride WH, Iwamoto KS, Syljuasen R, Pervan M, Pajonk F. The role of the ubiquitin/proteasome system in cellular responses to radiation. Oncogene, 2003, 22(37): 5755-5773. DOI:10.1038/sj.onc.1206676 |
| [52] | Ravanan P, Srikumar IF, Talwar P. Autophagy:The spotlight for cellular stress responses. Life Sciences, 2017, 188: 53-67. DOI:10.1016/j.lfs.2017.08.029 |
| [53] | Franssens V, Smagghe G, Simonet G, Claeys I, Breugelmans B, De Loof A, Vanden Broeck J. 20-Hydroxyecdysone and juvenile hormone regulate the laminarin-induced nodulation reaction in larvae of the flesh fly, Neobellieria bullata. Developmental and Comparative Immunology, 2006, 30(9): 735-740. DOI:10.1016/j.dci.2005.10.010 |
| [54] | Flatt T, Heyland A, Rus F, Porpiglia E, Sherlock C, Yamamoto R, Garbuzov A, Palli SR, Tatar M, Silverman N. Hormonal regulation of the humoral innate immune response in Drosophila melanogaster. The Journal of Experimental Biology, 2008, 211(16): 2712-2724. DOI:10.1242/jeb.014878 |
| [55] | Scott JG, Liu NN, Wen ZM. Insect cytochromes P450:diversity, insecticide resistance and tolerance to plant toxins. Comparative Biochemistry and Physiology Part C:Pharmacology, Toxicology and Endocrinology, 1998, 121(1/3): 147-155. |
| [56] | Greer EL, Brunet A. FOXO transcription factors at the interface between longevity and tumor suppression. Oncogene, 2005, 24(50): 7410-7425. DOI:10.1038/sj.onc.1209086 |
| [57] | Saraav I, Singh S, Sharma S. Outcome of Mycobacterium tuberculosis and Toll-like receptor interaction:immune response or immune evasion?. Immunology and Cell Biology, 2014, 92(9): 741-746. DOI:10.1038/icb.2014.52 |
| [58] | Myllym?ki H, R?met M. JAK/STAT pathway in Drosophila immunity. Scandinavian Journal of Immunology, 2014, 79(6): 377-385. DOI:10.1111/sji.12170 |
| [59] | Li YY, Zhang R, Liu SL, Donath A, Peters RS, Ware J, Misof B, Niehuis O, Pfrender ME, Zhou X. The molecular evolutionary dynamics of oxidative phosphorylation (OXPHOS) genes in Hymenoptera. BMC Evolutionary Biology, 2017, 17: 269. DOI:10.1186/s12862-017-1111-z |
| [60] | Xiong CL, Du Y, Wang HQ, Zheng YZ, Fu ZM, Wang HP, Zhang L, Chen DF, Guo R. Unraveling the mechanism regulating the Ascosphaera apis-resistance difference between Apis cerana cerana and Apis mellifera ligustica larvae based on comparative transcriptome analysis. Journal of China Agricultural University, 2019, 24(5): 106-114. (in Chinese) 熊翠玲, 杜宇, 王鸿权, 郑燕珍, 付中民, 王海朋, 张璐, 陈大福, 郭睿. 基于比较转录组学分析揭示中华蜜蜂及意大利蜜蜂幼虫的球囊菌抗性差异机制. 中国农业大学学报, 2019, 24(5): 106-114. |
| [61] | Valadkhan S, Gunawardane LS. lncRNA-mediated regulation of the interferon response. Virus Research, 2016, 212: 127-136. DOI:10.1016/j.virusres.2015.09.023 |
