魏鹏霖1,2, 张鹏2, 李伟2

 , 汪世华1
, 汪世华1
 , 尹文兵2
, 尹文兵2 1. 福建农林大学生命科学学院, 福建省病原真菌与真菌毒素重点实验室, 福建 福州 350002;
2. 中国科学院微生物研究所真菌学国家重点实验室, 北京 100101
收稿日期:2018-11-15;修回日期:2019-01-23;网络出版日期:2019-03-13
基金项目:国家重点研发计划(2016YFD0400105)
*通信作者:Tel/Fax:+86-10-64806170; 李伟, E-mail:liw@im.ac.cn;
汪世华, E-mail:wshyyl@sina.com.
摘要:[目的] 分生孢子色素是真菌细胞壁的重要成分,对真菌的生长发育极为重要,并有助于真菌抵御各种环境胁迫。本研究鉴定了黄曲霉分生孢子色素合成基因,并研究了分生孢子色素对黄曲霉生长发育及其对抗紫外照射和侵染能力的影响。[方法] 通过已知真菌孢子色素合成基因蛋白序列同源比对确定了黄曲霉分生孢子色素合成基因及其所在的基因簇,利用同源重组策略对目标基因进行敲除,获得了该色素合成基因缺失的突变菌株,并研究该基因敲除后对表型、产孢、菌核形成、黄曲霉毒素产生、抗紫外照射和侵染性等影响。[结果] 与野生型菌株相比,黄曲霉pks1基因缺失菌株的分生孢子颜色变为白色,生长速度、孢子产量、菌核形成和黄曲霉毒素B1的产生均没有显著性变化,但该基因的缺失导致孢子对紫外线照射的抵御能力明显减弱,降低了黄曲霉对玉米和花生种子的侵染能力。[结论] pks1(AFLA_006170)基因是黄曲霉分生孢子色素合成的关键基因,影响黄曲霉分生孢子对紫外线照射等不利环境因子的抵抗能力和对粮食种子的侵染能力。
关键词:黄曲霉分生孢子色素生长发育抗紫外性侵染性
Identification of conidial pigment synthetic gene pks1 and its effect on development and pathogenicity in Aspergillus flavus
Penglin Wei1,2, Peng Zhang2, Wei Li2

 , Shihua Wang1
, Shihua Wang1
 , Wen-Bing Yin2
, Wen-Bing Yin2 1. Fujian Key Laboratory of Pathogenic Fungi and Mycotoxins, School of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian Province, China;
2. State Key Laboratory of Mycology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
*Corresponding author: Tel/Fax:+86-10-64806170; Li Wei, E-mail:liw@im.ac.cn;
Wang Shihua, wshyyl@sina.com.
Foundation item: Supported by the the National Key Research and Development Program (2016YFD0400105)
Abstract: [Objective] The fungal conidial pigments are essential components for the formation of the fungal cell wall. The pigments play very important roles in fungal growth and resistance to various environmental stress responses. Here, the biosynthetic gene of conidial pigment was identified and characterized in Aspergillus flavus. [Methods] By the homologous search of the known pigment biosynthetic gene protein, the key gene involved in the pigment production was identified in A. flavus. The target gene was disrupted using homologous recombination strategy, and the effects of the gene deletion on fungal phenotype, sporulation, sclerotia formation, aflatoxin production, anti-ultraviolet irradiation and invasiveness were investigated. [Results] Compared with the wild-type strain, the Δpks1 mutant lost the conidial color instead of producing the white conidia. No obvious difference was detected in the growth rate, spore yield, formation of stress-resistant structure sclerotia and production of aflatoxin B1 between WT and Δpks1 mutant strains. However, the deletion of pks1 resulted in a decrease in resistance to UV radiation and the ability to infect corn and peanut seeds. [Conclusion] The pks1 (AFLA_006170) is a key gene for conidia pigment production in A. flavus. The conidial pigment biosynthetic pathway is related to fungal resistance against UV radiation and the infection to cereal crops.
Keywords: Aspergillus flavusconidial pigmentmorphological typeUV resistanceinfectivity
真菌孢子色素是真菌产生的一类重要的次级代谢产物,在真菌生长发育过程中发挥着重要的作用[1]。例如,类胡萝卜素(carotenoids)色素可起到抗氧化应激和对可见光或紫外线照射的保护作用[2];色素asparasone对于黄曲霉(Aspergillusflavus)的菌核存活非常重要[3];黑色素(melanin)对人类条件致病菌烟曲霉(Aspergillusfumigatus)分生孢子的完整性非常重要[4],也是烟曲霉致病过程中关键的毒力因子,通过阻碍宿主对LC3相关的吞噬来发挥作用[5-6];土曲霉(Aspergillusterreus)中的特征色素aspmelanin可以抵抗紫外线照射并能阻断土壤变形虫的吞噬作用[7];植物内生菌无花果拟盘多毛孢(Pestalotiopsisfici)的多细胞孢子中黑色素是分生孢子发育和保护分生孢子抵御各种环境胁迫所必不可少的成分[8]。真菌分生孢子色素合成机制的阐明对于理解这些次级代谢物影响真菌生长发育及其对抵御环境胁迫和致病的机理具有重要的意义。
黄曲霉是一种重要的腐生土壤真菌,是导致玉米和花生等粮食农作物和食物霉变的重要因素,黄曲霉产生的黄曲霉毒素(aflatoxin)会导致人类和动物中毒或诱发肝癌[9-10]。黄曲霉也是重要的动物和人类条件致病菌,通过侵入性生长导致免疫功能低下人群曲霉菌病(aspergillosis)发病率的增加[10]。分生孢子是黄曲霉分散传播及感染食物和宿主的主要形态,而致病因子与其孢子形成和次级代谢相关[10]。目前已知黄曲霉分生孢子的形成受到brlA、veA和velB等基因以及全局调控因子LaeA和DmtA甲基转移酶的调控[11-13],但其分生孢子色素的生物合成基因尚未确定。本研究以黄曲霉分生孢子色素合成基因为研究目标,利用黄曲霉基因组序列和已知真菌孢子色素合成基因进行序列分析比对,获得黄曲霉孢子色素合成基因pks1及其所在基因簇,并研究了pks1基因缺失后对黄曲霉表型、产孢、菌核形成、黄曲霉毒素产生、抗紫外照射和侵染性等的影响。
1 材料和方法 1.1 菌株和培养基 本研究所用的黄曲霉菌株见表 1,其中黄曲霉A. flavus CA14 PTs作为受体菌株用于构建目标基因缺失菌株,A. flavus Wild-type菌株作为对照菌株。PDA培养基(BD Difco,United States)用于菌株活化和孢子收集;GMM培养基(葡萄糖10.00 g/L,NaNO3 6.00 g/L,KCl 0.52 g/L,MgSO4·7H2O 0.52 g/L,KH2PO4 1.52 g/L,酒石酸铵10 mmol/L,pH 6.5)用于菌丝生长和分生孢子数量测定[14];YES培养基(Yeast extract 20 g/L,Sucrose 150 g/L,MgSO4·7H2O 1 g/L)用于产毒素检测[15];WKM培养基(Yeast extract 2 g/L,Peptone 3 g/L,Corn steep solids 5 g/L,Dextrose 2 g/L,Sucrose 30 g/L,NaNO3 2 g/L,K2HPO4·3H2O 1.0 g/L,MgSO4·7H2O 0.5 g/L,KCl 0.2 g/L,FeSO4·7H2O 0.1 g/L,pH 5.5)用于产菌核培养[11]。
表 1. 本研究所用的菌株 Table 1. Fungal strains used in this study
| Strain | Description | References |
| A. flavus CA14 PTs | Δku70; ΔpyrG | [16] |
| A. flavus Wild-type | Δku70; ΔpyrG::AfpyrG | [17] |
| TYPL1.1 | Δku70; ΔpyrG; Δpks1::AfpyrG | This study |
| TYPL 2.1 | Δku70; ΔpyrG; Δrum1::AfpyrG | This study |
| TXX = original transformant. | ||
表选项
1.2 目标基因的生物信息学和系统进化分析 在NCBI数据库中获得已知真菌分生孢子色素合成基因的蛋白序列,利用NCBI中的BLASTp在线软件对已报道的黑曲霉(Aspergillusniger)孢子色素合成骨架基因albA (ANI_1_726084)蛋白序列比对分析,确认黄曲霉中分生孢子色素合成基因,并通过antiSMASH在线软件分析进而获得该基因所在的基因簇信息[18]。根据NCBI已有的真菌孢子色素合成基因蛋白序列信息,利用MEGA 6软件中ClustalW初步处理蛋白序列,并用Maximum likelihood tree方法构建进化树,通过分析100个重复计算Bootstrap支持值。
1.3 目标基因缺失菌株的构建 根据获得的黄曲霉假定分生孢子色素合成基因序列设计引物,以黄曲霉基因组DNA为模板进行PCR扩增,扩增敲除基因的同源臂片段,以pyrG基因作为选择标记基因,通过Double-Joint PCR法构建目标基因同源重组片段[19]。利用原生质体和PEG介导转化法获得黄曲霉敲除转化子[20],并通过多对引物进行PCR鉴定,获得正确的目标基因缺失菌株。PCR仪为Bio-Rad公司的T100TM Thermal cycler,所用酶为TransStart FastPfu DNA聚合酶(Transgene Biotech,北京)。本研究所用引物序列见表 2。
表 2. 本研究中所用的引物 Table 2. PCR primer sets used in this study
| Name | Oligonucleotide sequence (5′→3′) | Uses |
| KOpks1_5f_F | GCATCGCTATGAAACTCG | pks1 deletion and probe |
| KOpks1_5f_R | GGGTGAAGAGCATTGTTTGAGGCCCTATCGTTCATATGGC | |
| KOpks1_3f_F | GCATCAGTGCCTCCTCTCAGACGGTCAAGAAAGTGCCAGTC | |
| KOpks1_3f_R | TTGCCGTTGCTCCGATGT | |
| pks1 5F_F | AGACGGCTCGGGAAGATA | |
| pks1 3F_R | GGCATACTGCGTGGAAAA | |
| KOrum1_5f_F | CGGGGTTCAAATTACTGC | rum1 deletion and probe |
| KOrum1_5f_R | GGGTGAAGAGCATTGTTTGAGGCATTGGCGCTTCATGGGTT | |
| KOrum1_3f_F | GCATCAGTGCCTCCTCTCAGACCGATCTGATCGTGAGCGGAAG | |
| KOrum1_3f_R | CAGATGGACCTGTGAAGATGC | |
| rum1 5F_F | GCGTGCGGCATATCTCATTG | |
| rum1 3F_R | CTCGTCACATCTCACCGTTTG | |
| pyrG_F | GCCTCAAACAATGCTCTTCACCC | gene deletion |
| pyrG_R | GTCTGAGAGGAGGCACTGATGC | |
| pks1 RT_F | GGTTACCACTGTGGCATCTC | pks1 mutant screen |
| pks1 RT_R | CATGATCCACGGTCTTTTTG | |
| rum1 RT_F | CAACTGGAGCCATTGACCTG | rum1 mutant screen |
| rum1 RT_R | GCCAATCGCAGATAGGACAG | |
| aflO/qRT-F | GTCTTTGCCAACTCGGATGC | RT-PCR for AFB1 biosynthesis cluster |
| aflO/qRT-R | ACATGGTTAGGTCACTGCCG | |
| aflS/qRT-F | CCAGACTCGGCCTTAGCTTC | |
| aflS/qRT-R | CGTGGAGGATACGCTCACTC | |
| aflR/qRT-F | CTCAAGGTGCTGGCATGGTA | |
| aflR/qRT-R | GGCTGGTCTTCCTCATCCAC | |
| aflD/qRT-F | TCCTTGGTCGCCCATATCAG | |
| aflD/qRT-R | CCAACATGCACGACTATGCG | |
| aflC/qRT-F | ACGACGCTTACCATGGAGTG | |
| aflC/qRT-R | GTACTCAGACACAGACCGGC | |
| 6120/qRT-F | TTATCGGTTCCAGCTCGGC | RT-PCR for conidial pigment biosynthesis cluster |
| 6120/qRT-R | AATACGATGCCAGTAGCGCA | |
| 6150/qRT-F | AAGAGAAGTGCTGTCGGTGG | |
| 6150/qRT-R | GCGTACTCGTCCAGATACTCC | |
| 6160/qRT-F | ATGCAAACCCGCTTACAGGA | |
| 6160/qRT-R | GCGCTCATCCTTTAGCTTGC | |
| 6170/qRT-F | GCCTTTGAACCGCCAACAG | |
| 6170/qRT-R | CGCACTTATGTGAGGATGCAG | |
| 6180/qRT-F | AGGTGGAACCTTTGAGTCGT | |
| 6180/qRT-R | GTACATTGTCCTGGAGCCGA | |
| 6230/qRT-F | GTGGGAGTGGAAGTACCGTG | |
| 6230/qRT-R | CAATGAGATATGCCGCACGC | |
| 6240/qRT-F | CGCCAATCGCAGATAGGACA | |
| 6240/qRT-R | GGGAAATCACGGAAGCGAGA | |
| actin-F | ACGGTGTCGTCACAAACTGG | |
| actin-R | CGGTTGGACTTAGGGTTGATAG |
表选项
1.4 生长发育测定 根据文献提供的多种方法对突变株分生孢子产量、菌落生长速度和菌核形成及产量研究黄曲霉生长发育情况进行了研究[13-15]。在加有20 mL GMM培养基的直径为90 mm培养皿中央点接种1 μL浓度为106 conidia/mL的黄曲霉分生孢子悬浮液,在29 ℃黑暗培养7 d后分别测量分生孢子产量和菌落直径[14]。在加有20 mL WKM培养基的直径为90 mm的培养皿中央点接种1 μL浓度为106 conidia/mL的黄曲霉分生孢子悬浮液,37 ℃黑暗培养7 d后,用75%乙醇喷洒平板表面去除分生孢子,然后在Asana显微镜下计数菌核数[17]。上述实验均重复3次。
1.5 RNA提取和定量RT-PCR 将黄曲霉对照菌株和突变株分别接种在GMM和YES固体培养基(培养基表面放玻璃纸便于收集菌体)上25 ℃培养7 d,用TranZolTM试剂盒(Transgen Biotech,China)提取菌丝体总RNA,利用Fast Quant RT试剂盒(Tiangen Biotech,China)进行反转录获得单链cDNA,利用CFX96实时系统(Bio-Rad)进行qRT-PCR分析。具体操作参照KAPA SYBR FAST qPCR试剂盒说明书进行,反应条件为:95 ℃ 3 min;95 ℃ 3 s,60 ℃ 20 s,72 ℃ 20 s,40个循环;然后65 ℃循环5 s,并保持95 ℃以计算解离曲线。每个cDNA样品一式3份进行,并计算平均阈值循环。使用2–ΔΔCt方法计算相对表达水平[8],最后将结果标准化为Actin蛋白基因(GenBank No.:AFLA_055230)的表达水平。所用引物在表 2列出。
1.6 抗紫外线照射能力测定 黄曲霉菌株在PDA培养基上37 ℃培养4 d后,用浓度为0.1% Tween-80水溶液收集分生孢子,取40 μL浓度为2.5×103 conidia/mL的孢子悬液接种在含有PDA培养基的90 mm培养皿上,并将培养皿分别暴露于0、20、50、100 J/m2的剂量紫外线(UV)下,然后在37 ℃培养24 h,显微镜观察孢子萌发[21]。所有实验均以3次重复进行,未照射的平板作为对照。UV光由UV交联机(UVP-CL1000 Ultraviolet Crosslinker)提供,紫外光波长为254 nm,照射时间由UV剂量决定。使用软件GraphPad Prism 7.0根据Two-Way ANOVA多重比较测试在P < 0.001下分析数据。
1.7 黄曲霉毒素分析 利用薄层色谱法(thin-layer chromatography,TLC)确定菌株黄曲霉毒素(aflatoxin B1,AFB1)的产生[22]。先用氯仿提取培养物中的次级代谢产物,然后在TLC薄层层析板上用丙酮:氯仿(1:9,V/V)溶剂系统作为展开剂对其中的AFB1进行分析鉴定,同时用AFB1标准品作为对照,AFB1在365 nm的紫外激活下显示出蓝色特异斑点。
1.8 种子侵染测定 以花生和玉米种子作为实验材料进行黄曲霉对种子侵染测验,具体操作参照文献进行[23]。除去花生和玉米种子胚芽而不损坏任何子叶组织后进行表面消毒,消毒步骤为:0.05%次氯酸钠溶液灭菌3 min →无菌水洗涤30 s → 70%乙醇灭菌5 s →无菌水洗涤30 s。种子消毒后沥干,然后每40粒种子分别浸入40 mL 0.1% Tween-80分生孢子悬浮液中(105 conidia/mL),浸入不含分生孢子的40 mL 0.1% Tween-80无菌水溶液的种子为空白对照,在摇床上以50 r/min摇动30 min,然后将花生和玉米种子分别置于90 mm培养皿中,每个培养皿摆放10粒玉米或10瓣花生,每个菌株3个平行,在29 ℃黑暗条件下孵育7 d。所用培养皿衬有1片湿滤纸(形成湿度室)以保持高湿度,滤纸每天用无菌水润湿,最后将种子收集到50 mL离心管中,加入10 mL 0.1% Tween-80无菌水涡旋3 min洗涤种子表面分生孢子,取100 μL孢子悬浮液稀释后使用血球计数板计数。分别再加入10 mL氯仿提取黄曲霉毒素,使用薄层色谱法进行结果分析。上述实验重复3次。
2 结果和分析 2.1 黄曲霉分生孢子色素合成基因的生物信息学分析 以黑曲霉孢子色素合成骨架基因albA (ANI_1_726084)蛋白序列为探针搜索黄曲霉A. flavus NRRL3357的基因组测序数据(GenBank No.:AAIH00000000.2),获得假定的分生孢子色素生物合成基因pks1 (AFLA_006170),然后利用antiSMASH在线分析预测黄曲霉pks1基因所在基因簇。结果显示,该基因簇含有NAD合成酶(AFLA_006120)、NADH焦磷酸酶(AFLA_006130)、黄素依赖性单加氧酶(AFLA_006150)、预测的HDA1复合亚基(AFLA_006160)、PKS基因(AFLA_006170,pks1)、外切聚磷酸酶(AFLA_006220)、羧酸脱氨酶(AFLA_006230)和转录因子(AFLA_006240,rum1)各1个,以及2个氧化酶(AFLA_006180和AFLA_006190) (图 1)。该基因簇的10个基因与米曲霉(Aspergillus oryzae)的孢子色素合成基因簇基因高度保守,其中pks1为该基因簇的骨架基因,与A.niger (ANI_1_726084,albA)、A.nidulans (AN8209,wA)和A.fumigatus (AFUA_2G17600,alb1)的黑色素生物合成基因相似性均在73%以上,而与非曲霉属的真菌P.fici黑色素生物合成基因(PFICI_07101,PfmaE)的相似性仅为44% (表 3)。将pks1基因与已知真菌分生孢子色素合成基因的编码蛋白序列进行系统发育进化分析,结果显示,曲霉属真菌属于同一个进化分支,且A.flavus与A.oryzae的距离最近,与已知的Altermariaalternate (Alm)、Magnaportheoryzae (MGG_07219)及P.fici黑色素合成基因距离较远(图 2)。上述结果表明黄曲霉中分生孢子色素合成途径应为类似于黑色素的1-DHN合成途径。
 |
| 图 1 黄曲霉分生孢子色素合成基因pks1基因簇的同源性比对 Figure 1 Homologue alignment of pks1 cluster in A. flavus |
| 图选项 |
 |
| 图 2 不同真菌中PKS1同源蛋白的系统进化关系分析 Figure 2 Phylogenetic analysis of PKS1 homologous protein from different fungi |
| 图选项 |
表 3. 黄曲霉分生孢子色素合成基因簇与不同真菌的比对分析 Table 3. Gene prediction through deep bioinformatic analysis of the pks1 cluster in different fungi
| Protein of Aspergillus flavus | Coverage/identity to gene of strains/% | Putative function | ||||
| Aspergillus oryzae | Aspergillus niger | Aspergillus fumigatus | Aspergillus nidulans | Pestalotiopsis fici | ||
| AFLA_006120 | AO090102000540 (100/93) | ANI_1_710084 (100/89) | – | AN8203 (100/83) | – | Glutamine-dependent NAD(+) synthetase |
| AFLA_006130 | AO090102000541 (100/99) | ANI_1_1498084 (100/79) | – | AN8204 (100/79) | – | NADH pyrophosphatase |
| AFLA_006150 | AO090102000543 (95/90) | ANI_1_1502084 (95/59) | – | AN8206 (95/56) | – | Flavin-containing monooxygenase |
| AFLA_006160 | AO090102000544 (100/99) | ANI_1_724084 (98/68) | – | AN8208 (99/61) | – | HDA1 complex subunit |
| AFLA_006170 (pks1) | AO090102000545 (100/99) | ANI_1_726084 (100/75) | AFUA_2G17600 (100/73) | AN8209 (100/73) | PFICI_07101 (99/44) | Polyketide synthetase PksP |
| AFLA_006180 | AO090102000546 (99/99) | – | AFUA_2G17530 (99/48) | – | PFICI_07103 (11/80) | Conidial pigment biosynthesis oxidase Arb2/brown2 |
| AFLA_006220 | AO090102000550 (100/100) | ANI_1_1506084 (98/53) | – | – | – | Exopolyphosphatase |
| AFLA_006230 | AO090102000551 (100/99) | ANI_1_1508084 (88/57) | – | – | – | 1-aminocyclopropane-1-carboxylate deaminase |
| AFLA_006240 (rum1) | AO090102000552 (100/99) | ANI_1_734084 (100/87) | AFUA_2G17640 (9/26) | AN8211 (99/79) | PFICI_07104 (11/43) | PHD transcription factor |
表选项
2.2 黄曲霉分生孢子色素合成基因(pks1)缺失菌株的构建 生物信息学分析表明,黄曲霉分生孢子色素合成基因pks1 (AFLA_006170)可读阅读框为6651 bp,编码蛋白为2141个氨基酸。利用分子遗传技术及原生质体转化方法构建了pks1缺失菌株(图 3-A),提取转化子的基因组DNA,通过PCR的方法验证阳性转化子(图 3-B)。采用相同的方法获得转录因子Rum1基因缺失的菌株。
 |
| 图 3 黄曲霉pks1缺失株的构建 Figure 3 Generation of pks1 deletion mutant in A. flavus. A: Schematic illustration for pks1 disruption; B: Verification of Δpks1 mutants by diagnostic PCR. The two specific bands were detected in Δpks1 mutants (T1–T3) but not in WT |
| 图选项 |
2.3 pks1是黄曲霉分生孢子色素合成的关键基因 为验证pks1基因是否是黄曲霉分生孢子色素合成的关键基因,将WT和Δpks1菌株接到GMM培养基上于29 ℃培养7 d。结果显示Δpks1突变株的生长菌落变为白色,即分生孢子没有绿色色素产生(图 4-A)。结果表明,pks1是黄曲霉分生孢子色素合成的关键基因。在相同的培养条件下测定菌落生长直径和分子孢子产量,结果发现,Δpks1突变株与WT对照菌株相比没有显著的差异(图 4-B,C),表明pks1基因的缺失不影响菌落生长和分生孢子产生。
 |
| 图 4 亲本菌株和Δpks1突变株菌落生长及分生孢子产生 Figure 4 Morphology, sporulation and growth of WT and Δpks1 mutant. A: Phenotypes of WT and Δpks1 strains; B: Colony diameter of WT and Δpks1 strains; C: Conidia production of WT and Δpks1 strains |
| 图选项 |
2.4 pks1基因不参与菌核的形成 为确定缺失pks1基因是否对黄曲霉菌核形成产生影响,将WT和Δpks1菌株接到WKM培养基于29 ℃黑暗培养7 d后,用75%酒精洗去菌落表面菌丝和孢子,观察其菌核表型变化,并进行菌核计数。结果显示,Δpks1菌株和WT对照菌株相同,均产生菌核(图 5-A),而且两者产生的菌核数目也没有显著的差异(图 5-B)。可见,pks1基因的缺失不参与黄曲霉菌核的形成。
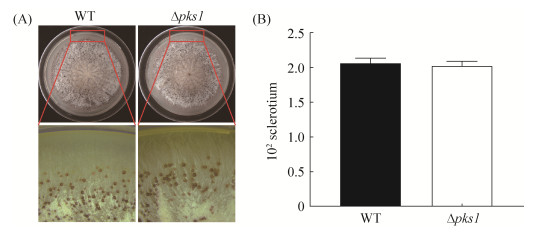 |
| 图 5 基因缺失对黄曲霉菌核形成的影响 Figure 5 Effect of pks1 disruption on sclerotia production in A. flavus. A: Colony morphology of WT and Δpks1 strains; B: Sclerotia production of WT and Δpks1 strains |
| 图选项 |
2.5 黄曲霉分生孢子色素影响对紫外线照射的耐受性 利用不同的UV剂量照射分生孢子测定萌发率。结果显示,在不进行UV照射的情况下(0 J/m2),WT对照菌株和Δpks1突变菌株分生孢子的萌发率没有差异。当UV能量提高到20和50 J/m2时,与WT对照菌株相比,Δpks1菌株的萌发率显著降低;当UV能量提高到100 J/m2时,Δpks1菌株的孢子萌发率急剧下降(图 6)。由此表明,pks1的缺失严重影响了黄曲霉分生孢子抵御紫外线辐射的能力。
 |
| 图 6 Δpks1突变菌株影响黄曲霉分生孢子对紫外线耐受性 Figure 6 Δpks1 mutant affects the sensitivity of spores to UV in A. flavus. ***P≤0.01 |
| 图选项 |
2.6 转录因子Rum1是黄曲霉分生孢子色素的生物合成非关键性调控因子 为研究黄曲霉中转录因子Rum1是否对分生孢子色素合成有调控作用,利用qRT-PCR分析了WT和Δrum1 (TYPL2.1)菌株分生孢子色素合成基因所在基因簇中的基因(AFLA_006120、AFLA_006150、AFLA_006160、pks1、AFLA_006180和AFLA_006230)的转录水平的表达情况。结果发现,分生孢子色素生物合成的关键基因pks1的表达在rum1缺失突变菌株中呈下调趋势(图 7),但rum1缺失突变菌株分生孢子色素与亲本及菌株相比并没有明显变化,表明转录因子Rum1并非是黄曲霉分生孢子色素生物合成的关键性调控因子。
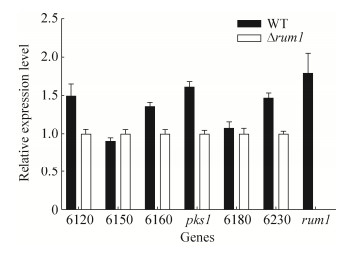 |
| 图 7 转录因子Rum1缺失对黄曲霉色素合成基因转录水平的影响 Figure 7 Effect of rum1 deletion on transcriptional levels of conidial pigment biosynthesis genes in A.flavus |
| 图选项 |
2.7 pks1对黄曲霉毒素B1的生物合成无显著性影响 为探究pks1缺失对黄曲霉毒素B1生物合成的作用,将WT和Δpks1菌株接到YES固体培养基中于29 ℃培养7 d,收集样品并利用TLC法测定AFB1的产量。结果表明,与WT对照菌株相比,Δpks1突变菌株中AFB1产生量没有显著变化(图 8-A,B)。利用qRT-PCR分析AFB1生物合成相关基因(aflR、aflS、aflC、aflD和af1O)的表达水平,结果发现,与WT对照菌株相比,调控基因aflR/S和甲基转移酶基因af1O表达水平在Δpks1菌株中显著上调,还原酶基因aflD的表达水平下调,而突变菌株中AFB1合成途径中关键前体Norsolorinic acid (NOR)合成基因aflC的表达水平并无显著性差异(图 8-C)。由此表明,pks1缺失对AFB1的产量无明显影响。
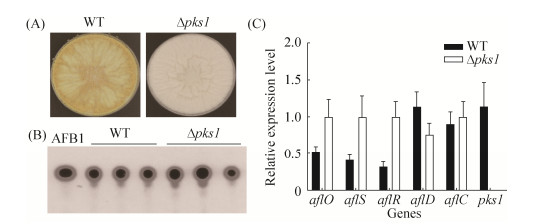 |
| 图 8 Δ pks1突变菌株对黄曲霉毒素合成的影响 Figure 8 Effect of pks1 deletion on the aflatoxin biosynthesis in A. flavus. A: Phenotypic characterization of WT and Δpks1 strains; B: AFB1 production of WT and Δpks1 strains by TLC assay; C: Transcriptional levels of aflatoxin biosynthesis related genes in WT and Δpks1 strains |
| 图选项 |
2.8 pks1缺失影响黄曲霉对农作物种子的侵染能力 为检测pks1缺失是否影响黄曲霉分生孢子对农作物种子侵染性,分别将玉米和花生与WT和Δpks1突变菌株的分生孢子悬浮液混合,在29 ℃培养7 d后观察和计数分生孢子,并检测黄曲霉侵染后的玉米和花生种子中AFB1的产生。结果显示,与WT对照菌株相比,Δpks1突变菌株侵染的玉米和花生中AFB1产量与WT菌株相比均没有显著的变化(图 9-C,D),但Δpks1突变菌株产生分生孢子数量明显减少(图 9-A,B)。可见,Δpks1突变菌株的分生孢子对玉米和花生种子的侵染传播效果显著下降,分生孢子色素合成基因pks1的缺失影响了黄曲霉对农作物种子的侵染能力。
 |
| 图 9 Δpks1突变菌株对玉米和花生种子的侵染性及产黄曲霉毒素的影响 Figure 9 Effect of pks1 deletion on infectivity and aflatoxin production on the maize and peanut seeds. A: Infectivity on peanut and maize seeds by WT and Δpks1 strains; B: Quantification of conidia from the infected peanut and maize seeds; C: AFB1 production in infected maize seeds by TLC assay; D: AFB1 production in infected peanut seeds by TLC analysis. **P≤0.01 |
| 图选项 |
3 讨论 真菌分生孢子色素参与细胞壁的形成,并帮助真菌抵御氧化胁迫、紫外线照射、土壤变形虫吞噬、菌核的生存胁迫等各种环境胁迫因子[24],真菌孢子色素合成机制的阐明对于理解重要真菌类群如何抵御环境胁迫及其致病性十分重要。本研究在对已知真菌孢子色素合成基因进行生物信息学分析的基础上,获得黄曲霉中孢子色素合成的同源基因pks1及其所在基因簇,通过基因敲除的方法获得pks1基因缺失的突变菌株,并证实了黄曲霉pks1基因是其分生孢子色素合成的关键基因,pks1基因缺失不能影响分生孢子产量和真菌的生长,但降低了黄曲霉对于紫外线照射的抵抗能力和粮食种子的侵染性。
真菌分生孢子色素的合成机制比较复杂,其中黑色素生物合成途径主要包括1, 8-二羟基萘(DHN)和1-3, 4-二羟基苯丙氨酸(1-DOPA)两种途径,前者需要聚酮合成酶基因、scytalone脱水酶基因和1, 3, 8-THN还原酶基因的参与。基因组序列分析结果表明,1, 8-DHN-黑色素生物合成途径在曲霉属真菌中多是保守的[24]。然而,近年来发现土曲霉缺乏合成DHN类型黑色素的基因,而其色素aspmelanin是由NRPS类型基因melA参与负责合成的[7],构巢曲霉中NRPS类型基因ivoA(AN10576)则与其灰棕色分生孢子色素的生物合成有关[25],这些研究丰富了我们对真菌色素合成机制的认识。本研究所鉴定的黄曲霉孢子色素合成基因pks1与黑曲霉albA基因、烟曲霉alb1基因、构巢曲霉wA基因具有高度的保守性和相似性(表 3和图 2),分生孢子色素合成途径属于黑色素的1, 8-二羟基萘(DHN)途径。
孢子色素影响真菌分生孢子的发育、紫外线耐受性及致病性。如:链格孢菌(Alternariaalternata)中黑色素生物合成基因alm和brm2均可以恢复稻瘟病菌(Magnaporthegrisea)黑色素缺陷型突变体的致病性[26],敲除brm2基因则影响链格孢菌分生孢子的发育和紫外线耐受性[27];链格孢菌黑色素生物合成基因pks在昆虫病原菌绿僵菌(Metarhiziumanisopliae)中的异源表达增加了菌株的毒力[28];小孢拟盘多毛孢菌(Pestalotiopsis microspora)pks基因参与的黑色素生物合成对分生孢子具有负面作用,但对维持细胞的完整性和生存能力也非常重要[29]。本研究中发现色素基因pks1的缺失虽不影响黄曲霉毒素的产量,但能降低黄曲霉的侵染性,这与烟曲霉中同源基因alb1敲除后降低了其致病传染性的结论相似[30]。此外,真菌分生孢子色素的合成受转录因子等多方面因素的调控,如烟曲霉分生孢子黑色素的产生受bHLH DevR和MADS-box RlmA两个转录因子的调控[31]。我们发现黄曲霉中的转录调控因子Rum1与分生孢子色素合成基因pks1在同一基因簇中,该转录因子在菌丝体、分生孢子、菌核生长以及黄曲霉毒素生物合成中起作用[17],然而本研究发现转录因子Rum1的缺失并未影响突变菌株分生孢子色素的产生,表明该转录因子Rum1不是黄曲霉分生孢子色素生物合成的关键性调控因子。因此,黄曲霉分生孢子色素合成基因pks1的表达调控机制有待于进一步深入研究。
构巢曲霉的白色和黄色孢子颜色突变菌株已被广泛用于鉴定和表征基因及其编码的色素合成的蛋白质,而烟曲霉孢子颜色突变菌株则被用于研究DHN-黑色素的生物合成途径以及黑色素在其致病性中的作用[32]。因此,黄曲霉孢子色素合成基因的确定和白色孢子突变菌株的获得为深入研究黄曲霉的产毒和侵染机制提供了重要的研究工具。
References
| [1] | Liu GY, Nizet V. Color me bad: microbial pigments as virulence factors. Trends in Microbiology, 2009, 17(9): 406-413. |
| [2] | Avalos J, Carmen Limón M. Biological roles of fungal carotenoids. Current Genetics, 2015, 61(3): 309-324. DOI:10.1007/s00294-014-0454-x |
| [3] | Cary JW, Harris-Coward PY, Ehrlich KC, di Mavungu JD, Malysheva SV, de Saeger S, Dowd PF, Shantappa S, Martens SL, Calvo AM. Functional characterization of a veA-dependent polyketide synthase gene in Aspergillus flavus necessary for the synthesis of asparasone, a sclerotium-specific pigment. Fungal Genetics and Biology, 2014, 64: 25-35. DOI:10.1016/j.fgb.2014.01.001 |
| [4] | Pihet M, Vandeputte P, Tronchin G, Renier G, Saulnier P, Georgeault S, Mallet R, Chabasse D, Symoens F, Bouchara JP. Melanin is an essential component for the integrity of the cell wall of Aspergillus fumigatus conidia. BMC Microbiology, 2009, 9: 177. DOI:10.1186/1471-2180-9-177 |
| [5] | Akoumianaki T, Kyrmizi I, Valsecchi I, Gresnigt MS, Samonis G, Drakos E, Boumpas D, Muszkieta L, Prevost MC, Kontoyiannis DP, Chavakis T, Netea MG, van de Veerdonk FL, Brakhage AA, El-Benna J, Beauvais A, Latge JP, Chamilos G. Aspergillus cell wall melanin blocks LC3-associated phagocytosis to promote pathogenicity. Cell Host & Microbe, 2016, 19(1): 79-90. |
| [6] | Jacobson ES. Pathogenic roles for fungal melanins. Clinical Microbiology Reviews, 2000, 13(4): 708-717. DOI:10.1128/CMR.13.4.708 |
| [7] | Geib E, Gressler M, Viediernikova I, Hillmann F, Jacobsen ID, Nietzsche S, Hertweck C, Brock M. A non-canonical melanin biosynthesis pathway protects Aspergillus terreus conidia from environmental stress. Cell Chemical Biology, 2016, 23(5): 587-597. DOI:10.1016/j.chembiol.2016.03.014 |
| [8] | Zhang P, Wang XN, Fan AL, Zheng YJ, Liu XZ, Wang SH, Zou HX, Oakley BR, Keller NP, Yin WB. A cryptic pigment biosynthetic pathway uncovered by heterologous expression is essential for conidial development in Pestalotiopsis fici. Molecular Microbiology, 2017, 105(3): 469-483. DOI:10.1111/mmi.2017.105.issue-3 |
| [9] | Sudhakar P, Latha P, Sreenivasulu Y, Reddy BVB, Hemalatha TM, Balakrishna M, Reddy KR. Inhibition of Aspergillus flavus colonization and aflatoxin (AfB1) in peanut by methyleugenol. Indian Journal of Experimental Biology, 2009, 47(1): 63-67. |
| [10] | Amaike S, Keller NP. Aspergillus flavus. Annual Review of Phytopathology, 2011, 49: 107-133. |
| [11] | Chang PK, Scharfenstein LL, Mack B, Ehrlich KC. Deletion of the Aspergillus flavus orthologue of A. nidulans fluG reduces conidiation and promotes production of sclerotia but does not abolish aflatoxin biosynthesis. Applied and Environmental Microbiology, 2012, 78(21): 7557-7563. |
| [12] | Chang PK, Scharfenstein LL, Li P, Ehrlich KC. Aspergillus flavus VelB acts distinctly from VeA in conidiation and may coordinate with FluG to modulate sclerotial production. Fungal Genetics and Biology, 2013, 58-59: 71-79. DOI:10.1016/j.fgb.2013.08.009 |
| [13] | Yang KL, Liang LL, Ran FL, Liu YH, Li ZG, Lan HH, Gao PL, Zhuang ZH, Zhang F, Nie XY, Kalayu Yirga S, Wang SH. The DmtA methyltransferase contributes to Aspergillus flavus conidiation, sclerotial production, aflatoxin biosynthesis and virulence. Scientific Reports, 2016, 6: 23259. DOI:10.1038/srep23259 |
| [14] | Lan HH, Sun RL, Fan K, Yang KL, Zhang F, Nie XY, Wang XN, Zhuang ZH, Wang SH. The Aspergillus flavus histone acetyltransferase AflGcnE regulates morphogenesis, aflatoxin biosynthesis, and pathogenicity. Frontiers in Microbiology, 2016, 7: 1324. |
| [15] | Yang KL, Qin QP, Liu YH, Zhang LM, Liang LL, Lan HH, Chen CH, You YC, Zhang F, Wang SH. Adenylate cyclase AcyA regulates development, aflatoxin biosynthesis and fungal virulence in Aspergillus flavus. Frontiers in Cellular and Infection Microbiology, 2016, 6: 190. |
| [16] | Chang PK, Scharfenstein LL, Wei QJ, Bhatnagar D. Development and refinement of a high-efficiency gene-targeting system for Aspergillus flavus. Journal of Microbiological Methods, 2010, 81(3): 240-246. |
| [17] | Hu YL, Yang G, Zhang DP, Liu YJ, Li Y, Lin GL, Guo ZQ, Wang SH, Zhuang ZH. The PHD transcription factor Rum1 regulates morphogenesis and aflatoxin biosynthesis in Aspergillus flavus. Toxins, 2018, 10(7): 301. DOI:10.3390/toxins10070301 |
| [18] | Medema MH, Blin K, Cimermancic P, de Jager V, Zakrzewski P, Fischbach MA, Weber T, Takano E, Breitling R. AntiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research, 2011, 39(S2): W339-W346. |
| [19] | Yu JH, Hamari Z, Han KH, Seo JA, Reyes-Domínguez Y, Scazzocchio C. Double-joint PCR: a PCR-based molecular tool for gene manipulations in filamentous fungi. Fungal Genetics and Biology, 2004, 41(11): 973-981. |
| [20] | Zhou ZY, Liu R, Jiang MY, Zhang L, Niu Y, Zhu YC, Dong ZJ, Liu JK. Two new cleistanthane diterpenes and a new isocoumarine from cultures of the Basidiomycete Albatrellus confluence. Chemical & Pharmaceutical Bulletin, 2009, 57(9): 975-978. |
| [21] | Eisenman HC, Casadevall A. Synthesis and assembly of fungal melanin. Applied Microbiology and Biotechnology, 2012, 93(3): 931-940. DOI:10.1007/s00253-011-3777-2 |
| [22] | Liu YH, Yang KL, Qin QP, Lin GN, Hu TR, Xu ZL, Wang SH. G protein α subunit gpaB is required for asexual development, aflatoxin biosynthesis and pathogenicity by regulating cAMP signaling in Aspergillus flavus. Toxins, 2018, 10(3): 117. DOI:10.3390/toxins10030117 |
| [23] | Kale SP, Milde L, Trapp MK, Frisvad JC, Keller NP, Bok JW. Requirement of LaeA for secondary metabolism and sclerotial production in Aspergillus flavus. Fungal Genetics and Biology, 2008, 45(10): 1422-1429. DOI:10.1016/j.fgb.2008.06.009 |
| [24] | Braga GUL, Rangel DEN, Fernandes KK, Flint SD, Roberts DW. Molecular and physiological effects of environmental UV radiation on fungal conidia. Current Genetics, 2015, 61(3): 405-425. |
| [25] | Sung CT, Chang SL, Entwistle R, Ahn G, Lin TS, Petrova V, Yeh HH, Praseuth MB, Chiang YM, Oakley BR, Wang CCC. Overexpression of a three-gene conidial pigment biosynthetic pathway in Aspergillus nidulans reveals the first NRPS known to acetylate tryptophan. Fungal Genetics and Biology, 2017, 101: 1-6. DOI:10.1016/j.fgb.2017.01.006 |
| [26] | Kawamura C, Moriwaki J, Kimura N, Fujita Y, Fuji SI, Hirano T, Koizumi S, Tsuge T. The melanin biosynthesis genes of Alternaria alternata can restore pathogenicity of the melanin-deficient mutants of Magnaporthe grisea. Molecular Plant-Microbe Interactions, 1997, 10(4): 446-453. DOI:10.1094/MPMI.1997.10.4.446 |
| [27] | Kawamura C, Tsujimoto T, Tsuge T. Targeted disruption of a melanin biosynthesis gene affects conidial development and UV tolerance in the Japanese pear pathotype of Alternaria alternata. Molecular Plant-Microbe Interactions, 1999, 12(1): 59-63. |
| [28] | Tseng MN, Chung PC, Tzean SS. Enhancing the stress tolerance and virulence of an entomopathogen by metabolic engineering of dihydroxynaphthalene melanin biosynthesis genes. Applied and Environmental Microbiology, 2011, 77(13): 4508-4519. DOI:10.1128/AEM.02033-10 |
| [29] | Yu X, Huo L, Liu H, Chen LF, Wang Y, Zhu XD. Melanin is required for the formation of the multi-cellular conidia in the endophytic fungus Pestalotiopsis microspora. Microbiological Research, 2015, 179: 1-11. DOI:10.1016/j.micres.2015.06.004 |
| [30] | Jahn B, Boukhallouk F, Lotz J, Langfelder K, Wanner G, Brakhage AA. Interaction of human phagocytes with pigmentless Aspergillus conidia. Infection and Immunity, 2000, 68(6): 3736-3739. DOI:10.1128/IAI.68.6.3736-3739.2000 |
| [31] | Valiante V, Baldin C, Hortschansky P, Jain R, Thywiβen A, Straβburger M, Shelest E, Heinekamp T, Brakhage AA. The Aspergillus fumigatus conidial melanin production is regulated by the bifunctional bHLH DevR and MADS-box RlmA transcription factors. Molecular Microbiology, 2016, 102(2): 321-335. DOI:10.1111/mmi.13462 |
| [32] | J?rgensen TR, Park J, Arentshorst M, van Welzen AM, Lamers G, Vankuyk PA, Damveld RA, van den Hondel CAM, Nielsen KF, Frisvad JC, Ram AFJ. The molecular and genetic basis of conidial pigmentation in Aspergillus niger. Fungal Genetics and Biology, 2011, 48(5): 544-553. DOI:10.1016/j.fgb.2011.01.005 |
