徐巧林, 宋艳钊, 郭岩彬


中国农业大学资源与环境学院, 生物多样性与有机农业北京市重点实验室, 北京 100193
收稿日期:2017-09-22;修回日期:2017-12-26;网络出版日期:2018-01-24
基金项目:国家自然科学基金(31470531,31200386)
*通信作者:郭岩彬, Tel:+86-10-62731457, Fax:+86-10-62732498, E-mail:guoyb@cau.edu.cn
摘要:细菌在生存过程中要面对复杂多样的环境,在长期进化过程中,细菌逐渐形成不同的应答机制来感应环境信号的变化,并通过精确的基因表达来调控生理生化反应。基因表达调控可分为转录水平和转录后水平两个方面,对于细菌来说,非编码RNA在转录后调控上发挥着重要的作用,而大多数非编码RNA与靶标mRNA的相互作用过程又离不开Hfq蛋白的辅助。本文综述了非编码RNA的分类、调控特点,伴侣蛋白Hfq的结构、功能以及两者相互作用的机制,以期深入了解非编码RNA及其伴侣蛋白Hfq在转录后调控中发挥的作用。
关键词: 转录后调控 非编码RNA Hfq
Non-coding RNA and RNA chaperone Hfq in bacteria
Qiaolin Xu, Yanzhao Song, Yanbin Guo


College of Resources and Environmental Sciences, China Agricultural University, Beijing Key Laboratory of Biodiversity and Organic Farming, Beijing 100193, China
Received 22 September 2017; Revised 26 December 2017; Published online 24 January 2018
*Corresponding author: Yanbin Guo, Tel:+86-10-62731457, Fax:+86-10-62732498, E-mail:guoyb@cau.edu.cn
Supported by the National Natural Science Foundation of China (31470531, 31200386)
Abstract: Bacteria have to adapt complex environment for survival in nature. For this environmental fitness, bacteria generate different response mechanisms to perceive environmental signals for regulating cell physiological and biochemical reaction in the long-term evolution process. Gene expression regulation is one of the response mechanisms with the transcription level and the post-transcriptional level. Non-coding RNAs (ncRNAs) play an important role in the post-transcriptional regulation in bacteria. Hfq is required for the action of many ncRNAs that act by base-pairing with target mRNAs. This review summarizes the function of ncRNAs and Hfq in post-transcriptional regulation, including:1. the classification and characteristics of ncRNAs; 2. the structure and function of Hfq protein; 3. non-coding RNAs and Hfq interaction.
Key words: post-transcriptional regulation non-coding RNAs Hfq
细菌虽然是单细胞生物,没有复杂的生命机制,但在长期的进化过程中也形成了一套相对简单却可以迅速响应外界环境变化的生存机制。细菌根据信号物质来感应环境的变化,并依靠精确的基因调控来适应环境[1-2]。细菌的基因调控在分子水平上可分为转录和转录后2个层面,转录后调控是指对mRNA的识别及翻译能否起始,其过程受到多种因素的影响,包括SD序列(Shine-Dalgarno sequence)、翻译产物(如核糖体蛋白或翻译终止因子)、反义RNA以及mRNA的稳定性等,非编码RNA也是其中重要的一部分,它通过起始或抑制翻译以及影响mRNA的稳定性来实现调控[3]。在大多数细菌中,Hfq作为RNA分子伴侣在转录后水平调控上发挥了关键性的作用[4]。
1 非编码RNA 在细菌中,除了三类传统的RNA,即转运RNA (tRNA)、核糖体RNA (rRNA)和信使RNA (mRNA)外,还有一类起调节作用的RNA,叫做小RNA (small RNA,sRNA),或者非编码RNA (non-coding RNA,ncRNA),长度在50–300个核苷酸的范围内,一般不被翻译成蛋白质[5]。非编码RNA大多数作为应答环境压力的调节元件,通过一定的机制调控基因的表达,在多种细胞生理过程中起着关键作用[6]。
1.1 非编码RNA的分类 在细菌中,非编码RNA分为两类,一类是持家RNA,第二类是调节RNA。持家RNA在细胞内均一持续表达。在大肠杆菌(Escherichia coli)中有3个具有持家功能的非编码RNA,分别为M1 RNA、tmRNA和4.5S RNA。M1 RNA是内切核糖核酸酶RNase P其中的一个亚基,RNase P作为一个催化RNA加工的典型核酶,能够将tRNA前体加工成具有成熟5′-端的tRNA[7]。tmRNA参与翻译的质量控制过程,能够将被受损mRNA占据的核糖体释放出来,重新循环利用,同时降解未完成合成的非正常多肽[8]。4.5S RNA作为信号识别颗粒(signal recognition particle,SRP)参与蛋白分泌的过程[9]。
调节RNA又可分为两大类,一类RNA与蛋白结合改变它的活性。例如,在E. coli中,6S RNA与RNA聚合酶结合,改变它对启动子识别的特异性,从而调节σ70因子的活性[10]。CsrA,是一个碳储存调节相关蛋白,至少受到CsrB和CsrC两个sRNA的调节,这2个sRNA分子作为CsrA蛋白的拮抗物,严格控制它的活性[11-12]。另一类调节RNA通过与同源的靶mRNA碱基配对来影响它的稳定性和翻译。根据配对方式的差别,sRNA分为顺式编码(cis-acting)和反式编码(trans-acting)两类[13]。顺式编码的sRNA与它的靶mRNA相应区段完全互补配对,它们分别以同一个基因的两条互补模板反向转录得到。反式编码的sRNA与它的靶mRNA的互补区域很短且不相邻,它定位于基因间,有多个靶标mRNA。在多数情况下,因为配对的碱基数有限,反式编码的sRNA与mRNA作用时需要Hfq的帮助。
1.2 非编码RNA的调控特点 非编码RNA的调节具有几点明显优势,一是非编码RNA很小(一般认为50–300个核苷酸的范围内),对细胞来说产生它的耗损少;二是非编码RNA产生速度快,不需要额外的翻译步骤,对信号反应迅速且灵敏[14];三是RNA能够支持阈值线性响应,基因表达与否或者表达的程度取决于sRNA和mRNA的相对水平[15-17];四是非编码RNA比蛋白产生速度快,在转录后水平发挥作用,几种动力模型表明,短期来看,它比依赖蛋白的转录调节因子更快地启动或者关闭基因表达[17-18]。
非编码RNA与靶标基因或mRNA的作用方式是多种多样的。例如,反式编码的sRNA能够调节多个不同的基因,使得这些sRNA能够在不同水平上控制整个生理网络。同时,sRNA与mRNA碱基配对的程度和特性可以区分不同mRNA调控的优先顺序,这样细胞能够在基因表达水平上整合不同的信号输入[19]。另外,同一个sRNA可以调节多个靶标mRNA,它们的丰度和结合的紧密性能够通过相互间的通讯强烈程度影响到另一个的表达。同时,多个sRNA也有可能作用于同一个靶标mRNA,例如,DsrA与低温应激有关,RprA与渗透压应激有关,OxyS与氧化应激有关,3个RNA在不同的条件下均激活或抑制同一个靶标RpoS的翻译[20-21]。此外有些mRNA调控作用是直接降解靶标mRNA,使得调控效果不可逆转[22]。
但是,并不是说sRNA的转录后调控作用时刻优于转录因子的转录调控,只有当外界输入大量和持久的信号时,sRNA能够比转录因子更强烈和稳定地控制基因表达,同时过滤掉干扰信号;而当输入信号很弱或者为瞬时信号时,sRNA调控在放大信号或者快速传递信号方面不如蛋白作用灵敏有效[13]。总之,sRNA的转录后调控不可或缺,是作为蛋白调控的补充,在转录因子调控之后和蛋白酶发挥效应之前起着重要作用。
2 分子伴侣Hfq Hfq (host factor for RNA phage Qβ replicase)又叫HF-1,是一个RNA分子伴侣蛋白,最早在E. coli Qβ噬菌体的RNA复制过程中作为必需的寄主因子被发现[23]。该蛋白是一个保守的同源六聚体蛋白,广泛存在于革兰氏阴性和阳性菌中,无论是生物信息学还是晶体结构分析都发现,它与真核生物的Sm、Sm-like (LSm)蛋白结构具有高度的同源性,因此也可称作类Sm蛋白[24]。在E. coli中,Hfq的每个单体由一个保守的N端Sm结构域和一个C末端尾巴构成,而C末端尾巴的长度和序列在不同的物种间差别较大[25]。
2.1 Hfq的结构特征 Hfq的单体呈α-β1–5折叠,是一个保守的核心结构,有Sm1和Sm2两个基序。Sm1基序包括β1–3折叠片,由32个氨基酸组成;Sm2基序包括β4–5折叠片,由14个氨基酸组成(图 1-A)[26-27]。Sm结构域六聚化后,Hfq形成一个圆盘状六聚体,圆盘中心有孔,并存在2个RNA结合面(图 1-B、C)。N端的α螺旋(α1)所在的一面不平整并带有大量的正电荷,这一面称为近侧面(proximal face);与近侧面相对应的一面称为远侧面(distal face),这一面比较平坦,与近侧面类似,远侧面也带有大量的正电荷(图 1-D)[28]。近侧面识别富含尿嘧啶(U-rich)的单链RNA序列,能够将其结合到靠近中心孔的结合口袋内, 多数sRNA都具有富含尿嘧啶的区域,因此Hfq多用近侧面与sRNA相互作用[26]。远侧面则识别多聚腺苷酸尾(polyA),优先与ARN、ARNN或者AAYAAYAA序列结合(R代表嘌呤,Y代表嘧啶,N可以是任何碱基)[28]。富含A的序列在mRNA的多聚腺苷酸尾和非翻译区很常见,因此远侧面对mRNA的结合很关键[29]。很早就有人推测,一个Hfq六聚体可以同时结合sRNA富含U的序列以及mRNA富含A的序列,从而帮助两者的配对。在Wang等的结果中这个猜测也得到了证实,富含腺嘌呤的单链RNA结合在Hfq的远端,而富含尿嘧啶的单链RNA结合在Hfq的近端[30]。
 |
| 图 1 Hfq的结构和它的两个RNA结合面模式图[26] Figure 1 The structure of Hfq and its interactions with RNA[26]. A: Secondary structural elements of the Hfq protomer, highlighting the conserved Sm1 and Sm2 sequence motifs; B: Each protomer is a compact α-β1-5 structural unit; C: The ring-like architecture of Hfq; D: Two faces of Hfq, proximal face and distal face. |
| 图选项 |
近年来发现在Hfq上还有第3个结合表面,即Hfq环状蛋白边缘的侧面。研究表明第3个结合表面对一些sRNA的结合是必需的,可能作用于sRNA逐步与mRNA结合、而伴侣蛋白Hfq从sRNA-Hfq-mRNA三元复合物中解离的过程[31],同时这个模型在边缘残基突变体体外实验中得到了支持[32]。
在Hfq蛋白中,紧接Sm结构域的C端,有一个柔性的尾巴Hfq CDT (C-terminal domain,CDT)。虽然Hfq的N端高度保守并形成Sm结构域,但其C-端尾巴的序列保守性相对来说较差,在不同的菌种中长度不一甚至缺失[24]。因此,人们曾一度认为Hfq CDT对于Hfq蛋白来说并没有确切的功能。然而,Arluison等在电子显微镜中观察到,缺失CTD的Hfq六聚体的形状与Hfq FL相比有一些变化;同时,在傅里叶变换红外光谱中也发现,缺失尾巴的Hfq六聚体β折叠的含量比全长蛋白的要少。总之,全长Hfq (Hfq FL)六聚体比缺失CTD的突变体要稳定[33]。因此,他们推测,CDT可以保护Hfq亚基间界面并稳定其六聚体结构。最新的研究发现,在E. coli中Hfq CDT对于加速碱基配对作用不明显,但对于双链RNA (double-stranded RNA,dsRNA)的释放是必需的。同时,Hfq CDT也可以调控sRNAs之间的竞争,提供sRNAs与近侧面和远侧面结合的动力学优势[34]。近年来的研究确认了CTD的一些功能,其重要性也逐渐被人们所认识到。
2.2 Hfq的功能 Hfq是一个全局性的调控因子,在细菌转录后水平发挥着重要的作用。大量研究表明,Hfq直接或间接参与了细菌多种代谢的调控,影响多种生理生化过程的正常进行,包括细菌的生长、游动性、趋化性、毒力、对环境压力的适应能力等等。
Hfq与生长有着密切的关系。大多数菌株的hfq突变体表现为生长缺陷,包括细菌的生长速率降低,很快进入稳定期,细胞的形态和大小发生变化等[35]。与E. coli类似,在鼠伤寒沙门氏菌(Salmonella typhimurium)和流产布鲁氏菌(Brucella abortus)中,hfq突变体的生长受损、生长速率降低,而在希瓦氏菌(Shewanella oneidensis)中,也有类似的结果,但不同的是,突变hfq后,该菌主要是指数期的生长速率降低,同时稳定期的细菌数量减少[35-36]。hfq的突变也并不一定都会影响细菌的生长,例如,在单核细胞增多性李斯特氏菌(Listeria monocytogenes)和金黄色葡萄球菌(Staphylococcus aureus)中,hfq突变后并没有观察到明显的生长缺陷[37]。
Hfq影响细菌的运动能力。细菌拥有快速适应环境变化的能力,游动性和趋化性对其生存发展以及正常的生理功能不可或缺。Hfq对运动能力的影响在许多细菌中都有报道,例如在S. typhimurium、铜绿假单胞菌(Pseudomonas aeruginosa)和水生拉恩氏菌(Rahnella aquatilis)中,hfq的突变会对游动性造成不同程度的损伤[37-38]。此外,在奇异变形菌(Proteus mirabilis)中发现hfq突变体游动性下降,鞭毛减少,鞭毛蛋白表达量下降,不容易形成生物膜[39]。研究表明,hfq主要通过影响细菌鞭毛和趋化基因的表达从而影响细菌的运动能力。然而,在阪崎肠杆菌(Cronobacter sakazakii)中则发现hfq缺失后细菌的游动性反而增强,鞭毛数量增加[40]。
Hfq调控基因的整体表达。Hfq作为关键的转录后调控因子,不仅能够直接调控某个基因的表达,而且能够通过调控转录子的表达来广泛调控一些基因的表达。高通量焦磷酸测序[high-throughput pyrosequencing (HTPS) technology]结果表明,在Salmonella中至少有18%的基因受到Hfq直接或者间接的调控[41]。然而,有研究表明,在芽孢杆菌(Bacillus)的hfq突变体中,转录组并没有受到全局性的影响,表型与E. coli和Salmonella的突变体有所不同[42]。蛋白组学研究结果表明,Hfq影响了细菌各类蛋白的表达,包括细菌的生长、定殖、鞭毛合成、运动、群体感应、物质转运和代谢、环境适应能力以及致病菌的毒力等多种功能蛋白[41]。
此外,Hfq在细菌生物膜形成、毒力基因表达以及对环境胁迫的敏感性方面也发挥着关键性的作用[43]。
3 非编码RNA与Hfq的相互作用 近年来的研究表明,Hfq在sRNA介导的转录后调控过程中处于核心地位(图 2),虽然在sRNA的调控过程中是否需要Hfq的辅助取决于多重因素,但Hfq的存在对大多数细菌sRNA与靶标mRNA的配对起到了促进作用。目前,关于Hfq参与sRNA调控机制的研究很多,但Hfq的作用机制仍不清楚。
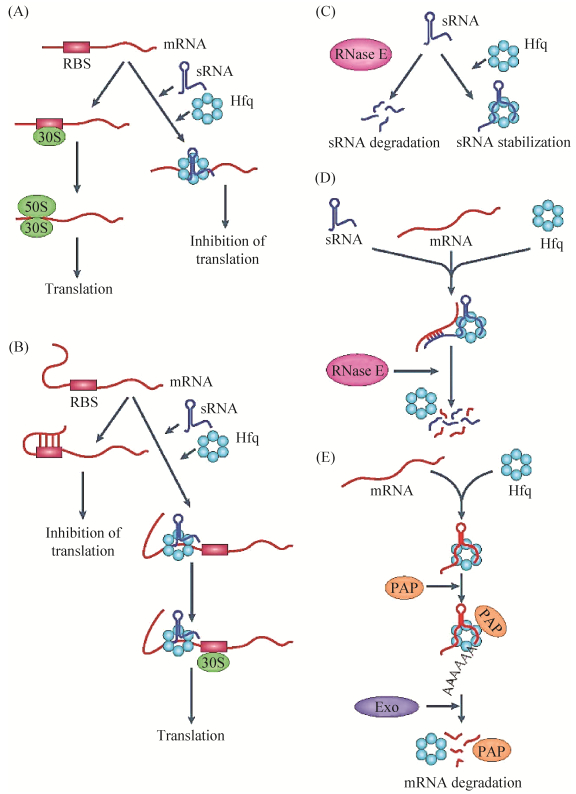 |
| 图 2 Hfq参与转录后调控的模式图[26] Figure 2 Widely accepted mode of Hfq activity[26]. 30S and 50S: Two of ribosomal subunits; RBS: Ribosome-binding site; RNase E: Ribonuclease E; PAP: Poly(A) polymerase; Exo: Exoribonuclease. |
| 图选项 |
3.1 非编码RNA的调控需要Hfq辅助 Hfq是一种RNA分子伴侣蛋白,在一些细菌中能够促进sRNA和靶标mRNA的配对。研究表明在接近一半的细菌基因组中存在Hfq蛋白,它也是E. coli中最丰富的蛋白之一,大约每个细胞中就有30000–60000个,它参与调节很多mRNA的稳定性、翻译和多聚腺苷酸化,也参与RNA的加工过程,缺失Hfq基因后会造成生长缺陷、对环境压力抵抗能力下降和毒性的改变[35, 44]。
序列分析表明,在E. coli和Salmonella中超过30%的sRNA与Hfq结合在一起,Hfq蛋白参与细菌的多种生命活动[32]。在S. aureus中,虽然存在hfq基因,但是在生长过程中的表达量很低,研究表明Hfq对于S. aureus的sRNA调控基因表达来说不是必要的[45]。但在弧菌属(Vibrio)和李斯特菌属(Listeria)中,Hfq对于一些sRNA发挥调控作用是必需的。目前认为,反式编码的sRNA与mRNA作用需要Hfq辅助,而顺式编码的sRNA则不需要。还有研究表明,在sRNA的调控过程中是否需要Hfq与以下几点相关:一是细菌基因组的GC含量,一般认为GC含量要高于50%;二是sRNA和mRNA配对的自由能,配对长度与配对自由能差值△G相关,通过生物信息学分析sRNA-mRNA配对的自由能,得出的规律是较低△G值的需要Hfq,较高△G值的不需要Hfq;三是基因组的大小,细菌中不存在Hfq的基因组相对较小;四是Hfq蛋白的结构特征变化,Hfq蛋白有一个核心的保守区域,而羧基端变化很大[46]。
总之,Hfq蛋白对于大多数sRNA介导的调控过程来说是必需的,它通过调节sRNA与靶标mRNA的互作来调节mRNA的稳定性。
3.2 Hfq参与非编码RNA调控过程的途径 目前的研究表明,Hfq发挥功能主要通过促进sRNA和mRNA的碱基配对,从而影响翻译水平和RNA的稳定性。虽然有很多关于Hfq参与sRNA调控机制的研究,但Hfq的作用机制仍不清楚。一种推测是,Hfq与sRNA结合后,改变了sRNA原来的结构,暴露出能与靶标配对的序列,或者是维持sRNA结构稳定,使得互补序列更容易配对,提高了配对效率。例如,在结合Hfq之后,OxyS的结构发生了变化[22]。另一种推测是,Hfq与sRNA和靶标mRNA同时结合,起到聚集的作用,增加了sRNA的局部浓度,使得sRNA与靶标的配对更加容易,Hfq起到一种“桥梁”的作用。
在转录水平和RNA稳定性上,Hfq介导的调节途径主要包括以下5种情况[26]:第一种情况,Hfq与sRNA结合后,使得靶标mRNA的核糖体结合位点(RBS)被隔绝,阻碍30S和50S核糖体亚基的结合,翻译不能够起始,从而起到抑制翻译的作用,这类sRNA有MicF、OxyS、Spot42、SgrS、RyhB等。而在Hfq不存在的时候,30S和50S核糖体亚基能顺利结合到mRNA上,翻译起始。第二种情况,某些mRNA在其5′-端非翻译区存在二级结构,可以掩饰住核糖体结合位点,抑制翻译[47-48]。当Hfq和特定的sRNA结合后,形成的复合物可以打开靶标mRNA核糖体结合位点的二级结构,从而使30S核糖体亚基可以结合到mRNA上,激活翻译,这种情况的sRNA有DsrA、RprA等,它们通过打开二级结构帮助靶标mRNA起始翻译[49-50]。第三种情况,Hfq能够保护一些sRNA不被核糖核酸酶降解,在多数情况下是核糖核酸酶E (RNase E),即Hfq不存在时,RNase E降解这些sRNA,Hfq存在时,Hfq与sRNA形成复合物,不能被RNase E降解。第四种情况,Hfq可以诱导一些sRNA和它们的靶标mRNA同时被RNase E降解[23, 51]。第五种情况,Hfq可以激活某个mRNA被poly(A)聚合酶(PAP)多聚腺苷酸化,修饰之后触发mRNA从3′-端到5′-端被核糖核酸外切酶(Exo)降解,也就是说Hfq促进某些mRNA被核糖核酸外切酶降解。
3.3 Hfq与非编码RNA的应用 sRNA与Hfq在生物技术上的应用也受到了广泛的关注。目前,基因工程的方法难以完成乳酸菌(Oenocuccus oeni)和产丁醇菌(Clostridium acetobutylicum)的基因改造,而sRNA却成功地解决了这一难题,因此,sRNA被认为是合成生物学中重要的一种基因编辑工具[52]。Hfq在病原菌的致病性、毒力等方面扮演了重要的角色,缺失hfq会使生物膜形成能力和细胞运动能力降低,同时对多种抗生素的耐药性也受到严重的影响,因此,Hfq为抗菌药物的开发提供了新的思路[53]。在未来的研究中,还需要进一步挖掘更多的sRNA,深入研究Hfq和sRNA的作用机制,为sRNA与Hfq在合成生物学、致病菌防治以及其他生物技术上的应用提供重要的理论指导。
4 小结和展望 本文从非编码RNA的分类、调控特点以及伴侣蛋白Hfq的结构、功能以及两者的相互作用机制3个方面综述了非编码RNA和Hfq在转录后调控中的重要作用。近年来,虽然sRNA与Hfq在细菌转录后调控中扮演的角色日益引起了人们的关注,但仍然有很多问题有待解决,未来需要对以下几个方面做进一步研究。
(1) sRNA主要通过与靶标mRNA配对来调控基因表达,而Hfq发挥功能主要是作为分子伴侣促进sRNA和mRNA的碱基配对。那么,sRNA是如何在几秒钟或几分钟内从数以千计的mRNAs中找到它们特定的靶标mRNA的?Hfq与sRNA的作用机制是什么,此过程是否具有特异性?有哪些因素影响Hfq与sRNA的结合?这些问题都还有待进一步研究。
(2) 由于Hfq参与细菌生理活动的多样性,使得该蛋白具有很高的研究价值。但由于Hfq调控方式的复杂性也增加了其功能及机制研究的难度。Hfq存在一个核心的保守区,但在不同的细菌中其C末端的长度和电荷分配会有所变化,这种差异是否具有生物学意义?如果有,这种差异又是如何影响Hfq的功能的?在一些细菌中有单个或多个hfq基因,而有些细菌则没有,是否存在Hfq的非同源替代蛋白?
(3) Hfq作为全局性的转录后调控因子,参与了细菌多种代谢的调控,这些代谢途径之间是否具有关联性?如果有,如何构建Hfq与sRNA的代谢调控网络?目前,关于Hfq和sRNA的研究主要集中在转录组上,还缺乏在蛋白组和代谢组上的研究。同时,对于Hfq在细菌生理和代谢中的调控网路还缺乏较深入的研究。
(4) Hfq因其调控方式的复杂性,对其作用机制还未完全揭示清晰,使其应用的研究相对滞后。如何利用Hfq与sRNA互作研究细菌功能基因组、遗传改良、基因编辑等应用,将成为后续Hfq与sRNA互作研究的热点。
References
| [1] | Papenfort K, Vogel J. Regulatory RNA in bacterial pathogens. Cell Host and Microbe, 2010, 8(1): 116-127. DOI:10.1016/j.chom.2010.06.008 |
| [2] | Jeong Y, Kim JN, Kim MW, Bucca G, Cho S, Yoon YJ, Kim BJ, Roe JH, Kim SC, Smith CP, Cho BK. The dynamic transcriptional and translational landscape of the model antibiotic producer Streptomyces coelicolor A3(2). Nature Communications, 2016, 7: 11605. DOI:10.1038/ncomms11605 |
| [3] | Assche EV, van Puyvelde S, Vanderleyden J, Steenackers HP. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology, 2015, 6: 141. |
| [4] | Leskinen K, Pajunen MI, Varjosalo M, Fernández-Carrasco H, Bengoechea JA, Skurnik M. Several Hfq-dependent alterations in physiology of Yersinia enterocolitica O:3 are mediated by derepression of the transcriptional regulator RovM. Molecular Microbiology, 2017, 103(6): 1065-1091. DOI:10.1111/mmi.2017.103.issue-6 |
| [5] | Storz G, Vogel J, Wassarman KM. Regulation by small RNAs in Bacteria:Expanding frontiers. Molecular Cell, 2011, 43(6): 880-891. DOI:10.1016/j.molcel.2011.08.022 |
| [6] | Michaux C, Verneuil N, Hartke A, Giard JC. Physiological roles of small RNA molecules. Microbiology, 2014, 160(6): 1007-1019. |
| [7] | Frank DN, Pace NR. Ribonuclease P:Unity and diversity in a tRNA processing ribozyme. Annual Review of Biochemistry, 1998, 67(1): 153-180. DOI:10.1146/annurev.biochem.67.1.153 |
| [8] | Keiler KC, Waller PRH, Sauer RT. Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA. Science, 1996, 271(5251): 990-993. DOI:10.1126/science.271.5251.990 |
| [9] | Herskovits AA, Bochkareva ES, Bibi E. New prospects in studying the bacterial signal recognition particle pathway. Molecular Microbiology, 2000, 38(5): 927-939. |
| [10] | Wassarman KM, Storz G. 6S RNA regulates E. coli RNA polymerase activity. Cell, 2000, 101(6): 61-623. |
| [11] | Weilbacher T, Suzuki K, Dubey AK, Wang X, Gudapaty S, Morozov I, Baker CS, Georgellis D, Babitzke P, Romeo T. A novel sRNA component of the carbon storage regulatory system of Escherichia coli. Molecular Microbiology, 2003, 48(3): 657-670. DOI:10.1046/j.1365-2958.2003.03459.x |
| [12] | Parker A, Cureoglu S, de Lay N, Majdalani N, Gottesman S. Alternative pathways for Escherichia coli biofilm formation revealed by sRNA overproduction. Molecular Microbiology, 2017, 105(2): 309-325. DOI:10.1111/mmi.2017.105.issue-2 |
| [13] | Waters LS, Storz G. Regulatory RNAs in bacteria. Cell, 2009, 136(4): 615-628. DOI:10.1016/j.cell.2009.01.043 |
| [14] | Beisel CL, Storz G. Base pairing small RNAs and their roles in global regulatory networks. FEMS Microbiology Reviews, 2010, 34(5): 866-882. DOI:10.1111/j.1574-6976.2010.00241.x |
| [15] | Legewie S, Dienst D, Wilde A, Herzel H, Axmann IM. Small RNAs establish delays and temporal thresholds in gene expression. Biophysical Journal, 2008, 95(7): 3232-3238. DOI:10.1529/biophysj.108.133819 |
| [16] | Levine E, Zhang Z, Kuhlman T, Hwa T. Quantitative characteristics of gene regulation by small RNA. PLoS Biology, 2007, 5(9): e229. DOI:10.1371/journal.pbio.0050229 |
| [17] | Mehta P, Goyal S, Wingreen NS. A quantitative comparison of sRNA-based and protein-based gene regulation. Molecular Systems Biology, 2008, 4: 221. |
| [18] | Shimoni Y, Friedlander G, Hetzroni G, Niv G, Altuvia S, Biham O, Margalit H. Regulation of gene expression by small non-coding RNAs:a quantitative view. Molecular Systems Biology, 2007, 3: 138. |
| [19] | Mitarai N, Andersson AMC, Krishna S, Semsey S, Sneppen K. Efficient degradation and expression prioritization with small RNAs. Physical Biology, 2007, 4(3): 164-171. DOI:10.1088/1478-3975/4/3/003 |
| [20] | Lease RA, Belfort M. A trans-acting RNA as a control switch in Escherichia coli:DsrA modulates function by forming alternative structures. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(18): 9919-9924. DOI:10.1073/pnas.170281497 |
| [21] | Battesti A, Majdalani N, Gottesman S. The RpoS-mediated general stress response in Escherichia coli. Annual Review of Microbiology, 2011, 65(1): 189-213. DOI:10.1146/annurev-micro-090110-102946 |
| [22] | Zhang AX, Wassarman KM, Ortega J, Steven AC, Storz G. The Sm-like Hfq protein increases OxyS RNA interaction with target mRNAs. Molecular Cell, 2002, 9(1): 11-22. DOI:10.1016/S1097-2765(01)00437-3 |
| [23] | Massé E, Escorcia FE, Gottesman S. Coupled degradation of a small regulatory RNA and its mRNA targets in Escherichia coli. Genes & Development, 2003, 17(19): 2374-2383. |
| [24] | Sun XG, Zhulin I, Wartell RM. Predicted structure and phyletic distribution of the RNA-binding protein Hfq. Nucleic Acids Research, 2002, 30(17): 3662-3671. DOI:10.1093/nar/gkf508 |
| [25] | Schumacher MA, Pearson RF, M?ller T, Valentin-Hansen P, Brennan RG. Structures of the pleiotropic translational regulator Hfq and an Hfq-RNA complex:a bacterial Sm-like protein. The EMBO Journal, 2002, 21(13): 3546-3556. DOI:10.1093/emboj/cdf322 |
| [26] | Vogel J, Luisi BF. Hfq and its constellation of RNA. Nature Reviews Microbiology, 2011, 9(8): 578-589. DOI:10.1038/nrmicro2615 |
| [27] | Updegrove TB, Zhang AX, Storz G. Hfq:the flexible RNA matchmaker. Current Opinion in Microbiology, 2016, 30: 133-138. DOI:10.1016/j.mib.2016.02.003 |
| [28] | Link TM, Valentin-Hansen P, Brennan RG. Structure of Escherichia coli Hfq bound to polyriboadenylate RNA. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(46): 19292-19297. DOI:10.1073/pnas.0908744106 |
| [29] | De Lay N, Schu DJ, Gottesman S. Bacterial small RNA-based negative regulation:Hfq and its accomplices. Journal of Biological Chemistry, 2013, 288(12): 7996-8003. DOI:10.1074/jbc.R112.441386 |
| [30] | Wang WW, Wang LJ, Wu JH, Gong QG, Shi YY. Hfq-bridged ternary complex is important for translation activation of rpoS by DsrA. Nucleic Acids Research, 2013, 41(11): 5938-5948. DOI:10.1093/nar/gkt276 |
| [31] | Sauer E, Schmidt S, Weichenrieder O. Small RNA binding to the lateral surface of Hfq hexamers and structural rearrangements upon mRNA target recognition. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(24): 9396-9401. DOI:10.1073/pnas.1202521109 |
| [32] | Zhang AX, Schu DJ, Tjaden BC, Storz G, Gottesman S. Mutations in interaction surfaces differentially impact E. coli Hfq association with small RNAs and their mRNA targets. Journal of Molecular Biology, 2013, 425(19): 3678-3697. DOI:10.1016/j.jmb.2013.01.006 |
| [33] | Arluison V, Folichon M, Marco S, Derreumaux P, Pellegrini O, Seguin J, Hajnsdorf E, Regnier P. The C-terminal domain of Escherichia coli Hfq increases the stability of the hexamer. European Journal of Biochemistry, 2004, 271(7): 1258-1265. DOI:10.1111/ejb.2004.271.issue-7 |
| [34] | Santiago-Frangos A, Kavita K, Schu DJ, Gottesman S, Woodson SA. C-terminal domain of the RNA chaperone Hfq drives sRNA competition and release of target RNA. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(41): 6089-6096. DOI:10.1073/pnas.1613053113 |
| [35] | Tsui HCT, Leung HCE, Winkler ME. Characterization of broadly pleiotropic phenotypes caused by an hfq insertion mutation in Escherichia coli K-12. Molecular Microbiology, 1994, 13(1): 35-49. DOI:10.1111/mmi.1994.13.issue-1 |
| [36] | Brennan CM, Keane ML, Hunt TM, Goulet MT, Mazzucca NQ, Sexton Z, Mezoian T, Douglas KE, Osborn JM, Pellock BJ. Shewanella oneidensis Hfq promotes exponential phase growth, stationary phase culture density, and cell survival. BMC Microbiology, 2013, 13: 33. DOI:10.1186/1471-2180-13-33 |
| [37] | Chao YJ, Vogel J. The role of Hfq in bacterial pathogens. Current Opinion in Microbiology, 2010, 13(1): 24-33. DOI:10.1016/j.mib.2010.01.001 |
| [38] | Song YZ. Preliminary study on the function of hfq gene in Rahnella aquatilis HX2. Master's Thesis of China Agricultural University, 2016(in Chinese). 宋艳钊. 水生拉恩氏菌Rahnella aquatilis HX2中hfq基因生物学功能的初步研究. 中国农业大学硕士学位论文, 2016. |
| [39] | Wang MC, Chien HF, Tsai YL, Liu MC, Liaw SJ. The RNA chaperone Hfq is involved in stress tolerance and virulence in uropathogenic Proteus mirabilis. PLoS One, 2014, 9(1): e85626. DOI:10.1371/journal.pone.0085626 |
| [40] | Kim S, Hwang H, Kim KP, Yoon H, Kang DH, Ryu S. Hfq plays important roles in virulence and stress adaptation in Cronobacter sakazakii ATCC 29544. Infection and Immunity, 2015, 83(5): 2089-2098. DOI:10.1128/IAI.03161-14 |
| [41] | Sittka A, Lucchini S, Papenfort K, Sharma CM, Rolle K, Binnewies TT, Hinton JCD, Vogel J. Deep sequencing analysis of small noncoding RNA and mRNA targets of the global post-transcriptional regulator, Hfq. PLoS Genetics, 4(8): 1000163. |
| [42] | H?mmerle H, Amman F, Ve?erek B, Stülke J, Hofacker I, Bl?si U. Impact of Hfq on the Bacillus subtilis transcriptome. PLoS One, 2014, 9(6): e98661. DOI:10.1371/journal.pone.0098661 |
| [43] | Bak G, Lee J, Suk S, Kim D, Young Lee J, Kim KS, Choi BS, Lee Y. Identification of novel sRNAs involved in biofilm formation, motility, and fimbriae formation in Escherichia coli. Scientific Reports, 2015, 5: 15287. DOI:10.1038/srep15287 |
| [44] | Kajitani M, Kato A, Wada A, Inokuchi Y, Ishihama A. Regulation of the Escherichia coli hfq gene encoding the host factor for phage Q beta. Journal of Bacteriology, 1994, 176(2): 531-534. DOI:10.1128/jb.176.2.531-534.1994 |
| [45] | Jousselin A, Metzinger L, Felden B. On the facultative requirement of the bacterial RNA chaperone, Hfq. Trends in Microbiology, 2009, 17(9): 399-405. DOI:10.1016/j.tim.2009.06.003 |
| [46] | Pandey SP, Minesinger BK, Kumar J, Walker GC. A highly conserved protein of unknown function in Sinorhizobium meliloti affects sRNA regulation similar to Hfq. Nucleic Acids Research, 2011, 39(11): 4691-4708. DOI:10.1093/nar/gkr060 |
| [47] | Kozak M. Regulation of translation via mRNA structure in prokaryotes and eukaryotes. Gene, 2005, 361: 13-37. DOI:10.1016/j.gene.2005.06.037 |
| [48] | Ellis MJ, Trussler RS, Haniford DB. Hfq binds directly to the ribosome-binding site of IS10 transposase mRNA to inhibit translation. Molecular Microbiology, 2015, 96(3): 633-650. DOI:10.1111/mmi.2015.96.issue-3 |
| [49] | Fr?hlich KS, Vogel J. Activation of gene expression by small RNA. Current Opinion in Microbiology, 2009, 12(6): 674-682. DOI:10.1016/j.mib.2009.09.009 |
| [50] | Soper T, Mandin P, Majdalani N, Gottesman S, Woodson SA. Positive regulation by small RNAs and the role of Hfq. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(21): 9602-9607. DOI:10.1073/pnas.1004435107 |
| [51] | Morita T, Maki K, Aiba H. RNase E-based ribonucleoprotein complexes:mechanical basis of mRNA destabilization mediated by bacterial noncoding RNAs. Genes & Development, 2005, 19(18): 2176-2186. |
| [52] | Thorsing M, dos Santos PT, Kallipoliti BH. Small RNAs in major foodborne pathogens:from novel regulatory activities to future applications. Current Opinion in Biotechnology, 2018, 49: 120-128. DOI:10.1016/j.copbio.2017.08.006 |
| [53] | Kakoschke TK, Kakoschke SC, Zeuzem C, Bouabe H, Adler K, Heesemann J, Rossier O. The RNA chaperone Hfq is essential for virulence and modulates the expression of four adhesins in Yersinia enterocolitica. Scientific Reports, 2016, 6: 29275. DOI:10.1038/srep29275 |
