江承亮1, 李鸿毅1, 王行1, 腾昌运1,2, 冯诗韵1,3, 楼莉萍1, 张志剑1,3


1.浙江大学环境与资源学院, 浙江 杭州 310058;
2.杭州吾蚨美生物科技有限公司, 浙江 杭州 311113;
3.杭州谷胜农业科技有限公司, 浙江 杭州 311108
收稿日期:2017-06-28;修回日期:2017-09-18;网络出版日期:2017-12-04
基金项目:国家水体污染控制与治理科技重大专项(2012ZX07101);浙江省重点研发计划(2015C03009)
作者简介:张志剑, 男, 1973年生。2002年获得浙江大学环境工程博士学位, 现同时供职于浙江大学, 副教授、博士生导师, 担任浙大环境健康研究所副所长、浙江大学-中国西部发展研究院水生态与流域建设研究中心主任, 浙江省"钱江人才"计划获得者(2006)、浙江省优秀科技工作者(省委组织部等, 2013), 中国昆虫学会-昆虫产业专业委员会委员、浙江省生态学会副秘书长。2012年4-8月, 赴美国依利诺伊大学(UIUC)开展"污染治理与流域生态建设"的联合研究; 2011年8月-2012年4月, 赴美国哥伦比亚大学(纽约市)开展"环境微生物学"的联合研究; 2002年3月-2004年8月, 赴美国明尼苏达大学从事"农牧废弃物处理与利用"的博士后研究。长期致力于有机废弃物处理与再利用、区域污染减排与增值型循环经济构建等科研与教学, 获得专利12项、发表论文50余篇, 获得省部科技进步一等奖2项, 央视CCTV-10"走进科学"专题报道1次
*通信作者:张志剑, E-mail:zhangzhijian@zju.edu.cn
摘要:[目的]过度使用抗生素作为动物饲料添加剂,导致畜禽粪便已成为抗生素抗性基因的主要蓄积库,为了研究蝇蛆(Musca domestica)对猪粪中残留抗生素及抗性基因的影响,本文动态采集了实际农场条件下蝇蛆转化过程中猪粪堆体及虫体样本。[方法]利用qPCR、液相色谱-电喷雾质谱、同位素内标法、Illumina高通量测序以及局部相似性研究蝇蛆生物转化过程中残留抗生素降解效能及相关抗性基因组变化的微生物生态机制。[结果]6 d周期内,猪粪中四环素、土霉素、金霉素、强力霉素、磺胺嘧啶、诺氟沙星、氧氟沙星、环丙沙星以及恩诺沙星等9种残留抗生素含量显著下降,累积减量为34.3%–58.1%,每日减量百分比介于7.8%–57.4%之间。猪粪中共检测到的158种抗性基因,其中有118种大幅衰减,衰减量平均达79.3%;23种抗性基因存在富集现象,富集倍数平均为3.48。在蝇蛆肠道的作用下,粪源微生物群落中Bacteroidetes相对丰度下降,Proteobacteria相对丰度增加,尤其是Ignatzschineria增幅最大。网络分析发现,抗性基因的增减与微生物群落的变化显著相关,与抗性基因衰减相关的微生物主要属于Clostridiales和Bacteroidales,而与抗性基因富集相关的微生物主要为Alcaligenaceae、[Weeksellaceae]及Bacillales。[结论]蝇蛆可有效削减猪粪中的残留抗生素及防控抗性基因向环境扩散。
关键词: 猪粪 蝇蛆 抗性基因 肠道菌群
Housefly larvae gut microbiome may attenuate veterinary antibiotics and alter the resistome in swine manure
Chengliang Jiang1, Hongyi Li1, Hang Wang1, Changyun Teng1,2, Shiyun Feng1,3, Liping Lou1, Zhijian Zhang1,3


1.College of Environment and Source Sciences, Zhejiang University, Hangzhou 310058, Zhejiang Province, China;
2.Hangzhou Wufumei Biotechnology co., LTD, Hangzhou 311113, Zhejiang Province, China;
3.Hangzhou Gusheng Agricultural Science and Technology co., LTD, Hangzhou 311108, Zhejiang Province, China
Received 28 June 2017; Revised 18 September 2017; Published online 4 December 2017
*Corresponding author: Zhijian Zhang, E-mail:zhangzhijian@zju.edu.cn
Supported by the National Water Pollution Control and Treatment Science and Technology Major Project (2012ZX07101) and by Primary Research & Developement Plan of Zhejiang Province (2015C03009)
Abstract: [Objective]The overuse of antibiotics as veterinary feed additives is contributing to a significant reservoir of antibiotic resistance in livestock manure. A housefly larvae (Musca domestica) vermireactor has been increasingly adopted for swine manure value-added bioconversion and pollution control. In order to investigate its efficiency on attenuations of antibiotic and the associated resistome during manure vermicomposting, we implemented dynamic sampling during a typical cycle (6 days).[Methods]High-capacity qPCR, liquid chromatography-electrospray ionization tandem mass spectrometry, the isotope-labeled internal standard method and Illumina Hiseq sequencer were used to explore the effectiveness of antibiotic degradation and microbial mechanisms on dynamics of the associated resistome of full-scale larvae manure vermicomposting.[Results]Nine antibiotics (tetracycline, oxytetracycline, chlortetracycline, doxycycline, sulfadiazine, norfloxacin, ofloxacin, ciprofloxacin and enrofloxacin) were dramatically removed. The cumulative removal of these tested antibiotics reached 34.3%-58.1%, along with the daily reduction percentages ranging from 7.8% to 57.4%. In total, 113 of 158 antibiotic resistance genes (ARGs) were significantly attenuated (by 79.3%), while 23 were significantly enriched (3.48-fold) following vermicomposting. The manure-borne bacterial community showed a decrease in the relative abundance of Bacteroidetes, and an increase in Proteobacteria, specifically Ignatzschineria, following gut transit, while ARG attenuation was significantly correlated with changes in microbial community structure. Bacteria correlated with attenuated ARGs mainly belonged to order Clostridiales and Bacteroidales. Enriched ARGs, were mostly correlated with family Alcaligenaceae, [Weeksellaceae] and order Bacillales.[Conclusion]Housefly larvae vermicomposting is an efficient way to attenuate antibiotics and ARGs in swine manure within the 6-day treatment.
Key words: animal manure larvae vericomposting resistome gut microbiome
中国是世界上最大的抗生素生产国和消费国,有超过46%的抗生素[1]作为疾病的预防治疗剂或者促生长剂被用于畜牧养殖[2],其中四环素类、磺胺类与喹诺酮类是目前应用最广泛的三类广谱抗生素[3-4]。兽用抗生素进入动物体内后,只有少量被吸收和降解,30%–90%的抗生素以母体化合物的形式[5]连同其在体内诱导出的抗性基因[6] (antibiotic resistance genes,ARGs)随粪便一并进入各类受纳环境;加之抗生素的滥用和过度使用,加重了猪粪中微生物的选择压力以及抗性基因的转移速度[7],导致抗性基因和耐药菌的大量富集,畜禽粪便已成为抗生素抗性基因重要的蓄积库[8]。
相比传统堆肥,蝇蛆共生微生物的复杂性促使其具备更高效的生物降解能力[9]。近年来,科学家利用蝇蛆(Musca domestica)设计废弃物生物反应器(图 1),研发了高效能的蝇蛆猪粪生物转化技术,并在国内多处实际应用[10]。张志剑等研究发现,蝇蛆在5–7 d内可将新鲜猪粪转化成可施用于土壤的有机肥[11],猪粪减量速率平均达106±17 kg/(m3·d),总体减量超过70%,水分总量减少80%;收获的成熟蝇蛆其蛋白质含量为57%,虫粪堆肥理化性质及微生物特性(如潜在的酶活性,异养呼吸)变化明显[11-12]。
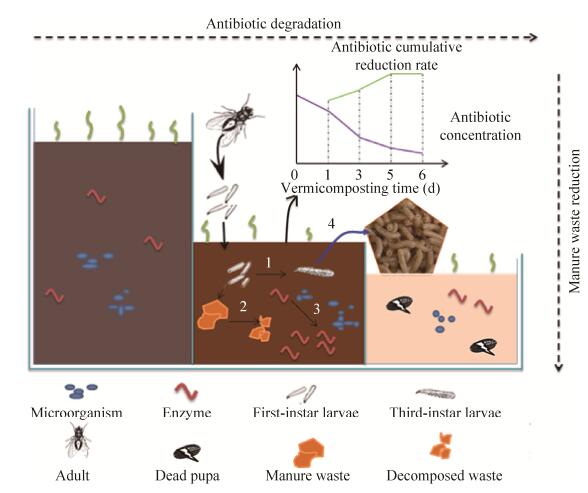 |
| 图 1 5–7日周期内猪粪/污染物(例如抗生素等)-蝇蛆反应器生物降解过程示意图 Figure 1 The diagrammatic sketch on manure/waste biodegradation (i.g., antibiotics in a housefly larvae (Musca domestica) vermireactor within 5–7 days. 1: The metabolism and growth of housefly larvae. 2: The physical reduction process of waste materials. 3: The synergy of manure enzymes and microorganisms. 4: Larvae harvest. |
| 图选项 |
微生物是环境系统中抗性基因的主要宿主[13-14],这意味着微生物群落结构的改变很可能引起环境抗性基因种类及其丰度的相应变化,特别需要注意的是粪源微生物中的抗性基因可赋存与转移到环境中的土著细菌,甚至转归至潜在的人类病原菌[15]。目前,我们对蝇蛆可以在多大程度上影响猪粪中微生物在分类学和功能的分布,这些微生物特征变化又与相关抗性基因存在怎样的联系等方面知之甚少。与此同时,研究粪源细菌与蝇蛆肠道共生细菌潜在的相互作用,进而探讨抗性基因环境持久性及其防控策略,更具有生态学意义。
本文动态监测了6 d周期内蝇蛆生物转化猪粪过程中的残留抗生素、抗性基因和微生物组的动态变化,旨在:(1)评估猪粪残留抗生素降解效能及阐明猪粪中抗性基因组的变化特征;(2)了解蝇蛆肠道如何改变粪源微生物的群落结构;(3)阐明与抗性基因动态变化相关的微生物生态演替机制。
1 材料和方法 1.1 实地农场蝇蛆生物反应器的设计与操作 选取位于杭州市萧山区(30°49'47.02″N,120°39'22.12″E)的杭州天元农业开发有限公司为试验点,该公司的蝇蛆生物转化技术已有5年以上的实际应用,设计最大日处理猪粪容量150 t。生猪粪取自附近养猪场,无需固液分离,经几小时储存后直接用于蝇蛆堆肥。
该农场采用温室大棚辅助蝇蛆生物猪粪全季节运行[11-12]。周期为6 d的蝇蛆生物反应器采用半连续投料模式(图 2),猪粪在蝇蛆接种前按11.0 kg/m2的面密度平铺于水泥槽中(7.2 m×2.5 m×0.2 m),厚度约为5 cm;再均匀接种孵化12 h后的幼蛆,接种密度平均为580000只/m2。接种12 h后的第1日、第2日、第3日、第4日分别按5.56、15.60、15.60、5.56 kg/m2的面密度添加新鲜猪粪,蝇蛆堆体厚度累计增至8–10 cm。
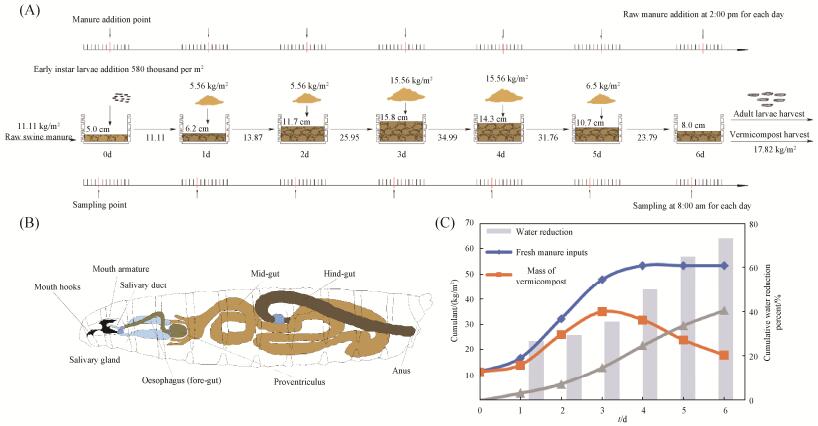 |
| 图 2 蝇蛆堆肥过程中猪粪的投加模式(A)、蝇蛆肠道中转化示意图(B)以及蝇蛆堆肥过程物量平衡(C) Figure 2 The larvae vermicomposting module with swine manure input during larvae gut passage. A: Operation of the semi-continuous feeding larvae vermireactor. B: The anatomy of the gut transit of housefly larvae (Musca domestica). C: The cumulative amount of fresh manure input, remaining residues and cumulative manure reduction mass (green curve). |
| 图选项 |
1.2 猪粪及蝇蛆肠道采样 猪粪样品随机取自未加蝇蛆前的反应器,标记为S0d;如图 2所示,6 d周期内,每日早上8点采集蝇蛆处理组样品,标记为S1d、S2d、S3d、S4d、S5d、S6d;同时收集蝇蛆样品,标记为L1d、L2d、L3d、L4d、L5d、L6d。每日下午2点按2.1设置的面密度投加生猪粪。设置对照组,除不添加蝇蛆外,其他操作均与处理组相同,采集的样品分别标记为C0d、C1d、C3d、C6d。每日取样后快速封存于便携式制冷设备,2 h内送至浙江大学,储存于–20 ℃冰箱,以便后续实验。样品使用前冷冻干燥,研磨后过0.15 mm筛。
1.3 抗生素的提取与分析 本文检测了3类广谱抗生素中的9种,均属于环境中常见的抗生素[3-4],分别为:(1)四环素类(TCs):四环素(tetracycline, TC),土霉素(oxytetracycline, OTC),金霉素(chlortetracycline, CTC),强力霉素(doxycycline, DOC);(2)磺胺类(SAs):磺胺嘧啶(sulfadiazine, SDZ);(3)喹诺酮类(QLs):诺氟沙星(norfloxacin, NFC),氧氟沙星(ofloxacin, OFC),环丙沙星(ciprofloxacin, CFC),恩诺沙星(enrofloxacin, EFC)。
提取与分析:准确称取样品2.00 g于50 mL聚乙烯离心管中,加入20 mL提取剂(磷酸盐缓冲液和乙腈(Mg(NO3)2-NH3·H2O)按体积比3:1混合),黑暗处涡旋混合10 min,超声提取15 min后,置于黑暗处12 h后取出,以5000 r/min离心10 min,转移上清液于新离心管中,残渣再重复提取2次,所得全部提取液过0.7 mm的玻璃微纤维过滤器后加入500 mL的超纯水稀释,使有机溶剂含量少于5%。利用全自动固相萃取仪,按仪器设定程序操作对所得溶液进行净化处理。抗生素的分离与检测采用液相色谱-电喷雾质谱联用技术,用同位素内标法进行定量。所有抗生素的校准范围为5.0–300 μg/L。
猪粪残留抗生素的累积减量百分比与每日减量百分比的计算按照公式(1–4)计算。
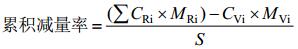 | 公式(1) |
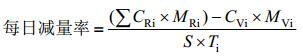 | 公式(2) |
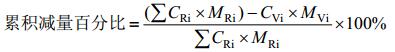 | 公式(3) |
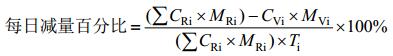 | 公式(4) |
1.4 DNA提取 蝇蛆肠道DNA提取前,先将蝇蛆饥饿处理8 h,达到空腹状态后解剖,获得的中肠及后肠用于DNA提取。猪粪样品DNA提取采用强力土壤DNA提取试剂盒(PowerSoilTM DNA Isolation Kit,MoBio,USA),按照试剂盒说明书步骤进行操作,所得DNA样品均一化至相同浓度后再用于下游利用。
1.5 高通量qPCR测定抗性基因 利用Wafergen智能芯片实时PCR平台测定猪粪DNA样品中的285种抗性基因,每个样品做3个重复,Ct值31作为检出限。当一个基因被多种引物标记时,只算作单个抗性基因。样品的抗性基因相对丰度参考Pfaffl[16]提出的数学模型计算;C0d作为标准样品,用于进一步计算所有其他样品抗性基因的倍数变化。在计算抗性基因倍数变化时,若抗性基因没有扩增,则使用Ct值31。检测到的抗性基因按照抗性机制以可耐抗生素的种类进行划分。
1.6 16S rRNA测序、数据处理及分析 DNA样品利用515F/806R通用引物片段扩增细菌16S rDNA V4区段后,由Illumina Hiseq测序仪完成测序。原始数据上传至DDBJ Sequence Read Archive数据库(Accession number: SRP050167)。先由QIIME软件(v1.8.0)[17]对原始数据进行前处理(过滤和分样后的样品C6d.3与L5d.2所含片段少于100,因此没有用于下游分析);再运用Usearch软件(v6.1)去除嵌合体,每个重复平均剩余54250条高质量片段,平均长度为253 bp;然后,利用Uclust软件(v1.2.22q)将有效序列在97%的相似性水平上进行聚类,得6436个OTUs,再与Greengenes数据库(13_8 release)[18]比对,最后利用RDP Classifier[19]对序列进行分类,获得分类学信息。
进一步分析前,将序列进行均一化处理以适应不同的分析深度。微生物群落结构的α和β多样性通过QIIME软件计算得出;运用SourceTracker追溯微生物的来源[20],过滤掉在小于5个样品中出现的物种。6 d周期内蝇蛆转化的猪粪作为“汇”,蝇蛆肠道以及新鲜猪粪中的微生物为“源”,“未知”表示“汇”中来源不明的“源”。
此外,利用Kruskal-Wallis检验及Dunn’s事后检验对数据进行差异统计分析,并以C0d的数据作为参考底值,检验数据变化的显著性(即P值)。
1.7 细菌类群与抗性基因的局部相似性分析 基于局部相似性分析(Local Similarity Analysis,LSA)[21]考察6日周期内OTUs或已注释细菌类群的相对丰度与猪粪中抗性基因的关系,相对的局部相似值和P值分别设为 > 0和 < 0.01。获取基础网络参数来构建原始结构关系,为减少网络结构的复杂度,进一步去除相对丰度小于0.1%的物种。网络的可视化通过Cytoscape 3.3实现。
2 结果和分析 2.1 蝇蛆转化猪粪过程中残留抗生素的动态变化 蝇蛆堆肥可显著影响猪粪中抗生素的浓度。在蝇蛆转化过程中四环素类和磺胺类抗生素持续减少,其中的土霉素、金霉素、磺胺嘧啶的累积减量率分别为23.8 mg/m2、8.34 mg/m2和4.09 mg/m2 (表 1),累积减量分别占投加生猪粪中相应抗生素总量的68.6%、69.8%和72.3% (图 3)。由于在蝇蛆成熟前(第6日)投加了生猪粪,每日减量率出现部分下降。周期内喹诺酮类抗生素中的氧氟沙星和恩诺沙星减量并没有持续,但氧氟沙星、环丙沙星累积减量率持续上升,至第6日分别为1.17 mg/m2和32.9 mg/m2,累积减量达34.3%–58.1%。本实验检测的9种抗生素接种蝇蛆后均快速减少,每日减量百分比在7.8%–57.4%之间,但减量速度随时间推移逐渐下降。
表 1. 蝇蛆处理猪粪的第1 (S1d)、3(S3d)、5 (S5d)、6(S6d)天的累积/每日抗生素减量率(n=3) Table 1. Cumulative and daily reduction rates of antibiotics after 1 (S1d), 3 (S3d), 5 (S5d), and 6 days (S6d) during swine manure vermicomposting using the housefly larvae (n=3)
| Item | TC | OTC | CTC | DOC | SDZ | NFZ | OFC | CFC | EFC |
| Cumulative reduction rate/(mg/m2) | |||||||||
| S1d | 0.186±0.005c | 5.23±0.34c | 3.67±0.03d | 0.204±0.014d | 0.945±0.30d | 0.476±0.031b | 0.119±0.004d | 10.7±0.5d | 3.20±0.40d |
| S3d | 0.452±0.0027b | 14.5±1.0b | 6.65±0.43c | 0.332±0.016c | 2.74±0.06c | 0.356±0.017c | 0.523±0.017c | 16.8±0.5c | 6.57±2.24c |
| S5d | 0.542±0.046a | 23.7±0.4a | 7.62±0.25b | 0.644±0.010b | 3.82±0.03b | 1.17±0.04a | 1.51±0.050a | 29.9±1.4b | 8.58±0.56a |
| S6d | 0.595±0.037a | 23.8±0.5a | 8.34±0.06a | 0.713±0.011a | 4.09±0.12a | 1.17±0.02a | 0.857±0.036b | 32.9±1.4a | 7.92±0.18b |
| Daily reduction rate/[mg/(m2·d)] | |||||||||
| S1d | 0.194±0.007a | 2.24±0.34a | 3.67±0.00a | 0.216±0.019a | 0.935±0.033a | 0.476±0.031a | 0.123±0.051c | 10.9±0.6a | 3.20±0.40a |
| S3d | 0.145±0.020b | 5.26±0.15a | 2.20±0.18b | 0.104±0.010c | 0.913±0.020a | 0.119±0.007d | 0.174±0.006b | 5.36±0.26b | 2.19±0.08b |
| S5d | 0.108±0.009c | 4.82±0.14a | 1.43±0.67c | 0.144±0.003b | 0.808±0.027b | 0.238±0.011b | 0.298±0.012a | 5.94±0.27b | 1.72±0.11c |
| S6d | 0.099±0.006c | 3.92±0.08b | 1.39±0.10c | 0.112±0.007c | 0.630±0.11c | 0.196±0.003bc | 0.139±0.008c | 5.49±0.22b | 1.33±0.33d |
| n is the number of repetitions. | |||||||||
表选项
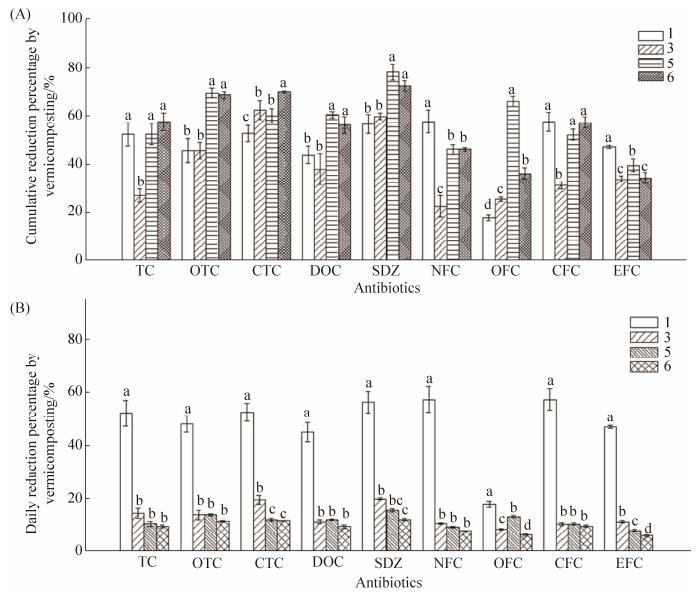 |
| 图 3 蝇蛆处理猪粪的第1、3、5、6天的累积(A)和每日抗生素减量百分比(B) Figure 3 Cumulative (A) and daily (B) antibiotic reduction in vermicompost after 1 (S1d), 3 (S3d), 5 (S5d) and 6 days (S6d) during swine manure larvae vermicomposting. Lowercase letters represent significant levels of each group. |
| 图选项 |
2.2 蝇蛆转化猪粪过程中抗性基因动态变化 利用抗性基因集成芯片技术,本研究样品中共检测到了158种不同的抗性基因(图 4),其耐药机制主要有三类:(1)降解或取代活性基团,致使抗生素失活;(2)利用外排泵将细胞内的抗生素排出,从而降低胞内抗生素浓度;(3)胞外保护,即在受体细胞的细胞膜上形成多糖类屏障阻止抗生素进入[22]。
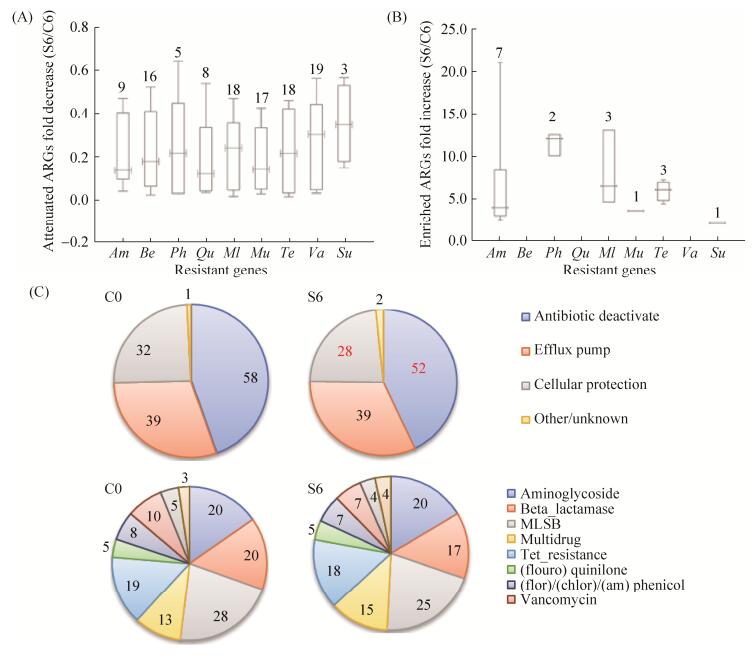 |
| 图 4 抗性基因的衰减(A)、富集(B)及相关耐药机制的分布(C) Figure 4 Attenuation or enrichment of antibiotic resistance genes and the associated mechanism of resistome. A: The descriptive statistics for attenuated resistance genes. B: The mechanism of resistance to the absence/presence of antibiotic resistant genes. C: The antibiotic to which they confer resistance. |
| 图选项 |
猪粪中可对氨基糖苷类、氯霉素类、大环内酯类、四环素类以及磺胺类抗生素产生耐药性的抗性基因均有存在。如图 4所示,相比对照组,处理组中113种抗性基因在6日周期内衰减79.3% (P < 0.05),16种抗性基因富集5.6倍,23种抗性基因富集3.48倍。这些基因主要通过失活以及胞外保护机制对β-内酰胺类、大环内酯类、四环素类以及万古霉素类抗生素产生耐药性。
2.3 蝇蛆肠道重塑堆体的微生物群落组成 如图 5所示,对照组相比,蝇蛆处理组的猪粪细菌物种丰度随转化时间显著下降。所有样品中,蝇蛆肠道菌群的多样性最低,并且第1日至第6日群落结构(β多样性)的变化最小。处理组堆体中的Proteobacteria(变形菌门)显著增加(P < 0.05),Bacteroidetes(拟杆菌门)下降(P < 0.05)。变形菌门为蝇蛆肠道的优势菌群,占所有序列的92%,主要由50%的Ignatzschineria(依格纳季氏菌属)、21%的Providencia(普罗维登斯菌属)以及12%的Pseudomonas (假单胞菌属)组成;但蝇蛆堆体中普罗维登斯菌属并不丰富,主要为30%的依格纳季氏菌属和6.3%的假单胞菌属。蝇蛆处理后堆体中的依格纳季氏菌属和假单胞菌属分别比猪粪丰富28倍和2倍。Bacteroides(拟杆菌属)和Prevotella (普氏菌属)是已知猪肠道的优势菌群,但在处理组堆体中却十分稀少。处理组前后堆体的微生物群落结构差异显著,前期与猪粪最为接近,后期与蝇蛆肠道共生菌群更相似。
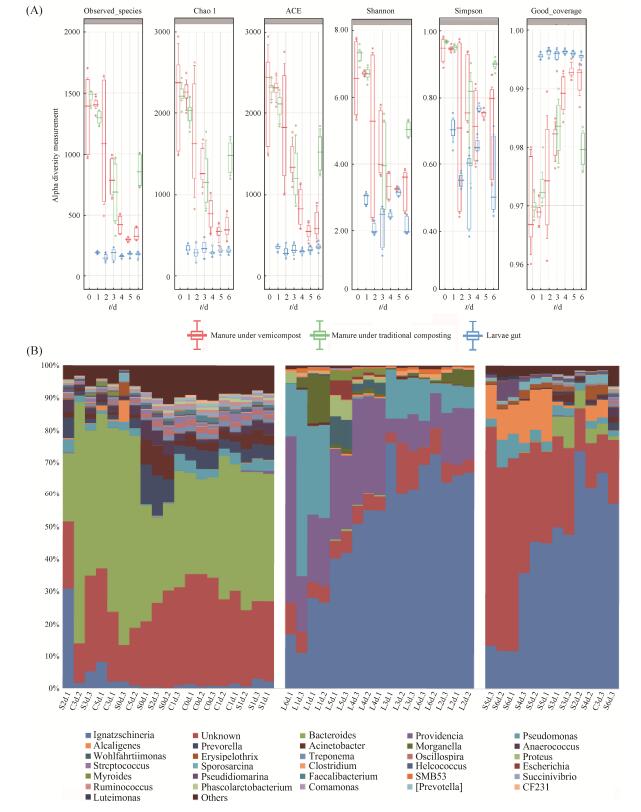 |
| 图 5 微生物多样性(A)及群落结构(B) Figure 5 Microbial diversity (A) and community structure (B) determined among studied samples by 16S rRNA analysis. |
| 图选项 |
2.4 蝇蛆肠道共生微生物组与抗性基因的关系 基于局部相似性分析发现(图 6),某些衰减或富集抗性基因与特定细菌类群的动态变化显著相关;此外,富集抗性基因的网络结构比衰减的更复杂,每个衰减抗性基因平均连接了30.3个节点,而富集抗性基因只有5.4个。
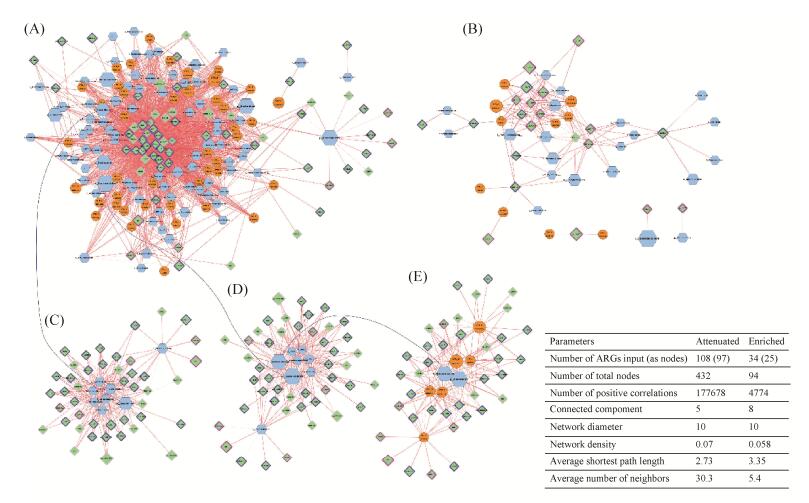 |
| 图 6 衰减、富集抗性基因与多水平菌群的网络分析图 Figure 6 Local similarity analyses showing the potential linkage between dynamics of antibiotic resistance genes (ARGs) and taxa/OTU changing patterns in manure under vermicomposting (day 1 to day 6). A: Subnetwork organized between significantly attenuated ARGs and taxa/OTUs. B: Subnetwork organized between significantly enriched ARGs and taxa/OTUs. C: Subnetwork organized between significantly attenuated ARGs and members belonging to the order Clostridiales. D: Subnetwork organized between significantly attenuated ARGs and family members belonging to the order Bacteroidales. E: Subnetwork organized between significantly attenuated ARGs and OTUs/taxa belonging to the family Bacteroidaceae. |
| 图选项 |
如图 6-A所示,分类等级从门到OTUs的细菌都与抗性基因存在关联,说明抗性基因在生物分类学上分布很广。如ermJ/ermD、vgaA-01、blaGES、vanXB与拟杆菌门相关(P < 0.01),cfr和vatE-01分别与Acinetobacter (不动杆菌属)、Prevotellaceae(普雷沃氏菌科)相关(P < 0.01)。而与衰减抗性基因相联系的细菌主要属于Clostridiales (梭菌目)和拟杆菌目,均属于猪粪的优势菌群。由图 6-C和6-D可知,除拟杆菌目的普雷沃氏菌科以及梭菌目中的Tissierellaceae(泰式菌科)外,这两个目中其他大部分科水平菌群存在相同的抗生素耐药机制,且与普雷沃氏菌科和泰式菌科相关的抗性基因主要赋存于移动遗传元件中。此外,通过图 6-C可知,丰度最大的Bacteroidales菌目与上述情况相同。这表明族系相同的物种之间具有类似的抗生素耐药机制,这也就解释了抗性基因可以在环境中长期存在的原因。如图 6-B所示,大部分衰减的抗性基因不赋存于移动遗传元件内,可能与某些细菌类群呈线性关系。mphA、aadA2、aadA5-2、aadA9-02、tetG-01、cmlA1、tetR-02等富集抗性基因,主要与Alcaligenaceae (产碱菌科)、[Weeksellaceae]和Bacillales (芽孢杆菌目)相关,它们在蝇蛆堆肥过程中丰度明显增加,分别从0.18%、0.38%、0.64%增至13%、26%、4.5%(图 5-B)。相比衰减抗性基因,与富集菌群相关的抗性基因更可能赋存于移动遗传元件内。假单胞菌属与ampC-07、tet-xtet、L-02等富集抗性基因相关,OTU 109756属于Pseudomonadaceae(假单胞菌科),与mphA、aadA2、aadA5-2、aadA9-02和tetG-01正相关。依格纳季氏菌属是猪粪经蝇蛆转化后堆肥中丰度最大的菌群,但它仅与aacC1相联系。
3 讨论 本文发现,在蝇蛆与微生物共同作用下猪粪中残留抗生素浓度显著下降。同样,已有众多研究发现传统堆肥也可去除动物粪便中兽用抗生素。Ramaswamy等实验发现,家禽粪便经38 d堆肥后,盐霉素削减99%[23];鸡粪进行堆肥处理40 d后发现,超过99%的NFCs被去除[24]。Dolliver等研究猪粪堆肥22–35 d后莫能菌素和泰乐菌素的变化,发现这两者减量可达54%–76%[25]。但相比传统堆肥,蝇蛆可在6 d内削减猪粪中70%的抗生素,显著缩短了处理周期,在处理时长和效率等方面具有显著优势。有研究表明,温度、pH等环境因子与抗生素的生物降解及转化存在正相关关系[26-27]。课题组前期研究发现,与猪粪减量关联的化学/生物化学因子(如有机质含量、酶活等)也会影响抗生素的衰减[28]。
蝇蛆肠道微生物组对猪粪中抗性基因组的动态变化起着至关重要的作用,蝇蛆肠道筛选特定的微生物物种(如依格纳季氏菌属、假单胞菌属和产碱杆菌属)定殖成为后期蝇蛆堆体的优势菌群。通过Sourcetracking分析发现,富集菌群来源于肠道微生物组和/或环境,例如,产碱杆菌属仅在后期蝇蛆堆体中检测到;蝇蛆堆体中富集的依格纳季氏菌属也主要来源于肠道微生物组,这说明处理组中的某些细菌类群可被追溯回蝇蛆肠道。同时,蝇蛆堆肥还可以改变生猪粪的物理化学性质[11-12, 28],导致部分不属于猪粪的微生物组细菌富集。这些富集的类群主要属于需氧/兼性厌氧细菌,因此这些细菌可在有氧的蝇蛆堆肥中生存和繁殖[29]。总的来说,蝇蛆处理后蝇蛆堆体的细菌群落结构类似于猪粪与蝇蛆肠道之间的一个过渡状态(图 5)。
通过局部相似性分析可知,相比富集抗性基因,衰减抗性基因与细菌类群的关系更紧密(图 6),这意味着细菌多样性减少的同时,伴随着某些抗性基因的削减,而且这些微生物可能主要属于粪源微生物。Heck等研究发现,生活垃圾和剩余污泥堆肥过程会富集抗生素耐药微生物,在蝇蛆堆体中的假单胞菌属和苍白杆菌内发现了多种耐药机制并存的抗性基因[30]。类似地,Riber等在粪便有机肥改良后的土壤中发现,假单胞菌属可通过水平基因转移长期存在,并获得多种耐药基因[31]。假单胞菌属内的基因盒隐藏着可对氨基糖苷类和四环素产生耐药的抗性基因。此外,可对氯霉素类、大环内酯类抗生素产生耐药性的富集抗性基因与芽孢杆菌目、产碱菌科和[Weeksellaceae]密切相关(图 6)。芽孢杆菌目不仅可以分泌耐热的纤维素酶和多酚氧化酶促进蝇蛆堆体的腐熟,还能在恶劣环境中生存成为新的有效定殖细菌。蝇蛆转化过程中产碱菌科和[Weeksellaceae]快速繁殖,在第6天成为优势菌群。此外,蝇蛆转化过程中发生水平基因转移,使得富集的细菌类群成为抗性基因的受体[32-33]。
动物粪便的减量化与无害化利用是畜牧业粪便管理的主要目标[34]。蝇蛆堆肥可在6 d周期内有效削减猪粪中残留的大量抗生素以及大部分抗性基因,但由于蝇蛆肠道微生物也存在潜在的抗性基因水平转移的风险,这使得蝇蛆处理所得的虫粪有机肥的成分更加复杂,因此在农田施用过程中进行一定的风险评估。
4 结论 蝇蛆在6 d周期内可有效削减猪粪中的残留抗生素以及大部分抗性基因。
(1) 检测的9种抗生素的累积减量可达34.3%–58.1%,每日减量百分比介于7.8%–57.4%。
(2) 相比对照组,猪粪经蝇蛆转化后113种抗性基因衰减79.3%,16种抗性基因富集了5.6倍,23种抗性基因增加了3.48倍。这些抗性基因主要通过编码失活以及胞外保护机制对β-内酰胺类、大环内酯类、四环素类以及万古霉素类抗生素产生抗性。
(3) 蝇蛆堆体中变形菌门明显增加,而拟杆菌门大幅减少(P < 0.05),而变形菌门是蝇蛆肠道的优势菌群(主要由50%的依格纳季氏菌属、21%的普罗维登斯菌属以及12%的假单胞菌属组成),但堆体中的普罗维登斯菌属并不丰富,主要为30%的依格纳季氏菌属及6.3%的假单胞菌属。
(4) 相比富集抗性基因,衰减抗性基因与细菌类群的网络结构更复杂,每个衰减抗性基因平均与30.3个节点相关,而富集的只有5.4个。与衰减抗性基因相关的细菌主要属于梭菌目菌目和拟杆菌目,与富集抗性基因相关的属水平细菌主要属于产碱菌科、Weeksellaceae和芽孢杆菌目。
References
| [1] | Hvistendahl M. Public health. China takes aim at rampant antibiotic resistance. Science, 2012, 336(6083): 795. DOI:10.1126/science.336.6083.795 |
| [2] | Wang J, Wang XH, Xu H. Antimicrobial resistance and dissemination of multidrug resistant organisms-A review. Acta Microbiologica Sinica, 2016, 56(11): 1671-1679. (in Chinese) 王娟, 王新华, 徐海. 多重耐药菌在人类、动物和环境的耐药和传播机制. 微生物学报, 2016, 56(11): 1671-1679. |
| [3] | Huang YJ, Cheng MM, Li WH, Wu LH, Chen YS, Luo YM, Christie P, Zhang HB. Simultaneous extraction of four classes of antibiotics in soil, manure and sewage sludge and analysis by liquid chromatography-tandem mass spectrometry with the isotope-labelled internal standard method. Analytical Methods, 2013, 5(15): 3721-3731. DOI:10.1039/c3ay40220g |
| [4] | Nan W, Min Q, Bing Z, Cheng WD, Zhu YG. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China. Environmental Science & Technology, 2010, 44(18): 6933-6939. |
| [5] | Sarmah AK, Meyer MT, Boxall ABA. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics (VAs) in the environment. Chemosphere, 2006, 65(5): 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
| [6] | Zou W, Luo Y, Zhou QX. Pollution and environmental regulation of antibiotic resistance genes (ARGs) in livestock manure. Journal of Agro-Environment Science, 2014, 33(12): 2281-2287. (in Chinese) 邹威, 罗义, 周启星. 畜禽粪便中抗生素抗性基因(ARGs)污染问题及环境调控. 农业环境科学学报, 2014, 33(12): 2281-2287. DOI:10.11654/jaes.2014.12.001 |
| [7] | Sun DC, Wang B, Zhu LH. Advances in molecular mechanisms of bacterial resistance caused by stress-induced transfer of resistance genes-A review. Acta Microbiologica Sinica, 2013, 53(7): 641-647. (in Chinese) 孙东昌, 王兵, 竺利红. 胁迫诱导抗性基因转移导致细菌耐药的分子机制研究进展. 微生物学报, 2013, 53(7): 641-647. |
| [8] | Whitehead TR, Cotta MA. Stored swine manure and swine faeces as reservoirs of antibiotic resistance genes. Letters in Applied Microbiology, 2013, 56(4): 264-267. DOI:10.1111/lam.12043 |
| [9] | ?i?ková H, Pastor B, Kozánek M, Martínez-Sánchez A, Rojo S, Taká? P. Biodegradation of pig manure by the housefly, Musca domestica:A viable ecological strategy for pig manure management. PLoS One, 2012, 7(3): e32798. |
| [10] | Zhang ZJ, Liu M, Zhu J. Organic waste treatment by earthworm vemicomposting and larvae bioconversion:Review and perspective. Environment Science, 2013, 34(5): 1679-1686. (in Chinese) 张志剑, 刘萌, 朱军. 蚯蚓堆肥及蝇蛆生物转化技术在有机废弃物处理应用中的研究进展. 环境科学, 2013, 34(5): 1679-1686. |
| [11] | Zhang ZJ, Wang H, Zhu J, Suneethi S, Zheng JG. Swine manure vermicomposting via housefly larvae (Musca domestica):The dynamics of biochemical and microbial features. Bioresource Technology, 2012, 118: 563-571. DOI:10.1016/j.biortech.2012.05.048 |
| [12] | Wang H, Zhang ZJ, Czapar GF, Winkler MKH, Zheng JG. A full-scale house fly (Diptera:Muscidae) larvae bioconversion system for value-added swine manure reduction. Waste Management & Research, 2013, 31(2): 223-231. |
| [13] | D'Costa VM, McGrann KM, Hughes DW, Wright GD. Sampling the antibiotic resistome. Science, 2006, 311(5759): 374-377. DOI:10.1126/science.1120800 |
| [14] | Ghosh S, Lapara TM. The effects of subtherapeutic antibiotic use in farm animals on the proliferation and persistence of antibiotic resistance among soil bacteria. The ISME Journal, 2007, 1(3): 191-203. DOI:10.1038/ismej.2007.31 |
| [15] | Forsberg KJ, Reyes A, Wang B, Selleck EM, Sommer MOA, Dantas G. The shared antibiotic resistome of soil bacteria and human pathogens. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761 |
| [16] | Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research, 2001, 29(9): e45. DOI:10.1093/nar/29.9.e45 |
| [17] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Pe?a AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [18] | McDonald D, Price MN, Goodrich J, Nawrocki EP, DeSantis TZ, Probst A, Andersen GL, Knight R, Hugenholtz P. An improved Greengenes taxonomy with explicit ranks for ecological and evolutionary analyses of bacteria and archaea. The ISME Journal, 2012, 6(3): 610-618. DOI:10.1038/ismej.2011.139 |
| [19] | Cole JR, Wang Q, Cardenas E, Fish J, Chai B, Farris RJ, Kulam-Syed-Mohideen AS, McGarrell DM, Marsh T, Garrity GM, Tiedje JM. The Ribosomal Database Project:improved alignments and new tools for rRNA analysis. Nucleic Acids Research, 2009, 37(Database issue): D141-D145. |
| [20] | Knights D, Kuczynski J, Charlson ES, Zaneveld J, Mozer MC, Collman RG, Bushman FD, Knight R, Kelley ST. Bayesian community-wide culture-independent microbial source tracking. Nature Methods, 2011, 8(9): 761-763. DOI:10.1038/nmeth.1650 |
| [21] | Xia LC, Steele JA, Cram JA, Cardon ZG, Simmons SL, Vallino JJ, Fuhrman JA, Sun F. Extended local similarity analysis (eLSA) of microbial community and other time series data with replicates. BMC Systems Biology, 2011, 5(S2): S15. |
| [22] | Alekshun MN, Levy SB. Molecular mechanisms of antibacterial multidrug resistance. Cell, 2007, 128(6): 1037-1050. DOI:10.1016/j.cell.2007.03.004 |
| [23] | Ramaswamy J, Prasher SO, Patel RM, Hussain SA, Barrington SF. The effect of composting on the degradation of a veterinary pharmaceutical. Bioresource Technology, 2010, 101(7): 2294-2299. DOI:10.1016/j.biortech.2009.10.089 |
| [24] | Ho YB, Zakaria MP, Latif PA, Saari N. Degradation of veterinary antibiotics and hormone during broiler manure composting. Bioresource Technology, 2013, 131: 476-484. DOI:10.1016/j.biortech.2012.12.194 |
| [25] | Dolliver H, Gupta S, Noll S. Antibiotic degradation during manure composting. Journal of Environmental Quality, 2008, 37(3): 1245-1253. DOI:10.2134/jeq2007.0399 |
| [26] | Ali M, Wang JJ, Delaune RD, Seo DC, Dodla SK, Hernandez AB. Effect of redox potential and pH status on degradation and adsorption behavior of tylosin in dairy lagoon sediment suspension. Chemosphere, 2013, 91(11): 1583-1589. DOI:10.1016/j.chemosphere.2012.12.050 |
| [27] | Chee-Sanford JC, Mackie RI, Koike S, Krapac IG, Lin YF, Yannarell AC, Maxwell S, Aminov RI. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [28] | Zhang ZJ, Shen JG, Wang H, Liu M, Wu LH, Ping F, He Q, Li HY, Zheng CF, Xu XH. Attenuation of veterinary antibiotics in full-scale vermicomposting of swine manure via the housefly larvae (Musca domestica). Scientific Reports, 2014, 4: 6844. |
| [29] | Mendes LW, Kuramae EE, Navarrete AA, van Veen JA, Tsai SM. Taxonomical and functional microbial community selection in soybean rhizosphere. The ISME Journal, 2014, 8(8): 1577-1587. DOI:10.1038/ismej.2014.17 |
| [30] | Heck K, de Marco éG, Duarte MW, Salamoni SP, van der Sand S. Pattern of multiresistant to antimicrobials and heavy metal tolerance in bacteria isolated from sewage sludge samples from a composting process at a recycling plant in southern Brazil. Environmental Monitoring and Assessment, 2015, 187: 328. DOI:10.1007/s10661-015-4575-6 |
| [31] | Riber L, Poulsen PHB, Al-Soud WA, Skov Hansen LB, Bergmark L, Brejnrod A, Norman A, Hansen LH, Magid J, S?rensen SJ. Exploring the immediate and long-term impact on bacterial communities in soil amended with animal and urban organic waste fertilizers using pyrosequencing and screening for horizontal transfer of antibiotic resistance. FEMS Microbiology Ecology, 2014, 90(1): 206-224. DOI:10.1111/fem.2014.90.issue-1 |
| [32] | Boucher Y, Labbate M, Koenig JE, Stokes HW. Integrons:mobilizable platforms that promote genetic diversity in bacteria. Trends in Microbiology, 2007, 15(7): 301-309. DOI:10.1016/j.tim.2007.05.004 |
| [33] | Ramsden SJ, Ghosh S, Bohl LJ, LaPara TM. Phenotypic and genotypic analysis of bacteria isolated from three municipal wastewater treatment plants on tetracycline-amended and ciprofloxacin-amended growth media. Journal of Applied Microbiology, 2010, 109(5): 1609-1618. |
| [34] | Zhongqi HE, Pagliari PH, Waldrip HM. Applied and environmental chemistry of animal manure:A review. Pedosphere, 2016, 26(6): 779-816. DOI:10.1016/S1002-0160(15)60087-X |
