黎志凤1

 , 张文燕1, 刘杨2, 曲绍峰1, 王岩1, 朱丽萍1, 李越中1
, 张文燕1, 刘杨2, 曲绍峰1, 王岩1, 朱丽萍1, 李越中1

1.山东大学生命科学学院, 微生物技术国家重点实验室, 山东 济南 250100;
2.山东大学数学学院, 山东 济南 250100
收稿日期:2016-06-28;修回日期:2016-08-05;网络出版日期:2016-09-05
基金项目:国家自然科学基金(31370123,30900027);高等学校博士学科点专项科研基金(200804221017);山东大学实验室建设软件项目(sy2008023)
*通信作者:黎志凤,Tel:+86-531-88363735;Fax:+86-531-88378067;E-mail:lizhifeng@sdu.edu.cn
李越中,Tel:+86-531-88363735;Fax:+86-531-88378067;E-mail:lilab@vip.163.com
摘要: [目的]以遗传片段分析仪内标法替代传统放射性标记引物延伸技术进行样本转录起始位点(TSS)分析,并弥补引物延伸技术应用于未知样本缺乏前期预测和后期评估环节,形成一套基于遗传片段分析仪内标法分析未知样品TSS的完整技术方案。[方法]以粘球菌Myxococcus DK1622来源的双拷贝GroELs基因为素材;首先从预测出发,利用数据库进行启动子和转录起始位点预测;其次,根据预测结果设计合成荧光标记引物进行靶标mRNA的反转录;再次,应用遗传片段分析技术内标法鉴定分析粘球菌来源的双拷贝GroELs基因转录起始位点(TSS)及其丰度;最后,应用正态分布理论进行鉴定结果评估。[结果]明确了转录起始位点的数量、转录丰度及最可能的TSS位点:粘球菌DK1622基因组中GroEL1拷贝存在1个启动子,TSS位点为TSS286;GroEL2拷贝存在2个启动子,TSS位点分别为TSS548和TSS502,其中TSS548转录丰度是TSS502的13.8倍,GroEL1的TSS286丰度是groEL2的TSS548丰度的14.3倍。[结论]预测结果指明了实验设计的范围,遗传片段分析仪内标检测法替代传统放射性标记法使实验更加简便、安全、自动、准确,正态分布理论进一步评估了实验结果的可信度,三者接合形成了完善的转录起始位点鉴定技术方案。
关键词: 片段分析系统 转录起始位点(TSS) 转录丰度 荧光标记引物 引物延伸 粘球菌 GroEL
Transcriptional start site analysis based on genetic fragment analysis system: from prediction to data evaluation
Li Zhifeng1

 , Zhang Wenyan1, Liu Yang2, Qu Shaofeng1, Wang Yan1, Zhu Liping1, Li Yuezhong1
, Zhang Wenyan1, Liu Yang2, Qu Shaofeng1, Wang Yan1, Zhu Liping1, Li Yuezhong1

1.State Key Laboratory of Microbial Technology, College of Life Science, Shandong University, Jinan 250100, Shandong Province, China;
2.School of Mathematics, Shandong University, Jinan 250100, Shandong Province, China
Received 28 June 2016; Revised 05 August 2016; Published online 05 September 2016
*Corresponding author: Tel:+86-531-88363735;Fax:+86-531-88378067; E-mail:lizhifeng@sdu.edu.cn
Tel:+86-531-88363735;Fax:+86-531-88378067; E-mail:lilab@vip.163.com
Supported by the National Natural Science Foundation of China (31370123, 30900027), by the Research Fund for the Doctoral Program of Higher Education of China (200804221017) and by the Laboratory Construction Software Project of Shandong University (sy2008023)
Abstract: [Objective] To establish a pipeline for unknown transcriptional start site (TSS) identification without radioactivity, we used genetic fragment analysis system and replenished two steps regarding prediction and evaluation.[Methods] We used unknown TSSs of GroEL genes from M. xanthus as a case. Firstly, we predicted the potential TSSs through bioinformatics databases. According to the prediction, we designed and synthesized fluorescence labeled primers to carry out the reverse transcription reactions. Further, we took advantage of the genetic fragment analysis system to identify TSSs with internal standards. Finally, we applied the normal distribution theory to evaluate the data.[Results] We determined the numbers, abundances and accurate sites of the TSSs:GroEL1 has one promoter and the site is TSS286, whereas GroEL2 has two promoters, and the sites are TSS548 and TSS502. TSS286 is 14.3 times more abundant than TSS548 and TSS548 is 13.8 times more than TSS502.[Conclusion] The bioinformatics analyzing indicates the range for the experimental design. TSS determination through genetic fragment analysis system is safer, more automatic and accurate. Normal distribution theory further refines the reliability of results. Combination of the three techniques establishes a more complete pipeline of primer extension for unknown TSS determination.
Key words: genetic fragment analysis system transcriptional start site (TSS) transcriptional abundances fluorescently labeled primer primer extension Myxococcus GroEL
基因转录起始位点(Transcription start site,TSS)分析是遗传学研究的经典内容[1-2]。相关工作对了解并进一步研究基因的表达和调控机制有重要意义。引物延伸实验是进行TSS鉴定的经典技术[1-3]。引物延伸法基本原理是:待测基因mRNA与过量的5?末端标记的互补DNA引物杂交,引物在反转录酶作用下进行延伸,直到TSS终止,通过检测延伸的cDNA产物长度和产量,即可判断引物5?末端与mRNA 5?起始位点的距离(据此确 定TSS)和目标RNA的丰度。经典的引物延伸技术涉及放射性标记、跑变性聚丙烯胶、曝光干胶等操作,报道始于1987年并一直沿用至今[1-9]。1991年Voss等应用单色荧光标记引物和自动测序仪进行技术改进[10],样品分析涉及延伸产物和作为参照的四碱基测序产物共5个泳道的检测,该方案在安全性和自动性两方面进行了很大的改善从而被****们采用[5];1993年My?h?nen等进一步在参照的选择和标记上进行了改进,将延伸引物的测序结果选作参照,用4色荧光标记实现了检测参照仅需1个泳道[11]。1999年Altermann等以已知启动子为实验材料,对基于测序仪的自动荧光引物延伸实验中的模板和引物浓度进行了优化摸索[12]。上述方案中,引物延伸产物和参照均是在独立平行的毛细管中进行,通过比较两根毛细管中峰的滞留时间来判断TSS,其结果的一致性和准确性取决于毛细管间的平行度。2003年Fekete等用添加内标参照法对检测技术进一步改进,然而测试结果仍需辅助平行测序反应进行校正,作者通过对齐延伸反应和测序反应中的参照来判断TSS[13]。2007年Qi等利用地高辛标记替代传统放射性标记[14],对传统技术进行了安全性改进,为精确性要求不高且无法做测序或片段分析检测的实验室提供了另一选择方案。
综上,自1987年引物延伸技术报道应用以来,经历了多次技术革新与改进,其中自动荧光引物延伸技术在安全、省时和自动性方面作出了很大的改进,但已报道的自动荧光引物延伸技术目前尚依赖平行测序反应,且缺乏前期预测分析和后期结果处理评估环节。为完善当前相关技术,论文拟以粘球菌DK1622来源的双拷贝groELLs基因为研究素材[15-17],基于片段分析系统,建立一套基于荧光内标法鉴定未知样本TSS的完整技术方案。
1 材料和方法 1.1 材料
1.1.1 菌株: Myxococcus xanthus DK1622[18](粘球菌模式菌株,D. Kaiser,Stanford University友好馈赠)。
1.1.2 软件或数据库: NCBI Genome Database (检索获取目标序列信息);NNPP (Neural Network Promoter Prediction,http://www.fruitfly.org/seq_tools/promoter.html,启动子预测识别)[19];BPROM (http://www.softberry.com/berry.phtml,启动子预测识别);SAK (http://nostradamus.cs.rhul.ac.uk/~leo/sak_demo/,启动子预测识别)[20];PPP (http://bioinformatics.biol.rug.nl/websoftware/ppp/ppp_start.php,启动子预测识别);Primer Premier 5(引物设计);SimVector 4 (序列注释);Clustalx (多序列比对);BioEdit (序列分析编辑);NanoDrop software (RNA质量检测);GeXP Multiplexed Gene Expression Analysis System (片段分析系统)[21]。
1.1.3 试剂: RNAlater (Qiagen);SV Total RNA Isolation System (Promega);DNA-free kit (Takara);PrimeScriptTM Reverse Transcriptase试剂盒 (TaKaRa);Sample loading solution (SLS,Beckman Coulter Inc);Size Standard-600(Beckman Coulter Inc);5×RNA固定/保护缓冲液(95%无水乙醇+5% pH 4.5的酸酚);分子生物学及生化试剂均为分析纯.
1.1.4 仪器: GeXP Multiplexed Gene Expression Analysis System (Beckman);ND-1000Spectrophotometer (NanoDrop Technologies,Inc.);其它常规分子生物学仪器等。 1.2 启动子预测分析与引物设计 登入NCBI数据库,由Myxococcus xanthus DK 1622菌株基因组序列记录号(CP000113),检索并获取GroEL1和GroEL2,及其上下游序列(groELL1:6125113-6127957;groELL2:5532695-5530361),提交多个启动子预测识别数据库执行启动子及TSS在线预测。借助Primer Premier 5软件在预测TSS下游设计并合成反向Cy5荧光标记引物(*GroEL1R:5?-TCCGTTGGCAACCGAAAGC-3?和*GroEL2R:5?-GCGGACTGATGGAAGAAAAT-3?),同时在预测的TSS上游设计正向常规引物(GroEL1F:5?-GGAGACCTGGGAAGCGTAGC-3?和GroEL2F: 5?-TGGGTTCACGGTCTACTC-3?),荧光标记借由磷氨基团连接器(phosphoramidite linker)连接至引物末端,引物由上海生物工程公司合成。借助BioEdit软件将DK 1622基因组数据库本地化并考查反向引物特异性。借助SimVector 4软件对预测结果进行注释。
1.3 RNA的制备纯化与质检 将DK 1622转接到CTT液体培养基中,30°C培养至对数期(细胞密度达108-109 个/mL),取1 mL培养菌体,加入250 μL RNAlater[22]或者5×RNA固定/保护缓冲液[23-24],离心,弃上清;依RNA提取试剂盒制备RNA;用DNA-free kit对RNA样品进行除DNA处理。RNA纯化后,稀释10倍,用ND-1000分光光度计进行质量评估。
1.4 反转录扩增与产物纯化 取5μg RNA依据PrimeScriptTM Reverse Transcriptase试剂盒说明书配制反转录操作体系,于50 °C下反转录1 h。加入Glycogen、NaOAC和乙醇对样品进行除盐纯化处理,并重悬于等体积的SLS中。
1.5 荧光cDNA片段检测与数据分析处理 40μLSLS样品中加入0.5μL Size Standard-600进行混合,载入上样板,编辑程序,设置仪器参数(毛细管温度:50°C预热→变性:90°C120s→注射电压:2.0KV30s→分离电压:4.8KV80min),启动片段分析仪进行毛细管电泳分离与检测。输出结果用遗传片段分析系统GeXP Multiplexed Gene Expression Analysis System进行处理分析,设置分析参数(染料迁移校正:PAver.1;斜率阈值:50;置信水准:99%;染料谱图:使用校正的染料谱;内标:SizeStandard-600;使用的染料:D1;模型:Quartic)。
1.6 TSS位点定位概率分析 根据内标SizeStandard-600标准曲线,软件分析给出的标准偏差值,定义为σ;根据待测荧光cDNA片段迁移时间,依标准曲线由软件分析给出的片段测量大小值,定义为随机变量ε;可能的实际片段大小值定义为a;对于ε~N(a,σ2),记
2 结果和分析 2.1 启动子预测与注释 Myxococcus xanthus DK 1622菌株基因组中含有2个groELL基因。groELL1(MXAN_4895)与上游groES形成操纵子,而groELL2(MXAN_4467)上游无groES。利用多个数据库对启动子及TSS进行了预测。综合预测结果显示(图 1),在0.4的阈值条件下,groELL1上游非编码区预测出一个可能的启动子,TSS位于280bp处,TSS280与翻译起始位点(Translation start site,TLS)ATG的距离TSS280-TLS为128 bp。而groELL2上游非编码区预测出2个可能的启动子,TSS分别位于502 bp和550 bp处,TSS502-TLS为68 bp,TSS550-TLS距离为20 bp。
 |
| 图 1. 双拷贝groELLs启动子预测注释结果及设计的引物位置分布图 Figure 1. The predicted promoters annotation of the two groELLs and the location of designed primers (The shadow labelled the predicted TSS sites and the bold bases indicate the most likely TSS from experiments). A: GroEL1; B: GroEL2. |
| 图选项 |
2.2 引物设计与特异性 在预测的TSS下游100bp左右的位置择优选择设计了反向Cy5荧光标记引物*GroEL1R和*GroEL2R。其中*GroEL1R距预测的Tss距离长度为95 bp,*GroEL2R距预测的2个TSS位点的距离长度分别为55 bp和103 bp (图 1)。Primer premier分析结果显示:引物*GroEL2R无任何二级结构,而引物*GroEL1R则在自身5?端形成二聚体,能值为-5.2 kcals/mol。Bioedit建库本地化比对Myxococcus xanthus DK 1622基因组数据分析结果显示:*GroEL2R存在3?处潜在的错配位点(6764101-6764087,8556774-8556762和5611885- 5611873),这些错配位点在引物的3?末端存在连续的2至7不匹配碱基,理论上不易引发错配延伸反应。*GroEL1R在整个基因组中预测无任何错配信息。利用常规PCR扩增检测引物效率和特异性,groELL1验证引物对GroEL1F-*GroEL1R扩增产物长491bp,groELL2验证引物对GroEL2F-*GroEL2R扩增产物长340 bp。电泳结果显示上述引物有很好的特异性,GroEL1F-*GroEL1R扩增产物前端存在引物二聚体,目标产物扩增效率较GroEL2F-*GroEL2R低(图 2)。
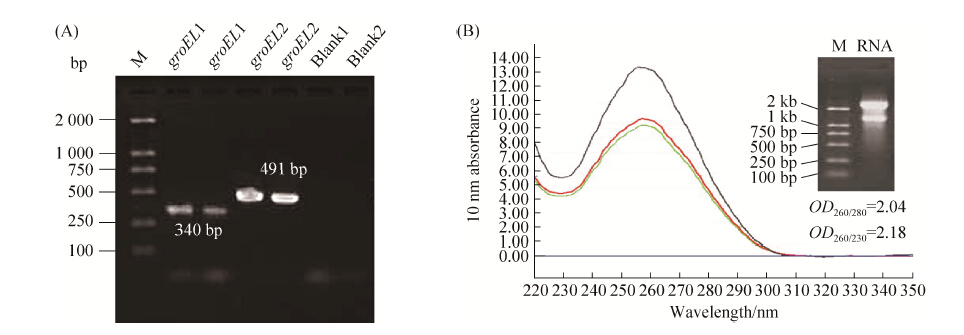 |
| 图 2. 引物扩增效率与RNA质量检测 Figure 2. Detections of nucleic acid samples. A: efficiency of primers amplification; B: RNA quality detection. |
| 图选项 |
2.3 反转录产物荧光cDNA片段检测 制备的RNA样品检测值为:样品原始浓度3.83μg/μL,23S rRNA亮度约为16S rRNA的2倍,OD260/280值为2.04,OD260/230为2.18,因此从完整性和纯度方面,RNA样本质量较理想(图 2)。
反向荧光标记引物与纯化后的RNA样品执行反转录并纯化后,加入内标执行荧光片段延伸产物检测分析,样品在Beckman CEQ片段分析仪中检测结果见图 3,蓝色为样品峰,红色为内标。内标的线性标准曲线图拟合很好,groELL1的相关系数值为0.999996,标准偏差在0.38bp,groELL2相关系数值为0.999997,标准偏差在0.35 bp。图 3-A内标中60 bp参照峰的面积为2.58×104 rfu×mm (相对荧光强度单位rfu乘以半峰宽单位mm),样品中检测到1个启动子峰,该groELL1-P峰面积高达1.57×105 rfu×mm,片段测量大小89.14 bp。图 3B内标中60 bp参照峰的面积为2.36×104 rfu×mm,检测到2个明显的样品峰,高丰度片段groELL2-P2信号面积值为5.30×104 rfu×mm,测量大小为56.92 bp;低丰片段groELL2-P1信号强度为3.84×103 rfu×mm,测量大小为102.24 bp。
 |
| 图 3. 基于遗传片段分析系统的荧光片段检测分析结果 Figure 3. The fragment analysis results, the blue is sample peak, the red is standard. |
| 图选项 |
2.4 TSS 最可能位点定位的概率分析 依“3σ原则”:测量值落在(a-3σ,a+3σ )之中的概率是99.73%,落在该区间之外的概率几乎为零。因此,测量值ε 满足:a-3σ<ε< a+3σ 。就groEL2-P2 而言,测量值ε=56.92,σ=0.35,即得55.87< a< 57.97,而 a 表征的是片段的实际长度,取整数,因此a 值只能是56 或者57。更进一步,对测量值ε =56.92 而言,当55< a< 57 时,设:a ~ N(56.92,0.352 ),则
| $\begin{align} & P(55<a<57)=\left( P\frac{55-56.92}{0.35}<\eta <\frac{57-56.92}{0.35} \right) \\ & =P(-5.48571<\eta <0.228571) \\ & =\Phi (0.228571)-\Phi (-5.48571) \\ & =\Phi (0.228571)+\Phi (5.48571)-1\approx 0.591 \\ \end{align}$ |
| $\begin{align} & P(56<a<58)=\left( P\frac{56-56.92}{0.35}<\eta <\frac{58-56.92}{0.35} \right) \\ & =\Phi (3.085714)+\Phi (2.62857)-1\approx 0.9957 \\ \end{align}$ |
同理,groEL2-P1 测量值ε =102.24,σ =0.35,由“3σ 原则”及取整,groEL2-P1 中的a 值只能是102或者103;当101<a<103时,对测量值ε =102.24而言,设:a ~ N(102.24,0.352 ),则P(101<a<103) Φ(2.171429) -Φ(-3.54285714) ≈ 0.9848;而当102<a<104时,P(102< a<104)=0.7549;由此,测量值ε =102.24,由实际值102 测量波动而来的概率是97.66%,而由103 测量波动而来的概率是75.49%,因此groEL-P1 TSS 最可能是102 bp,概率为97.66%。
同理,groEL1-P 测量值ε =89.14,σ =0.38,由“3σ原则”及取整,groEL1-P 的a 值只能是89 或者90,对测量值ε =89.14而言,设a ~ N(89.14,0.382 )则P(88< a< 90) ≈ 0.9868;而P(89< a< 91) ≈ 0.6443;因此groEL1-P TSS 最可能是89 bp,概率为98.68%。
3 讨论 以粘球菌GroELs基因启动子为素材,鉴定结果表明:粘球菌DK1622基因组中的GroEL1拷贝中仅存在1个启动子,groELL1-P引物延伸产物长度和TSS位点分别为89bp和TSS286;GroEL2拷贝存在2个启动子,其引物延伸产物长度和TSS位点分别为groELL2-P1:57 bp,TSS548和groELL2-P2:102 bp,TSS502;在所检测的RNA状态中groELL2-P1 TSS548转录丰度是groELL2-P2 TSS502的13.8倍,groELL1-P的TSS286丰度是groELL2-P1 TSS548丰度的14.3倍,与前期基因表达功能实验吻合[15-16]。
对于未知样品,开展实验前,充分的生物信息学预测分析对实验顺利开展有很好的方向性指导。然而,没有一种预测方法能保证100%的敏感性和正确性。借助几种不同的方法进行预测,若能得出类似的结果,则该预测结果在一定程度上可参考。目前,原核生物启动子TSS预测注释资源包括基于TATA box和Inr局部信息预测的NNPP[15],基于功能motifs和寡核苷酸信息预测的BPROM,Gordon等开发的基于整个启动子信号的SAK[20],基于隐马模型预测的PPP。NNPP给出了预测的启动子区段及其TSS;BPROM给出了预测的TSS和可能的-10、-35区;而SAK则给出了所有可能的TSS碱基区段,及各碱基预测为TSS的得分情况。以粘球菌groELL2为例,NNPP数据库在0.4的阈值下预测出2个可能的启动子;BPROM数据库仅预测到1个启动子,该启动子与NNPP数据库预测结果之一吻合;SAK数据库预测结果中包含了NNPP、BPROM中预测的TSS。实验鉴定结果表明groELL2确实存在2个启动子,但预测的TSS位点存在偏差。因此,生物信息学预测能够很好地帮我们定位延伸引物的设计范围,但预测结果准确性需实验修正。
从荧光片段分析结果看,无论*GroEL2R还是*GroEL1R,均无明显的非特异性产物。因此,当引物与基因组模板存在错配,而引物5?末端存在连续的2-7个不匹配碱基时,并不影响荧光引物延伸反应特异性。当反向荧光引物存在自身二聚体时,PCR扩增效率会将低,但因延伸反应中引物较模板完全过量且是线性扩增,似乎*GroEL1R的部分二聚体并未显著影响到引物延伸反应的定位和丰度测量,GroEL1的表达丰度显著高于GroEL2,与前期功能实验吻合[15-16]。
合成荧光标记引物时,需明确荧光标记物与引物间的连接器。在做精确片段分析时,荧光基团的连接方式决定了校正参数的选择。磷氨基团连接器需选择PA-Ver1染料迁移校正,而激活的酯连接器则选AE-Ver2染料迁移校正。针对本文的groELLs,由于Cy5荧光标记用的是磷氨基团连接器,所以选PA-Ver1染料迁移校正,此参数下分析的groELL2引物延伸片段大小分别为56.92bp和102.24 bp,groELL1引物延伸片段大小为89.14 bp。如不进行校正则分析所得groELL2片段大小分别为54.88 bp和100.54 bp,groELL1片段大小为87.93 bp,分别较实际值小1-3 bp左右。而误选AE-Ver2校正则groELL2片段大小为50.41 bp和97.38 bp,groELL1片段大小为83.98 bp,片段较实际值小 4-7 bp左右。推测2003年Fekete等进行片段分析时因未能进行参数校正,所以检测结果存在几碱基偏差[13]。由此,正确校正参数进行分析,可省却辅助的平行测序反应,使基于片段分析的TSS检测技术仅在一根毛细管中即可完成,技术流程更为简便、高效。
片段分析仪以内标中片段迁移时间和片段大小为坐标绘制标准曲线,并得出标准曲线的共线性和标准偏差参数,由标准曲线及未知荧光片段的迁移时间即可得出片段的大小。然而通过标准曲线计算得到的片段大小并非整数,而实际的片段大小应该是整数。由此可见检测结果不可避免地存在微小测量误差,这种测量误差是由为数众多相互独立的各随机因素的微小影响叠加而成,因而符合正态分布[25-26]。应用正态分布理论,我们可以评估所测结果匹配上某个真实整数值的概率并明确最可能的实际片段长度。
论文建立了一套从生物信息预测出发,基于片段分析系统的启动子转录起始位点鉴定和转录起始水平检测的完善技术方案,涉及8个技术环节:启动子预测分析、荧光标记引物的设计与合成、RNA制备、RT-PCR扩增、扩增产物纯化、荧光片段分离检测、数据分析处理和以及误差评估分析。我们已将该技术成功应用于埃博霉素生物合成基因簇启动子分析[27],该技术方案可广泛应用于各物种来源的未知基因启动子起始位点鉴定分析和不同状态下相关基因转录起始丰度分析。
参考文献
| [1] | Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual (2nd ed.). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory, 1989. http://www.oalib.com/references/4929497 |
| [2] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory, 2001. http://www.oalib.com/references/10425460 |
| [3] | Walmsley M, Leonard M, Patient R. Primer extension analysis of mRNA//Rapley R.NJ: Humana Press,, 2000: 195-199. |
| [4] | Wang J, Dong XB, Gao LX, Zhou DS, Yin Z, Zhang YQ. Transcriptional regulation of hcp1 by H-NS in Vibrio parahaemolyticus.Acta Microbiologica Sinica, 2016, 56(1): 143–149(in Chinese).王洁, 董新波, 高丽晓, 周冬生, 殷喆, 张义全. H-NS蛋白对副溶血弧菌hcp1的转录调控.微生物学报, 2016, 56(1): 143–149. |
| [5] | Wang JY, Wang WS, Li X, Zhao H, Yang KQ. Progress in transcriptional studies.Chinese Journal of Biotechnology, 2015, 31(8): 1141–1150(in Chinese).王俊阳, 王为善, 李肖, 赵华, 杨克迁. 转录调控研究的方法学进展.生物工程学报, 2015, 31(8): 1141–1150. |
| [6] | Ni B, Zhang YQ, Huang XX, Yang RF, Zhou DS. Transcriptional regulation of dps by OxyR protein in Yersinia pestis.Acta Microbiologica Sinica, 2013, 53(7): 685–690(in Chinese).倪斌, 张义全, 黄新祥, 杨瑞馥, 周冬生. 鼠疫菌OxyR调控子蛋白对dps的转录调控机制.微生物学报, 2013, 53(7): 685–690. |
| [7] | Adolph MB, Webb J, Chelico L. Retroviral restriction factor APOBEC3G delays the initiation of DNA synthesis by HIV-1 reverse transcriptase.PLoS One, 2013, 8(5). |
| [8] | Yang YT, Yang GD, Liu SJ, Guo XQ, Zheng CC. Isolation and functional analysis of a strong specific promoter in photosynthetic tissues.Science in China (Series C), 2003, 33(4): 298–306(in Chinese).杨予涛, 杨国栋, 刘石娟, 郭兴启, 郑成超. 一个光合组织特异表达强启动子的分离及功能分析.中国科学(C辑:生命科学), 2003, 33(4): 298–306. |
| [9] | Therit B, Cheung JK, Rood JI, Melville SB. NanR, a transcriptional regulator that binds to the promoters of genes involved in sialic acid metabolism in the anaerobic pathogen Clostridium perfringens.PLoS One, 2015, 10(7). |
| [10] | Voss H, Wirkner U, Jakobi R, Hewitt NA, Schwager C, Zimmermann J, Ansorge W, Pyerin W. Structure of the gene encoding human casein kinase II subunit beta.Journal of Biological Chemistry, 1991, 266(21): 13706–13711. |
| [11] | My?h?nen S, Wahlfors J. Automated fluorescent primer extension.Biotechniques, 1993, 14(1): 16–17. |
| [12] | Altermann E, Klein JR, Henrich B. Synthesis and automated detection of fluorescently labeled primer extension.Biotechniques, 1999, 26(1): 96–98. |
| [13] | Fekete RA, Miller MJ, Chattoraj DK. Fluorescently labeled oligonucleotide extension:a rapid and quantitative protocol for primer extension.Biotechniques, 2003, 35(1): 90–94. |
| [14] | Qi XT, Chai XQ, Chai TY. An improved primer extension method for detection of mRNA start-points using non-radioactive digoxigenin-labeling primers.Biotechnology Letters, 2007, 29(7): 1125–1128DOI:10.1007/s10529-007-9356-z. |
| [15] | Wang Y, Zhang WY, Zhang Z, Li J, Li ZF, Tan ZG, Zhang TT, Wu ZH, Liu H, Li YZ. Mechanisms involved in the functional divergence of duplicated GroEL chaperonins in Myxococcus xanthus DK1622.PLoS Genetics, 2013, 9(2). |
| [16] | Li J, Wang Y, Zhang CY, Zhang WY, Jiang DM, Wu ZH, Liu H, Li YZ. Myxococcus xanthus viability depends on GroEL supplied by either of two genes, but the paralogs have different functions during heat shock, predation and development.Journal of Bacteriology, 2010, 192(7): 1875–1881DOI:10.1128/JB.01458-09. |
| [17] | Wang Y, Li X, Zhang W, Zhou X, Li YZ. The groEL2 gene, but not groEL1, is required for biosynthesis of the secondary metabolite myxovirescin in Myxococcus xanthus DK1622.Microbiology, 2014, 160. |
| [18] | Goldman BS, Nierman WC, Kaiser D, Slater SC, Durkin AS, Eisen JA, Ronning CM, Barbazuk WB, Blanchard M, Field C, Halling C, Hinkle G, Iartchuk O, Kim HS, Mackenzie C, Madupu R, Miller N, Shvartsbeyn A, Sullivan SA, Vaudin M, Wiegand R, Kaplan HB. Evolution of sensory complexity recorded in a myxobacterial genome.Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(41): 15200–15205DOI:10.1073/pnas.0607335103. |
| [19] | Reese MG. Application of a time-delay neural network to promoter annotation in the Drosophila melanogaster genome.Journal of Computational Chemistry, 2001, 26(1): 51–56DOI:10.1016/S0097-8485(01)00099-7. |
| [20] | Gordon L, Chervonenkis AY, Gammerman AJ, Shahmuradov IA, Solovyev VV. Sequence alignment kernel for recognition of promoter regions.Bioinformatics, 2003, 19(15): 1964–1971DOI:10.1093/bioinformatics/btg265. |
| [21] | Beckman Coulter Inc.. GenomeLab Fragment Analysis protocol. USA: Beckman Coulter Inc., 2007. |
| [22] | Henry R, Crane B, Powell D, Deveson LD, Li ZF, Aranda J, Harrison P, Nation RL, Adler B, Harper M, Boyce JD, Li J. The transcriptomic response of Acinetobacter baumannii to colistin and doripenem alone and in combination in an in vitro pharmacokinetics/pharmacodynamics model.Journal of Antimicrobial Chemotherapy, 2015, 70(5): 1303–1313DOI:10.1093/jac/dku536. |
| [23] | Pan HW, Liu H, Liu T, Li CY, Li ZF, Cai K, Zhang CY, Zhang Y, Hu W, Wu ZH, Li YZ. Seawater-regulated genes for two-component systems and outer membrane proteins in Myxococcus.Journal of Bacteriology, 2009, 191(7): 2102–2111DOI:10.1128/JB.01556-08. |
| [24] | Liu H, Dong J, Liu MQ, Jin Q. A method of RNA isolation of bacterial from infected mammalian cells.Acta Microbiologica Sinica, 2004, 44(5): 672–675(in Chinese).刘红, 董杰, 刘墨青, 金奇. 一种从感染的培养细胞中分离细菌RNA的方法.微生物学报, 2004, 44(5): 672–675. |
| [25] | Tamhane AC, Dunlop DD. Statistics and data analysis: from elementary to intermediate. New Jersey: Prentice Hall, 2000. http://cn.bing.com/academic/profile?id=3f0b5c5f776dc2d32dfe9a5063aea111&encoded=0&v=paper_preview&mkt=zh-cn |
| [26] | Liang XY. Normality Test.Beijing: China Statistics Press, 1997(in Chinese).梁小筠. 正态性检验.北京: 中国统计出版社, 1997. |
| [27] | Zhu LP, Li ZF, Sun X, Li SG, Li YZ. Characteristics and activity analysis of epothilone operon promoters from Sorangium cellulosum strains in Escherichia coli.Applied Microbiology and Biotechnology, 2013, 97(15): 6857–6866DOI:10.1007/s00253-013-4830-0. |
