刘舒婷, 苏杨, 姚玉峰


上海交通大学医学院, 上海 200025
收稿日期:2017-04-14;修回日期:2017-07-17;网络出版日期:2017-08-04
基金项目:国家“973计划”(2015CB554203)
作者简介:姚玉峰,上海交通大学医学院基础医学院博士生导师。现任中国微生物学会分析微生物专业委员会委员、美国微生物学会会员、上海市微生物学会医学微生物专业委员会委员等。涉及的研究领域主要包括:蛋白质乙酰化修饰调节病原菌毒力,宿主免疫分子乙酰化修饰与抗菌感染,细菌Ⅵ型分泌系统的结构组成以及分泌蛋白功能研究,以及新型结核病诊断技术开发。目前主持科技部“973计划”课题、国家自然科学基金、“十一五”传染病重大专项以及上海市科委启明星计划等课题。校内主讲医学本科生《医学微生物学》和研究生《细胞微生物学》等课程。近三年来发表数篇SCI论文,也曾受邀撰写综述,科研成果瞩目
*通信作者:姚玉峰, Tel:+86-21-63846590, Fax:+86-21-64671226, E-mail:yfyao@sjtu.edu.cn
摘要:蛋白质翻译后修饰是调控蛋白质生物学功能的重要步骤之一。甲基化修饰作为蛋白质翻译后修饰的一种重要形式,参与了真核生物和原核生物的多种细胞进程。本文综述了目前蛋白质甲基化的研究进展,包括真核生物、原核生物,组蛋白和非组蛋白,以及多种氨基酸位点的甲基化修饰。这些发现丰富了人们对蛋白质甲基化修饰的认识,对深入了解蛋白质翻译后修饰的功能具有重要意义。
关键词: 甲基化修饰 O-甲基化 N-甲基化
Progress in protein methylation
Shuting Liu, Yang Su, Yufeng Yao


School of Medicine, Shanghai Jiao Tong University, Shanghai 200025, China
Received 14 April 2017; Revised 17 July 2017; Published online 4 August 2017
*Corresponding author: Yufeng Yao, Tel:+86-21-63846590, Fax:+86-21-64671226, E-mail:yfyao@sjtu.edu.cn
Supported by the National Basic Research Program of China (973 Program) (2015CB554203)
Abstract: Protein post-translational modifications, as an important step in protein maturation, participate in the regulation of diverse protein biological functions. Methylation is one of the modifications and plays multiple roles in cellular processes, ranging from eukaryotes to prokaryotes. Protein methylation occurs on various amino acid residues, such as lysine, arginine, glutamine and others. This review summarized the progress in protein methylation, including both eukaryote and prokaryote, as well as histone and non-histone proteins.
Key words: protein methylation O-methylation N-methylation
翻译后修饰(Post-translational modifications,PTMs),是指蛋白质在合成之后发生的共价修饰,一般指酶学的修饰,也可以由小分子物质修饰产生。肽段在形成之后经过修饰成为成熟蛋白质,并在细胞生物进程中发挥重要作用。因此,PTMs作为细胞信号通路的重要组成部分,是蛋白质动态反应以及相互作用的分子基础,同时也是细胞信号调控的重要靶点。
目前已经发现蛋白质存在多种PTMs,从不同程度改变蛋白质的结构和功能,从而对生命活动产生一系列影响。甲基化作为蛋白质PTMs的一种,最早发现于20世纪60年代的鼠伤寒沙门菌鞭毛蛋白的N-甲基化赖氨酸[1],引起了极大关注,但此后甲基化研究发展缓慢,很快不再作为主流方向。进入21世纪,得益于分子生物学技术的发展,甲基化研究有了较为显著的进展。作为细胞生物进程中的一个重要组成部分,蛋白质甲基化在细胞的生理及致病过程中均发挥不可或缺的调节作用[2]。
甲基化反应由甲基转移酶(Methyltransferases,MTs)催化发生,MTs作用的底物可以是核酸,也可以是蛋白质。本文仅围绕蛋白质的甲基化进行阐述。根据MTs作用的底物氨基酸种类差异,蛋白质甲基转移酶(Protein methyltransferases,PMTs)主要分为蛋白质赖氨酸甲基转移酶(Protein lysine methyltransferases,PKMTs)、蛋白质精氨酸甲基转移酶(Protein arginine methyltransferases,PRMTs)等,此外,能发生甲基化修饰的氨基酸还包括组氨酸、天冬氨酸[3]等。另外,MTs作用底物除核酸和蛋白质之外,还包括一些小分子物质如邻苯二酚和甘氨酸等,以及砷等一些化学元素[4]。甲基转移酶具有位点特异性并在多种生理活动中发挥重要作用,或可作为潜在的药物靶点,用于治疗癌症、病毒或细菌感染等。
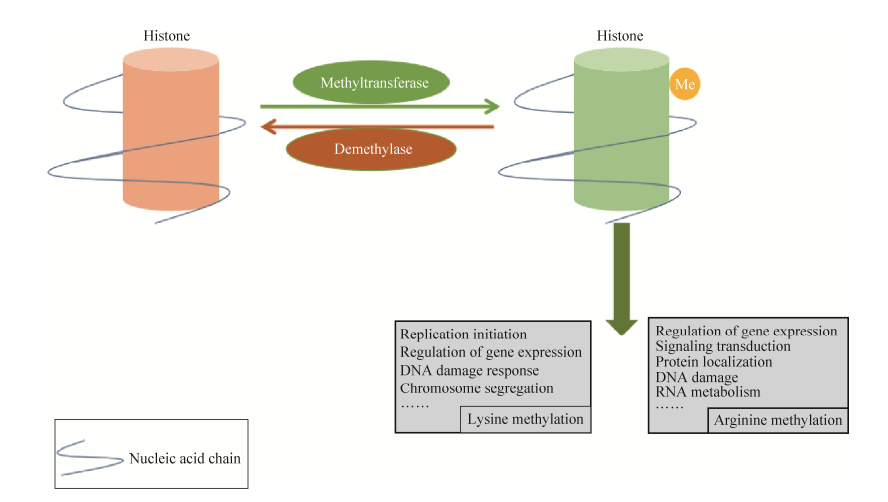
1 真核生物蛋白质甲基化修饰 1.1 组蛋白甲基化修饰 真核生物的染色质结构对基因表达的调控非常重要,组蛋白的N-端尾巴突出在核小体的外面,为多种PTMs的产生创造了条件。组蛋白的甲基化最早发现于1964年[5],源于蛋白侧链与S-腺苷甲硫氨酸(SAM)相互作用后发生的翻译后修饰[6],是一种相对稳定的PTM类型。组蛋白在很多赖氨酸和精氨酸位点上均存在甲基化修饰,但其甲基化主要发生于H3、H4的N-端赖氨酸和精氨酸残基,这些位点的甲基化修饰影响染色质的结构特性,进而对基因的转录进行调控。组蛋白的甲基化是一种非常重要且广泛存在的染色质修饰,通过影响染色质的精密整合,或与其他蛋白质复合物之间的信号传递来调控基因的表达,进而影响细胞的多种生理过程[2]。
最初认定组蛋白甲基化是不可逆的蛋白质修饰,后来随着组蛋白H3K4去甲基化酶(Demethylase) LSD1的发现[7],表明组蛋白的甲基化也是可逆的。目前已经鉴定出相当数量的甲基转移酶和去甲基化酶,二者协同参与介导氨基酸的甲基化与去甲基化修饰。
1.1.1 组蛋白赖氨酸甲基化:组蛋白的赖氨酸甲基化通常发生在组蛋白H3的第4、9、27、36、79位等赖氨酸位点,以及H4的第20和59位等赖氨酸位点上[4]。从单细胞生物到哺乳动物,几乎所有组蛋白的赖氨酸甲基转移酶(Lysine methyltransferases,KMTs)都含有高度保守的氨基酸序列,根据其保守序列差异大致可分为两类,以下将分别进行阐述。
大多数的KMTs含有一个高度保守的催化结构域,从酿酒酵母到人,甚至细菌和病毒的多个蛋白质序列中都检测到其存在。该结构域最初发现于黑腹果蝇的蛋白质中,是一个存在于修饰子SUV (Suppressor of variegation) 3–9、多梳蛋白家族E(Z) (Enhancer of Zeste)、以及TRX (Trithorax)蛋白家族的模体序列,称为SET催化结构域[8]。除了SET结构域之外,不同的KMTs还含有不同的蛋白结构域,根据其初级氨基酸序列、结构域以及作用底物的不同,可以将KMTs分为8种不同类别[9]。
另一类组蛋白KMTs含有保守的酵母Dot1 (Disruptor of telomeric silencing 1) 蛋白,其同源序列称为DOT1L (Dot1-like protein)。Dot1/DOT1L催化组蛋白H3K79甲基化的发生,后者参与细胞分裂的多种时期。甲基化修饰影响染色质的结构,进而调控基因表达、复制起始、DNA损伤应答、微管重组、染色体分离及异染色质形成。因此Dot1/DOT1L对于维持基因组和染色体的稳定性是必需的[10]。
含有SET结构域的KMTs通常甲基化组蛋白N-端的赖氨酸,而含有Dot1和DOT1L的KMTs则对组蛋白的球状中心部分H3进行甲基化,即只能够甲基化核小体组蛋白H3,而非游离的H3[10]。
赖氨酸可以被KMTs单甲基化、双甲基化或三甲基化[11]。SUV39H1是最早发现的组蛋白KMT,它的主要甲基化位点是H3K9。研究发现,H3K9双甲基化修饰后,该位点几乎不再具有酶学活性,而其三甲基化肽段则完全不再作为甲基化酶的底物;另外,H3K9的乙酰化能够抑制该位点甲基化的发生,而H3S10的去磷酸化是H3K9发生甲基化修饰的前提条件,因此H3S10的磷酸化能够抑制相邻位点K9的甲基化。
根据其修饰位点氨基酸种类的不同,组蛋白的甲基化修饰表现为对基因转录的激活或者抑制。举例来说,组蛋白H3K4、H3K36、H3K79位点的甲基化能够有效地激活相应基因的表达,而H3K9、H3K27、H4K20的双甲基化或三甲基化通常与基因沉默相关[12]。
组蛋白的赖氨酸甲基化对于构建和维持异染色质和常染色质区域有重要作用[13]。综合看来,赖氨酸的甲基化主要通过两种机制来调控蛋白质的功能,一方面能够与其他形式的PTMs相互影响,另一方面则通过影响蛋白质之间的相互作用来对蛋白质功能进行调控[14](图 1)。
 |
| 图 1 组蛋白甲基化 Figure 1 Histone methylation. |
| 图选项 |
在临床治疗中,赖氨酸特异性的组蛋白甲基转移酶和甲基水解酶,可以作为慢性疾病(如炎症和癌症)的药物靶点。PMTs的抑制剂可以与甲基供体SAM相互竞争其结合位点,如DOT1L的抑制剂已经进入癌症的临床试用[15]。
1.1.2 组蛋白精氨酸甲基化:组蛋白精氨酸的甲基化最早发现于20世纪70年代,其发生位点包括H3第2、8、17、26以及H4第3位的精氨酸等。目前已发现并鉴定出的蛋白质精氨酸甲基转移酶(Protein arginine methyltransferases,PRMTs)共有9种,依次命名为PRMT1–9。在PRMTs的催化下,甲基基团从甲硫氨酸转移至胍基氮原子,实现该氨基酸位点的精氨酸甲基化修饰。哺乳动物体内的精氨酸存在有三种类型的甲基化修饰,分别产生单甲基化精氨酸(Mono-methylation arginine,MMA)、非对称型双甲基化精氨酸(Asymmetric-dimethylation arginine,ADMA)、以及对称型双甲基化精氨酸(Symmetric-dimethylation arginine,SDMA),ADMA指2个甲基基团连接在同一胍基氮原子上,而SDMA则指2个甲基基团分别连接在胍基的2个氮原子上。这两种不同形式的精氨酸双甲基化的存在,暗示了其在生物进程中的不同作用,及其不同的识别机制。根据甲基化程度及类型的差异,除都可以产生MMA[16]之外,可以将PRMTs分为3种类型:Ⅰ型PRMTs,包括PRMT1、2、3、4、6、8,可催化产生ADMA[17];而Ⅱ型PRMTs,主要是PRMT5、9,可催化产生SDMA[18];Ⅲ型PRMT,即PRMT7,则专一性得催化产生MMA[16]。
精氨酸胍基基团含有5个氢键供体,能够结合于氢键受体并与之形成氢键[19]。甲基化修饰并不会改变精氨酸的正电荷,而是通过消除自身氢键供体来改变蛋白质之间连接的氢键结构,或是通过改变精氨酸庞大的侧链结构,来影响蛋白质相互之间的作用[20]。精氨酸的甲基化在多种生理进程中发挥重要作用,如信号转导、蛋白质定位、基因表达等的调控,DNA损伤修复以及RNA的代谢等。此外也有研究表明PRMTs参与肿瘤的发生及转移过程,PRMTs的过表达会触发参与肿瘤发生的相关底物的异常甲基化,进而促进肿瘤的恶化[21]。
PRMTs结构各不相同,但均含有1个高度保守的约310个氨基酸构成的中心结构域,除此之外,每个PRMT都含有1个独特的长短不一的N-端结构域。组蛋白甲基转移酶N-端结构域的缺失会损害其六聚体结构的稳定性并减弱其催化活性,类似地,PRMT5的N-端和C-端结构域的相互作用也会影响其同质复合物的形成。另外,PRMT3的N-端结构域包含有1个锌指模体,缺失该部分则会损坏蛋白质的活性并改变其底物特异性[13]。
PRMTs的亚细胞定位也暗示了其在多种细胞进程中的重要作用(图 2)。如PRMT1主要存在于细胞核[22],同时其在多种细胞的多种亚细胞结构中也都具有动力学活性,是细胞内最重要的MTs。由PRMTs催化的核小体组蛋白甲基化在细胞核介导的转录调控、染色质重塑以及基因表达的某些方面发挥重要作用。组蛋白精氨酸甲基化也能通过与多种PTMs共同作用来实现对特定蛋白的调控,改变染色质结构并调控转录进程[23]。
 |
| 图 2 大肠杆菌趋化性信号通路 Figure 2 Chemotaxis signaling pathway in E. coli. |
| 图选项 |
精氨酸的甲基化增加了相应氨基酸的大小,阻断了氢键的形成,但是不改变电荷,而电荷的维持相当重要,因为带正电荷的精氨酸通常能够结合蛋白或核酸。精氨酸的甲基化对蛋白本身的影响要弱于其他的PTM,只是调控某些蛋白而不是作为一种开关存在,因此常伴随有其他的PTMs存在,如乙酰化、磷酸化等[19]。
对比看来,赖氨酸和精氨酸的甲基化参与调控基因表达是通过以下两方面实现的,或是甲基化改变了染色质的结构,或是甲基化提供了转录调节因子的结合位点,影响的是蛋白质之间的相互作用[24]。添加于精氨酸残基的甲基基团更是通过消除氢键的形成并改变巨大的精氨酸侧链,进而对蛋白之间的相互作用产生影响[25]。
1.2 非组蛋白甲基化 随着蛋白组学技术尤其是质谱分析技术的发展,越来越多的非组蛋白甲基化已经被发现。非组蛋白靶位点多为转录因子、信号通路分子和肿瘤抑制蛋白。大多数的组蛋白MTs也可以同时参与非组蛋白的甲基化修饰。目前已鉴定到的存在甲基化与去甲基化修饰的非组蛋白包括参与细胞信号通路转录调控的成分,如受体激酶和效应蛋白、效应蛋白激活子和抑制子、转录调控因子等,肿瘤抑制因子p53,另外如RNA结合蛋白TAF10[26]等也可以发生甲基化修饰。下文将举例说明。
p53是人类肿瘤细胞中最易产生突变的肿瘤抑制因子,p53能够结合到特异的DNA序列,激活靶基因的转录,进而调控若干生物学进程,包括细胞周期、细胞凋亡以及DNA修复等。p53的赖氨酸甲基化主要发生于其C-端的赖氨酸位点,同时由于其本身可发生甲基化修饰的赖氨酸位点距离很近,上游位点的甲基化会对下游赖氨酸位点产生影响。p53能够发生多种形式的PTMs,其修饰的多样性在其调控蛋白稳定性、蛋白互作以及自身反式激活中发挥重要作用,增强自身稳定性及其在细胞核内的定位[27]。
p53第372位赖氨酸(K372) 是最早发现的p53甲基化修饰位点[28],该位点的甲基化修饰导致DNA损伤增加,但同时又能够稳定p53蛋白本身的结构并促进其对靶基因的激活。K372的甲基化能促进其K382位发生乙酰化,而后者能够激活p53转录调控活性,但同时K382自身的甲基化则抑制其转录调控活性[27]。K372的甲基化与K370的甲基化相互抑制,K370的甲基化由甲基转移酶KMT3C介导产生,而K372的甲基化则由KMT7介导产生,K372的甲基化能够直接阻断p53和KMT3C之间的作用导致K370甲基化受到抑制,而K370的甲基化虽然能够抑制K372的甲基化,却并不能够阻断其与KMT7之间的相互作用。尽管在培养条件下,K372的甲基化对于稳定p53以及调控其转录调节活性具有重要作用,但是也有研究发现,在体内实验中,K372并不影响p53相关的细胞进程[29]。
KMT7/SET7介导的p53甲基化是最早被发现的KMT介导的非组蛋白的赖氨酸甲基化,此后,越来越多的KMTs或KDMs被发现能够甲基化或去甲基化p53[28]。
一些RNA结合蛋白的甲基化参与RNA在核-质间的分布[13]。异质性核糖核蛋白(Heterogeneous nuclear ribonucleoproteins,hnRNPs)是最早发现的能够发生精氨酸甲基化的一类RNA结合蛋白,在基因转录过程中与RNA形成复合物。hnRNPs的形成表示mRNA处于未成熟的状态,蛋白质与mRNA前体的结合,既可以防止RNA前体发生折叠而影响RNA与蛋白质的相互作用,又能够促进RNA的有效剪接,并将成熟的RNA从细胞核内转运出去。而蛋白质结合在被剪切掉的内含子部分,介导后者的降解。hnRNP A1是其中一种hnRNPs,其在富含RGG序列的结构域有4个位点的精氨酸发生了甲基化,导致其对单链核苷酸结合能力的减弱,增加了对胰蛋白酶的敏感性。类似的,hnRNP A2的RGG结构域也能发生甲基化修饰,影响其在细胞内的分布。A2通常定位于细胞核内,而在敲除RGG结构域后则定位于细胞质内。同样的,在RGG结构域的精氨酸甲基化修饰被阻断之后,A2也定位于细胞质中,这表明RGG结构域的甲基化促进其在细胞核内的分布[26]。
2 原核生物蛋白质甲基化 与真核生物不同的是,原核生物的蛋白质甲基化知之甚少,已有的甲基化研究也仅集中于少数的微生物中(如大肠杆菌、沙门氏菌等)。原核生物的蛋白质甲基化据其功能差异性可以分为两种形式:发生于赖氨酸、精氨酸、谷氨酰胺的N-甲基化,以及发生于谷氨酸的侧链羧基的O-甲基化。N-甲基化涉及末端氨基酸上1到3个甲基基团的添加,而且甲基基团数量很大程度上取决于末端氨基酸的类型,赖氨酸最多可接受3个甲基基团而精氨酸最多接受2个甲基基团,而O-甲基化则仅参与羧酸盐侧链之间甲酯的形成[30]。
2.1 N-甲基化 OMPs (Outer membrane proteins)是表达于革兰阴性菌表面的一类与毒力相关的蛋白质,是细菌与外部环境信号交换的第一道防线,参与生物分子的转运以及细菌的感染和致病等过程。其赖氨酸甲基化能够影响细菌与宿主的相互作用,抑制其乙酰化、泛素化等修饰的发生。因此,此类蛋白的甲基化对细菌自身在巨噬细胞内的存活,以及对真核细胞的入侵都是具有重要意义的。
立克次氏体Omps参与其对宿主细胞的黏附、入侵及在宿主细胞内的分布等生理过程,并能够引发宿主的免疫应答。OmpB是立克次氏体内高度保守的一种OMP,是自主转运蛋白OMPs家族的一员。OmpB前体由1个信号肽、1个N-端效应结构域和1个C-端结构域三部分组成。OmpB的甲基化发生在其N-端结构域,并且在有毒株中OmpB的甲基化修饰更为普遍[31]。赖氨酸的甲基化与细菌毒力密切相关,Abeykoon等在立克次氏体内分离出两种OmpB特异性的MTs,即rRP789和rRP027–028,也是最早在革兰阴性菌中鉴定到的能够修饰外膜蛋白的MTs。二者都可以甲基化OmpB的多个氨基酸位点,但是rRP027–028优先催化蛋白质三甲基化的生成,而rRP0789则可以单甲基化、二甲基化和三甲基化[32]。
OmpB是强酸性蛋白,赖氨酸的甲基化增强了其局部电荷带电量,影响了蛋白质之间的静电作用和氢键连接,以及OmpB的功能。OmpB参与介导立克次氏体对内皮细胞的黏附和入侵,赖氨酸的甲基化会改变其与宿主细胞表面的相互作用,也将影响其在进入细胞之后与胞内蛋白的相互作用。OmpB的甲基化增强了自身的抗原性,刺激血清产生强烈的信号,某些病人来源的血清只能够识别甲基化的OmpB,甲基化的OmpB增加了其在免疫反应中的效价,可作为诊疗试剂进行诊断治疗。
2.2 O-甲基化 O-甲基化参与细菌的趋化性信号转导通路,是一种保守存在的甲基化修饰形式[33]。
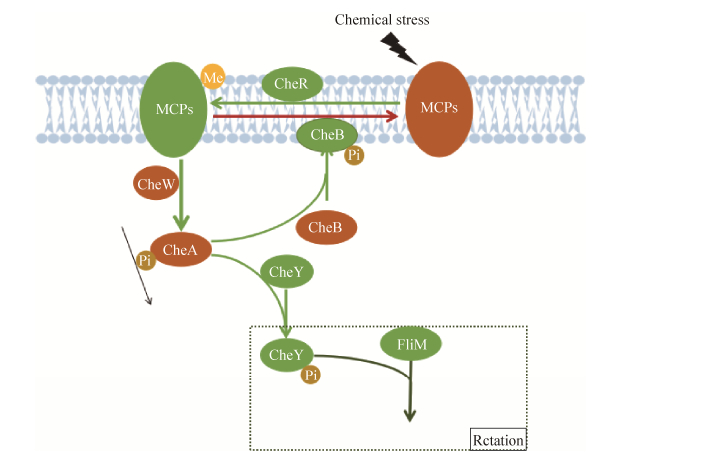
细菌鞭毛通过化学感应系统来改变细菌游走的方向。为了感应外界化学效应物的浓度,应答调节子CheY被组氨酸激酶磷酸化为CheY-P,后者随之结合鞭毛的转换蛋白FliM并引起不同的动力输出,或停止或减缓,在大肠杆菌中则会引起细菌运动方向由逆时针方向转换为顺时针方向[34]。
大肠杆菌的趋化性信号通路能够感知外界环境的变化,信号被专一性的跨膜趋化性蛋白MCPs (Methyl-accepting chemotaxis proteins)监测到,CheW协助MCPs与组氨酸自主磷酸激酶CheA结合。随后,CheA引起下游2个应答调节子的磷酸化,分别是CheY和CheB,CheB是一个MCP特异性的甲基酯酶和脱酰胺酶,其甲基酯酶活性能够水解MCPs的甲基基团[35]。磷酸化的CheB甲基酯酶活性增强,使得MCPs发生去甲基化,导致后者与CheA之间的相互作用减弱,CheA的自主磷酸化水平降低,不能诱导CheY磷酸化的发生,细菌运动方向将不发生改变。诱导剂浓度的增加也会抑制CheA的自身磷酸化,导致CheY磷酸化的减少,细菌运动方向将不发生改变。另外,CheB的磷酸化活性减弱,会使得组成型甲基转移酶CheR甲基化修饰MCPs,促进CheA的自身磷酸化并将磷酸基团转移至CheY。磷酸化后的CheY结合于FliM,进而引起回转方向的改变[33](图 2)。即使在细胞趋化因子持续存在的情况下,高度磷酸化的MCPs也能够刺激CheA的磷酸化,反过来又使CheA发生自身磷酸化,细菌运动方向得以恢复。受体信号状态的重置,使得细菌能够适应当前诱导剂的浓度并感知其连续的变化[30]。在此过程中,CheB和CheR协同作用调控MCPs的甲基化修饰状况,并与其上发生的其他翻译后修饰相互作用,进而帮助细菌有效地调整其运动方向,以更好地适应外界环境的变化。
趋化性以外的O-甲基化方面的研究还很有限。曹行军等在问号钩端螺旋体内的蛋白质组分析发现,在104个蛋白质上有共135个Glx (Glu/Gln)位点存在甲基化修饰[36]。Eshghi等也检测到,在问号钩端螺旋体的LipL32中11个位点存在多种多样的Glx-甲基化[37]。生物信息学分析也表明,蛋白质中包含Glx-甲基化的区域与其潜在的抗原表位相关,暗示了甲基化在免疫逃逸中的作用。
3 展望 沙门菌作为食源性疾病的主要致病菌,无论是在发达国家还是发展中国家都具有相当的危害。沙门菌全局性双组分调控系统(Two-component regulatory system,TCS) PhoP-PhoQ非常保守,其中调节蛋白PhoP调控了沙门菌基因组5%基因的表达,参与沙门菌的多种细胞活动,包括细菌的毒力、对弱酸环境和低浓度Mg2+环境的适应等。PhoP的第52位天冬氨酸(D52) 的磷酸化是激活PhoP蛋白的关键。我们课题组已有研究发现,PhoP的多个位点存在乙酰化修饰,其中第201位赖氨酸的乙酰化参与调控PhoP对下游基因启动子的结合,而第102位赖氨酸的乙酰化则与PhoP的磷酸化相互影响[38]。我们在对PhoP进行质谱分析后,发现其在多个氨基酸位点存在多种不同的PTM类型,包括甲基化修饰(未发表的数据)。在对甲基化可能位点进行定点突变后,突变菌株表现出与野生菌株的差异,因此我们推测PhoP的甲基化修饰或许参与调控PhoP的活性,进而影响沙门菌的毒力及其在细胞内复制与生存。但其具体的工作机制仍有待进一步深入探究,揭示甲基化修饰与沙门菌毒力之间的关系,为认识沙门菌的致病机制提供新的思路。
甲基化修饰能够发生在不同的氨基酸位点,或是在同一个氨基酸位点产生不止一个甲基化修饰。因此对研究蛋白质功能来说,确定其甲基化位点是研究其功能的首要任务。通过各种分子生物学实验技术,并结合蛋白质组学等方法,了解蛋白质甲基化的功能,并进一步深入探究其与不同PTMs之间的相互影响与作用,进而了解其在细胞信号通路、动物发展及疾病治疗中的作用,将能够帮助我们更好地了解原核生物生命活动的规律,而通过对关键环节进行阻断干扰也将能够更好地为对抗相关疾病提供有力的科学依据和措施。关于原核生物蛋白质甲基化在细菌生物进程中发挥何种作用,尚需要我们更加深入地进行探索研究。此外,翻译后修饰种类的多样性也暗示着其对蛋白质生物功能调控的复杂性,多种修饰类型的相互作用与影响共同调控了细菌的生理过程,尤其是其在细菌致病性中发挥关键作用。
References
| [1] | Ambler RP, Rees MW. ε-N-Methyl-lysine in bacterial flagellar protein. Nature, 1959, 184(4679): 56-57. |
| [2] | Biggar KK, Li SSC. Non-histone protein methylation as a regulator of cellular signalling and function. Nature Reviews Molecular Cell Biology, 2015, 16(1): 5-17. |
| [3] | Greer EL, Shi Y. Histone methylation:a dynamic mark in health, disease and inheritance. Nature Reviews Genetics, 2012, 13(5): 343-357. DOI:10.1038/nrg3173 |
| [4] | Kaniskan Hü, Szewczyk MM, Yu ZT, Eram MS, Yang XB, Schmidt K, Luo X, Dai M, He F, Zang I, Lin Y, Kennedy S, Li FL, Dobrovetsky E, Dong AP, Smil D, Min S J, Landon M, Lin-Jones J, Huang XP, Roth BL, Schapira M, Atadja P, Barsyte-Lovejoy D, Arrowsmith CH, Brown PJ, Zhao KH, Jin J, Vedadi M. A potent, selective and cell-active allosteric inhibitor of protein arginine methyltransferase 3(PRMT3). Angewandte Chemie:International Edition, 2015, 54(17): 5166-5170. DOI:10.1002/anie.v54.17 |
| [5] | Murray K. The occurrence of iε-N-methyl lysine in histones. Biochemistry, 1964, 3(1): 10-15. DOI:10.1021/bi00889a003 |
| [6] | Kim S, Paik WK. Studies on the origin of epsilon-N-methyl-L-lysine in protein. Journal of Biological Chemistry, 1965, 240(12): 4629-4634. |
| [7] | Shi YJ, Lan F, Matson C, Mulligan P, Whetstine JR, Cole PA, Casero RA, Shi Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell, 2004, 119(7): 941-953. DOI:10.1016/j.cell.2004.12.012 |
| [8] | Rea S, Eisenhaber F, O'Carroll D, Strahl BD, Sun ZW, Schmid M, Opravil S, Mechtler K, Ponting CP, Allis CD, Jenuwein T. Regulation of chromatin structure by site-specific histone H3 methyltransferases. Nature, 2000, 406(6796): 593-599. DOI:10.1038/35020506 |
| [9] | Alam H, Gu BN, Lee MG. Histone methylation modifiers in cellular signaling pathways. Cellular and Molecular Life Sciences, 2015, 72(23): 4577-4592. DOI:10.1007/s00018-015-2023-y |
| [10] | Kim W, Choi M, Kim JE. The histone methyltransferase Dot1/DOT1L as a critical regulator of the cell cycle. Cell Cycle, 2014, 13(5): 726-738. DOI:10.4161/cc.28104 |
| [11] | Smith BC, Denu JM. Chemical mechanisms of histone lysine and arginine modifications. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2009, 1789(1): 45-57. DOI:10.1016/j.bbagrm.2008.06.005 |
| [12] | Martin C, Zhang Y. The diverse functions of histone lysine methylation. Nature Reviews Molecular Cell Biology, 2005, 6(11): 838-849. DOI:10.1038/nrm1761 |
| [13] | Lee DY, Teyssier C, Strahl BD, Stallcup MR. Role of protein methylation in regulation of transcription. Endocrine Reviews, 2005, 26(2): 147-170. DOI:10.1210/er.2004-0008 |
| [14] | Lanouette S, Mongeon V, Figeys D, Couture JF. The functional diversity of protein lysine methylation. Molecular Systems Biology, 2014, 10(4): 724. DOI:10.1002/msb.134974 |
| [15] | Kaniskan Hü, Konze KD, Jin J. Selective inhibitors of protein methyltransferases. Journal of Medicinal Chemistry, 2015, 58(4): 1596-1629. DOI:10.1021/jm501234a |
| [16] | Zurita-Lopez CI, Sandberg T, Kelly R, Clarke SG. Human protein arginine methyltransferase 7(PRMT7) is a type Ⅲ enzyme forming ω-NG-monomethylated arginine residues. Journal of Biological Chemistry, 2012, 287(11): 7859-7870. DOI:10.1074/jbc.M111.336271 |
| [17] | Bedford MT, Richard S. Arginine methylation:an emerging regulatorof protein function. Molecular Cell, 2005, 18(3): 263-272. DOI:10.1016/j.molcel.2005.04.003 |
| [18] | Branscombe TL, Frankel A, Lee JH, Cook JR, Yang ZH, Pestka S, Clarke S. PRMT5(Janus kinase-binding protein 1) catalyzes the formation of symmetric dimethylarginine residues in proteins. Journal of Biological Chemistry, 2001, 276(35): 32971-32976. DOI:10.1074/jbc.M105412200 |
| [19] | Bedford MT, Clarke SG. Protein arginine methylation in mammals:who, what, and why. Molecular Cell, 2009, 33(1): 1-13. DOI:10.1016/j.molcel.2008.12.013 |
| [20] | Tripsianes K, Madl T, Machyna M, Fessas D, Englbrecht C, Fischer U, Neugebauer KM, Sattler M. Structural basis for dimethylarginine recognition by the Tudor domains of human SMN and SPF30 proteins. Nature Structural & Molecular Biology, 2011, 18(12): 1414-1420. |
| [21] | Yang YZ, Bedford MT. Protein arginine methyltransferases and cancer. Nature Reviews Cancer, 2013, 13(1): 37-50. |
| [22] | Frankel A, Yadav N, Lee J, Branscombe TL, Clarke S, Bedford MT. The novel human protein arginine N-methyltransferase PRMT6 is a nuclear enzyme displaying unique substrate specificity. Journal of Biological Chemistry, 2002, 277(5): 3537-3543. DOI:10.1074/jbc.M108786200 |
| [23] | Pahlich S, Zakaryan RP, Gehring H. Protein arginine methylation:cellular functions and methods of analysis. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2006, 1764(12): 1890-1903. DOI:10.1016/j.bbapap.2006.08.008 |
| [24] | Sprung R, Chen Y, Zhang K, Cheng DM, Zhang T, Peng JM, Zhao YM. Identification and validation of eukaryotic aspartate and glutamate methylation in proteins. Journal of Proteome Research, 2008, 7(3): 1001-1006. DOI:10.1021/pr0705338 |
| [25] | Ng SS, Yue WW, Oppermann U, Klose RJ. Dynamic protein methylation in chromatin biology. Cellular and Molecular Life Sciences, 2009, 66(3): 407-422. DOI:10.1007/s00018-008-8303-z |
| [26] | Blackwell E, Ceman S. Arginine methylation of RNA-binding proteins regulates cell function and differentiation. Molecular Reproduction and Development, 2012, 79(3): 163-175. DOI:10.1002/mrd.v79.3 |
| [27] | Carr SM, Roworth AP, Chan C, Thangue NBL. Post-translational control of transcription factors:methylation ranks highly. The FEBS Journal, 2015, 282(23): 4450-4465. DOI:10.1111/febs.13524 |
| [28] | Chuikov S, Kurash JK, Wilson JR, Xiao B, Justin N, Ivanov GS, McKinney K, Tempst P, Prives C, Gamblin SJ, Barlev NA, Reinberg D. Regulation of p53 activity through lysine methylation. Nature, 2004, 432(7015): 353-360. DOI:10.1038/nature03117 |
| [29] | Huang J, Berger SL. The emerging field of dynamic lysine methylation of non-histone proteins. Current Opinion in Genetics & Development, 2008, 18(2): 152-158. |
| [30] | Cain JA, Solis N, Cordwell SJ. Beyond gene expression:the impact of protein post-translational modifications in bacteria. Journal of Proteomics, 2014, 97: 265-286. DOI:10.1016/j.jprot.2013.08.012 |
| [31] | Abeykoon AH, Wang GH, Chao CC, Chock PB, Gucek M, Ching WM, Yang DCH. Multimethylation of Rickettsia OmpB catalyzed by lysine methyltransferases. Journal of Biological Chemistry, 2014, 289(11): 7691-7701. DOI:10.1074/jbc.M113.535567 |
| [32] | Abeykoon AH, Chao CC, Wang GH, Gucek M, Yang DCH, Ching WM. Two protein lysine methyltransferases methylate outer membrane protein B from Rickettsia. Journal of Bacteriology, 2012, 194(23): 6410-6418. DOI:10.1128/JB.01379-12 |
| [33] | Brown MT, Delalez NJ, Armitage JP. Protein dynamics and mechanisms controlling the rotational behaviour of the bacterial flagellar motor. Current Opinion in Microbiology, 2011, 14(6): 734-740. DOI:10.1016/j.mib.2011.09.009 |
| [34] | Porter SL, Wadhams GH, Armitage JP. Signal processing in complex chemotaxis pathways. Nature Reviews Microbiology, 2011, 9(3): 153-165. DOI:10.1038/nrmicro2505 |
| [35] | Parkinson JS, Hazelbauer GL, Falke JJ. Signaling and sensory adaptation in Escherichia coli chemoreceptors:2015 update. Trends in Microbiology, 2015, 23(5): 257-266. DOI:10.1016/j.tim.2015.03.003 |
| [36] | Cao XJ, Dai J, Hao X, Nie S, Chang X, Hu BY, Sheng QH, Wang LS, Ning ZB, Li YX, Guo XK, Zhao GP, Zeng R. High-coverage proteome analysis reveals the first insight of protein modification systems in the pathogenic spirochete Leptospira interrogans. Cell Research, 2010, 20(2): 197-210. DOI:10.1038/cr.2009.127 |
| [37] | Eshghi A, Pinne M, Haake DA, Zuerner RL, Frank A, Cameron CE. Methylation and in vivo expression of the surface-exposed Leptospira interrogans outer-membrane protein OmpL32. Microbiology, 2012, 158(Pt 3): 622-635. |
| [38] | Ren J, Sang Y, Tan YC, Tao J, Ni JJ, Liu ST, Fan X, Zhao W, Lu J, Wu WJ, Yao YF. Acetylation of lysine 201 inhibits the DNA-Binding ability of PhoP to regulate Salmonella virulence. PLoS Pathogens, 2016, 12(3): e1005458. DOI:10.1371/journal.ppat.1005458 |
