彭政, 丁俊美, 杨云娟, 李俊俊, 慕跃林, 黄遵锡


云南师范大学生命科学学院, 生物能源持续开发利用教育部工程中心, 云南省生物质能与环境生物技术 重点实验室, 云南省酶工程技术研究中心, 云南 昆明 650500
收稿日期:2016-04-29;修回日期:2016-05-16;网络出版日期:2016-06-03
*通信作者:Tel: +86-871-5920830; Fax: +86-871-5920952,E-mail:huangzunxi@163.com.
摘要: [目的] 克隆芽孢杆菌HJ14的酯酶基因EstZ1并利用大肠杆菌表达得到相应的酯酶,分析重组酯酶的酶学性质和对邻苯二甲酸二乙酯(Diethyl phthalate,DEP)的降解。[方法] 特异性扩增酯酶基因EstZ1并对其全长测序,分析其氨基酸序列。利用pEASY-E2表达系统将EstZ1转化到Escherichia coli BL21(DE3)中完成异源表达。根据组氨酸标签纯化EstZ1,研究其酶学性质并利用HPLC和LC/MS检测系统定性分析其对DEP的降解。[结果] EstZ1全长903 bp,编码300个氨基酸残基,蛋白分子量33.84 kDa。EstZ1氨基酸序列分析结果显示,与NCBI数据库收录的HSL-like家族酯酶相似度最高可达到98%。酶学性质分析结果显示,EstZ1可水解碳链长度较短的p-NP底物,最适底物为p-NPC4(p-NP butyrate)。EstZ1的最适pH和最适温度分别为9.0和50℃,并且在pH 7.0–9.5和40–70℃范围内保持50%以上的酶活,为耐热碱性酯酶。EstZ1对多数金属离子和化学试剂保有良好的抗性。EstZ1可将DEP水解生成相应的单酯和醇。[结论] 本文报道了Bacillus sp.HJ14来源的酯酶基因并对其在大肠杆菌中表达获得的重组酶的酶学性质进行研究,EstZ1具有良好的碱性pH耐受性和热稳定性,能够部分降解DEP,本研究对邻苯二甲酸酯类的生物降解有一定的参考意义。
关键词: 耐热酯酶 异源表达 酶学性质 DEP降解
Cloning, heterologous expression and characterization of a thermostable esterase from Bacillus sp. HJ14 for diethyl-phthalate degradation
Peng Zheng, Ding Junmei, Yang Yunjuan, Li Junjun, Mu Yuelin, Huang Zunxi


College of Life Sciences, Yunnan Normal University, Engineering Research Center of Sustainable Development and Utilization of Biomass Energy, Ministry of Education, Key Laboratory of Yunnan for Biomass Energy and Biotechnology of Environment, Key Laboratory of Enzyme Engineering, Kunming 650500, Yunnan Province China
Received 29 April 2016; Revised 16 May 2016; Published online 03 June 2016
*Corresponding author: Tel: +86-871-5920830; Fax: +86-871-5920952. E-mail:huangzunxi@163.com.
Abstract: [Objective] A thermostable esterase EstZ1 from Bacillus sp. HJ14 able to degrade diethyl-phthalate (DEP) was heterologously expressed in Escherichia coli BL21(DE3) and characterized.[Methods] Full-length EstZ1 was obtained based on specific amplification and genome sequencing, and amino acid sequence of EstZ1 was analyzed. EstZ1 was expressed in Escherichia coli BL21(DE3) using the pEASY-E2 expression system. EstZ1 was purified to electrophoretic homogeneity by Ni2+-NTA metal chelating affinity chromatography, and the enzyme was characterized. The degradation products from DEP were detected by high-pressure liquid chromatography and electrospray ionization mass spectrometry.[Results] The 903 bp full-length EstZ1 encoded 300 amino acid residues (EstZ1:33.84 kDa). EstZ1 showed the highest identity of 98% with hormone-sensitive lipase (HSL)-like family in NCBI databases. The optimal temperature and pH was 50℃ and 9.0, respectively, with p-NP butyrate as the best substrate. Meanwhile, it was stable between 40 and 70℃, pH 7.0 to 9.5. Most of metal ions, chemical agents had little impact. DEP could partially be degraded by EstZ1 to its corresponding monoalkyl and alcohol.[Conclusion] Our findings may serve as reference for phthalate esters degradation.
Key words: thermostable esterase heterologous expression enzyme characterizations DEP degradation
酯酶(Esterase,EC 3.1.1.x)是指能够催化酯键水解的一类水解酶。根据其作用的底物类型可以把酯酶分为脂肪酶(Triacylglycerol lipase,E.C.3.1.1.3)、羧酸酯酶(Carboxylesterase,EC 3.1.1.1)和芳香酯酶(Arylesterase,EC 3.1.1.2)等[1]。习惯意义上的酯酶是除脂肪酶以外能够水解酯键的水解酶。酯酶的空间结构属于α/β水解酶折叠,包含了6个α螺旋(αA-αF)和8个不同的β折叠(β1-β8),Ser通常是其活性位点,位于保守的五肽结构域内(GXSXG)[2]。在酯酶的分子结构中还存在二硫键,由4-5个半胱氨酸形成,对于保持活性中心的稳定具有重要作用。对于分泌型的羧酸酯酶C-末端还含有由His-X-Glu-Leu (HXEL)组成的信号序列[3]。
酯酶广泛存在动物、植物和微生物体内,其中微生物来源的酯酶分布最为广泛,包括细菌、真菌和放线菌的多个属种[4-6]。特别是来自极端环境的微生物,如嗜热、嗜酸碱和嗜盐等微生物,它们在长期进化过程中形成了对极端环境的良好抗性,这些嗜极微生物蕴含了丰富的嗜极酶,是发掘酯酶的良好材料。酯酶因其耐热性在食品工业、洗涤行业和废弃物处理等方面的应用潜力收到越来越多的关注[7]。
邻苯二甲酸酯类化合物(Phthalate esters,PAEs)作为改变塑料柔韧性和可塑性的添加剂在增塑剂市场中占主导地位,PAEs被世界卫生组织(WTO)认定为是一种内分泌干扰物(Endocrine-disrupting chemicals,EDCs),长期接触会对动物生殖系统造成严重的危害。生物降解是影响邻苯二甲酸酯类(Phthalate esters,PAEs)在环境中的行为和归宿的重要途径[8]。在以往的研究中,对PAEs生物降解的过程、机理以及微生物的代谢特性有了一些了解,许多微生物对PAEs及其代谢的中间产物有降解能力[9-11]。Mathur和Rouatt将DEHP和DOP作为唯一碳源分离出Marcescens、Serratia等菌,其初级降解产物为邻苯二甲酸和相应的醇,这些初级降解产物可被其他微生物继续降解,最后转化成CO2和水[12]。自然环境中邻苯二甲酸酯类降解菌普遍存在,在已经报道的研究中,PAEs降解菌的来源有土壤[13-14]、水体沉积物[15-16]、活性污泥[17-18]和固体废弃物[19-20]等。在这些研究中关于酯酶直接作用来降解PAEs,特别是具有较好的热稳定性并且对PAEs有水解活性的酯酶很少,然而高温环境下PAEs污染的情况却很普遍,例如农业生产使用塑料薄膜覆盖促进作物生长,这样的密闭环境在夏季可达到50 ℃甚至更高;还有在垃圾处理时常用到堆肥的方法,堆肥的过程通常会达到很高的温度才能发生预想的生化反应[21]。所以研究和开发耐高温并且对PAEs有水解活性的酯酶具有重要的实际意义。
随着分子生物学技术的不断革新,建立高通量筛选模型,对目标菌株进行全基因组测序,通过定向进化技术对酶的特性进行改造,获得高效表达的基因工程菌,已成为酶制剂开发应用的重要手段[22-23]。本研究菌株Bacillus sp. HJ14筛选自盐碱土壤,从中克隆得到HSL-like家族酯酶基因EstZ1,成功利用大肠杆菌表达并研究其酶学性质,旨在挖掘极端环境下具有应用潜力的酯酶。
1 材料和方法 1.1 材料 1.1.1 实验菌株和载体:基因来源菌株Bacillus sp. HJ14为本实验室筛选;表达宿主菌E. coli BL21 (DE3)购于Novagen公司;载体和克隆宿主菌pEASY-E2载体和Trans-I感受态细胞购自北京全式金公司。
1.1.2 主要试剂:ExTaqDNA聚合酶、dNTPs、DNA Marker均购自TaKaRa公司;蛋白Marker购自Fermentas公司;对硝基苯酚乙酸酯(p-NPC2)、对硝基苯酚丁酸酯(p-NPC4)、对硝基苯酚己酸酯(p-NPC6)、对硝基苯酚辛酸酯(p-NPC8)、对硝基苯酚癸酸酯(p-NPC10)、对硝基苯酚月桂酸酯(p-NPC12)购自Sigma公司;Nickel-NTA agarose购自Qiagen公司;邻苯二甲酸二乙酯(AR)和邻苯二甲酸二乙酯标品(99%)购自百灵威公司;正己烷、乙酸乙酯、甲醇等有机溶剂均为色谱纯,购自Merck LiChrosolv公司;基因组和质粒提取试剂盒购自Omega公司。
1.1.3 实验仪器:PCR扩增仪(Bio-Rad);离心机(Eppendorf);凝胶成像仪(Bio-Rad);核酸电泳仪(Bio-Rad);蛋白电泳仪(Amersham Bioscience);酶标仪(Bio-Rad);超声波细胞破碎仪(宁波新芝科技有限公司);移液器(Eppendorf);气质联用仪(GC/MS)(Agilent GC7890A/MS5975C);高效液相色谱仪(HPLC)(Agilent 1100);超高效液相色谱串联三重四级杆质谱仪(UPLC-TQ-MS)(Waters AcQuity,Waters Xevo TQ-S)。
1.2 序列分析 分别使用BLASTn和BLASTp在线分析程序(http://blast.ncbi.nlm.nih.gov/Blast/)分析DNA和蛋白序列;信号肽预测使用软件SignalP 4.0[24];进化树构建运用邻接法,使用分子进化遗传分析软件MEGA(version 6.0)[25];多序列比对使用软件ESPript;蛋白结构预测分析使用软件VADAR[26];二硫键预测使用软件DIANNA1.1[27]。
1.3 EstZ1的克隆和转化 根据Bacillus sp. HJ14全基因组测序及基因片段注释结果,针对EstZ1设计引物并由华大基因公司合成(Forward: 5′-AACTTAGAAGAACAAATC AAAATC GC-3′;Reverse: 5′-AGGTCTCTGTT CAAACGC-3′)。以Bacillus sp. HJ14基因组为模板扩增EstZ1基因片段,扩增条件为:94 ℃ 90 s,94 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,28个循环;94 ℃ 30 s,44 ℃ 30 s,72 ℃ 100 s,7个循环;72 ℃ 10 min。
将扩增产物连接到pEASY-E2载体上并转化克隆宿主菌Trans-I (方法可见说明书),过夜培养并提取阳性克隆子混合质粒(方法可见说明书),得到重组质粒pEASY-E2-EstZ1。将重组质粒转化表达宿主BL21(DE3),培养后测序验证。
1.4 目标蛋白的表达与纯化 将含有重组质粒的E. coli BL21 (DE3)菌株和只含有pEASY-E2空质粒的E. coli BL21 (DE3) 菌株,以1/1000的接种量接种于含Amp (100 μg/mL)的新鲜LB液体培养基中,在37 ℃的恒温摇床中培养16 h,转速180 r/min;再以1/100的接种量将活化的新鲜菌液转接到含Amp (100 μg/mL)的新鲜LB液体培养基中,快速振荡2-3 h,OD600大约在0.6时加入终浓度为0.4 mmol/L的IPTG,继续在20 ℃和140 r/min转速下培养约20 h诱导表达。收集菌体并用适量的50 mmol/L McIlvaine 缓冲液(pH 7.0)悬浮菌体后,在冰水混合物水浴下超声波破碎菌体。将破碎的菌体于4 ℃、12000 r/min条件下高速离心得到大肠杆菌胞内表达的粗酶。利用Nickel-NTA Agarose纯化粗酶得到目的蛋白,并对纯化蛋白验证(SDS-PAGE)和定量(Bradford法)。
1.5 EstZ1酶学特性测定 纯化重组酶EstZ1采用对硝基苯酚法(p-nitrophenol)进行活性测定:总体系为500 μL,含有420 μL pH 7.5的50 mmol/L缓冲液,30 μL 10 mmol/L底物p-NPC4和50 μL适当稀释倍数的酯酶EstZ1。底物和缓冲液在反应温度下预热2 min后加入酶液反应5 min,最后加入0.1 mmol/L的Na2CO3终止反应,在405 nm波长下测定OD值。使用的缓冲液为:50 mmol/L McIlvaine 缓冲液(pH 5.0-8.0)、50 mmol/L Tris-HCl(pH 7.0-9.0)、50 mmol/L硼砂-NaOH(pH 9.0-11.0)。酶活单位定义为一定条件下每分钟水解底物p-NPC4生成1 μmoL对硝基苯酚所需要的酶量。
EstZ1的底物特异性:将纯化酶在37 ℃、pH 7.0的缓冲液中和50 mmol/L不同碳链长度的p-NP底物进行酶促反应;EstZ1的最适pH:将适当稀释的纯化酶EstZ1在37 ℃下和pH 2.0-11.0范围内进行酶促反应;EstZ1的最适温度:将适当稀释的纯化酶EstZ1在pH 9.0下,于0-95 ℃范围内进行酶促反应;EstZ1的pH稳定性:将适当稀释的纯化酶EstZ1加入pH 2.0-11.0的缓冲液中,在37 ℃下耐受1 h,然后在pH 9.0和37 ℃下进行酶促反应,以未处理的稀释酶液作为100%;EstZ1的温度稳定性:将适当稀释的纯化酶EstZ1分别在37、60和80 ℃耐受1 h,耐受酶液在pH 9.0和37 ℃下进行酶促反应,同样以未处理的稀释酶液作为100%;EstZ1对金属离子和有机试剂的抗性:在反应体系中加入终浓度为1 mmol/L的Ag+、K+、Ca2+、K+、Fe2+、Zn2+、Mg2+、Na+、Mn2+、Fe3+、Pb2+、Ni2+、Co2+、Cu2+、Hg2+、Al3+、EDTA、SDS、β- mercaptoethanol和终浓度为1% (V/V)的Triton X-100、Tween-80、Methylalcohol、Alcohol和Acetone在pH 9.0和37 ℃下进行酶促反应,以不加任何物质的反应体系作为100%。
1.6 EstZ1对邻苯二甲酸二乙酯(DEP)的降解产物分析 在10 mmol/L pH 9.0的Tris-HCl缓冲液中加入终浓度为50 mg/mL的DEP和纯化的重组酯酶EstZ1 5 U,总反应体积1 mL,以不加酶相同处理作为对照组。在50 ℃的水浴锅中反应2 h,用100 μL 1 mol/L HCl终止反应,用等体积的乙酸乙酯萃取,回收下层的乙酸乙酯蒸发干净,用等体积的甲醇重新定容,产物样品用HPLC和UPLC-MS分析。HPLC检测条件:色谱柱选用Agilent zorbax SB-Aq (4.6 mm× 250 mm,5 μm),流动相:乙腈,H3PO4水溶液 (0.1%),进样量5 μL,柱温30 ℃,流速1 mL/min,检测器 (DAD),检测波长230 nm。UPLC检测条件:色谱柱选用Agilent Zorbax SB-C18 (4.6 mm×250 mm,5 μm),流动相:乙腈,CH3COOH水溶液 (0.1%),进样量2 μL,柱温30 ℃,流速0.6 mL/min检测器(PDA),检测波长210 nm。
ESI-MS采用正负离子模式检测,检测条件同上所述。
2 结果和分析 2.1 菌株和基因登录号 菌株Bacillus sp. HJ14和EstZ1基因在GenBank数据库的登录号分别为KC787602和KU680808。
2.2 基因克隆和序列分析 利用PCR扩增得到酯酶基因(EstZ1)全长,共903 bp,编码300个氨基酸残基,蛋白理论分子量33.84 kDa,不含信号肽。系统发育进化树分析结果(图 1)显示EstZ1属于HSL-like酯酶家族。与NCBI数据库收录的酯酶基因进行多序列比对结果(图 2)显示EstZ1预测含有催化三残基分别是Ser145、Asp240和His270。将EstZ1与已发表的耐热酯酶EstS1进行蛋白二级和三级结构的比较发现在可能影响酯酶热稳定性的因素中,EstZ1的脯氨酸残基和甘氨酸残基数与EstS1相近,α-螺旋含量高于EatS1,此外EstZ1还含有EstS1没有的二硫键,这些都可以从一定程度上证明EstZ1的耐热属性,但只是利用生物信息学进行预测的结果,今后还需要利用实验进行验证(表 1)。
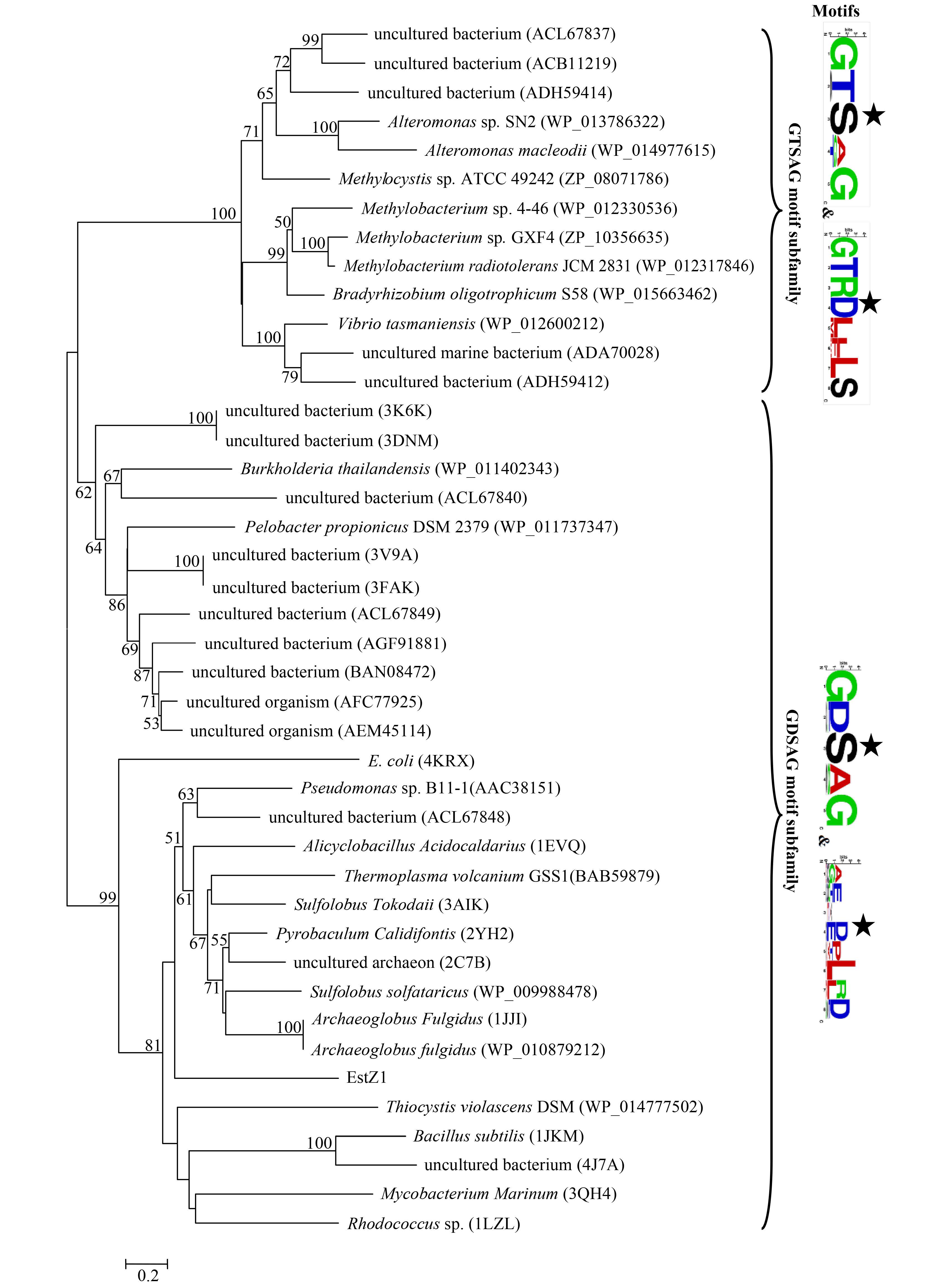 |
| 图 1. EstZ1的系统进化树分析 Figure 1. Phylogenetic analysis of EstZ1. The tree was built by the Neighbor-Joining method with a JTT matrix-based model. Bootstrap analysis of 1000 replicates was conducted, and values above 50% are shown. GenBank accession numbers or protein IDs are shown at the end of each name. The number on branch point means similarity. Scaleplate could be considered as the difference between two sequences |
| 图选项 |
 |
| 图 2. EstZ1的多序列比对分析 Figure 2. Multiple sequence alignment of EstZ1. Sequences retrieved from the NCBI server were aligned in CLUSTAL W and rendered using ESPript output. Sequences are grouped according to similarity. EstZ1 from Bacillus sp.HJ14; 3AIK from Ferroplasma; KPN14852 from Bacillus sp. NH7I_1; AAG09918 from Bacillus pumilus; KOO40129 from Bacillus okuhidensis; KOS02876 from Paenibacillus polymyxa; five-pointed star indicate amino acid residues belonging to the catalytic triad |
| 图选项 |
表 1. 影响EstZ1耐热性的可能因素 Table 1. Possible factors contributing to protein thermotolerance
| Factor | Value | |
| EstZ1 | EstS1 | |
| helix residues% | 34 | 31.91 |
| Pro residues% | 8.67 | 9.5 |
| Gly residues% | 5.67 | 7.2 |
| No. of Cys residues/disulfide bonds | 4/1 | 2/0 |
表选项
2.3 蛋白表达和纯化 重组质粒pEASY-E2-EstZ1成功转化表达载体大肠杆菌BL21(DE3)并表达,为胞内表达,无胞外酶活性。细胞破碎得到EstZ1粗酶,经Nickel-NTA树脂纯化,得到纯化蛋白,纯化蛋白浓度为2.68 mg/mL。纯化蛋白进行12%的SDS-PAGE分析,结果显示目的蛋白大小与理论分子量大小一致(图 3)。
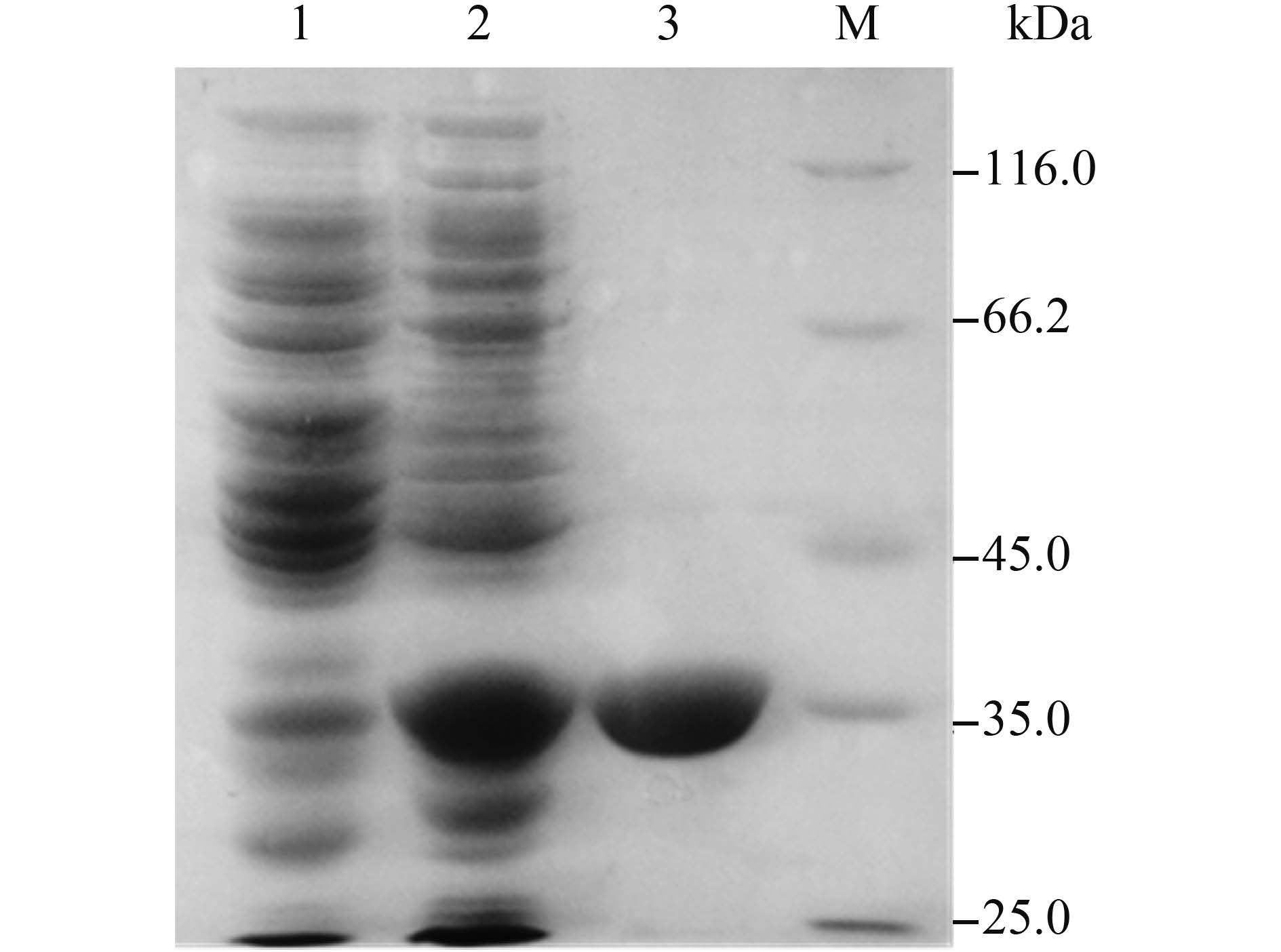 |
| 图 3. 纯化酶EstZ1的SDS-PAGE分析 Figure 3. Purification of EstZ1. Lane 1: total proteins of E. coli BL21(DE3) with blank vector pEASY-E2; Lane 2: total proteins of E. coli BL21(DE3) harboring the recombinant plasmid pEASY-E2-EstZ1; Lane 3: purified EstZ1; Lane M: protein molecular weight marker |
| 图选项 |
2.4 EstZ1的酶学特性 2.4.1 底物特异性及动力学参数:Km米氏常数,反应了酶与底物的亲和能力,Km值越小表示与底物亲和能力越强,Kcat为反应常数,Kcat/Km值是表征酶对底物的催化效率,比值越高说明酶对该底物的选择性越强。表 2结果表明EstZ1的最适底物是p-NPC4,Km和Kcat/Km值分别为0.14 mmol/L和161.07/s。
表 2. EstZ1的动力学参数 Table 2. Kinetic parameters of recombinant EstZ1
| Substrates | Km/ (mmol/L) | Vmax/ (U/mg) | Kcat/ (/s) | Kcat/Km [L/(s·mmol)] |
| p-NPC2 | 0.62±0.16 | 34.76±4.18 | 61.63±7.41 | 99.40 |
| p-NPC4 | 0.14±0.03 | 12.72±0.64 | 22.55±1.14 | 161.07 |
| p-NPC6 | 0.20±0.04 | 4.12±0.28 | 7.30±0.49 | 36.50 |
| p-NPC8 | 0.28±0.10 | 3.07±0.40 | 5.45±0.71 | 19.46 |
| p-NPC10 | 0.50±0.47 | 1.70±0.69 | 3.02±1.23 | 6.04 |
表选项
2.4.2 最适pH和pH稳定性:EstZ1的最适pH为9.0(图 4-A),在pH 7.0-9.0处理1 h酶活仍保持60%以上(图 4-B),在pH 9.0时的100%绝对活力值为(17.94±0.38) U/mL。
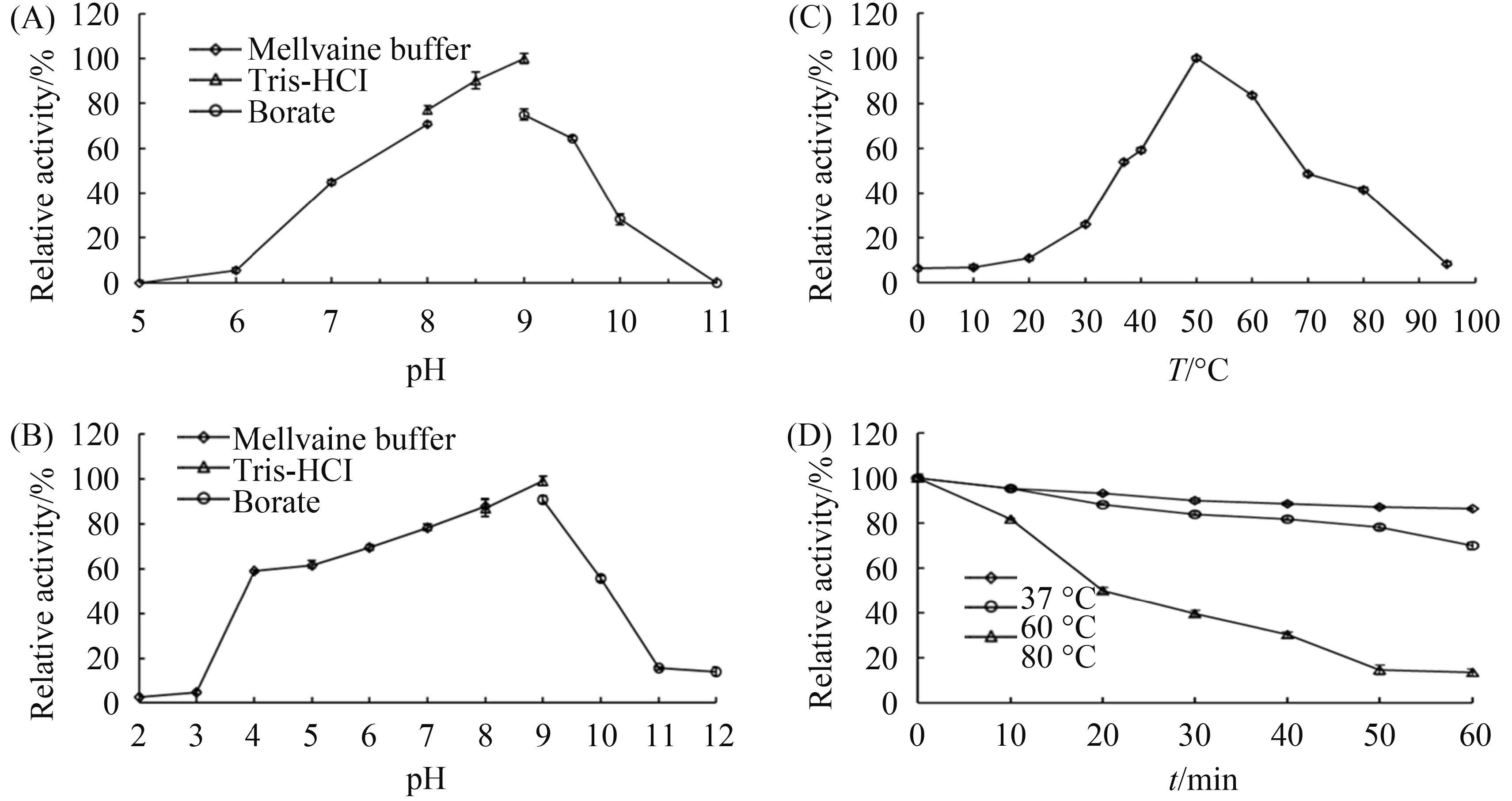 |
| 图 4. 纯化酶EstZ1的酶学特性 Figure 4. The enzymatic characteristics of purified EstZ1. A: Effect of pH on EstZ1 activity. Reactions were performed in different buffers with the concentration of 50 mmol/L range from 5.0–11.0 (McIlvaine buffer for pH 5.0–8.0, Tris-HCl buffer for pH 7.0–9.0 and Borate buffer for pH 9.0–11.0) at 37 ℃, respectively. B: pH stability of EstZ1. The enzyme was incubated for 1 h in buffers of various pH values (pH 2.0–12.0) at 37 ℃. C: Effect of temperature on EstZ1 activity was determined using p-NPC4 as substrate in the temperature range of 0 ℃ to 95 ℃ in Tris-HCl buffer (50 mmol/L, pH 9.0). D: Thermostability of EstZ1. The enzyme was incubated for 1 h at 37 ℃, 60℃ and 80 ℃ |
| 图选项 |
2.4.3 最适温度和温度稳定性:EstZ1的最适温度为50 ℃ (图 4-C),在40-70 ℃酶活仍保持50%以上。EstZ1在37 ℃、60 ℃和80 ℃处理1 h,80 ℃下半衰期为20 min,其余温度下1 h后活性保持80%以上(图 4-D)。
2.4.4 金属离子和有机试剂抗性:如表 3显示,Fe3+,Al3+,EDTA,TritonX-100对EstZ1的活性有部分促进(大约11.1%-46.5%);Na+,Pb2+,Mn2+,Ni2+,Cu2+,methyl alcohol,acetone对EstZ1的活性影响较小(保持在88.0%-105.5%);Co2+,Hg2+,SDS,β-mercaptoethanol,Tween-80,Ethyl alcohol对EstZ1的活性有部分抑制(大约15.3%-78.8%)。其中可破坏二硫键的β-mercaptoethanol对EstZ1有强烈抑制,说明二硫键对于EstZ1的稳定性和活性有较大影响。
表 3. 金属离子和化学试剂对EstZ1的活力影响 Table 3. Effect of various metal ions and chemical agents on enzyme activity
| Reagents | Concentrate | Relative activity/% |
| None | 0 mmol/L | 100.0±0.4 |
| Ag+ | 1 mmol/L | 113.2±0.7 |
| Ca2+ | 1 mmol/L | 93.1±0.1 |
| K+ | 1 mmol/L | 85.8±0.4 |
| Fe2+ | 1 mmol/L | 111.2±0.3 |
| Zn2+ | 1 mmol/L | 34.0±0.2 |
| Mg2+ | 1 mmol/L | 91.5±0.4 |
| Na+ | 1 mmol/L | 103.0±0.2 |
| Mn2+ | 1 mmol/L | 93.7±0.3 |
| Fe3+ | 1 mmol/L | 146.5±0.0 |
| Pb2+ | 1 mmol/L | 88.0±0.1 |
| Ni2+ | 1 mmol/L | 103.1±0.9 |
| Co2+ | 1 mmol/L | 84.7±0.1 |
| Cu2+ | 1 mmol/L | 105.5±0.4 |
| Hg2+ | 1 mmol/L | 39.7±0.1 |
| Al3+ | 1 mmol/L | 128.3±0.3 |
| EDTA | 1 mmol/L | 125.8±0.2 |
| SDS | 1 mmol/L | 60.4±1.0 |
| β-mercaptoethanol | 1 mmol/L | 21.2±0.1 |
| Triton X-100 | 1% V/V | 111.1±0.1 |
| Tween-80 | 1% V/V | 67.1±0.7 |
| Methylalcohol | 1% V/V | 91.4±0.5 |
| Alcohol | 1% V/V | 75.1±0.4 |
| Acetone | 1% V/V | 95.4±0.5 |
表选项
2.5 EstZ1对邻苯二甲酸二乙酯的降解 HPLC分析结果显示邻苯二甲酸二乙酯(DEP)标品的保留时间为5.080 (图 5-A),产物中除了在相同的保留时间出现峰还出现了新的峰,表明产物中除了DEP (分子量222)还有新物质生成(图 5-B)。酯酶水解邻苯二甲酸酯类的降解途径为酯键逐个水解或者同时水解(图 6),所以DEP的可能降解产物有邻苯二甲酸单乙酯(MEP,分子量194)和邻苯二甲酸(PTH,分子量166)。LC/MS的分析结果显示,可以得到加Na+的分子离子峰m/z为245(图 7-A),减H+的分子离子峰m/z为193 (图 7-B),根据分子量推算可以推测出EstZ1水解DEP可生成MEP。
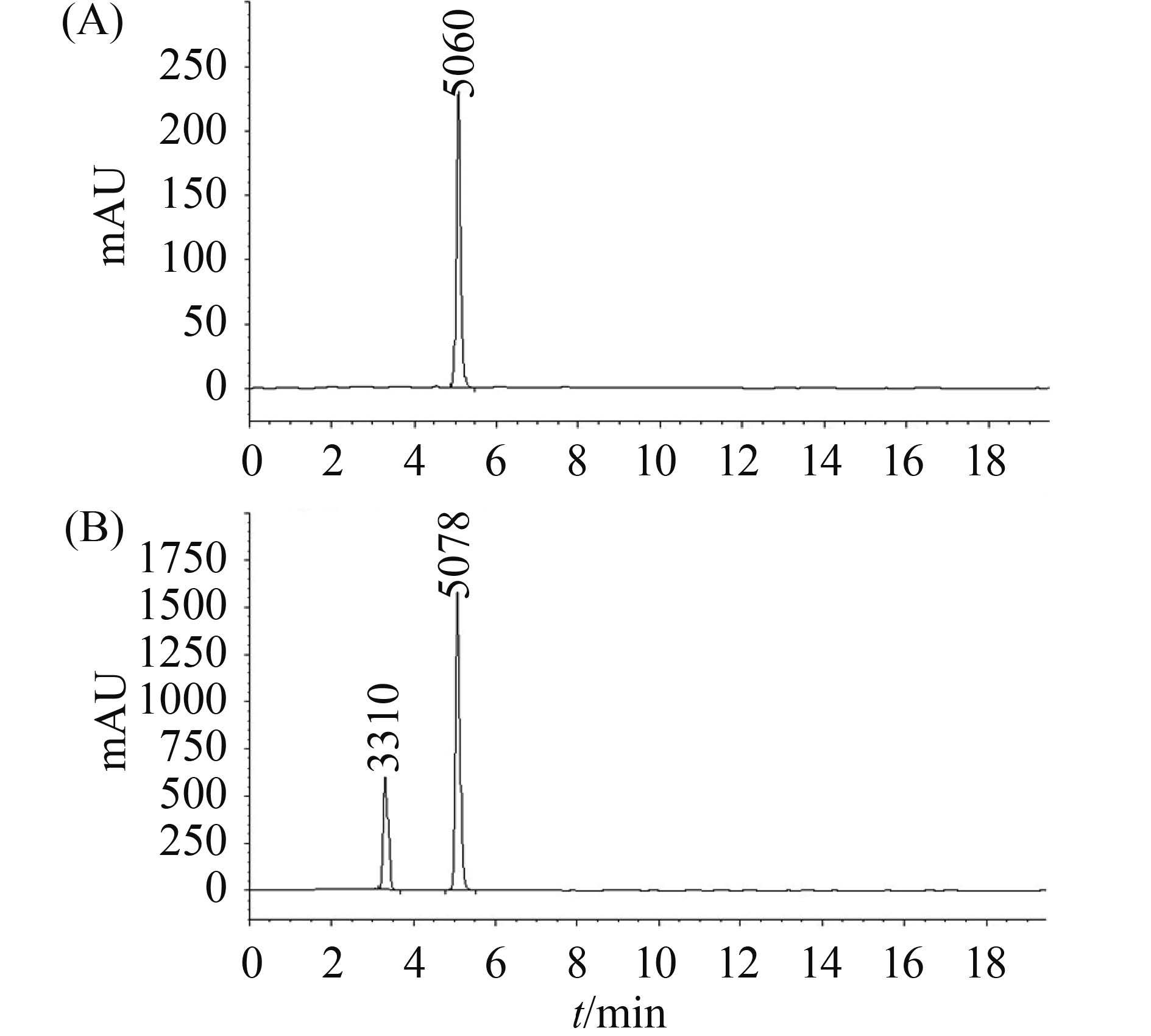 |
| 图 5. HPLC分析结果 Figure 5. The results of HPLC analyses. A: DEP standard sample. B: The results of DEP degradation after incubation with EstZ1 |
| 图选项 |
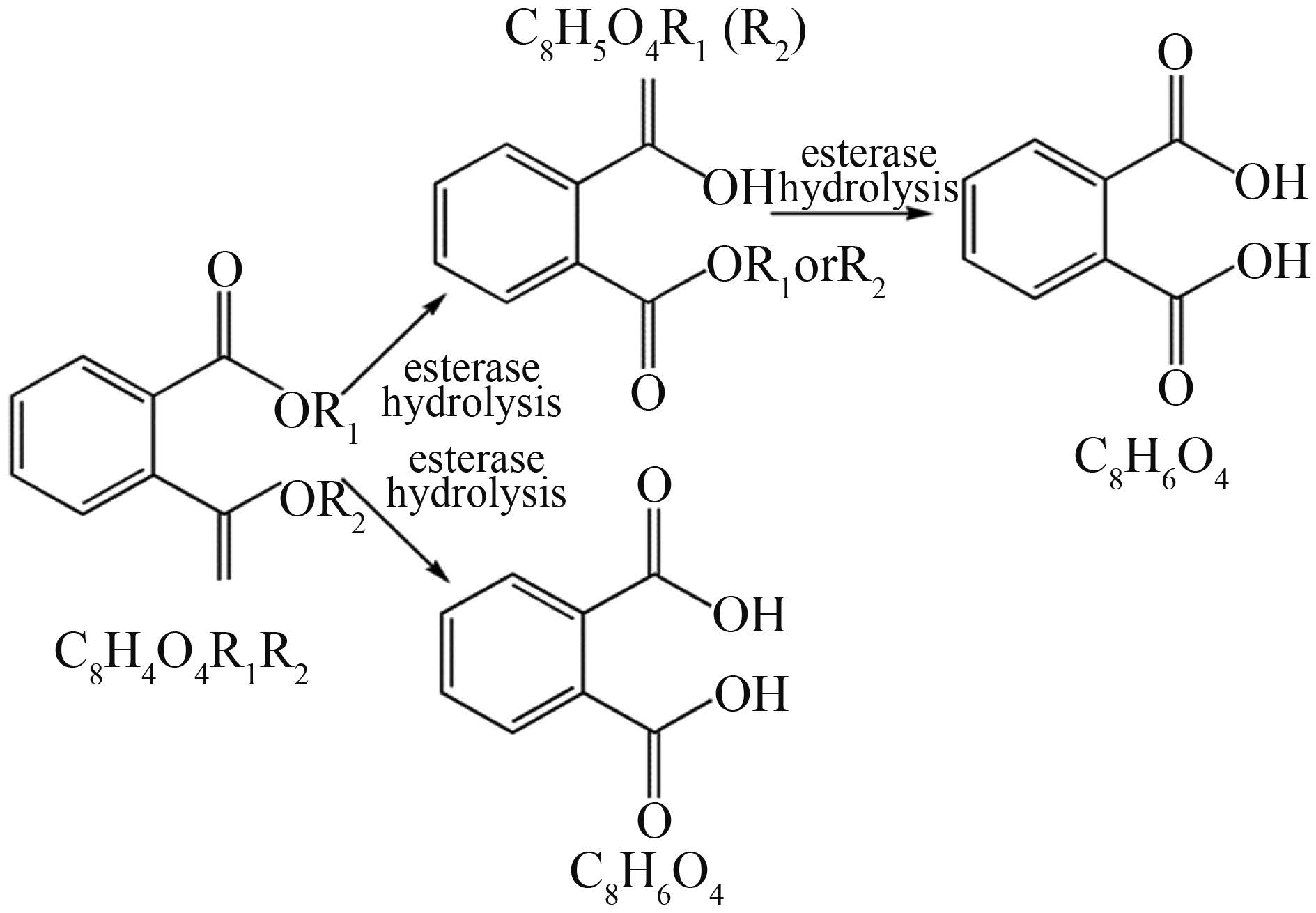 |
| 图 6. 酯酶对邻苯二甲酸酯水解 Figure 6. Degradation of PAEs by esterase |
| 图选项 |
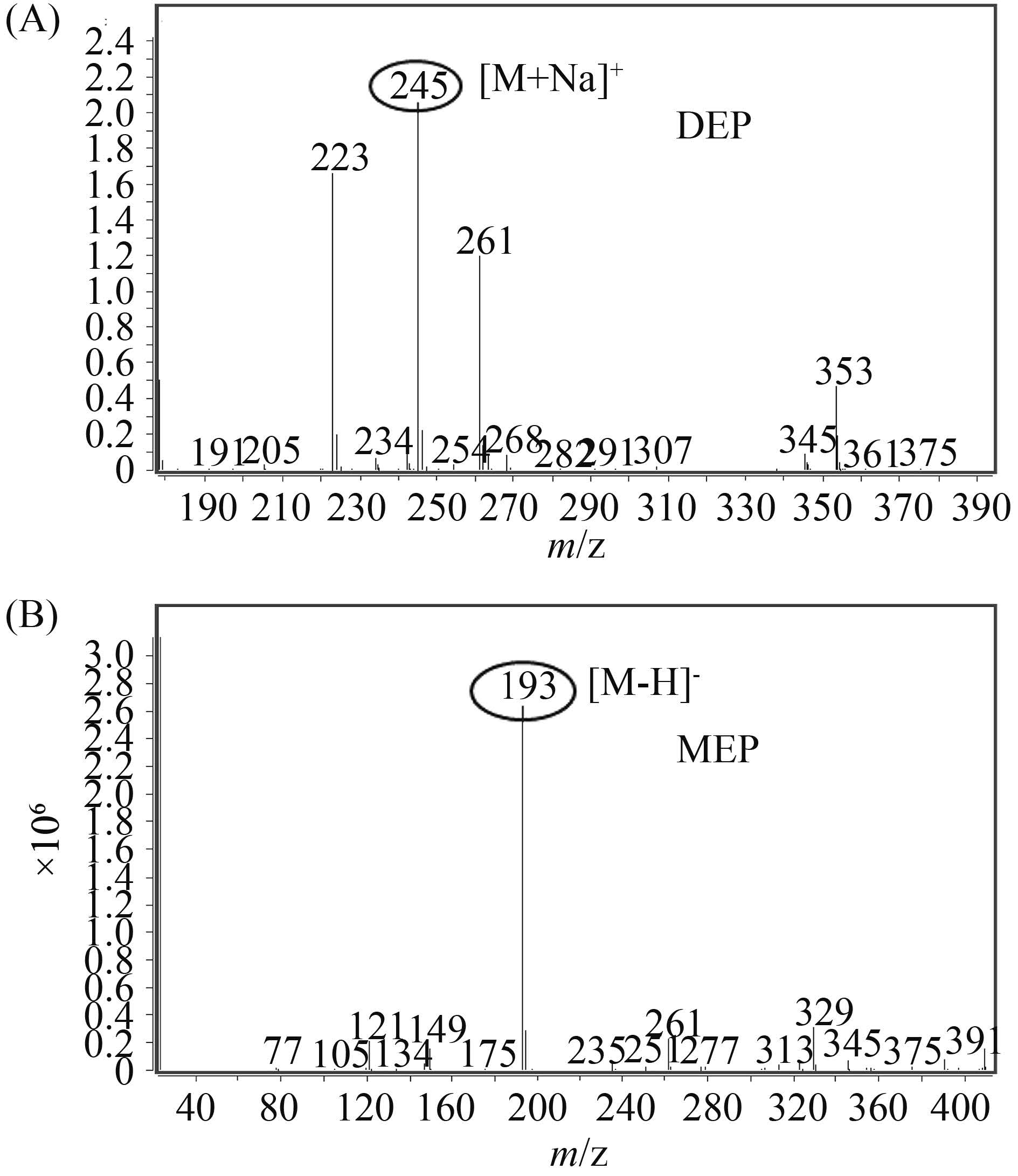 |
| 图 7. LC/MS分析结果 Figure 7. The results of LC-MS analyses. A: DEP (m/z 245) which detected in the products. B: MEP (m/z 193) which also detected in the products |
| 图选项 |
3 讨论 本研究得到了来自Bacillus sp. HJ14的酯酶EstZ1并研究了其酶学性质和对环境激素类化合物邻苯二甲酸二乙酯(常用作增塑剂)的降解。EstZ1表现出对底物对硝基苯酚酯类的较强活性并可将邻苯二甲酸二乙酯(DEP)水解生成邻苯二甲酸单乙酯(MEP)和相应的醇。综上,EstZ1的酶学性质和应用实验表明EstZ1是一个能够降解增塑剂的新型耐热HSL-like家族酯酶。
从EstZ1的序列分析中发现,EstZ1存在类环境激素敏感脂肪酶(Hormone-sensitive lipase,HSL-like)的保守结构域表明它属于环境激素敏感脂肪酶或者酯酶家族[28]。环境激素敏感酯酶包括了脂肪酶和羧酸酯酶,类似于哺乳动物的环境激素敏感脂肪酶,这个家族的耐热酶在生物催化方面具有巨大的潜力。底物动力学参数显示EstZ1对p-NPC2和p-NPC4有着较高的活性,对pNPC6和p-NPC8的活力很低,当碳链长度大于10时基本没有酶活,与已报道的HSL-like家族酯酶E25[29]性质相似,然而来自Alicyclobacillus acidocaldarius的HSL-like家族酯酶Est2[30]的酶促反应速率随着碳链长度的增加而变快。EstZ1的最适温度为50 ℃与E25一样,低于Est2的70 ℃,但是对EstZ1热稳定性的研究中发现其有着很强的耐热性,在60 ℃下耐受一小时酶活还剩余80%以上,在80 ℃下的半衰期为20 min,这样耐热性要好于来自Archaeoglobus fulgidus DSM4304的耐热酯酶Est-AF[31]、来自Aspergillus fumigatus的酯酶EstQ[32]和来自Sulfobacillus acidophilus DSM10332的酯酶EstS1[33]。目前为止我们知道EstZ1是耐热性质非常好的HSL-like家族酯酶。此外,EstZ1还表现出了广阔的pH (5.0-11.0)范围,与E25 (6.0-11.0)相似,高于Est2 (5.6-8.0)。综上EstZ1表现出的耐热性和嗜碱性与已报道的HSL-like家族酯酶以及耐热酯酶性质大致符合,这使得EstZ1在环境激素类化合物或污染物的生物降解中具有着别的HSL-like家族酯酶无法比拟的优势。
根据先前的研究,酯酶是一类催化合成或者水解酯键的水解酶,并且HSL-like家族酯酶与哺乳动物HSL在生物催化方面有着相似的功能。结合这两方面我们试着用EstZ1来降解被当做增塑剂广泛应用于各个行业的邻苯二甲酸二乙酯(DEP),结果表明EstZ1可以将DEP水解成邻苯二甲酸单乙酯(MEP)和相应的醇。但是值得注意的是EstZ1只能水解DEP的1个酯键,而对邻苯二甲酸单乙酯(MEP)没有活性,并不能将其水解生成邻苯二甲酸(PTH)。在已报道的其他家族酯酶降解苯二甲酸酯类的研究中大多数酯酶仅能水解一个酯键,例如,来源于Fusarium sp. DMT53的酯酶仅能水解对苯二甲酸二甲酯(DMT)的1个酯键,而对于对苯二甲酸单甲酯(MMOL/LT)则没有水解活性,不能水解单酯[34]。少数可以同时水解2个酯键,Ding等[35]克隆表达的酯酶CarEW,可将邻苯二甲酸二异丁酯(DiBP)水解生成邻苯二甲酸单丁酯(MiBP),又进一步将MiBP水解生成邻苯二甲酸(PTH),所以对于EstZ1水解DEP的分子机制我们尚不清楚,同时EstZ1能否水解其他种类的底物还需要进一步深入。另外,我们还可以尝试利用分子定向进化技术定向改造EstZ1来提高其稳定性和抗性,以便在实际应用中获得更大前景。
参考文献
| [1] | Sayali K, Sadichha P, Surekha S. Microbial esterases:an overview.International Journal of Current Microbiology and Applied Sciences, 2013, 2(7): 135–146. |
| [2] | Jaeger KE, Reetz MT. Microbial lipases form versatile tools for biotechnology.Trends in Biotechnology, 1998, 16(9): 396–403DOI:10.1016/S0167-7799(98)01195-0. |
| [3] | 徐红娜.利用定向进化提高羧酸酯酶E.coli BioH水解活性和酰化活性的研究.杭州:浙江大学硕士学位论文,2013. |
| [4] | Panda T, Gowrishankar B. Production and applications of esterases.Applied Microbiology and Biotechnology, 2005, 67(2): 160–169DOI:10.1007/s00253-004-1840-y. |
| [5] | Riley DJS, Kyger EM, Spilburg CA, Lange LG. Pancreatic cholesterol esterases.2.Purification and characterization of human pancreatic fatty acid ethyl ester synthase.Biochemistry, 1990, 29(16): 3848–3852DOI:10.1021/bi00468a007. |
| [6] | Weng X, Meng L, Liu CJ. Study on part purification of botanical esterase for detection of pesticide residue.Chemistry & Bioengineering, 2006, 23(2): 22–24(in Chinese).翁霞, 孟玲, 刘长江. 用于农药残留测定的植物酯酶部分纯化的研究.化学与生物工程, 2006, 23(2): 22–24. |
| [7] | Contesini FJ, Lopes DB, Macedo GA, da Gra?a Nascimento M, de Oliveira Carvalho P. .Aspergillus sp.lipase:potential biocatalyst for industrial use.Journal of Molecular Catalysis B:Enzymatic, 2010, 67(3/4): 163–171. |
| [8] | Fang CR, Chen CL, Xiang S. Review of degradation of plasticizers (Phthalic Acid Esters).Journal of Zhejiang University of Science and Technology, 2006, 18(4): 286–290(in Chinese).方程冉, 陈川龙, 项硕. 邻苯二甲酸酯类增塑剂降解研究进展.浙江科技学院学报, 2006, 18(4): 286–290. |
| [9] | Keyser P, Pujar BG, Eaton RW, Ribbons DW. Biodegradation of the phthalates and their esters by bacteria.Environmental Health Perspectives, 1976, 18: 159–166DOI:10.1289/ehp.7618159. |
| [10] | Nomura Y, Takada N, Oshima Y. Isolation and identification of phthalate-utilizing bacteria.Journal of Fermentation and Bioengineering, 1989, 67(4): 297–299DOI:10.1016/0922-338X(89)90235-3. |
| [11] | Yan H, Ye CM, Yin CQ. Kinetics of phthalate ester biodegradation by Chlorella pyrenoidosa.Environmental Toxicology and Chemistry, 1995, 14(6): 931–938DOI:10.1002/etc.v14:6. |
| [12] | Mathur SP, Rouatt JW. Utilization of the pollutant di-2-ethylhexyl phthalate by a bacterium.Journal of Environmental Quality, 1975, 4(2): 273–275. |
| [13] | Yuan SY, Lin YY, Chang BV. Biodegradation of phthalate esters in polluted soil by using organic amendment.Journal of Environmental Science and Health,Part B, 2011, 46(5): 419–425DOI:10.1080/03601234.2011.572512. |
| [14] | Zhang Y, Wang L, Du N, Ma GP, Yang AM, Zhang H, Wang ZG, Song QX. Effects of diethylphthalate and di-(2-ethyl) hexylphthalate on the physiology and ultrastructure of cucumber seedlings.Environmental Science and Pollution Research, 2014, 21(2): 1020–1028DOI:10.1007/s11356-013-1884-6. |
| [15] | Chang BV, Liao CS, Yuan SY. Anaerobic degradation of diethyl phthalate,di-n-butyl phthalate,and di-(2-ethylhexyl) phthalate from river sediment in Taiwan.Chemosphere, 2005, 58(11): 1601–1607DOI:10.1016/j.chemosphere.2004.11.031. |
| [16] | Yuan SY, Huang IC, Chang BV. Biodegradation of dibutyl phthalate and di-(2-ethylhexyl) phthalate and microbial community changes in mangrove sediment.Journal of Hazardous Materials, 2010, 184(1/3): 826–831. |
| [17] | Lu Y, Tang F, Wang Y, Zhao JH, Zeng X, Luo QF, Wang L. Biodegradation of dimethyl phthalate,diethyl phthalate and di-n-butyl phthalate by Rhodococcus sp.L4 isolated from activated sludge.Journal of Hazardous Materials, 2009, 168(2/3): 938–943. |
| [18] | Wu XL, Liang RX, Dai QY, Jin DC, Wang YY, Chao WL. Complete degradation of di-n-octyl phthalate by biochemical cooperation between Gordonia sp.strain JDC-2 and Arthrobacter sp.strain JDC-32 isolated from activated sludge.Journal of Hazardous Materials, 2010, 176(1/3): 262–268. |
| [19] | Boonyaroj V, Chiemchaisri C, Chiemchaisri W, Theepharaksapan S, Yamamoto K. Toxic organic micro-pollutants removal mechanisms in long-term operated membrane bioreactor treating municipal solid waste leachate.Bioresource Technology, 2012, 113: 174–180DOI:10.1016/j.biortech.2011.12.127. |
| [20] | Ejlertsson J, Meyerson U, Svensson B. Anaerobic degradation of phthalic acid esters during digestion of municipal solid waste under landfilling conditions.Biodegradation, 1996, 7(4): 345–352DOI:10.1007/BF00115748. |
| [21] | Amir S, Hafidi M, Merlina G, Hamdi H, Jouraiphy A, El Gharous M, Revel JC. Fate of phthalic acid esters during composting of both lagooning and activated sludges.Process Biochemistry, 2005, 40(6): 2183–2190DOI:10.1016/j.procbio.2004.08.012. |
| [22] | Aharoni A, Amitai G, Bernath K, Magdassi S, Tawfik DS. High-throughput screening of enzyme libraries:thiolactonases evolved by fluorescence-activated sorting of single cells in emulsion compartments.Chemistry & Biology, 2005, 12(12): 1281–1289. |
| [23] | Zhou YX, Zhu SY, Cai CZ, Yuan PF, Li CM, Huang YY, Wei WS. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells.Nature, 2014, 509(7501): 487–491DOI:10.1038/nature13166. |
| [24] | Zhang LH, Zhao CX, Ohta WY, Wang YJ. Inhibition of glucose on an exoinulinase from Kluyveromyces marxianus expressed in Pichia pastoris.Process Biochemistry, 2005, 40(5): 1541–1545DOI:10.1016/j.procbio.2004.01.057. |
| [25] | Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:molecular evolutionary genetics analysis version 6.0.Molecular Biology and Evolution, 2013, 30(12): 2725–2729DOI:10.1093/molbev/mst197. |
| [26] | Willard L, Ranjan A, Zhang HY, Monzavi H, Boyko RF, Sykes BD, Wishart DS. VADAR:a web server for quantitative evaluation of protein structure quality.Nucleic Acids Research, 2003, 31(13): 3316–3319DOI:10.1093/nar/gkg565. |
| [27] | Ferrè F, Clote P. Disulfide connectivity prediction using secondary structure information and diresidue frequencies.Bioinformatics, 2005, 21(10): 2336–2346DOI:10.1093/bioinformatics/bti328. |
| [28] | Angkawidjaja C, Koga Y, Takano K, Kanaya S. Structure and stability of a thermostable carboxylesterase from the thermoacidophilic archaeon Sulfolobus tokodaii.FEBS Journal, 2012, 279(17): 3071–3084DOI:10.1111/j.1742-4658.2012.08687.x. |
| [29] | Li PY, Ji P, Li CY, Zhang Y, Wang GL, Zhang XY, Xie BB, Qin QL, Chen XL, Zhou BC, Zhang YZ. Structural basis for dimerization and catalysis of a novel esterase from the GTSAG motif subfamily of bacterial hormone-sensitive lipase (HSL) family.The Journal of Biological Chemistry, 2014, 289(27): 19031–19041DOI:10.1074/jbc.M114.574913. |
| [30] | Manco G, Febbraio F, Adinolfi E, Rossi M. Homology modeling and active-site residues probing of the thermophilic Alicyclobacillus acidocaldarius esterase 2.Protein Science, 1999, 8(9): 1789–1796DOI:10.1110/(ISSN)1469-896X. |
| [31] | Kim SB, Lee W, Ryu YW. Cloning and characterization of thermostable esterase from Archaeoglobus fulgidus.The Journal of Microbiology, 2008, 46(1): 100–107DOI:10.1007/s12275-007-0185-5. |
| [32] | Zhang SS, Wu GB, Feng SY, Liu ZD. Improved thermostability of esterase from Aspergillus fumigatus by site-directed mutagenesis.Enzyme and Microbial Technology, 2014, 64-65: 11–16DOI:10.1016/j.enzmictec.2014.06.003. |
| [33] | Zhang XY, Fan X, Qiu YJ, Li CY, Xing S, Zheng YT, Xu JH. Newly identified thermostable esterase from Sulfobacillus acidophilus:properties and performance in phthalate ester degradation.Applied and Environmental Microbiology, 2014, 80(22): 6870–6878DOI:10.1128/AEM.02072-14. |
| [34] | Luo ZH, Wu YR, Chow RKK, Luo JJ, Gu JD, Vrijmoed LLP. Purification and characterization of an intracellular esterase from a Fusarium species capable of degrading dimethyl terephthalate.Process Biochemistry, 2012, 47(5): 687–693DOI:10.1016/j.procbio.2012.01.015. |
| [35] | Ding JM, Wang CF, Xie ZR, Li JJ, Yang YJ, Mu YL, Tang XH, Xu B, Zhou JP, Huang ZX. Properties of a newly identified esterase from Bacillus sp.K91 and its novel function in diisobutyl phthalate degradation.PLoS One, 2015, 10(3): e0119216DOI:10.1371/journal.pone.0119216. |
