Possible technical elements for the ultimate nucleotide sequencers
Jun Yu1,2编委: 包其郁
收稿日期:2018-07-2修回日期:2018-09-26网络出版日期:2018-11-20
| 基金资助: |
Received:2018-07-2Revised:2018-09-26Online:2018-11-20
| Fund supported: |

摘要
关键词:
Abstract
Keywords:
PDF (345KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
于军. 实现“终极版”核苷酸测序仪的技术要素[J]. 遗传, 2018, 40(11): 929-937 doi:10.16288/j.yczz.18-179
Jun Yu.
除了学科基础和人才考量外,对各科学学科发展方向的把握(学科前沿的识别)、对科学领域拓展优先权的考量(新学科的识别)、对实现科学目标途径的选择(路线图)构成驾驭科学动态发展的基本要素。除了学科基础和布局外,满足学科发展与新颖性科学发现的需求是技术、工程、方法和资源(包括人与物)等支撑要素积累与发展的原动力。因此,对于技术的评价,对于关键前沿技术、核心工程技术、颠覆性集成技术等技术分类的识别构成科学发展的重要基石。近半个世纪以来的科技发展史充分地证明了这一点,尤其是物理学与生命科学的发展历程更充分地证明了这些判断。例如,来自这两个领域的科学家,美国理论物理学家巴丁(John Bardeen, 1908~ 1991)和英国生物化学家桑格(Fredrick Sanger, 1918~ 2013),他们在各自领域发明的关键前沿技术(晶体管和超导效应、蛋白质和DNA测序原理)分别开启了物理学的信息时代和生物学的分子时代,对科学重大转折、高速发展的时代做出了伟大贡献。同时,诺贝尔奖官方网站(https://www.nobelprize.org)显示,他们也是诺贝尔奖历史中独有的两位同一领域两次获得诺奖(均分别在20世纪50年代和70年代)的科学家。
生命科学领域曾经发生了一次从观察生物学到分子生物学的重大转折,相应的更宏观的是从宏观生物圈(the Macrobiosphere)到微观生物圈(the Microbiosphere)的转折。这个转折引出了更深刻的理论和技术的综合思考[1,2]。在微观生物圈里,生命科学从分子水平不断认识自身发展的需求,不断发现新的现象、原理和理论、乃至新的地貌(landscape),而其基本特征是与物理、化学、工程学、信息学等在概念、关系和参数上实现实质性的汇聚、融合,至最后的融会贯通。从量子力学出发,到量子物理和量子化学的应用,再到量子生物学的应用,构成微观生物圈的基本科学基础和途径。而在这个过程中,技术的汇聚与应用(即仪器与设备的制造)成为关键中的关键,用以支持和引领数据导向(data- driven)、发现导向(discovery-driven)的科学研究活动,比如DNA序列的测定和蛋白质机器的结构确定。
生命科学在科学的重大转折中同样有两条基本主线:其一是自身发展和相应的技术突破;其二是与其他学科的融合并进。生命科学进入分子水平是从20世纪40年代人类对DNA(即遗传物质)和DNA序列编码基因的认知开始。70年代以来的寡聚核甘酸合成技术、DNA测序技术、核苷酸链式杂交技术,以及80年代的PCR技术构成了基因组科学发展的核心技术,这些技术的延伸和翻新一直延续至今,也体现在几代DNA测序仪的核心技术组分之中[3,4,5,6]。可见,就基因组学所引领的生命科学前沿而言,DNA测序技术(连同RNA测序一起可统称为核苷酸测序技术)无疑已成为重中之重,其涉及的核心元素就成为核心技术要素,其聚焦的突破性研究活动就成为前沿研究,而与其一起发展起来的“孪生兄弟”之一即是生物信息学,后者又带上了“互联网+”和人工智能等信息技术前沿要素。
纵观人类文明发展史,科学从来没有像今天这样表现出对技术发展的渴求。首先,就科学总体而言,本身的进步与技术的发展产生了无法摆脱的相互依赖性。也就是说,科学发展的速度越快,遇到技术瓶颈的机会就越多,也越难突破,突破的需求也越强烈。比如分子生物学对DNA测序的需求,细胞生物学对RNA测序仪和高灵敏度质谱仪的需求。其次,人类科学知识的进步提供了预测科学进步与
技术发展可能进程与重要转折的能力。比如从宏观生物圈到微观生物圈的转折。第三,成功的划时代大科学计划突破了科学研究的自由探索和假说导向的经典模式,开拓了数据、发现导向的新模式,奠定了规模化生物学研究的理论基础和方法学。如果说国际“人类基因组计划”代表了人类自20世纪70年代以来对破解生命奥秘的理想追求,那么各国纷纷启动的“精准医学”计划则代表了人类21世纪以来致力于保障自身健康需求的美好愿景。任何大科学计划必有“五位一体”的发展规划:发展新学科(或学科前沿)、建立相应的新科研机构、突破关键技术、完成特定引领项目以及造就新业态(或促进现有产业发展)。核苷酸序列测定作为其中一个核心的关键技术在推动生命科学研究领域发展过程中发挥着不可替代的作用。第四,知识产权日益受到尊敬和重视,而在未来一定会成为一种“硬通货”,科学和技术成果将会在未来的某个商业化体系中实现估值、流通和兑现。第五,由于国际层面的多领域竞争、国家层面的领域发展优先权决策、研究经费投入分配等复杂原因,科技投入不仅日益增加,而且与国家、社会和国民经济发展绑定在一起。由此可见,生命科学在诸多科学领域竞争中的地位决定了实现其技术需求的优先权,而不仅仅是学术界的共识。考虑到技术投入的实效性、持续性和总量评估,生命科学相关技术的开发投入需要国家战略的介入,尤其是与健康保障领域的支出挂钩。
在此,笔者从40年来亲历的本学科领域科学发展和技术需求,分四个方面介绍对核苷酸测序技术的发展、面临困境和必由之路的体会和认识:首先,阐述生命科学从宏观生物圈到微观生物圈转折中最后阶段所需要进行的努力和基本目标;其次,探讨生命科学实验技术平台的发展和需求;然后,展望核苷酸测序技术未来的成长;最后,探讨“终极版”核苷酸测序仪所要汇聚的技术要素。
1 生命科学研究的大转折:从宏观生物圈到微观生物圈
人类早在300多年前即已知道微观生物圈的存在,近60年来的科学发展,尤其是细胞生物学和分子生物学的发展,让人们认识到细胞在分子水平更深层次(如基因表达与调控)的活动和变化。然而,人们到目前为止还没有在物理大小上将细胞和组成细胞的大、小分子真正联系起来。换句话讲,目前还没有一个细胞的CPK (Corey-Pauling-Koltun,空间充满式)模型,来模拟细胞的结构存在、时空匹配、运动平衡、新陈代谢等,更没有实现从头组装一个细胞器,或者原核或真核的活细胞。如果要提出这样宏大的科学设想,启动这样划时代的科学计划和分解项目,不汲取启动和操作“人类基因组计划”的经验,不深思熟虑技术难题的解决方案和计划是不可能发生的。生命科学在微观生物圈实现与其他学科在科学与技术层面的汇聚至少要从以下四个方面努力:首先是用(生物)化学法从头合成生命,目前的初步努力是从头合成信息的载体——DNA。尽管人类合成生命的步伐已经迈出了坚实的脚步[7],不过目前的合成仅限于信息流研究,将来一定会延伸到操作流中的DNA与RNA、蛋白质相互作用调控染色质结构部分,那时就要建立更多的模式底盘生物(chassis)。其次是体外模拟生命操作(流)、稳态(平衡流)、分室(流)等更复杂的生物过程。例如,利用微纳流控装置在单分子(机器)和单细胞水平设计和进行精准的体外无细胞系统(cell-free)实验[8]。第三是利用以上两个体系,再加上谱系特异(lineage-specific)的自然体系,来观察和训练生命体系对环境的应答和适应,也就是可塑性(流)揭示其分子机制。第四是利用计算机科学和信息科学的进展,实现虚拟细胞,最终实现生命的全方位虚拟化。
实现微观生物圈需要与物理、化学、计算机科学在尺度上汇聚主要体现在分子水平的机制研究和实际应用的牵引,这两个问题都非常重要。前者的重要性在于提出和凝聚科学问题,后者则是路线图和短期(大项目)目标的制定。比如,生命体系的“力”是目前要彻底研究的问题(表1),而生命体内最基本的化学键——氢键力的准确测定,分离氢键力与碱基叠加力等还没能得到科学界的共识[9,10]。其复杂性在于水作为生命的物质载体的最大特征是与周围的生物分子、有机分子、甚至很多无机分子形成复杂、多样、多变的氢键系统。由此可见,人们要走的科学之路还很漫长,需要深刻思考和解决问题的方案。不过,几个可能的大科学项目还是可以设想出来的。例如,除了合成生命体外,还可以启动“单细胞RNA组计划”(测定每一种细胞的RNA表达谱与调控动态,可与“细胞组图谱计划”交汇;https://www. humancellatlas.org)、“虚拟线粒体计划”(主要是能量产生的控制和代谢平衡)、RNA修饰组计划(构建RNA修饰组数据库)等。
Table 1
表1
表1 分子间共价键和弱相互作用力
Table 1
| 作用力类型 | 举例 | 作用力(pN) |
|---|---|---|
| 打开共价键 | 碳-碳键(C-C) | 1600 |
| 碳-硅键(C-Si) | 370 | |
| 二硫桥(—C-S-S-C—) | 100 | |
| ATP水解 | 80 | |
| 打开弱键 | 氢键 | 4 |
| 打开非共价作用力 | 生物素(Biotin-SA) | 160 |
| 抗体(Ab-Ag) | 100 | |
| 受体(Receptor-ligand) | 50 | |
| DNA与核小体 | 20 | |
| 细胞运动 | 哺乳动物细胞 | 80 |
| 驱动蛋白(kinesin) | 5 |
新窗口打开|下载CSV
2 细胞与分子生化实验平台的时代演进:单细胞单分子时代
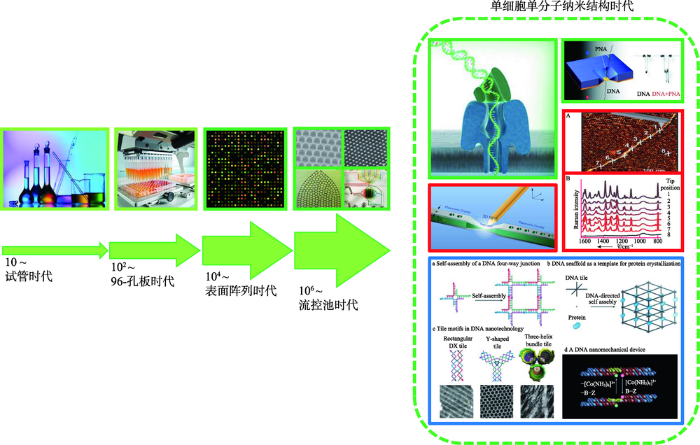
生命科学的实验平台是从化学实验平台等逐步衍生而来的,并从70年代的“试管时代”开始(第一代),经过了80年代的“96-孔板时代”(第二代),到后来90年代的“表面阵列时代”(第三代),00年代的“流控池时代(第四代)”,再到了今天的单细胞单分子——“双单时代”(第五代) (图1)。这五代实验平台的变迁不仅仅改变了实验室的基本布局,也改变了基本实验工具、仪器和设备,更改变了实验平台和构成实验平台的基本模式、技术元素、实验规模和通量(即效率)。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1核苷酸研究实验技术平台发展示意图
核苷酸研究实验平台的演变。箭头表示各种平台测序通量的增加。第五代测序技术包含的元素如右图所示(自左上角起:蛋白质纳米孔、固态纳米孔、原子力显微镜、增强拉曼光谱、DNA多角结构)。
Fig. 1The evolution of experimental platforms for nucleic acids research
在这些技术的变迁中,一些技术的应用被逐渐抛弃,而有些则实现了新平台上的“咸鱼翻身”,还有些则退守“小众市场”(niche market)。比如,寡聚核苷酸合成实现从单一合成到96-孔板合成,再到阵列合成和微流控合成等一系列的“翻身”,其生命力犹在。PCR也完成了从普通PCR到定量PCR,再到数字PCR的进步,走进了“双单时代”。然而DNA和RNA分离等技术还没有达到单细胞水平,比如,目前还不能直接从单细胞开始制备其中的DNA或RNA。尽管流式细胞仪可以高通量分离单个细胞,但是需要小型化的配套设备来接轨。基于电泳、荧光分光技术等的DNA/RNA定量在这个环节的取代品——高灵敏度定量技术,则变得尤为重要了。
可见,在向“双单时代”的转变中,识别、培育、创造有潜力的单细胞、单分子技术成为关键环节,是技术汇聚的核心。就生物体系而言,这些技术可简单分为体内(in vivo)、体外(in vitro)、活体(ex vivo)和模拟(in silico)等形式。例如,电子显微镜可以用来观察细胞,它的“衍生物”冷冻电镜则可以用来观察生物大分子的结构,但是得到的数据基本上是体外数据。又如,微纳流控装置就其尺度而言,可以实现很多单细胞活体水平的实验操作,而且精准度超过经典的显微注射。蛋白质机器和活细胞模拟技术其实就是算法、数据库、制图软件包等的集成。目前可用的单分子技术很有限,比如原子力显微镜(atomic force microscope, AFM)、扫描隧道显微镜(scanning tunneling microscope, STM)、扫描探针显微镜(scanning probe microscope, SPM)、纳米尺度的光镊(optical tweezers)和磁镊(magnetic tweezers)、纳米孔技术(nanopores)、基于拉曼增强技术的表面增强(surface-enhanced Raman scattering, SERS)和针尖增强(tip-enhanced Raman scattering, TERS)、超分辨光学成像技术(super-resolution imaging)等。这些单分子分辨技术有些是直接法,直接观察分子的运动、测量分子间力的变化;有些是间接法,通过荧光标记来判断分子的运动轨迹,也可以通过图像的处理来实现高分辨率。
DNA测序技术可以按照前述五个实验平台时代的技术内涵来确定其时代更替(表2)。在这里,第一代测序技术对应的是第一代、第二代实验平台;第二代测序技术,即所谓下一代(next-generation sequencing,NGS)技术对应的是第三代、第四代实验平台;第三代测序技术对应的是第四代、第五代实验平台。目前的单分子测序平台共有3个,分别来自美国太平洋生物科学公司(Pacific BioSciences, PacBio)、英国牛津纳米孔技术有限公司(Oxford Nanopores Technologies, ONT)和深圳市瀚海基因生物科技有限公司(Direct Genomics;其技术源于美国斯坦福大学Stephen Quake实验室)。其中,ONT的技术其实应该属于第五代实验平台,因为只有它没有用到第四代的微流控反应池(flowcell)技术,用的是微纳加工技术。其他两家公司的产品则用的是间接法荧光标记技术和微流控反应池。另外,ONT的单分子测序平台用的是纳米孔阵列(低通量),而PacBio和瀚海基因分别用的是ZMW(zero-mode waveguide)阵列和表面随机阵列。
Table 2
表2
表2 各代核苷酸测序平台(仪)的主要技术要素
Table 2
| 核心技术元素 | 第一代 (桑格法) | 第二代 (循环法) | 第三代 (单分子 分辨率) | 第四代 (直接 读取) |
|---|---|---|---|---|
| 蛋白质与酶学 | √ | √ | √ | √ |
| 荧光核苷酸标记 | √ | √ | √ | |
| 模版PCR扩增 | √ | √ | ||
| 凝胶电泳 | √ | |||
| 流控反应池 | √ | √ | √ | |
| 半导体加工 | √ | √ | √ | |
| 超分辨荧光显微镜 | √ | √ | ||
| 纳米光电 | √ | √ | ||
| 纳米材料 | √ | √ | ||
| 蛋白质纳米孔 | √ | √ | ||
| 固体纳米孔 | √ | |||
| 单分子核磁共振 | √ | |||
| 单分子增强拉曼光谱 | √ |
新窗口打开|下载CSV
3 DNA测序技术从“初级版”到“终极版”
DNA测序技术其实只是获取线性的遗传信息, 是满足科学数据诸多需求中的初级部分。如果扩大到核苷酸测序,那就包括了RNA序列的测定。遗传信息的初级部分是线性形式(信息流)来存储的,是以核苷酸的线性序列为基础。因此,核苷酸测列的测定技术成为基因组学研究的基本技术之一。DNA测序与RNA测序在本质上是有差别的,只是目前的RNA测序其实就是DNA测序,是将RNA转录成cDNA(互补DNA)后,通过测定cDNA的序列,最后关联到RNA序列的,不是用逆转录酶催化的RNA直接测序。在直接测序得到的信息方面,DNA测序与RNA测序也有很大的差别。首先,人体的每个细胞一般都会带有相同的DNA序列信息(除了DNA共价修饰信息、生殖系与体细胞系遗传差异、遗传嵌合体问题等微小不同外),但是却带有不同的RNA序列信息,包括RNA种类、每个种类的数量分布、每个RNA分子上的修饰位点等。每个细胞RNA的信息量(总核苷酸数)大约是DNA的3倍。其次,RNA分子是以单链的形式存在的,不仅有复杂的高级结构,大小变化也很大(成熟RNA分子通常在20~10 000 nt),因此在从RNA制备cDNA时,其序列信息就会有很大的缺失,直接关系到RNA信息的完整性。再者,在从RNA到cDNA的逆转录过程中,几乎所有的共价修饰核苷酸信息均被丢掉。虽然DNA中共价修饰核苷酸很少,但是RNA的这种共价修饰核苷酸则有百余种,而且在单分子水平就有变化(位点的有无,构成该RNA分子中该位点的修饰程度等)。最后,科学界对RNA组学的研究从时间来看很久远了,但是从研究的进展(尤其是信息的完整性)来看几乎是零。比如,人们可以精准地测定一个人的基因组序列。但是却没有一个或一种细胞的RNA组,甚至仅是转录组或是mRNA组被完成。其实,在RNA组学的框架下,一种细胞的RNA组是不存在的,但是其动态范围是可以是有限的,也可以以统计学参数来界定的。以此类推的原因还有很多,如诸多的表达标签EST研究,但归根结底的原因在于缺乏行之有效的RNA序列直接测定技术。
简单来说,DNA测序技术的发展传承有序——从桑格法到纳米孔测序,实现了基因组序列数据的实际、有效的积累。这与技术的完善和方法学的开发都有关系,当然也受到政策、法律等因素的影响。然而,RNA序列的积累却经历了诸多曲折,以至于现在也没有令人满意的可积累的基于细胞特异性的RNA数据。近10年来,科学界逐渐意识到RNA直接测序的迫切性,使RNA直测技术有了实际资助[11],同时也有了极其初步,但是实质性的进展[12]。
DNA或RNA的线性序列测定技术评价要包括诸多生物信息与仪器参数:(1) 通量相关参数包括平均读长(average read length)、一次测序时间(turneround time)、每台仪器每次测定碱基数总和(total output);(2) 精准度相关参数包括碱基错误率、测序覆盖度对准确度的贡献、覆盖度与准确度的关系;(3) 价格相关参数包括一次测序总产量与覆盖度的关系、试剂与耗材占测序总价格的百分比。
在这些参数里,对读长、通量、价格和错误率的基本要素的评估尤为重要(表3)。这些要素的高低也直接决定了测序技术和相关仪器在市场中的表现、状态和优化目标。首先,目前还没有一台理想的仪器具有所有优良指标,但有了一匹“驮马”(workhorse),Illumina的HiSeq系列,其通量可以基本满足高通量测序的需求。但这些仪器产出数据的读长较短(150~200 bp),因此在应用推广上存在短板。例如,在用于宏基因组研究时,短读长的序列导致基因注释有较大比例的不确定性。其次,PacBio和ONT平台的读长有一定优势,但通量和错误率却都偏高,尤其是通量,导致基本价格很难降低。尽管它们的这两个“硬伤”有改进的余地,但是最终可能会定格在小众市场和大众市场之间,也许会有些杀手级应用(killer application),但在可预见的区间(10年)内可能很难有机会成为“龙头”,至少它们很难逾越错误率这个短板。
Table 3
表3
表3 各代代表性DNA测序仪及技术参数比较
Table 3
| 产品名称 | 生产厂家 | 核心技术(代次) | 各参数比较 | ||||
|---|---|---|---|---|---|---|---|
| 测序通量 | 单轮需时 | 测序读长 | 准确率 | 价格优势 | |||
| ABI3730XL | Thermo-Fischer | 桑格法(第一代) | + | + | ++ | +++ | + |
| HiSeq4000 | Illumina | 循环法(第二代) | +++ | +++ | + | ++ | +++ |
| GenoCare | 深圳瀚海基因生物 科技有限公司 | 全内反射荧光成像 (第三代) | + | ++ | + | + | ++ |
| MinION | Oxford Nanopores Technologies | 蛋白质纳米孔(第三代) | + | + | +++ | + | + |
| PacBio RS | Pacific BioSciences | 零模波导(第三代) | + | + | +++ | + | + |
| 期待中 | Yet-to-come | 固体纳米孔(第四代) | +++ | + | +++ | +++ | +++ |
新窗口打开|下载CSV
4 核苷酸测序技术“终极版”的“硬科技”挑战
值得人们深思的是:未来会有哪些真正的“硬科技”可以用来实现核苷酸测序技术的根本性革命,乃至实现市场的爆发式突破?在回答这个问题之前,作为在这个领域长期工作的科学家,我们首先要检讨自己是否设身处地,深刻地思考了相关问题?在这个领域里,我们到底有没有几个到数十个坚持不懈的研究团队?有没有一个实现有限目标的路线图?其次,我们有没有意识到研制核苷酸测序仪所涉及的科技前沿有哪些?这些前沿目前所研究和意欲突破的科学问题是什么?如果在这些方面连基本的小目标也没有,其实就相当于没有“报名”参加这个“比赛”项目。第三是要解决技术的宏观定位问题。DNA测序技术引来了划时代的大科学计划,大科学计划又推动了这项技术的深入发展,循环往复的技术发展必将推动新的业态诞生。例如,在卫生与健康保障领域,DNA测序技术成为体外诊断(in-vitro diagnostics,IVD)领域中分子诊断的产业热点。最后是要绘出产品地貌(product landscape),别人已经做了什么,我们还能做什么,将来会是什么样子的。就分子诊断市场而言,核苷酸分析仍然是一个朝阳产业,它在许多方面会取代过去的传统诊断方式和分子诊断仪器设备。比如,DNA和RNA的可扩增性是蛋白质研究很难比拟的,无形中提高了检测的灵敏度,而这个扩增的方法就是PCR技术。目前的“终极版”的核苷酸测序技术要与基因分型技术、PCR技术直接竞争分子诊断市场。就市场应用而言,需求(问题)和价格(问题的解决方案和价格)是首要决定因素。也就是说,最便宜、最准确和最有效的检测会在三者中胜出。其次是政策问题,比如基于这3类技术的检测服务是否能够进入到医保的报销单。就技术发展本身而言,基因分型技术和PCR技术也是两个正在发展的技术前沿。这两个技术都面临向第四代和第五代实验平台转移的问题,因此在在竞争的同时“终极版”核苷酸测序仪也可与之合作,共享一些前沿技术。
第三代和第四代核苷酸测序仪研发会面选择哪些技术要素,又会面临哪些科技前沿的挑战呢?尽管一个技术体系的兴衰是很难预测,但是技术的发展一般需要10年乃至以上的孵育和开发期。桑格法从发明到实现自动化,Illumina测序平台、PacBio和ONT的测序平台从实验室雏形到产品上市的过程都均是如此。目前可规划的技术和方法至少包括以下几个重要的方向:首先是单分子光学。利用光学显微镜(如全内反射荧光显微镜)的超分辨功能,可实现单分子水平的核苷酸序列测定尤其是RNA序列的直接测定。由3个因素决定:分辨率足够、技术平台趋于成熟、因此价格趋于可控。但是,单分子光学技术的缺点是仪器较大,很难实现便携。其次是纳米孔技术。蛋白质孔已经被证明可用于核苷酸的直接测序,下一个则是固体纳米孔的开发问题。纳米孔的信号检测基本上是单分子电学,检测的灵敏度足够、技术平台趋于成熟、价格也趋于可控。纳米孔技术出道时主打优势就是便携,但是准确度和通量问题始终是瓶颈。前者有待于操作系统和软件系统的升级、序列识别和质量评估软件的不断成熟,后者主要由市场的需求来牵引。纳米孔,尤其是固体纳米孔技术,允许直接测定DNA或者RNA的序列,尤其是测定RNA序列上的共价修饰核苷酸。这是用其他方法都不能直接实现的。这个方法的局限是通量问题和上游配套的单分子核苷酸分流系统(只有这个系统才能实现单分子RNA的制备,并将其直接导入纳米孔)。第三是微纳流控技术在核苷酸测序技术领域的应用和深度开发。这个系统的建立必须要从特定分离的单细胞出发,既可以以各类人类细胞为主体,也可以构建体外(in vitro)模式生物的细胞系统,比如非人类哺乳动物细胞等。进而,按大小分离和分流细胞内的RNA组分,也可以采取捕获的方法,也可以制备某种特定的RNA群,例如tRNA或其他类别的小RNA。第四是利用信息科学的进步,将核苷酸测序相关的软件和数据库系统置于信息科学发展之中,充分利用领域前沿的新进展,甚至给予优先发展的地位。第五是单分子定性技术的探索性研究。比如运用拉曼增强、原子力显微镜、散射探针显微镜和等离激元光子学(plasmonics)等识别核苷酸在单分子水平的结构与相互作用。这些技术要素涉及诸多纳米技术,包括纳米物理学、纳米化学、纳米材料学和纳米结构设计与加工等。第六是产品工程与工艺改良,包括工程系统优化、组件加工、生产工艺、化学试剂改良和基于市场需求的产品定制等。
5 结语与展望
决定核苷酸测序仪研发的牵引来自于科学发展和预期的应用。地球上的所有物种(包括已知和未知)都有自己特定的基因组,而且每个物种的每个体都对种群的多态性有贡献,这些信息都是科学发展不可或缺的。从人类健康的角度出发,我们也可以根据自己的基因组信息来评价疾病风险、用药风险、健康生活方式等,而这需要获取和存贮每个人的遗传信息。在疾病的诊断和治疗上,我们还需要基因表达和调控的信息;在新生儿出生时,我们需要检测他们是否携带罕见疾病和其他疾病风险。纵览生命科学基础研究(包括生物化学、分子生物学、细胞生物学等)技术的发展轨迹,我们不难看出核苷酸测序技术将成为一个普适性的实验技术,有着广泛的应用前景和价值,需要深度开发其应用。就目前市场的现状而言,还有很多应用没有实现,尤其是在医学方面的应用。仪器的进一步研发和科研、医疗市场的进一步应用可能会使其成为一项永久性产业。微观生物圈的研究目标远大、前途广阔,因此也很容易聚焦成科学与技术发展的前沿。这样的前沿研究往往会发生技术的汇聚和资源的集中。因此,为来自不同层面的技术汇聚提供一个可利用、可深度合作的环境,可以达到事半功倍的作用,如生命科学与物理科学在纳米空间里的汇聚[12]。在拥挤的纳米空间,DNA和RNA以及其建筑单元核苷酸的物理行为已经不再遵从大小和电荷所具备的规律。再如,生命孕育于水,然而我们对水、生物大分子与水、生物结构与水等操作层面中氢键的分布、相互作用等问题一直没有很深刻、很精准的认识,目前物理学家们正在利用现有工具揭示水与水合物的结构关系[13]。此外,生命科学也会很快进入微观生物圈的研究活动,在“五流”(信息流、操作流、平衡流、分室流、可塑流)的思维框架下从多维度(强调时空关系)、多方向(强调目的和途径的关系)、多层次(强调整体和局部的关系)、多功能(强调目标和实施的关系)、多谱系(强调谱系间和物种内的差异)入手来充分认识生命现象和分子基础[14]。
核苷酸测序技术的发展需要不断的孵育和支持,形成联合体[15]或利用大科学工程和项目来推动其发展。DNA测序技术一直在大科学项目的推动下发展,RNA直接测序也需要动力,需要需求明确的大科学项目。可能的出发点是单细胞水平小分子RNA的定量、共价修饰的动态变化等。例如,可以将tRNA组、小RNA组、乃至RNA组研究等作为先导项目。
由于技术平台发展的阶段性和技术从发明到制造成仪器的周期较长,越早参加,发展就会越快。尽管仪器在市场上的存亡是一个渐变的过程,我们目前还没有看到任何一个(代)仪器有取代所有的可能性。除了同类(代)竞争中退出的仪器外,大部分仪器有一个从大众市场退到小众市场的环节,但是同样也有被新来者全部颠覆的可能,因为核苷酸测序仪的诸多共性参数(长读长、高通量、低价格)与未来需求(从单细胞出发、具有单分子分辨率、定量识别RNA共价修饰等)还远没有同时被满足。乐观的一面是诸多学科技术汇聚速度加快,相关学科向生命科学靠近的驱使增强,科学前沿的更新换代在相似和相近领域之间也具有一定的协同性。
技术本身并不是业态,也不是产业。但是,只有置身于两者之中参与竞争、检验和创造价值,技术才有更有活力,才能实现高速度、不间断的进步。核苷酸测序技术可以是仪器和耗材,也就是产品;也可以是服务,成为分子诊断设备的一部分。在科技发展的道路上,对高速发展产业的选择和取舍是非常困难的,但是基本原则应该不可缺失对关键技术的投入。就技术的应用和市场开发而言,核苷酸测序技术在造福人类福祉上主要聚焦于卫生与健康领域,因此应当联合医学院、医院和医生共同开发。就需求而言,核苷酸测序技术具有普适性,在精准医疗的方向上大有作为。市场的核心推动力是基于需求和价值之间的合理价格,核苷酸测序技术以及其他等价技术(如基因分型和PCR)的发展在于不断地挖掘和拓宽其实际应用,有效和合理地分类使用,在政策与需求中找到高速发展的空间。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 1]

A scientific dream proposed some 20 years ago has been realized-the completion of the DNA sequence for the Human Genome Project (HGP) in 2004. As a result, an entirely new field of biological research has arisen: genome biology or genomics is celebrated for its unprecedented scale, intrinsically digital output, and systematic approach to getting all the data.Its sequel, the HapMap Project, will reach fruition later this year. These projects established new precedents for international collaborations and open data access.
URL [本文引用: 1]
URL [本文引用: 1]
URLPMID:4875313 [本文引用: 1]

As one of the key technologies in biomedical research, DNA sequencing has not only improved its productivity with an exponential growth rate but also been applied to new areas of application over the past few years. This is largely due to the advent of newer generations of sequencing platforms, offering ever-faster and cheaper ways to analyze sequences. In our previous review, we looked into technical characteristics of the next-generation sequencers and provided prospective insights into their future development. In this article, we present a brief overview of the advantages and shortcomings of key commercially available platforms with a focus on their suitability for a broad range of applications.
URLPMID:20596955 [本文引用: 1]

As one of the most powerful tools in biomedical research, DNA sequencing not only has been improving its productivity in an exponential growth rate but also been evolving into a new layout of technological territories toward engineering and physical disciplines over the past three decades. In this technical review, we look into technical characteristics of the next-gen sequencers and provide prospective insights into their future development and applications. We envisage that some of the emerging platforms are capable of supporting the $1000 genome and $100 genome goals if given a few years for technical maturation. We also suggest that scientists from China should play an active role in this campaign that will have profound impact on both scientific research and societal healthcare systems.
URLPMID:4411503 [本文引用: 1]

Nanopore-based sequencers, as the fourth-generation DNA sequencing technology, have the potential to quickly and reliably sequence the entire human genome for less than $1000, and possibly for even less than $100. The single-molecule techniques used by this technology allow us to further study the interaction between DNA and protein, as well as between protein and protein. Nanopore analysis opens a new door to molecular biology investigation at the single-molecule scale. In this article, we have reviewed academic achievements in nanopore technology from the past as well as the latest advances, including both biological and solid-state nanopores, and discussed their recent and potential applications.
URL [本文引用: 1]

Historians of the future may well describe 2018 as the year that the world's first functional synthetic eukaryotic genome became a reality. Without the benefit of hindsight, it might be hard to completely grasp the long-term significance of a breakthrough moment in the history of science like this. The role of synthetic biology in the imminent birth of a buddingSaccharomyces cerevisiaeyeast cell carrying 16 man-made chromosomes causes the world of science to teeter on the threshold of a future-defining scientific frontier. The genome-engineering tools and technologies currently being developed to produce the ultimate yeast genome will irreversibly connect the dots between our improved understanding of the fundamentals of a complex cell containing its DNA in a specialised nucleus and the application of bioengineered eukaryotes designed for advanced biomanufacturing of beneficial products. By joining up the dots between the findings and learnings from the internationalSynthetic Yeast Genomeproject (known as theYeast 2.0orSc2.0project) and concurrent advancements in biodesign tools and smart data-intensive technologies, a future world powered by a thriving bioeconomy seems realistic. This global project demonstrates how a collaborative network of dot connectors riven by a tinkerer's indomitable curiosity to understand how things work inside a eukaryotic cell re using cutting-edge biodesign concepts and synthetic biology tools to advance science and to positively frame human futures (i.e. improved quality of life) in a planetary context (i.e. a sustainable environment). Explorations such as this have a rich history of resulting in unexpected discoveries and unanticipated applications for the benefit of people and planet. However, we must learn from past explorations into controversial futuristic sciences and ensure that researchers at the forefront of an emerging science such as synthetic biology remain connected to all stakeholders concerns about the biosafety, bioethics and regulatory aspects of their pioneering work. This article presents a shared vision of constructing a synthetic eukaryotic genome in a safe model organism by using novel concepts and advanced technologies. This multidisciplinary and collaborative project is conducted under a sound governance structure that does not only respect the scientific achievements and lessons from the past, but that is also focussed on leading the present and helping to secure a brighter future for all. The international Synthetic Yeast Genome (Sc2.0) project is discovering the dots that connect basic eukaryotic biology, biodesign, bioengineering and biomanufacturing with a budding bioeconomy of the future.
URLPMID:29509709 [本文引用: 1]

Synthetic biology is a rapidly growing multidisciplinary branch of science which aims to mimic complex biological systems by creating similar forms. Constructing an artificial system requires optimization at the gene and protein levels to allow the formation of entire biological pathways. Advances in cell-free synthetic biology have helped in discovering new genes, proteins, and pathways bypassing the complexity of the complex pathway interactions in living cells. Furthermore, this method is cost- and time-effective with access to the cellular protein factory without the membrane boundaries. The freedom of design, full automation, and mimicking of in vivo systems reveal advantages of synthetic biology that can improve the molecular understanding of processes, relevant for life science applications. In parallel, in vitro approaches have enhanced our understanding of the living system. This review highlights the recent evolution of cell-free gene design, proteins, and cells integrated with microfluidic platforms as a promising technology, which has allowed for the transformation of the concept of bioprocesses. Although several challenges remain, the manipulation of biological synthetic machinery in microfluidic devices as suitable omes for in vitro protein synthesis has been proposed as a pioneering approach for the development of new platforms, relevant in biomedical and diagnostic contexts towards even the sensing and monitoring of environmental issues.
.
URLPMID:4360479 [本文引用: 1]

Abstract As one of the most crucial properties of DNA, the structural stability and the mechanical strength are attracting a great attention. Here, we take advantage of high force resolution and high special resolution of Atom Force Microscope and investigate the mechanical force of DNA duplexes. To evaluate the base pair hydrogen bond strength and base stacking force in DNA strands, we designed two modes (unzipping and stretching) for the measurement rupture forces. Employing k-means clustering algorithm, the ruptured force are clustered and the mean values are estimated. We assessed the influence of experimental parameters and performed the force evaluation for DNA duplexes of pure dG/dC and dA/dT base pairs. The base binding strength of single dG/dC and single dA/dT were estimated to be 20.0 00± 0.2 pN and 14.0 00± 0.3 pN, respectively, and the base stacking interaction was estimated to be 2.0 00± 0.1 pN. Our results provide valuable information about the quantitative evaluation of the mechanical properties of the DNA duplexes.
.
URLPMID:27609897 [本文引用: 1]

We directly measured at the single-molecule level the forces and lifetimes of DNA base-pair stacking interactions for all stack sequence combinations. Our experimental approach combined dual-beam optical tweezers with DNA origami components to allow positioning of blunt-end DNA helices so that the weak stacking force could be isolated. Base-pair stack arrays that lacked a covalent backbone connection spontaneously dissociated at average rates ranging from 0.02 to 500 per second, depending on the sequence combination and stack array size. Forces in the range from 2 to 8 piconewtons that act along the helical direction only mildly accelerated the stochastic unstacking process. The free-energy increments per stack that we estimate from the measured forward and backward kinetic rates ranged from –0.8 to –3.4 kilocalories per mole, depending on the sequence combination. Our data contributes to understanding the mechanics of DNA processing in biology, and it is helpful for designing the kinetics of DNA-based nanoscale devices according to user specifications.
URLPMID:29334379 [本文引用: 1]

Abstract Sequencing the RNA in a biological sample can unlock a wealth of information, including the identity of bacteria and viruses, the nuances of alternative splicing or the transcriptional state of organisms. However, current methods have limitations due to short read lengths and reverse transcription or amplification biases. Here we demonstrate nanopore direct RNA-seq, a highly parallel, real-time, single-molecule method that circumvents reverse transcription or amplification steps. This method yields full-length, strand-specific RNA sequences and enables the direct detection of nucleotide analogs in RNA.
URLPMID:3780799 [本文引用: 2]

78 Nanopores are a recent addition to the toolbox of single-molecule biophysics. 78 Force can be applied to single molecules without using any chemical label or surface immobilization. 78 Nucleic acid transport through small pores is a complex yet highly informative process. 78 Pore-based genotyping/sequencing can revolutionize personalized medicine, forensics, and research.
URLPMID:27081066 [本文引用: 1]

We report the quantitative assessment of nuclear quantum effects on the strength of a single hydrogen bond formed at a water-salt interface, using tip-enhanced inelastic electron tunneling spectroscopy based on a scanning tunneling microscope. The inelastic scattering cross section was resonantly enhanced by “gating” the frontier orbitals of water via a chlorine-terminated tip, so the hydrogen-bonding strength can be determined with high accuracy from the red shift in the oxygen-hydrogen stretching frequency of water. Isotopic substitution experiments combined with quantum simulations reveal that the anharmonic quantum fluctuations of hydrogen nuclei weaken the weak hydrogen bonds and strengthen the relatively strong ones. However, this trend can be completely reversed when a hydrogen bond is strongly coupled to the polar atomic sites of the surface.
.上海交通大学出版社.
URL [本文引用: 1]

随着人类基因组计划的完成、肿瘤基因组计划的实施和一系列靶向药物的出现,肿瘤的诊断与治疗已开始迈入精准医学的时代。该文简要回顾了从人类基因组计划到精准医学计划的历史、肿瘤基因组学和靶向治疗的主要成就,并提出了未来肿瘤研究的方向。
URL [本文引用: 1]
