 ,1,2,*
,1,2,*Advances in techniques and methods of wildlife monitoring
Wen-Hong XIAO1, Qing-Song ZHOU1, Chao-Dong ZHU1, Dong-Hui WU3, Zhi-Shu XIAO ,1,2,*
,1,2,*通讯作者: *xiaozs@ioz.ac.cn
编委: 温学发
责任编辑: 赵航
收稿日期:2019-06-27接受日期:2019-10-14网络出版日期:2020-04-20
| 基金资助: |
Corresponding authors: *xiaozs@ioz.ac.cn
Received:2019-06-27Accepted:2019-10-14Online:2020-04-20
| Fund supported: |

摘要
野生动物是生态系统研究和保护管理的重要生物类群, 对调控生态系统结构和功能, 维持生态系统健康平衡具有显著作用。科学监测数据是野生动物研究、保护、管理决策的前提基础, 但由于传统监测技术的限制, 野生动物的多样性特征及其与环境、生态系统的平衡机制未得到充分关注和研究。随着自动化、智能化、信息化技术的发展及应用, 野生动物监测技术和方法出现较大突破和变革。该文论述了近年来野生动物监测研究领域的重要新技术, 包括红外相机技术、全球定位系统(GPS)追踪技术、DNA条形码技术和高通量测序技术等。通过综合介绍相关的基础概念、基本原理, 该文总结了新技术的应用优势和重大应用进展, 同时探讨了新技术应用中存在的问题, 并对未来野生动物监测技术的发展趋势进行了展望。
关键词:
Abstract
Wildlife as one major group in ecosystem research and conservation management, play a critical role in regulating the structure and function of ecosystems and maintaining the health and balance of ecosystems. Scientific monitoring data are the basis for wildlife research, protection and management decisions. However, the wildlife diversity, their relationship and related mechanisms with the environment and ecosystem balance have been paid insufficient attention due to the limitations of traditional monitoring technologies. With the development and application of automatical and information technologies, wildlife monitoring technology has achieved great breakthroughs and changes. In this paper, we described four new technologies widely used for wildlife monitoring recently, including camera-trapping technology, Global Positioning System (GPS) tracking technology, DNA-barcode technology and next-generation sequencing technology. We introduced the basic concepts and principles, then summarized the advantages and major application progress of these four key technologies as well as the problems existing in the application. Finally, we discussed the trend of the wildlife monitoring technologies.
Keywords:
PDF (952KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
引用本文
肖文宏, 周青松, 朱朝东, 吴东辉, 肖治术. 野生动物监测技术和方法应用进展与展望. 植物生态学报, 2020, 44(4): 409-417. DOI: 10.17521/cjpe.2019.0165
XIAO Wen-Hong, ZHOU Qing-Song, ZHU Chao-Dong, WU Dong-Hui, XIAO Zhi-Shu.
野生动物是生态系统研究和保护管理的重要生物类群, 具有调控生态系统结构和功能, 维持生态系统健康平衡的作用。其中, 脊椎动物、土壤动物以及昆虫等节肢动物类群是目前生物种群监测和生物多样性监测的重要指示类群。
传统的动物监测方法依赖人工观测, 需投入大量的人力, 且受环境条件因素影响大, 和动物的数量密度间很难建立稳定的关系, 降低了结果的可信度(Li et al., 2012)。借助现代先进科技手段, 目前野生动物监测技术和方法突破了时间、环境的限制, 并越来越少地依赖于人工观测, 逐渐向非入侵性、智能化、自动化的方向发展, 减少了对动物及其栖息地的影响, 可以更好地服务于野生动物监测和生态系统研究(Pimm et al., 2015)。
对于野生脊椎动物而言, 传统调查方法通常是通过样线、样方调查搜集动物实体或出现的证据。 由于多数动物善于奔走, 活动隐秘性高, 调查效率低, 往往需要投入大量的人力才能保证结果的可靠, 大规模的人力调查也会惊扰动物的正常生活。研究者一直期盼高效、对动物干扰小的监测技术出现。近些年, 红外相机技术和全球定位系统(GPS)追踪技术在脊椎动物监测中的作用日渐凸显, 采集的物种信息和位置与运动数据更加准确、标准、高效, 减少了对动物及其栖息地的影响(Pimm et al., 2015), 能够更好地服务于野生动物监测和生态系统研究。
然而, 昆虫等无脊椎动物的个体较小, 生活史复杂, 且分布极广。因此, 这些小型动物的多样性和种群监测面临一些明显的挑战(Ollerton et al., 2014)。通常, 可靠的物种鉴定是进行无脊椎动物多样性监测的重要基础, 但如何首先完成快速而有效的类群及种类初筛和初分, 进而进行重点类群分流及精确的物种鉴定, 是国内外无脊椎动物监测与研究不可回避的难题与挑战。目前, 多样性监测工作中节肢动物多样性监测的新方法和新设施的开发研究受到重视和鼓励, 对无脊椎动物多样性的保护有着重要意义(Didham et al., 2010)。随着分子生物学技术的发展, 测序费用的降低, 分子分类技术也逐渐应用在土壤动物和昆虫等无脊椎动物的监测研究中。然而, 无脊椎动物的类群众多, 想要获取较为完善的DNA数据库仍需要大量的人力、财力和时间。目前, 高通量测序正在发展成为这些生物类群监测研究的主要手段。众多的生物信息分析流程也在不断完善分析结果, 逐步提高物种鉴定的准确度。
本文结合近年来野生动物监测需求, 对红外相机技术、GPS追踪技术、DNA条形码技术和高通量测序技术4种重要新技术进行综述, 阐述相关基础概念和基本原理, 总结重大应用进展, 同时探讨各技术方法的优势和监测中存在的问题, 并展望今后野生动物监测技术的发展趋势和应用走向。
1 监测对象及技术分类
1.1 监测对象
1.1.1 野生脊椎动物脊椎动物通常位于食物链的中上层, 对生态系统的其他动植物种有重要调控功能。对其开展有效监测, 不仅有助于掌握种群和分布动态, 推动野生动物保护管理, 同时有利于保护其所在生态系统的生物多样性(Ripple et al., 2014, 2015)。
1.1.2 土壤动物
土壤动物是陆地生态系统最活跃的地下生物组成部分之一, 土壤动物种类和个体数量均十分惊人, 是土壤中重要的消费者和分解者, 具有极大生态价值、环境价值和经济价值。
1.1.3 昆虫及小型节肢动物
昆虫等节肢动物是生物界的一个主要类群, 如目前已记述的昆虫种类达1 020 007种, 约占动物界物种数量的66% (张蕾等, 2011)。虽然个体较小, 但是许多昆虫类群在不同生态系统中发挥着十分重要的生态功能, 如传粉、生物防治和环境指示等(Bonada et al., 2006)。
如非特别注明, 本文中所涉及的脊椎动物主要指陆生哺乳动物, 土壤动物是指生活在土层中的脊椎和节肢动物, 昆虫及小型节肢动物主要指生活在地上的类群, 划分依据是中国生物多样性监测与研究网络的设置。
1.2 技术分类
1.2.1 红外相机技术红外相机是一种自动拍摄设备, 它配有运动传感器或红外传感器, 使野生动物在经过相机的感应区域时利用动物与环境之间的温度差来自动触发拍摄, 用于收集动物照片或视频信息作为动物出现、分布等的证据。红外相机主要应用于陆生大中型兽类和地栖鸟类。
1.2.2 无线电追踪技术
无线电追踪技术是利用无线电信号的传输来定位目标动物佩戴的发射器, 以获取动物的位置信息而达到追踪动物的目的。应用在野生动物监测领域的无线电追踪技术主要包括高频率(VHF)、Argo等卫星和GPS追踪3种类型, 近年也出现了融合了GPS和卫星追踪技术的无线电项圈。无线电追踪技术应用对象主要包括兽类、鸟类、两栖类及爬行动物等脊椎动物。
1.2.3 DNA条形码技术
由Hebert等(2003)首次提出, 使用线粒体细胞色素c氧化酶亚基I (COI)片段作为识别动物的条形码, 根据条形码序列在物种种内和种间遗传变异的差异对物种进行界定, 并与已有条形码序列的物种进行比较来对物种进行鉴定。由于生物类群的不同, 条形码的序列也不尽相同。
1.2.4 DNA宏条形码技术(DNA metabarcoding)
DNA条形码技术与高通量测序技术的结合。通过高通量测序技术对混合样品的PCR扩增产物进行测序, 并通过生物信息学手段分析获得样品内分类操作单元的数量和相对多度, 并以此进行多样性分析。
2 野生动物监测应用进展
2.1 红外相机技术的应用进展
通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(李晟等, 2014; 肖治术等, 2014)。对于大部分猫科动物等一些个体具有独特天然标记的动物, 红外相机拍摄的图像是鉴别其个体的重要依据, 可基于标记-重捕框架获得动物种群数量、密度、存活率、死亡率等种群参数。在准确掌握虎(Panthera tigris)(Wang et al., 2016)、雪豹(Uncia)(Alexander et al., 2015)等珍稀濒危物种的种群数量和变化趋势(Duangchantrasiri et al., 2016), 评估保护的威胁方面发挥了重要作用。例如, 基于红外相机网络的东北虎豹监测平台, 经过多年监测, 弄清了我国东北虎豹及其野生猎物资源情况(Wang et al., 2017, 2018), 为东北虎豹国家公园的建立提供了强大的科学依据, 有效推动了濒危猫科动物的保护与管理。此外, 红外相机监测数据也非常适合占域模型框架, 即在考虑探测率的情况下, 分析目标物种的空间分布以及环境等因素对栖息地选择或分布的影响(MacKenzie et al., 2006; Wang et al., 2018)。红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(Wang et al., 2015; Bu et al., 2016), 群落多样性和动态(Rovero et al., 2014; van der Weyde et al., 2018), 行为模式和特征(Frey et al., 2017), 对比不同区域多个物种分布和多度的动态变化(O’Brien et al., 2010)发挥了优势, 为深入研究生物多样性的变化规律提供了新的视角(肖治术, 2016)。除了数据, 影像本身可以帮助决策者加强科学决策和科学管理, 也可促进与公众之间的信息交流和科学传播。
2.2 GPS追踪技术的应用进展
利用GPS追踪技术可以提供大型、连续、高频率的动物运动数据, 与地表传统的高频率三角测量技术和VHF通过飞行器或Argos卫星多普勒定位方法相比, 具有精确度和可重复性高, 受天气影响小等明显优势(Tomkiewicz et al., 2010)。该技术有效帮助科学家了解动物的去向, 从个体、种群、群落和生态系统多个水平理解动物生态学关键问题, 目前广泛用于兽类和鸟类的行为和运动监测与研究。早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(Millspaugh et al., 2012)。随着GPS和遥感技术的发展, 高频率的动物运动信息结合高分辨率的遥感数据为深入了解环境对动物运动的影响以及运动决策机制(Moorcroft & Lewis, 2006), 精确分析动物家域的分布和利用模式(Horne et al., 2007), 研究栖息地异质性对动物种群动态的影响(Morales et al., 2010)创造了机遇。通过追踪多个个体和多个物种的运动, 拓展对动物个体生活史, 动物社群行为(Nagy et al., 2010), 扩散与定居之间的关系(Jonsson et al., 2016), 种间作用(如捕食者-猎物)的机制(Wilson et al., 2013)和生态系统服务(C?rtes & Uriarte, 2013)的理解, 这种高分辨率的运动数据甚至可以将追踪的动物作为环境传感器以监测生态环境的变化及其产生的生物学影响(Kays et al., 2015), 为了解动物运动背后的行为、生理、进化等生态学机制提供了有力工具。
2.3 DNA条形码技术的应用进展
COI基因片段作为标识物种的DNA条形码, 具有简便、快捷、高效、准确等优点(Hebert et al., 2003)。在最近的10多年里, DNA条形码的应用极大地促进了以昆虫为代表的无脊椎动物的物种研究, 也促进了种类和个体数量巨大但传统分类专家缺乏的土壤动物多样性的研究。与传统形态分类学相比, 以DNA条形码技术为代表的分子分类学能解决以形态为代表的传统分类学所面临的许多难题, 如隐存种、雌雄异形、生活史不同阶段, 且对研究人员的分类学知识要求不多(Hebert & Gregory, 2005)。分子技术不仅为无脊椎动物多样性监测提供了遗传多样性的评估方法, 而且以其快速、易掌握的特点可以在无脊椎动物分类鉴定方面发挥重要作用。Liu等(2017)对扩增引物进行标记, 将若干样品的PCR产物混合进行高通量测序, 并通过生物信息学手段获取每个样品的DNA条形码序列, 能够降低常规DNA条形码的费用, 适用于常规DNA条形码的研究领域, 能够快速、准确、经济地建立参考DNA条形码数据库。
虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(Cristescu, 2014; Gibson et al., 2015)。随着下一代测序(Next-Generation Sequencing, NGS, 既高通量测序) 平台的发展, DNA宏条形码技术也能辅助用于几乎所有生物类群的多样性监测和评估(Ji et al., 2013), 通过短序列片段的高通量测序评估物种数量及其相对多度, 相比DNA条形码效率更高, 成本更低(Yu et al., 2012)。短序列COI、18S、16S、ITS片段可以覆盖包括细菌、真菌、植物、动物等在内的原/真核生物多样性(Taberlet et al., 2012; Yu et al., 2012; Drummond et al., 2015), 并根据测序目标基因片段的不同可对不同营养级生物群落进行分析, 如通过使用动/植物通用条形码片段进行宏条形码测序可以评估植食性/捕食性动物的食谱, 结合肠道微生物群落的研究可以探讨食物网络结构互作关系等信息。
此外, 利用DNA条形码获取物种多样性的方法也得到了发展, Zhang等(2016)将DNA条形码研究中常用的方法集成了一个软件包(BarcodingR), 可以同时对数据集进行多方面的分析并对结果进行比较, 辅助我们获得一个最优结果。Shi等(2018)开发的FuzzyID软件能够对DNA条形码和DNA宏条形码技术获取的基因片段进行准确、快速的物种鉴定, 能够极大地提升通过高通量技术手段获取类似昆虫物种多样性的速率。Jin等(2018)通过“两步法” (two-step barcoding analysis pipeline)利用多个基因片段对形态学鉴定和COI条形码鉴定结果进行辅助校准, 有助于提高鉴定准确率, 提高获知昆虫物种多样性的效率。
2.4 高通量测序技术的应用进展
高通量测序已经成为生态学研究中一个常被提起的专业术语, 描述了许多不同的基于分子的方法论。最常见的高通量测序方法有扩增子测序(metagenetics)和鸟枪法测序(宏基因组学)。这些方法用于研究所有生命体共有的核酸, 并用此识别来自任何生物群落、栖息地和环境(例如, 水、土壤、气态等)的样本中的生物类群及其功能。因此, 高通量测序数据可以作为通用的、环境独立的生物监测系统的基础, 该系统能够通过划分(分类物种或操作分类单元(OTU))和关联(功能相互作用)序列中相应的节点来解决生态上复杂的网络关系。在高通量测序中, 首先从采样环境中分离出DNA (或RNA)。提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段。“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(Carugati et al., 2015; Lanzén et al., 2016))。这种方法也可以用于靶向标记功能性基因, 尤其是参与生物地球化学循环的基因(Gaby & Buckley, 2012; Barbi et al., 2014; Papaspyrou et al., 2014; Li et al., 2015; Lansdown et al., 2016), 并以此来提供生态系统功能的评估。然后对这些系统发育或功能标记基因进行扩增子测序(D’Amore et al., 2016)。相对而言, 宏基因组学在靶向扩增这一步骤, 取而代之的是将提取的DNA随机分裂成能覆盖所有基因的小片段(D’Amore et al., 2016)。然后对它们与扩增遗传扩增子一样进行测序。这避免了宏基因组学扩增步骤中可能发生的潜在的分类学上的偏差(Cotton et al., 2014)。然而, 群落内的全基因覆盖需要提取大量的DNA, 并且宏基因组学的成本目前远高于宏基因测序。
高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(Caporaso et al., 2010; Edgar, 2013; Dumbrell et al., 2016), 相同序列的数量也可以用来估计相对多度(Thomas et al., 2012)。这易受到引物偏好性的影响, 一些引物会优先扩增某些OTU类群, 而使得其他的OTU数量变相地降低。这种偏差可以通过相应的分子方法(Polz & Cavanaugh, 1998; Sipos et al., 2007)减少, 也可以通过量化来判断更为通用的引物(Cotton et al., 2014)。此外, 数据的测序误差、噪声和统计假象(例如极端数据、非正态分布)的问题都被进行了详细研究, 并且有合适的生物信息学方法来处理这些问题(Dumbrell et al., 2016)。一个主要的挑战是如何通过高通量测序序列数据库来识别这些不同DNA序列所代表的物种或者功能。尽管16S rRNA基因数据库富含强大的信息, 但其他基因数据库也可能包含错配的序列或者缺少一些类群的数据(Tedersoo et al., 2011)。因此, 在全球范围建立一个强大、精密和完善的分子序列数据库是迫切需要的。
近年来, 高通量测序技术已经引起极大的关注。其最大的特点是采用单分子读取技术, 数据读取速度更快, 测序通量和测序读长都大大提高。另外, 高通量测序技术不依赖于PCR扩增, 解决了PCR扩增偏向的制约问题。因此, 高通量测序技术可以对无脊椎动物样本中各物种的DNA总量进行推断, 以此对各类群生物量和相对多度进行评估, 将极大地改变生态学家对生物多样性监测方法的认知。高通量测序技术在无脊椎动物多样性监测研究中的发展, 也使得其应用范围在不断扩大, 如可以直接从环境中提取DNA (环境DNA、eDNA)进行高通量测序并通过生物信息学手段(如统计建模(Di Bella et al., 2013)和机器学习(Bohan et al., 2011))重建生态网络, 将极大地促进无脊椎动物多样性监测研究并显著降低研究成本, 也将有助于从更深层次来探究无脊椎动物多样性及其复杂的生态网络。
3 展望
3.1 加强监测技术的交叉融合
野生动物监测是生态系统监测管理和保护的重要内容。随着技术的不断创新发展, 野生动物监测技术和方法出现变革, 逐渐从主要依赖人工观测向自动化、信息化、智能化发展。尤其是近年来, DNA条形码技术和高通量测序技术的运用, 使得对无脊椎动物多样性大规模监测成为了可能, 对无脊椎动物多样性的研究在不断地全面和深入。新技术让我们可以突破时间、环境等因素的限制, 获取高频率、高精确度的科学数据, 实现从动物个体、行为、种群、群落到生态系统多个尺度的观测和研究(图1)。监测技术的发展促进了野生动物的监测与调查, 也相应地促进了一些统计分析方法的发展, 比如目前许多红外相机监测采用基于占域框架的网格布设(Li et al., 2018), 促进了占域模型的应用和完善。图1
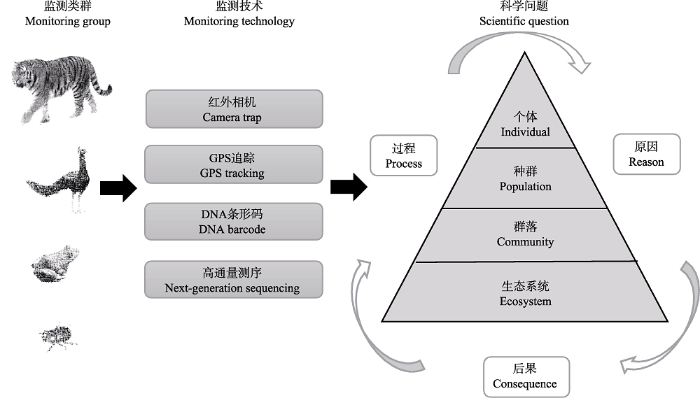 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1野生动物监测新技术的应用示意图。
Fig. 1Application of new technologies for wildlife monitoring.
新的技术方法也存在一些局限性, 比如红外相机和GPS追踪技术多用于数量相对较少, 较难捕捉动物个体的脊椎动物, 而DNA条形码技术更适合较易获得组织样本的无脊椎动物。一些微型设备的价格高昂, 特别是GPS追踪和具有无线传输功能的红外相机需使用卫星或移动蜂窝网络接收或传输数据, 需要支付大笔额外费用, 造成难以获得较多的样本量而最终影响推论的质量(Rodgers, 2001)。DNA宏条形码技术在实际使用过程中存在PCR扩增的偏向性。由于无脊椎动物样品的复杂性和多重性, 单个分子标记难以涵盖所有已知物种序列数据库。目前节肢动物各物种的参考序列数据库仍未建立完善, 很多物种DNA条形码未知, 甚至存在对应物种的错误, 这极大影响了高通量数据的比对分析。因此, 需要加强方法的交叉融合, 弥补相互间的不足, 有效促进野生动物调查的准确性, 达到从分子进化、种群变迁、群落组成等多个层次揭示生态系统维持机制的目的。
3.2 建立监测数据智能化管理平台
在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力。回收的大批量数据也为模型建立、统计分析提供了新的难题。随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(建立综合性监测数据信息服务共享平台将成为未来野生动物监测的发展趋势。该平台可高质量优化集成现有关键监测技术, 同时整合了物联网、智能技术、云计算与大数据等新一代信息技术, 以全面感知、实时传送和智能在线处理为运行方式, 开展多源数据实时采集、网络化、智能化等天地一体化综合观测。通过智能平台可实现数据综合管理、数据综合展示和用户智能化管理(Urbano et al., 2010; Steenweg et al., 2017)。Bohan等(2017)认为在未来10年内将出现一种新的全球规模的生态监测方法, 从而准确、经济、广泛地监测生态系统变化。通过对地球环境中采集的DNA进行高通量测序将会展现出更加丰富的分类操作单元以及生态功能数据, 机器学习方法将依据高通量测序数据重现生态网络内的各种相互作用。并设想开发一种自动采样器, 对核苷酸进行采样并将高通量测序序列数据上传到云系统以重建生态网络, 在全球规模尺度上大量运用这些采样器将实现以空间和时间尺度对地球主要生态系统进行高分辨率生物多样性自动监测, 从而彻底改变我们对生态系统变化的理解。
我们相信, 在研究者、技术开发人员和统计学家思想的不断碰撞下, 野生动物监测数据的采集、管理、分析会更加便利, 为野生动物动态监测和生态系统变化提供快速、准确的预警, 更好地服务于科学家、决策者、保护组织和公众。随着监测技术的完善和规范化应用, 也将促进科学数据的标准化管理和全面使用, 野生动物监测研究将会进入到一个崭新的时代, 为探究动物与环境的作用关系和生态系统的平衡机制提供新的视野和机遇。

参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
DOI:10.1371/journal.pone.0134815URLPMID:26322682 [本文引用: 1]

When densities of large carnivores fall below certain thresholds, dramatic ecological effects can follow, leading to oversimplified ecosystems. Understanding the population status of such species remains a major challenge as they occur in low densities and their ranges are wide. This paper describes the use of non-invasive data collection techniques combined with recent spatial capture-recapture methods to estimate the density of snow leopards Panthera uncia. It also investigates the influence of environmental and human activity indicators on their spatial distribution. A total of 60 camera traps were systematically set up during a three-month period over a 480 km2 study area in Qilianshan National Nature Reserve, Gansu Province, China. We recorded 76 separate snow leopard captures over 2,906 trap-days, representing an average capture success of 2.62 captures/100 trap-days. We identified a total number of 20 unique individuals from photographs and estimated snow leopard density at 3.31 (SE = 1.01) individuals per 100 km2. Results of our simulation exercise indicate that our estimates from the Spatial Capture Recapture models were not optimal to respect to bias and precision (RMSEs for density parameters less or equal to 0.87). Our results underline the critical challenge in achieving sufficient sample sizes of snow leopard captures and recaptures. Possible performance improvements are discussed, principally by optimising effective camera capture and photographic data quality.
DOI:10.1371/journal.pone.0116264URLPMID:25545363 [本文引用: 1]

Plant biomass degradation in soil is one of the key steps of carbon cycling in terrestrial ecosystems. Fungal saprotrophic communities play an essential role in this process by producing hydrolytic enzymes active on the main components of plant organic matter. Open questions in this field regard the diversity of the species involved, the major biochemical pathways implicated and how these are affected by external factors such as litter quality or climate changes. This can be tackled by environmental genomic approaches involving the systematic sequencing of key enzyme-coding gene families using soil-extracted RNA as material. Such an approach necessitates the design and evaluation of gene family-specific PCR primers producing sequence fragments compatible with high-throughput sequencing approaches. In the present study, we developed and evaluated PCR primers for the specific amplification of fungal CAZy Glycoside Hydrolase gene families GH5 (subfamily 5) and GH11 encoding endo-beta-1,4-glucanases and endo-beta-1,4-xylanases respectively as well as Basidiomycota class II peroxidases, corresponding to the CAZy Auxiliary Activity family 2 (AA2), active on lignin. These primers were experimentally validated using DNA extracted from a wide range of Ascomycota and Basidiomycota species including 27 with sequenced genomes. Along with the published primers for Glycoside Hydrolase GH7 encoding enzymes active on cellulose, the newly design primers were shown to be compatible with the Illumina MiSeq sequencing technology. Sequences obtained from RNA extracted from beech or spruce forest soils showed a high diversity and were uniformly distributed in gene trees featuring the global diversity of these gene families. This high-throughput sequencing approach using several degenerate primers constitutes a robust method, which allows the simultaneous characterization of the diversity of different fungal transcripts involved in plant organic matter degradation and may lead to the discovery of complex patterns in gene expression of soil fungal communities.
URLPMID:22242111 [本文引用: 1]
URLPMID:28359573 [本文引用: 1]
URLPMID:16332221 [本文引用: 1]
DOI:10.1371/journal.pone.0164271URLPMID:27723772 [本文引用: 1]

Understanding the interactions between species and their coexistence mechanisms will help explain biodiversity maintenance and enable managers to make sound conservation decisions. Mesocarnivores are abundant and diverse mid-sized carnivores and can have profound impacts on the function, structure and dynamics of ecosystem after the extirpation of apex predators in many ecosystems. The moist temperate forests of Southwest China harbor a diverse community of mesocarnivores in the absence of apex predators. Sympatric species tend to partition limited resources along time, diet and space to facilitate coexistence. We determined the spatial and temporal patterns for five species of mesocarnivores. We used detection histories from a large camera-trap dataset collected from 2004-2015 with an extensive effort of 23,313 camera-days from 495 camera locations. The five mesocarnivore species included masked palm civet Paguma larvata, leopard cat Prionailurus bengalensis, hog badger Arctonyx collaris, yellow-throated marten Martes flavigula, and Siberian weasel Mustela sibirica. Only the masked palm civet and hog badger tended to avoid each other; while for other pairs of species, they occurred independently of each other, or no clear pattern observed. With regard to seasonal activity, yellow-throated marten was most active in winter, opposite the pattern observed for masked palm civet, leopard cat and hog badger. For diel activity, masked palm civet, leopard cat and hog badger were primarily nocturnal and crepuscular; yellow-throated marten was diurnal, and Siberian weasel had no clear pattern for most of the year (March to November), but was nocturnal in the winter (December to February). The seasonal shift of the Siberian weasel may be due to the high diet overlap among species in winter. Our results provided new facts and insights into this unique community of mesocarnivores of southwest China, and will facilitate future studies on the mechanism determining coexistence of animal species within complex system.
URLPMID:20383131 [本文引用: 1]
URLPMID:25957694 [本文引用: 1]
DOI:10.1111/j.1469-185X.2012.00250.xURLPMID:23136896 [本文引用: 1]

General principles about the consequences of seed dispersal by animals for the structure and dynamics of plant populations and communities remain elusive. This is in part because seed deposition patterns emerge from interactions between frugivore behaviour and the distribution of food resources, both of which can vary over space and time. Here we advocate a frugivore-centred, process-based, synthetic approach to seed dispersal research that integrates seed dispersal ecology and animal movement across multiple spatio-temporal scales. To guide this synthesis, we survey existing literature using paradigms from seed dispersal and animal movement. Specifically, studies are discussed with respect to five criteria: selection of focal organisms (animal or plant); measurement of animal movement; characterization of seed shadow; animal, plant and environmental factors included in the study; and scales of the study. Most studies focused on either frugivores or plants and characterized seed shadows directly by combining gut retention time with animal movement data or indirectly by conducting maternity analysis of seeds. Although organismal traits and environmental factors were often measured, they were seldom used to characterize seed shadows. Multi-scale analyses were rare, with seed shadows mostly characterized at fine spatial scales, over single fruiting seasons, and for individual dispersers. Novel animal- and seed-tracking technologies, remote environmental monitoring tools, and advances in analytical methods can enable effective implementation of a hierarchical mechanistic approach to the study of seed dispersal. This kind of mechanistic approach will provide novel insights regarding the complex interplay between the factors that modulate animal behaviour and subsequently influence seed dispersal patterns across spatial and temporal scales.
URLPMID:25275629 [本文引用: 2]
URLPMID:25175416 [本文引用: 1]
DOI:10.1186/s12864-015-2194-9URLPMID:26763898 [本文引用: 2]

BACKGROUND: In the last 5 years, the rapid pace of innovations and improvements in sequencing technologies has completely changed the landscape of metagenomic and metagenetic experiments. Therefore, it is critical to benchmark the various methodologies for interrogating the composition of microbial communities, so that we can assess their strengths and limitations. The most common phylogenetic marker for microbial community diversity studies is the 16S ribosomal RNA gene and in the last 10 years the field has moved from sequencing a small number of amplicons and samples to more complex studies where thousands of samples and multiple different gene regions are interrogated. RESULTS: We assembled 2 synthetic communities with an even (EM) and uneven (UM) distribution of archaeal and bacterial strains and species, as metagenomic control material, to assess performance of different experimental strategies. The 2 synthetic communities were used in this study, to highlight the limitations and the advantages of the leading sequencing platforms: MiSeq (Illumina), The Pacific Biosciences RSII, 454 GS-FLX/+ (Roche), and IonTorrent (Life Technologies). We describe an extensive survey based on synthetic communities using 3 experimental designs (fusion primers, universal tailed tag, ligated adaptors) across the 9 hypervariable 16S rDNA regions. We demonstrate that library preparation methodology can affect data interpretation due to different error and chimera rates generated during the procedure. The observed community composition was always biased, to a degree that depended on the platform, sequenced region and primer choice. However, crucially, our analysis suggests that 16S rRNA sequencing is still quantitative, in that relative changes in abundance of taxa between samples can be recovered, despite these biases. CONCLUSION: We have assessed a range of experimental conditions across several next generation sequencing platforms using the most up-to-date configurations. We propose that the choice of sequencing platform and experimental design needs to be taken into consideration in the early stage of a project by running a small trial consisting of several hypervariable regions to quantify the discriminatory power of each region. We also suggest that the use of a synthetic community as a positive control would be beneficial to identify the potential biases and procedural drawbacks that may lead to data misinterpretation. The results of this study will serve as a guideline for making decisions on which experimental condition and sequencing platform to consider to achieve the best microbial profiling.
URLPMID:24029734 [本文引用: 1]
[本文引用: 1]
URLPMID:26445670 [本文引用: 1]
URLPMID:27153529 [本文引用: 1]
//McGenity TJ, Timmis KN, Nogales B.
[本文引用: 2]
DOI:10.1038/NMETH.2604URL [本文引用: 1]

Amplified marker-gene sequences can be used to understand microbial community structure, but they suffer from a high level of sequencing and amplification artifacts. The UPARSE pipeline reports operational taxonomic unit (OTU) sequences with <= 1% incorrect bases in artificial microbial community tests, compared with >3% incorrect bases commonly reported by other methods. The improved accuracy results in far fewer OTUs, consistently closer to the expected number of species in a community.
[本文引用: 1]
URLPMID:22848735 [本文引用: 1]
[本文引用: 1]
URLPMID:26488407 [本文引用: 1]
[本文引用: 2]
URLPMID:16243770 [本文引用: 1]
URLPMID:17918412 [本文引用: 1]
URLPMID:23910579 [本文引用: 1]
[本文引用: 1]
URLPMID:26852171 [本文引用: 1]
URLPMID:26068858 [本文引用: 1]
[本文引用: 1]
DOI:10.1038/srep28257URLPMID:27321429 [本文引用: 1]

Mountain elevation gradients are invaluable sites for understanding the effects of climate change on ecosystem function, community structure and distribution. However, relatively little is known about the impact on soil microbial communities, in spite of their importance for the functioning of the soil ecosystem. Previous studies of microbial diversity along elevational gradients were often limited by confounding variables such as vegetation, pH, and nutrients. Here, we utilised a transect in the Pyrenees established to minimise variation in such parameters, to examine prokaryotic, fungal, protist and metazoan communities throughout three consecutive years. We aimed to determine the influences of climate and environmental parameters on soil microbial community structure; as well as on the relationships between those microbial communities. Further, functional diversity of heterotrophic bacteria was determined using Biolog. Prokaryotic and fungal community structure, but not alpha-diversity, correlated significantly with elevation. However, carbon-to-nitrogen ratio and pH appeared to affect prokaryotic and protist communities more strongly. Both community structure and physicochemical parameters varied considerably between years, illustrating the value of long-term monitoring of the dynamic processes controlling the soil ecosystem. Our study also illustrates both the challenges and strengths of using microbial communities as indicators of potential impacts of climate change.
DOI:10.1128/AEM.02654-14URLPMID:25326303 [本文引用: 1]

Nitrification, mediated by ammonia-oxidizing bacteria (AOB) and ammonia-oxidizing archaea (AOA), is important in global nitrogen cycling. In estuaries where gradients of salinity and ammonia concentrations occur, there may be differential selections for ammonia-oxidizer populations. The aim of this study was to examine the activity, abundance, and diversity of AOA and AOB in surface oxic sediments of a highly nutrified estuary that exhibits gradients of salinity and ammonium. AOB and AOA communities were investigated by measuring ammonia monooxygenase (amoA) gene abundance and nitrification potentials both spatially and temporally. Nitrification potentials differed along the estuary and over time, with the greatest nitrification potentials occurring mid-estuary (8.2 mumol N grams dry weight [gdw](-1) day(-1) in June, increasing to 37.4 mumol N gdw(-1) day(-1) in January). At the estuary head, the nitrification potential was 4.3 mumol N gdw(-1) day(-1) in June, increasing to 11.7 mumol N gdw(-1) day(-1) in January. At the estuary head and mouth, nitrification potentials fluctuated throughout the year. AOB amoA gene abundances were significantly greater (by 100-fold) than those of AOA both spatially and temporally. Nitrosomonas spp. were detected along the estuary by denaturing gradient gel electrophoresis (DGGE) band sequence analysis. In conclusion, AOB dominated over AOA in the estuarine sediments, with the ratio of AOB/AOA amoA gene abundance increasing from the upper (freshwater) to lower (marine) regions of the Colne estuary. These findings suggest that in this nutrified estuary, AOB (possibly Nitrosomonas spp.) were of major significance in nitrification.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:29048539 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:20376149 [本文引用: 1]
DOI:10.1111/acv.2010.13.issue-4URL [本文引用: 1]
DOI:10.1126/science.1257259URLPMID:25504719 [本文引用: 1]

Pollinators are fundamental to maintaining both biodiversity and agricultural productivity, but habitat destruction, loss of flower resources, and increased use of pesticides are causing declines in their abundance and diversity. Using historical records, we assessed the rate of extinction of bee and flower-visiting wasp species in Britain from the mid-19th century to the present. The most rapid phase of extinction appears to be related to changes in agricultural policy and practice beginning in the 1920s, before the agricultural intensification prompted by the Second World War, often cited as the most important driver of biodiversity loss in Britain. Slowing of the extinction rate from the 1960s onward may be due to prior loss of the most sensitive species and/or effective conservation programs.
DOI:10.1371/journal.pone.0094111URLPMID:24728381 [本文引用: 1]

Denitrification and dissimilatory nitrate reduction to ammonium (DNRA) are processes occurring simultaneously under oxygen-limited or anaerobic conditions, where both compete for nitrate and organic carbon. Despite their ecological importance, there has been little investigation of how denitrification and DNRA potentials and related functional genes vary vertically with sediment depth. Nitrate reduction potentials measured in sediment depth profiles along the Colne estuary were in the upper range of nitrate reduction rates reported from other sediments and showed the existence of strong decreasing trends both with increasing depth and along the estuary. Denitrification potential decreased along the estuary, decreasing more rapidly with depth towards the estuary mouth. In contrast, DNRA potential increased along the estuary. Significant decreases in copy numbers of 16S rRNA and nitrate reducing genes were observed along the estuary and from surface to deeper sediments. Both metabolic potentials and functional genes persisted at sediment depths where porewater nitrate was absent. Transport of nitrate by bioturbation, based on macrofauna distributions, could only account for the upper 10 cm depth of sediment. A several fold higher combined freeze-lysable KCl-extractable nitrate pool compared to porewater nitrate was detected. We hypothesised that his could be attributed to intracellular nitrate pools from nitrate accumulating microorganisms like Thioploca or Beggiatoa. However, pyrosequencing analysis did not detect any such organisms, leaving other bacteria, microbenthic algae, or foraminiferans which have also been shown to accumulate nitrate, as possible candidates. The importance and bioavailability of a KCl-extractable nitrate sediment pool remains to be tested. The significant variation in the vertical pattern and abundance of the various nitrate reducing genes phylotypes reasonably suggests differences in their activity throughout the sediment column. This raises interesting questions as to what the alternative metabolic roles for the various nitrate reductases could be, analogous to the alternative metabolic roles found for nitrite reductases.
URLPMID:26437636 [本文引用: 2]
[本文引用: 1]
DOI:10.1126/science.1241484URLPMID:24408439 [本文引用: 1]

Large carnivores face serious threats and are experiencing massive declines in their populations and geographic ranges around the world. We highlight how these threats have affected the conservation status and ecological functioning of the 31 largest mammalian carnivores on Earth. Consistent with theory, empirical studies increasingly show that large carnivores have substantial effects on the structure and function of diverse ecosystems. Significant cascading trophic interactions, mediated by their prey or sympatric mesopredators, arise when some of these carnivores are extirpated from or repatriated to ecosystems. Unexpected effects of trophic cascades on various taxa and processes include changes to bird, mammal, invertebrate, and herpetofauna abundance or richness; subsidies to scavengers; altered disease dynamics; carbon sequestration; modified stream morphology; and crop damage. Promoting tolerance and coexistence with large carnivores is a crucial societal challenge that will ultimately determine the fate of Earth's largest carnivores and all that depends upon them, including humans.
DOI:10.1126/sciadv.1400103URLPMID:26601172 [本文引用: 1]

Large wild herbivores are crucial to ecosystems and human societies. We highlight the 74 largest terrestrial herbivore species on Earth (body mass >/=100 kg), the threats they face, their important and often overlooked ecosystem effects, and the conservation efforts needed to save them and their predators from extinction. Large herbivores are generally facing dramatic population declines and range contractions, such that ~60% are threatened with extinction. Nearly all threatened species are in developing countries, where major threats include hunting, land-use change, and resource depression by livestock. Loss of large herbivores can have cascading effects on other species including large carnivores, scavengers, mesoherbivores, small mammals, and ecological processes involving vegetation, hydrology, nutrient cycling, and fire regimes. The rate of large herbivore decline suggests that ever-larger swaths of the world will soon lack many of the vital ecological services these animals provide, resulting in enormous ecological and social costs.
[本文引用: 1]
URLPMID:25054806 [本文引用: 1]
DOI:10.1111/1755-0998.12738URLPMID:29154499 [本文引用: 1]

Species identification through DNA barcoding or metabarcoding has become a key approach for biodiversity evaluation and ecological studies. However, the rapid accumulation of barcoding data has created some difficulties: for instance, global enquiries to a large reference library can take a very long time. We here devise a two-step searching strategy to speed identification procedures of such queries. This firstly uses a Hidden Markov Model (HMM) algorithm to narrow the searching scope to genus level and then determines the corresponding species using minimum genetic distance. Moreover, using a fuzzy membership function, our approach also estimates the credibility of assignment results for each query. To perform this task, we developed a new software pipeline, FuzzyID2, using Python and C++. Performance of the new method was assessed using eight empirical data sets ranging from 70 to 234,535 barcodes. Five data sets (four animal, one plant) deployed the conventional barcode approach, one used metabarcodes, and two were eDNA-based. The results showed mean accuracies of generic and species identification of 98.60% (with a minimum of 95.00% and a maximum of 100.00%) and 94.17% (with a range of 84.40%-100.00%), respectively. Tests with simulated NGS sequences based on realistic eDNA and metabarcode data demonstrated that FuzzyID2 achieved a significantly higher identification success rate than the commonly used Blast method, and the TIPP method tends to find many fewer species than either FuzztID2 or Blast. Furthermore, data sets with tens of thousands of barcodes need only a few seconds for each query assignment using FuzzyID2. Our approach provides an efficient and accurate species identification protocol for biodiversity-related projects with large DNA sequence data sets.
URLPMID:2083845 [本文引用: 1]
DOI:10.1002/fee.2017.15.issue-1URL [本文引用: 1]
URLPMID:22486824 [本文引用: 1]
URLPMID:21949797 [本文引用: 1]
URLPMID:22587947 [本文引用: 1]
DOI:10.1098/rstb.2010.0090URL [本文引用: 1]
DOI:10.1098/rstb.2010.0081URL [本文引用: 1]
DOI:10.1016/j.biocon.2018.01.033URL [本文引用: 1]
DOI:10.1016/j.biocon.2015.03.032URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
URLPMID:23765495 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
Face value: towards robust estimates of snow leopard densities
1
2015
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
PCR primers to study the diversity of expressed fungal genes encoding lignocellulolytic enzymes in soils using high-throughput sequencing
1
2014
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
Automated discovery of food webs from ecological data using logic-based machine learning
1
2011
... 近年来, 高通量测序技术已经引起极大的关注.其最大的特点是采用单分子读取技术, 数据读取速度更快, 测序通量和测序读长都大大提高.另外, 高通量测序技术不依赖于PCR扩增, 解决了PCR扩增偏向的制约问题.因此, 高通量测序技术可以对无脊椎动物样本中各物种的DNA总量进行推断, 以此对各类群生物量和相对多度进行评估, 将极大地改变生态学家对生物多样性监测方法的认知.高通量测序技术在无脊椎动物多样性监测研究中的发展, 也使得其应用范围在不断扩大, 如可以直接从环境中提取DNA (环境DNA、eDNA)进行高通量测序并通过生物信息学手段(如统计建模(
Next-generation global biomonitoring: large-scale, automated reconstruction of ecological networks
1
2017
... 建立综合性监测数据信息服务共享平台将成为未来野生动物监测的发展趋势.该平台可高质量优化集成现有关键监测技术, 同时整合了物联网、智能技术、云计算与大数据等新一代信息技术, 以全面感知、实时传送和智能在线处理为运行方式, 开展多源数据实时采集、网络化、智能化等天地一体化综合观测.通过智能平台可实现数据综合管理、数据综合展示和用户智能化管理(
Developments in aquatic insect biomonitoring: a comparative analysis of recent approaches
1
2006
... 昆虫等节肢动物是生物界的一个主要类群, 如目前已记述的昆虫种类达1 020 007种, 约占动物界物种数量的66% (
Spatial co-occurrence and activity patterns of mesocarnivores in the temperate forests of Southwest China
1
2016
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
QIIME allows analysis of high-throughput community sequencing data
1
2010
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Metagenetic tools for the census of marine meiofaunal biodiversity: an overview
1
2015
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
Integrating frugivory and animal movement: a review of the evidence and implications for scaling seed dispersal
1
2013
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
What goes in must come out: testing for biases in molecular analysis of arbuscular mycorrhizal fungal communities
2
2014
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
From barcoding single individuals to metabarcoding biological communities: towards an integrative approach to the study of global biodiversity
1
2014
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
A comprehensive benchmarking study of protocols and sequencing platforms for 16S rRNA community profiling
2
2016
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
... ).相对而言, 宏基因组学在靶向扩增这一步骤, 取而代之的是将提取的DNA随机分裂成能覆盖所有基因的小片段(
High throughput sequencing methods and analysis for microbiome research
1
2013
... 近年来, 高通量测序技术已经引起极大的关注.其最大的特点是采用单分子读取技术, 数据读取速度更快, 测序通量和测序读长都大大提高.另外, 高通量测序技术不依赖于PCR扩增, 解决了PCR扩增偏向的制约问题.因此, 高通量测序技术可以对无脊椎动物样本中各物种的DNA总量进行推断, 以此对各类群生物量和相对多度进行评估, 将极大地改变生态学家对生物多样性监测方法的认知.高通量测序技术在无脊椎动物多样性监测研究中的发展, 也使得其应用范围在不断扩大, 如可以直接从环境中提取DNA (环境DNA、eDNA)进行高通量测序并通过生物信息学手段(如统计建模(
Research needs in insect conservation and diversity
1
2010
... 然而, 昆虫等无脊椎动物的个体较小, 生活史复杂, 且分布极广.因此, 这些小型动物的多样性和种群监测面临一些明显的挑战(
Evaluating a multigene environmental DNA approach for biodiversity assessment
1
2015
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
Dynamics of a low-density tiger population in Southeast Asia in the context of improved law enforcement
1
2016
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
Microbial community analysis by single-amplicon high-throughput next generation sequencing: data analysis—From raw output to ecology
2
2016
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
... ).此外, 数据的测序误差、噪声和统计假象(例如极端数据、非正态分布)的问题都被进行了详细研究, 并且有合适的生物信息学方法来处理这些问题(
UPARSE: highly accurate OTU sequences from microbial amplicon reads
1
2013
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Investigating animal activity patterns and temporal niche partitioning using camera-trap data: challenges and opportunities
1
2017
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
A comprehensive evaluation of PCR primers to amplify the nifH gene of nitrogenase
1
2012
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
Automated species identification: Why not?
1
2004
... 在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力.回收的大批量数据也为模型建立、统计分析提供了新的难题.随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(
Large-scale biomonitoring of remote and threatened ecosystems via high-throughput sequencing
1
2015
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
Biological identifications through DNA barcodes
2
2003
... 由
... COI基因片段作为标识物种的DNA条形码, 具有简便、快捷、高效、准确等优点(
The promise of DNA barcoding for taxonomy
1
2005
... 与传统形态分类学相比, 以DNA条形码技术为代表的分子分类学能解决以形态为代表的传统分类学所面临的许多难题, 如隐存种、雌雄异形、生活史不同阶段, 且对研究人员的分类学知识要求不多(
Analyzing animal movements using Brownian bridges
1
2007
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding
1
2013
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
A two-step DNA barcoding approach for delimiting moth species: moths of Dongling Mountain (Beijing, China) as a case study
1
2018
... 此外, 利用DNA条形码获取物种多样性的方法也得到了发展,
Tracking animal dispersal: from individual movement to community assembly and global range dynamics
1
2016
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
Terrestrial animal tracking as an eye on life and planet
1
2015
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
Importance and controls of anaerobic ammonium oxidation influenced by riverbed geology
1
2016
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
Multi-targeted metagenetic analysis of the influence of climate and environmental parameters on soil microbial communities along an elevational gradient
1
2016
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
amoA gene abundances and nitrification potential rates suggest that benthic ammonia- oxidizing bacteria not archaea dominate N cycling in the Colne Estuary, United Kingdom
1
2015
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
A direct comparison of camera-trapping and sign transects for monitoring wildlife in the Wanglang National Nature Reserve, China
1
2012
... 传统的动物监测方法依赖人工观测, 需投入大量的人力, 且受环境条件因素影响大, 和动物的数量密度间很难建立稳定的关系, 降低了结果的可信度(
红外相机技术在我国野生动物研究与保护中的应用与前景
1
2014
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
红外相机技术在我国野生动物研究与保护中的应用与前景
1
2014
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
Using large spatial scale camera trap data and hierarchical occupancy models to evaluate species richness and occupancy of rare and elusive wildlife communities in southwest china
1
2018
... 野生动物监测是生态系统监测管理和保护的重要内容.随着技术的不断创新发展, 野生动物监测技术和方法出现变革, 逐渐从主要依赖人工观测向自动化、信息化、智能化发展.尤其是近年来, DNA条形码技术和高通量测序技术的运用, 使得对无脊椎动物多样性大规模监测成为了可能, 对无脊椎动物多样性的研究在不断地全面和深入.新技术让我们可以突破时间、环境等因素的限制, 获取高频率、高精确度的科学数据, 实现从动物个体、行为、种群、群落到生态系统多个尺度的观测和研究(
Filling reference gaps via assembling DNA barcodes using high-throughput sequencing—Moving toward barcoding the world
1
2017
... 与传统形态分类学相比, 以DNA条形码技术为代表的分子分类学能解决以形态为代表的传统分类学所面临的许多难题, 如隐存种、雌雄异形、生活史不同阶段, 且对研究人员的分类学知识要求不多(
Insect species recognition using discriminative local soft coding
1
2012
... 在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力.回收的大批量数据也为模型建立、统计分析提供了新的难题.随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(
1
2006
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
1
2012
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
1
2006
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
Building the bridge between animal movement and population dynamics
1
2010
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
Hierarchical group dynamics in pigeon flocks
1
2010
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
The wildlife picture index: monitoring top trophic levels
1
2010
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
Extinctions of aculeate pollinators in Britain and the role of large-scale agricultural changes
1
2014
... 然而, 昆虫等无脊椎动物的个体较小, 生活史复杂, 且分布极广.因此, 这些小型动物的多样性和种群监测面临一些明显的挑战(
Nitrate reduction functional genes and nitrate reduction potentials persist in deeper estuarine sediments. Why?
1
2014
... 在高通量测序中, 首先从采样环境中分离出DNA (或RNA).提取DNA后, 通过宏基因组技术使用基因特异性引物来扩增目标系统发育或功能标记基因片段.“通用引物”适用于扩增生物体中所有3个主要的系统遗传信息标记基因(例如16S或18S rRNA基因(
Emerging technologies to conserve biodiversity
2
2015
... 传统的动物监测方法依赖人工观测, 需投入大量的人力, 且受环境条件因素影响大, 和动物的数量密度间很难建立稳定的关系, 降低了结果的可信度(
... 对于野生脊椎动物而言, 传统调查方法通常是通过样线、样方调查搜集动物实体或出现的证据. 由于多数动物善于奔走, 活动隐秘性高, 调查效率低, 往往需要投入大量的人力才能保证结果的可靠, 大规模的人力调查也会惊扰动物的正常生活.研究者一直期盼高效、对动物干扰小的监测技术出现.近些年, 红外相机技术和全球定位系统(GPS)追踪技术在脊椎动物监测中的作用日渐凸显, 采集的物种信息和位置与运动数据更加准确、标准、高效, 减少了对动物及其栖息地的影响(
Biasin template-to-product ratios in multi template PCR
1
1998
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Status and ecological effects of the world’s largest carnivores
1
2014
... 脊椎动物通常位于食物链的中上层, 对生态系统的其他动植物种有重要调控功能.对其开展有效监测, 不仅有助于掌握种群和分布动态, 推动野生动物保护管理, 同时有利于保护其所在生态系统的生物多样性(
Collapse of the world’s largest herbivores
1
2015
... 脊椎动物通常位于食物链的中上层, 对生态系统的其他动植物种有重要调控功能.对其开展有效监测, 不仅有助于掌握种群和分布动态, 推动野生动物保护管理, 同时有利于保护其所在生态系统的生物多样性(
1
2001
... 新的技术方法也存在一些局限性, 比如红外相机和GPS追踪技术多用于数量相对较少, 较难捕捉动物个体的脊椎动物, 而DNA条形码技术更适合较易获得组织样本的无脊椎动物.一些微型设备的价格高昂, 特别是GPS追踪和具有无线传输功能的红外相机需使用卫星或移动蜂窝网络接收或传输数据, 需要支付大笔额外费用, 造成难以获得较多的样本量而最终影响推论的质量(
Estimating species richness and modelling habitat preferences of tropical forest mammals from camera trap data
1
2014
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
FuzzyID2: a software package for large data set species identification via barcoding and metabarcoding using hidden Markov models and fuzzy set methods
1
2018
... 此外, 利用DNA条形码获取物种多样性的方法也得到了发展,
Effect of primer mismatch, annealing temperature and PCR cycle number on 16S rRNA gene-targetting bacterial community analysis
1
2007
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Scaling-up camera traps: monitoring the planet’s biodiversity with networks of remote sensors
1
2017
... 建立综合性监测数据信息服务共享平台将成为未来野生动物监测的发展趋势.该平台可高质量优化集成现有关键监测技术, 同时整合了物联网、智能技术、云计算与大数据等新一代信息技术, 以全面感知、实时传送和智能在线处理为运行方式, 开展多源数据实时采集、网络化、智能化等天地一体化综合观测.通过智能平台可实现数据综合管理、数据综合展示和用户智能化管理(
Towards next-generation biodiversity assessment using DNA metabarcoding
1
2012
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
Tidying up international nucleotide sequence databases: ecological, geographical and sequence quality annotation of ITS sequences of mycorrhizal fungi
1
2011
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Metagenomics—A guide from sampling to data analysis
1
2012
... 高通量测序的数据以序列间的差异来提供分类学或功能多样性的评估标准(
Global positioning system and associated technologies in animal behaviour and ecological research
1
2010
... 利用GPS追踪技术可以提供大型、连续、高频率的动物运动数据, 与地表传统的高频率三角测量技术和VHF通过飞行器或Argos卫星多普勒定位方法相比, 具有精确度和可重复性高, 受天气影响小等明显优势(
Wildlife tracking data management: a new vision
1
2010
... 建立综合性监测数据信息服务共享平台将成为未来野生动物监测的发展趋势.该平台可高质量优化集成现有关键监测技术, 同时整合了物联网、智能技术、云计算与大数据等新一代信息技术, 以全面感知、实时传送和智能在线处理为运行方式, 开展多源数据实时采集、网络化、智能化等天地一体化综合观测.通过智能平台可实现数据综合管理、数据综合展示和用户智能化管理(
Multi-species occupancy modelling of a carnivore guild in wildlife management areas in the Kalahari
1
2018
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
Shared resources between giant panda and sympatric wild and domestic mammals
1
2015
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
Amur tigers and leopards returning to China: direct evidence and a landscape conservation plan
1
2016
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
A science-based approach to guide amur leopard recovery in china
1
2017
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
Living on the edge: opportunities for Amur tiger recovery in China
2
2018
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
... ;
Locomotion dynamics of hunting in wild cheetahs
1
2013
... 早期的动物追踪数据主要用于解答动物家域和栖息地偏好等问题(
红外相机技术促进我国自然保护区野生动物资源编目调查
1
2016
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
红外相机技术促进我国自然保护区野生动物资源编目调查
1
2016
... 红外相机可同时监测多个物种, 在研究种间关系(如捕食者-猎物, 同域物种)时空作用机制(
红外相机技术在我国野生动物监测研究中的应用
1
2014
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
红外相机技术在我国野生动物监测研究中的应用
1
2014
... 通过红外相机所获得动物的影像, 可以更准确地辨识物种, 近年来被广泛应用于动物种群和群落相关的研究中(
An insect imaging system to automate rice light-trap pest identification
1
2012
... 在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力.回收的大批量数据也为模型建立、统计分析提供了新的难题.随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(
Biodiversity soup: metabarcoding of arthropods for rapid biodiversity assessment and biomonitoring
2
2012
... 虽然对个体标本的鉴定依然重要, 但DNA条形码已经开始应用于对整个群落生物的鉴定(
... ;
BarcodingR: an integrated R package for species identification using DNA barcodes
1
2016
... 此外, 利用DNA条形码获取物种多样性的方法也得到了发展,
实蝇科果实蝇属昆虫数字图像自动识别系统的构建和测试
2
2011
... 昆虫等节肢动物是生物界的一个主要类群, 如目前已记述的昆虫种类达1 020 007种, 约占动物界物种数量的66% (
... 在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力.回收的大批量数据也为模型建立、统计分析提供了新的难题.随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(
实蝇科果实蝇属昆虫数字图像自动识别系统的构建和测试
2
2011
... 昆虫等节肢动物是生物界的一个主要类群, 如目前已记述的昆虫种类达1 020 007种, 约占动物界物种数量的66% (
... 在实际应用中, 野生动物监测新技术与新方法带来的最突出的问题是高效率、高密度的数据采集方式产生了大量的数据, 但数据后期的清洗、筛选、管理、整合和分析目前主要依靠人工完成, 比如数以万计的红外相机图片的物种鉴别, 大批量追踪信息的过滤需要投入大量的人力.回收的大批量数据也为模型建立、统计分析提供了新的难题.随着现代计算机技术的高速发展(如处理性能的飞速提高、并行计算技术的成熟), 模式识别理论的日益完善, 大规模数据库系统和人工智能技术在专业领域的成功应用, 网络和数据检索技术的完善等, 逐渐出现“世界有害实蝇自动识别系统”、世界鞘翅目昆虫智能分类系统(甲天下(BICS) v0.1)(
