大豆(Glycine max (L.) Merr.)是世界上食用植物油和植物蛋白的主要来源,生长过程中受多种病害侵染。其中,大豆花叶病毒(soybean mosaic virus,SMV)病对大豆的产量和品质均可造成严重危害。由于SMV分布广、危害重、化学药剂难以防治,一直是大豆种植面临的主要问题。选育和种植抗病品种是控制该病最经济、有效且对环境友好的防治方法。
筛选新的抗病种质并发掘新的抗病基因是拓宽大豆抗病遗传基础、成功培育抗SMV品种的关键。国家和各省大豆新品种审定实行SMV抗性“一票否决”制[1],对抗SMV育种起到了极大的推进作用。随着大豆抗SMV研究的深入,王大刚等[2]、李凯等[3]曾对国内外****在SMV抗源筛选、抗性遗传方式和抗病基因分子标记定位等方面的研究进行了阐述。
本文主要聚焦SMV抗病基因的标记定位、候选抗病基因的分析及抗SMV相关基因的功能鉴定和参与的调控网络等研究,通过系统的梳理和总结,以期为大豆分子设计育种、抗SMV功能基因的利用和新基因的发掘提供参考。
1 大豆对SMV抗病基因的标记定位
大豆对SMV的抗性主要存在数量抗性和质量抗性两类[4]。数量抗性遗传主要由1对加性主基因+加性-显性多基因共同控制[5],利用中豆29×中豆32的重组自交系RIL(recombinant inbred lines,RIL)群体,郭丹丹等[6]研究还发现大豆对SMV株系SC3的数量抗性遗传由2对等加性主基因控制。大豆对SMV质量抗性遗传,国内外普遍认为是由1对显性抗病基因控制[7,8,9,10,11]。通过抗病品种与感病品种的正反交组合研究发现,大白麻对SMV株系SC4及齐黄22对SC8均是由单显性核基因控制,且抗性不受细胞质影响[12]。迄今为止,已经命名和定位的大豆抗SMV单显性基因位点主要有“Rsv”(resistance of soybean mosaic virus,Rsv)系列和“RSC”(resistance of soybean mosaic virus in China,RSC)系列两类。其中“Rsv”系列抗病基因位点主要有Rsv1、Rsv3、Rsv4和Rsv5等,由美国、加拿大和韩国等****鉴定、命名和定位[13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28](表1);“RSC”系列抗病基因位点主要有RSC3Q、RSC4—RSC8、RSC10—RSC12、RSC14Q、RSC15、RSC17—RSC18、RSC20等,由中国和印度****鉴定、命名和定位[27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47](表1)。
Table 1
表1
表1大豆花叶病毒(SMV)抗病基因位点的标记定位
Table 1The markers location of resistance gene loci to soybean mosaic virus in soybean
| 抗病基因位点 Resistance gene locus | 染色体 Chr. | 亲本及群体 Parent and population | 侧翼标记及物理距离 The flanking markers and physical distance (kb) | 参考文献 Reference |
|---|---|---|---|---|
| Rsv1 | 13 | J05×Essex F2 | Sat_154-3296.6-Satt510 | [25] |
| Rsv3 | 14 | L29×Sowon F2 | S156l-207.5-BS141417 | [26] |
| Rsv4 | 2 | Sowon×V94-5152 BC3F2 | Rat2-119.4-S6ac | [27] |
| Rsv5 | 13 | PI96983×York F2 | Rsv1-440.0-Rsv5 | [28] |
| RSC3Q | 13 | Qihuang No.1×Nannong1138-2 F2 | BS131114-649.7-BS131136 | [39] |
| RSC4 | 14 | Dabaima×Nannong1138-2 F2 | BS141413-63.3-BS141416 | [34] |
| RSC5 | 2 | Kefeng No.1×Nannong1138-2 RIL | Bin352-750.0-Bin353 | [44] |
| RSC6 | 2 | Q0923×Nannong1138-2 F2 | BS020617-50.5-BS020621 | [38] |
| RSC7 | 2 | Kefeng No.1×Nannong1138-2 RIL | BS020621-157.1-BS020632 | [41] |
| RSC8 | 2 | Kefeng No.1×Nannong1138-2 RIL | ZL-42-30.7-ZL-52 | [43] |
| RSC10 | 2 | Kefeng No.1×Nannong1138-2 RIL | Satt634-589.8-Gm020580 | [36] |
| RSC11 | 13 | Qihuang No.1×Nannong1138-2 F2 | Sat_234-4295.2-Sct_033 | [11] |
| RSC12 | 13 | Qihuang22×Nannong1138-2 F2 | Satt334-2342.6-Sct_033 | [31] |
| RSC14Q | 13 | Qihuang No.1×Nannong1138-2 RHL | MY525-617.6-MY750 | [32] |
| RSC15 | 6 | RN-9×7605 RIL | SSR_06_17-95.2-BS060835 | [46] |
| RSC17 | 2 | Zhongdou35×Nannongcaidou No.5 F2 | BS020622-60.2-BS020627 | [38] |
| RSC18 | 6 | RN-9×7605 RIL | Satt286-997.6-Satt277 | [47] |
| RSC18A | 2 | Kefeng No.1×Nannong1138-2 RIL | BS020667-77.7-BS020670 | [40] |
| RSC18B | 13 | Qihuang22×Nannong1138-2 F2 | Satt334-567.9-SOYHSP176 | [40] |
| RSC20 | 13 | Qihuang No.1×Nannong1138-2 RHL | gm-ssr_13-14-80.3-gm-indel_13-3 | [45] |
| RSC-pm | 13 | PI96983×Nannong1138-2 F2 | BS131114-345.1-BS131136 | [37] |
| RSC-ps | 13 | PI96983×Nannong1138-2 F2 | BS131140-378.4-BS131185 | [37] |
新窗口打开
由表1可知,目前国内外已标记定位的SMV抗病基因位点有22个,主要分布在大豆第2、6、13和14染色体上,其中第13染色体上基因位点最多,有10个,定位在第2染色体上的基因位点有8个,而在第6和14染色体上均有2个SMV抗病基因位点。除少数几个基因位点两侧分子标记的物理距离较远外,多数抗病基因位点两侧分子标记的物理距离均在1 Mb以内(表1)。
SMV抗病基因位点Rsv1最早由YU等[15]利用SSR(simple sequence repeats,SSR)标记SM176定位在大豆F连锁群(第13染色体)上,遗传距离为0.5 cM。利用RFLP(restriction fragment length polymorphism,RFLP)、RAPD(random amplified polymorphic DNA,RAPD)和SSR标记,GORE等[18]构建了包含Rsv1和花生斑驳病毒抗病基因Rpv1位点在内的6.8 cM精细遗传图谱。SHI等[25]构建J05与Essex杂交的F2群体,进一步将Rsv1位点定位在SSR标记Sat_154和Satt510之间,遗传距离分别为0.2和2.3 cM(表1)。参考大豆Willimas82全基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),通过分析发现SMV抗病基因位点Rsv1所在的物理区间起止位置为28 506 083—31 802 676 bp,物理距离3 296.6 kb(表1)。Rsv1-h尽管是Rsv1位点的一个等位基因[20],但其在大豆第13染色体上的物理位置并不确定[48]。利用Suweon97和Willimas82的F2群体,MA等[48]研究发现Suweon97携带的抗病基因Rsv1-h定位在标记BARCSOYSSR_13_1114和BARCSOYSSR_13_1115之间,与PI96983携带的Rsv1位点属于不同的区域。两标记间的物理距离为97.5 kb(http://www.phytozome.net/soybean,Wm82.a2.v1)。
Rsv3位点被定位在大豆连锁群B2(第14染色体)上标记A519和M3Satt之间,遗传距离分别为0.9和0.8 cM[19],而基于标记A519开发的3个SNP(single nucleotide polymorphism,SNP)标记A519-460、A519-956和A519-1165与Rsv3紧密连锁[24]。SHI等[25]发现SSR标记Sat_424与大豆J05携带的Rsv3紧密连锁,遗传距离为1.5 cM。利用新开发的标记,SUH等[26]进一步将Rsv3定位在分子标记S156l和BARCSOYSSR_14_1417之间,遗传距离分别为0.3和0.5 cM,候选区域在第14染色体上的起止位置为46 852 283—47 059 763 bp(http://www.phytozome.net/soybean,Wm82.a2.v1),物理距离207.5 kb(表1)。
HAYES等[49]利用BSA(bulked segregant analysis,BSA)法和F2群体将抗病基因Rsv4定位在大豆D1b连锁群(第2染色体)上,SSR标记Satt558和Satt542与Rsv4之间的遗传距离分别为7.8和4.7 cM。利用比较作图策略,HWANG等[50]进一步将Rsv4定位在EST(expressed sequence tag,EST)标记AI856415-g和AW471852A之间,遗传距离分别为2.8和2.4 cM。基于大豆基因组鸟枪测序法,SAGHAI MAROOF等[51]将Rsv4锁定在1.3 cM的精细作图区域内。2016年,ILUT等[27]对Rsv4区域进行了深度重测序,发现Rsv4与两侧的标记Rat2和S6ac的遗传距离均为0.2 cM(表1),起止位置为12 044 285—12 163 994 bp(http://www.phytozome.net/soybean,Wm82.a2.v1),物理区间119.7 kb(表1)。
美国大豆品种York携带的抗病基因早先被命名为Rsv1-y[52],被推测是PI96983携带抗病基因Rsv1位点的等位基因,2个品种对SMV株系G1-G7的抗性反应存在差异[52]。KLEPADLO等[28]利用PI96983(Rsv1)×York(Rsv1-y)构建包含3 000个F2:3家系的群体检测了2个抗病基因之间的等位性关系,结果发现Rsv1和Rsv1-y是2个紧密连锁的基因位点,遗传距离为2.2 cM,物理距离约为440 kb,最后作者将York携带的Rsv1-y抗病基因重新命名为Rsv5[28]。
科丰1号是中国科学院遗传与发育生物学研究所从河北地方大豆品种系统选择育成,对中国的18个SMV株系(SC1-SC13、SC16和SC18—SC21)和美国的7个株系(G1—G7)均具有抗性[53,54,55]。利用科丰1号×南农1138-2组配的F2、RIL和剩余杂合系RHL(residual heterozygous lines,RHL)群体,ADHIMOOLAM等[44]、FU等[29]、YAN等[41]、WANG等[33]、ZHAO等[43]、李春燕等[36]、郭东全等[56]和LI等[40]先后将科丰1号对SMV株系SC5、SC7、SC8、SC10、SC13、SC18携带的抗病基因RSC5、RSC7、RSC8、RSC10、RSC13、RSC18A定位在大豆的第2染色体上(表1和图1)。此外,阳小凤等[38]将大豆品系Q0923对SC6株系具有的抗病基因RSC6和中豆35携带对SC17的抗病基因RSC17也均定位在第2染色体上(表1和图1)。
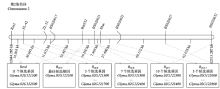 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1大豆抗SMV基因在第2染色体上的遗传、物理图谱及候选抗病基因虚线箭头部分表示单个SMV抗病基因的物理区间;最下面的框内是SMV抗病候选基因的个数及范围。下同
-->Fig. 1Resistance candidate genes, genetic and physical maps of SMV resistance loci on chromosome 2The dashed lines arrow indicated that the physical distance of single SMV resistance gene; the number and range of SMV resistance candidate genes in the bottom box. The same as below
-->
根据大豆Willimas82的参考基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),结合上述标记定位的结果分析可以发现,大豆第2染色体上的SMV抗病基因位点主要分布在11 188 722—13 104 514 bp(表1,图1)。其中有6个SMV抗病基因位点的候选区段交叉重叠,集中分布在12 044 285—12 313 444 bp约269.2 kb的区间内,推测这6个基因位点可能紧密连锁或具有等位关系。进一步对这一区间分析发现,该区间内有20个候选基因(Glyma.02G121500—Glyma.02G123400)(图1,表2),但均不具有NBS-LRR(nucleotide binding site-leucine rich repeat,NBS-LRR)抗病基因保守结构域。
Table 2
表2
表2抗病基因位点Rsv4、RSC5、RSC6、RSC7、RSC8和RSC17在大豆第2染色体上可能的候选基因a
Table 2Putative candidate gene loci on the chromosome 2 region containing Rsv4, RSC5, RSC6, RSC7, RSC8, and RSC17a
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location (bp) | 方向 Orientation | 外显子 Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) | ||||
|---|---|---|---|---|---|---|---|---|---|---|
| Glyma.02G121400(Rsv4) | Glyma02g13380 | 12028930-12030635 | 反向 Reverse | 1 | 355 | 未知蛋白 Unknown protein | ||||
| Glyma.02G121500(RSC8) | Glyma02g13401 | 12065640-12082937 | 正向 Forward | 8 | 244 | MADS-box蛋白 MADS-box protein | ||||
| Glyma.02G121600(RSC8) | Glyma02g13420 | 12084714-12089043 | 正向 Forward | 8 | 243 | MADS-box蛋白 MADS-box protein | ||||
| Glyma.02G121700 | - | 12093068-12095722 | 反向 Reverse | 4 | 128 | RING锌指蛋白 RING zinc finger protein | ||||
| Glyma.02G121800(Rsv4) | Glyma02g13450 | 12106073-12107969 | 正向 Forward | 4 | 254 | 普遍应急蛋白 Universal stress protein | ||||
| Glyma.02G121900(Rsv4) | Glyma02g13460 | 12112034-12115054 | 正向 Forward | 1 | 820 | 丝氨酸/苏氨酸蛋白激酶 Serine/threonine protein kinase | ||||
| Glyma.02G122000(Rsv4) | Glyma02g13470 | 12115287-12118397 | 正向 Forward | 1 | 647 | 丝氨酸/苏氨酸蛋白激酶 Serine/threonine protein kinase | ||||
| Glyma.02G122100(Rsv4, RSC5) | Glyma02g13495 | 12134374-12137612 | 正向 Forward | 3 | 121 | 铜转运相关蛋白ATOX1 Copper transport protein ATOX1 related | ||||
| Glyma.02G122200(RSC7) | Glyma02g13520 | 12141974-12149160 | 反向 Reverse | 4 | 556 | 含DnaJ结构域的伴侣蛋白 Chaperone DnaJ-domain containing protein | ||||
| Glyma.02G122300 | - | 12143960-12145950 | 反向 Reverse | 1 | 78 | 未知蛋白 Unknown protein | ||||
| Glyma.02G122400 | Glyma02g13530 | 12150906-12151220 | 反向 Reverse | 1 | 104 | 未知蛋白 Unknown protein | ||||
| Glyma.02G122500 | Glyma02g13540 | 12158735-12163084 | 反向 Reverse | 7 | 452 | 含ACT结构域的蛋白5 ACT domain-containing protein 5 | ||||
| Glyma.02G122600 | Glyma02g13555 | 12195454-12196275 | 反向 Reverse | 2 | 224 | SWIM锌指/MULE转座酶结构域 SWIM zinc finger/MULE transposase domain | ||||
| Glyma.02G122700 | Glyma02g13570 | 12226519-12231374 | 正向 Forward | 4 | 485 | Gar2相关蛋白 Gar2-related protein | ||||
| Glyma.02G122800 | Glyma02g13580 | 12250250-12252534 | 正向 Forward | 4 | 136 | 未知蛋白 Unknown protein | ||||
| Glyma.02G122900 | Glyma02g13590 | 12259463-12264960 | 正向 Forward | 6 | 304 | 含BSD结构域的蛋白 BSD domain-containing protein | ||||
| Glyma.02G123000 | Glyma02g13616 | 12271389-12276607 | 反向 Reverse | 7 | 422 | 羧肽酶相关的X蛋白 Pro-X carboxypeptidase-related | ||||
| Glyma.02G123100 | Glyma02g13630 | 12279586-12286860 | 正向 Forward | 11 | 794 | 类脯氨酰内肽酶 Prolyl Endopeptidase-like | ||||
| Glyma.02G123200 | Glyma02g13640 | 12289339-12294930 | 正向 Forward | 8 | 493 | GDP岩藻糖蛋白 GDP-fucose protein | ||||
| Glyma.02G123300 | Glyma02g13655 | 12298608-12305535 | 正向 Forward | 6 | 272 | 香叶基转移酶2α亚基 Geranylgeranyl transferase type-2 subunit alpha | ||||
| Glyma.02G123400 | Glyma02g13670 | 12306442-12311749 | 反向 Reverse | 7 | 336 | 固醇调节元件结合蛋白 Sterol regulatory element- binding protein | ||||
新窗口打开
齐黄1号是山东省农业科学院从山东寿张地方品种系统选择育成的另一个对SMV具有广谱抗性的品种,对中国的18个SMV株系(SC1—SC8、SC11—SC14、SC16—SC18和SC20—SC22)和美国的7个株系(G1—G7)也均具有抗性[53,54,55]。利用F2、RIL和RHL群体,ZHENG等[39]、白丽等[11]、LI等[30]、MA等[31,32]、LI等[40]和ADHIMOOLAM等[45]将齐黄1号及其衍生系齐黄22携带的抗SMV基因RSC3Q、RSC11、RSC12、RSC14Q、RSC18B和RSC20均定位在大豆的第13染色体上(表1和图2)。YANG等[37]将美国抗源PI96983携带对SMV株系SC3、SC6、SC17的抗病基因Rsc-pm以及对SC7的抗病基因Rsc-ps也均定位在第13染色体上相邻的区域内(表1和图2)。
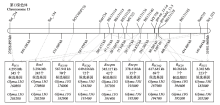 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2大豆抗SMV基因在第13染色体上的遗传、物理图谱及抗病候选基因
-->Fig. 2Resistance candidate genes, genetic and physical maps of SMV resistance loci on chromosome 13
-->
基于大豆Willimas82的参考基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),结合标记定位结果分析发现,国内外定位于第13染色体上的SMV抗病基因位点的候选区段主要在27 656 895—31 951 960 bp成簇存在(图2),物理距离为4.295 Mb,且这一区段内富集着许多具NBS-LRR结构域的候选基因(表3)。从图2可以发现,在4.295 Mb的物理区段内,SMV抗病基因位点的候选区段均存在相互重叠的现象,相较于第2染色体上候选区段内的基因数,第13染色体上候选区段内的基因数量更多,推测该候选区段内的多数基因连锁较为紧密,难以发生交换。
Table 3
表3
表3SMV抗病基因位点Rsv1、Rsv5、RSC3Q、RSC11、RSC12、RSC14Q、RSC18B、RSC20、Rsc-pm和Rsc-ps在大豆第13染色体上部分可能的候选基因
Table 3Putative resistance candidate gene loci on the chromosome 13 region containing Rsv1, Rsv5, RSC3Q, RSC11, RSC12, RSC14Q, RSC18B, RSC20, Rsc-pm, and Rsc-ps
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location (bp) | 方向 Orientation | 外显子 Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) |
|---|---|---|---|---|---|---|
| Glyma.13G184400 | - | 29818371-29819472 | 正向 Forward | 2 | 335 | 类MYB DNA结合域 MYB-like DNA-binding domain |
| Glyma.13G184800 (Rsv1-h) | Glyma13g25420 | 29858000-29863047 | 反向 Reverse | 2 | 1263 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G184900 (Rsv1-h) | Glyma13g25440 | 29873181-29877088 | 反向 Reverse | 2 | 1155 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G187500 | Glyma13g25716 | 30129159-30133972 | 正向 Forward | 3 | 533 | 类MYB DNA结合域 MYB-like DNA-binding protein |
| Glyma.13G187600 | Glyma13g25724 | 30134637-30143817 | 反向 Reverse | 9 | 672 | 非特异性丝氨酸/苏氨酸蛋白激酶 Non-specific serine/threonine protein kinase |
| Glyma.13G187900 (RSC3Q) | Glyma13g25750 (6gG9) | 30174410-30180072 | 正向 Forward | 1 | 1184 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G188300 | Glyma13g25780 | 30207091-30212726 | 正向 Forward | 2 | 1181 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G188800 | Glyma13g25800 | 30236854-30239729 | 反向 Reverse | 2 | 133 | G型凝集素S-类受体丝氨酸/苏氨酸蛋白激酶 G-type lectin S-receptor-like serine/threonine- protein kinase |
| Glyma.13G188900 | Glyma13g25811 | 30244450-30249368 | 反向 Reverse | 8 | 642 | G型凝集素S-类受体丝氨酸/苏氨酸蛋白激酶 G-type lectin S-receptor-like serine/threonine- protein kinase |
| Glyma.13G189000 | Glyma13g25820 | 30251712-30254986 | 正向 Forward | 7 | 669 | G型凝集素S-类受体丝氨酸/苏氨酸蛋白激酶 G-type lectin S-receptor-like serine/threonine- protein kinase |
| Glyma.13G189400 | Glyma13g25860 | 30286518-30295433 | 正向 Forward | 6 | 287 | 类组蛋白转录因子 Histone-like transcription factor |
| Glyma.13G190000 (RSC3Q) | Glyma13g25920 (5gG3) | 30355157-30359208 | 正向 Forward | 4 | 1244 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G190300 (RSC3Q) | Glyma13g25950 | 30388583-30392233 | 正向 Forward | 2 | 1201 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G190400 (RSC3Q) | Glyma13g25970 | 30402029-30409606 | 正向 Forward | 1 | 2199 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G190800 (RSC3Q, Rsv1) | Glyma13g26000 (3Gg2) | 30423894-30430435 | 正向 Forward | 1 | 1280 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G192100 | Glyma13g26141 | 30530165-30536199 | 反向 Reverse | 1 | 1232 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G192200 | Glyma13g26150 | 30542364-30549789 | 正向 Forward | 4 | 414 | GRF锌指蛋白 GRF zinc finger protein |
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location (bp) | 方向 Orientation | 外显子 Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) |
| Glyma.13G192700 | Glyma13g26201 | 30595116-30597770 | 反向 Reverse | 3 | 816 | 锌指蛋白 Zinc finger protein |
| Glyma.13G193100 | Glyma13g26230 | 30643486-30648668 | 正向 Forward | 1 | 1188 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G193300 | Glyma13g26250 | 30662869-30665830 | 反向 Reverse | 1 | 874 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G193700 | Glyma13g26280 | 30682181-30695761 | 反向 Reverse | 11 | 469 | 转录因子 Transcription factor |
| Glyma.13G194100 | Glyma13g26310 | 30726074-30730604 | 正向 Forward | 1 | 1129 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G194500 | Glyma13g26380 | 30763823-30769223 | 正向 Forward | 1 | 1223 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G194600 | Glyma13g26400 | 30777645-30787581 | 反向 Reverse | 3 | 435 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.13G194700 (RSC20) | Glyma13g26420 | 30793151-30800421 | 反向 Reverse | 4 | 1095 | TIR-NB-LRR蛋白 TIR-nucleotide-binding leucine-rich repeat protein |
| Glyma.13G194800 | Glyma13g26450 | 30806038-30808277 | 反向 Reverse | 3 | 490 | TIR-NB-LRR蛋白 TIR-nucleotide-binding leucine-rich repeat protein |
| Glyma.13G194900 | Glyma13g26650 | 30832071-30838537 | 反向 Reverse | 4 | 530 | TIR-NB-LRR蛋白 TIR-nucleotide-binding leucine-rich repeat protein |
| Glyma.13G195100 (RSC20) | Glyma13g26460 | 30850341-30856173 | 正向 Forward | 4 | 1095 | TIR-NB-LRR蛋白 TIR-nucleotide-binding leucine-rich repeat protein |
| Glyma.13G195200 | Glyma13g26470 | 30862185-30877782 | 正向 Forward | 16 | 1421 | 丝氨酸/苏氨酸蛋白激酶 Serine/threonine protein kinase |
新窗口打开
RN-9是继科丰1号和齐黄1号后发现的又一个广谱抗源,对15个SMV株系均表现极端抗性,特别是对能感染所有鉴别寄主的SC15株系表现抗病[35,47]。利用RN-9×7605的RIL群体,YANG等[35]将RN-9携带的对SMV株系SC15具有的抗病基因RSC15定位在大豆第6染色体上,该基因与SSR标记Sat_213和Satt286之间的遗传距离分别为8.0和6.6 cM。2017年,REN等[46]进一步将RSC15锁定在标记SSR_06_17和BS060835之间,遗传距离分别为1.44和0.63 cM(表1),起止位置为15 635 537—15 730 967 bp(http://www.phytozome.net/soybean,Wm82.a2.v1),物理区间为95.2 kb(表1,图3-a)。
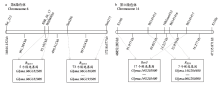 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3大豆抗SMV基因在第6和14染色体上的遗传、物理图谱及抗病候选基因
-->Fig. 3Resistance candidate genes, genetic and physical maps of SMV resistance loci on chromosome 6 and 14
-->
此外,李凯等[47]将RN-9携带对SC18的抗性基因RSC18定位在标记Satt286和Satt277之间,遗传距离分别为6.12和4.69 cM,物理区间为997.6 kb。参考大豆Willimas82的基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),分析发现抗病基因RSC15和RSC18在第6染色体上的候选区段没有重叠的区域(图3-a)。其中,在95.4 kb的RSC15候选基因区段内存在5个候选基因(Glyma.06G182500—Glyma.06G182900),且均不具有NBS-LRR抗病基因保守结构域(表4,图3-a),在RSC18候选基因区段内,有73个候选基因存在(Glyma.06G186300—Glyma. 06G193500)。
Table 4
表4
表4抗病基因RSC15在大豆第6染色体上可能的候选基因
Table 4Putative candidate genes on the chromosome 6 region containing RSC15
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location(bp) | 方向 Orientation | 外显子 Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) |
|---|---|---|---|---|---|---|
| Glyma.06G182500 | Glyma06g19380 | 15642471-15643990 | 正向 Forward | 1 | 307 | 未知蛋白 Unknown protein |
| Glyma.06G182600 (RSC15) | Glyma06g19390 | 15654270-15659464 | 反向 Reverse | 12 | 459 | 过氧化物酶体膜蛋白 Peroxisomal membrane protein |
| Glyma.06G182700 | Glyma06g19400 | 15677974-15682343 | 正向 Forward | 8 | 261 | 碳酸酐酶 Carbonic anhydrase |
| Glyma.06G182800 | Glyma06g19430 | 15705290-15709591 | 反向 Reverse | 2 | 753 | 轻链驱动蛋白 Kinesin light chain |
| Glyma.06G182900 | - | 15722947-15727315 | 反向 Reverse | 4 | 342 | 非特异性丝氨酸/苏氨酸蛋白激酶 Non-specific serine/threonine protein kinase |
新窗口打开
利用大白麻×南农1138-2的F2作图群体,WANG等[34]将大白麻携带的抗病基因RSC4精细定位在大豆第14染色体上,紧密连锁的2个基因组SSR标记BARCSOYSSR_14_1413和BARCSOYSSR_14_1416与RSC4的遗传距离分别为0.17和0.27 cM(表1),物理距离63.4 kb(表1,图3-b)。参考大豆Willimas82的基因组序列(http://www.phytozome.net/soybean,Wm82.a2.v1),结合RSC4和Rsv3定位结果分析可以发现,在大豆第14染色体46 852 283—47 135 871 bp共有17个可能的候选基因(表5),其中有5个候选基因具NBS-LRR保守结构域。从图3-b还可以发现,2个SMV抗病基因位点RSC4和Rsv3的候选基因位置具有重叠的物理区段,推测RSC4与Rsv3可能是等位或紧密连锁的。
Table 5
表5
表5抗病基因位点Rsv3和RSC4在大豆第14染色体上可能的候选基因
Table 5Putative candidate gene loci on the chromosome 14 region containing Rsv3, and RSC4
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location(bp) | 方向 Orientation | 外显子 Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) |
|---|---|---|---|---|---|---|
| Glyma.14G203800 | Glyma14g38420 | 46841491-46853116 | 反向 Reverse | 9 | 780 | 核苷酸转移酶家族相关蛋白 Nucleotidyl transferase family protein-related |
| Glyma.14G203900 | Glyma14g38430 | 46856580-46859641 | 反向 Reverse | 3 | 254 | EXPA8蛋白 Expansin-A8 protein |
| Glyma.14G204000 | Glyma14g38445 | 46883204-46883614 | 反向 Reverse | 1 | 136 | 未知蛋白 Unknown protein |
| Glyma.14G204100 | Glyma14g38460 | 46886862-46890243 | 反向 Reverse | 4 | 362 | bZIP转录因子 bZIP transcription factor |
| Glyma.14G204200 | Glyma14g38468 | 46893813-46897703 | 反向 Reverse | 4 | 279 | 未知蛋白 Unknown protein |
| Glyma.14G204300 | Glyma14g38476 | 46909226-46909477 | 正向 Forward | 1 | 83 | 未知蛋白 Unknown protein |
| Glyma.14G204400 | Glyma14g38492 | 46856580-46859641 | 反向 Reverse | 3 | 254 | CW型锌指卷曲螺旋结构域蛋白3 Zinc finger CW-type coiled-coil domain protein 3 |
| Glyma.14G204500 (Rsv3) | Glyma14g38500 | 46946496-46957734 | 正向 Forward | 3 | 1315 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.14G204600 (Rsv3, RSC4) | Glyma14g38516 | 46968705-46974585 | 正向 Forward | 4 | 1236 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.14G204700 (Rsv3) | Glyma14g38533 | 46981104-46996696 | 正向 Forward | 2 | 1307 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.14G204800 | - | 46984060-46984741 | 正向 Forward | 1 | 112 | 未知蛋白 Unknown protein |
| Glyma.14G204900 | Glyma14g38550 | 47000358-47001315 | 正向 Forward | 1 | 222 | 未知蛋白 Unknown protein |
| Glyma.14G205000 (Rsv3, RSC4) | Glyma14g38561 | 47005574-47019661 | 正向 Forward | 3 | 1311 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.14G205100 | Glyma14g38570 | 47020646-47026852 | 正向 Forward | 3 | 623 | 类KIP1家族蛋白 KIP1-like family protein |
| Glyma.14G205200 (RSC4) | Glyma14g38580 | 47041931-47046048 | 正向 Forward | 4 | 505 | 细胞色素P450 CYP2亚家族 Cytochrome P450 CYP2 subfamily |
| Glyma.14G205300 (Rsv3) | Glyma14g38586 | 47046209-47053652 | 反向 Reverse | 4 | 1350 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein |
| Glyma.14G205400 | Glyma14g38593 | 47059088-47060670 | 正向 Forward | 4 | 171 | 未知蛋白 Unknown protein |
新窗口打开
2 SMV抗病候选基因预测
随着大豆对SMV抗病基因不断地发现、标记和精细定位,大豆对SMV候选抗病基因保守结构域的生物信息学分析、qRT-PCR(quantitative real time polymerase chain reaction,qRT-PCR)表达分析及克隆测序分析等也在不断地拓展和深入,极大地缩小了候选基因的筛选范围。在第2染色体上,SAGHAI MAROOF等[51]对Rsv4的候选基因序列(http://www.phytozome.net/soybean,Wm82.a1.v1)进行分析发现,这些基因均不具有NBS-LRR结构域(图1,表2),推测Rsv4属于一种新类型的抗病基因,其可能是通过干扰病毒感染和细胞间运动,并延迟病毒在维管束间移动,从而达到抗病的目的。ILUT等[27]通过对SMV株系G1和G7具有不同抗性反应的6个野生大豆和13个栽培大豆Rsv4位点所在的120 kb的区域进行深度测序分析发现,这一区域至少存在2个Rsv4单倍型,其中最可能的一个单倍型包含4个候选基因:1个胁迫响应基因Glyma.02G121800、2个RD类受体蛋白酶基因(Glyma.02G121900和Glyma.02G122000)和1个铜转运蛋白基因(Glyma.02G122100)(表2)。从基因结构可以看出,这几个候选基因均不具有经典的NBS-LRR型抗病基因结构,验证了Rsv4可能属于一种新类型抗病基因的推测[27,50]。作者对该区域的单倍型系统发育分析发现Rsv4位点是最新才从野生大豆进化到栽培大豆中的[27]。不过,KLEPADLO等[28]通过对10个大豆品种的测序却证实Glyma.02G121400可能是Rsv4的候选基因(表2)。
ADHIMOOLAM等[44]将SNP标记及抗性鉴定表型数据进行关联分析和qRT-PCR表达分析发现,科丰1号携带对SC5最有可能的抗病候选基因是Glyma02g13495(Glyma.02G122100)(表2)。
根据候选基因的结构分析,YAN等[41]推测具NBS-LRR结构域的基因Glyma02g13600,具有热休克蛋白40(HSP40)结构的基因Glyma02g13520(Glyma.02G122200)和具有丝氨酸羧肽酶型(Serine carboxypeptidase-type)的基因Glyma02g13620可能是RSC7的候选基因,不过基因Glyma02g13600和Glyma02g13620在最新的大豆参考基因组序列中已经被重新进行整合(http://www.phytozome.net/soybean,Wm82.a2.v1,2018年3月12日)。CHE等[57]通过GWAS(genome wide association study,GWAS)和qRT-PCR技术研究发现,与拟南芥转录调控因子WRKY72、真核生物延伸因子eEF1Bβ和RLP9同源的3个大豆基因(Glyma.01G043300、Glyma.09g063800和Glyma.12g233700)可能参与了大豆抗SC7株系的过程。
WANG等[33]运用qRT-PCR技术对精细定位的RSC8抗病候选基因分析发现,其中5个候选基因的表达量在抗感品种间的响应模式或时间上有很大的差异,推测这些基因可能参与了大豆对SMV株系的抗性反应。ZHAO等[43]进一步将RSC8的候选基因确定为具MADS-box保守结构域的基因Glyma.02G121500和Glyma.02G121600(表2)。
通过对SC15的抗病品种RN-9和感病品种7605的5个候选基因(Glyma.06G182500— Glyma.06G182900)DNA和cDNA进行测序和qRT-PCR分析,REN等[46]推测过氧化物膜蛋白基因GmPEX14(Glyma.06G182600)是RSC15最有可能的候选基因[46]。根据测序和表达分析,LI等[40]研究发现Glyma.02G127800、Glyma.02G128200和Glyma.02G128300可能是科丰1号对SMV株系SC18的抗病候选基因(表4)。
通过分析大豆抗病基因Rsv1位点定位区域的NBS-LRR基因簇,HAYES等[58]从抗SMV品种PI96983中克隆到一个在结构上具有non-TIR(toll interleukin1 receptor,TIR)-NBS分子特征的抗病候选基因3gG2(Glyma.13G190800),该基因全长3 390 bp,包含一个完整的编码框,SHAO等[59]从抗病基因进化的角度证实了Glyma13g26000(Glyma.13G190800)是Rsv1的一个候选基因。MA等[48]对抗病基因Rsv1-h的8个候选基因序列进行分析发现,Glyma13g184800和Glyma13g184900具有CC(coiled-coil,CC)-NBS-LRR抗病基因的保守结构,推测这两个基因可能是Rsv1-h的最佳候选基因(表3)。
在精细定位的基础上,ZHENG等[39]通过qRT-PCR分析发现,Glyma13g25730、Glyma13g25750(Glyma.13G187900)、Glyma13g25950(Glyma.13G190300)、Glyma13g25970(Glyma.13G190400)和Glyma13g26000(Glyma.13G190800)等5个基因可能是RSC3Q的候选基因(表3)。LI等[42]进一步通过转录组和qRT-PCR研究表明齐黄1号携带对SC3抗病基因RSC3Q可能的候选基因为Glyma13g25920(Glyma.13G190000)、Glyma13g25950(Glyma. 13G190300)、Glyma13g25970(Glyma.13G190400)、Glyma13g26000(Glyma.13G190800)等4个具NBS-LRR抗病基因保守结构域的基因(表3),其中有3个候选基因与ZHENG等[39]的研究结果相一致。
ADHIMOOLAM等[45]综合候选基因的测序结果和qRT-PCR表达分析发现,齐黄1号携带对SC20最有可能的抗病候选基因是Glyma.13G194700和Glyma.13G195100(表2)。
在大豆的第14染色体上,基于抗病基因RSC4精细定位的结果,WANG等[34]参考Willimas82的参考基因组序列和qRT-PCR技术发现3个抗病候选基因Glyma14g38510(Glyma.14G204600)、Glyma14g38560(Glyma.14G205000)和Glyma14g38580(Glyma.14G205200)在抗感品种间不同时间点的表达量具有差异,初步推测这些基因可能参与了大豆对SC4株系的抗性反应(表5)。
对Rsv3抗病基因所在的154 kb区段进行分析,SUH等[26]发现这个区段是一个富集多个抗病基因的基因簇,其中有5个抗病候选基因Glyma14g38500(Glyma.14G204500)、Glyma14g38510(Glyma.14G204600)、Glyma14g38540(Glyma.14G204700)、Glyma14g38560(Glyma.14G205000)和Glyma14g38590(Glyma.14G205300)编码NB-LRR结构的抗病蛋白(表5),作者认为Rsv3可能是NB-LRR家族中的一员[26]。值得注意的是,SMV抗病基因RSC4和Rsv3均在这个区段内,说明RSC4和Rsv3属于同一个抗病基因簇。SUH等[26]推测的基因Glyma.14G204600和Glyma.14G205000与WANG等[34]通过qRT-PCR获得的抗病候选基因是一样的,不过2个基因位点是不是同一个基因仍需进一步的研究分析。
通过分析抗病品种L29接种SMV株系G7后1—3 h的表达量,REDEKAR等[60]发现,基因Glyma14g38533(Glyma.14G204700)是5个Rsv3候选基因中表达量最大的一个基因。比较测序研究表明,Glyma. 14G204700在抗病基因型(Rsv3型)和感病基因型(rsv3型)间有150个SNP和6个Indel差异,而且这种差异主要集中在LRR编码区。因此,作者推测Glyma.14G204700最有可能是Rsv3的候选基因[60](表5)。
3 SMV抗病相关基因筛选
大豆对SMV抗病基因的发掘除了利用精细定位图位克隆外,还有通过同源克隆等方法获得了一系列抗SMV相关基因的报道。贺超英等[61]和WANG等[62]利用同源克隆的方法从科丰1号中获得2个SMV抗性相关基因KR1和KR4,它们都具有NBS和LRR等抗病基因的结构特征,RT-PCR分析表明二者均受SMV株系Sa接种和水杨酸处理的影响,推测KR1和KR4可能参与了大豆抗SMV的过程[62,63]。此外,WANG等[64]研究发现大豆同源异型-亮氨酸拉链蛋白基因GmHZ1具有HD-Zip结构域,是植物特有的转录因子。接种SMV株系N3后,该基因在感病品种中的表达量上调,而在抗病品种中则下调,初步证实GmHZ1可能作为转录激活因子参与大豆对SMV的抗性(表6)。
Table 6
表6
表6参与大豆抗大豆花叶病毒可能的相关基因
Table 6Putative related genes to soybean mosaic virus in soybean
| 基因名称 Gene name | 别名(Wm82.a1.v1) Alias (Wm82.a1.v1) | 染色体位置 Chromosome location(bp) | 方向 Orientation | 外显子Exon | 氨基酸 AA | 基因注释 Gene annotation (BLASTX) | 参考文献 References |
|---|---|---|---|---|---|---|---|
| Glyma.19G054900 | KR1 | Chr.19: 9162156-9166831 | 反向 Reverse | 5 | 1124 | TIR-NB-LRR蛋白 TIR-nucleotide-binding leucine-rich repeat protein | [63] |
| Glyma.15G232800 | KR4 | Chr.15: 43743901-43747977 | 反向 Reverse | 2 | 1158 | NB-LRR蛋白 Nucleotide-binding leucine-rich repeat protein | [62] |
| Glyma.06G190200 | GmHZ1 | Chr.6: 16703967-16706492 | 反向 Reverse | 3 | 326 | 同源异型域相关亮氨酸拉链蛋白 Homeobox associated leucine zipper protein | [64] |
| Glyma.07G202300 | IFS1 | Chr.7: 37166660-37169027 | 正向 Forward | 2 | 521 | 细胞色素P450/异黄酮合酶Ⅰ Cytochrome P450/Isoflavonoid synthaseⅠ | [65] |
| Glyma.13G236500 | GmERF4 | Chr.13: 34675713-34677522 | 正向 Forward | 1 | 222 | 含AP2结构域的转录因子 AP2 domain-containing transcription factor | [66] |
| Glyma.09G040600 | GmPR10 | Chr.9: 3385940-3388420 | 正向 Forward | 2 | 158 | Bet Ⅵ家族病程相关蛋白 Pathogenesis-related protein Bet Ⅵ family | [67] |
| Glyma.06G044400 | GmSN1 | Chr.6: 3381869-3382860 | 反向 Reverse | 2 | 88 | 赤霉素调节蛋白 Gibberellin regulated protein | [68] |
| Glyma.16G032900 | GmMPK4 | Chr.16: 3110022-3117619 | 正向 Forward | 6 | 373 | 促分裂原活化蛋白激酶4 Mitogen-activated protein kinase 4 | [69] |
| Glyma.02G138800 | GmMPK6 | Chr.2: 14379790-14384864 | 正向 Forward | 6 | 391 | 促分裂原活化蛋白激酶6 Mitogen-activated protein kinase 6 | [70] |
| Glyma15g062900 | GmHSP40 | Chr.15: 4450904-4452397 | 反向 Reverse | 1 | 497 | DnaJ同源亚家族C成员 DnaJ homolog subfamily C member | [71] |
| Glyma.14G162100 | GmPP2C3a | Chr.14: 38860097-38863074 | 正向 Forward | 3 | 386 | 蛋白磷酸酶2C Protein phosphatase 2C | [72] |
| Glyma.08G187600 | GmAKT2 | Chr.8: 15041718-15048454 | 正向 Forward | 11 | 834 | 钾通道AKT2 Potassium channel AKT2 | [73] |
| Glyma.02G272700 | GmCnx1 | Chr.19: 45640500-45647671 | 反向 Reverse | 12 | 652 | 蝶呤型钼辅因子蛋白 Molybdopterin biosynthesis protein | [74] |
| Glyma.17G186600 | GmEF1A | Chr.17: 23719358-23722187 | 正向 Forward | 2 | 447 | 真核延伸因子1A Eukaryotic elongation factor 1A | [77] |
| Glyma.02G112400 | GmeIF5A | Chr.2: 10905625-10907687 | 反向 Reverse | 5 | 159 | 真核翻译起始因子5A Eukaryotic translation initiation factor 5A | [78] |
| Glyma.01G043300 | - | Chr.1: 4707961-4714222 | 正向 Forward | 4 | 509 | WRKY DNA结合结构域 WRKY DNA-binding domain | [57] |
| Glyma.09g063800 | - | Chr.9: 6145341-6146535 | 正向 Forward | 5 | 126 | 伸长因子1B Elongation factor 1B | [57] |
| Glyma.12g233700 | - | Chr.12: 39288169-39296830 | 反向 Reverse | 8 | 1045 | 类受体蛋白13 Receptor-like protein 13 | [57] |
新窗口打开
关联分析研究发现,大豆异黄酮合成酶基因IFS1与大豆对SC3的抗性显著相关[65]。表达分析发现接种SC3后7 d,IFS1基因在抗病品种中的表达量显著上调,而在感病品种中没有变化,推测该基因可能参与了大豆抗SMV侵染的过程[65]。ZHANG等[66]对大豆中一个耐胁迫基因GmERF4研究发现,其能够被SMV诱导表达,推测该基因可能是大豆对SMV抗性反应的一个相关因子。GmPR10是大豆中的一个PR10蛋白基因,RT-PCR分析表明,大豆接种SMV后该基因在叶片中被强烈诱导并高效表达,推测其可能在病毒诱导下使大豆获得系统抗性以抵抗外来病原菌的侵袭[67]。
通过转录组分析,HE等[68]发现GAST(GA- stimulated transcript,GAST)基因亚家族成员在接种SMV后,在感病品种(感病型:Ssmv1)中的表达量下调,而在抗病品种(抗病型:Rsmv1)中没变化。进一步测序和聚类比较分析表明,GAST家族成员之一GmSN1基因与土豆中的抗病基因同源,可能参与了大豆抗SMV的过程[68]。
4 大豆抗SMV相关基因的功能鉴定
在利用生物信息学、qRT-PCR及克隆测序等一系列技术对SMV抗病候选基因进行预测和抗SMV相关基因进行筛选的基础上,相关****进一步通过病毒诱导的基因沉默VIGS(virus induced gene silencing,VIGS)、转基因操作等技术对抗SMV相关基因的功能进行了鉴定和研究。与空载对照相比,LIU等[69,70]研究发现沉默大豆蛋白激酶GmMPK4(Glyma.16G032900)和GmMPK6(Glyma.02G138800)的植株均对SMV表现更好的抗性;双分子荧光互补(bimolecular fluorescence complementation,BiFC)试验表明,GmMKK1和GmMKK2与GmMPK4相互作用,同时GmMKK1和GmMKK2在GmMPK4的上游起作用;GmMKK4与GmMPK6相互作用,GmMKK4在GmMPK6的上游起作用[70](表6)。
热激蛋白在植物防御生物及非生物胁迫中具有至关重要的作用,LIU等[71]研究发现沉默具有DnaJ结构域的大豆热激蛋白基因GmHSP40的植株能够增加大豆对SMV的感病性,说明GmHSP40在大豆抗SMV的过程中具有正调控作用。SEO等[72]通过大豆与SMV互作研究表明大豆2C型蛋白磷酸酶基因GmPP2C3a(Glyma.14G162100)是Rsv3介导的对SMV抗性中的一个调控因子。
ZHOU等[73]研究表明在多施钾肥的情况下能够提高大豆对SMV的抗性,过表达大豆钾离子通道基因GmAKT2发现,转基因阳性植株对SMV的抗性明显增强。其可能的原因是大豆GmAKT2的过量表达导致其韧皮部发生改变,进而不利于外部病毒的侵染、移动和复制[73]。转基因研究发现SMV对过表达大豆硝酸还原酶及黄嘌呤脱氢酶辅助因子GmCnx1转基因植株的侵染率显著低于对照品种,转基因植株显著增强大豆对SMV株系的抗性[74]。
Glyma.14G204500、Glyma.14G204600和Glyma.14G204700在抗感品种间表达量有显著差异且具有NB-LRR抗病基因保守结构域,它们被作为抗病基因RSC4的3个候选基因。LI等[75]研究发现,沉默3个基因后的抗病品种显著感病,3个基因在抗感品种间的序列比对发现它们在抗感品种间均有氨基酸上的差异,推测这3个基因与大豆对SMV的抗病均有关系[75]。
TRAN等[76]对Rsv3的候选基因Glyma.14G204700进行研究发现,在缺失Rsv3基因型的大豆品种中过量表达基因Glyma.14G204700后SMV的侵染被明显抑制;而对携带Rsv3基因型的大豆品种L29进行瞬时沉默后发现,原来对L29不致病的病毒株系表现较多的SMV积累[76]。将SMV抗病候选基因GmSN1过表达转化到拟南芥和大豆中,HE等[68]发现与对照相比,过表达转基因拟南芥植株能够增强对萝卜花叶病毒的抗性,转基因大豆植株能够增强对SMV的抗性。
通过酵母双杂交和VIGS研究发现,大豆转录延伸因子家族GmEF1A(Glyma.17G186600)蛋白基因能够增进SMV的复制,调控病原侵染;沉默该基因可降低SMV的积累,提高大豆对SMV的抗性[77]。敲除大豆真核翻译起始因子GmeIF5A(Glyma. 02G112400),CHEN等[78]研究发现植株减少了系统过敏性坏死反应,病毒积累增强,说明GmeIF5A在Rsv1介导的对SMV株系G7的系统过敏性坏死反应信号通路中起重要作用。
5 SMV抗病候选基因的调控网络
对Rsv1介导的极端抗性(extreme resistance,ER)候选基因3gG2、5gG3和6gG9进行沉默,ZHANG等[79]研究发现沉默后的植株表现感病,由Rsv1介导的ER丧失。同时,通过VIGS研究发现基因GmEDR1、GmEDS1、GmHSP90、GmJAR1、GmPAD4和2个WRKY转录因子(GmWRKY6和GmWRKY30)都参与了Rsv1介导的抗性[79]。基于上述研究结果并结合以前的研究,ZHANG等[79]形成了一个由Rsv1介导的ER调控网络:其中non-TIR-NBS-LRR基因Rsv1及热激蛋白基因HSP90和伴侣蛋白基因SGT1和RAR1位于调控网络顶端,EDS1和PAD4脂肪酶位于Rsv1的下游但在WRKY转录因子的上游,JAR1和EDR1最有可能在与EDS1和PAD4到WRKY转录因子的通路中是平行起作用的,最终表现对SMV的极端抗性(图4-a)。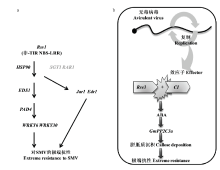 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4Rsv1和Rsv3介导对SMV抗性的调控网络图(引自ZHANG等[
-->Fig. 4Proposed signaling network for Rsv1- and Rsv3-mediated resistance to soybean mosaic virus (Cited from ZHANG et al.[
-->
SEO等[72]构建了一个Rsv3介导对SMV的ER调控模型:在大豆最初感染的细胞中,SMV复制并产生效应因子CI。Rsv3通过R蛋白识别CI,从而导致激素脱落酸(abscisic acid,ABA)在大豆体内的积累,进而激活ABA信号通路。ABA信号通路与Rsv3介导的极端抗性相关联,通过上调信号传导正调控因子GmPP2C3a,进而刺激胼胝质沉积。最终,在胞间连丝处的胼胝质沉积抑制病毒从最初侵染的细胞向健康细胞的移动并限制病毒的积累[72,81](图4-b)。
通过对GmSN1转基因拟南芥进行转录组表达分析,HE等[68]研究发现,该基因主要参与植物抗病的信号传导和免疫反应,而且在转基因大豆中发现另外一个与SMV抗性相关的基因GmAKT2也能够上调表达,表明这两个基因协同提高大豆对SMV的抗性。
6 展望
近年来,随着对大豆抗SMV研究的不断深入,在抗病基因的分子标记及候选基因的鉴定方面取得很大的进展。在大豆第2、6、13和14染色体上研究发现的SSR、SNP、Indel等分子标记与SMV抗病基因之间均紧密连锁或共分离,为分子标记辅助选择及分子设计育种奠定了良好的基础。MA等[31]研究表明:单独使用标记Satt334或Sct_033对RSC12在齐黄22×南农1138-2的3个世代以及对RSC14Q在齐黄1号×南农1138的2个世代分子标记辅助选择的准确率均在85%以上,同时使用2个标记的准确率达95%以上。SAGHAI MAROOF等[82]、SHI等[83]和WANG等[84]利用与SMV抗病基因紧密连锁的分子标记均成功地将分布在3条不同染色体上抗病基因精准地聚合在同一个大豆品系中,为广谱高抗SMV育种提供了优异的抗源材料。齐黄1号对中国的18个SMV株系和美国的7个株系均具有抗性[53,54,55],其所携带的单显性抗病基因均定位在第13染色体上[11,32,39-40,45]。郑桂杰[85]收集整理骨干亲本齐黄1号的衍生品种(系)发现,齐黄1号在全国11个省市衍生出92个高产优质品种和优异品系,其中黄淮海地区衍生出87个品种(系)[86]。通过对90个衍生品种(系)接种SC3和SC7发现,生产上大面积推广的品种徐豆1号、跃进5号、中黄19、豫豆19和齐黄22等对两个株系均表现抗病[85]。此外,对SMV具有广谱抗性的科丰1号及大白麻的衍生品种在生产上的推广也对控制SMV的危害起到了关键的作用[87]。
然而,在日本和韩国相继发现了使具有单个抗病基因的品种失去抗性的SMV毒株[88,89,90],特别是在加拿大发现的SMV分离物L-RB能够攻克对美国所有7个株系都表现抗病的Rsv4基因[91]。最近,WANG等[92]研究发现大豆花叶病毒P3蛋白单个氨基酸的改变就能使无毒株系攻克Rsv4的抗性而成为致病株系。此外,中国报道的强致病株系及一些新发现的株系和重组株系也预示着SMV与大豆寄主之间一直处在动态平衡中[53-54,93-95]。因此,持续寻找新的抗病材料并发掘新的抗病基因是抗SMV育种的一个长期目标。
中国是大豆的起源地,不仅具有大量的栽培大豆类型,而且野生资源丰富多样,目前发现的野生大豆占世界90%以上[96]。WEN等[97]通过野生大豆与地方品种的遗传多样性比较发现,野生大豆中仅有51%的等位基因在地方大豆品种中保留下来,大豆的重测序研究结果也印证了这一发现[98]。可见,经过多次驯化和人工选择,野生大豆中大量优异的遗传变异没有保留在栽培大豆中[97,98]。因此,利用野生大豆资源,发掘新的SMV抗病基因,对减少SMV危害,提高抗SMV育种成效具有关键性的作用。
大豆抗SMV的研究为确定SMV抗病基因和阐明大豆抗SMV调控网络奠定了扎实的基础[27-28,42-46]。以往由于大豆遗传转化及再生比较困难,对SMV抗病基因的功能验证研究较少[68,73,76]。目前,随着现代生物技术的发展,利用不断完善的大豆转基因技术、候选基因沉默技术及CRISPR/Cas9基因编辑技术等,对标记定位的SMV抗病候选基因进行功能验证,对尽快确定抗SMV过程中的关键基因将发挥重要作用。此外,从SMV侵染大豆的角度出发,研究者筛选出一些能够协助SMV侵染大豆的基因,通过沉默这些基因发现,大豆增强了对SMV的抗性[77,78]。因此,后期如果能够通过CRISPR/Cas9基因编辑技术对研究发现的这些基因或一些新发现的负调控关键因子进行改造,将是培育高效广谱抗SMV大豆品种的一个新途径。
解决抗SMV育种最根本的方法是在明确SMV抗病基因功能的基础上,阐明大豆抗SMV过程中调控的信号网络。SMV抗病基因的标记定位及分析对理解大豆抗SMV的调控信号网络以及发掘新的大豆抗病基因具有重要价值。ZHANG等[79]和SEO等[72]对SMV抗病基因Rsv1和Rsv3在抗病过程中的调控网络进行了构建和模拟,为进一步验证提供了重要线索。因此,探索大豆抗SMV的调控网络及对大豆的抗病性进行改良也是一个很重要的研究方向。
(责任编辑 李莉)
The authors have declared that no competing interests exist.
