0 引言
【研究意义】作物种质资源是开展作物品种选育的物质基础,稀有特效种质对推动育种进程具有决定性作用,新的育种目标能否实现决定于育种单位所拥有的种质资源,此外,种质资源也是开展生物学理论研究和分子遗传的重要基础材料,因此,开展种质资源的收集、评估、保存和利用对中国农业研究和发展具有十分重要的意义。黍稷(Panicum miliaceum L.),属于禾本科黍属(Panicum L.)[1],一年生草本植物[2]。它是最古老的农作物之一,有10 000多年的栽培历史,在世界范围内都有种植[3,4]。黍稷生长周期短,抗旱耐贫瘠,是干旱半干旱地区的重要作物[5,6]。中国目前保存黍稷资源8 800多份,准确评估黍稷资源在形态学和分子水平的遗传多样性,有利于其合理开发与利用,并促进其育种工作和基础理论方面的研究[7]。【前人研究进展】遗传多样性是生物多样性的重要组成部分,通过遗传多样性分析可以为各稀有物种的收集、育种应用以及起源进化研究提供有利数据。早期关于黍稷遗传多样性的研究多集中于形态学特征。胡兴雨等[8]和董孔军等[9]分别对国家种质库的8 016份黍稷种质资源和甘肃省600份黍稷地方资源的11个农艺性状进行主成分分析和聚类分析,评估了参试材料在形态学水平上的遗传多样性并以此为基础构建了黍稷初级核心种质。近年来,随着分子生物技术的快速发展,分子标记在黍稷遗传多样性研究方面也得到了较广泛的应用。在不同类型的分子标记中,SSR标记具有多等位基因、共显性和高度多态性等优点[10],广泛应用于作物的遗传多样性评价中,是评估遗传多样性的最佳选择[11,12,13]。CHO等[14]、王银月等[15]和RAJPUT等[16]采用SSR-PCR技术分别开发了25对、116对、254对多态性较好的黍稷特异性SSR标记。王瑞云等[17]开发了85对高基元SSR标记,其分辨能力强,扩增多态性好;朱建楚[18]、董俊丽等[19]将分子水平和表型结合对黍稷资源进行了遗传多样性分析;HU等[20]、HU等[21]、连帅等[22],LIU等[23]和王瑞云等[24]分别利用SSR标记评估了不同地理来源的黍稷资源的遗传多样性,研究表明聚类结果与地理分布密切相关,王瑞云等[17]和HU等[21]的研究结果表明黄土高原地区的遗传多样性最丰富,推测黄土高原可能是黍稷的起源地。连帅等[25]利用63对SSR多态性引物对192份黍稷资源进行遗传多样性分析,结果表明,国内外群体遗传分化不明显,群体间亲缘关系较近,个体间存在相互渗透。【本研究切入点】目前,黍稷遗传多样性研究多数集中于栽培品种与地方品种的研究,有关黍稷野生资源和国外地方品种的遗传多样性,特别是分子水平群体遗传结构的研究鲜有报道。【拟解决的关键问题】本研究利用本课题组开发的黍稷基因组SSR标记对近年来新引进的国内主栽区的黍稷野生资源以及国外资源共计146份材料进行SSR标记水平的遗传多样性分析,旨在揭示不同来源黍稷种质资源的遗传多样性和群体遗传结构差异水平,为进一步开展黍稷起源地、传播途径以及性状进化等研究奠定基础。1 材料与方法
1.1 材料
所用的146份黍稷资源(12个群体)均由中国农业科学院作物科学研究所小宗作物课题组近几年新收集和引进,包括85份国内野生资源和新引进的61份国外地方品种(电子附表1)。Table S1
附表1
附表1参试材料
Table S1Broomcorn millet varieties and landraces used in this study
| 序号 Number | 群体编号与名称 Group number and name | 材料编号 Material number | 材料来源 Material source |
|---|---|---|---|
| 1 | 1中国山西 Shanxi, China | 大同-1 | 中国山西大同 Datong Shanxi, China |
| 2 | 中国山西 Shanxi, China | 大同-2 | 中国山西大同 Datong Shanxi, China |
| 3 | 中国山西 Shanxi, China | 山西岗县野糜子 | 中国山西岗县 Gangxian Shanxi, China |
| 4 | 2中国 宁夏 Ningxia, China | 宁夏-1 | 中国宁夏 Ningxia, China |
| 5 | 中国宁夏 Ningxia, China | 宁夏-2 | 中国宁夏 Ningxia, China |
| 6 | 中国宁夏 Ningxia, China | 宁夏-3 | 中国宁夏 Ningxia, China |
| 7 | 中国宁夏 Ningxia, China | 宁夏-4 | 中国宁夏 Ningxia, China |
| 8 | 中国宁夏 Ningxia, China | 宁夏-5 | 中国宁夏 Ningxia, China |
| 9 | 中国宁夏 Ningxia, China | 宁夏-6 | 中国宁夏 Ningxia, China |
| 10 | 中国宁夏 Ningxia, China | 宁夏-7 | 中国宁夏 Ningxia, China |
| 11 | 中国宁夏 Ningxia, China | 宁夏-8 | 中国宁夏 Ningxia, China |
| 12 | 中国宁夏 Ningxia, China | 宁夏-9 | 中国宁夏 Ningxia, China |
| 13 | 中国宁夏 Ningxia, China | 宁夏-10 | 中国宁夏 Ningxia, China |
| 14 | 中国宁夏 Ningxia, China | 宁夏-11 | 中国宁夏 Ningxia, China |
| 15 | 中国宁夏 Ningxia, China | 宁夏-12 | 中国宁夏 Ningxia, China |
| 16 | 中国宁夏 Ningxia, China | 宁夏-13 | 中国宁夏 Ningxia, China |
| 17 | 中国宁夏 Ningxia, China | 宁夏-14 | 中国宁夏 Ningxia, China |
| 18 | 中国宁夏 Ningxia, China | 宁夏-15 | 中国宁夏 Ningxia, China |
| 19 | 3 中国内蒙古 Inner Mongolia, China | 赤峰-2 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 20 | 中国内蒙古 Inner Mongolia, China | 赤峰-4 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 21 | 中国内蒙古 Inner Mongolia, China | 赤峰-5 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 22 | 中国内蒙古 Inner Mongolia, China | 赤峰-6 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 23 | 中国内蒙古 Inner Mongolia, China | 赤峰-7 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 24 | 中国内蒙古 Inner Mongolia, China | 赤峰-8 | 中国内蒙古赤峰 Chifeng Inner Mongolia, China |
| 25 | 中国内蒙古 Inner Mongolia, China | 内蒙-2 | 中国内蒙古 Inner Mongolia, China |
| 26 | 中国内蒙古 Inner Mongolia, China | 内蒙-3 | 中国内蒙古 Inner Mongolia, China |
| 27 | 中国内蒙古 Inner Mongolia, China | 内蒙-4 | 中国内蒙古 Inner Mongolia, China |
| 28 | 中国内蒙古 Inner Mongolia, China | 内蒙-5 | 中国内蒙古 Inner Mongolia, China |
| 29 | 中国内蒙古 Inner Mongolia, China | 内蒙-6 | 中国内蒙古 Inner Mongolia, China |
| 30 | 4 中国河北 Hebei, China | 承德-2 | 中国河北承德 Chengde Hebei, China |
| 31 | 中国河北 Hebei, China | 承德-3 | 中国河北承德 Chengde Hebei, China |
| 32 | 中国河北 Hebei, China | 承德-5 | 中国河北承德 Chengde Hebei, China |
| 33 | 中国河北 Hebei, China | 承德-A | 中国河北承德 Chengde Hebei, China |
| 34 | 中国河北 Hebei, China | 承德-6 | 中国河北承德 Chengde Hebei, China |
| 35 | 中国河北 Hebei, China | 承德-7 | 中国河北承德 Chengde Hebei, China |
| 36 | 中国河北 Hebei, China | 承德-8 | 中国河北承德 Chengde Hebei, China |
| 37 | 中国河北 Hebei, China | 承德-9 | 中国河北承德 Chengde Hebei, China |
| 38 | 中国河北 Hebei, China | 承德B | 中国河北承德 Chengde Hebei, China |
| 39 | 中国河北 Hebei, China | 承德-10 | 中国河北承德 Chengde Hebei, China |
| 40 | 中国河北 Hebei, China | 承德-11 | 中国河北承德 Chengde Hebei, China |
| 41 | 中国河北 Hebei, China | 承德C | 中国河北承德 Chengde Hebei, China |
| 42 | 中国河北 Hebei, China | 承德-12 | 中国河北承德 Chengde Hebei, China |
| 43 | 中国河北 Hebei, China | 承德D | 中国河北承德 Chengde Hebei, China |
| 44 | 中国河北 Hebei, China | 河北-4 | 中国河北 Hebei, China |
| 45 | 中国河北 Hebei, China | 河北-5 | 中国河北 Hebei, China |
| 46 | 中国河北 Hebei, China | 河北-6 | 中国河北 Hebei, China |
| 47 | 中国河北 Hebei, China | 河北-9 | 中国河北 Hebei, China |
| 48 | 中国河北 Hebei, China | 河北-10 | 中国河北 Hebei, China |
| 49 | 中国河北 Hebei, China | 河北-14 | 中国河北 Hebei, China |
| 50 | 中国河北 Hebei, China | 河北-15 | 中国河北 Hebei, China |
| 51 | 中国河北 Hebei, China | 河北-16 | 中国河北 Hebei, China |
| 52 | 中国河北 Hebei, China | 河北-17 | 中国河北 Hebei, China |
| 53 | 中国河北 Hebei, China | 河北-19 | 中国河北 Hebei, China |
| 54 | 中国河北 Hebei, China | 河北-20 | 中国河北 Hebei, China |
| 55 | 中国河北 Hebei, China | 河北-7 | 中国河北 Hebei, China |
| 56 | 5 中国辽宁 Liaoning, China | 辽宁-1 | 中国辽宁 Liaoning, China |
| 57 | 中国辽宁 Liaoning, China | 辽宁-2 | 中国辽宁 Liaoning, China |
| 58 | 中国辽宁 Liaoning, China | 辽宁-3 | 中国辽宁 Liaoning, China |
| 59 | 中国辽宁 Liaoning, China | 辽宁-6 | 中国辽宁 Liaoning, China |
| 60 | 中国辽宁 Liaoning, China | 辽宁-7 | 中国辽宁 Liaoning, China |
| 61 | 中国辽宁 Liaoning, China | 辽宁-8 | 中国辽宁 Liaoning, China |
| 62 | 中国辽宁 Liaoning, China | 辽宁-9 | 中国辽宁 Liaoning, China |
| 63 | 6 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-1 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 64 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-2 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 65 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-3 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 66 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-4 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 67 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-5 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 68 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-6 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 69 | 中国黑龙江 Heilongjiang, China | 齐齐哈尔野-7 | 中国黑龙江齐齐哈尔 Qiqihar Heilongjiang, China |
| 70 | 中国黑龙江 Heilongjiang, China | 黑龙江245-1 | 中国黑龙江 Heilongjiang, China |
| 71 | 中国黑龙江 Heilongjiang, China | 黑龙江245-2 | 中国黑龙江 Heilongjiang, China |
| 72 | 中国黑龙江 Heilongjiang, China | 黑龙江245-3 | 中国黑龙江 Heilongjiang, China |
| 73 | 中国黑龙江 Heilongjiang, China | 黑龙江245-4 | 中国黑龙江 Heilongjiang, China |
| 74 | 中国黑龙江 Heilongjiang, China | 黑龙江245-5 | 中国黑龙江 Heilongjiang, China |
| 75 | 中国黑龙江 Heilongjiang, China | 黑龙江245-6 | 中国黑龙江 Heilongjiang, China |
| 76 | 中国黑龙江 Heilongjiang, China | 黑龙江245-7 | 中国黑龙江 Heilongjiang, China |
| 77 | 中国黑龙江 Heilongjiang, China | 黑龙江246-2 | 中国黑龙江 Heilongjiang, China |
| 78 | 中国黑龙江 Heilongjiang, China | 黑龙江246-3 | 中国黑龙江 Heilongjiang, China |
| 79 | 中国黑龙江 Heilongjiang, China | 黑龙江246-4 | 中国黑龙江 Heilongjiang, China |
| 80 | 中国黑龙江 Heilongjiang, China | 黑龙江246-5 | 中国黑龙江 Heilongjiang, China |
| 81 | 中国黑龙江 Heilongjiang, China | 黑龙江246-6 | 中国黑龙江 Heilongjiang, China |
| 82 | 中国黑龙江 Heilongjiang, China | 黑龙江246-8 | 中国黑龙江 Heilongjiang, China |
| 83 | 中国黑龙江 Heilongjiang, China | 黑龙江247-1 | 中国黑龙江 Heilongjiang, China |
| 84 | 中国黑龙江 Heilongjiang, China | 黑龙江247-2 | 中国黑龙江 Heilongjiang, China |
| 85 | 中国黑龙江 Heilongjiang, China | 黑龙江248-1 | 中国黑龙江 Heilongjiang, China |
| 86 | 7 亚洲 Asia | AMES32315 | 格鲁吉亚 Georgia |
| 87 | 亚洲 Asia | AMES32316 | 格鲁吉亚 Georgia |
| 88 | 亚洲 Asia | PI163298 | 印度 India |
| 89 | 亚洲 Asia | PI163300 | 印度 India |
| 90 | 亚洲 Asia | PI170592 | 土耳其 Turkey |
| 91 | 亚洲 Asia | PI207501 | 阿富汗 Afghanistan |
| 92 | 亚洲 Asia | PI211058 | 阿富汗 Afghanistan |
| 93 | 亚洲 Asia | PI211059 | 阿富汗 Afghanistan |
| 94 | 亚洲 Asia | PI211060 | 阿富汗 Afghanistan |
| 95 | 亚洲 Asia | PI212108 | 阿富汗 Afghanistan |
| 96 | 亚洲 Asia | PI202317 | 阿富汗 Afghanistan |
| 97 | 亚洲 Asia | PI253953 | 阿富汗 Afghanistan |
| 98 | 亚洲 Asia | PI253955 | 阿富汗 Afghanistan |
| 99 | 亚洲 Asia | PI649381 | 阿富汗 Afghanistan |
| 100 | 亚洲 Asia | PI269955 | 巴基斯坦 Pakistan |
| 101 | 亚洲 Asia | PI269957 | 巴基斯坦 Pakistan |
| 102 | 亚洲 Asia | PI269958 | 巴基斯坦 Pakistan |
| 103 | 亚洲 Asia | PI269959 | 巴基斯坦 Pakistan |
| 104 | 亚洲 Asia | PI346939 | 哈萨克斯坦 Kazakhstan |
| 105 | 亚洲 Asia | PI427247 | 尼泊尔 Nepal |
| 106 | 亚洲 Asia | PI427247 | 尼泊尔 Nepal |
| 107 | 亚洲 Asia | PI427248 | 尼泊尔 Nepal |
| 108 | 亚洲 Asia | PI427249 | 尼泊尔 Nepal |
| 109 | 亚洲 Asia | PI427250 | 尼泊尔 Nepal |
| 110 | 亚洲 Asia | PI433381 | 中国台湾 Taiwan, China |
| 111 | 8 欧洲 Europe | PI649372 | 法国 France |
| 112 | 欧洲 Europe | PI477123 | 德国 Germany |
| 113 | 欧洲 Europe | PI209790 | 德国 Germany |
| 114 | 欧洲 Europe | PI531407 | 德国 Germany |
| 115 | 欧洲 Europe | PI232929 | 匈牙利 Hungary |
| 116 | 欧洲 Europe | PI289320 | 匈牙利 Hungary |
| 117 | 欧洲 Europe | PI289321 | 匈牙利 Hungary |
| 118 | 欧洲 Europe | PI289324 | 匈牙利 Hungary |
| 119 | 欧洲 Europe | PI290726 | 英国 England |
| 120 | 欧洲 Europe | PI346934 | 乌克兰 Ukraine |
| 121 | 欧洲 Europe | PI346941 | 乌克兰 Ukraine |
| 122 | 欧洲 Europe | PI346942 | 乌克兰 Ukraine |
| 123 | 欧洲 Europe | PI346933 | 苏联 Soviet Union |
| 124 | 欧洲 Europe | PI346935 | 苏联 Soviet Union |
| 125 | 欧洲 Europe | PI346944 | 苏联 Soviet Union |
| 126 | 欧洲 Europe | PI346945 | 苏联 Soviet Union |
| 127 | 欧洲 Europe | PI442533 | 比利时 Belgium |
| 128 | 欧洲 Europe | PI516181 | 罗马尼亚 Romania |
| 129 | 欧洲 Europe | PI531399 | 保加利亚 Bulgaria |
| 130 | 欧洲 Europe | PI531402 | 捷克斯洛伐克 Czechoslovakia |
| 131 | 欧洲 Europe | PI531406 | 捷克斯洛伐克 Czechoslovakia |
| 132 | 9 南美洲 South America | PI202294 | 阿根廷 Argentina |
| 133 | 南美洲 South America | PI202295 | 阿根廷 Argentina |
| 134 | 10 北美洲 North America | PI296376 | 加拿大 Canada |
| 135 | 北美洲 North America | PI677103 | 美国 America |
| 136 | 北美洲 North America | PI649385 | 美国 America |
| 137 | 北美洲 North America | PI649384 | 美国 America |
| 138 | 北美洲 North America | PI649383 | 美国 America |
| 139 | 11 大洋洲 Oceania | PI365040 | 澳大利亚 Australia |
| 140 | 大洋洲 Oceania | PI365842 | 澳大利亚 Australia |
| 141 | 大洋洲 Oceania | PI365845 | 澳大利亚 Australia |
| 142 | 大洋洲 Oceania | PI367683 | 澳大利亚 Australia |
| 143 | 大洋洲 Oceania | PI367684 | 澳大利亚 Australia |
| 144 | 12 非洲 Africa | PI517016 | 摩洛哥 Morocco |
| 145 | 非洲 Africa | PI517017 | 摩洛哥 Morocco |
| 146 | 非洲 Africa | PI531419 | 肯尼亚 Kenya |
新窗口打开
Table S2
附表2
附表2所用SSR引物的序列及退火温度
Table S2Sequence of SSR primers and annealing temperature(Supplement)
| 位点 Locus | 正向引物 Forward primer (5'-3') | 反向引物 Reverse primer (5'-3') | 退火温度 Tm (℃) |
|---|---|---|---|
| BM5 | CCCTTCTTCCCTACTTTGCC | GTGTGCGTGCATGGGTGT | 55 |
| BM114 | ATCGTAGAAACCATTGGCCC | TGACCCATGGACACTTTTCA | 55 |
| BM136 | AATGTCACAGGTTTCCCTCG | GCGAGAAAGAGGAGAGGGTT | 55 |
| BM289 | TGGGACAATATGGCAAGGTT | ACAAATGCCTGATGGTAGGC | 55 |
| BM295 | CACACAGATATTTGGCACCG | TGAGGATCCGAAAAGATTGG | 55 |
| BM306 | ATTTTCTGGGCAATTCAACG | GTCCTCATCCCTTCCCTCTC | 55 |
| BM344 | AGCACTGAGGCACAATTCCT | GTGCTGGGGTTTGTGACTTT | 55 |
| BM374 | CTACCGCTTCAAAACGAAGG | TGTCCCACTCTCCTACCTACTACC | 55 |
| BM378 | ATGGGATGCACAGGTACACA | TCCTTAGGTCATCGTCCTATTTG | 55 |
| BM396 | TTGATTATGCTTTGGAGGGG | CCTCTCCTTACACGGGGATT | 55 |
| BM484 | GGACAGGATGAGGAGGATCA | CGGTTACCATCGCCTTCTTA | 55 |
| BM552 | GCAGCAATAGATTTGCCTCC | TCTAGCCTACCCGAACTTGC | 55 |
| BM637 | CACACTTGTGCTGTTGGGTC | TCTAGCCTACCCCAACTTGC | 55 |
| BM787 | TCCGTGACATGATGCAGACT | GCACTATTTCTGCAGGAGCC | 55 |
| BM994 | AACAGAACCGCACATCAGTG | TCAGGTGAGCGATTCTTCCT | 55 |
| BM1303 | TTGACGCGTACAACAAGAGC | ACCGAAGAGAAGGTTGCTGA | 55 |
| BM1332 | CGGTTCAGTATTCAGGGCAT | CACTACACGTACCCGGTCCT | 55 |
| BM1419 | CGGTTCAGTATTCAGGGCAT | CACAAGGATCAGGCCAAAGT | 55 |
| BM1477 | GCGAAAGATGAACTGCCAAT | GCTGCGACGGATACTGATCT | 55 |
| BM1533 | CTCTTGTCGTCTTGGTCCGT | CGTGCGTGTGTCGAGAGAG | 55 |
| BM1745 | CAGCTGATCATTTGAAAAGTATGG | TAGTTGTCGCGATCATGAGG | 55 |
| BM4724 | TGCATGAAGAAGTGCAGGTG | ATGATGCCCAAGAGATCGAC | 55 |
| BM4739 | GGATGAGAGGAAGGGGTTGT | CTTCCCTCCAAACTTCCTCC | 55 |
| BM4741 | GTCTGCGTACCTCGGAAGTC | TTCCCTCCAAACTTCCTCCT | 55 |
| BM4749 | TCTGCGTACCTCGGAAGTCT | TTCCCTCCAAACTTCCTCCT | 55 |
| BM4761 | GATCACAAAGGGGAACAGGA | ACATGAGCCAGCTCTCCACT | 55 |
| BM4830 | GTTGAAGGCAGACACCTTCC | CTCCTCCCTCTCCCTCCC | 55 |
| BM4846 | TCTAGCCTACCCCAACTTGC | ACAAAACCGCTGATCTGCTT | 55 |
| BM4847 | GGACGAGTCGGTGAAGAGAG | AATTTCTGTAGCGGTGTCGC | 55 |
| BM4851 | TTCAATCCAATCCAAGGAGC | AACCTTGAAGACATGAGCTGG | 55 |
| BM4858 | CCCAACTTGCTTGGGATAAA | ACCTGATGGCATATTGCTCC | 55 |
| BM4871 | CCAGTCGGTGAAGAGAGACC | CTCTGTACTCGAGGGCGGT | 55 |
| BM4877 | CGCTCCTTTCCTTTCTGTTG | AGGAGCCTACTGCTGGAACA | 55 |
| BM4882 | TGGGTGTCAGCTTGACTCAG | TGCTTACATCCACTGCTTGC | 55 |
| BM4947 | GATTTGGGACGAGTCGGTTA | AAAGAATTTTCGGGCGACTT | 55 |
| BM4954 | GGCGGATCGATGATTGTAGT | TAAACGAGGGCAGAAAGGAA | 55 |
| BM4956 | GGCTGTAAATCCGCTCGTAG | TCCCTCTAATGCAGCCAGTT | 55 |
| BM4958 | TCCATAGCTTCCATAGCATTTTC | CCCAAAGGATATGGTTCCAA | 55 |
| BM4962 | TCCATTGCTAGCGTTCTCCT | TAGCGCCAACATGGTGTAGA | 55 |
| BM4965 | CCTCCATCGAGAAACCAACT | TGCAGAGACATGCATCACAA | 55 |
| BM4967 | CTTCACCCACCACACCTTCT | AGAATCTGACACCTGCACCA | 55 |
| BM4969 | CTTCACCCACCACACCTTCT | GAATCTGACACCTGCACCAA | 55 |
| BM4973 | TTATCTTCCTCCGCCTCATC | TGCATCACAACTGAAGACGA | 55 |
| BM4997 | TTCTATCGCTGTGGTGGTCA | CAACTGCTTCTACAACGGCA | 55 |
| BM5037 | ATTCCACCCTTGACCTCCTT | TGCATTGACTGCCTTGAAAC | 55 |
| BM5038 | GGTCTCTTAAGGAGGGCTGG | TGCATTGACTGCCTTGAAAC | 55 |
| BM5043 | TGACGAGATCATCTGGCTTG | GAAAAGACAGGAGCGCAAAC | 55 |
| BM5181 | CCTCACGGTGCATCAGAGT | CCAGAGAGCTAAGATATAGGGAATC | 55 |
| BM5190 | ACGATCACGAATAAATCCGC | TTGAGATGATTCCTGTGTGTGTC | 55 |
| BM5191 | GCCCGATAGCCAACGTAATA | ATGTTTTGAAGGCCCTATGC | 55 |
| BM5197 | CTCTGCAACACTGCCACCTA | TTTACACCGTCGAATTTTATGA | 55 |
| BM5198 | CGGAAATAAAGCAACCATGC | ATTGTGTGCTAGGCCACCTC | 55 |
| BM5199 | TCACGTCCCTACACTTCACG | GAGGTCGACAGGAAATGCTC | 55 |
| BM5200 | CGGAAATAAAGCAACCATGC | GACTGACGTAGGGCGTTCTC | 55 |
| BM5222 | TGGAGTGTGTGTGTATGTGTGTG | CTTGTAGGGCTTGTGGCAAT | 55 |
| BM5233 | CTGCTAGAAAAGGACGGGTG | CTAGCTAGCCAGGCCCATC | 55 |
| BM5236 | CATGCCTCATCCCCTTATTC | CGAAACGGCAAACACAAATA | 55 |
| BM5255 | GTAAATTCAGATGCTTGCCG | CCATCCGAGTTCAAGTTGGT | 55 |
| BM5259 | CATACTTCCACACGTGCCTG | GGACTATTCTTTGCTACTCCGGT | 55 |
| BM5260 | GCGTGCGATAATCATTCGT | GCCTACAGAAATATCAGCTGCG | 55 |
| BM5278 | CGCCGTTTCATTTTTCTTTC | TCACACACACACTACACTCACACTC | 55 |
| BM5279 | CGCCGTTTCATTTTCTTTCT | CTCGCTCACACACACACTCA | 55 |
| BM5292 | GCATGCATGTGTGTGTGTGT | AACATATGTGGACGGTTGGC | 55 |
| LMX258 | GTGTCTCTTTCGTCTTGCCC | GGGACACTTCCACCATCATC | 55 |
| LMX503 | GTGGTACAGCTGCTCGTTCA | AGGAGGAACCAGGAAGCAAT | 55 |
| LMX515 | CGTTTTCTCGCTACACACGA | TGGACAACGGAAAACGTACA | 55 |
| LMX621 | ATGAATCACCCGATCCACAT | ACGCCAACATCAGCATATCA | 55 |
| LMX632 | GCTGTCGGTCAGTCCTGTTT | ACGCCAACATCAGCATATCA | 55 |
| LMX635 | GCACACGCATCATCACAAGT | GCTCATTCAACGACAGATGC | 55 |
| LMX645 | GCTAGGTGTCGTTTTCTCGC | GGGGCGTCTAAGCTTGATATT | 55 |
| LMX653 | CGATGAACGAAAATTCACCC | GTTCATTCGTCCAAATGCCT | 55 |
| LMX684 | GGCCTCCTGTGGTCTTCTCT | GACCCTCTCCTCCTACCACC | 55 |
| LMX786 | CCTGGACACACACACACACA | TCTTGTCACTGTCGGCGTAG | 55 |
| LMX691 | ACTCATGGTTACGGCAACTG | GCGCGAGAGAGAGAGAGAGA | 55 |
| LMX836 | GCGCAGTAATATATTTCAGTAATTCA | GCATCATCGTCAAGACCTCA | 55 |
| LMX1067 | ATCGACGACTAGGCCCTGTA | TGCGGAGTGTCTTTGTTCTG | 55 |
| LMX1080 | ATCGACGACTAGGCCCTGTA | GGCCGTCACTATATCTGTCACC | 55 |
| LMX1220 | AGTTACTTTTCCACACCCGC | TTTATTGGGAGTTCGGATGG | 55 |
| LMX1251 | GCAGTTTTGTTTCGATCGGT | TGGTGTCAAGACATGTGGATG | 55 |
| LMX1380 | GCCTCCTGTCTTGTAGCGTC | AGGGTAGGCTGAGAGCCTGT | 55 |
| LMX1387 | TTTCAGGGACTGGACTGGAC | GTAGGGGGTAGCTGAGAGCC | 55 |
| LMX1400 | AGCAACGGAGGTGAGAGAGA | TCGACACACACGACACACAC | 55 |
| LMX1429 | AATATCCCTTTTGTCGCACG | ATGCATTGATGGGCTTGATT | 55 |
| LMX1625 | ACCCAACCGTATATCCAACG | TGTCACAGTTGTCCTGGCAT | 55 |
| LMX1672 | ATTTGACCTGTGACCTCGCT | CCTTTCTGTTTCTGCAAGCC | 55 |
| LMX1760 | TGTGGGAGAGAAGTGGGC | CAAGGAAGGAATAAACCGCA | 55 |
| LMX1749 | CTCAACACCACACCTTCCCT | CTTTTGATCCATGGGCGTAG | 55 |
| LMX1761 | GAGATGGTGCGGATTCTGAG | TCATTTCCACTGTCACTGCC | 55 |
| LMX1959 | GTGGTGAGAAGAAGCCTTGC | GATGGGTCGATCAGCAGAAT | 55 |
| LMX2175 | GTGACCGACGACTACCGTTT | CACCCCTTTCCTCTCGCTAT | 55 |
| LMX2410 | CTCAACACCACACCTTCCCT | CTTTTGATCCATGGGCGTAG | 55 |
| LMX1940 | GCAGTGGGTCAGCTTATGGT | TCTCTCTGTGTGTGTGCGTG | 55 |
| LMX2019 | CCTCTCCTTACACGGGGATT | TTGATTATGCTTTGGAGGGG | 55 |
| LMX2068 | TGAGATTGGCATCAAGCAAG | TTTCTGGTCAGTTCGGTCAG | 55 |
| LMX2074 | GCCACACTAAATAAGCTTTGTGTC | TGGTCGTCACTGATTACGGA | 55 |
| LMX2185 | GAGTTAGAGGACAGCGTGGC | TGCAGCAGAGAATGTGCTACT | 55 |
| LMX2281 | TTCTCCGTCAGCTCACATTG | TCCATTGTTCATTTAGTAGAAACCT | 55 |
| LMX2305 | CGCACTAGCCCTTGTCTTTC | CGCCCTACGAACAAATCACT | 55 |
| LMX2370 | CCAAGTCCCAAGGTTTTTCA | GAGAGAGAGAGGCGTGTGCT | 55 |
| LMX2382 | CAACAAGGTTGGTTGGCTTT | ATGCTGCTGCAGATGTTTTG | 55 |
| LMX2540 | TATTCGAGCCCCATTTCTTG | GCGTTATCCGGATGATGAAG | 55 |
| LMX2734 | CACACAGATATTTGGCACCG | TGAGGATCCGAAAAGATTGG | 55 |
| LMX2782 | GCCGGAGTATAGATCCGACA | GTCAGGCCGTGAACGTTATT | 55 |
新窗口打开
1.2 DNA的提取与检测
选取50 mg室内花盆培养出苗20 d左右的幼叶,用DNA提取试剂盒(北京鼎国昌盛生物有限公司)提取基因组DNA。采用1%的琼脂糖凝胶电泳检测DNA的质量,用超微量分光光度计(P360)测定其浓度。调试其终浓度至30 ng·μL-1,-20℃保存备用。1.3 SSR分析
选用6份地理差异显著的黍稷种质资源对137对小宗作物课题组开发的具有多态性的SSR引物进行初步筛选,最终筛选103对条带清晰、扩增良好且多态性稳定的SSR引物,用于后续遗传多样性分析。PCR扩增反应在MY-CYCLER PCR仪上进行。PCR反应体系包括1.6 μL 10×PCR buffer、0.8 μL 2.5 mmol·L-1 dNTP、0.1 μL Taq酶(5.0 U·μL-1)、0.5 μL引物、0.1 μL DNA模版和5.5 μL ddH2O。PCR扩增程序为94℃ 5 min;94℃ 45 s,55℃ 50 s,72℃ 1 min,39个循环;72℃ 10 min;4℃保存。用8%的聚丙烯酰胺凝胶电泳分离PCR产物,硝酸银染色显影。
1.4 数据分析
分析SSR标记扩增条带,如果在至少2份资源中观察到不同大小的2条DNA条带,则SSR标记被认为是多态性的,有条带记为“1”,无条带记为“0”。利用以下公式计算每个SSR标记物的分辨率(resolving power,Rp值):Rp=∑Ib,Ib=1-(2×︱0.5-P︱),其中Ib为某等位基因信息量,P为某等位基因在6份材料中出现的频率。分别按照地理来源与国家来源,将146份黍稷资源分为12个群体和30个群体,利用PowerMarker3.25和MEGA7计算不同群体间和群体内各位点的PIC值及遗传距离,并绘制聚类图。利用PopGen1.32计算不同群体间和群体内各位点的遗传多样性指标,包括观测等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)及Shannon-Weaver指数(I)。利用Structure2.3.4对黍稷资源进行遗传结构分析。2 结果
2.1 SSR标记分析
使用103对SSR标记对146份糜子资源进行检测(表1),共检测出308个等位基因(Na),平均值为2.9903,其中有效等位基因(Ne)为236.55个,平均值为2.2966,所占比重为76.8%。检测的等位基因数的范围为1(BM994、BM5191和LMX635)—6(BM5038),其中,62对标记检测到多个等位基因(3—6),38对标记检测到2个等位基因,3对标记检测到单个等位基因。Table 1
表1
表1103对SSR标记的遗传参数
Table 1Genetic parameters from the 103 polymorphic SSR markers used in this study
| 位点 Locus | 观测等位基因 Na | 有效等位基因 Ne | 多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | Nei's期望 杂合度 Nei | 多态性信息 含量指数 PIC | 分辨率 Rp |
|---|---|---|---|---|---|---|---|---|
| BM5 | 2 | 1.2565 | 0.3576 | 0.2308 | 0.2053 | 0.2041 | 0.5269 | 1.001 |
| BM114 | 2 | 1.9915 | 0.6910 | 0.0000 | 0.5002 | 0.4979 | 0.5847 | 1.001 |
| BM136 | 3 | 1.3930 | 0.5512 | 0.0000 | 0.2835 | 0.2821 | 0.4847 | 0.668 |
| BM289 | 3 | 1.8225 | 0.6754 | 0.6562 | 0.4531 | 0.4513 | 0.4968 | 2.001 |
| BM295 | 2 | 1.8502 | 0.6521 | 0.4715 | 0.4614 | 0.4595 | 0.6325 | 2.001 |
| BM306 | 3 | 2.7101 | 1.0391 | 0.2500 | 0.6336 | 0.6310 | 0.7642 | 1.668 |
| BM344 | 2 | 1.7415 | 0.6170 | 0.0246 | 0.4275 | 0.4258 | 0.5282 | 1.001 |
| BM374 | 4 | 2.3928 | 1.0022 | 0.0948 | 0.5846 | 0.5821 | 0.6966 | 1.334 |
| BM378 | 3 | 2.2358 | 0.9262 | 0.0089 | 0.5552 | 0.5527 | 0.6360 | 1.669 |
| BM396 | 4 | 3.6430 | 1.3358 | 0.6064 | 0.7294 | 0.7255 | 0.6398 | 1.336 |
| BM484 | 4 | 2.3389 | 0.9574 | 0.8871 | 0.5748 | 0.5725 | 0.3753 | 2.002 |
| BM552 | 2 | 1.9912 | 0.6909 | 0.0833 | 0.4999 | 0.4978 | 0.6173 | 1.334 |
| BM637 | 3 | 2.3849 | 0.9628 | 0.0781 | 0.5830 | 0.5807 | 0.6654 | 1.001 |
| BM787 | 2 | 1.9716 | 0.6859 | 0.0400 | 0.4953 | 0.4928 | 0.6137 | 1.001 |
| BM994 | 1 | 1.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0640 | 1.000 |
| BM1303 | 4 | 2.2497 | 0.9907 | 0.3566 | 0.5577 | 0.5555 | 0.5251 | 0.668 |
| BM1332 | 4 | 2.8550 | 1.1246 | 0.7327 | 0.6530 | 0.6497 | 0.5680 | 2.002 |
| BM1419 | 4 | 1.6182 | 0.6285 | 0.4706 | 0.3834 | 0.3820 | 0.4808 | 2.003 |
| BM1477 | 3 | 1.9241 | 0.7037 | 0.0942 | 0.4820 | 0.4803 | 0.5392 | 3.335 |
| BM1533 | 3 | 2.2618 | 0.8869 | 0.7523 | 0.5604 | 0.5579 | 0.5590 | 0.334 |
| BM1745 | 3 | 2.3735 | 0.9358 | 0.0690 | 0.582 | 0.5787 | 0.6554 | 2.668 |
| BM4724 | 2 | 2.0000 | 0.6931 | 0.9167 | 0.5021 | 0.5000 | 0.3595 | 0.668 |
| BM4739 | 2 | 1.0083 | 0.0268 | 0.0083 | 0.0083 | 0.0082 | 0.2554 | 0.334 |
| BM4741 | 2 | 1.9917 | 0.6910 | 0.9353 | 0.4997 | 0.4979 | 0.1914 | 0.334 |
| BM4749 | 2 | 1.9975 | 0.6925 | 0.9648 | 0.5011 | 0.4994 | 0.1142 | 0.668 |
| BM4761 | 2 | 1.8248 | 0.6443 | 0.6901 | 0.4536 | 0.4520 | 0.3752 | 1.001 |
| 位点 Locus | 观测等位基因 Na | 有效等位基因 Ne | 多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | Nei's期望 杂合度 Nei | 多态性信息 含量指数 PIC | 分辨率 Rp |
| BM4830 | 2 | 1.9976 | 0.6925 | 0.9653 | 0.5011 | 0.4994 | 0.0898 | 0.668 |
| BM4846 | 2 | 1.9879 | 0.6901 | 0.8594 | 0.5009 | 0.4969 | 0.4484 | 0.668 |
| BM4847 | 2 | 1.9996 | 0.6930 | 0.0141 | 0.5017 | 0.4999 | 0.4327 | 1.001 |
| BM4851 | 2 | 1.9999 | 0.6931 | 0.9920 | 0.5020 | 0.5000 | 0.2280 | 0.668 |
| BM4858 | 3 | 2.7031 | 1.0374 | 0.0709 | 0.6323 | 0.6300 | 0.6300 | 2.001 |
| BM4871 | 2 | 1.3308 | 0.4147 | 0.2909 | 0.2497 | 0.2486 | 0.5378 | 1.667 |
| BM4877 | 2 | 1.9929 | 0.6914 | 0.9403 | 0.5001 | 0.4982 | 0.2309 | 2.000 |
| BM4882 | 3 | 1.9758 | 0.8215 | 0.1615 | 0.4958 | 0.4939 | 0.6368 | 1.335 |
| BM4947 | 3 | 2.0764 | 0.7801 | 0.8043 | 0.5203 | 0.5184 | 0.3802 | 0.334 |
| BM4954 | 2 | 1.9991 | 0.6929 | 0.9648 | 0.5015 | 0.4998 | 0.1152 | 1.001 |
| BM4956 | 5 | 3.4708 | 1.3661 | 0.2114 | 0.7148 | 0.7119 | 0.8035 | 3.335 |
| BM4958 | 4 | 3.4478 | 1.2918 | 0.2743 | 0.7131 | 0.7100 | 0.7883 | 2.335 |
| BM4962 | 3 | 2.8703 | 1.0762 | 0.0230 | 0.6554 | 0.6516 | 0.6699 | 2.001 |
| BM4965 | 3 | 1.3579 | 0.4981 | 0.2302 | 0.2645 | 0.2635 | 0.4106 | 2.668 |
| BM4967 | 3 | 2.9301 | 1.0862 | 0.0672 | 0.6615 | 0.6587 | 0.7226 | 2.334 |
| BM4969 | 2 | 1.4776 | 0.5041 | 0.3784 | 0.3254 | 0.3232 | 0.5539 | 0.667 |
| BM4973 | 2 | 1.9987 | 0.6928 | 0.3590 | 0.5029 | 0.4997 | 0.6401 | 0.668 |
| BM4997 | 2 | 1.4516 | 0.4901 | 0.3853 | 0.3125 | 0.3111 | 0.5699 | 2.002 |
| BM5037 | 3 | 2.8324 | 1.0669 | 0.1463 | 0.6509 | 0.6469 | 0.6741 | 1.668 |
| BM5038 | 6 | 4.0419 | 1.4982 | 0.1484 | 0.7555 | 0.7526 | 0.7900 | 3.002 |
| BM5043 | 3 | 2.3529 | 0.9352 | 0.0439 | 0.5775 | 0.5750 | 0.6650 | 1.334 |
| BM5181 | 5 | 4.8801 | 1.5964 | 0.0204 | 0.7992 | 0.7951 | 0.7777 | 1.335 |
| BM5190 | 2 | 1.9980 | 0.6926 | 0.8421 | 0.5021 | 0.4995 | 0.4943 | 1.001 |
| BM5191 | 1 | 1.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.2499 | 1.000 |
| BM5197 | 4 | 3.3698 | 1.2706 | 0.0816 | 0.7069 | 0.7032 | 0.7446 | 1.668 |
| BM5198 | 5 | 4.137 | 1.4859 | 0.0306 | 0.7622 | 0.7583 | 0.7582 | 0.668 |
| BM5199 | 3 | 2.2909 | 0.9548 | 0.0286 | 0.5662 | 0.5635 | 0.6581 | 1.335 |
| BM5200 | 3 | 2.7989 | 1.0603 | 0.0444 | 0.6463 | 0.6427 | 0.6837 | 0.668 |
| BM5222 | 2 | 1.9623 | 0.6835 | 0.0146 | 0.4922 | 0.4904 | 0.4688 | 1.667 |
| BM5233 | 3 | 2.1754 | 0.8627 | 0.1071 | 0.5436 | 0.5403 | 0.6430 | 1.001 |
| BM5236 | 5 | 3.8616 | 1.4480 | 0.0093 | 0.7445 | 0.7410 | 0.7622 | 2.003 |
| BM5255 | 4 | 3.6971 | 1.3394 | 0.0851 | 0.7334 | 0.7295 | 0.7467 | 2.668 |
| BM5259 | 2 | 1.8682 | 0.6574 | 0.0469 | 0.4684 | 0.4647 | 0.5271 | 1.001 |
| BM5260 | 4 | 3.8839 | 1.3702 | 0.2400 | 0.7455 | 0.7425 | 0.7789 | 3.001 |
| BM5278 | 2 | 1.9117 | 0.6699 | 0.2243 | 0.4791 | 0.4769 | 0.6784 | 1.001 |
| BM5279 | 4 | 2.328 | 0.9643 | 0.4015 | 0.5726 | 0.5704 | 0.6987 | 1.669 |
| BM5292 | 2 | 1.4356 | 0.4811 | 0.2460 | 0.3047 | 0.3034 | 0.5285 | 1.001 |
| LMX258 | 3 | 2.1764 | 0.9221 | 0.2520 | 0.5427 | 0.5405 | 0.6832 | 1.668 |
| 位点 Locus | 观测等位基因 Na | 有效等位基因 Ne | 多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | Nei's期望 杂合度 Nei | 多态性信息 含量指数 PIC | 分辨率 Rp |
| LMX503 | 4 | 3.3528 | 1.3002 | 0.3571 | 0.7053 | 0.7017 | 0.6855 | 1.669 |
| LMX515 | 5 | 2.0152 | 0.9363 | 0.1967 | 0.5058 | 0.5038 | 0.5872 | 2.668 |
| LMX621 | 5 | 3.7329 | 1.375 | 0.5283 | 0.7356 | 0.7321 | 0.6647 | 0.668 |
| LMX632 | 3 | 2.3143 | 0.9109 | 0.8889 | 0.5703 | 0.5679 | 0.4042 | 1.334 |
| LMX635 | 1 | 1.0000 | 0.0000 | 0.0000 | 0.0000 | 0.0000 | 0.2981 | 0.334 |
| LMX645 | 3 | 2.9129 | 1.0834 | 0.4701 | 0.6595 | 0.6567 | 0.6549 | 2.002 |
| LMX653 | 2 | 1.7016 | 0.6027 | 0.5128 | 0.4141 | 0.4123 | 0.5904 | 1.334 |
| LMX684 | 2 | 1.7813 | 0.6304 | 0.6496 | 0.4402 | 0.4386 | 0.4325 | 1.335 |
| LMX786 | 2 | 1.2215 | 0.3269 | 0.0672 | 0.1821 | 0.1813 | 0.4216 | 0.668 |
| LMX691 | 2 | 1.2575 | 0.3585 | 0.2316 | 0.2058 | 0.2048 | 0.5274 | 1.667 |
| LMX836 | 5 | 2.8613 | 1.1653 | 0.3780 | 0.6531 | 0.6505 | 0.7013 | 2.668 |
| LMX1067 | 5 | 2.546 | 1.1267 | 0.2913 | 0.6102 | 0.6072 | 0.6619 | 2.000 |
| LMX1080 | 4 | 3.1104 | 1.2160 | 0.4615 | 0.6814 | 0.6785 | 0.6495 | 3.335 |
| LMX1220 | 5 | 2.1885 | 1.1030 | 0.2266 | 0.5452 | 0.5431 | 0.5782 | 2.668 |
| LMX1251 | 5 | 2.2345 | 1.0430 | 0.3119 | 0.5550 | 0.5525 | 0.6052 | 2.002 |
| LMX1380 | 2 | 1.9996 | 0.6930 | 0.1000 | 0.5017 | 0.4999 | 0.5392 | 1.334 |
| LMX1387 | 2 | 1.9844 | 0.6892 | 0.0242 | 0.4981 | 0.4961 | 0.5566 | 1.334 |
| LMX1400 | 3 | 2.4945 | 0.9791 | 0.0408 | 0.6022 | 0.5991 | 0.6757 | 2.000 |
| LMX1429 | 2 | 1.3888 | 0.4532 | 0.0396 | 0.2814 | 0.2800 | 0.5090 | 0.334 |
| LMX1625 | 3 | 2.9380 | 1.0880 | 0.5464 | 0.6631 | 0.6596 | 0.6223 | 2.000 |
| LMX1672 | 2 | 1.9642 | 0.6840 | 0.8492 | 0.4929 | 0.4909 | 0.3918 | 0.668 |
| LMX1760 | 4 | 2.6827 | 1.1341 | 0.2286 | 0.6295 | 0.6272 | 0.6324 | 1.335 |
| LMX1749 | 3 | 1.9862 | 0.8570 | 0.1574 | 0.4988 | 0.4965 | 0.6170 | 3.001 |
| LMX1761 | 4 | 3.1087 | 1.2338 | 0.1971 | 0.6808 | 0.6783 | 0.6482 | 4.002 |
| LMX1959 | 3 | 2.5044 | 1.0042 | 0.0312 | 0.6031 | 0.6007 | 0.6492 | 1.001 |
| LMX2175 | 4 | 2.5274 | 1.0572 | 0.2238 | 0.6065 | 0.6043 | 0.6920 | 1.668 |
| LMX2410 | 3 | 2.7092 | 1.0394 | 0.7128 | 0.6343 | 0.6309 | 0.5818 | 2.000 |
| LMX1940 | 3 | 2.1372 | 0.8179 | 0.9432 | 0.5351 | 0.5321 | 0.4158 | 1.334 |
| LMX2019 | 3 | 2.7286 | 1.0436 | 0.1304 | 0.6370 | 0.6335 | 0.7104 | 1.668 |
| LMX2068 | 5 | 3.1166 | 1.3036 | 0.0286 | 0.6824 | 0.6791 | 0.7290 | 2.336 |
| LMX2074 | 3 | 1.4252 | 0.5352 | 0.1385 | 0.2995 | 0.2983 | 0.4854 | 1.001 |
| LMX2185 | 3 | 2.4666 | 0.9893 | 0.0103 | 0.5977 | 0.5946 | 0.6571 | 1.335 |
| LMX2281 | 2 | 1.9944 | 0.6917 | 0.0088 | 0.5008 | 0.4986 | 0.5806 | 1.001 |
| LMX2305 | 4 | 3.4614 | 1.3025 | 0.1639 | 0.7170 | 0.7111 | 0.5894 | 0.668 |
| LMX2370 | 2 | 1.1542 | 0.2587 | 0.0682 | 0.1341 | 0.1336 | 0.3140 | 1.000 |
| LMX2382 | 2 | 1.9668 | 0.6847 | 0.0649 | 0.4948 | 0.4916 | 0.5882 | 1.001 |
| LMX2540 | 2 | 1.0973 | 0.1881 | 0.0930 | 0.0890 | 0.0887 | 0.3112 | 0.667 |
| LMX2734 | 4 | 3.8637 | 1.3679 | 0.3077 | 0.7440 | 0.7412 | 0.7392 | 2.669 |
| LMX2782 | 3 | 1.7210 | 0.7103 | 0.3985 | 0.4205 | 0.4189 | 0.5599 | 2.000 |
| 平均值Mean | 2.9903 | 2.2966 | 0.8478 | 0.4871 | 0.5105 | 0.3642 | 0.5544 | |
| 标准差SD | 1.0891 | 0.7841 | 0.3440 | 0.1784 | 0.1775 | 0.1434 |
新窗口打开
Shannon-Weaver指数范围(I)为0(BM994、BM5191和LMX635)—1.5964(BM5181),平均值为0.8478.。观测杂合度(Ho)范围为0(BM114、BM136、BM994、BM5191和LMX635)—0.9920(BM4851),平均值为0.4871。期望杂合度(He)范围为0(BM994、BM5191和LMX635)—0.7992(BM5181),平均值为0.3642。Nei's期望杂合度范围为0(BM994、BM5191和LMX635)—0.7951(BM5181)。PIC值范围为0.0640(BM994)—0.8035(BM4956),平均值为0.5544。一些SSR标记(BM114、BM136、BM994、BM5191和LMX635)的Shannon-Weaver指数、观测杂合度、期望杂合度、Nei's期望杂合度为0,表明个体之间存在普遍异交。
引物分辨率(Rp值)指不同标记对不同基因型的辨别能力,引物分辨率越高,其对不同基因的分辨能力越强,因此从各标记的高分辨能力也可以观察到这些标记的高多态性。103对SSR标记的Rp值范围为0.334(BM1533、BM4739、BM4741、BM4947、LMX635和LMX1429)—4.002(LMX1761)。Rp值的分布区间为0—1、1—2、2—3、3—4和4—5,分布频率分别为21.36%、45.63%、26.21%、5.83%和0.97%(图1),由以上数据可知,77.67%的标记分布区间1—4,具有适度分辨力,选择有效的标记利于后续遗传多样性的分析。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1103个SSR标记的分辨率(Rp值)
-->Fig. 1Resolving power values of 103 SSR marker in broomcorn millet
-->
2.2 不同群体间的遗传多样性分析
利用PopGen1.3.2软件分析12个不同群体间黍稷资源的遗传多样性(表2),结果表明,各群体间等位基因(Na)变化范围为1.4947—2.6602,最大的为中国河北群体,最小的为南美洲群体。有效等位基因(Ne)变化范围为1.4510—2.0961,均值为1.8037。就国内外两大群体而言(表3),国内各群体的有效等位基因数(2.2620±0.7862)大于国外群体(2.0429±0.6601),国内群体间的有效等位基因范围为1.6302—2.0961(河北>黑龙江>辽宁>宁夏>内蒙古>山西),均值为1.8740,比全部参试群体的平均有效等位基因高0.0703。国外群体间的有效等位基因范围为1.4510—1.9860(欧洲>亚洲>北美洲>大洋洲>非洲>南美洲),均值为1.7334,比全部参试群体的平均有效等位基因低0.0703。由以上数据可知国内外群体间遗传丰富度差异不大,国内野生糜子遗传丰富度略大于国外地方品种。Table 2
表2
表212个群体的遗传多样性分析
Table 2Estimates of genetic diversity within 12 populations
| 群体 Population | 观测等位基因 Na | 有效等位基因 Ne | 多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | Nei's期望杂合度 Nei | 多态性信息含量指数 PIC |
|---|---|---|---|---|---|---|---|
| 中国山西 Shanxi, China | 1.7083±0.6475 | 1.6302±0.6012 | 0.4393±0.3741 | 0.3177±0.4168 | 0.4073±0.3534 | 0.3005±0.2489 | 0.2910 |
| 中国宁夏 Ningxia, China | 2.3069±0.8688 | 1.8465±0.6811 | 0.6145±0.3762 | 0.3206±0.3429 | 0.4020±0.2382 | 0.3816±0.2257 | 0.3845 |
| 中国内蒙古 Inner Mongolia, China | 1.9406±0.7723 | 1.7138±0.6396 | 0.5076±0.3839 | 0.3079±0.3914 | 0.3533±0.2586 | 0.3320±0.2432 | 0.3419 |
| 中国河北 Hebei, China | 2.6602±0.8695 | 2.0961±0.6902 | 0.7608±0.3409 | 0.2818±0.3376 | 0.4789±0.1975 | 0.4659±0.1915 | 0.4728 |
| 中国辽宁 Liaoning, China | 2.3168±0.8238 | 1.9491±0.6763 | 0.6730±0.3498 | 0.3443±0.3573 | 0.4754±0.2299 | 0.4239±0.2020 | 0.4305 |
| 中国黑龙江Heilongjiang, China | 2.5534±0.8270 | 2.0086±0.6633 | 0.7203±0.3376 | 0.2727±0.3204 | 0.4590±0.2010 | 0.4435±0.1941 | 0.4898 |
| 亚洲(除中国)Asia | 2.5534±0.8826 | 1.9263±0.5939 | 0.6967±0.3271 | 0.3255±0.3371 | 0.4390±0.1937 | 0.4278±0.1889 | 0.4771 |
| 欧洲 Europe | 2.5392±0.8286 | 1.9860±0.6475 | 0.7054±0.3280 | 0.3075±0.3594 | 0.4534±0.2001 | 0.4391±0.1937 | 0.4385 |
| 南美洲 South America | 1.4947±0.5433 | 1.4510±0.5037 | 0.3246±0.3497 | 0.3263±0.4422 | 0.3123±0.3388 | 0.2289±0.2457 | 0.1383 |
| 北美洲 North America | 1.9903±0.6492 | 1.7557±0.5554 | 0.5551±0.3393 | 0.3134±0.3889 | 0.4241±0.2565 | 0.3657±0.2180 | 0.3562 |
| 大洋洲Oceania | 1.9706±0.6815 | 1.7293±0.5367 | 0.5416±0.3419 | 0.3348±0.3946 | 0.4073±0.2505 | 0.3576±0.2199 | 0.3426 |
| 非洲Africa | 1.6700±0.6039 | 1.5522±0.5157 | 0.4097±0.3516 | 0.2950±0.3927 | 0.3590±0.3123 | 0.2804±0.2375 | 0.2886 |
新窗口打开
Table 3
表3
表3国内外群体的遗传多样性分析
Table 3Estimates of genetic diversity of populations of broomcorn millet from domestic and abroad
| 群体 Population | 观测等位基因 Na | 有效等位基因 Ne | 多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | Nei's 期望杂合度 Nei | 多态性信息含量指数 PIC |
|---|---|---|---|---|---|---|---|
| 国内 Domestic | 2.9126±1.0582 | 2.2620±0.7862 | 0.8302±0.3595 | 0.2972±0.3030 | 0.5023±0.1909 | 0.4980±0.1891 | 0.5278 |
| 国外 Abroad | 2.7670±0.9922 | 2.0429±0.6601 | 0.7434±0.3417 | 0.3172±0.3405 | 0.4581±0.1967 | 0.4533±0.1946 | 0.4827 |
新窗口打开
Shannon-Weaver指数(I)范围为0.3246—0.7608,最大的为中国河北群体,最小的为南美洲群体。国内群体Shannon-Weaver指数(0.8302)大于国外群体(0.7434),其不同群体的变化趋势与Ne一致。从Nei’s基因杂合度分析,观测杂合度(Ho)最大的是中国辽宁群体(0.3443),最小的是中国黑龙江群体(0.2727),均值为0.3123。期望杂合度(He)最大的是中国河北群体(0.4789),最小的是南美洲群体(0.3123),均值为0.4143。国内外不同群体的观测杂合度和期望杂合度的变化趋势不一致,国内群体的Ho值(0.2972±0.3030)小于国外群体(0.3172±0.3405),He值(0.5023±0.1909)大于国外群体(0.4581±0.1967)。各参试群体的PIC值变化范围为0.1383(南美洲)—0.4898(中国黑龙江),国内群体的PIC值大于国外群体。
综合以上遗传参数可知,国内资源的遗传多样性高于国外资源。国内群体中河北、黑龙江的遗传多样性较高,国外群体中亚洲、欧洲的遗传多样性较高。
2.3 不同群体间遗传相似性分析
利用PopGen1.3.2软件分析12个群体的遗传相似性(表4),结果表明,遗传距离的变化范围为0.0783—0.5762,均值为0.2938。遗传一致度变化范围为0.5620—0.9247,均值为0.7500。欧洲群体和亚洲群体的遗传距离最小(0.0783),遗传一致度最大(0.9247),说明这两个群体的亲缘关系较近。而南美洲群体和中国山西群体的遗传距离最大(0.5762),遗传一致度最小(0.5620),两者的亲缘关系较远。由以上分析可知,遗传相似性与地理分布具有一定相关性,地理分布越近,遗传距离越小,遗传一致度越高,亲缘关系越近。Table 4
表4
表412个群体遗传距离和遗传一致度
Table 4Nei's unbiased measures of genetic identity and distance within 12 populations
| 群体 Population | 中国山西 Shanxi, China | 中国宁夏 Ningxia, China | 中国内蒙古 Inner Mongolia, China | 中国河北 Hebei, China | 中国 辽宁 Liaoning, China | 中国 黑龙江 Heilongjiang, China | 亚洲 (除中国) Asia (excluding China) | 欧洲 Europe | 南美洲 South America | 北美洲 North America | 大洋洲 Oceania | 非洲 Africa |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 中国山西 Shanxi, China | - | 0.6718 | 0.7028 | 0.7772 | 0.7062 | 0.7252 | 0.7248 | 0.6897 | 0.562 | 0.6762 | 0.6718 | 0.6388 |
| 中国宁夏 Ningxia, China | 0.3978 | - | 0.7939 | 0.8398 | 0.8082 | 0.7817 | 0.7816 | 0.7569 | 0.6318 | 0.7108 | 0.7181 | 0.6759 |
| 中国内蒙古 Inner Mongolia, China | 0.3527 | 0.2307 | - | 0.8281 | 0.8159 | 0.7627 | 0.7336 | 0.6914 | 0.5794 | 0.6709 | 0.6887 | 0.6141 |
| 中国河北 Hebei, China | 0.2521 | 0.1746 | 0.1887 | - | 0.8753 | 0.881 | 0.8434 | 0.8211 | 0.6856 | 0.7807 | 0.7643 | 0.7411 |
| 中国辽宁 Liaoning, China | 0.3479 | 0.213 | 0.2035 | 0.1332 | - | 0.8522 | 0.7758 | 0.7555 | 0.6038 | 0.7409 | 0.7003 | 0.6709 |
| 中国黑龙江 Heilongjiang, China | 0.3213 | 0.2463 | 0.2709 | 0.1267 | 0.1599 | - | 0.8158 | 0.7918 | 0.6818 | 0.7592 | 0.7337 | 0.6929 |
| 亚洲(除中国) Asia (excluding China) | 0.3218 | 0.2465 | 0.3098 | 0.1703 | 0.2538 | 0.2036 | - | 0.9247 | 0.7813 | 0.8855 | 0.8736 | 0.8172 |
| 欧洲Europe | 0.3715 | 0.2785 | 0.3691 | 0.1971 | 0.2803 | 0.2334 | 0.0783 | - | 0.7523 | 0.904 | 0.8932 | 0.8375 |
| 南美洲 South America | 0.5762 | 0.4592 | 0.5458 | 0.3774 | 0.5044 | 0.383 | 0.2468 | 0.2847 | - | 0.6678 | 0.6644 | 0.6646 |
| 北美洲 North America | 0.3912 | 0.3414 | 0.3992 | 0.2476 | 0.2999 | 0.2755 | 0.1216 | 0.1009 | 0.4037 | - | 0.8418 | 0.8251 |
| 大洋洲 Oceania | 0.3978 | 0.3311 | 0.3729 | 0.2688 | 0.3563 | 0.3096 | 0.1351 | 0.1129 | 0.4089 | 0.1722 | - | 0.7716 |
| 非洲Africa | 0.4482 | 0.3917 | 0.4875 | 0.2997 | 0.3991 | 0.3669 | 0.2018 | 0.1774 | 0.4086 | 0.1923 | 0.2592 | - |
新窗口打开
2.4 UPMGA聚类分析
基于UPMGA对12个群体进行聚类(图2),遗传距离0.15处,12个群体分为4个组群,分别为组群Ⅰ、组群Ⅱ、组群Ⅲ、组群Ⅳ。组群Ⅰ和组群Ⅱ为国外群体,组群Ⅲ和组群Ⅳ为国内群体。遗传距离为0.18处南美洲群体独立分为一支,与其余国内外群体遗传关系均较远,从地理上看,它与别的群体相距均较远,各种环境因素、气候条件的不同可能是造成该结果的主要原因。遗传距离为0.15处,中国山西群体独立分为一支,与其他国内群体相差较远。国内群体中,中国河北群体、黑龙江群体、辽宁群体遗传关系较近,值得注意的是,河北群体和黑龙江群体尽管地理分布相距较远,但两者的遗传距离最小,且聚在同一个亚群。国外群体中,亚洲群体、欧洲群体和北美洲群体遗传关系较近,大洋洲群体次之。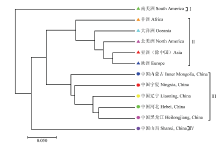 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2基于遗传距离的12个群体黍稷资源聚类图
-->Fig. 2Cluster diagram of 12 populations of broomcorn millet based on genetic distance
-->
将国外群体按不同的国家分为24个群体,然后与国内6个群体进行聚类(图3),结果显示,遗传距离为0.20处,国内群体与国外群体有明显的分离,与以上结果一致。但是,从图中可以看出,遗传距离为0.10处,国外群体被分为若干个小群,几乎没有分为大的聚类,总体而言,亚洲与欧洲各群体的遗传关系较近,与12个群体间聚类的结果吻合。
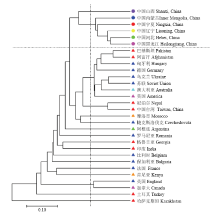 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3基于遗传距离的30个群体黍稷资源聚类图
-->Fig. 3Cluster diagram of 30 populations of broomcorn millet based on genetic distance
-->
基于UPGMA对146份个体进行聚类(图4),结果显示国内外资源划分非常显著。在遗传距离为0.63处,146份黍稷资源分为3大组群,组群Ⅰ和组群Ⅱ为国外资源,组群Ⅲ为国内资源。组群Ⅰ包括2份资源,均为亚洲资源。在遗传距离为0.39处,组群Ⅱ可分为3个亚群,分别为亚群A、亚群B和亚群C。亚群A包括29份资源,分别为亚洲6份、欧洲15份、大洋洲3份、北美洲3份和非洲2份。亚群B包括24份资源,分别为亚洲13份、欧洲5份、南美洲2份、大洋洲2份、非洲1份和北美洲1份。亚群C包括6份资源,分别为亚洲4份、欧洲1份和北美洲1份。在遗传距离为0.45处,组群Ⅲ可分为5个亚群,分别为亚群D、亚群E、亚群F、亚群G和亚群H。亚群D包括4份资源,中国山西1份和中国河北3份。亚群E包括20份资源,分别为中国辽宁2份和中国黑龙江18份。亚群F包括10份资源,分别为中国河北2份、中国辽宁2份和中国黑龙江5份。亚群G包括21份资源,分别为中国宁夏15份、中国内蒙古5份和中国河北1份。亚群H包括30份资源,分别为中国河北20份、中国山西2份、中国内蒙古6份和中国辽宁2份。从各组群和亚群分布可以看出,亚洲与欧洲资源、中国河北与中国山西、中国内蒙古资源的遗传关系较近,各组群分布与地理来源有一定关联。
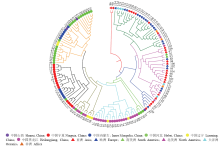 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4基于遗传距离的146份黍稷资源聚类图
-->Fig. 4Cluster diagram of 146 accessions of broomcorn millet based on genetic distance
-->
2.5 遗传结构分析
利用Structure2.3.4软件对146份黍稷资源进行群体结构分析(图5),并根据对数似然方差Var[Lnp](D)]对K值绘制折线图,最佳分组应为9组。国内外群体间存在明显的遗传分化(表5),组群2、组群5、组群6、组群7和组群9是国内野生资源特有基因型,各省份群体分布较为分散,其中中国河北、中国黑龙江、中国辽宁资源集中分布在组群5、组群7和组群9,中国宁夏、中国内蒙古资源集中分布在组群2和组群6,各组群分布与地理来源基本相符。组群1和组群4为国外材料特有基因型,各群体分布更为集中。组群3中除2份亚洲资源外,其余均为国内野生资源(80%)组群8中除1份中国山西资源外,其余均为国外资源(97%),中国山西野生糜子和亚洲其他国家地方品种存在不同程度的遗传混杂,遗传关系相对较近。此外,中国宁夏资源全部分布在组群2,南美洲资源全部聚集在组群8,表明这两个地区的黍稷资源的群体结构趋向单一化。相反,中国河北资源的群体结构最复杂,趋向多元化,分别被聚到6个组群中;其次是中国黑龙江和亚洲资源,分别被聚到4个组群中。这些结果很好地印证了UPGMA聚类结果,均于各群体的地理分布有一定的相关性。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5146份黍稷种质资源群体遗传结构图
-->Fig. 5Population genetic structure graph of 146 broomcorn millet
-->
Table 5
表5
表512个群体的遗传结构分析
Table 5Distribution of 12 populations based on STRUCTURE analysis (K = 9)
| 群体 Population | 组群1 Group1 | 组群2 Group2 | 组群3 Group3 | 组群4 Group4 | 组群5 Group5 | 组群6 Group6 | 组群7 Group7 | 组群8 Group8 | 组群9 Group9 | 总计 Total |
|---|---|---|---|---|---|---|---|---|---|---|
| 中国山西 Shanxi, China | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 3 |
| 中国宁夏 Ningxia, China | 0 | 15 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 15 |
| 中国内蒙古Inner Mongolia, China | 0 | 5 | 0 | 0 | 0 | 6 | 0 | 0 | 0 | 11 |
| 中国河北 Hebei, China | 0 | 1 | 1 | 0 | 6 | 1 | 9 | 0 | 8 | 26 |
| 中国辽宁 Liaoning, China | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 5 | 7 |
| 中国黑龙江 Heilonhjiang, China | 0 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | 20 | 23 |
| 亚洲(除中国) Asia (excluding China) | 4 | 0 | 2 | 2 | 0 | 0 | 0 | 17 | 0 | 25 |
| 欧洲 Europe | 5 | 0 | 0 | 9 | 0 | 0 | 0 | 7 | 0 | 21 |
| 南美洲 South America | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 2 |
| 北美洲North America | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 2 | 0 | 5 |
| 大洋洲Oceania | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 2 | 0 | 5 |
| 非洲Africa | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 2 | 0 | 3 |
| 总计Total | 11 | 21 | 5 | 16 | 6 | 9 | 12 | 33 | 33 | 146 |
新窗口打开
3 讨论
3.1 黍稷SSR标记的遗传多样性
SSR标记是分析遗传多样的有效标记,已被广泛应用于多种作物的遗传多样性研究,如玉米[11]、小麦[12]、燕麦[13]、水稻[26]等。近年来,不少报道中开发了黍稷特异性SSR标记,并分析了不同黍稷资源的遗传多样性。CHO等[14]开发了25对多态性SSR标记,共检测到110个等位基因,平均每个位点4.4个。连帅等[25]采用63对SSR标记对192份黍稷地方品种和野生品种进行遗传多样性分析,共扩增出161个等位基因,平均每个位点2.56个,PIC值为0.0877—0.8020,平均0.4855。HOU等[27]采用14个多态性SSR标记检测到43个等位基因,平均每个位点3个。王瑞云等[24]利用15个糜子特异性荧光SSR标记分析了132份国内黍稷资源的遗传多样性,检测到107个等位变异,平均每个位点变异数为7个,PIC值范围为0.0893—0.8538,平均0.4864。本研究中利用103对SSR标记对146份黍稷资源进行遗传多样性分析,扩增出308个等位基因,平均每个位点2.99个,PIC值为0.064—0.8035,平均0.5544,Rp值为0.334—4.002,77.67%的标记具有适度分辨力。等位基因数低于王瑞云等[24]的7个,CHO等[14]的4.4个,高于连帅等[25]的2.56个,与HOU等[27]的3个相当,这可能与引物筛选标准不同有关。平均PIC值大于0.5,高于连帅等[25]和王瑞云等[24]的报道,表明所选引物为高度多态性信息引物,可用于黍稷遗传多样性和起源进化的进一步研究。3.2 参试材料的遗传多样性以及与地理分布的关系
本研究中MEGA结果显示,个体间聚类和群体间聚类均将国内外群体分为两大组群,国内群体中,河北、黑龙江、辽宁资源亲缘关系较近,各遗传参数也表明这些地区黍稷资源的遗传多样性丰富。国外群体中亚洲、欧洲遗传距离最小,遗传一致度最大,亲缘关系最近。中国山西群体与南美洲群体的遗传距离最大,两者均独立分为一支,与其他材料遗传差异较大,且两者地理分布相距远,环境气候有很大差异,表明聚类分析与地理分布相关。STRUCTURE分析结果显示国内外群体间存在明显的基因型分型,国内外群体间地理位置相距较远,地理距离的隔离对各群体的基因流具有一定阻碍。此外还发现中国山西资源与国外亚洲资源存在部分遗传混杂,表明两者亲缘关系较近,且中国山西与亚洲其他国家地理相距较近,暗示中国山西与亚洲其他国家的黍稷资源间可能存在基因流。黍稷群体间可能存在遗传距离与地理距离的相关性,该研究结果与前人[10,21,23-24]报道相似。本研究聚类分析和遗传结构分析结果均表明国外群体遗传分化不明显,没有大的聚类,个体间存在相互渗透,这与连帅等[25]研究结果相似,这可能与国外黍稷资源遗传背景比较狭窄有关,因此,国外黍稷在育种上需加强种质资源的引进与创新。3.3 黍稷起源进化研究现状
黍稷是干旱半干旱地区的主要作物之一,具有重要的经济价值。研究黍稷野生资源是开展黍稷起源进化研究的重要根据之一。自1753年,提出黍稷原生于印度起[29],国内外****对黍稷的分类和进化进行了一系列的研究。目前,中国是世界上黍稷考古发现最早、资料最丰富的国家,以黄河中下游为中心,西至新疆,东至黑龙江,多处发现黍稷的遗迹,是公认的黍稷起源中心[28,29,30]。高俊山等[31]对中国620份栽培黍稷品种酯酶同工酶谱类型地理分布的研究表明中国的栽培黍稷起源于黄土高原。HU等[21]和王星玉等[32]也得出了相同的结论。中国黍稷专家魏仰浩将野黍定位为黍稷的亚种[29,30]。通过从酯酶同工酶谱和生态性状分析栽培黍稷与野生黍稷的关系,表明栽培黍稷是以具有各种原始性状的野生黍稷及其近缘种进化而来[29,30,31]。本研究利用SSR标记,从DNA分子水平分析了黍稷野生资源的遗传多样性,结果显示黍稷野生资源中中国河北群体的遗传多样性最丰富,其群体结构趋向多元化,与黑龙江、辽宁群体遗传关系较近,与内蒙古、山西资源个体间存在相互渗透。与栽培黍稷相比较,野生黍稷的遗传多样性更为丰富,在很大程度上保持着原先的遗传状态。一方面可能是因为来自于河北的参试材料较多;另一方面,河北一带野生糜子分布广泛,目前栽培黍稷种植区域也较大,因此,推测河北也有可能是栽培黍稷的初级或次级起源中心,关于黍稷起源中心尚需丰富野生资源进一步研究确定。4 结论
国内材料野生资源的遗传多样性高于国外材料,其中河北、黑龙江的遗传多样性最丰富,河北可能是黍稷的起源中心。聚类结果与遗传结构分析结果相似,均与地理分布有一定的相关性。(责任编辑 李莉)
The authors have declared that no competing interests exist.
