基因组编辑技术是指可以在基因组水平上对DNA序列进行定点改造的遗传操作技术,其在基因功能研究和改造、生物医学和植物遗传改良等方面都具有重大的应用价值[1-2]。科学家自20世纪90年代末就开始探索基因组定点编辑技术,但直到2002年,也仅在小鼠[3]和果蝇[4]等少数模式生物中实现了同源重组介导的基因组定点编辑,且因同源重组的效率很低,限制了其应用前景。进入21世纪后,随着蛋白质结构与功能研究的新突破和人工核酸内切酶(engineered endonuclease,EEN)技术的出现,将特异识别并结合DNA的蛋白结构域和EEN融合,创造出能够特异切割DNA序列的核酸酶(sequence-specific nucleases,SSNs),从而可以对基因组特定位点进行高效和精确的靶向编辑[5]。
目前,SSNs主要包括锌指核酸酶(Zinc finger nucleases,ZFNs)[6]、类转录激活因子效应物核酸酶(transcription activator-like effector nucleases,TALENs)[7]、成簇的规律间隔的短回文重复序列及其相关系统(clustered regularly interspaced short palindromic repeats/CRISPR-associated Cas9,CRISPR/Cas9 system)[8]和CRISPR/Cpf1系统[9]。这些SSNs的共同特点是都能在基因组特定部位精确切割DNA双链,造成DNA双链断裂(DNA double-strand breaks,DSBs);而DSBs能够极大地提高染色体重组事件发生的概率[10]。DSBs的修复机制在真核生物细胞中高度保守,主要包括同源重组(homology- directed repair,HDR)和非同源末端连接(non-homologous end joining,NHEJ)[11]2种修复途径(图1)。当存在同源序列供体DNA时,以HDR方式的修复能够产生精确的定点替换或插入;而没有供体DNA时,细胞则通过NHEJ途径修复[11]。因NHEJ方式的修复往往不够精确,在DNA链断裂位置常会产生少量核酸碱基的插入或缺失(insertion-deletion,InDel),从而导致基因突变[11]。
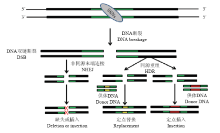 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1SSNs介导的DSBs及其修复途径
-->Fig. 1SSNs-induced DSBs and repair pathways
-->
SSNs自2002年出现以后[12],就掀起了一股席卷全球的基因组定点编辑研究热潮[13]。尤其是2010年出现TALENs和2013年出现CRISPR/Cas9技术后,因它们相对简单、精确、高效,很快被广泛应用于医学、农业、基础研究等领域[2,14]。因TALENs和CRISPR/Cas9潜在的巨大应用价值,它们相继被《科学》杂志评为2012年和2013年的十大科学突破之一。本文将重点综述基因组编辑技术在作物遗传改良上的应用进展、提出基因组编辑技术应用于作物改良的一般原则,最后讨论该技术应用于作物育种的机遇和挑战。
1 基因组编辑技术在作物遗传改良上的应用进展
目前,在作物遗传改良上应用的基因组编辑技术主要包括ZFNs、TALENs和CRISPR/Cas系统3种类型。其中CRISPR/Cas系统包括CRISPR/Cas9、CRISPR/Cpf1、CRISPR/C2c1[15]和CRISPR/C2c2[16]等亚类型,但应用最多的是CRISPR/Cas9,而CRISPR/ C2c1和CRISPR/C2c2尚无在作物改良上应用的报道。上述3类基因组编辑技术均能对植物基因组进行精准的定点敲除、插入和替换,因此,其对于控制作物重要农艺性状基因的功能鉴定、作物重要性状的遗传改良具有巨大的应用潜力。相比传统的常规育种,基因组编辑技术能直接对目标性状基因进行修饰,有可能大幅度提高目标性状聚合的精准度和加快聚合育种进程[17]。而相比传统的转基因育种,基因组编辑在靶向修饰特定基因后,能通过自交或杂交剔除外源基因以消除转基因安全顾虑。因此,自基因组编辑技术成功应用于植物后,对该技术的优化及在作物遗传改良上的应用已成为世界各国和国际农业生物技术公司投资与研发的重点。目前,基因组编辑技术主要集中应用于作物产量、品质、抗性、育性等4个方面的遗传改良(表1)。现就技术类型和目标性状分述如下。
1.1 ZFNs技术及其应用
ZFNs技术被称为第一代基因组编辑技术。ZFN是一种人工改造而成的核酸内切酶,由锌指蛋白的DNA结合域和核酸内切酶FokⅠ的切割结构域组成[49]。其中,DNA结合域通常由3—6个锌指结构域(Zinc finger domain,ZFD)串联而成[50]。一个ZFD能特异识别DNA链上3个连续的核苷酸碱基,多个ZFD串联后就能特异识别较长的核苷酸序列。FokⅠ是一种非特异性核酸内切酶,在二聚体状态时才有内切酶活性[50]。因此,实际应用ZFNs打靶时,需要在靶点的两侧各设计一个ZFN。待2个ZFN结合到结合位点后,2个FokⅠ相互作用形成二聚体,从而在靶点处切割DNA,产生DSBs[6]。2002年,ZFNs首次成功应用于果蝇内源基因的定向突变[12]。迄今为止,ZFNs技术已成功应用于人类干细胞[51]、大鼠[52]、果蝇[53]、斑马鱼[54]、拟南芥[55]、烟草[56]和玉米[18]等生物体的基因组编辑。在作物改良方面,ZFNs技术迄今仅在玉米性状改良上有一例报道,即2009年《Science》报道SHUKLA等[18]利用ZFNs技术靶向突变玉米IPK1,获得低植酸含量的玉米突变体。植酸盐是谷物饲料的抗营养组分,低植酸盐含量的谷物饲料能降低来自动物饲养作业所产生废物流中的磷酸盐污染。由于ZFNs的构建难度较大,普通分子实验室难以操作,且成本高,预测其很难广泛应用于植物基因组编辑。
1.2 TALENs技术及其应用
TALENs是继ZFNs之后的第二代基因组编辑技术。TALEN结构与ZFN类似,也由DNA结合域和FokⅠ的切割结构域融合而成[57]。TALEN的DNA结合域为天然的或经改造的TAL效应子(TAL effector,TALE)蛋白结构域。天然的TALEs都具有高度统一的结构:即高度保守的N端和C端、中间部分的重复区。不同TALEs之间的差异主要在中间的重复区,其由33—35个氨基酸的重复单元组成,各重复单元除了第12和第13位氨基酸高度可变外,其他都高度保守,而这两个高度可变的氨基酸组合被称为重复可变区(repeat-variable diresidue,RVD)[58,60]。2009年,BOCH等[59]和MOSCOU等[60]相继破解RVDs与核苷酸碱基之间的识别密码(即NI或NS—A,NG—T,NN或NK—G和HD—C),由此打开了人工构建靶向基因组任意位点TALENs的大门。TALEN的工作原理也与ZFN相似,TALE重复区的每个重复单元能特异识别一个特定的核甘酸碱基,多个重复单元串联后就能特异识别较长的核苷酸序列。实际利用TALENs打靶时,同样需要在靶点两侧设计一对TALENs。与ZFN相比,TALEN载体的构建相对简单、成本也更低、普通的分子生物实验室都能操作;此外,TALEN技术基于重复单元与核苷酸碱基一对一的识别模式,特异性更强,靶向编辑的效率更高[61]。因此,自2010年TALENs技术出现后[7, 57],便迅速取代ZFNs成为新一代的基因组编辑技术。Table 1
表1
表1基因编辑技术在作物遗传改良中的应用
Table 1Application of genome editing in crop genetic improvement
| SSN类型 SSN type | 物种 Species | 基因 Gene | 修饰方式 Modification type | 突变体表型 Mutant phenotype | 参考文献 References | |
|---|---|---|---|---|---|---|
| ZFNs | 玉米 Zea mays | IPK1 | 敲除Knockout | 低植酸含量Low phytate | [18] | |
| TALENs | 水稻 Rice | Os11N3 | 敲除Knockout | 抗白叶枯病Bacterial blight resistant | [19] | |
| 水稻Rice | OsBADH2 | 敲除Knockout | 具有香味Fragrance | [20] | ||
| 水稻Rice | Lox3 | 敲除Knockout | 耐贮藏性Storage tolerance | [21] | ||
| 水稻Rice | SWEET14 | 敲除Knockout | 抗白叶枯病Bacterial blight resistant | [22] | ||
| 小麦Wheat | MLO | 敲除Knockout | 抗白粉病Powdery mildew resistant | [23] | ||
| 大豆Soybean | FAD2-1A, FAD2-1B | 敲除Knockout | 高油酸含量 High oleic acid | [24] | ||
| 马铃薯Potato | VInv | 敲除Knockout | 耐冷藏性Cold storage tolerance | [25] | ||
| CRISPR/Cas9 | 水稻Rice | OsERF922 | 敲除Knockout | 稻瘟病抗性增强Rice blast resistance improvement | [26] | |
| 水稻Rice | IPA1 | 敲除Knockout | 分蘖和穗粒数改变 Tillers and grain number per panicle change | [27] | ||
| 水稻Rice | DEP1 | 敲除Knockout | 直立穗密度增加Dense erect panicles increasement | [27-28] | ||
| 水稻Rice | Gn1a | 敲除Knockout | 主穗粒数增加Spike grain number increasement | [27, 29] | ||
| 水稻Rice | GS3 | 敲除Knockout | 谷粒变长 Long grain | [27, 29] | ||
| 水稻Rice | Gn1a | 敲除Knockout | 穗粒数增加 Grain number per panicle increasement | [29] | ||
| 水稻Rice | GW2, GW5, TGW6 | 敲除Knockout | 粒重增加 Grain weight enhancement | [30] | ||
| 水稻Rice | csa | 敲除Knockout | 光敏核雄性不育Photo-sensitive genic male sterile | [31] | ||
| 水稻Rice | TMS5 | 敲除Knockout | 温敏核雄性不育Thermo-sensitive genic male sterile | [32] | ||
| 水稻Rice | OsWaxy | 敲除Knockout | 低直链淀粉含量Low amylose | [33] | ||
| 水稻Rice | BEIIb | 敲除Knockout | 高直链淀粉含量High amylose | [34] | ||
| 水稻Rice | ALS | 定点替换Replacement | 抗除草剂Herbicide resistant | [35-36] | ||
| 水稻Rice | OsEPSPS | 定点替换Replacement | 抗除草剂Herbicide resistant | [37] | ||
| 水稻Rice | ALS | 定点替换Replacement | 抗除草剂Herbicide resistant | [38] | ||
| 玉米maize | ARGOS8 | 定点插入Insertion | 抗旱 Drought resisting | [39] | ||
| 玉米maize | ALS | 定点替换Replacement | 抗除草剂Herbicide resistant | [40-41] | ||
| 玉米maize | Ms26, Ms45 | 敲除Knockout | 雄性不育Male sterile | [41] | ||
| 玉米maize | LIG | 敲除Knockout | 无叶舌Liguleless | [41] | ||
| 小麦Wheat | TaGASR7 | 敲除Knockout | 粒重增加Grain weight enhancement | [42] | ||
| 小麦Wheat | TaDEP1 | 敲除Knockout | 植株变矮Dwarf plant | [42] | ||
| 大豆Soybean | GmFT2a | 敲除Knockout | 花期推迟Flowering time delay | [43] | ||
| 番茄Tomato | SP5G | 敲除Knockout | 花期提前、产量增加 Flowering time in advance and gain in yield | [44] | ||
| 柑桔Citrus | CsLOB1 | 敲除Knockout | 溃疡病的抗性增强Canker resistant improvement | [45] | ||
| 双孢菇 Mushroom | PPO | 敲除Knockout | 抗褐变Anti-browning | [46] | ||
| CRISPR/Cpf1 | 水稻Rice | OsPDS | 敲除Knockout | 植株白化Albino plant | [47] | |
| 水稻Rice | OsBEL | 敲除Knockout | 除草剂敏感Herbicide susceptible | [47] | ||
| 水稻Rice | OsCAO1 | 敲除Knockout | 植株黄化Etiolated plant | [48] | ||
新窗口打开
TALENs技术应用于作物遗传改良最早见于水稻。2012年,LI等[19]首次利用TALENs技术对水稻白叶枯病感病基因Os11N3(SWEET14)启动子中的效应蛋白结合元件(effector-binding element,EBE)进行定点编辑,有效阻止了水稻白叶枯菌(Xanthomonas oryzae pv. oryzae,Xoo)分泌的效应蛋白(AvrXa7和PthXo3)与Os11N3启动子结合,使该基因不受Xoo的诱导表达,从而提高了水稻的白叶枯病抗性。随后,TALENs技术相继在小麦[23]、大豆[24]和马铃薯[25]等主要作物的重要性状改良上获得成功。
MLO(MILDEW-RESISTANCE LOCUS)是大麦、小麦等作物中白粉病菌成功侵染所必需的基因,该基因突变后,大麦表现出对白粉病菌的持久、广谱抗性[62]。2014年,中国科学院遗传与发育生物学研究所高彩霞团队利用TALENs技术同时靶向突变小麦MLO的3个拷贝,获得了3个拷贝都突变的纯合突变体,该突变体小麦对白粉病菌表现极为显著的抗性[23],证明TALENs技术能对小麦这样的多倍体植物进行定向遗传改良。同年,美国Cellectis公司研究人员通过TALENs技术靶向突变大豆脂肪酸脱氢酶基因FAD2-1A和FAD2-1B,获得的双基因突变体中油酸含量由20%提高到80%、亚油酸含量由50%降至4%,极大地改善了大豆油的品质[24];随后,该公司又通过TALENs技术靶向修饰马铃薯的VInv,改良了马铃薯的耐冷藏和加工性[25]。此外,SHAN等[20]利用TALENs技术敲除水稻品种日本睛的OsBADH2,使无香味的稻米产生了香味;MA等[21]利用该技术敲除水稻脂肪氧化酶基因Lox3,改良了水稻种子的耐贮藏性;BLANVILLAIN-BAUFUME等[22]利用该技术对水稻白叶枯病感病基因SWEET14启动子中效应蛋白AvrXa7和Tal5、TalC的EBEs进行定点编辑,使感病水稻品种的白叶枯病抗性提高到中抗以上水平。
1.3 CRISPR/Cas9技术及其应用
CRISPR/Cas是细菌和古细菌抵御病毒和外源DNA入侵的适应性免疫系统[63]。目前,CRISPR/Cas系统可分为两大类。第一大类由多个Cas蛋白组成的复合体行使生物学功能,第二大类由单个Cas蛋白(如Cas9、Cpf1、C2c1和C2c2)行使生物学功能[64]。CRISPR/Cas9属于Ⅱ类,仅需要成熟的crRNA(CRISPR-derived RNA)、tracrRNA(trans-activating RNA)和Cas9蛋白就能实现对外源DNA的切割[65-66]。经人工改造的CRISPR/Cas9系统主要由引导RNA(single guide RNA,sgRNA)和Cas9蛋白结构域组成。其中,sgRNA起精确定位作用,其5′端前20个核苷酸碱基决定Cas9蛋白特异性切割基因组DNA的部位;Cas9蛋白包含类HNH核酸酶结构域和类RuvC核酸酶结构域,分别负责切割靶DNA互补链和非互补链DNA[66-67]。CRISPR/Cas9系统在工作时,sgRNA和Cas9形成一个嵌合蛋白,该嵌合蛋白切割与sgRNA 5′端前20-nt互补的基因组DNA双链,产生DSBs,最后通过HDR或NHEJ修复途径产生基因突变。2013年,CPISPR/Cas9系统成功实现人类细胞的基因组定向编辑[8, 68],随后被迅速应用于人类、动物和植物等的基因组编辑[69-70],并超越TALENs成为第三代基因组编辑技术。CRISPR/Cas9系统具有构建简单、编辑效率高、容易实现多基因编辑等优势,现已成为应用最广泛的基因组编辑技术,在作物遗传改良和品种培育上具有重大应用潜力[71]。目前,CRISPR/Cas9技术已成功应用于多种作物如水稻[26-38]、玉米[39-41]、小麦[42]、大豆[43]、番茄[44]、柑桔[45]和蘑菇[46]的重要农艺性状遗传改良。
1.3.1 在作物产量和品质改良上的应用 LI等[27]以水稻品种中花11为材料,利用CRISPR/Cas9技术对水稻产量负调控基因Gn1a(每穗实粒数)、DEP1(直立型密穗)、GS3(粒长和粒重)和IPA1(穗粒数、分蘖相关)进行定点修饰,发现Gn1a或DEP1、GS3突变后水稻的每穗实粒数或着粒密度、粒长都明显增加,但DEP1突变体在着粒密度增加的同时有半矮化现象,而GS3突变体在粒长增加的同时芒也显著增长。类似地,SHEN等[29]利用CRISPR/Cas9技术对5个粳稻品种的GS3和Gn1a进行定向敲除,构建了5个粳稻背景的gs3和gs3gn1a突变系,所有突变系稻谷粒长相比其野生型都增加,所有gs3gn1a突变系的主穗粒数相比其野生型也都增加,单株产量增幅最高达14%。此外,XU等[30]通过该技术靶向突变水稻GW2(粒宽和粒重)、GW5 (粒宽和粒重)和TGW6(千粒重)基因后发现,gw2gw5tgw6和gw2gw5纯合突变系的粒长、粒宽和粒重相比其野生型都显著增加。WANG等[28]利用CRISPR/Cas9技术对籼稻DEP1进行定点突变发现,获得的大片段缺失突变体水稻着粒密度增加、植株变矮,产量增加。
直链淀粉含量与水稻品质密切相关。MA等[33]利用CRISPR/Cas9技术靶向突变直链淀粉合成酶基因OsWaxy,突变体直链淀粉含量从14.6%下降至2.6%,由此获得了糯性品质。SUN等[34]利用该技术对水稻糖苷水解酶基因BE1和淀粉分支酶Ⅱb基因BEIIb进行定点编辑,BE1突变株相比野生型在粒型、总淀粉含量、直链淀粉含量等指标上未有显著差异,但BEIIb突变株相比野生型粒型显著变小、总淀粉含量和直链淀粉含量显著增加,后者最高达到25%。
在小麦产量改良上,ZHANG等[42]利用瞬时表达CRISPR/Cas9系统对小麦粒长和粒重负调控基因TaGASR7和直立型密穗基因TaDEP1定点编辑,获得的TaGASR7纯合突变株粒重显著增加,TaDEP1纯合突变株明显变矮。
在番茄生育期和产量改良上,SOYK等[44]通过CRISPR/Cas9技术对一种感光野生番茄的抗成花基因SP5G进行定向修饰。结果显示SP5G突变后,番茄开花和成熟时间提前了约2周,且挂果量增加、产量显著提升。
双孢菇(Agaricus bisporus)非常容易褐变,从而影响其品质。宾夕法尼亚州立大学伯克分校的杨亦农实验室利用CRISPR/Cas9技术对双孢菇的1个多酚氧化酶基因PPO进行定向修饰,获得的DNA-free突变双孢菇中多酚氧化酶的活性降低了30%,并具有了抗褐变能力。而抗褐变蘑菇于2016年4月成为第一个豁免美国农业部监管的CRISPR编辑作物[46]。
综上,利用CRISPR/Cas9技术对控制作物产量、品质相关的负调控基因进行定向修饰,能不同程度地改良作物的产量、品质和株型等,是作物高产优质育种的新途径。
1.3.2 在作物抗病、抗逆性改良上的应用 稻瘟病是危害水稻最严重的病害之一。水稻OsERF922是一个ERF(ethylene responsive factors)类转录因子基因,负调控水稻对病瘟病的抗性[72]。中国农业科学院作物科学研究所赵开军团队以稻瘟病感病品种空育131为材料,利用CRISPR/Cas9技术靶向敲除OsERF922,获得的T2纯合突变系在苗期和分蘖期对稻瘟病菌的抗性相比野生型都有显著提高[26]。柑桔溃疡病是柑桔生产上最严重的病害之一,CsLOB1是导致柑桔溃疡病的关键感病基因[73]。JIA等[45]利用CRISPR/Cas9技术对CsLOB1的编码区进行定点编辑,获得的突变体植株对溃疡病的抗性显著增强。
随着作物轻简化栽培技术的普及,除草剂在作物生产上的使用日益广泛,因此培育抗除草剂的作物新品种成为重要的育种目标。乙酰乳酸合酶(acetolactate synthase,ALS)是支链蛋白质合成通路中的第一个常见酶,ALS中特定氨基酸突变可以提高植物对乙酰乳酸合酶类除草剂的抗性[74]。2015年,美国杜邦先锋公司通过CRISPR/Cas9技术将玉米ALS2编码区的第165位脯氨酸突变为丝氨酸,获得了抗氯磺隆的玉米突变体[40-41]。通过类似的策略,中国农业科学院作物科学研究所夏兰琴团队和美国加州大学圣地亚哥分校赵云德团队合作,将ALS编码区特定碱基定点替换,导致2个氨基酸(W548L和S627I)变异,获得了抗磺酰脲类除草剂的水稻[35]。同时,ENDO等[36]也通过类似的方法,获得了ALS编码区W548L和S627I位置处碱基定点替换的水稻突变体。此外,中国科学院遗传与发育生物学研究所的高彩霞团队和李家洋团队合作,利用NHEJ修复方式建立了基于CRISPR/Cas9的基因组定点插入及替换系统,并利用该系统获得了在OsEPSPS基因保守区2个氨基酸定点替换(T102I和P106S)的杂合突变体,其对草甘膦具有抗性[37]。最近,SHIMATANI等[38]等通过基于CRISPR/ Cas9的Target-AID(target-activation-induced cytidine deaminase)方法,将水稻ALS编码区的第96位丙氨基酸突变成缬氨酸,获得了抗磺酰脲类除草剂的水稻突变体。
玉米ARGOS8是一个乙烯响应的负调控因子,在干旱胁迫过表达ARGOS8的玉米比野生型显著增产[75]。SHI等[39]利用CRISPR/Cas9技术,将玉米GOS2启动子(能赋予中等水平的组成型表达)定点插入ARGOS8的5′-非翻译区或直接替换ARGOS8的启动子,获得ARGOS8表达量显著增加的突变体。这些突变体在干旱环境下,其最终产量相比野生型玉米显著提升。这是目前首次报道利用CRISPR/Cas9技术通过调节靶标基因的表达量来改良作物遗传性状的案例。
总之,通过CRISPR/Cas9技术定点敲除作物抗性负调控因子基因,或定点修饰抗逆性正调控基因的启动子以增强基因的表达,或对抗性相关基因编码区定点替换改变基因功能,都能在不同程度上改良作物的抗病或抗逆性,是作物抗性分子育种的有效途径。
1.3.3 在创制水稻雄不育系上的应用 光温敏核雄性不育系(P/TGMS)的发现使杂交水稻由三系法向两系法发展。使用传统的方法,培育一个新的光温敏核雄性不育系通常需要几年甚至十年以上时间,但利用CRISPR/Cas9技术可以大大加快培育水稻雄不育系的进程。2016年,上海交通大学生命科学技术学院张大兵团队利用CRISPR/Cas9技术靶向编辑粳稻品种空育131内源基因csa(Carbon Starved Anther),获得了粳型光敏核雄性不育系[31]。同年11月,华南农业大学生命科学学院的庄楚雄团队利用CRISPR/Cas9技术对水稻温敏核雄性不育基因TMS5进行特异性编辑,创制了一批温敏核雄性不育系[32]。可见,CRISPR/Cas9技术为水稻两系不育系的培育提供了一条新的便捷途径。
1.4 CRISPR/Cpf1技术及其应用
CRISPR/Cpf1隶属于Ⅱ类type V-A CRISPR系统[9],是迄今发现最简单、有效的Ⅱ类CRISPR/Cas系统。CRISPR/Cpf1特异切割DNA双链的原理与CRISPR/Cas9相似,但其作用机制不同[76]。Cpf1是Cas蛋白的一种,其功能与Cas9类似,目前AsCpf1(Acidaminococcus sp. Cpf1)、LbCpf1(Lachnospiraceae bacterium Cpf1)和FnCpf1(Francisella novicida Cpf1)已被证实具有DNA切割及执行RNA加工的活性[77-79]。Cpf1蛋白由具有识别sgRNA功能的a-REC区域和具有核酸酶功能的NUC区域构成。a-REC包含REC1和REC2 2个识别结构域;NUC主要包含RuvC结构域、WED(wedge)结构域、一种推定的核酸酶(Nuc)结构域和PI(PAM-interacting)结构域,其中,Ruv-C和Nuc核酸酶分别负责切割靶DNA的非互补链及互补链的不同位点,由此产生具粘性末端的DSBs[79]。不同于SpCas9(Streptococcus pyogenes Cas9),Cpf1仅需一个42-nt的crRNA(3′端有23-nt与靶DNA序列互补),就能对靶DNA双链进行切割[76]。另AsCpf1和LbCpf1的PAM识别区碱基为5′-TTTN-3′,FnCpf1的PAM识别区碱基为5′-TTN-3′,不同于SpCas9的5′-NGG-3′ [80]。此外,Cpf1与SpCas9切割机制也不一样,SpCas9切割靶DNA互补链的4位和非互补链的16位,形成一个平末端;而Cpf1切割靶DNA互补链的23位和非互补链的18位,形成一个5-nt的粘性末端[76]。但最新的研究结果表明,Cpf1并非只切割靶DNA非互补链的18位,而是在非互补链的14—18位间多个位点切割。当spacer序列长度≥20-nt时,Cpf1倾向于切割互补链的18位;当spacer序列长度<20-nt时,Cpf1倾向于切割互补链的14位[81]。此外,研究人员还证明Cpf1核酸酶在人类细胞中与SpCas9具有相当的切割效率,也证实Cpf1核酸酶在人类细胞中具有高度的特异性[82-83]。目前,CRISPR/Cpf1技术已成功应用于烟草[77]、大豆[84]和水稻[47-48, 77, 85-86]的基因组编辑。2016年,ENDO等[77]首次将CRISPR/Cpf1系统成功移植于烟草和水稻的基因组编辑,他们将FnCpf1和crRNA融合开发了Cpf1-crRNA系统,在烟草和水稻中的编辑效率最高可达90%。随后,XU等[47]将LpCpf1分别与pre-crRNA和加长pre-crRNA融合构建了crRNA- Cpf1系统,在水稻中的编辑效率最高可达41.2%;HU等[86]也将LpCpf1和crRNA整合开发了CRISPR-Cpf1系统,并利用该系统成功实现水稻内源基因的定点编辑;KIM等[84]开发了Cpf1-RNP(ribonucleoprotein)系统,并成功利用该系统对大豆和烟草的内源基因进行定点突变。此外,BEGEMANN等[48]将Cpf1、crRNA和供体片段构成融合表达载体,对水稻叶绿素a加氧酶基因OsCAO1进行定点编辑,获得了在靶点产生的预期InDel突变和片段插入,此外纯合突变植株整株都产生黄化现象。TANG等[85]针对Cpf1蛋白及crRNA的表达特性,构建了PolⅡ型启动子融合核酶(ribozyme)驱动的 Cpf1和crRNA植物表达单元,并成功对水稻内源基因OsPDS、OsDEP1和OsROC5进行定点突变。WANG等[87]将4个DR (direct repeats)-guide单元组成的crRNA分别与FnCpf1和LbCpf1融合构建CRISPR/Cpf1系统,并利用该系统简单、高效地实现了水稻多基因定点编辑。
CRISPR/Cpf1与CRISPR/Cas9一样,都是目前最为简捷、高效和最易实现多基因编辑的基因组编辑技术。两者表达载体的构建都相当简单,且都只需要串联多个crRNA即可实现多位点编辑,而编辑效率和特异性也都相当,另因都受PAM识别位点的限制,编辑范围也都有一定的局限性。因此,CRISPR/Cpf1与CRISPR/Cas9可以说是不相伯仲的基因组编辑技术,相当于CRISPR/Cas技术“厨房”两把效果一样但又能互补的“剪刀”,作为“厨师”的我们,只要能达到我们的目标,选择任一“剪刀”都可。
1.5 利用CRISPR/Cas系统在基因组编辑方式上的技术创新
1.5.1 植物基因组单碱基编辑系统创新和应用 单碱基突变可引起作物许多农艺性状的改变,因此,实施单碱基编辑对作物遗传改良具有十分重要的意义。2016年5月,《Nature》报道了KOMOR等[88]将大鼠胞嘧啶脱氨酶(APOBEC1,一种碱基修饰酶能改变DNA序列)、Cas9变体(D10A)和尿嘧啶糖基化酶抑制剂(UGI)融合构建了大鼠基因组单碱基编辑系统,并用该系统成功纠正了小鼠细胞中阿尔茨海默病相关的突变,单碱基替换效率高达75%。随后,有4个研究团队将该系统成功改造并用于粮食和蔬菜作物(水稻、小麦、玉米、番茄)的基因组单碱基编辑[38, 89-91]。2016年12月,中国科学院上海生命科学研究院朱健康团队[89],以及夏兰琴和赵云德合作团队[90]同时在《Molecular Plant》上发表了其在植物基因组单碱基编辑上取得的最新成果。朱健康团队将APOBEC1通过非结构化的16残基肽XTEN作为接头,融合到Cas9(D10A)的N末端,并将核定位信号(NLS)肽添加到Cas9(D10A)的C末端,构建了APOBEC1-XTEN-Cas9(D10A)植物基因组单碱基编辑系统[89]。利用该系统对水稻NRT1.1B和SLR1进行编辑,结果表明该系统能在靶位点产生预期的C→T(G→A)碱基替换,NRT1.1B和SLR1在靶位点产生预期突变的效率分别为2.7%和13.3%。夏兰琴和赵云德合作团队也利用APOBEC1、Cas9变体(nCas9-D10A)和UGI的融合基因BE3[88]构建了植物基因组单碱基编辑系统pCXUN-BE3[90]。利用pCXUN-BE3系统编辑水稻八氢番茄红素脱氢酶基因OsPDS和淀粉分支酶Ⅱb基因OsSBEIIb,获得在3个靶位点产生预期突变(靶序列5′端4—8位置有G→A或C→T)的水稻植株,效率最高可达20%[90]。
2017年2月,《Nature Biotechnology》杂志报道了高彩霞实验室在水稻、小麦和玉米基因组中实现高效、精确的单碱基编辑的成果。他们同样利用APOBEC1、nCas9-D10A和UGI,构建了植物基因组单碱基编辑系统nCas9-PBE。利用该系统对3种作物5个内源基因7个靶位点的定点突变结果显示,在靶序列5′端3—9位置能产生预期的C→T的替换,其中,单个C的替换效率为0.39%—7.07%,多个C的替换效率高达12.48%;而遗传转化的结果表明,该系统在小麦、水稻和玉米中获得靶区域单碱基替换的效率最高可达43.48%[91]。同年3月,SHIMATANI等[38]在《Nature Biotechnology》杂志发表了其在水稻和番茄中实现高效的单碱基编辑的最新成果。将nCas9-D10A(nCas9)、PmCDA1(Petromyzon marinus cytidine deaminase)和sgRNA融合构建了植物基因组单碱基编辑系统nCas9-PmCDA1,并利用该系统对水稻ALS和FTIP1e以及番茄DELLA和ETR1进行定点突变。结果表明,在水稻中该系统在靶区域诱导单个或多个预期单碱基替换(C→T)的效率最高可达50%,在番茄中最高可达80%。
1.5.2 DNA-free植物基因组编辑系统创新和应用 生物安全问题是限制转基因作物商业化的主要因素。虽然CRISPR/Cas对基因进行定点修饰后能通过自交后代分离剔除外源DNA,获得无外源DNA基因(DNA-free)的编辑植株。但目前只有美国对CRISPR编辑作物安全实行监管豁免[92],其他国家都还处于观望状态。因此,建立全程DNA-free的植物基因组编辑系统对于推动基因编辑作物的商业化利用具有重要意义。目前,DNA-free植物基因组编辑系统主要有瞬时表达CRISPR/Cas9编辑系统[42]和RGEN(RNA- guided engineered nuclease)核糖核蛋白(RGEN ribonucleoproteins,RGEN RNPs)编辑系统[93]。
瞬时表达CRISPR/Cas9编辑系统的操作流程和原理是:将CRISPR/Cas9质粒DNA(TECCDNA)或其转录的RNA(TECCRNA)通过基因枪法直接转入植物愈伤组织,因是环状质粒或RNA,故不易被整合进植物基因组中;在完成切割使命后,质粒DNA或RNA会被细胞内源核酸酶分解,从而实现全程DNA-free的基因组编辑[42]。研究人员对该系统在小麦基因组中的定向修饰特性进行了详细的研究。通过对4个小麦品种的7个内源基因(共9个靶位点)靶向修饰发现,TECCDNA在小麦T0转基因植株中诱导突变的效率为1.0%—9.5%,DNA-free突变植株的比例为43.8%—86.8%。对比分析显示,TECCDNA和经典CRISPR/ Cas9系统的定向编辑效率和脱靶效应上没有明显差别,但都显著高于TECCRNA[42]。
RGEN RNPs技术是将CRISPR-Cas蛋白和gRNA在体外组装成核糖核蛋白复合体,该复合体进入细胞后能迅速行使DNA切割功能,然后被细胞内源蛋白酶快速分解,从而实现全程无外源DNA整合的基因组编辑[94-95]。RGEN RNPs技术最先应用于低等动物[76]和人类细胞[77],随后被应用于拟南芥[93]、烟草[93]、莴苣[93]、水稻[93]、矮牵牛花[96]、玉米[41]、小麦[97]和大豆[84]的基因组编辑。
2015年,WOO等[93]首先将RGEN RNPs技术成功用于植物的基因组编辑。研究表明,RGEN RNPs在拟南芥、烟草和水稻的原生质体中定向编辑的效率分别为16%—19%、17%—44%和8.4%—23%,在转化莴苣的再生植株中定向编辑的效率为45.7%,且没有检测到脱靶突变[93]。此外,SUBBURAJ等[96]通过RGEN RNPs技术对矮牵牛花内源基因PhNR(nitrate reductase)的6个位点(NR1-6)进行定向修饰,发现在原生质体中RGEN RNPs诱导定点突变的平均效率为(11.5±2)%。显然,这两项研究只聚焦在植物原生质体层面的基因组定向修饰,还未真正涉及作物的遗传改良。
2016年11月,《Nature Communications》报道了美国杜邦先锋公司利用RGEN RNPs技术获得了无叶舌、雄性不育和抗氯磺隆的玉米改良系,标志着RGEN RNPs技术在主要作物遗传改良上的真正应用[41]。研究人员通过基因枪法将组装好的RGEN RNPs,及RGEN RNPs和同源供体片段转入玉米愈伤组织,通过NHEJ途径实现对玉米无叶舌基因(LIG)、雄性育性基因(Ms26和Ms45)进行定点突变,通过HDR途径实现对ALS2基因定点替换(编码区的第165位脯氨酸突变为丝氨酸),最终获得了全程无外源DNA作为载体的DNA-free基因编辑玉米,表现为无叶舌、雄性不育和抗氯磺隆[41];对比分析发现,RGEN RNPs和Cas9-gRNA质粒(即TECCDNA)定向编辑效率基本一致,但RGEN RNPs诱导的脱靶效应显著低于Cas9-gRNA质粒诱导的[41]。类似地,LIANG[97]等利用RGEN RNPs技术对小麦TaGW2进行定向编辑,深度测序结果表明CRISPR/Cas9 RNP定向编辑的效率较CRISPR/Cas9 DNA要低,脱靶的机率也更低,且在突变植株中未检测到脱靶现象。
上述报道都是基于CRISPR/Cas9 RNP的DNA- free植物基因组编辑。2017年,KIM等[84]首次利用CRISPR/Cpf1 RNP成功对大豆FAD2和烟草AOC实现定点编辑,效率最高达11.7%,且在大豆基因组潜在脱靶位点未检测到显著突变。
2 基因组编辑技术应用于作物改良的基本原则
2.1 ZFNs、TALENs和CRISPR/Cas如何抉择
ZFNs、TALENs和CRISPR/Cas都是有效的基因组编辑技术,但三者在设计、特异性和效率上各有不同。ZFNs由于特异性不高、脱靶问题严重及获得ZFN蛋白非常困难,严重阻碍了其广泛应用。TALENs的优点是特异高、脱靶效应低,但载体构建较繁琐[98]、编辑效率不是很高、且难以同时对多个基因进行编辑[23, 99-100]。CRISPR/Cas系统的优点是编辑效率非常高[33, 101],设计和构建极其简单(只需2—3 d),仅需设计、合成靶点识别序列,且也只需将sgRNA串联就能实现多基因编辑[102];但CRISPR/Cas系统的特异性稍差,存在较明显的脱靶效应[103-105],另受PAM识别位点限制。目前,ZFNs已基本被TALENs和CRISPR/Cas系统所取代,而TALENs在植物基因组编辑中也已逐渐失去优势。因此,在作物遗传改良上,优先推荐使用CRISPR/Cas系统,如CRISPR/Cas系统无合适的靶位点或对特异性要求极高的情况下可使用TALENs技术。2.2 基因敲除还是基因替换
目前,基因编辑技术应用于作物遗传改良主要有两种方式,一是通过靶向敲除目标性状负调控基因,造成该基因功能缺失,以改良目标性状;二是通过对目标性状控制基因进行定点替换,导致该基因功能发生改变,从而获得新的目标性状。因此,在实践中运用基因组编辑技术对作物进行遗传改良时也要分以下2种情况:一是对于目标性状起负调控作用的基因采取基因敲除的方式,目前,绝大部分基于基因组编辑技术的作物遗传改良都是通过此方式实现的,如WANG等[26]对水稻稻瘟病抗性的改良,ZHOU等[32]创制水稻温敏核雄性不育系等;二是对于目标性状的获得是因目标基因突变导致基因功能发生改变的基因,采用基因定点替换的方式,如抗除草剂水稻、玉米等都是通过基因定点替换而获得的[35-38, 41]。2.3 靶位点如何选择
目前,已有多个专门设计SSNs靶位点和搜索脱靶位点的网站和工具[106]。而在作物遗传改良的实际操作中,是敲除多基因还是单基因、靶位点在基因上的位置及数目都是需要考虑的。如果需要同时改良多个目标性状,以及目标性状是由微效多基因或多等位基因控制的情况,最好针对每个目标性状控制基因,或微效多基因等设计多个靶位点进行定向敲除,能最快、最省地获得改良目标性状;而如果控制目标性状的是主效基因,则敲除单个基因一般即可达到改良目的。靶位点在基因上的位置也比较重要,进行基因敲除时,靶位点应优先选择在起始密码子附近,或在特定的功能域(可能引起密码子缺失和移码);而如果是基因替换,靶位点则需选择在基因特定功能区(突变后基因功能发生改变的区域)。
此外,靶位点的数量也关系到定向编辑的效率和遗传转化的规模,笔者在试验中发现,敲除同一个基因时分别设计1个、2个和3个靶点进行修饰,突变的效率分别为42%、70%(2个靶点都突变的为63.3%)和90%(3个靶点都突变),获得纯合突变株的效率分别为14.3%、47.6%和40.7%[26]。因此,在基因敲除时,建议单个基因设计2个靶位点,能减轻遗传转化的工作量,也能更省、更快地获得纯合突变植株,极大地节省人力和物力。
2.4 如何快速获得纯合的DNA-free编辑植株
目前,获得纯合的DNA-free编辑植株主要有2种途径:一是通过经典的TALENs和CRISPR/Cas9技术获得基因得以修饰的植株后,通过自交或杂交的方式剔除外源基因;二是直接利用DNA-free植物基因组编辑系统对植物基因组进行定向编辑。2种途径都有各自的优缺点,通过自交或杂交剔除的途径相对来说操作简单、成本低,普通的分子实验室都能操作完成,且最快能在T1代获得纯合的DNA-free编辑植株[20, 26]。DNA-free植物基因组编辑系统的优势在于能得到全程无外源DNA整合的编辑植株,且在T0代即可获得纯合的DNA-free编辑植株[41-42],但相对经典的CRISPR/Cas9系统操作较复杂、成本更高。因此,想要最快获得纯合的DNA-free编辑植株,有条件的实验室优先推荐使用RGEN RNPs编辑系统,其次推荐瞬时表达CRISPR/Cas9编辑系统中的TECCRNA方式,而TECCDNA方式因质粒DNA被细胞内源核酸酶分解后的小片段DNA有时会插入靶位点和非靶位点处,如果插入的DNA片段过小,目前的检测手段还不能对其进行有效鉴定,故不推荐;没有条件利用DNA-free植物基因组编辑系统的情况下,则使用经典的CRISPR/Cas系统。3 基因组编辑技术在作物遗传改良上应用的机遇与挑战
作物转基因育种的历史已有20多年,很多转基因作物如棉花、大豆、玉米等已在多个国家种植,但转基因生物(genetically modified organism,GMO)的生物安全性问题一直备受关注。基因编辑技术的出现为作物分子育种提供了新的机遇,但仍然面临政策法规和技术优化两方面的挑战。目前,国际上对基因编辑作物是否属于转基因生物尚未有明确一致的定论,其监管标准也存在较大争议。美国农业监管部门认为基因编辑作物(genome edited crop,GEC)是通过细胞自我修复机制而产生的突变体,与作物自然突变以及物理和化学诱导突变极其相似,都是少数几个核苷酸的改变,因此,GEC不被定义为GMO[107]。欧盟对GMO监管较为严格,其认为只要有外源DNA转入,而不论最终植物中是否还存在外源DNA,都被定义为GMO。从科学角度来讲,基因编辑诱导的突变与自然突变以及物理和化学诱导突变性质一样,实质无法区分,因此欧盟也有建议将GEC定义为非GMO。但基于政治考虑,欧盟尚未下明确定论,对GEC的监管标准存在不确定性[108]。目前,中国对于基因编辑作物的监管标准尚无明确的规定,因此亟需出台相关的政策法规以正确引导基因编辑育种。迄今,美国农业监管部门至少已认定5种基因编辑作物新品种不属于GMO范畴,即通过ZFNs技术创制的低植酸玉米品种,通过TALEN技术创制的耐冷藏马铃薯品种和高油酸、低亚油酸大豆,以及通过CRISPR/Cas9技术创制的抗褐化双孢菇和抗除草剂玉米。为正确引导基因编辑技术应用于作物育种,中国科学院的李家洋院士等针对基因编辑作物提出了一个监管框架,并发表在《Nature Genetics》杂志。文章对基因编辑作物的监管提出了几点建议[109]:一是在研究阶段应尽量减少GEC不受控的传播风险;二是要确保GEC中外源DNA被完全剔除;三是准确记录靶点DNA的变化,如果通过同源同组引入了新的序列,必须注明供体和受体之间的亲缘关系;四是基于参考基因组信息和全基因组测序技术检测并确定主要靶点没有意外的二次编辑事件和考虑潜在脱靶事件的后果;五是将上述四点信息在新品种资料中登记备案,除上述四点外,GEC只需执行常规作物品种的监管标准即可。同期,该杂志编辑部发文支持GEC管理框架的建议,并倡导GEC品种在完成透明的原则下,不需要进一步的监管,及与传统品种进行区分[110]。
基因组编辑技术从出现至今不过短短十多年时间,但其已广泛应用于生物医学和农业等领域。特别是TALENs和CRISPR/Cas9技术出现后,极大地加快了作物分子育种的研究步伐。目前,CRISPR/Cas技术已成为基因编辑应用的首选,虽然发明至今不到5年时间,但已被广泛应用于生物和农业的各个领域,而其本身也历经了多次技术优化。例如,利用Cas9变体将脱靶率降低到检测不到的水平[111-112],对Cas9和Cpf1蛋白进行定点突变扩大了PAM位点的选择范围[113-114],基于CRISPR/Cas9的植物基因组单碱基编辑系统[89-91],基于CRISPR/Cas9的植物基因组定点插入和替换系统[35-41, 115],以及DNA-free植物基因组编辑系统[41-42]等。
目前,基因编辑技术应用于作物遗传改良,主要是通过定点突变靶标基因,造成基因功能缺失(loss- of-function)以实现性状改良,故敲除的基因必须都是目标性状负调控基因。但作物许多性状改良都需要获得基因功能(gain-of-function),因此,基因组定点替换或插入的基因组编辑具有更广泛的应用前景,而植物细胞中同源重组效率很低,目前植物基因组编辑技术对基因单碱基替换、片段定点插入和替换等编辑的效率还很低。如目前已报道的植物单碱基定点替换技术在水稻、小麦和玉米中的编辑效率为0.39%—20%(大都低于10%),且只能是C→T(G→A)碱基替换[89-91];而已报道的植物基因组定点插入和替换技术在水稻、玉米和拟南芥中的编辑效率也极低,片段定点替换效率普遍低于1%[35-37, 40]。因此,对大部分控制重要农艺性状的正调控基因目前还无法高效、精准地进行编辑,从而极大地限制了基因编辑技术大规模应用于作物遗传改良的步伐。
虽然目前基因组编辑技术应用于作物遗传改良还存在政策法规和技术优化两方面的挑战,但在植物基因功能研究和作物遗传改良上其更具有其他分子技术无法比拟的巨大优势和机遇。放眼未来,我们预计GEC品种监管的法规肯定会宽松;实现基因定点替换和插入的基因组编辑技术一定会得到优化。随着大量作物品种全基因组测序的完成和越来越多重要农艺性状不利基因被发现,基因组编辑技术必将在作物遗传改良和品种培育上发挥巨大的作用。
(责任编辑 李莉)
The authors have declared that no competing interests exist.
