0 引言
【研究意义】紫花苜蓿(Medicago sativa L.)是世界上最重要的栽培牧草之一。秋眠性是其适应晚秋日照缩短、温度降低的一种生长特性[1]。紫花苜蓿秋眠性是其引种、区划及种植期选择的重要理论依据。因此,揭示紫花苜蓿秋眠性遗传规律,实现紫花苜蓿秋眠性的基因定位,并获得与之紧密连锁的分子标记,对紫花苜蓿正确引种及区划具有重要的指导意义。【前人研究进展】紫花苜蓿秋眠性的概念是Oakley和Westover在1921年提出的[2],至今已有近百年。在这期间,科学家们用大量时间对秋眠性测定的方法、理论开展了研究,形成了目前以秋季最后一次收割后植株生长高度为标准的9级/11级分级体系[3,4]。紫花苜蓿秋眠性被认为受到光照和温度的共同调控。因此,利用表型[5,6]、生理生态[7,8]、及分子生物学[9,10]手段,研究光照和低温对紫花苜蓿秋眠性调控也得到广泛开展。FAN等[11]利用转录组测序技术,比较分析了不同秋眠级紫花苜蓿在秋眠过程中的microRNA表达谱的差异,发现28个miRNA参与了秋眠性的调控,其中含20个已知miRNA和8个新报道miRNA。LI等[12]利用不同标记对苜蓿分离群体开展秋眠性的QTL定位研究,对揭示紫花苜蓿秋眠性分子遗传机制提供了重要的参考。紫花苜蓿秋眠性是典型的复杂数量性状,目前针对其QTL定位研究主要以连锁分析为主。以连锁不平衡为基础的关联分析以现有的自然群体为材料,无需构建专门的作图群体,多态性标记的覆盖面和多种等位基因型以及利用历史上的重组事件,使关联定位较传统的连锁分析具有精度高、广度大的优点[13,14,15]。【本研究切入点】近年来虽然紫花苜蓿关联分析已有应用[16,17],但对其秋眠性的研究还未见报道。【拟解决的关键问题】以来源于世界各地的紫花苜蓿种质为材料,采用均匀分布于紫花苜蓿全基因组的SSR引物,对紫花苜蓿秋眠性进行关联分析,旨在发掘与其相关联的位点,为揭示其遗传调控提供理论依据。1 材料与方法
试验于2012—2015年在河北省农林科学院旱作农业研究所实验基地内进行。1.1 试验材料
紫花苜蓿关联群体由75份四倍体紫花苜蓿种质构建[17],其中9个中国紫花苜蓿品种由国家草种质资源库提供,2份叙利亚种质,1份利比亚种质,1份苏丹种质由中国农业科学院北京畜牧兽医研究所提供,其余62份由美国国家种质资源系统(NPGS)提供。中国的紫花苜蓿品种,每份材料选取7—8个基因型;其余材料每份选取3—4个基因型,共321个基因型。试验为随机区组设计,每个基因型2个重复,每个重复包括6个无性扦插苗,株距30 cm,小区间距75 cm。1.2 表型鉴定
紫花苜蓿秋眠性用秋季株高表示,在一年中最后一次刈割约21 d后进行植物株高测量。2013—2015连续3年进行测定,由于2013年数据缺失较多,所以本试验对2014、2015两年数据进行分析。1.3 SSR标记分析
DNA提取采用CTAB法。试验所使用的85对SSR标记来源于EUJAYL等[18]和ROBINS等[19]。基因型鉴定采用DNA测序仪(ABI 3730XL)进行[17]。1.4 数据分析
利用SAS 8.02的GLM(General linear model)对两年秋季株高数据进行ANOVA分析,同时进行广义遗传力分析。群体结构采用STRUCTURE 2.2分析[20],群体结构分类标准为Q值>0.7,否则定为Mix群体。亲缘关系矩阵(K)采用SPAGeDi分析[21]。采用Tassel v2.1的GLM(general linear model)和MLM(mixed linear model)两种模型进行性状和标记之间的关联分析[22]。性状与标记之间显著关联的阈值设为P<0.005。2 结果
2.1 紫花苜蓿关联群体秋季株高变异分析
321个紫花苜蓿基因型秋季株高2014—2015两年结果统计如表1所示。2014年,秋季株高平均为22.4 cm,变异幅度在5.1—55.1 cm,变异系数为45.5%。2015年,秋季株高平均为15.2 cm,变异幅度在3.5—44.9 cm,变异系数为43.7%(表1)。方差分析(ANOVA)结果显示,紫花苜蓿秋季株高在321个基因型之间表现出极显著差异(P<0.01),说明不同基因型之间紫花苜蓿秋季株高多样性丰富(表1)。同时,该性状表现出极显著的年际及年际×基因型互作效应,说明该性状具典型的数量性状特性。如表1所示,该性状广义遗传力为0.71,说明该性状受遗传控制程度较大。年际间相比,除群体I年际之间差异不显著外,
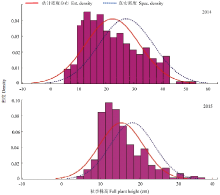
2015年所有基因型、群体II及Mix群体平均秋季株高均显著低于2014年(图1)。正态性检验结果显示,紫花苜蓿秋季株高两年均呈现接近正态分布特征(图2)。
Table 1
表1
表1紫花苜蓿秋季株高表型变异
Table 1Phenotypic variation of fall plant height in alfalfa
| 年 Year | 平均值±标准差 Mean±SD (cm) | 最小值 Min (cm) | 最大值 Max (cm) | 变异系数 CV | 遗传力 H2 | 基因型 Genotypes | 年 Year | 基因型×年 Genotype × year |
|---|---|---|---|---|---|---|---|---|
| 2014 | 22.4 ± 10.21 | 5.1 | 55.1 | 45.5 | 0.71 | ** | ** | ** |
| 2015 | 15.2 ± 6.67 | 3.5 | 44.9 | 43.7 |
新窗口打开
2.2 紫花苜蓿秋季株高关联分析
为了控制假阳性关联,利用GLM和MLM两种模型对紫花苜蓿秋季株高进行关联分析,结果如表2、图3所示。由表2可知,利用GLM模型2014和2015年分别鉴定51和34个显著关联的SSR位点,远高于MLM模型(12和11个)。这是由于MLM模型考虑了群体结构和亲缘关系2个方面,降低了假阳性关联,因此检测到的位点大幅降低。这个结论在图3的QQ-plot中得到进一步验证。由图3可知,MLM模型的观测P值比GLM模型更接近预期值,说明MLM模型很好的控制了紫花苜蓿秋季株高的假阳性关联。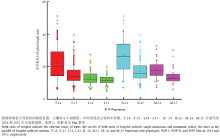 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图12014-2015年紫花苜蓿秋季株高Boxplot图
-->Fig. 1Boxplot of fall plant height in alfalfa from 2014 to 2015
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图22014—2015年紫花苜蓿秋季株高频率分布图
-->Fig. 2Histogram of fall plant height in alfalfa investigated from 2014 to 2015
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3紫花苜蓿秋季株高关联分析两种模型的QQ图
-->Fig. 3QQ plot of observed versus expected P-values using two models for alfalfa fall plant height
-->
基于MLM模型(表3),在2014年共找到12个显著关联的SSR位点,除Chr 8外,其余染色体上均有分布,表型贡献率为2.42%—6.73%。2015年共找到11个,分布在Chr1、2、3、4、6、8染色体上,表型贡献率为2.45%—4.81%(表3)。综合两年结果,分布于Chr 2和Chr 3上的均占到一半,说明调控紫花苜蓿秋季株高的等位基因位点及候选基因主要分布于这两条染色体上。在这些关联位点中,分布于Chr 2的m83_157、Chr 3的m525_230和m525_231、及Chr 4的m429_245,不仅在2014和2015年重复检测到(表3),而且在两种模型中均重复检测到。显著关联位点的等位基因效应见表3。在2014年中,除第5条染色体的m53_114为正效应外(4.61 cm),其余11个均为负效应,其中位于第4条染色体的m429_245负效应最强(-16.11 cm)。在2015年中,11个关联位点中有3个为正效应,8个为负效应。其中,位于Chr 6的m329_206正效应最强(1.96 cm),位于Chr 4的m429_245负效应最强(-19.19 cm)。
Table 2
表2
表2紫花苜蓿秋季株高不同模型关联分析
Table 2Association mapping of alfalfa fall plant height using two models in two years (P<0.005)
| 年 Year | GLM | MLM | ||
|---|---|---|---|---|
| 位点 No of loci | 表型解释率 R2 (%) | 位点 No of loci | 表型解释率 R2 (%) | |
| 2014 | 51 | 1.78—8.35 | 12 | 2.42—6.73 |
| 2015 | 34 | 2.23—9.96 | 11 | 2.45—4.81 |
新窗口打开
Table 3
表3
表3紫花苜蓿秋季株高关联位点及表型变异的贡献率
Table 3The SSR locus associated with alfalfa fall plant height and their explained phenotypic variation
| 位点 Loci | 染色体Chr | 位置 Location | 2014 | 2015 | ||||
|---|---|---|---|---|---|---|---|---|
| P值 P value | 贡献率R2 | 效应Effect | P值 P value | 贡献率R2 | 效应 Effect | |||
| m208_198 | 1 | 99 | 0.000671 | 3.6 | -5.12 | - | - | - |
| m243_206 | 1 | 169 | - | - | - | 0.001907 | 2.87 | 1.88 |
| m546_150 | 2 | 300 | - | - | - | 0.003098 | 2.72 | -1.77 |
| m546_176 | 2 | 313 | - | - | - | 0.00023 | 4.25 | -1.76 |
| m606_108 | 2 | 232 | - | - | - | 0.004879 | 2.45 | 1.61 |
| m83_149 | 2 | 337 | 0.001529 | 3.24 | -5.6 | - | - | - |
| m83_157 | 2 | 341 | 0.004332 | 2.62 | -15.20 | 8.54E-05 | 4.81 | -13.14 |
| m52_105 | 3 | 478 | 0.001665 | 3.25 | -3.35 | - | - | - |
| m525_230 | 3 | 358 | 0.000225 | 4.17 | -3.84 | 0.004121 | 2.45 | -1.94 |
| m525_231 | 3 | 359 | 3.25E-06 | 6.73 | -4.91 | 0.000306 | 3.91 | -2.36 |
| m525_236 | 3 | 362 | 0.001747 | 2.99 | -3.33 | - | - | - |
| m429_245 | 4 | 535 | 0.002746 | 2.73 | -16.11 | 0.000133 | 4.39 | -19.19 |
| m646_177 | 4 | 558 | - | - | - | 0.002097 | 3.04 | -0.16 |
| m53_114 | 5 | 697 | 0.004655 | 2.42 | 4.61 | - | - | - |
| m53_167 | 5 | 728 | 0.000185 | 4.26 | -8.80 | - | - | - |
| m298_177 | 6 | 757 | 0.001523 | 3.09 | -2.82 | - | - | - |
| m329_206 | 6 | 748 | - | - | - | 0.003837 | 2.57 | 1.96 |
| m2_140 | 7 | 825 | 0.004821 | 2.43 | -2.65 | - | - | - |
| m206_267 | 8 | 970 | - | - | - | 0.002271 | 2.88 | -8.31 |
新窗口打开
3 讨论
本试验检测到紫花苜蓿秋季株高广义遗传力为0.71,这与LI等[12]研究结果相似(0.74),而高于BROUWER等[23]的结果(0.45、0.63)。总的来说,紫花苜蓿秋眠性主要受遗传调控,因此适宜于通过遗传方法进行改良。紫花苜蓿是同源四倍体(2n=4X=32),具有异花授粉、高度杂合、自交不亲和及基因组信息少等特点,使紫花苜蓿遗传图谱构建和QTL 定位等领域进展缓慢。自YU等[24]1993年利用RAPD标记构建了第一张四倍体紫花苜蓿遗传图谱以来,迄今为止,已构建了十余张四倍体紫花苜蓿的遗传图谱。许多与产量/形态性状[18,25]、持久性[26]、抗寒性[12,23]、抗病虫[27,28]、水分利用效率及抗旱性[29,30]等性状相关的QTL已经在四倍体紫花苜蓿连锁图谱上进行了定位。目前国内外对秋眠性开展的研究并不多。BROUWER等[23]利用RFLP标记对两个苜蓿分离群体进行了秋眠性QTL分析,分布于Chr 1、Chr 3、和Chr 8的3个相关标记在多个环境中被重复检测到。LI等[16]报道指出,分布于Chr 1和Chr 7号染色体的2个秋眠相关QTL在多个环境中被重复检测到。紫花苜蓿秋眠性是受光温调控的复杂数量性状,关联分析是研究复杂数量性状的有效方法。本研究中,分别利用GLM和MLM两种模型进行关联分析。通过GLM模型,有15个显著关联位点在两年内均检测到,而在MLM模型中仅有4个被重复检测到,分别位于Chr 2、Chr 3、Chr 4号染色体上。这4个关联位点在GLM模型中也被重复检测到,表明这些标记较稳定。而且,这4个位点在前人研究中未见报道,为新发现。
4 结论
利用多样性丰富的321个紫花苜蓿基因型构成的关联群体,对其秋眠性状开展关联分析,利用GLM和MLM两种关联模型分别检测到85个和23个与紫花苜蓿秋眠性相关标记,其中4个新位点在两种模型和两年中均被检测到,分别位于Chr 2、Chr 3、Chr 4号染色体上。研究结果为克隆新的秋眠性相关基因以及通过分子标记辅助育种加速紫花苜蓿新品种培育提供了有用信息。The authors have declared that no competing interests exist.
