0 引言
【研究意义】研究表明,miRNA可在转录后通过降解或抑制黑色素形成相关基因的翻译过程来调控毛色性状的表达。通过高通量测序技术获得藏绵羊皮肤组织miRNA表达谱,并筛选和毛色性状相关miRNA,对系统研究转录后水平调控藏绵羊毛色性状的通路具有重要的意义。【前人研究进展】随着分子生物学和细胞生物学的快速发展,人们逐步认识到调控毛色的分子机制非常复杂,在黑色素细胞的迁移和分化以及黑色素的产生和分布等过程中受大量基因的协同调控[1,2,3],涉及到许多信号通路,如Wnt信号通路[4,5]、SCF/Kit信号通路[6,7]、Notch信号通路[8,9]等。miR-27a-3p可通过靶向作用于Wnt3a从而抑制黑色素的合成[10]。miR-21a-5p可通过抑制Sox5基因下调表达MITF基因和TYR基因使得黑色素合成减少[11]。miR-211[12]、miR-508-3p[13]等可通过靶向MITF基因降低黑色素细胞内黑色素的合成。miR-137则可通过多种通路靶向特定基因导致黑色素合成水平降低[14,15,16]。【本研究切入点】随着高通量测序技术的快速发展,应用测序技术在羊毛品质方面的研究越来越多,筛选出了一些和毛囊发育相关的miRNA[17,18],也有部分****利用高通量测序技术获得羊驼、山羊等物种不同毛色皮肤miRAN表达谱[19,20],然而在绵羊毛色方面的筛选工作鲜有报道。【拟解决的关键问题】拟通过miRNA-Seq技术,发现更多潜在调控绵羊毛色可能的miRNA,为后续研究毛色调控的分子机制奠定基础。1 材料与方法
1.1 材料
试验所用藏绵羊采自西藏自治区林芝市八一镇,分别采集2只具有黑白花毛色的藏绵羊皮肤组织(图1),每只藏绵羊分别取黑色和白色皮肤组织,编号分别为:1W,1B,2W,2B(数字代表藏绵羊编号,W表示白色皮肤组织,B表示黑色皮肤组织)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1白色和黑色皮肤组织样品采集于2只黑白花藏绵羊
-->Fig. 1The white and black skin tissues were collected from two Tibetan black and white sheep
-->
1.2 方法
1.2.1 样品采集 藏绵羊活体状态下,剪去藏绵羊体侧羊毛,使皮肤组织完全裸露,用75% 酒精对皮肤表面消毒后屠宰绵羊,在不同毛色部位快速割取皮肤组织,迅速置于组织样品保存液RNAstore中保存(购自TaKaRa宝生物工程(大连)有限公司)。组织保存液保存12h后置于-80℃永久保存。1.2.2 总RNA提取、文库构建与测序 将冻存在-80℃保存的皮肤组织取出,置于冰盒中解冻后,将皮肤组织剪成末状,加入1mL Trizol试剂后匀浆。室温放置10min,在12 000 r/min 4℃条件下离心10min。将上清液转移至新的RNAse Free的1.5mL离心管中。用RNeasy Micro kit进行提取和纯化,总RNA质量经Agilent2100检测。提取的藏绵羊皮肤组织RNA样品交由上海伯豪生物技术有限公司进行文库构建并在Illumina Hiseq 2000平台进行测序。
1.2.3 数据处理与分析 (1)FastQC(http://www.bioinformatics.babraha m.ac.uk/projects/fastqc/)和Fastx(fastx_toolkit-0.0.13.2)用于对测序数据进行质控和预处理。(2)Bowtie(http://bowtie-bio.sourceforge. net/),用于序列比对。(3)将比对到绵羊基因组(Ensemble 基因组数据库:http://www.ensembl.org/)上的reads,利用miRDeep2软件[21]与miRBase20.0[22]数据库(http://www.mirbase.org/)中绵羊及牛的miRNA序列进行比对分析,鉴定出已知保守的miRNA和预测novel miRNA。(4)用基于负二项分布的DESeq软件(http://www.bioconductor.org/ packages/release/bioc/html/edgeR.html/)对数据进行统计分析,获得差异表达miRNA。(5)用TargetScan、PicTar和DIANA-microT v3.0,用于microRNA靶基因预测:A. http://www.targetscan.org/(TargetScan);B. http://pictar.mdc-berlin.de/(PicTar);C. http://diana. cslab.ece.ntua.gr/microT/(DIANA-microT v3.0)。(6)DAVID软件(http://david.abcc.ncifcrf.gov/)用于KEGG通路分析和GO分析。
1.2.4 cDNA及引物的合成 对提取的总RNA用全式金(北京全式金生物技术有限公司)的miRNA反转录试剂盒(TransScript miRNA RT Enzyme Mix)进行cDNA的合成(加尾法)。委托北京擎科新业生物技术有限公司合成引物,并用于荧光定量PCR。引物序列信息见表1。
Table 1
表1
表1荧光定量引物基因列表
Table 1The list of gene primers of qRT-PCR
| miRNA | 上游引物序列 Primer sequences (5′-3′) | miRNA序列 miRNA sequences(5′-3′) | |
|---|---|---|---|
| oar-miR-411a-5p | ATAGTAGACCGTATAGCGTACG | AUAGUAGACCGUAUAGCGUACG | |
| oar-miR-30c | GCGTGTAAACATCCTACACTCTCA | UGUAAACAUCCUACACUCUCA | |
| oar-miR-423-3p | AGCTCGGTCTGAGGCCCCTCAGT | AGCUCGGUCUGAGGCCCCUCAGU | |
| oar-miR-628-5p | GCGCTGACATATTTACTAGAGGG | AUGCUGACAUAUUUACUAGAGGG | |
| oar-miR-151-5p | GCTCGAGGAGCTCACAGTCTAG | UCGAGGAGCUCACAGUCUAGU | |
| oar-miR-103 | AGCAGCATTGTACAGGGCTATG | AGCAGCAUUGUACAGGGCUAUG | |
| oar-miR-24-3p | TGGCTCAGTTCAGCAGGAAC | UGGCUCAGUUCAGCAGGAAC | |
| U6-F | CTCGCTTCGGCAGCACA | ||
| U6-R | AACGCTTCACGAATTTGCGT | ||
| miRNA反转录引物 miRNA reverse transcriptional primer | GATCGCCCTTCTACGTCGTATCGTCATCTGACCGTTATCGCTGCACGTTTTTTTTTTTTTTTTTTTT | ||
| qPCR下游通用引物 qPCR downstream generic primers | GATCGCCCTTCTACGTCGTAT | ||
新窗口打开
1.2.5 Real-time PCR 分析 Real-time PCR 用于检测藏绵羊黑色和白色皮肤组织中差异表达miRNA的相对表达量。反应体系包括:Nuclease-FreeWater(3.6μL)、上游引物(0.2μL)、下游引物(0.2μL)、qPCR Master Mix(5μL),样品1μL,构成总体积为10μL 反应体系。反应条件为95℃变性2 min,95℃10s,60℃ 30s,72℃ 25s,40 个PCR 循环,采用默认设置自动生成Ct 值。RT-PCR 相对定量的内参选择U6。每个样品设置3个重复。
2 结果
2.1 数据预处理及数据总体情况
应用Fastx(fastx_toolkit-0.0.13.2)对测序原始reads进行预处理,去除接头序列以及低质量序列,总共获得85 756 541条原始reads,经过滤后得到85 076 302条clean reads,结果见表2。筛选白色皮肤组织和黑色皮肤组织中获得的clean reads,选取一定长度范围内的small RNA进行长度统计分布,本试验过滤掉18bp以下的reads,结果见图2。大多数reads分布在21—24nt之间,白色皮肤组织中,22nt的reads占到总reads数的20.33%,24nt的reads数占到总reads数的22.01%,同样,黑色皮肤组织的22nt和24nt分别占到总reads数的20.16%和19.70%。
Table 2
表2
表2数据过滤情况信息
Table 2The information of raw data filtering
| 样本名称 Sample name | 总Reads数 Total reads | 过滤Reads数 Clean reads | 过滤百分比 Clean ratio | Q20值 Q20 | Q30值 Q30 |
|---|---|---|---|---|---|
| 1B | 21 242 319 | 21 098 740 | 99.32% | 95.55% | 90.11% |
| 2B | 21 190 378 | 20 936 910 | 98.80% | 95.14% | 89.56% |
| 1W | 23 296 720 | 23 203 857 | 99.60% | 96.06% | 90.96% |
| 2W | 20 027 124 | 19 836 795 | 99.05% | 95.59% | 90.22% |
新窗口打开
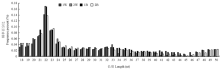 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2长度分布情况
-->Fig. 2The distribution of read lengths from Solexa sequencing data
-->
2.2 已知miRNA的鉴定
从miRBase 20.0数据库中下载绵羊和牛的已知miRNA成熟序列和前体,从Ensemble绵羊基因组数据库中下载绵羊的基因组数据,利用bowtie软件将大于18bp的clean reads比对至绵羊全基因组,应用MiRDeep2软件对测序数据进行注释分析,获得已知的miRNA。共334个保守的miRNA被鉴定,此外预测出59个新的novel miRNA。其中75个miRNA和牛的同源,占miRNA的19.02 %,100个miRNA和绵羊的同源,占miRNA的25.45 %,山羊的则占到41.39 %(图3)。其中,10个表达最高的miRNA见表3。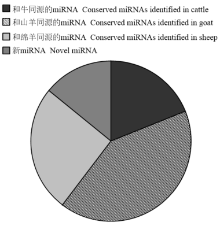 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3山羊miRNA注释情况
-->Fig. 3Annotation of miRNA
-->
Table 3
表3
表3藏绵羊皮肤中表达量前10的miRNA
Table 3The most abundantly expressed miRNA in Tibetan sheep skin tissues (Top 10)
| miRNA名称 miRNA name | 标准化后表达量 Normalized expression level | 成熟序列 Mature sequences | |||
|---|---|---|---|---|---|
| 1W | 2W | 1B | 2B | ||
| oar-miR-26a | 602845.5 | 310620.9 | 480900.6 | 468074.9 | uucaaguaauccaggauaggcu |
| oar-miR-143 | 438273.2 | 358600.1 | 456624.7 | 475479.6 | ugagaugaagcacuguagcucg |
| oar-miR-10b | 291169.5 | 221939.2 | 294070.4 | 306866.7 | uacccuguagaaccgaauuugu |
| oar-miR-27a-3p | 202229.4 | 143789.0 | 198022.4 | 181256.2 | uucacaguggcuaaguucug |
| oar-miR-181a | 182943.2 | 108167.6 | 155674.6 | 142686.0 | aacauucaacgcugucggugagu |
| oar-miR-22-3p | 115254.0 | 100157.4 | 95987.81 | 111496.4 | aagcugccaguugaagaacugu |
| oar-miR-205 | 125057.6 | 82819.1 | 111672.1 | 88451.08 | uccuucauuccaccggagucug |
| oar-miR-199a-3p | 63039.34 | 65631.51 | 72534.59 | 88648.49 | acaguagucugcacauugguu |
| oar-miR-25-3p | 73133.47 | 49057.62 | 62949.66 | 60981.26 | aauugcacgguauccaucug |
| oar-let-7a | 61053.65 | 48268.74 | 57625.1 | 50859.58 | ugagguaguagguuguauaguu |
新窗口打开
2.3 不同毛色皮肤组织差异表达miRNA分析
将获得的已知miRNA用DEGseq软件进行差异表达分析,将这些数据进行归一化处理后鉴定差异表达miRNA(图4)。总共鉴定出83个差异表达miRNA。通过以下标准进一步筛选差异表达miRNA:A. 标准化后的倍数差异(log2 Fold Change)大于0.5;B. 标准化后的表达量大于100;C. p值小于等于0.001。共获得23个差异表达miRNA,其中14个差异表达miRNA在白色皮肤组织中表达显著上调,9个则表达下调(表4)。由图4可以直观的看出,差异表达的基因不多,
并且以在黑色皮肤中上调表达为主。由表4可以看出,有2个miRNA(oar-miR-2284b和oar-miR-744)在黑色皮肤中不表达,有5个miRNA(oar-miR-23b,oar-miR-411a-5p,oar-miR-30c,oar-miR-423-3p,oar-miR-324-5p)在白色皮肤中不表达。部分miRNA表达量相对较低,仅有miR-23b相对表达量大于10 000。表达量相对较高的有oar-miR-103、oar-miR- 200b、oar-miR-486、oar-miR-24-3p、oar-miR-151-5p和oar-miR-10a,但倍数差异不大,其中oar-miR-10a表达量最高。
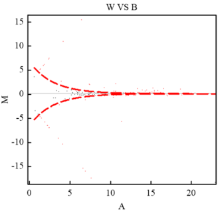 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4藏绵羊皮肤中已知miRNA平均值VS倍数变化作图
-->Fig. 4Plot of normalized mean versus log2-fold change for the miRNA in black and white skin
-->
Table 4
表4
表4藏绵羊皮肤组织差异表达miRNA
Table 4Differentially expressed miRNA among different Tibetan sheep skin sample
| 序号No. | MiRNA名称Gene names | 白色皮肤White skin | 黑色皮肤Black skin | 标准化后倍数差异log2(F C) normalized |
|---|---|---|---|---|
| 1 | oar-miR-2284b | 3214 | 0 | 12.7504034 |
| 2 | oar-miR-744 | 144 | 0 | 8.270174185 |
| 3 | oar-miR-2285b | 651 | 172 | 2.020498162 |
| 4 | oar-miR-505 | 179 | 81 | 1.244214958 |
| 5 | oar-miR-103 | 11042 | 5539 | 1.095553246 |
| 6 | oar-miR-200c | 2895 | 1503 | 1.045967523 |
| 7 | oar-miR-200b | 27099 | 14860 | 0.967054682 |
| 8 | oar-miR-10a | 109458 | 62866 | 0.900274709 |
| 9 | oar-miR-532-5p | 4029 | 2628 | 0.716705714 |
| 10 | oar-miR-423-5p | 5781 | 3849 | 0.687084585 |
| 11 | oar-miR-486 | 10914 | 7438 | 0.65344248 |
| 12 | oar-miR-24-3p | 14616 | 10012 | 0.646067527 |
| 13 | oar-miR-664b | 329 | 242 | 0.54332972 |
| 14 | oar-miR-708-3p | 4809 | 3620 | 0.509996413 |
| 15 | oar-miR-23b | 0 | 16561 | -14.91525299 |
| 16 | oar-miR-411a-5p | 0 | 6437 | -13.55192357 |
| 17 | oar-miR-30c | 0 | 4300 | -12.96987176 |
| 18 | oar-miR-423-3p | 0 | 4140 | -12.91516587 |
| 19 | oar-miR-324-5p | 0 | 134 | -7.965840007 |
| 20 | oar-miR-628-5p | 71 | 4014 | -5.720828598 |
| 21 | oar-miR-493-5p | 194 | 363 | -0.803663712 |
| 22 | oar-miR-151-5p | 21818 | 33898 | -0.535432113 |
| 23 | oar-miR-2284a | 636 | 973 | -0.513163856 |
新窗口打开
2.4 novel miRNA预测
miRDeep2会根据miRNA的结构和相近物种miRNA预测出新的miRNA,根据miRDeep2 score分值、成熟miRNA与星号miRNA比例以及预测出的miRNA二级结构进行筛选。下表列出了在毛囊中表达的前11个novel miRNA的相关信息(表5和表6)和预测的miRNA结构(图5)。在这些预测的novel miRNA中,仅Novel-1-3p表达量较高,其余novel miRNA表达量很低。图5中预测的novel miRNA的二级结构中,红色的表示novel miRNA的成熟序列,黄色表示颈环序列,蓝色表示星号序列,紫色表示miRNA的星号序列。Table 5
表5
表5藏绵羊皮肤组织中预测的novel miRNA表达量
Table 5The 11 most abundantly expressed novel miRNA in Tibetan sheep skin tissues (Top 11)
| MiRNA名称 MiRNA name | 标准化后的表达量 Normalized expression level | 成熟序列 Mature sequences | |||
|---|---|---|---|---|---|
| 1W | 2W | 1B | 2B | ||
| Novel-1-3p | 48168 | 51657 | 54418 | 59143 | guacaguacugugauaacuga |
| Novel-2-3p | 1033 | 701 | 884 | 790 | aagaaccugaaugaaccuuuugg |
| Novel-3-3p | 336 | 290 | 362 | 311 | caacaaaucacagucugccaua |
| Novel-4-5p | 233 | 137 | 133 | 121 | uccgagccugggucucccucu |
| Novel-5-5p | 222 | 179 | 175 | 205 | aaaaaccugaacgaacucuuug |
| Novel-6-3p | 68 | 54 | 78 | 47 | uggagccggagcugguuagcggc |
| Novel-7-3p | 40 | 53 | 59 | 37 | augcggcaccugcggauacgg |
| Novel-8-3p | 31 | 16 | 32 | 21 | uguacucugggacucggguguca |
| Novel-9-5p | 13 | 7 | 14 | 13 | acugcuuuaaaaccuuugacag |
| Novel-10-3p | 10 | 11 | 9 | 11 | gcugaggcugcucaggcuguga |
| Novel-11-3p | 10 | 10 | 5 | 15 | ugcugagccgucgcuuucacu |
新窗口打开
Table 6
表6
表6藏绵羊皮肤组织中预测的novel miRNA前体信息
Table 6Predicted precursor sequences and genome locations of 11 novel miRNA
| MiRNA名称MiRNA name | MiRNA前体序列 MiRNA precursor sequence | 染色体位置 Chromosome | Strand |
|---|---|---|---|
| Novel-1-3p | caguuaucacagugcugaugcuguccauucuaaagguacaguacugugauaacuga | chr1 | - |
| Novel-2-3p | agaaguucguuuggguuuucccguaagaugguacagaagaaccugaaugaaccuuuugg | chr10 | + |
| Novel-3-3p | uggaagacuagugauuuuguuguuuuuagauaacugaaucgacaacaaaucacagucugccaua | chr2 | + |
| Novel-4-5p | ggggguccccggugcucggaucucgagggugcuuauuguucgguccgagccugggucucccucu | chr3 | - |
| Novel-5-5p | aaaaguucauuugugguuuuucucuaauaucuuauggaaaaaccugaacgaacucuuug | chr3 | - |
| Novel-6-3p | cgcuggcccgccccgcuccaggaagugaccgaaaaccuggagccggagcugguuagcggc | chr1 | - |
| Novel-7-3p | auguccgcggguucccuaucccagguuggucgaguucacggaugcggcaccugcggauacgg | chr3 | + |
| Novel-8-3p | uccccgagucccuggcgugcacugacgccacgucacccgguguacucugggacucggguguca | chr1 | + |
| Novel-9-5p | acugcuuuaaaaccuuugacaggugcugggaugaucugucaaaaguuuuaaagcaguca | chr23 | - |
| Novel-10-3p | acacccugcucagcuucagaagcccaggcccucuccgcugaggcugcucaggcuguga | chr14 | - |
| Novel-11-3p | agagagcgacugcgcuuagaagaaggcuaacauaaauccugcugagccgucgcuuucacu | chr4 | + |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5预测novel miRNA的结构
-->Fig. 5Predicted secondary structures of novel miRNA
-->
2.5 miRNA靶基因预测及基因功能分析
高通量测序技术快速发展以来,对miRNA靶基因的预测也随之迅速开展,产生了大量的预测靶基因软件。为避免单一的软件预测可能造成较多的假阳性,本试验用Target Scan(http://www.targetscan. org/)、PicTar(http://pictar.mdc-berlin.de/)和DIANA- microT v3.0(http://diana.cslab.ece.ntua.gr/microT/) 3种方法预测靶基因,将任意两种软件都有交叉 的miRNA筛选出来,共预测出981个靶基因可能参与到调控当中。为了更好地将这些基因进行 归类并统计分析,对这些预测的靶基因进行了功能分析。将预测出的靶基因用DAVID软件进行GO分析和PATHWAY通路分析。共发掘出24个通路,下图列出了前17个可能作用于毛色的通路(图6)。根据信号通路中包含的靶基因数量及可能的生物学意义,在这些通路当中,MAPK信号通路、WNT信号通路、ErbB信号通路及Melanogenesis通路与毛色调控相关,这些通路分别包含预测出的70个、60个、35个和33个靶基因。
在Melanogenesis通路中(图7),发现许多基因和WNT信号通路、SCF/Kit信号通路相关,通过影响MITF基因的表达起到调控毛色的作用。大量研究表明,WNT信号通路和MAPK通路及ErbB通路联系密切,因此推测,色素的合成同时受到多种信号通路共同的作用。
将预测得到的靶基因进行GO分析,结果显示这些靶基因涉及116个生物学过程,包括Regulation of transcription(21%)、Transcription(16%)、Regulation of RNA metabolic process(14%)、Regulation of transcription, DNA-dependent(13%)、Intracellular signaling cascade(10%)等。这些生物学过程多与基因的转录调控、细胞周期及增殖相关。表7中列出了前5个所涉及到的生物学过程。同时表8和表9分别列出了前5个cellular component中的分类和molecular function中的分类(表9)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6藏绵羊白色皮肤VS黑色皮肤样品差异表达miRNA靶基因KEGG Pathway通路分析
-->Fig. 6KEGG Pathway analysis of predicted target genes of differently expressed miRNA between white and black skin sample in Tibtan sheep (Top 17)
-->
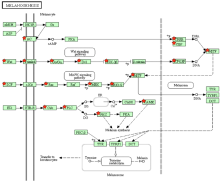 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7Melanogenesis Pathway信号通路
-->Fig. 7Melanogenesis signal Pathway
-->
Table 7
表7
表7藏绵羊白色皮肤VS黑色皮肤样品差异表达miRNA靶基因GO分析
Table 7GO analysis of predicted target genes of differently expressed miRNA between white and black skin sample in Tibetan sheep (Biological process)
| GO编号 GO accession | GO分类 GO terms | 基因数目 Gene count | 百分比 Percentage (%) | P值P value |
|---|---|---|---|---|
| GO:0045449 | 转录调控Regulation of transcription | 642 | 19.80 | 2.46E-23 |
| GO:0006350 | 转录 Transcription | 521 | 16.07 | 6.10E-19 |
| GO:0051252 | RNA代谢过程调控 Regulation of RNA metabolic process | 441 | 13.60 | 5.23E-14 |
| GO:0006355 | DNA依赖性的转录调控 Regulation of transcription, DNA-dependent | 427 | 13.17 | 7.90E-13 |
| GO:0007242 | 细胞内信号放大级联Intracellular signaling cascade | 300 | 9.25 | 1.08E-08 |
新窗口打开
Table 8
表8
表8藏绵羊白色皮肤VS黑色皮肤样品差异表达miRNA靶基因GO分析(Cellular Component)
Table 8GO analysis of predicted target genes of differently expressed miRNA between white and black skin sample in Tibetan sheep (Cellular Component)
| GO编号 GO accession | GO分类 GO terms | 基因数目 Gene count | 百分比 Percentage (%) | P值P value |
|---|---|---|---|---|
| GO:0043228 | 非膜界限的细胞器Non-membrane-bounded organelle | 507 | 15.63 | 8.37E-06 |
| GO:0043232 | 细胞内非膜界限的细胞器 Intracellular non-membrane-bounded organelle | 507 | 15.63 | 8.37E-06 |
| GO:0044459 | 浆膜部分 Plasma membrane part | 447 | 13.78 | 4.93E-07 |
| GO:0031974 | 膜封闭的内腔 Membrane-enclosed lumen | 371 | 11.44 | 2.82E-05 |
| GO:0043233 | 细胞器内腔 Organelle lumen | 364 | 11.22 | 3.26E-05 |
新窗口打开
Table 9
表9
表9藏绵羊白色皮肤VS黑色皮肤样品差异表达miRNA靶基因GO分析
Table 9GO analysis of predicted target genes of differently expressed miRNA between white and black skin sample in Tibetan sheep (Molecular Function)
| GO编号 GO accession | GO分类 GO terms | 基因数目 Gene count | 百分比 Percentage (%) | P值P value |
|---|---|---|---|---|
| GO:0043167 | 离子结合 Ion binding | 859 | 26.49 | 9.00E-06 |
| GO:0043169 | 阳离子结合Cation binding | 844 | 26.03 | 1.84E-05 |
| GO:0046872 | 金属离子结合Metal ion binding | 839 | 25.87 | 1.15E-05 |
| GO:0046914 | 过渡金属离子结合 Transition metal ion binding | 566 | 17.45 | 5.32E-04 |
| GO:0003677 | DNA结合DNA binding | 513 | 15.82 | 1.09E-07 |
新窗口打开
2.6 差异表达miRNA定量PCR的验证
选取藏绵羊黑色皮肤和白色皮肤中7个差异表达miRNA(oar-miR-411a-5p,oar-miR-30c,oar-miR-423-3p,oar-miR-628-5p,oar-miR-151-5p,oar-miR-103,oar- miR-24-3p)进行定量验证,结果表明:oar-miR- 411a-5p,oar-miR-30c,oar-miR-423-3p,oar-miR-628-5p在黑色皮肤组织中上调表达,而oar-miR-103和oar-miR-24-3p两个miRNA在白色皮肤组织中下调。与高通量测序结果一致(图8)。其中,oar-miR-411a-5p倍数差异最大。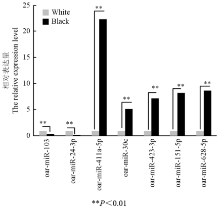 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8差异表达miRNA在藏绵羊黑色和白色皮肤组织中的相对表达情况
-->Fig. 8The relative expression of miRNAs in black and white skin tissues of Tibetan sheep
-->
3 讨论
本研究利用高通量测序技术对白色和黑色皮肤组织中的miRNA进行测序并分析了差异表达miRNA,筛选出23个差异表达miRNA,其中miR-23b、miR-411a-5p、miR103、miR-10a和miR-200b可能和毛色调控关系密切。有研究表明,miR-23a可通过调控TGF-β信号通路促进骨细胞的分化[23],而TGF-β信号通路可间接激活Wnt/β-catenin信号通路[24],而miR-23b则与Notch信号通路有关[25]。SUN等[26]在横纹肌肉瘤增殖分化的研究中发现,miR-411-5p与TGF-β信号通路和MAPK信号通路有关。所获得的miR103、miR-10a和miR-200b差异表达miRNA在毛色调控方面还鲜有报道。这些miRNA均是参与调节多种信号通路的多功能miRNA,其作用涉及增殖、分化、凋亡、细胞粘附等各个方面,而且关于其功能及作用机制的研究仍在不断扩展。通过靶基因预测并进行功能分析,发现毛色调控和Wnt信号通路、MAPK信号通路、EDNRB信号通路及Melanogenesis通路均有关,暗示调控毛色性状的分子机制多样而复杂。大量研究表明,WNT信号通路和MAPK信号通路共同影响着色素沉积[27,28]。KIM等[29]通过芯片发掘了地方牛光鼻和暗色鼻子的差异表达基因,发现MAPK和WNT信号通路可能通过独特的方式影响两种性状的表达。其中MAPK信号通路可能通过激活cAMP而导致真黑素的合成。YE[30]等发现,经典的WNT信号通路的激活可使小鼠毛囊毛球部位的成熟黑色素细胞数量明显增多,并且色素沉积现象明显。但也有报道表明,WNT通路可以抑制黑色素的沉积,PARK等[31]通过WIF(Wnt Inhibitory Factor)在体外培养的人正常黑色素细胞中抑制WNT通路,发现MITF和TYR基因的表达量显著上升,因此,WIF可以促进黑色素细胞的色素沉积。YAMADA等[32]通过激光显微切割技术分离了毛囊的四个部位(毛球部、毛囊隆突部、毛囊隆突部上部区域和下部区域),研究了Frizzled (Fzd)-4、 Fzd7、 Lrp5和Lrp6的mRNA表达水平,发现在毛囊隆突部这些基因均高表达。而毛囊隆突部是黑色素干细胞定居的场所。这些基因又是WNT信号通路关键基因,因此推测WNT信号通路在黑色素干细胞分化为成熟黑色素细胞的过程中发挥了重要的作用。本研究中,大量基因指向多种信号通路,因此具体何种信号通路起到主要调控毛色性状仍需进一步的研究。
4 结论
本研究获得藏绵羊不同毛色皮肤组织miRNA表达谱及23个差异表达miRNA,这些miRNA功能较广且调节多种信号通路。靶基因预测和功能分析表明,调控毛色的信号通路可能涉及Wnt信号通路、MAPK信号通路等多种途径。这为进一步鉴定调控藏绵羊毛色的miRNA和信号通路奠定了理论基础。The authors have declared that no competing interests exist.
