0 引言
【研究意义】‘中矮1号’是中国农业科学院果树研究所选育的中国第一个优良梨树矮化砧木,其株型矮化紧凑,且具有使嫁接树矮化、早果的作用[1],在梨树矮化密植栽培中发挥着重要作用,但其矮生和矮化机制尚不清楚,限制了新的矮生型品种和矮化砧木的选育及改良。植株高矮与茎的伸长紧密相关,生长素是调节植物茎伸长的重要激素之一,而植物对生长素的反应受许多基因的调控,生长素响应因子(auxin response factor,ARF)则是其中一类较为重要的调节基因,它能够通过调节植物对生长素的反应而调控植株的生长发育[2,3]。因此,鉴定梨基因组中ARF转录因子成员并分析其结构及其在矮生梨树不同组织器官中的表达规律,将有助于解析该家族基因在矮生梨树中的作用机制,同时,也为进一步克隆梨树ARF和研究其在生长素信号途径中的作用奠定基础。【前人研究进展】ARF转录因子调节生长素反应是通过调节生长素反应基因的表达来实现的,它可以与植物生长素反应基因启动子上的生长素反应元件(auxin response elements,AuxRE)TGTCTC特异结合,进而调节生长素反应基因的表达[4]。在植物中,ARF转录因子是以基因家族的形式存在的,AtARF1是第一个被鉴定出来的ARF[5],继而在拟南芥中共鉴定出23个ARF[6],随着越来越多植物基因组测序的完成,很多大田作物、蔬菜和果树中的ARF也已经被鉴定出来,如玉米中鉴定出31个[7],水稻25个[8],大豆51个[9],番茄17个[10],黄瓜18个[11],葡萄19个[12],苹果29个[13],甜橙19个[14],番木瓜11个[15],香蕉47个[16]。【本研究切入点】梨基因组测序数据的发布已使梨基因组中ARF的分离与鉴定成为可能[17],但还未见相关研究报道,尤其缺少不同ARF的表达与植株高矮关系的研究。【拟解决的关键问题】本研究利用生物信息学方法鉴定梨基因组中所有ARF,以明确该家族基因在梨树中的结构特点与进化特征,通过荧光定量PCR技术分析该基因家族成员在梨树不同组织器官和不同品种中的表达差异,为进一步研究其在梨树矮生性状形成过程中的作用奠定基础。1 材料与方法
1.1 植物材料
供试材料‘中矮1号’(矮生)、‘锦香’(半矮生,‘中矮1号’母本)及‘早酥’(非矮生),树龄20年以上,基砧均为杜梨(Pyrus betulifolia Bge.),每个品种3株。分别于2016年4月27日采集‘中矮1号’的花(每花序只采集1朵边花)和根(基砧杜梨),6月21日采集上述3个品种新梢顶部幼叶、新梢中部的韧皮部和木质部(每个单株采集新梢3个,摘除叶片,剪去枝条基部和顶部幼嫩处,将韧皮部和木质部分离,分别迅速剪碎混匀),6月30日采集‘中矮1号’的果实(每个单株采集果实3个,切成小块混匀),锡箔纸包裹,液氮速冻后保存于-80℃冰箱中待用。1.2 梨基因组中ARF家族基因鉴定
由于梨与苹果均属于蔷薇科仁果类果树,二者亲缘关系较近,因此,本研究以苹果和模式植物拟南芥中已鉴定出的ARF序列为诱饵,利用NCBI网站的BLAST在线软件(http://blast.ncbi.nlm.nih.gov/Blast. cgi)对梨基因组Pyrus bretschneideri(taxid:225117)进行定向tblastn检索,数据库选择RefSeq RNA,从而获得梨候选ARF的mRNA、蛋白和对应的基因序列。利用SMART(http://smart.embl-heidelberg.de/)和PROSITE(http://prosite.expasy.org/)在线软件对候选基因的蛋白序列进行分析,以确定其含有ARF所特有的Auxin_resp保守结构域。1.3 梨ARF蛋白序列的保守结构域及保守元件分析
利用本地软件DNAMAN v6、在线软件WebLogo 3(http://weblogo.threeplusone.com/)和MEME Suite 4.11.1(http://meme-suite.org/tools/meme)对梨ARF蛋白序列的保守结构域和保守元件进行进一步分析。1.4 梨ARF蛋白的进化分析
以获取的梨ARF蛋白序列和文献报道的苹果、葡萄和拟南芥等植物的ARF蛋白序列进行进化树分析,利用MEGA5软件构建系统进化树,采用NJ 方法(执行参数:bootstrap:1 000次重复、Poission model和pairwise deletion)。1.5 梨ARF的内含子、外显子结构分析
根据BLAST鉴定获得梨ARF的CDS序列和对应的基因序列,采用在线软件GSDS2.0(http://gsds.cbi.pku.edu.cn/)对其进行内含子、外显子结构分析。
1.6 梨ARF表达分析
将1.1中采自不同单株的相同样品等量均匀混合研磨,以Plantol高效总RNA提取试剂盒(深圳莱伯克生物科技有限公司)提取各样品的总RNA,使用DNaseⅠ(大连宝生物科技有限公司)去除总RNA中的DNA,用Prime ScriptTM RT reagent Kit(大连宝生物科技有限公司)对RNA进行反转录,采用SYBR®Premix Ex TaqTMⅡ(大连宝生物科技有限公司)进行RT-qPCR反应,试验过程均按各试剂盒的使用说明书进行。RT-qPCR反应采用20 μL体系:SYBR Premix Ex Taq II 10 μL,正、反向引物各1 μL(10 mmol·L-1),cDNA 1 μL,ddH2O 7 μL。反应程序为:95℃,5 min;95℃,30 s;55—60℃(表1),30 s;72℃,30 s,32个循环。以ACTIN(GU830958.1)为内参,所用引物见表1。采用2-ΔΔCT方法计算各基因的相对表达量。RT-qPCR反应设置3次技术重复,采用DPS 7.05软件对结果进行差异显著性分析。Table 1
表1
表1qPCR所用引物序列及退火温度
Table 1Primer sequences and their annealing temperature used in qPCR
| 基因 Gene name | 正向引物(5′→3′) Forward primer | 反向引物(5′→3′) Reverse primer | 退火温度 Annealing temperature (℃) |
|---|---|---|---|
| PbARF1 | TCATCTACACCAGAGAGGAAGTCCA | ATCCACACCCTCCGCACCT | 60 |
| PbARF2/3 | CCTTGAGAGCCAGTTTTGCG | CCATTATTCCCGAAGCCTGA | 60 |
| PbARF4 | TTGATGCGGAATCTGACCA | CCTTCCAACAGCAATCCCT | 57 |
| PbARF5 | TATGATGCTTATGGGTGATGAC | AACTTCTTGTGACGACATTGG | 55 |
| PbARF6 | TGATGCTTATGGGCGATGAC | GATGACCGTCCCCCCTTC | 60 |
| PbARF7/8 | GAATGTAACTATGGGGTCTGTGC | ACAACCTAAAACCTGAAGTGCC | 58 |
| PbARF9 | TCTTGTACACTGATAGTGAGAACGA | ATTACAACATCACAATGACCACC | 57 |
| PbARF10 | ACAAGCGCCGGTCATGC | CCTCGGAACTCCATACAATACATAA | 55 |
| PbARF11 | TGGAGTGGAGGTTCCGACAT | ACCACATCGGTGACAGTGCTAT | 60 |
| PbARF12 | AGGACCATAGAATAAATCGGACTC | GCTGCCACTTATTGTTGATTGA | 58 |
| PbARF13 | AAGCGTTGATGTTGGTAGCG | TGCCGAGGTAATCTGCGAC | 59 |
| PbARF14 | CTTTTCCGGGGCACTCA | ACACAAGGGTCAACGACAGTAC | 57 |
| PbARF15 | GCTGCCCACACAGATTACTTG | CCTTGCTAACCCCACCACA | 58 |
| PbARF16 | ACTCATCAATAGGGGAGGACATA | CGCTGCACCGTTCATGTAT | 57 |
| PbARF17 | CGGCAACATTCCAGTCCC | CATTCGAACCCTTACCGATTAA | 59 |
| PbARF18 | ACGCCAAGAGACATTGAGACAG | CCCACGATCCATTGCCTAA | 59 |
| PbARF19 | CGTTGACAGGGAGGATGATG | AGTGAGCCAAGAGACGGTAGC | 58 |
| PbARF20 | CTTTTAGCCCAAATACAGGTCA | GAGCCGAGAGATGGTAGCC | 57 |
| PbARF21 | ATTTGTTGACAGGGAGAATGATG | TTGGGACAGACGCCACACT | 60 |
| PbARF22/23 | AAGGTTCATAAGTCAGGGTCC | GATCTCACAGGGTCGTCCA | 55 |
| PbARF24 | AAACTGAAGAAGGGCACAACC | CCCAACAATAAACACTTCACTACTG | 58 |
| PbARF25/26 | CCTCAGGCGTGCACTTACAA | AGCAGACTGGTTTGTATGAATGATC | 60 |
| PbARF27 | GCCATCATACAGGGAGCC | GTCTTTATCTCATCATTATCCTTCA | 55 |
| PbARF28 | CTTGGAAGACAGATGACAGCG | TTTCCTTGCCAAACATACCAG | 58 |
| PbARF29 | TGCAGAAAATGGACTGGGAG | CATTCATTCAGTATTCTCAAGCATC | 58 |
| PbARF30 | TCAACAAAGCATACAAGAGCG | TCATCATAGGACCCAAGAAGTG | 57 |
| PbARF31 | AAATGAAACCGAACTAAGTCTGG | CGTCAATCTCCTTGCCGTT | 58 |
| PbACTIN | GGAATGGAAGCTGCTGGC | CGTCATACTCACCCTTGGAAATC | 60 |
新窗口打开
2 结果
2.1 梨基因组中ARF家族基因鉴定
根据苹果和拟南芥ARF蛋白的序列信息,在梨基因组中检索,并通过结构域分析去除不含Auxin_resp结构域的基因,最终共获得31个梨ARF,为了便于后续分析,将其以PbARF1—31的方式进行命名,其中,将同一基因位点出现的多个转录变异体视为同一基因,后续分析时仅以转录变异体(transcript variant)X1对应的序列作为该基因的代表。分析表明,PbARFs的长度在587—1 154 aa,分子量在64.78—128.88 kD,等电点在5.07—7.82(表2),除PbARF2和PbARF3、PbARF7和PbARF8等2对序列完全相同外,其他序列均各不相同。Table 2
表2
表2梨ARF
Table 2ARF genes in pear
| 基因名 Gene name | a基因 ID Gene ID | 基因位点 Gene location | 染色体位置Chromosome location | 基因注释 Gene annotation | 编码蛋白 Deduced protein | ||
|---|---|---|---|---|---|---|---|
| 长度 Length (aa) | 分子量 kD MW | 等电点 PI | |||||
| PbARF1 | XM_009370525.2 | LOC103958273 | scaffold172.0 | bARF 2-like transcript variant X1 | 844 | 93.84 | 6.74 |
| PbARF2 | XM_009375357.2 | LOC103962621 | scaffold168.0.1 | ARF 2-like transcript variant X1 | 843 | 93.86 | 6.82 |
| PbARF3 | XM_009339237.2 | LOC103929963 | scaffold44.2.16 | ARF 2-like transcript variant X1 | 843 | 93.86 | 6.82 |
| PbARF4 | XM_009357101.2 | LOC103946402 | scaffold43.0.1 | ARF 1 | 677 | 75.30 | 6.38 |
| PbARF5 | XM_009360363.2 | LOC103949259 | scaffold67.0 | ARF 9 | 682 | 75.57 | 6.69 |
| PbARF6 | XM_009343599.2 | LOC103933897 | scaffold626.0 | ARF 9-like | 684 | 75.63 | 6.43 |
| PbARF7 | XM_009377135.2 | LOC103964226 | scaffold275.0 | ARF 18 transcript variant X1 | 695 | 77.58 | 7.07 |
| PbARF8 | XM_009338480.2 | LOC103929308 | scaffold450.0 | ARF 18 transcript variant X1 | 695 | 77.58 | 7.07 |
| PbARF9 | XM_009336773.2 | LOC103927821 | scaffold407.0 | ARF 4-like | 799 | 88.18 | 6.81 |
| PbARF10 | XM_009348760.2 | LOC103938724 | scaffold957.0 | ARF 4-like | 799 | 88.66 | 6.40 |
| PbARF11 | XM_009354565.2 | LOC103944151 | scaffold24.0 | ARF 3-like transcript variant X1 | 709 | 77.17 | 6.50 |
| PbARF12 | XM_018644940.1 | LOC103936579 | scaffold1.0.1 | ARF 3-like transcript variant X1 | 714 | 77.85 | 6.57 |
| PbARF13 | XM_009339384.2 | LOC103930094 | scaffold464.0 | ARF 5-like transcript variant X1 | 896 | 98.39 | 5.93 |
| PbARF14 | XM_018642727.1 | LOC103927986 | scaffold13.0.1 | ARF 5 | 896 | 97.98 | 5.07 |
| PbARF15 | XM_009371744.2 | LOC103959396 | scaffold187.0.1 | ARF 19-like transcript variant X1 | 1112 | 122.58 | 6.90 |
| PbARF16 | XM_009363741.2 | LOC103952195 | scaffold93.0 | ARF 19-like transcript variant X1 | 1118 | 123.74 | 6.80 |
| PbARF17 | XM_009361435.2 | LOC103950246 | scaffold76.0 | ARF 19 | 1108 | 123.35 | 7.15 |
| PbARF18 | XM_009338903.2 | LOC103929678 | scaffold458.0.1 | ARF 7 | 1154 | 128.88 | 6.83 |
| PbARF19 | XM_009343591.2 | LOC103933889 | scaffold618.0 | ARF 8-like transcript variant X1 | 844 | 93.83 | 6.25 |
| PbARF20 | XM_009372313.2 | LOC103959932 | scaffold194.0.1 | ARF 8-like transcript variant X1 | 848 | 94.34 | 6.25 |
| PbARF21 | XM_018651961.1 | LOC103965484 | scaffold11.0 | ARF 6-like transcript variant X1 | 931 | 102.99 | 6.69 |
| PbARF22 | XM_009362673.2 | LOC103951320 | scaffold85.0.1 | ARF 6 | 893 | 98.90 | 6.60 |
| PbARF23 | XM_009379210.2 | LOC103966082 | scaffold314.0 | ARF 6-like | 893 | 98.89 | 6.54 |
| PbARF24 | XM_018649812.1 | LOC103956998 | scaffold152.0.1 | ARF 17 | 597 | 65.71 | 6.01 |
| PbARF25 | XM_009368745.2 | LOC103956717 | scaffold149.0.1 | ARF 17-like | 587 | 64.81 | 6.03 |
| PbARF26 | XM_009345978.2 | LOC103936134 | scaffold756.0 | ARF 17-like | 587 | 64.78 | 6.03 |
| PbARF27 | XM_009346490.2 | LOC103936643 | scaffold98.0.12 | ARF 18-like transcript variant X1 | 668 | 73.60 | 7.81 |
| PbARF28 | XM_009347111.2 | LOC103937192 | scaffold828.0 | ARF 18-like | 665 | 73.31 | 6.57 |
| PbARF29 | XM_009368787.2 | LOC103956759 | scaffold150.0 | ARF 18 transcript variant X1 | 723 | 79.38 | 7.82 |
| PbARF30 | XM_009344648.2 | LOC103934885 | scaffold15.0.1 | ARF 18-like transcript variant X1 | 702 | 77.25 | 7.01 |
| PbARF31 | XM_009375254.2 | LOC103962541 | scaffold243.0.1 | ARF 18-like transcript variant X1 | 703 | 77.24 | 6.76 |
新窗口打开
2.2 梨ARF结构域分析
通过SMART和PROSITE在线软件对所获得的蛋白序列进行检索分析,所有候选蛋白的N端除含有1个AFR蛋白所特有的Auxin_resp结构域外,还含有1个B3结构域,除PbARF11、12、24、25和26外,其他序列的C末端还存在一个PB1(AUX_IAA)结构域。通过DNAMAN比对和Weblogo在线分析,发现上述3个结构域在梨中高度保守。其中,B3结构域中有40个氨基酸在所有序列中是完全保守的,约占结构域总长度(103 aa)的38.8%;Auxin_resp结构域中有9个氨基酸在所有序列中是完全保守的,约占结构域平均长度(83 aa)的10.8%;PB1结构域中有10个氨基酸在含有该结构域的序列中是完全保守的,约占结构域平均长度(83 aa)的12.0%(图1)。利用在线软件MEME对梨ARF蛋白序列进行分析,结果显示,梨ARF蛋白含有15个保守元件。其中,元件1、2和10属于B3结构域,它们与元件3和元件5共同构成DNA结合结构域;元件6、7、8和14属于Auxin_resp结构域;元件4、9和15属于PB1结构域;元件11、12和13为未知结构域。除PbARF18缺失元件3和5外,所有序列均含有DNA结合结构域的5个元件和Auxin_resp结构域4个元件中的至少3个元件(PbARF5—8缺失元件8,PbARF24—31缺失元件14)。PbARF1—10和13—23含有PB1结构域中的所有3个元件,而PbARF11和12则缺失了其中的元件4和9,PbARF27—31则缺失了其中的元件15,PbARF27—28除缺失元件15外,还缺失元件4。此外,除PbARF11和12外,所有序列均含有未知元件11,而未知元件12仅在PbARF24和25—31中出现,未知元件13只在PbARF13和15—24中出现(图2)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1梨ARF蛋白的保守结构域
-->Fig. 1The conserved domains of ARF proteins in pear
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2梨ARF家族蛋白序列保守元件分布
-->Fig. 2Distribution of the identified motifs in pear ARF family proteins
-->
2.3 梨ARF蛋白的进化分析
利用MEGA5.05软件对本研究获得的31个梨及文献报道的29个苹果[13]、19个葡萄[12]和23个拟南芥[6]的共102个AFR氨基酸序列进行进化分析。结果表明,所有102个ARF成员可以被清晰分成4组:ClassⅠ、ClassⅡ、ClassⅢ和ClassⅣ,每组中都含有上述4个物种中的成员。其中,PbARF1—8等8个成员在ClassⅠ中,PbARF9—12等4个成员在ClassⅡ中,PbARF13—23等11个成员在ClassⅢ中,PbARF24—31等8个成员在ClassⅣ中。在所有31个PbARF中,除PBARF19、20、27和28等4个成员外,其他成员均能在苹果中找到与其配对的同源序列,苹果中也仅有MdARF1、4、5和14等4个成员在梨中没有与其配对的同源序列。在ClassⅠ中,AtARF12—15、20—23等8个拟南芥成员单独聚在一个小分支上,在梨、苹果和葡萄等其他3种果树中均未发现与其高度同源的序列(图3)。2.4 梨ARF的内含子、外显子结构分析
对获得的31个PbARF蛋白序列进行了进一步的进化分析,发现这些蛋白也可以被清晰的聚为4组,这与图3的分组结果基本一致,只是不同组间的进化关系略有不同。对获得的PbARF的CDS序列和对应的基因序列采用在线软件GSDS2.0(http:// gsds.cbi.pku.edu.cn/)进行内含子和外显子结构分析,发现PbARF家族基因外显子的数量在2—15,其中ClassⅣ成员的外显子数量最少,仅为2—3个,ClassⅡ成员的外显子数量为11—12,ClassⅠ和ClassⅢ成员的外显子数量多为14个,仅个别成员为13或15个(图4)。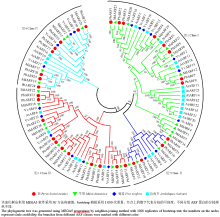 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3梨、苹果、葡萄和拟南芥ARF蛋白的进化关系与分类
-->Fig. 3Phylogenetic relationships and classification of ARF proteins from pear, apple, grape and Arabidopsis
-->
2.5 梨ARF表达分析
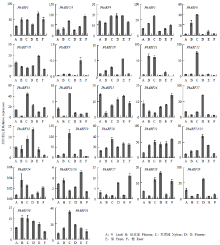
运用荧光定量PCR的方法对PbARF基因家族成员在矮生梨品种‘中矮1号’不同组织器官中的表达情况进行分析。由于PbARF2和PbARF3、PbARF7和PbARF8、PbARF22和PbARF23、PbARF25和PbARF26等4对基因的序列相同或或高度相似,难以设计特异表达引物加以区分,因此,表达量的检测结果的是1对基因的共同表达量。组织表达结果表明,所检测的PbARF基因家族成员几乎在‘中矮1号’所有组织中均有表达。其中,PbARF24相对于其他成员基因表达量较低;PbARF9和PbARF10在果实中的表达量相对高于其他组织;PbRF17、18、22/23、28、29在花中的表达量相对高于其他组织;PbARF6、11、12、13、14、19、31等7个成员则在木质部中的表达量相对较高,PbRF11除在木质部中表达相对较高外,在韧皮部中的表达量也相对较高(图5)。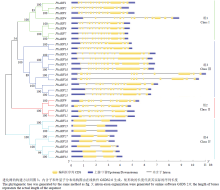 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4PbARF家族基因内含子和外显子分布结构图
-->Fig. 4Intron-exon structures of PbARF genes
-->
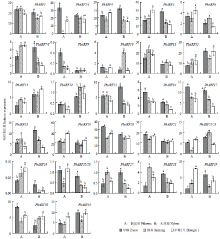
进一步比较PbARF基因家族成员在‘早酥’(乔化)、‘锦香’(‘中矮1号’母本,半矮化)和‘中矮1号’(矮化)等3个梨品种木质部和韧皮部中的表达情况。结果表明,不同PbARF在3个品种韧皮部和木质部中的表达各有高低,多无明显规律,仅PbARF29在3个品种韧皮部中的表达表现为植株越矮表达量越高的趋势,PbARF16、17、18、27等4个基因在3个品种木质部中的表达表现为植株越矮表达量越低的趋势(图6)。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5PbARF家族基因在梨‘中矮1号’不同组织器官和基砧杜梨根系中的相对表达量
-->Fig. 5The expression of PbARF family genes in different tissues from ‘Zhongai1’ pear and root from Pyrus betulifolia rootstock
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6PbARF家族基因在3个梨品种韧皮部和木质部中的相对表达量
-->Fig. 6The expression of PbARF family genes in phloem and xylem from 3 pear cultivars
-->
3 讨论
随着高通量测序和生物信息技术的发展,越来越多物种的基因组序列已得到解析,使得从基因组层面上对某一家族基因进行全面的鉴定和分析成为可能。ARF是生长素信号途径中一类重要的转录因子,其在基因组中即以家族的形式存在,分别调控植物生长发育的许多重要环节。ARF基因家族成员已在多个物种中被分离鉴定出来,但不同物种中ARF基因家族成员数量不同,本研究自梨基因组中鉴定出PbARF基因31个,与玉米[7]中的成员数量一致,比拟南芥、苹果[6,8,10-15]等多个物种中的成员数量均多,但少于大豆(51个)[9]和香蕉(47个)[16]。虽然梨的ARF数量与玉米中的数量一样多,但其基因组大小为527 Mb[17],远小于玉米的2 300 Mb[18];虽然基因组大小与香蕉的523 Mb[19]接近,但ARF数量却比香蕉中的少的多。由此可见,ARF在基因组中的数量多少不完全取决于基因组大小。有研究表明基因复制对基因家族的形成起着重要作用[20],在苹果中的研究即发现串联复制、片段复制和全基因组复制对苹果ARF基因组的扩展具有重要作用[13]。梨与苹果同属蔷薇科果树,ARF蛋白的进化分析结果表明,二者的亲缘关系较近,虽然梨基因组小于苹果(742 Mb[21]),但二者ARF基因数量接近,仅各有4个ARF蛋白没有形成互相配对的同源序列,其他成员均能在另一个基因组中找到与之配对的同源序列。因此,推测梨基因组中ARF的进化也经历了与苹果类似的过程,但由于基因组大小和组成不同,个别基因的进化过程和功能可能又略有不同。通过系统进化和基因内含子、外显子组成结构等分析,本研究中的PbARF蛋白可被划分为4组,该分组结果与水稻、葡萄和甜橙等[8,12,19]的研究结果一致,与苹果、玉米等[7,19]的研究结果略有不同,其不同的主要原因是他们对成员较多的组又进行了细致划分。但与梨一致的是,在其他物种中也有一个外显子数量较少的分组,其他分组成员的外显子数量均较多,可见,ARF在外显子和内含子进化上具有较大的一致性。
本研究鉴定的梨AFR蛋白多含有3个保守结构域,1个AFR蛋白所特有的Auxin_resp结构域、1个B3结构域和1个PB1结构域,但PbARF11、12、24、25和26等5个成员仅有2个保守结构域,PB1结构域缺失,与前人研究结果类似[22]。研究表明,B3结构域具有DNA结合活性[23],其可以与生长素反应基因启动子上的作用元件结合,进而通过与B3结构域相连的Auxin_resp结构域激活或抑制相关基因的表达[3]。PB1结构域是一个介导蛋白质-蛋白质互作的结构域,另一个在生长素信号途径中起抑制作用的AUX/IAA蛋白中也具有类似结构域,2个蛋白可以通过该结构域的互作而形成ARF-ARF、ARF-Aux/IAA和 Aux/IAA-Aux/IAA同源或异源寡聚体。当生长素浓度低时,ARF与Aux/IAA形成异源寡聚体,抑制生长素反应基因的表达;当生长素浓度升高时,Aux/IAA蛋白被泛素化途径降解,抑制作用解除[22]。但PbARF11、12、24、25和26等PB1结构域缺失的ARF是否有类似的功能还有待进一步研究。
荧光定量PCR表达分析结果表明,本研究中各PbARF在梨树根、韧皮部、木质部、叶、花和果实等不同组织器官中均可表达,在苹果[13]和番茄[24]等作物上也获得了类似结果。通过对拟南芥突变体的研究表明,AtARF6/AtARF8可以对开花时间和花器官的质量产生影响[25],本研究中PbARF19—23与其处于同一进化分枝,其中,PbARF22/23即在花器官中具有相对较高的表达量,可能与AtARF6/AtARF8具有类似的功能。AtARF10/AtARF16对根冠的形成具有重要作用[26],本研究中PbARF27—31与其处于较近的分枝上,PbARF27和PbARF28在根中均具有相对较高的表达量,可能与根的发育相关。AtARF7/AtARF19在侧根的形成、根系和下胚轴的向重力性中起重要作用[27],番茄中AtARF7的同源基因SlARF7则在花中具有较高的表达量,调控授粉受精过程[28],本研究中PbARF17、18与AtARF7、19处于较近的分枝上,在花中均有相对较高的表达量,与番茄中的结果一致,其功能或许与拟南芥不同。
‘中矮1号’不仅自身矮化,其作中间砧对嫁接品种也具有矮化作用,而中间砧仅由木质部和韧皮部两部分组成,可见控制其矮化性状的基因只能在木质部和韧皮中起作用;前期研究结果表明,新梢停长早是‘中矮1号’矮生的原因之一,6月中下旬是‘中矮1号’即将停长的关键时期[29]。因此,本研究还于6月21日采集3个不同生长势梨品种新梢的木质部和韧皮部,比较了PbARF在其中的表达情况,发现PbARF29在3个品种韧皮部中的表达表现为植株越矮表达量越高的趋势,PbARF16、17、18、27等4个基因在3个品种木质部中的表达表现为植株越矮表达量越低的趋势。PbARF16、17、18与AtARF7和AtARF19处于同一分枝,PbARF27、29与AtARF10和AtARF16处于较近分枝上,研究表明,上述基因在拟南芥中均可对根系的生长发育产生影响[26,27],其中,AtARF7和AtARF19功能有重叠,拟南芥arf7和arf19双突变体具有侧根发育严重缺陷和根及下胚轴向地性异常等表型,但各自的单突变体均无此表型[27];番茄中AtARF7的同源基因SlARF7具有调控果实授粉受精的功能[28];AtARF10和AtARF16也具有一定程度的功能冗余,他们均为microRNA160的靶基因,受其负调控,主要影响根冠的发育,同时,AtARF10还在种子的萌发过程中起重要作用[30],但均未见对植株高度影响的报道。尽管如此,由于梨树是木本植物,与拟南芥和番茄等物种的差异极大,这些基因是否能够影响梨树植株的生长还需进一步研究。
4 结论
梨基因组中含有31个ARF基因家族成员;所有PbARF蛋白均含有Auxin_resp和B3等2个高度保守的结构域;PbARF蛋白可以被划分为4组,基因结构进化高度保守;所有PbARF在‘中矮1号’不同组织器官及‘早酥’(乔化)、‘锦香’(‘中矮1号’母本,半矮化)和‘中矮1号’(矮化)3个梨品种的木质部和韧皮部中均有表达,其中,PbARF29、16、17、18、27等5个基因的表达可能与梨树植株的高矮有关。The authors have declared that no competing interests exist.
