0 引言
【研究意义】RNA剪接是从DNA模板链转录出的最初转录产物中除去内含子,并将外显子连接起来形成一个连续的RNA分子的过程,其在真核生物基因表达过程中具有重要意义[1]。在高等生物中,可以通过对同一个基因转录的前体RNA(pre-RNA)进行不同的选择性剪接(alternative splicing),从而产生不同的mRNA亚型,最终产生多种相似却又独特的蛋白质以达到调节基因表达的目的[2-3]。通过选择性剪接这一过程,增加了高等生物基因表达的复杂程度,同时对高等生物的细胞分化与器官发育也具有重要的调控作用[2]。【前人研究进展】细胞周期素依赖性蛋白激酶11(CDK11)是一类丝/苏氨酸蛋白激酶,在细胞周期调控、肿瘤的发生以及细胞凋亡等过程中扮演重要角色[4-5]。CDK11有两种主要的亚型CDK11P110和CDK11P58,其中,CDK11P110能够与cyclinL相互作用,参与前体RNA(pre-RNA)的剪接以及转录调控,在细胞增殖的过程中也具有重要作用[4-6]。CDK11P58是由CDK11P110mRNA编码区里的内部核糖体进入位点(internal ribosome entry site,IRES)翻译而来,在G2/M期特异表达,与中心体成熟、姐妹染色单体内聚和胞质分裂相关[7-9]。核糖核酸结合蛋白S1(RNA binding protein with serine rich domain 1,RNPS1)是一种具有核质穿梭功能的剪接因子,参与了mRNA剪接、mRNA的核监管与输出以及无义介导的mRNA衰变[10-11]。富含丝氨酸和精氨酸的剪接因子7(serine and arginine rich splicing factor 7,9G8)是一类重要的磷酸蛋白,能促进mRNA由核向质的输出[12],并且能够参与mRNA选择性剪接的过程[13]。RNPS1和9G8都属于SR蛋白家族,该蛋白家族成员都具有一个富含丝氨酸/精氨酸(S/R)重复序列的RS结构域,并且在RNA剪接体的组装和选择性剪接的调控过程中起着重要作用[14-16]。在对人类的研究中发现,CDK11能与cyclinL1、cyclinL2以及剪接因子相互作用,参与pre-RNA剪接的调控[17]。进一步研究表明,CDK11P110复合物能够分别与pre-RNA剪接因子RNPS1、9G8以及cyclinL相互作用,表明CDK11参与了pre-RNA剪接的过程并具有重要作用[6]。【本研究切入点】笔者课题组前期已经克隆并鉴定了家蚕(Bombyx mori)CDK11的两种重要剪切体CDK11A和CDK11B,并且验证了其与cyclinL的相互作用,表明CDK11在家蚕的细胞周期调控中起重要作用[18]。然而,其是否参与了pre-RNA剪接的过程,还未见明确报导。【拟解决的关键问题】在家蚕中克隆并鉴定SR家族的两种重要剪切因子RNPS1和9G8,并分别验证RNPS1和9G8与CDK11两种剪切体的相互作用,为进一步探索CDK11在家蚕pre-RNA剪接中的功能打下基础。1 材料与方法
试验于2016—2017年在西南大学家蚕基因组生物学国家重点实验室完成。1.1 试验材料
供试家蚕品种大造(Dazao)由西南大学家蚕基因资源库提供,在25℃下用新鲜桑叶饲育。家蚕胚胎细胞系为BmN-SWU1[19],由西南大学家蚕基因组生物学国家重点实验室保存,在27℃培养箱中用含10%胎牛血清的TC-100昆虫细胞培养基培养。1.2 主要试剂
TaKaRa反转录试剂盒购自宝生物工程有限公司;Trizol试剂购自Invitrogen公司;BCA蛋白浓度测定试剂盒、HRP标记山羊抗小鼠/兔抗体IgG(H+L)、DEPC水均购自碧云天生物技术研究所;蛋白分子量Marker购自Bio-Rad公司;质粒提取试剂盒购自Promega公司产品;Ex Taq酶、pMD19-T Simple载体、限制性内切酶和T4连接酶为TaKaRa公司产品;PVDF膜、转染试剂购自Roche公司;Trans1-T1 Phage Resistant感受态细胞购自北京全式金生物有限公司;Flag、HA标签单克隆抗体购自Sigma公司;蛋白A磁珠购自Invitrogen有限公司;引物均由北京六合华大基因科技股份有限公司合成;测序由北京六合华大基因科技股份有限公司完成;其他试剂均为国产或进口分析纯试剂。1.3 基因序列的生物信息学分析
本研究克隆的家蚕基因序列检索自家蚕基因组数据库SilkDB(http://www.silkdb.org/silkdb/);其他物种的相关序列均从NCBI(National Center for Biotechnology Information,https://www.ncbi.nlm.nih. gov/)检索并下载获得;结构域预测使用在线预测软件SMART(http://smart.embl-heidelberg.de/);引物设计的软件采用Primer 5.0;预测同源序列比对软件采用ClustalX 1.83和GENEDOC 3.2;系统发生树构建使用MEGA 6.0软件。1.4 总RNA提取及cDNA的合成
取5龄第3天的全蚕组织材料经液氮研磨后使用TRIZOL Reagent试剂盒提取总RNA。1%琼脂糖凝胶电泳检测RNA质量,并按照反转录试剂盒说明书反转录合成cDNA,保存于-20℃备用。1.5 家蚕RNPS1与9G8的克隆
以家蚕大造品系的全蚕cDNA作为模板,按表1所示的TA克隆引物进行目的片段的扩增,PCR反应体系(25 µL):cDNA 1 µL;Ex Taq酶0.2 µL;10×Ex Taq Buffer 2.5 µL;2.5 mmol·L-1 MgCl2 2 µL;2.5 mmol·L-1 dNTP 2 µL;上下游引物(10 µmol·L-1)各1 µL;ddH2O 16.3 µL。PCR条件:94℃预变性4 min,然后94℃变性40 s、57℃退火40 s、72℃延伸1 min,共35个循环,最后72℃延伸10 min。将扩增的产物进行胶回收,将回收的片段与pMD19-T载体进行连接,进行TA克隆,挑选阳性克隆送测序。将测序正确的TA克隆按表1所示亚克隆引物进行目的片段的扩增,经酶切、连接和转化后通过菌液PCR筛选阳性克隆,并送测序。Table 1
表1
表1引物列表
Table 1Primers used in this study
| 名称Name | 序列Sequence |
|---|---|
| RNPS1-T-F | 5′-ATGGCTAGAAATAAATCGCCTT-3′ |
| RNPS1-T-R | 5′-CTATCGTGAAGAGGATGAAGAAGA-3′ |
| RNPS1- BamHI-F | 5′-CGGGATCCATGGCTAGAAATAAATCGCCTTT-3′ |
| RNPS1-XbaI-R | 5′-GCTCTAGATCGTGAAGAGGATGAAGAAGAC-3′ |
| 9G8-T-F | 5′-ATGTCACGCTATGGTGACTG-3′ |
| 9G8-T-R | 5′-CTAATTAGATTTCTTGGAATCTTTAG-3′ |
| 9G8-BamHI-F | 5′-CGGGATCCATGTCACGCTATGGTGACTGCA-3′ |
| 9G8-XbaI-R | 5′-GCTCTAGAATTAGATTTCTTGGAATCTTTAGA-3′ |
新窗口打开
1.6 免疫荧光共定位
待家蚕BmN-SWU1细胞密度达到80%—90%时,将其转移到铺有爬片的24孔培养板中。将RNPS1-Flag和CDK11A-HA,RNPS1-Flag和CDK11B-HA,9G8-Flag和CDK11A-HA,9G8-Flag和CDK11B-HA分别共同转染,按质粒与转染试剂比例1﹕3(m/v)转染0.5 µg质粒,在无抗生素的细胞培养基中孵育30 min后,转染家蚕BmN-SWU1细胞。转染48 h后,进行免疫荧光处理。吸掉培养基,用PBS润洗2—3次,每个孔添加400 µL多聚甲醛对细胞进行固定;15 min后,弃掉液体,加入300 µL PBS,摇晃清洗5次,每次5 min;分别加入400 µL 0.1% Triton X-100,室温孵育15 min;吸掉Triton X-100,PBS摇晃清洗3次,每次5 min;分别加入封闭液(10%羊血清原液+3%牛血清蛋白的PBS液),37℃孵育1—1.5 h;吸掉封闭液,分别加入250 µL Flag、HA的抗体稀释液(稀释比例为1﹕200),37℃恒温培养箱孵育1 h;加入300 µL PBS,摇晃清洗5次,每次5 min;向每个孔中加入不同荧光标记山羊抗鼠的二抗,于37℃恒温培养进行孵育1 h;用封闭液将DAPI原液以1﹕500稀释,并加入孔中,室温染色15 min;加入300 µL PBS,摇晃清洗5次,每次5 min;用带钩的针头轻轻挑起爬片,将其铺在载玻片上,用指甲油封片,随后用激光共聚焦显微镜进行观察。1.7 免疫共沉淀验证蛋白相互作用
当细胞密度达到80%左右时进行转染,将每一对质粒进行共转,每一对质粒转两瓶;将转染72 h后的细胞用PBS润洗2—3次,加入1 mL的IP裂解液和10 µL的PMSF,在冰上摇动裂解30 min,然后液氮反复冻融2次,使细胞完全裂解,取出30 µL作为总蛋白样品;将细胞裂解液于4℃,12 000×g离心30 min,收集上清到1.5 mL EP管中备用,同时取出30 µL作为上清液样品;在两个新的1.5 mL EP管中分别加入50 µL protein A磁珠;将加有磁珠的EP管放到磁力架上,待磁珠完全被吸附住之后,将上清吸出弃掉,PBS清洗两次;分别在两个EP管中加入400 µL孵育液(PBST),同时,一个管中加入1﹕250稀释的鼠/兔IgG,另一个加入1﹕250稀释的鼠/兔标签抗体,在旋转摇床上孵育30 min;对磁珠进行清洗,将得到的两管细胞裂解上清液分别加入两管中,在旋转摇床上孵育1 h;继续清洗磁珠3次,然后分别加入60 µL PBS和12 µL的5×蛋白上样缓冲液,同时在总蛋白和上清液样品中也加入5×蛋白上样缓冲液,经沸水煮10 min;瞬时离心后,再次放到磁力架上,将煮好的蛋白溶液,转移至新的1.5 mL EP管,-20℃保存备用。1.8 Western blot分析
制备12% SDS-PAGE胶,将上述制备好的蛋白点入孔中,以10 mA恒定电流进行蛋白浓缩,等样品跑出浓缩胶进入分离胶后将电流调至恒流15 mA;根据蛋白大小,适时终止电泳,将凝胶中的蛋白通过转膜仪转至PVDF膜上;随后将PVDF膜用封闭液(含5%脱脂奶粉的TBST溶液)于37℃封闭1.5 h以上;一抗孵育,用对应鼠源Flag/HA抗体(用封闭液1﹕3 000稀释),于37℃恒温培养进行孵育1 h;PVDF膜用TBST溶液在摇床上摇动洗涤5 min×5次;二抗孵育,向每个孔中加入HRP标记的山羊抗小鼠IgG(H+L)(用封闭液1﹕ 5 000稀释),于37℃恒温培养进行孵育1 h;PVDF膜再次用TBST溶液摇动洗涤5 min×5次;最后使用ECL显色液显影并通过蛋白曝光仪观察试验结果。2 结果
2.1 RNPS1和9G8克隆及氨基酸序列分析
成功克隆了家蚕RNPS1和9G8的CDS序列,其中RNPS1(GenBank登录号:NP_001040150.1)的开放阅读框(open reading frame,ORF)为882 bp,编码293个氨基酸,预测的蛋白分子量为32.71 kD,等电点为11.76。9G8(GenBank登录号:NP_001091768.1)的开放阅读框为531 bp,编码176个氨基酸,预测的蛋白分子量为20.17 kD,等电点为11.42。同时,分别对RNPS1和9G8的蛋白结构域进行预测,分析发现这两个基因分别在149—223、8—76氨基酸处含有一个RNA识别基序(RNA recognition motif,RRM),并且RNPS1和9G8都具有一个富含丝氨酸/精氨酸(S/R)重复序列的RS结构域(图1)。此外,9G8在103—119氨基酸处还含有一个锌指结构域(zinc finger domain)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1RNPS1(A)和9G8(B)的核苷酸序列及其编码的氨基酸序列(黄色部分为RRM结构域,绿色部分为RS结构域)
-->Fig. 1The nucleotide sequence and deduced amino acid sequence of RNPS1 (A) and 9G8 (B)( The RRM domain was shown in yellow background, the RS domain was shown in green background)
-->
2.2 家蚕RNPS1和9G8的同源序列比对及系统进化分析
分别选取了人(Homo sapiens)、小鼠(Mus musculus)、非洲爪蟾(Xenopus tropicalis)、斑马鱼(Danio rerio)和棉红铃虫(Pectinophora gossypiella)等进行同源氨基酸序列比对。结果表明,家蚕RNPS1和9G8在进化中高度保守,与其他物种中的同源基因在RRM结构域的氨基酸序列相似性接近80%(图2)。同时,9G8在锌指结构域的氨基酸序列相似性也高达80%(图3)。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2家蚕与其他物种RNPS1的多序列比对(红色线条区域的序列表示保守的RRM结构域;蓝色背景区表示在所有比对物种中一致的氨基酸残基;绿色背景区表示在75%或以上的比对物种中一致的氨基酸残基。
-->Fig. 2Multiple sequence alignment of RNPS1 from B. mori and other species(家蚕Bombyx mori(NP_001040150.1);人Homo sapiens(AAL56665.1);小鼠Mus musculus(AAH02061.1);非洲爪蟾Xenopus tropicalis(AAI18692.1);斑马鱼Danio rerio(AAH65953.1);黑腹果蝇Drosophila melanogaster(NP_649903.1);金凤蝶Papilio machaon(XP_014367106.1);柑橘凤蝶Papilio xuthus(KPI94450.1);棉红铃虫Pectinophora gossypiella(JAT86686.1);豆荚盲蝽Lygus hesperus(JAG18174.1))
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3家蚕与其他物种9G8的多序列比对(紫色线条区域的序列表示保守的锌指结构域Sequences in the purple line area indicated the zinc finger domain家蚕Bombyx mori(NP_001091768.1);柑橘凤蝶Papilio xuthus(NP_001299268.1);媒斑蚊Culex tarsalis(JAV19565.1);致倦库蚊Culex quinquefasciatus(XP_001863306.1);人Homo sapiens(AAA35495.1);小鼠Mus musculus(AAH25529.1);非洲爪蟾Xenopus tropicalis(NP_001007487.1);黑腹果蝇Drosophila melanogaster(AAF43414.1);斑点木蝶Pararge aegeria(JAA87011.1))
-->Fig. 3Multiple sequence alignment of 9G8 from B. mori and other species
-->
为了进一步分析家蚕RNPS1和9G8同其他物种在进化上的亲缘关系,通过同源检索获得多个物种的RNPS1和9G8氨基酸序列,并利用软件Clustalx 1.83和MEGA 6.0构建系统进化树。结果表明,系统进化树分为脊椎动物和无脊椎动物两大支,而家蚕RNPS1聚类于无脊椎动物一支,与同为鳞翅目昆虫的斑点木蝶(Pararge aegeria)、黑脉金斑蝶(Danaus plexippus)等亲缘关系较近(图4)。9G8也聚类于无脊椎动物一支,与同为鳞翅目昆虫的黑脉金斑蝶、金凤蝶(Papilio machaon)等关系较近(图5)。以上结果表明,家蚕RNPS1和9G8与鳞翅目昆虫中的同源基因都具有高度保守的RNA识别基序和RS结构域,且都与鳞翅目昆虫聚为一类。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4家蚕RNPS1蛋白与其他同源蛋白的进化树
-->Fig. 4Unrooted phylogenetic tree of RNPS1 in B. mori and other homologous
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5家蚕9G8蛋白与其他同源蛋白的进化树
-->Fig. 5Unrooted phylogenetic tree of 9G8 in B. mori and other homologous
-->
2.3 荧光共定位分析CDK11A、CDK11B与RNPS1和9G8的相互作用
构建了融合HA标签的pIZ-RNPS1-OE、pIZ-9G8-OE真核过表达载体以及融合Flag标签的pIZ-CDK11A- OE、pIZ-CDK11B-OE真核过表达载体。转染家蚕BmN-SWU1细胞系,转染48 h后进行免疫荧光分析,同时以DAPI作为细胞核染料。激光共聚焦结果显示,RNPS1分别与CDK11A和CDK11B共定位于细胞核中,且具有点状共聚集现象;同时发现,RNPS1还具有胞质定位,点状聚集于核外周;这主要由于RNPS1属于核质穿梭蛋白,需要同时在核和胞质中行使功能(图6-A)。9G8分别与CDK11A、CDK11B在细胞核中也具有共定位现象(图6-B)。以上结果表明,CDK11A和CDK11B可分别与RNPS1和9G8相互作用。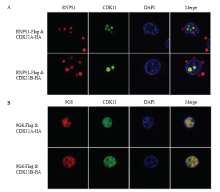 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6荧光共定位分析CDK11A、CDK11B与RNPS1和9G8的相互作用,DAPI染细胞核
-->Fig. 6Immunofluorescence analysis of the co-localization of CDK11A and CDK11B with RNPS1 and 9G8, nuclei were stained with DAPI
-->
2.4 免疫共沉淀分析CDK11A、CDK11B与RNPS1和9G8的相互作用
分别将RNPS1-Flag与CDK11A-HA,RNPS1-Flag与CDK11B-HA,9G8-Flag与CDK11A-HA以及9G8-Flag与CDK11B-HA,在家蚕细胞系BmN-SWU1中共同转染,72 h后进行免疫共沉淀试验。首先,分别用Flag抗体去沉淀RNPS1-Flag、9G8-Flag,随后用HA抗体进行Western blot分别检测CDK11A-HA、CDK11B-HA,观察CDK11A和CDK11B是否能被RNPS1和9G8沉淀下来;然后,进行反向验证,用HA抗体分别沉淀CDK11A-HA和CDK11B-HA,再用Flag抗体进行Western blot分析RNPS1和9G8能否被CDK11沉淀下来。结果显示,在所有的总蛋白和细胞裂解离心后的上清液样品中,均能检测到对应目的条带的表达,表明共转染的细胞均能正常表达目的蛋白,并且蛋白溶于上清。同时,在所有的共沉淀条带中,均能检测到对应目的条带,表明CDK11A、CDK11B均与RNPS1和9G8具有相互作用(图7)。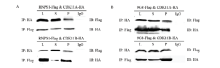 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7免疫共沉淀和Western blot分析CDK11A、CDK11B与RNPS1和9G8的相互作用(L:细胞裂解液Cell lysate;S:上清Supernatant;P:免疫沉淀Immunoprecipitation;IgG:对照Control)
-->Fig. 7Co-immunoprecipitation (Co-IP) and Western blot analysis of the interaction of CDK11A and CDK11B with RNPS1 and 9G8
-->
3 讨论
CDK11是由p34cdc2(CDC2L1和CDC2L2)编码的一类重要的细胞周期调控蛋白,能够形成不同的亚型,主要包括CDK11P110、CDK11P58和CDK11P46[1]。不同亚型行使不同的功能,CDK11P110能够与cyclinL相互作用参与转录调控、pre-RNA剪接和自噬[4-5,13,20-21]。CDK11P58在G2/M期特定表达,能够与cyclinD3相互作用,参与有丝分裂过程中中心体的成熟、纺锤体的形成和姐妹染色单体结合[7-9]。在细胞凋亡时,由CDK11P110和CDK11P58切割产生第3种异构体CDK11P46,CDK11P46异常表达将会诱导凋亡[21]。小鼠中敲除CDK11(CDK11P110和CDK11P58均被敲除)后,胚泡发育的过程中出现增殖缺陷和早期胚胎死亡,暗示了CDK11在细胞增殖和转录调控等过程具有必不可少的作用[22]。在人类中,CDK11p110是研究最广泛的CDK11剪切体,报道最多的是其参与转录与mRNA剪接。而笔者课题组之前的研究已经克隆并鉴定了家蚕CDK11的两种剪切体CDK11A和CDK11B[18],在本研究中也发现CDK11A和CDK11B与剪接因子RNPS1和9G8具有相互作用,因此推测CDK11可能参与pre-RNA的剪接成熟过程。据报道,CDK11P110能够与RNA聚合酶II、转录延伸因子和pre-RNA结合形成大分子聚合物来参与转录和pre-RNA剪接等生物过程[23-24]。进一步研究表明,CDK11p110定位于两个剪接因子隔室(splicing factor compartments,SFC)和核质,与其相互作用的剪接因子包括RNPS1和9G8,其参与了pre-RNA的剪接成熟过程[23,25]。RNPS1属于SR蛋白家族一员,作为剪接的普遍活化剂,能够以底物特异性方式促进可选择剪接[10,26]。RNPS1也能通过磷酸化反应影响pre-RNA剪接和mRNA翻译[27]。9G8是一个核质穿梭蛋白,能够促进mRNA的核质输出[3]。9G8的磷酸化状态对于其剪接和导出功能至关重要,9G8能够以高磷酸化形式募集到mRNA,而在剪接期间变为低磷酸化[3,12]。SR蛋白能够通过其RS结构域或特有的结构域实现与pre-RNA特异性序列或者其他剪接因子的相互作用,协同完成剪切位点的正确选择,并促进剪切体的形成[14,28-30]。基于这些已有研究,笔者推测在家蚕中蛋白激酶CDK11能够磷酸化剪接因子RNPS1和9G8,进而参与pre-RNA的剪接过程,但具体机制仍需深入研究。许多重要基因的表达需要通过RNA选择性剪接进行调控,同时很多疾病的产生是由于RNA剪接发生紊乱导致的[2-3,31]。虽然目前有关RNA剪接的研究有很多,但具体调控机制仍不清楚,下一步拟通过磷酸化检测以及基因过表达与敲除等策略进行深入研究;也可通过基因工程的手段改变SR蛋白或它的作用底物,以进行更多方面的应用研究。
4 结论
克隆并鉴定了两个剪切因子RNPS1和9G8,并且通过同源序列比对和系统进化分析显示,家蚕RNPS1和9G8具有典型的RRM结构域以及RS结构域,属于SR蛋白家族,与其他鳞翅目昆虫具有较高的同源性。通过免疫荧光和免疫共沉淀均证明CDK11A和CDK11B能够与剪切因子RNPS1和9G8相互作用。The authors have declared that no competing interests exist.
