0 引言
【研究意义】面粉颜色是评价小麦品质的重要指标[1]。小麦籽粒中多酚氧化酶(polyphenol Oxidase,PPO)、过氧化物酶(peroxidase,POD)和脂肪氧化酶(lipoxidase,LOX)等是面粉和面制品在加工、储藏过程中被漂白和发生褐变的主要原因[2-3]。POD具有与LOX类似的对胡萝卜素等色素类物质的漂白作用,并可以作为主要的天然漂白剂与LOX同时加入到面粉中,使面团得以漂白,因此,高POD活性的小麦面粉白度更高[4]。另外,籽粒中的POD能催化阿魏酸等主要酚酸的氧化,并产生发色基团(如醌式结构),从而使面制品在制作和贮存过程中发生褐变[5-7]。目前,小麦籽粒LOX和PPO活性与颜色相关的指标在育种实践中已受到重视,但对POD与颜色相关性的研究较少[8]。鉴于POD对面制品色泽存在双重作用,深入开展普通小麦POD研究,对明确面制品颜色形成机制、影响因素及其改良策略具有重要意义。【前人研究进展】ŽILIĆ等[9]研究表明,普通小麦POD活性显著高于硬粒小麦(P<0.05)。在普通小麦的不同品种间POD活性可相差3—10倍[10]。因此,通过遗传途径改良POD活性是可行的。WEI等[11]利用豆麦/石4185重组自交系(recombinant inbred lines,RIL)群体的214个株系和7 391个SNP标记及一个新开发的STS标记对普通小麦POD活性进行了QTL分析,共检测到3个QTL QPod.caas-3AL、QPod.caas-4BS和QPod.caas-5AS,它们在不同环境下分别解释5.3%—9.2%、9.3%—21.2%和5.8%—11.7%的表型变异。连锁分析理论上可以应用于所有性状的遗传解析,但受遗传群体亲本的差异度和群体大小等限制,定位QTL数目有限[12]。而关联作图则是一种能有效克服连锁作图局限的方法,连锁作图和关联作图相结合可以互为补充互为验证[13]。近年来,关联分析已被广泛应用于各种作物复杂农艺性状QTL的发掘。杨胜先等[14]、赖勇等[15]和张焕欣等[16]分别对大豆、大麦和玉米等作物进行了关联分析研究。而在小麦育种工作中,陈广凤等[17]利用24 355个SNP标记对205份中国冬小麦品种进行关联分析,共检测到38个与株高相关的SNP,其中11个位点在2个以上环境稳定表达。SUN等[18]利用90 K芯片对163份黄淮麦区冬小麦品种进行了产量相关性状GWAS,并发现了41个产量相关性状的QTL位点。DONG等[19]利用90K芯片对166份黄淮麦区冬小麦品种进行了碳水化合物进行GWAS,并发现了23个产量相关性状的QTL位点。【本研究切入点】虽然WEI等[11]用RIL群体对POD活性进行了全基因组连锁分析,并发现了3个POD活性位点,但迄今为止尚未有POD活性GWAS的报道。【拟解决的关键问题】本研究以151份黄淮麦区和82份北部冬麦区小麦品种(系)2个自然群体为材料,利用Wheat 90K iSelect SNP芯片,对小麦籽粒POD活性进行GWAS分析,以发掘新的POD活性位点,解析小麦POD遗传机制,为小麦POD活性的遗传改良提供可用分子标记。1 材料与方法
1.1 供试材料
供试材料包括233份冬小麦品种(系),其中151份来自黄淮冬麦区(Yellow and Huai River Valley Facultative Wheat Region, YHRVWWR),于2012— 2013和2013—2014年度种植于河南安阳和安徽濉溪;82份材料来自于北部冬麦区(Northern Winter Wheat Region,NWWR),于2012—2013和2013—2014年度种植于北京顺义和河北石家庄。试验采用随机区组设计,3次重复,行长2 m,行距25 cm。上述试验材料均由中国农业科学院作物科学研究所小麦品质课题组提供。1.2 POD活性检测
以25 μL的H2O2、5 μL 2%的愈创木酚和145 μL 0.05 mol·L-1的磷酸-柠檬酸缓冲液(pH 5.0)的混合液为底物,采用WEI等[13]的愈创木酚紫外分光光度法进行POD活性的检测,每个小麦品种的POD活性重复检测2次,2次检测结果相差超过10%的进行重复检测。1.3 表型数据统计
采用SAS v9.2的PROC UNIVARIATE、RROC CORR程序分别进行描述性统计变量分析、相关分析和方差分析。利用R 3.31程序进行遗传力计算。广义遗传力计算公式:h2 = σg2 / (σg2 + σge2/r +σε2 / re),其中σg2、σge2和σε2分别表示基因型方差、基因型与环境互作方差和误差方差,e和r分别表示环境个数和每个环境内的重复次数。1.4 90 K SNP芯片基因型分型
基于Illumina测序平台,应用小麦90 K iSelectSNP芯片(81 587个SNP)对233份小麦品种(系)进行SNP分型,由博奥生物技术有限公司(http://www. capitalbio.com)完成。利用Genome Studio软件(http://www.illumina.com)进行SNP分型,具体操作参照CAVANAGH等[20]方法。人工对分型结果进行质量控制,剔除数据缺失率>50%、杂合率>50%和最小等位基因频率(minor allele frequency,MAF)<0.05的SNP标记,保留高质量的SNP标记进行关联分析。使用Power Marker v3.25进行MAF和多态性信息含量(polymorphism information content,PIC)的运算[21]。
1.5 群体结构分析
关联分析前进行群体结构分析可有效降低结果中假阳性概率。采用Structure v2.3.4的Admixture Ancestry模型分析2个自然群体的群体结构。1.6 全基因组关联分析
采用Tassel v5.0对2个自然群体进行kinship matrix(K matrix)运算。运用Tassel v5.0中的混合线性模型(mixed linear model,MLM),在考虑群体结构和亲缘关系的情况下,进行SNP标记与POD活性的关联分析。关联分析结果中多个SNP标记是否位于同一位点由LD衰减距离决定。在P<0.001水平进行SNP标记和籽粒POD活性显著性检测。利用R语言对关联分析结果绘制Manhattan图和quantile-quantile(Q-Q)图。1.7 候选基因筛选
以与POD活性显著关联的SNP标记序列为探针,在NCBI(http://www.ncbi.nlm.nih.gov/;National center for biotechnology information)和ENA(European Nucleotide Archive;http://www.ebi.ac.uk/ena)数据库中进行BLASTx,筛选与POD活性相关的候选基因。e-值<10-5,序列一致性大于75%。2 结果
2.1 POD活性分析
151份黄淮麦区冬小麦材料在安阳2013、濉溪2013、安阳2014、濉溪2014及均值环境下的POD活性分别为507.0、513.8、781.4、和727.5 U·min-1g-1,变异系数为15.4%—21.8%。各环境之间的POD活性呈极显著正相关,相关系数为0.46—0.89(P<0.0001),遗传力为0.79。83份北部冬麦区材料在北京2013、石家庄2013、北京2014、石家庄2014及均值环境下POD活性分别为591.6、567.5、899.5、858.9和729.4 U·min-1g-1,变异系数为15.0%—19.9%。各环境之间POD活性呈极显著正相关,相关系数为0.50—0.87(P<0.0001),遗传力为0.82(表1)。以上结果表明中国冬小麦品种(系)的POD活性主要受遗传因素控制,在早期世代对其进行选择是有效的;且籽粒POD活性变异范围广,具有较大的选择潜力。通过育种途径选育高POD活性的品种,进而改良小麦面制品颜色是可行的。Table 1
表1
表1233份品种中POD含量统计分析
Table 1Statistical analysis of POD content of 233 wheat cultivars
| 环境 Environment | 平均值 Mean | 标准差 SD | 变异系数 CV (%) | 相关系数Correlation coefficient | 遗传力h2 | |||
|---|---|---|---|---|---|---|---|---|
| 安阳(2013) Anyang (2013) | 濉溪(2013) Suixi (2013) | 安阳(2014) Anyang (2014) | 濉溪(2014) Suixi (2014) | |||||
| 安阳(2013) Anyang (2013) | 507.03 | 110.64 | 21.8 | 0.79 | ||||
| 濉溪(2013) Suixi (2013) | 513.82 | 111.94 | 21.8 | 0.46*** | ||||
| 安阳(2014) Anyang (2014) | 781.35 | 129.89 | 16.6 | 0.49*** | 0.59*** | |||
| 濉溪(2014) Suixi (2014) | 894.16 | 154.29 | 17.3 | 0.6*** | 0.61*** | 0.65*** | ||
| 平均值 Average | 727.5 | 112.27 | 15.4 | 0.76*** | 0.80*** | 0.84*** | 0.89*** | |
| 北京(2013) Beijing (2013) | 石家庄(2013)Shijiazhuang (2013) | 北京(2014) Beijing (2014) | 石家庄(2014) Shijiazhuang (2014) | |||||
| 北京(2013) Beijing (2013) | 591.63 | 110.48 | 18.7 | 0.82 | ||||
| 石家庄(2013)Shijiazhuang (2013) | 567.45 | 113.01 | 19.9 | 0.50*** | ||||
| 北京(2014) Beijing (2014) | 899.49 | 166.06 | 18.5 | 0.58*** | 0.55*** | |||
| 石家庄(2014)Shijiazhuang (2014) | 858.88 | 137.33 | 16.0 | 0.56*** | 0.62*** | 0.63*** | ||
| 平均值 Average | 729.36 | 109.26 | 15.0 | 0.78*** | 0.79*** | 0.87*** | 0.86*** | |
新窗口打开
2.2 SNP标记的多态性及群体结构分析
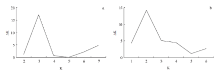
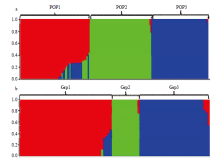
利用90 K SNP芯片对151份黄淮麦区及82份北部冬麦区品种(系)进行检测。其中黄淮麦区最终采用18 189个SNP标记进行GWAS分析,平均单条染色体包含866个标记,A、B和D组染色体分别包含1 007、1 338和254个标记;北部冬麦区品种GWAS分析选用18 417个标记,平均每条染色体含有877个标记,A、B和D组染色体平均包含1 005、1 357和269个SNP标记。在2个自然群体中,SNP标记图谱长为3 700 cM,每个标记之间的平均遗传距离为0.2 cM。在2个群体所选标记的A、B和D组染色体中,均表现为A与B组染色体标记密度显著高于D组染色体(表2)。2个自然群体SNP标记PIC值为0.09—0.38,MAF为0.05—0.50。利用Structure v 2.3.4分别对2个自然群体进行遗传结构分析。当ΔK=3时,2个自然群体的K值均达到最大值(图1)。因此,2个群体均可分为3个亚群(图2)。在黄淮麦区中,第一亚群(记为Pop1)含有57份品种,以山东品种(43.0%)为主,还包括陕西、河南及18份国外品种;第二亚群(记为Pop2)共含有48份品种,以河南品种(37.5%)为主,还包括部分陕西和安徽品种;第三亚群(记为Pop3)中含有46份品种,以河南品种(56.5%)为主。北部冬麦区自然群体的第一亚群(记为Grp1)含有39份品种,主要由国外品种(66.6%)组成,还包括8份北京和5份山西品种;第二亚群(记为Grp2)共有13份国内品种,主要由北京品种(84.6%)组成;第三亚群(记为Grp3)共有30份品种,以国外品种(46.6%)为主,还包含12份北京品种、1份河北品种、1份山西品种和1份宁夏品种。
Table 2
表2
表2黄淮麦区与北部冬麦区群体关联分析所用标记统计
Table 2Statistical analysis of SNP markers for analysis of wheat cultivars from Yellow & Huai Winter Wheat Region (YHRVWWR) and Northern China Plain Winter Wheat Region (NWWR)
| 染色体 Chromosome | 黄淮麦区YHRVWWR | 北部冬麦区NWWR | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 标记数量 No. of markers | 最小等位基因频率MAF | 多态信息含量PIC | 标记数量 No. of markers | 最小等位基因频率MAF | 多态信息含量PIC | |||||
| 平均 Mean | 范围 Range | 平均 Mean | 范围 Range | 平均 Mean | 范围 Range | 平均 Mean | 范围 Range | |||
| 1A | 1176 | 0.24 | 0.05-0.50 | 0.27 | 0.09-0.38 | 1053 | 0.27 | 0.06-0.50 | 0.29 | 0.10-0.38 |
| 1B | 1823 | 0.27 | 0.05-0.50 | 0.32 | 0.09-0.38 | 1871 | 0.23 | 0.05-0.50 | 0.27 | 0.08-0.38 |
| 1D | 463 | 0.27 | 0.05-0.50 | 0.25 | 0.09-0.37 | 454 | 0.25 | 0.06-0.47 | 0.27 | 0.10-0.38 |
| 2A | 1049 | 0.26 | 0.05-0.50 | 0.28 | 0.09-0.38 | 1068 | 0.23 | 0.06-0.50 | 0.26 | 0.10-0.38 |
| 2B | 1439 | 0.27 | 0.05-0.50 | 0.29 | 0.09-0.38 | 1458 | 0.29 | 0.06-0.50 | 0.30 | 0.10-0.38 |
| 2D | 552 | 0.26 | 0.05-0.50 | 0.31 | 0.09-0.38 | 611 | 0.38 | 0.05-0.50 | 0.29 | 0.10-0.38 |
| 3A | 874 | 0.26 | 0.05-0.50 | 0.28 | 0.09-0.38 | 955 | 0.25 | 0.05-0.50 | 0.27 | 0.09-0.38 |
| 3B | 1192 | 0.31 | 0.05-0.50 | 0.28 | 0.09-0.38 | 1217 | 0.26 | 0.05-0.50 | 0.29 | 0.09-0.38 |
| 3D | 212 | 0.18 | 0.05-0.47 | 0.25 | 0.09-0.37 | 249 | 0.27 | 0.05-0.49 | 0.29 | 0.08-0.38 |
| 4A | 737 | 0.28 | 0.05-0.50 | 0.28 | 0.09-0.38 | 779 | 0.27 | 0.05-0.50 | 0.29 | 0.09-0.38 |
| 4B | 718 | 0.24 | 0.05-0.50 | 0.28 | 0.09-0.38 | 743 | 0.26 | 0.06-0.50 | 0.29 | 0.10-0.38 |
| 4D | 49 | 0.30 | 0.05-0.50 | 0.26 | 0.09-0.37 | 63 | 0.29 | 0.06-0.48 | 0.30 | 0.10-0.38 |
| 5A | 1007 | 0.26 | 0.05-0.50 | 0.31 | 0.09-0.38 | 1011 | 0.25 | 0.05-0.50 | 0.28 | 0.09-0.38 |
| 5B | 1790 | 0.29 | 0.05-0.50 | 0.31 | 0.09-0.38 | 1778 | 0.24 | 0.06-0.50 | 0.26 | 0.10-0.38 |
| 5D | 167 | 0.18 | 0.05-0.47 | 0.29 | 0.09-0.37 | 169 | 0.24 | 0.06-0.50 | 0.25 | 0.10-0.38 |
| 6A | 1083 | 0.32 | 0.05-0.50 | 0.27 | 0.09-0.38 | 1093 | 0.30 | 0.06-0.50 | 0.29 | 0.10-0.38 |
| 6B | 1314 | 0.21 | 0.05-0.50 | 0.28 | 0.09-0.38 | 1314 | 0.26 | 0.06-0.50 | 0.28 | 0.10-0.38 |
| 6D | 166 | 0.25 | 0.05-0.48 | 0.28 | 0.10-0.37 | 166 | 0.24 | 0.06-0.49 | 0.26 | 0.10-0.38 |
| 7A | 1123 | 0.27 | 0.05-0.50 | 0.28 | 0.10-0.38 | 1076 | 0.29 | 0.06-0.50 | 0.29 | 0.10-0.38 |
| 7B | 1089 | 0.25 | 0.05-0.50 | 0.28 | 0.09-0.38 | 1121 | 0.28 | 0.05-0.50 | 0.29 | 0.08-0.38 |
| 7D | 166 | 0.17 | 0.05-0.47 | 0.25 | 0.09-0.37 | 168 | 0.22 | 0.06-0.47 | 0.24 | 0.10-0.38 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1使用deltak值估计黄淮冬麦区(a)和北部冬麦区(b)品种亚群数
-->Fig. 1Estimation of number of sub-populations (K) in Yellow & Huai Facultative Winter Wheat Region (a) and Northern China Plain Winter Wheat Region (b)
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2黄淮冬麦区(a)和北部冬麦区(b)品种的群体结构分析
-->Fig. 2Population structure analysis of 151 cultivars from Yellow & Huai Winter Wheat Region (a) and Northern China Plain Winter Wheat Region (b)
-->
2.3 POD活性全基因组关联分析
对黄淮麦区151份品种(系)及北部冬麦区82份品种(系)的籽粒POD活性进行全基因组关联分析。当P≤0.001时,认为该标记与POD活性显著关联,多个环境检测到的标记视为稳定遗传标记(图3)。由Q-Q图可以看出,2个自然群体的群体结构控制较好,可以避免假阳性的出现(图4)。黄淮麦区151份品种在A、B、D和全基因组水平下的LD衰减距离分别为5、7、11和7 cM,北部麦区82份品种在A、B、D和全基因组水平下的LD衰减距离分别为6、7、10和8 cM。GWAS结果中多个标记是否位于同一位点(Locus)由对应基因组的LD衰减距离决定。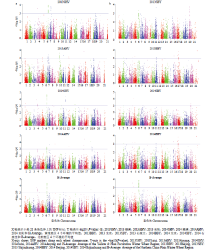 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3POD活性全基因组关联分析
-->Fig. 3Genome-wide association of POD activity with mixed linear model
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4黄淮冬麦区(a)和北部冬麦区(b)品种POD活性全基因组关联分析的Q-Q图
-->Fig. 4Quantile-quantile plot for POD activity of Yellow & Huai Facultative Winter Wheat Region (a) and Northern China Plain Winter Wheat Region (b)
-->
在黄淮麦区共检测到20个位点,包含86个与POD活性显著关联的SNP标记(P<0.001),分别位于1A、2A、2B、2D、3A、3B、3D、4A、4B、5A、5B、6A、6D和7A染色体上,单个位点可解释7.8%—13.7%的表型变异,其中4个位点至少在2个环境中稳定遗传,分布在2A(177 cM)、2D(97—103 cM)、5A(80 cM)和6A(7 cM)染色体上。在北部冬麦区群体中筛选到20个位点,包含51个与POD活性显著关联的SNP标记(P<0.001),分布在1A、1B、1D、2A、2B、2D、3A、3B、4B、6A、6B、7A、7B和7D染色体上,单个位点可解释14.4%—23.2%的表型变异,其中4个位点至少在2个环境中稳定遗传,分别位于2A(177 cM)、2D(97—99 cM)、7B(98—99 cM)和7D(182—184 cM)染色体上。在发现的所有POD活性相关位点中,2A(177cM,GENE-1003_357)和2D(99 cM,wsnp_Ex_c9619_15913632)2个位点在2个自然群体中均能检测到,且在多个环境下稳定存在(表3)。
Table 3
表3
表3黄淮冬麦区与北部冬麦区品种中与POD活性显著关联的SNP标记
Table 3Marker-trait association for POD activity in Yellow & Huai winter wheat region and Northern China Plain Winter Wheat Region
| 标记a Marker | 染色体b Chromosome | 位置c Position (cM) | MLM | 环境d ENV | ||
|---|---|---|---|---|---|---|
| P值P_value | R2(%) | |||||
| 黄淮冬麦区 YHRVWWR | ||||||
| 1 | RAC875_c39125_365 | 1A | 70 | 5.2E-04 | 8.9 | One |
| 2 | BobWhite_c48481_81 | 2A | 66 | 3.8E-04 | 8.5 | One |
| 3 | BS00073382_51 | 2A | 66 | 6.2E-04 | 8.5 | One |
| 4 | Excalibur_c15740_1106 | 2A | 66 | 5.3E-04 | 7.9 | One |
| 5 | Excalibur_rep_c67994_169 | 2A | 66 | 5.6E-04 | 7.9 | One |
| 6 | GENE-0749_215 | 2A | 66 | 2.9E-04 | 8.1 | One |
| 7 | IACX8060 | 2A | 66 | 5.6E-04 | 7.9 | One |
| 8 | RAC875_rep_c78518_198 | 2A | 66 | 5.6E-04 | 7.9 | One |
| 9 | GENE-1003_357* | 2A | 177 | 1.4E-04-3.0E-04 | 10.1-10.9 | Two |
| 10 | BS00086322_51 | 2B | 109 | 9.5E-04 | 7.8 | One |
| 11 | BobWhite_rep_c64049_232 | 2B | 109 | 9.5E-04 | 7.8 | One |
| 12 | CAP8_c2833_325 | 2D | 103 | 3.0E-05-8.6E-04 | 8.2-13.1 | Three |
| 13 | D_contig03185_174 | 2D | 101 | 8.4E-05-1.9E-04 | 10.4-11.5 | Two |
| 14 | D_contig13570_307 | 2D | 103 | 3.9E-05-6.8E-04 | 8.3-12.3 | Two |
| 15 | Excalibur_c2311_1116 | 2D | 97 | 4.3E-05-8.7E-04 | 8.8-12.8 | Three |
| 16 | Excalibur_c2311_1190 | 2D | 97 | 1.1E-04-2.5E-04 | 9.8-10.9 | Two |
| 17 | Excalibur_c2311_2260 | 2D | 97 | 4.0E-05-4.7E-04 | 9.0-12.3 | Two |
| 18 | Excalibur_c5193_2213 | 2D | 103 | 2.7E-05-8.6E-04 | 8.2-13.2 | Three |
| 19 | Excalibur_c4167_231 | 2D | 103 | 3.4E-05-9.8E-04 | 7.8-12.7 | Three |
| 20 | Excalibur_c7366_1475 | 2D | 97 | 2.5E-05-8.0E-04 | 8.5-13.4 | Three |
| 21 | Excalibur_c66045_388 | 2D | 103 | 3.3E-05-6.7E-04 | 8.4-12.9 | Two |
| 22 | Excalibur_c9619_1136 | 2D | 101 | 4.0E-05-4.7E-04 | 9.0-12.3 | Two |
| 23 | GENE-1304_646 | 2D | 99 | 3.5E-04-4.3E-04 | 9.3-9.4 | Two |
| 24 | GENE-0762_74 | 2D | 101 | 8.6E-05-2.0E-04 | 10.4-11.5 | Two |
| 25 | IAAV790 | 2D | 97 | 1.9E-04 | 8.4 | One |
| 26 | IAAV9128 | 2D | 103 | 3.8E-05-6.9E-04 | 8.3-12.5 | Two |
| 27 | Jagger_c7882_154 | 2D | 103 | 3.0E-05-6.5E-04 | 9.0-13.6 | Three |
| 28 | Kukri_c17_943 | 2D | 103 | 2.9E-05-7.8E-04 | 8.3-13.3 | Three |
| 29 | Kukri_c16094_496 | 2D | 103 | 3.9E-05~9.9E-04 | 7.8-12.8 | Three |
| 30 | Kukri_c5276_447 | 2D | 97 | 4.2E-05-9.7E-04 | 8.5-12.8 | Three |
| 31 | Kukri_c38203_354 | 2D | 103 | 3.7E-05-9.7E-04 | 8.0-12.5 | Two |
| 32 | RAC875_c22873_1129 | 2D | 97 | 7.2E-05-1.5E-04 | 10.6-11.7 | Two |
| 33 | RAC875_c15171_858 | 2D | 97 | 1.1E-04-2.0E-04 | 10.3-11.1 | Two |
| 34 | RAC875_c35807_595 | 2D | 101 | 8.4E-05-1.9E-04 | 10.4-11.5 | Two |
| 35 | RAC875_c4851_1600 | 2D | 103 | 3.8E-05-6.9E-04 | 8.3-12.5 | Two |
| 36 | RAC875_c64504_417 | 2D | 99 | 6.0E-04-9.0E-04 | 8.1-8.7 | Two |
| 37 | RAC875_c5998_1056 | 2D | 103 | 2.9E-05-8.7E-04 | 8.1-13.2 | Three |
| 标记a Marker | 染色体b Chromosome | 位置c Position (cM) | MLM | 环境d ENV | ||
| P值P_value | R2(%) | |||||
| 38 | RAC875_c67940_296 | 2D | 99 | 4.0E-05-4.7E-04 | 9.0-12.3 | Two |
| 39 | RAC875_rep_c101664_297 | 2D | 101 | 1.2E-04-2.2E-04 | 10.0-10.8 | Two |
| 40 | TA002335-0425 | 2D | 97 | 2.3E-05-7.9E-04 | 8.6-13.7 | Three |
| 41 | RAC875_rep_c77646_102 | 2D | 97 | 2.5E-05-8.0E-04 | 8.5-13.4 | Three |
| 42 | tplb0042o21_938 | 2D | 99 | 4.1E-05-4.7E-04 | 9.1-12.4 | Two |
| 43 | tplb0030l01_1677 | 2D | 99 | 1.2E-04-2.2E-04 | 10.0-10.7 | Two |
| 44 | tplb0060e06_1267 | 2D | 99 | 1.2E-04-2.3E-04 | 9.8-10.7 | Two |
| 45 | wsnp_Ex_c9619_15913632* | 2D | 99 | 1.6E-04-3.5E-04 | 9.6-10.5 | Two |
| 46 | Excalibur_c25043_618 | 2D | 97 | 4.9E-04 | 9.5 | One |
| 47 | wsnp_CAP11_rep_c4157_1965583* | 3A | 34 | 6.3E-04 | 8.6 | One |
| 48 | IACX2831 | 3A | 34 | 8.6E-04 | 8.1 | One |
| 49 | BobWhite_c35789_281 | 3A | 85 | 4.3E-04 | 9.3 | One |
| 50 | BS00022029_51 | 3A | 86 | 6.6E-04 | 8.3 | One |
| 51 | Ku_c17569_905 | 3A | 87 | 9.2E-04 | 8 | One |
| 52 | wsnp_Ex_c3478_6369892 | 3A | 87 | 9.5E-04 | 8 | One |
| 53 | wsnp_Ex_c12341_19693090 | 3A | 123 | 2.5E-04 | 9.7 | One |
| 54 | BS00064472_51 | 3A | 129 | 5.0E-04 | 8.7 | One |
| 55 | Excalibur_c49743_97 | 3A | 130 | 2.0E-04 | 10.4 | One |
| 56 | Excalibur_c78654_184 | 3A | 130 | 2.2E-04 | 10.2 | One |
| 57 | Tdurum_contig10548_1605 | 3A | 130 | 2.4E-04 | 9.8 | One |
| 58 | Excalibur_rep_c110501_525 | 3A | 130 | 3.2E-04 | 9.4 | One |
| 59 | wsnp_Ex_c700_1379957 | 3B | 97 | 9.8E-04 | 7.8 | One |
| 60 | Excalibur_c47973_67 | 3D | 148 | 1.8E-04 | 10.2 | One |
| 61 | Excalibur_c5624_1331 | 4A | 87 | 6.4E-04 | 8.5 | One |
| 62 | Kukri_c29142_747 | 4A | 91 | 2.6E-04 | 9.8 | One |
| 63 | Kukri_rep_c106490_583 | 4A | 91 | 4.8E-04 | 8.8 | One |
| 64 | RAC875_c8472_1174 | 4A | 91 | 5.4E-04 | 8.9 | One |
| 65 | Excalibur_c5624_1026 | 4A | 91 | 7.2E-04 | 8.2 | One |
| 66 | Kukri_c34435_265 | 4A | 91 | 7.2E-04 | 8.2 | One |
| 67 | Excalibur_c5624_845 | 4A | 91 | 8.1E-04 | 8.1 | One |
| 68 | Kukri_c29142_473 | 4A | 91 | 8.3E-04 | 8.3 | One |
| 69 | wsnp_Ex_c2288_4293430 | 4A | 91 | 9.1E-04 | 8 | One |
| 70 | Kukri_c29142_450 | 4A | 91 | 9.3E-04 | 7.9 | One |
| 71 | Tdurum_contig33737_157 | 4B | 56 | 4.7E-05 | 12.6 | One |
| 72 | Excalibur_c36630_2194 | 4B | 56 | 1.4E-04 | 10.6 | One |
| 73 | BobWhite_rep_c49034_132 | 4B | 56 | 1.8E-04 | 10.2 | One |
| 74 | BS00021984_51 | 4B | 56 | 2.6E-04 | 9.7 | One |
| 标记a Marker | 染色体b Chromosome | 位置c Position (cM) | MLM | 环境d ENV | ||
| P值P_value | R2(%) | |||||
| 75 | Excalibur_c42450_727 | 4B | 67 | 7.5E-04 | 8.3 | One |
| 76 | BS00021969_51 | 4B | 89 | 5.0E-04 | 8.9 | One |
| 77 | Excalibur_c766_462 | 5A | 80 | 4.2E-04-7.7E-04 | 8.4-9.3 | Two |
| 78 | TA001999-0444 | 5B | 180 | 7.0E-04 | 8.6 | One |
| 79 | Excalibur_c23801_115 | 5B | 183 | 6.3E-04 | 8.5 | One |
| 80 | Tdurum_contig66604_927 | 5B | 183 | 6.5E-04 | 8.4 | One |
| 81 | Tdurum_contig27939_357 | 6A | 7 | 3.3E-05-1.6E-04 | 10.7-13.3 | Two |
| 82 | Kukri_c14679_1082 | 6A | 30 | 2.8E-04 | 10.2 | One |
| 83 | Excalibur_c4789_2748 | 6D | 9 | 8.0E-05 | 9.4 | One |
| 84 | BS00078460_51 | 7A | 83 | 5.3E-04 | 8.6 | One |
| 85 | IAAV1265 | 7A | 83 | 5.4E-04 | 9 | One |
| 86 | Ex_c9615_574 | 7A | 83 | 5.9E-04 | 8.5 | One |
| 北部冬麦区 NWWR | ||||||
| 1 | Ra_c54249_1031 | 1A | 71 | 9.20E-04 | 14.4 | One |
| 2 | BS00067201_51 | 1B | 61 | 3.00E-04 | 17.5 | One |
| 3 | Excalibur_c28681_966 | 1B | 63 | 8.90E-04 | 15.7 | One |
| 4 | BS00083060_51 | 1B | 98 | 6.50E-04 | 15.4 | One |
| 5 | BS00077498_51 | 1B | 122 | 9.50E-04 | 15.9 | One |
| 6 | D_GA8KES402HUUGV_172 | 1D | 48 | 6.50E-04 | 15.4 | One |
| 7 | GENE-1003_357* | 2A | 177 | 3.0E-04-7.4E-04 | 16.5-18.5 | Two |
| 8 | Excalibur_c13413_165 | 2B | 52 | 8.50E-05 | 15.3 | One |
| 9 | Kukri_c52384_196 | 2B | 97 | 6.30E-04 | 16.5 | One |
| 10 | Excalibur_c12675_1666 | 2D | 103 | 1.50E-04 | 14.7 | One |
| 11 | wsnp_Ex_c7366_12631101 | 2D | 97 | 3.6E-04-6.1E-04 | 15.5-16.9 | Two |
| 12 | wsnp_Ex_c9619_15913632* | 2D | 99 | 9.7E-04-9.7E-04 | 15.5-18.2 | Two |
| 13 | RAC875_c64504_417 | 2D | 99 | 4.70E-04 | 16.8 | One |
| 14 | Kukri_c23833_181 | 3A | 86 | 1.20E-04 | 20.3 | One |
| 15 | wsnp_Ex_c12354_19711297 | 3A | 86 | 1.20E-04 | 20.3 | One |
| 16 | BS00022029_51 | 3A | 86 | 2.60E-04 | 18.9 | One |
| 17 | RAC875_rep_c75557_177 | 3A | 124 | 9.00E-04 | 14.7 | One |
| 18 | RAC875_rep_c112642_422 | 3B | 67 | 9.00E-04 | 14.5 | One |
| 19 | IACX5989 | 4B | 62 | 9.10E-04 | 15.1 | One |
| 20 | BS00022466_51 | 4B | 87 | 2.30E-04 | 18.6 | One |
| 21 | IAAV8633 | 6A | 3 | 5.50E-04 | 16.7 | One |
| 22 | BS00055293_51 | 6B | 0 | 5.30E-04 | 16.6 | One |
| 23 | BS00003852_51 | 6B | 39 | 1.50E-04 | 21.3 | One |
| 标记a Marker | 染色体b Chromosome | 位置c Position (cM) | MLM | 环境d ENV | ||
| P值P_value | R2(%) | |||||
| 24 | GENE-0418_209 | 6B | 39 | 3.80E-04 | 17.9 | One |
| 25 | IACX6021 | 6B | 39 | 3.80E-04 | 17.9 | One |
| 26 | RAC875_c536_872 | 6B | 39 | 4.20E-04 | 18.2 | One |
| 27 | BS00090070_51 | 6B | 39 | 4.30E-04 | 17.8 | One |
| 28 | TA004901-0137 | 6B | 39 | 8.80E-04 | 15.2 | One |
| 29 | BS00011530_51 | 6B | 39 | 9.20E-04 | 15 | One |
| 30 | IACX6023 | 6B | 39 | 9.30E-04 | 15 | One |
| 31 | BS00003891_51 | 6B | 39 | 9.80E-04 | 14.8 | One |
| 32 | BS00011365_51 | 6B | 39 | 9.80E-04 | 14.8 | One |
| 33 | Kukri_c35255_1234 | 6B | 39 | 9.80E-04 | 14.8 | One |
| 34 | Kukri_rep_c104879_103 | 6B | 39 | 9.90E-04 | 15.2 | One |
| 35 | RAC875_c13920_836 | 6B | 40 | 9.30E-04 | 15 | One |
| 36 | RAC875_c2532_64 | 7A | 213 | 4.60E-04 | 16.9 | One |
| 37 | RAC875_c11283_379 | 7A | 213 | 9.60E-04 | 14.4 | One |
| 38 | Excalibur_c12499_1907 | 7B | 98 | 1.9E-04-8.8E-04 | 14.8-19.6 | Two |
| 39 | Excalibur_c57808_355 | 7B | 99 | 2.0E-04-9.4E-04 | 15.6-19.7 | Two |
| 40 | Excalibur_c3309_1180 | 7B | 99 | 5.6E-04-6.5E-04 | 15.8-16.0 | Two |
| 41 | Tdurum_contig23504_196 | 7B | 98 | 1.7E-04-8.7E-04 | 14.8-19.5 | Two |
| 42 | Excalibur_rep_c68461_1046 | 7B | 99 | 1.8E-04-9.7E-04 | 14.7-19.5 | Two |
| 43 | wsnp_Ku_c854_1768062 | 7B | 99 | 5.3E-04-6.6E-04 | 15.8-16.2 | Two |
| 44 | wsnp_Ku_c854_1768346 | 7B | 99 | 5.3E-04-6.6E-04 | 15.8-16.2 | Two |
| 45 | wsnp_Ex_c10500_17163956 | 7B | 99 | 5.3E-04-6.4E-04 | 15.9-16.6 | Two |
| 46 | RAC875_c3361_2093 | 7B | 99 | 3.50E-04 | 18.8 | One |
| 47 | Excalibur_c12499_2075 | 7B | 99 | 5.80E-04 | 17.7 | One |
| 48 | TA006077-0786 | 7B | 100 | 8.30E-04 | 15.8 | One |
| 49 | Kukri_c5789_1029 | 7D | 182 | 1.2E-04-1.8E-04 | 19.2-21.4 | Two |
| 50 | BS00074120_51 | 7D | 184 | 1.3E-04-3.3E-04 | 18.0-20.6 | Two |
| 51 | BS00074121_51 | 7D | 183 | 1.3E-04-3.3E-04 | 18.0-20.6 | Two |
新窗口打开
2.4 优异等位变异与POD活性的回归分析
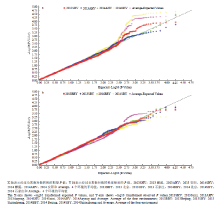
每个SNP标记含有2个等位变异,其中与高POD活性对应的等位变异则为优异等位变异。2个自然群体的优异等位变异的加性回归分析表明,黄淮麦区151份冬小麦品种(系)中含有1—16个优异等位基因,POD活性与优异等位基因数量呈正相关(y=25.493x+499.52,R2=0.9517);而北部冬麦区82份冬小麦品种(系)中含有3—17个优异等位变异,POD活性与优异等位基因数量呈正相关(y= 34.022x+536.06,R2=0.7554)(图5)。总之,优异等位基因数量越多,小麦籽粒POD活性越高。供试的233份材料中Soissons、SELYANKA、MV05-08、DONSKI-93、内乡188、鲁麦5号、鲁麦11号和泰山5号等品种含有较多的优异等位基因和较高的POD活性。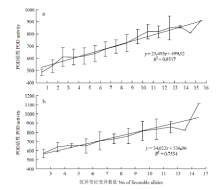 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5黄淮冬麦区(a)和北部冬麦区(b)品种POD活性与优异等位基因数量的加性回归分析
-->Fig. 5Regression of favorable POD alleles in 151 cultivars from Yellow & Huai Winter Wheat Region (a) and Northern China Plain Winter Wheat Region (b)
-->
2.5 候选基因分析
将51个稳定遗传的与POD活性显著关联的SNP标记序列在NCBI数据库中进行BLASTx,获得3个候选基因,即编码产物为磷酸甘露糖变位酶phosphomannomutase的PMM-D1,编码辣根过氧化物酶和相关的分泌性的植物过氧化物酶的PER40和编码烷基氢过氧化物还原酶(AhpC)的F775_31640(表4),为后续功能验证及深入揭示POD活性表达机理奠定基础。Table 4
表4
表4黄淮冬麦区和北部冬麦区品种筛选获得的候选基因
Table 4Candidate genes screened from cultivars from Northern China Plain Winter Wheat Region and Yellow & Huai Winter Wheat Region
| 区域 Area | 标记名称 SNP | 基因型 Genotype | 染色体 Chromosome | 遗传位置 Position (cM) | 候选基因 Candidate gene | 参考文献 Reference |
|---|---|---|---|---|---|---|
| 黄淮冬麦区 YHRVWWR | IAAV9128 | A/G | 2D | 103 | PMM-D1 | [22] |
| Excalibur_c5193_2213 | A/G | 2D | 103 | F775_31640 | [23] | |
| RAC875_c67940_296 | T/C | 2D | 99 | PER40 | [23] | |
| 北部冬麦区NWWR | RAC875_c67940_296 | T/C | 2D | 99 | PER40 | [23] |
新窗口打开
3 讨论
3.1 POD活性定位及应用
小麦面粉颜色是衡量面制品品质的重要指标。影响面粉颜色的原因有很多,如小麦类胡萝卜素合成限速酶八氢番茄红素合酶(phytoene synthase,PSY)、多酚氧化酶PPO(polyphenol oxidase)与脂肪氧化酶LOX(lipoxygenase)。对PSY、PPO和LOX相关基因进行的研究已经较成熟,但对过氧化物酶POD的研究较少。POD也是影响面粉颜色的重要指标,因此,研究POD活性基因对小麦品质改良具有重要意义。本研究利用90K SNP基因芯片对2个区域自然群体多个环境下的POD活性进行GWAS,检测到2A、2D、5A、6A、7B和7D等多个稳定遗传的位点。其中2D位点(97—100 cM)在3个环境中均能检测到,是比较稳定的遗传位点,可根据其连锁标记开发新的标记,用于分子标记辅助育种。将与其紧密连锁的12个SNP标记(CAP8_c2833_325、Excalibur_c2311_ 1116、Excalibur_c5193_2213、Kukri_c17_943、Excalibur_c4167_231、Excalibur_c7366_1475、Jagger_ c7882_154、RAC875_rep_c77646_102、Kukri_c16094_ 496、Kukri_c5276_447、RAC875_c5998_1056和TA002335-0425)转化为STARP或CAPS标记[24],将为分子标记辅助育种提供有效的工具。在检测到的多环境稳定遗传的标记位点中,7D位点的贡献率最大,为18.0%—21.4%。因此,在进一步研究中,可以考虑优先选择2D与7D染色体的关联位点进一步进行精细定位、图位克隆及功能标记开发,为分子标记辅助育种提供有益信息。此外,由于单个位点标记的选择效率有限,通过发现新的主效位点,将本研究发现的POD活性位点改进和开发的新的功能标记与小麦3A染色体的POD活性功能标记POD-3A1和POD-3A2[11]相结合,将多个主效位点结合起来进行分子标记辅助选择,能显著提高选择效率,最终选出高POD活性的材料。
另外POD活性与优异等位变异含量显著正相关,因此,含有较多的优异等位基因和较高的POD活性的品种,如Soissons、SELYANKA、MV05-08、DONSKI- 93、内乡188、鲁麦5号、鲁麦11号和泰山5等品种(系),可作为优良亲本用于育种工作,为POD活性改良工作提供基础。
3.2 基于90 K SNP基因芯片的关联分析与连锁分析比较
随着生物芯片技术的不断发展,利用高密度芯片对基因定位的研究越来越广泛。目前小麦中报道的芯片主要有小麦9K SNP芯片、小麦90K SNP芯片、小麦660K SNP芯片和小麦820K SNP芯片等。本研究所利用的小麦90K SNP 芯片:该芯片由美国Illumina公司与Akhunov E、Hayden M.及Cavanagh合作完成,包含81 587个SNP标记。其中有染色体信息的标记共42 104个,占51.61%[25]。90K芯片的分辨率已经很好,并且应用90K芯片来进行小麦基因定位的研究已经很成熟,虽然660K芯片与820K芯片的标记密度较90K芯片更高,但90K芯片较660K与820K芯片的成本较低,性价比更高。WEI等[11]利用豆麦/石4185 RIL群体结合小麦90K SNP芯片,首次对普通小麦POD活性进行了连锁分析,发现了3个主效QTL。其中5A位点与本研究一致;进一步证明关联分析与连锁分析均能得到与目标性状显著相关的QTL位点,通过自然群体进行关联分析与连锁分析技术互为补充,互为验证。ZHAI等[26]同样用90K SNP芯片对面粉颜色性状进行QTL定位,其中2D(84.3—94.0 cM)、5A(71.7—76.3 cM)和7B(109.2—114 cM)QTL与本研究检测到的2D(97—103 cM)、5A(80 cM)和7B(98和99 cM)位点相近。2种方法同时能检测到的重合位点是稳定遗传的关键位点。但是本研究发现大部分POD活性位点与QTL定位的位点并不相同,如GWAS结果中的7D等位点在2个麦区均为稳定遗传位点,但QTL定位并没有检测到,在排除假阳性和伪关联的前提下,可见以自然群体为材料的关联分析可以补充检测到更多与POD活性相关的新位点。近年来利用连锁分析进行QTL定位已经成为现代分子育种领域的研究重点,但受限于遗传群体亲本的差异度,单一以连锁分析的方法进行QTL定位往往会遗漏大量微效甚至主效基因[27]。而关联分析是一种利用自然群体中积累的历史重组结果,发掘性状关联位点的方法,此方法能达到比双亲分离群体更高的分辨率,是连锁分析的有益补充,能对连锁分析结果进行验证的同时,发现新的关键位点[28]。因此,基于高密度基因芯片,采用连锁分析结合关联分析技术发掘作物产量、品质、抗逆等数量性状基因已经成为分子标记研究领域的发展趋势[29-30]。
3.3 候选基因分析
PMM-D1编码产物为磷酸甘露糖变位酶(phosphomannomutase,PMM),在高等植物中,PMM是抗氧化抗坏血酸合成的重要前体物质[31],在高等植物POD调节途径中,PMM可以通过控制抗氧化抗坏血酸的合成来抑制POD活性[32]。PER40编码辣根过氧化物酶和相关的分泌性的植物过氧化物酶,分泌过氧化物酶属于植物血红素依赖性POD超家族Ⅲ类。F775_31640编码烷基氢过氧化物还原酶(AhpC),在植物体内,AhpC可以通过与POD竞争相同底物H2O2,来抑制POD活性[33]。以上候选基因编码不同蛋白,在植物代谢途径中直接或间接参与POD活性表达,故推测对小麦籽粒POD活性有调节作用,因此,在后续工作中对候选基因进行深入研究有助于为提高小麦籽粒POD活性提供依据。4 结论
黄淮麦区与北部冬麦区2个自然群体遗传多样性丰富,群体结构简单,适用于全基因组关联分析。在2个自然群体中分别发现20个POD活性位点,其中有2个位点在2个自然群体中均能稳定遗传,发现3个候选基因PMM-D1、PER40和F775_31640。含有越多优异等位变异的材料,其POD活性越高,可并发掘高POD活性品种为育种工作提供优质材料。The authors have declared that no competing interests exist.
