0 引言
【研究意义】小麦赤霉病(Fusarium head blight,FHB)是由禾谷镰孢(Fusarium graminearum)引起的一种小麦重要穗部病害,主要发生于潮湿及半潮湿地区[1]。在小麦赤霉病大流行年份,可以造成70%,甚至是100%的产量损失[2]。小麦赤霉病的发生不仅造成严重的产量损失,而且感病的小麦籽粒还含有各种毒素(尤其是DON毒素),严重威胁人和动物[3-4]的食品安全。近年来,随着全球气候变暖以及耕作制度的变化,小麦赤霉病逐渐由偶发病害上升为常发病害,发生面积也不断扩大。在中国,赤霉病已由常发的长江中下游麦区扩展到黄淮麦区和华北麦区[5-6]。目前还没有彻底防治小麦赤霉病的化学药剂,最为经济环保的控制小麦赤霉病危害的途径就是培育和选用抗赤霉病品种。对普通小麦-长穗偃麦草(Thinopyrum ponticum)衍生小麦新品种进行赤霉病抗性鉴定和抗性基因的分子鉴别,可为深入研究和应用长穗偃麦草的赤霉病抗性基因提供理论依据,对于小麦赤霉病抗性育种具有重要意义。【前人研究进展】赤霉病抗源相对较少,国内外****通过大量的基因资源研究,迄今从普通小麦及其近缘种属中仅鉴定出7个赤霉病抗性主效基因,分别是来自苏麦3号的Fhb1[7]和Fhb2[8]、大赖草的Fhb3[9]、望水白的Fhb4[10]和Fhb5[11]、披碱草属的Fhb6[12]和十倍体长穗偃麦草的Fhb7[13]。中国工作者经多年努力,培育出了扬麦系列、宁麦系列和鄂麦系列等优良抗性品种,但大面积生产中缺乏抗赤霉病的高产小麦品种[14]。纵观国内数十年的小麦抗赤霉病育种,其抗源仍然主要局限于苏麦3号、望水白和它们的衍生系。赤霉病抗源基础狭窄是小麦育种工作取得突破性进展的瓶颈,发掘新的小麦赤霉病抗源成为提高小麦赤霉病抗性的关键[15]。由此可见,鉴定和利用小麦近缘种属的赤霉病抗性基因尤为重要[16]。长穗偃麦草是小麦的近缘植物,其E基因组含有许多优异的抗性基因,如抗旱、抗寒能力很强,对白粉病[17]和锈病完全免疫(7E染色体)[18],高抗条纹花叶病和小麦黄矮病(2E、7E染色体)[19-20],对赤霉病表现较强的耐病性(1E、7E染色体)[18,21-23],且又能与小麦进行远缘杂交,实现抗病基因的遗传转移。因此,长穗偃麦草是小麦抗病育种的重要抗源亲本之一[24]。八倍体小偃麦小偃693和小偃7430染色体总数均为56条,但是它们分别含有8对和6对长穗偃麦草染色体,还分别含有4条和2条小麦-长穗偃麦草易位染色体[25]。小偃7430与普通小麦杂交、回交选育而成了许多抗白粉病和条锈病、秆锈病的小偃麦衍生新品种(系)[26-28]。然而,目前关于小偃693衍生后代抗病性的研究鲜有报道。小偃597是利用小偃693与普通小麦杂交、回交选育成功的含有十倍体长穗偃麦草遗传物质的普通小麦品种,对赤霉病表现抗性[29]。研究表明,长穗偃麦草中已发现3个赤霉病抗性主效基因,分别定位于二倍体长穗偃麦草1E染色体[30-32]和7E染色体[22,32],十倍体长穗偃麦草7E染色体[13,18,23]。其中十倍体长穗偃麦草7E染色体长臂上赤霉病抗性基因被正式命名为Fhb7[13],可解释抗赤霉病表型变异的30.46%[18]。GUO等[13]将Fhb7定位在7EL染色体末端1.7 cM范围内,且两侧紧密连锁的分子标记分别为Xcfa2240和XsdauK66;该抗病区间对应于水稻、短柄草和高粱的基因组大小分别为18.8、37.1和27.0 kb,并开发出与Fhb7紧密连锁的特异分子标记,如XsdauK60、XsdauK71等,这些分子标记可用于该基因的分子标记辅助选择。【本研究切入点】西农509、西农511和西农529是由西北农林科技大学农学院培育出的含有十倍体长穗偃麦草遗传物质的高产、优质、抗病的普通小麦新品种[33-34],田间赤霉病表现轻[6,33-34]。这些品种已在黄淮冬麦区南片的河南省(南部稻茬麦区除外)、安徽省沿淮及淮北地区、江苏省淮北地区和陕西省关中地区示范推广种植。2014—2016年在河南南阳和驻马店、江苏省徐州和安徽淮北生态区域等赤霉病重发区,这些品种赤霉病轻度发生,发病率仅为1%—3%,属于中抗或中感抗性水平[6,35-37]。然而,目前它们赤霉病的抗性遗传基础并不清楚。【拟解决的关键问题】对3个衍生新品种进行赤霉病抗性进行人工接种鉴定,然后用偃麦草E组染色体特异标记对西农509、西农511和西农529及其主要亲本小偃693、小偃597和十倍体长穗偃麦草进行分子鉴定,初步确定其赤霉病抗性基因的来源,在此基础上利用长穗偃麦草7EL染色体上的特异分子标记及与抗性基因Fhb7紧密连锁的分子标记对实验材料进一步进行分子鉴别,为这些抗赤霉病的长穗偃麦草衍生新品种的推广和利用提供理论依据。1 材料与方法
1.1 试验材料
普通小麦中国春(Triticum aestivum),十倍体长穗偃麦草;普通小麦-十倍体长穗偃麦草远缘杂交后代材料和品种:八倍体小偃麦小偃693(染色体组成为AABBDDEE)和普通小麦品种小偃597;新培育的普通小麦-十倍体长穗偃麦草衍生品种西农509、西农511和西农529。赤霉病抗性鉴定的对照品种苏麦3号、扬麦158、宁麦9号和安农8455。小偃693是本研究中3个普通小麦-长穗偃麦草衍生新品种的间接亲本,普通小麦品种小偃597是西农529和陕麦159的直接亲本,是西农511的间接亲本(图1)。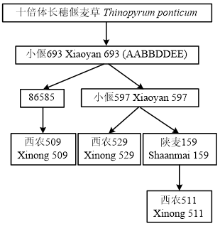 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1普通小麦-长穗偃麦草衍生小麦新品种长穗偃麦草遗传基因溯源
-->Fig. 1The source of Thinopyrum genes in three derived varieties of common wheat-Th. ponticum
-->
1.2 赤霉病人工接种鉴定
抗赤霉病人工接种鉴定由江苏省农业科学院植物保护研究所在南京鉴定完成,赤霉菌菌株为F0301、F0609、F0980和F1126,鉴定方法和调查记载标准参见“小麦抗病虫性评价技术规范”系列标准(NY/T 1443—2007)[38]。苏麦3号、扬麦158和宁麦9号为抗病对照品种,安农8455为感病对照品种。抗性分级标准:抗(R),平均严重度<1.5(苏麦3号的平均反应级+标准差);中抗(MR),1.5≤平均严重度<2.5(扬麦158的平均反应级+标准差);中感(MS),2.5≤平均严重度<3.5,感(S),平均严重度≥3.5。1.3 抗赤霉病基因的分子鉴别
小麦幼叶基因组DNA采用微量CTAB法 提取,干燥后溶于TE缓冲液。由于长穗偃麦草中已发现的赤霉病抗性主效基因均被定位于E基因组[13,18,21-23,30-32],本研究选用长穗偃麦草E组染色体特异分子标记[22,39-47]及7EL上Fhb7连锁的分子标记[13,18,48-49]对参试材料进行长穗偃麦草遗传物质和Fhb7的检测(附表1)。引物由生物工程(上海)有限公司合成。PCR扩增反应在GeneAmp 9700型热循环仪(ABI,USA)上进行,反应混合液总体积为10 μL,其中包括0.25 U Taq酶、1.0 μL 10×Buffer、1.5 mmol·L-1 MgCl2、0.2 mmol·L-1 dNTPs、0.5 μmol·L-1 primer(上下游引物)、50—100 ng模板DNA。扩增程序:94℃ 2 min;94℃ 1 min,50—60℃ 1 min,72℃ 1 min,35个循环;72℃ 5 min。PCR产物用8.0%的非变性聚丙烯酰胺凝胶上电泳检测,银染后照相观察并记录。Table 1
表1
表13个普通小麦-长穗偃麦草衍生小麦新品种的赤霉病病情指数和抗性评价
Table 1The disease index of resistance to FHB of three derived varieties of common wheat-Th. ponticum
| 小麦品种 Wheat variety | 病情指数和抗性评价 Disease index and resistance* | ||||
|---|---|---|---|---|---|
| 2009 | 2010 | 2014 | 2015 | 2016 | |
| 西农509 Xinong 509 | 1.44 R | 2.35 MR | |||
| 西农529 Xinong 529 | 2.42 MR | 2.45 MR | |||
| 西农511 Xinong 511 | 2.47 MR | ||||
| 扬麦158 Yangmai 158 | 1.82 MR | 3.67 S | 2.11 MR | 2.27 MR | 2.22 MR |
| 苏麦3号 Sumai 3 | 1.65 R | 1.62 R | 1.39 HR | 1.27 HR | 1.16 HR |
| 宁麦9号 Ningmai 9 | 1.67 MR | 2.36 R | 1.90 MR | 1.65 MR | 1.65 MR |
| 安农8455 Annong 8455 | 3.98 S | 3.95 S | 3.83 HS | 3.56 HS | 3.02 HS |
新窗口打开
2 结果
2.1 3个普通小麦-长穗偃麦草衍生新品种的赤霉病抗性表现
江苏省农业科学院植物保护研究所先后对西农509(陕麦509)(2008—2010)、西农529(2013—2015)和西农511(2015—2016)进行了赤霉病抗性人工接种鉴定。由表1调查结果可以看出,西农509、西农529和西农511的赤霉病鉴定结果均与中抗对照品种扬麦158的抗性水平相当,表现为中抗。2.2 普通小麦-长穗偃麦草衍生新品种外源遗传物质的分子标记检测
用105个长穗偃麦草E基因组的特异标记(其中1E、2E、3E、4E、5E、6E和7E染色体上特异标记分别有29、12、10、5、14、10和25个)分别对中国春、十倍体长穗偃麦草、西农509、西农511和西农529进行分子检测。结果显示,71个标记在中国春和十倍体长穗偃麦草间有多态性,但只有Xcfa2040(图2-A)、Xcfa2106、Xmag1759、Xpsp3123、Xwmc273(图2-B)、Xbarc154和Xgwm156等7个标记在3个普通小麦-十倍体长穗偃麦草衍生新品种中均扩增出长穗草的特异产物,其中前5个标记都位于7E长臂,Xbarc154和Xgwm156则位于7E短臂。E基因组分子标记分析结果表明,西农509、西农511和西农529携带来自于十倍体长穗偃麦草7E染色体的遗传物质。2.3 普通小麦-长穗偃麦草衍生新品种及其亲本的分子检测
用定位于7E染色体长、短臂上的75和22个分子标记(包括DArT标记、SSR标记、EST标记)[13,36,43,45,48-49]分别对中国春、十倍体长穗偃麦草、3个普通小麦-长穗偃麦草衍生新品种及它们重要的亲本小偃693和小偃597进行分子标记分析。结果表明,7ES 64.63到7EL 153.27长约89 cM之间共有20对引物在小偃693、小偃597和3个新品种中均能扩增出稳定的长穗偃麦草的特异条带,其中12个引物定位于7E长臂,8个定位于7E短臂(表2)。由此进一步推断西农509、西农511和西农529这3个普通小麦-长穗偃麦草衍生新品种含有十倍体长穗偃麦草7E染色体跨着丝粒区域的中段遗传物质。2.4 普通小麦-长穗偃麦草衍生新品种抗赤霉病基因的分子检测
用定位于7EL 140.03到160.00处的与Fhb7紧密连锁的40个XsdauK系列标记、7个TNAC系列标记、4个SSR标记(Xcfa2040、Xcfa2240、Xmag1932和Xswes19)[13,36,43,45]分别对中国春、十倍体长穗偃麦草、3个普通小麦-长穗偃麦草衍生新品种以及赤霉病抗性基因的供体亲本小偃693和小偃597进行基因标记分型分析。结果表明(表2),XsdauK8(图2-C)、XsdauK144(7EL 149.51)、XsdauK27(图2-D)、XsdauK99(图2-E)、XsdauK116和Xcfa2040(7EL 153.27)(图2-A)等6个标记在3个新品种及小偃693和小偃597两亲本中具有十倍体长穗偃麦草的特异性条带;XsdauK60(7EL 153.77)、Xmag1932(7EL 154.70)、Xcfa2240(7EL 156.27)(图2-F)和XsdauK66(7EL 158.02)(图2-G)4个标记能在八倍体小偃麦小偃693中扩增产生十倍体长穗偃麦草的特异性条带,而在小偃597和3个小麦新品种中没有十倍体长穗偃麦草的特异条带;XsdauK71(7EL 158.97)和Xsews19(7EL 160.00)(图2-H)在小偃693、小偃597和3个小麦新品种中均没有十倍体穗偃麦草的特异条带。Table 2
表2
表27E染色体上部分分子标记对3个小麦新品种及其主要亲本的扩增片段大小
Table 2Amplified fragment size of markers on 7E ?anking Fhb7 in three derived varieties of common wheat-Th. ponticum and their major donor parents
| 标记 Marker | 标记位置Position | 共线基因Orthologous genes | 中国春 Chinese Spring | 长穗偃麦草 Th. ponticum | 小偃 693 Xiaoyan 693 | 小偃 597 Xiaoyan 597 | 西农 509 Xinong 509 | 西农511 Xinong 511 | 西农 529 Xinong 529 |
|---|---|---|---|---|---|---|---|---|---|
| Xcfa2174 | 64.63 | 230 | 240 | 240 | 240 | 240 | 230 | 240 | |
| Xgwm44 | 71.35 | 160 | 140 | 160, 140 | 160, 140 | 160, 140 | 160, 140 | 160, 140 | |
| Xwmc653 | 75.10 | 160 | 190 | 160, 190 | 160, 190 | 160, 190 | 160 | 160, 190 | |
| Xcfd31 | 86.10 | 320 | 340 | 340 | 340 | 340 | 340 | 340 | |
| Xgwm350 | 87.32 | 130 | 160 | 130, 160 | 130, 160 | 130, 160 | 130, 160 | 130, 160 | |
| Xbarc154 | 89.76 | 600 | 520 | 600, 520 | 600, 520 | 600, 520 | 600, 520 | 600 | |
| Xcfd21 | 92.36 | 280 | 260 | 280, 260 | 280, 260 | 280, 260 | 280, 260 | 280, 260 | |
| Xgwm333 | 96.59 | 290 | 300 | 300 | 300 | 300 | 300 | 300 | |
| Xmag1759 | 98.22 | 200 | 250, 620 | 250, 620 | 250, 620 | 250 | 250, 620 | 250, 620 | |
| Xcfa2106 | 104.89 | 220 | 240 | 220, 240 | 220, 240 | 220, 240 | 220, 240 | 220, 240 | |
| Xpsp3123 | 110.39 | 150 | 120 | 150, 120 | 150, 120 | 150, 120 | 150, 120 | 150, 120 | |
| Xgwm635 | 111.32 | 110 | 130 | 130 | 130 | 130 | 130 | 130 | |
| Xgwm130 | 116.93 | 120 | 250 | 250 | 250 | 250 | 250 | 250 | |
| Xwmc273 | 131.76 | 210 | 200, 150 | 200, 150 | 200 | 200 | 200 | 210, 150 | |
| XsdauK8 | Bradi1g29460 | 765 | 985 | 765, 985 | 985 | 985 | 985 | 985 | |
| XsdauK144 | 149.51 | Bradi1g29441 | 460 | 440 | 440 | 460, 440 | 460, 440 | 440 | 440 |
| XsdauK27 | Os06g51420 Bradi1g29390 Sb10g031190 | 430 | 465 | 430, 465 | 430, 465 | 430, 465 | 430, 465 | 430, 465 | |
| XsdauK99 | Os06g51440 Bradi1g29140 | 580 | 660, 700 | 660, 700 | 660, 700 | 660 | 660 | 660 | |
| XsdauK116 | Os06g514370 Bradi1g29210 | 250 | 230 | 230 | 230 | 230 | 230 | 230 | |
| Xcfa2040 | 153.27 | 260 | 280, 240 | 280, 240 | 280, 240 | 280, 240 | 280, 240 | 280, 240 | |
| XsdauK60 | 153.77 | Os06g51490 Bradi1g29250 Sb10g031265 | 420 | 430 | 420, 430 | 420 | 420 | 420 | 420 |
| Xmag1932 | 154.70 | 275 | 285, 235 | 285, 275 | 275 | 275 | 275 | 275 | |
| Xcfa2240 | 156.27 | 280 | 250 | 280, 250 | 280 | 280 | 280 | 280 | |
| XsdauK66 | 158.02 | Os06g51500 Bradi1g29290 Sb10g031250 | 350 | 330 | 350, 330 | 350 | 350 | 350 | 350 |
| XsdauK71 | 158.97 | Os06g51510 Bradi1g29300 Sb10g031240 | 430 | 440 | 430 | 430 | 430 | 430 | 430 |
| Xsews19 | 160.00 | 195 | 275, 195 | 195 | 195 | 205, 192 | 205, 192 | 205, 192 |
新窗口打开
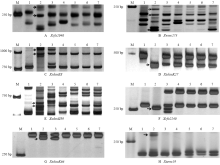 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图27E染色体上部分标记PCR 扩增产物的聚丙烯酰胺凝胶电泳图谱
M:DEL2000;1:中国春Chinese Spring (CS);2:十倍体长穗偃麦草Th. ponticum;3:小偃693 Xiaoyan 693;4:小偃597 Xiaoyan 597;5:西农509 Xinong 509;6:西农511 Xinong 511;7:西农529 Xinong 529。左侧为分子量标记条帯大小(bp)。箭头所示为偃麦草染色体的特异条带
-->Fig. 2Polyacrylamide gels of PCR products obtained with some specific markers located on the chromosome 7E of Th. Ponticum
The numbers at the top of the gels are lane numbers. The numbers on each photo is the marker bands size in bp. Arrows indicated the Thinopyrum specific bands linked to Fhb7
-->
3 讨论
在抗赤霉病育种方面,自20世纪70年代,中国研究者从普通小麦及其近缘物种3万多份材料中筛选出苏麦3号和望水白两个抗性强而稳定的普通小麦品种,但其农艺性状较差[50],应用过程中赤霉病抗性很难和丰产性结合起来,生产上亟需高产抗赤霉病的小麦品种。然而,自扬麦158后,大面积推广的品种均未解决好赤霉病抗性与丰产性负相关的问题[14]。因而开拓新的抗源以及如何高效利用小麦近缘种的抗病基因已成为小麦抗赤霉病育种亟待解决的重要问题[51]。长穗偃麦草是小麦远缘杂交育种的重要亲本材料之一,研究表明其1E和7EL染色体上携带有抗赤霉病基因[13,18,21-23,26]。JAUHAR等[31]将抗赤霉病的长穗偃麦草与硬粒小麦杂交,获得了赤霉病抗性提高显著的衍生系。张璐璐等[52]利用偃麦草7E染色体特异分子标记筛选结合GISH验证培育了小麦-长穗偃麦草7EL异位系和7ES附加系,赤霉病抗性鉴定表明7EL易位系的赤霉病抗性明显较好。西北农林科技大学培育的普通小麦-十倍体长穗偃麦草衍生新品种西农509、西农511和西农529目前已成为黄淮冬麦区南片和陕西省关中地区示范推广的高产优质的小麦新品种。据报道,上述3个小麦新品种在河南、安徽、江苏等赤霉病重发地田间表现赤霉病很轻[6,31-33]。本研究中,通过人工抗性鉴定,这3个品种的赤霉病抗性与扬麦158相当,属于中抗水平,且抗性稳定。西农509、西农511和西农529赤霉病轻且综合性状和丰产性好,有望在黄淮冬麦区南片赤霉病常发区和重发区大面积推广种植。
本研究利用长穗偃麦草E基因组特异的引物初步确定3个小麦新品种的长穗偃麦草的遗传物质来自7E染色体跨着丝粒区域的中段,说明其抗赤霉病基因来源于7E染色体,研究结果与前人的研究一致[13,18,22-23,52]。来自十倍体长穗偃麦草的抗赤霉病基因Fhb7已定位于7EL染色体上,Xcfa2240(7EL 156.27)和XsdauK66(7EL 158.02)分别与其紧邻[13]。为了探究上述3个小麦新品种中赤霉病抗性基因与Fhb7的关系,选用偃麦草7EL染色体(7EL 140.03—7EL 160.00)上与Fhb7连锁的分子标记对这3个新品种及其主要亲本小偃693、小偃597和十倍体长穗偃麦草进一步进行分子标记分析。结果发现,7EL上6个与Fhb7紧密连锁的分子标记在3个新品种中扩增产生长穗偃麦草的特异产物,其中,XsdauK99、XsdauK116和Xcfa2040(7EL 153.27)3个标记与Fhb7的遗传距离仅3.0—4.0 cM。GUO等[13]利用与抗赤霉病基因Fhb7紧密连锁的分子标记(Xcfa2240和XsdauK66)进行分子辅助选择,从济麦22与小麦-长穗偃麦草抗赤霉病短片段易位系SDAUl881杂交、回交后代中成功获得2个新的改良系SDAU2003和SDAU2028。然在本研究中,与Fhb7紧邻的4个标记XsdauK60(7EL 153.27)、Xmag1932(7EL 154.70)、Xcfa2240(7EL 156.27)和XsdauK66(7EL 158.02),在小偃597和3个长穗偃麦草衍生小麦新品种中均没有扩增出长穗偃麦草的特异性条带,只在八倍体小偃麦小偃693中有长穗偃麦草的特异性条带。同时,7EL末端的标记XsdauK71(7EL 158.97)和Xsews19(7EL 160.00)不仅在3个小麦新品种中没有偃麦草特异条带,而且在小偃693和小偃597中也没有。何方[25]以十倍体长穗偃麦草总基因组DNA为探针对小偃693根尖细胞染色体进行GISH分析,发现小偃693中不仅含有4对Ee组染色体和4对Eb组染色体,同时还含有4条小麦-长穗偃麦草异位染色体。本研究中,西农509、西农511和西农529 这3个普通小麦-十倍体长穗偃麦草衍生新品种中含有长穗偃麦草7E染色体跨着丝粒区域的中段的遗传物质,但缺失了Fhb7相关联的7EL末端片段,究其原因,也许是在八倍体小偃麦小偃693与普通小麦杂交、回交选育过程中,7E染色体与普通小麦染色体发生了异位,导致7EL末端片段丢失,这个推测有待借助GISH和FISH等技术进一步验证。
4 结论
西农509、西农511和西农529等3个普通小麦-十倍体长穗偃麦草衍生新品种在黄淮南片麦区赤霉病重发地区表现赤霉病很轻,同时人工接种鉴定表现为中抗赤霉病。赤霉病抗性基因分子鉴定表明,长穗偃麦草7ES 64.63—7EL 153.27之间有20对引物在3个新品种和它们的主要亲本小偃693和小偃597中均扩增出稳定清晰的长穗偃麦草特异条带,然而在这3个普通小麦-长穗偃麦草衍生新品种及小偃597中没有检测到7EL末端抗赤霉病主效QTL Fhb7的分子标记产物。推测这3个普通小麦-长穗偃麦草衍生新品种含有来自于十倍体长穗偃麦草的不同于Fhb7的抗赤霉病基因。Table S1
附表1
附表1所利用的长穗偃麦特异分子标记
Table S1 Specific molecular markers of Thinopyrum ponticum and their sequences used in this study
| 标记名称 Marker name | 引物序列 Nucleotides sequence of markers | 所在染色体 Genome location | 扩增片段大小 Fragment size (bp) | 参考文献 Reference | |
|---|---|---|---|---|---|
| 左引物 Left primer (5′-3′) | 右引物 Right primer (5′-3′) | ||||
| 1Ee STS241 | TAACTAGCACACCAAATCGTTTTTGCAAAC | AATTCATTGGGGTGGGTAAAGGAAGTGG | 1E | 250 | [41] |
| 1Ee STS318 | GAATTCATCATAAAAACAGATCAAAGG | TAACGTAAACTTCCCGTCTGCTAC | 1E | 280 | [41] |
| SP1E_No.1 | TATCAAATAATATGTGCATG | CATGACAGAGATATGAAAAG | 1E | 276 | [42] |
| SP1E_No.21 | CATCATATAAGAATGCAGGA | TCAACAGCACGCCAAC | 1E | 230 | [42] |
| SP1E_No.23 | TAGGAAAGAAATCTGAAATA | TGAGACAGCAGCAAAA | 1E | 220 | [42] |
| SP1E_No.27 | GGCTGATGCACTGACGT | TCCGAAATCTGAAAAGGAG | 1E | 273 | [42] |
| SP1E_No.29 | ACTGTTTTTTTTTTCGAG | AGCGCACAAGATGAGAC | 1E | 151 | [42] |
| SP1E_No.30 | TTAGAAAAATTCAACCCG | AATCTTGAAACATGGGAG | 1E | 80 | [42] |
| SP1E_No.31 | CCGGTTTGAAAGAAGAGA | CAACAAGCAAATGAGGGA | 1E | 158 | [42] |
| SP1E_No.33 | TCACCTATGAGATCGCT | TATCCGAAGAACCTGTAC | 1E | 285, 175 | [42] |
| SP1E_No.34 | TGTTCCTTGCATCATCT | AACTCACCTATCGGTCTAT | 1E | 158 | [42] |
| SP1E_No.35 | TTGACAACCCAATCTG | TCACTGATAAATAACAAGAG | 1E | 162 | [42] |
| SP1E_No.36 | TGACGGCAACGGAGTG | TCTGCCGAACATGGAATC | 1E | 150 | [42] |
| SP1E_No.37 | AGCACTTGGGTTTTCAT | GGATTGGAGGGTCACTA | 1E | 260 | [42] |
| SP1E_No.38 | TTCAAGAAGTATAGCTCGTT | AACCATAAAGTCCCAACA | 1E | 300 | [42] |
| SP1E_No.41 | GGAAAGGGCACTCCAACC | GCGTCACATCCTGGTTCG | 1E | 210 | [42] |
| SP1E_No.42 | CTCCATCCCTCCACTT | AACATCAGCAGTTGGG | 1E | 190 | [42] |
| SP1E_No.44 | TTGACTTCTCCCAAGTTG | AATGGAAAGGAAAATCTG | 1E | 203 | [42] |
| SP1E_No.45 | TCTATTTTGCTATTTGCC | TTTTGTCTTCATCACCAT | 1E | 568 | [42] |
| SP1E_No.47 | TACTCGCTACTTACAATCT | GAACATTATTGTGAAGCA | 1E | 255 | [42] |
| SP1E_No.48 | CGGAATGTCCTCACA | ATTGGCTAATGCTATGA | 1E | 280 | [42] |
| SP1E_No.8 | AATATTTATGGCGCACGCAG | TCATCAGGGGAGTGAGGAGC | 1E | 400, 200 | [42] |
| Xedm17 | TAGCTGCATCTCTGGTTTGG | CTAGTTGTTGTTGCTGCGAAA | 1E | 396 | [39] |
| Xedm74 | GTAAGTCGACCGAGGAGACG | CCTTCTTGCTTGGCATTCTC | 1EL | 360 | [39] |
| Xedm92 | ATCCGCGACGACAATGTAAG | CTGGTGTGGATCTTGACGTG | 1EL | 185 | [39] |
| Xmag2137 | GAGTTCGATGACATGGCTGA | CCGATAACAAAGTGCGTGAA | 1E | 580, 250 | [43-44] |
| Xmag3489 | CGATGTAAAGACTGTAGCCTGTAGG | CGTCCATTTGGTTCGGTGAG | 1E | 330 | [43-44] |
| Xmag834 | ACGCCCTCAAACAAGAAAGA | CTTCCTCTCCTTCCCCAGAG | 1E | 500 | [43-44] |
| Z34-1E200 | GATGTCACCAAGCCCTAC | TCCTACGAAACACCCAAG | 1E | 225 | [47] |
| 2Ee STS116 | TTAACCAGGGATACAAAAACACCATAATG | AATTCAATTACCTTGTGGATGCAAACAC | 2E | 150 | [41] |
| 2Ee STS182 | AATTCATTTCTAATAGATACTCGAATGAA | TTAACTGAGGAGAGGCTACAACATCC | 2E | 182 | [41] |
| SM2E-1 | CAGGTCCAAAAAACCAGCC | GAATTCGAGGTTCCGCAG | 2E | 480, 210 | [40] |
| SM2E-3 | GTGTGTGCGCATGAACTACTT | GGAGTCGTTGGTTGGACATG | 2E | 225 | [40] |
| SM2E-5 | ATGCTTTTCCCTTCCAACAG | CAAACGAAAAGCTGACCAAAC | 2E | 173 | [40] |
| Xmag2090 | AAAGGTGTAAGCGTGGCATC | ACACGCATAATGCACTCCAG | 2E | 300, 255 | [43-44] |
| Xedm28 | GCTCACTCACGCATCATAGC | GTTGGCGGAATCCTTCTTC | 2ES & 3EL | 150 | [39] |
| Xedm8 | GACCCCCACTTCCTGCTC | GGGCAAAACTGTGGCTATTG | 2ES & 3EL | 145 | [39] |
| Xedm96 | TCCAAGACTGTGCCTCAAGA | GCGTTGAACTCCTGTTCCAT | 2ES & 3ESL | 175 | [39] |
| Xmag3253 | CTGCTGCTTGGGATCATTCT | GCTGGTGAGAGTTGGAAACC | 2E | 600, 310 | [43-44] |
| Xmag3616 | GTCCGTGCCATTCAACTTCT | TCTATCCACTCCATTCCAAGC | 2E&3E | 260, 200 | [43-44] |
| Xmag4137 | CTCGTGTGGCTCATCAATGT | TGACATCTGGTTGGTGGTCA | 2E&3E | 490 | [43-44] |
| iPBS8 | TGCAGGTCGACGA | GCGGGTATGCTTTG | 3E | 428 | [46] |
| SM3E-1 | CACACGCAATTCAACAACTCAAG | ACGAATTGACCGTCGATCAGA | 3E | 195 | [40] |
| SM3E-2 | GCAGCAGAGAACGTACACTTGTA | TGGCAAAAGTATTCTGAACCGT | 3E | 154 | [40] |
| TNAC1248 | ATGATGCAGCAGCAAATTACA | CTGAGGAGCCTCTCCAACTCT | 3E | 700 | [43,45] |
| Xmag905 | ATGTGAATGGAAGGTCGGAG | AGCACTTGCAGGCTCTTCAT | 3E | 260 | [43-44] |
| SM4E-1 | ATTCCTAATGGTCCTACAGC | ATCACGCACGAACAAGTC | 4E | 580 | [40] |
| Xmag1682 | CGAATGCCAAGCTGTTCCCT | ACATGCCCCTTGAGAGTGTGG | 4E | 855 | [43-44] |
| Xmag2055 | TTCCCAGCAAAGTAGCACAA | TGTGGGATCGCCTAGAAATC | 4E | 550 | [43-44] |
| Xmag3733 | CACACAACTCCACGGAACAG | CATAAACCAGCAGCCCAACT | 4E&6E | 300 | [43-44] |
| Z8-4E374 | TGTTGCCAGTGGAGGAAG | TTGAACTACTGAACCGAAT | 4E | 368 | [47] |
| TNAC1503 | TGAGGTTGGTTCTCATCTGGA | CGTTGGAAACAATCTGAATGG | 5E | 700.00 | [43,45] |
| TNAC1559 | AAACAAGGCCCTGAAACACTT | CATTGTCAGGCTATGGGACAT | 5E | 1180 | [43,45] |
| Xmag1293 | GATTGTTCTTGGGGAGATGCTTG | GCCTTTTGGACCGTCAGAGAC | 5E | 260, 300 | [43-44] |
| Xmag1426 | GCGAGTTTTCGTAGCAAAGG | TCACAGGAGTGGAGGCTCAC | 5E | 650, 280 | [43-44] |
| Xmag1579 | AAGGCATTTTTCTCGGTCTC | CACACTTCTTGAGGATTTGAGC | 5E | 550 | [43-44] |
| Xmag4013 | TCGACTCCCATCCTCAAGAC | CACCACCATTCCTTTGGAG | 5E | 650, 400 | [43-44] |
| Xmag532 | CGACGATCCAACCAGCTAAT | TAGCCAGCCTATGGTAACGG | 5E | 250 | [43-44] |
| Xmag620 | TAGTTGCATGGTCGCTTCTG | CGTAGCTTTTCGTTGATCCC | 5E | 295 | [43-44] |
| Xedm54 | CGTCCTTGTTCTTGCTTCAA | TGAGCAGGCCATAAGGAAAC | 5ES | 150 | [39] |
| Xedm205 | AGTCGCCGTTGTATAGTGCC | CGACCCGGTTCACTTCAG | 5ES | 240 | [39,49] |
| Xgwm205 | CGACCCGGTTCACTTCAG | AGTCGCCGTTGTATAGTGCC | 5ES | 180 | [39] |
| Xedm46 | CCGCCGTCCGTCCT | GCAGGAGGCCTCGTTGTAG | 5EL & 7EL | 215 | [39] |
| Xedm335 | CGGTCCAAGTGCTACCTTTC | CGTACTCCACTCCACACGG | 5EL & 7EL | 150 | [39,44] |
| Xgwm335 | CGTACTCCACTCCACACGG | CGGTCCAAGTGCTACCTTTC | 5EL & 7EL | 150 | [39] |
| TNAC1674 | CCACCACAGAAGCAGATGAAT | GCTAGATGGCACACCAAGTG | 6E | 700 | [43,45] |
| Xedm140 | CCTCCGACGAGATCTGCTAC | GCTTTGGCTTCCGGTACG | 6E | 271 | [39] |
| Xgwm179 | AAGTTGAGTTGATGCGGGAG | CCATGACCAGCATCCACTC | 6E | 162 | [39] |
| Xmag2276 | CCGCCCCGATACAGTTTATTTC | TACCGCTCCGTCGTCAACTACTG | 6E | 320 | [43-44] |
| Xmag3017 | GGTGAGGGAGTGCGAGAAT | AGGCTTCCTGTTGCTGTGG | 6E | 260, 170 | [43-44] |
| Xmag3113 | TCATGCACCCATCTGGACTA | AGCTATTCGTGGCAAACACA | 6E | 210 | [43-44] |
| Xedm149 | ATCCACGCCAAGCAGAAG | CTGTGGGAAGAAGTGCCTTG | 6EL | 186 | [39] |
| Xedm80 | ATGCTCAAGCCGAGGAAGTA | TTGATGGAGCGACTGAAATG | 6ES | 126, 150 | [39] |
| Xgwm149 | CATTGTTTTCTGCCTCTAGCC | CTAGCATCGAACCTGAACAAG | 6ES | 85 | [39] |
| Xgwm292 | TCACCGTGGTCACCGAC | CCACCGAGCCGATAATGTAC | 6ES | 163 | [39] |
| Xedm158 | CTACGTCGTCTACCGCTGCT | CTTGGGCATCATCGATCTTC | 7E | 120 | [39] |
| Xbarc76 | ATTCGTTGCTGCCACTTGCTG | GCGCGACACGGAGTAAGGACACC | 7EL | 210 | [49] |
| TNAC1825 | GAAGCGGTTCAGGGTGAC | ACCCATCACGTTGCTGTAGTC | 7EL | 750 | [43,45] |
| TNAC1826 | CACATATGATGATGACGGCAAT | GGCAGGGAGGAAACTCTACTG | 7EL | 152.00 | [43,45] |
| TNAC1829 | GCCACTTCCTCCCTCCTC | GTCGGTCCTCCAGTATCAGC | 7EL | 210 | [43,45] |
| TNAC1834 | CCGAGTGTCATCGTTAGGAAA | CCAGCAATCCTACCTGTTGAA | 7EL | 1900 | [43,45] |
| TNAC1845 | AATGAACAGCTTGCTTTCTGC | CAGATGCTCTGGATTTCATGG | 7EL | 120, 90 | [43,45] |
| TNAC1956 | ACGAAGGACAATTGCTGCTAA | GTGCACTTCTTGCCCTACTTG | 7EL | 1074 | [43,45] |
| TNAC1957 | TCAACATTTGCAGGATTGTCA | TTTCACAGGAACCTCTGCATC | 7EL | 170 | [43,45] |
| XBE399084 | CCTACCTACGACGCAAGTCC | AGCGAGCAGAAAGCATCAAG | 7EL | 132 | [13] |
| Xcfa2040 | TCAAATGATTTCAGGTAACCACTA | TTCCTGATCCCACCAAACAT | 7EL | 280, 240 | [49] |
| Xcfa2106 | GCTGCTAAGTGCTCATGGTG | TGAAACAGGGGAATCAGAGG | 7EL | 240 | [49] |
| Xcfa2240 | TGCAGCATGCATTTTAGCTT | TGCCGCACTTATTTGTTCAC | 7EL | 250 | [49] |
| Xedm105 | ACCGCCAGGGAGCTCTGC | GATGTCCTTCTGGCCGTACT | 7EL | 240 | [39] |
| Xedm154 | GGAGAGGCCACTGTTTTTGA | GGCACATCACTCCCTTCTTC | 7EL | 230 | [39] |
| Xgwm130 | AGCTCTGCTTCACGAGGAAG | CTCCTCTTTATATCGCGTCCC | 7EL | 250 | [49] |
| Xgwm295 | CGTACTCCACTCCACACGG | CGGTCCAAGTGCTACCTTTC | 7EL | 288 | [49] |
| Xgwm333 | GCCCGGTCATGTAAAACG | TTTCAGTTTGCGTTAAGCTTTG | 7EL | 300 | [49] |
| Xgwm344 | CAAGGAAATAGGCGGTAACT | ATTTGAGTCTGAAGTTTGCA | 7EL | 130 | [49] |
| Xgwm573 | AAGAGATAACATGCAAGAAA | TTCAAATATGTGGGAACTAC | 7EL | 182 | [49] |
| Xgwm635 | TTCCTCACTGTAAGGGCGTT | CAGCCTTAGCCTTGGCG | 7EL | 130 | [49] |
| Xksum052 | GTCGCTGAGTGCGAACTCCG | CAGAGGAAGCCGTCTACTGG | 7EL | 240 | [49] |
| Xmag1759 | GTACGAGTATGGCAACAGC | CCAACTCTAGGGGCAGATG | 7EL | 620, 250 | [13,43-44] |
| Xmag1932 | TGGCGTCCTGCTGAAAAC | CCGAGCACAAACCCTTGG | 7EL | 285,235 | [13,43-44] |
| Xmag2931 | GGGGGCAGAACTTGTATGAA | TCTTGAGGGACGTGAGGAAC | 7EL | 300 | [13,43-44] |
| Xmag3283 | TGCCTGACCTACTGCAACAC | GAATAGCCATCAACCGCAAG | 7EL | 1820 | [13,43-44] |
| Xpsp3003 | GATCGACAAGGCTCTAATGC | CAGGAGGAGAGCCTCTTGG | 7EL | 200 | [13] |
| Xpsp3123 | AGATTAGATGGCATAAGCAAAGAGC | GATTAATCACCGCCCTTGGTCC | 7EL | 120 | [49] |
| Xpsr680 | GCTCCACTACTTCATCATCC | TGTTTCCTCTATCACTGACTTG | 7EL | 246 | [49] |
| XsdauK107 | AAGACGCAGTGTCTCGAGGT | TGCATCCTCAACATTCTCCA | 7EL | 100 | [13,48] |
| XsdauK116 | TCTGCGCCTTGTATCCTTCT | GGAGTTCCTGGATGTCGTGT | 7EL | 230 | [13,48] |
| XsdauK118 | GTTCAGTTCCACGCCCAATA | ATTATGCGCCGGGCTTCT | 7EL | 170 | [13,48] |
| XsdauK124 | CAGTGCTGCAGGAGCTACAA | AGAACCCTTCGGTGGAAACT | 7EL | 500 | [13,48] |
| XsdauK13 | GCCCTGTTACAGTCCACGAT | GAGCTCCTTGCTCTGATTGG | 7EL | 300 | [13,48] |
| XsdauK130 | TCACACAACCTGCATTGGAT | ATGCCATACGCATTTCCAG | 7EL | 900, 800 | [13,48] |
| XsdauK138 | GCAACCCAGTACGCCTAGTT | CAAATTCGAAAGCCCACATT | 7EL | 400 | [13,48] |
| XsdauK140 | AGGGTCTGTTTGGTTGGATG | AGGCGTGAGAAGTCAATGCT | 7EL | 390 | [13,48] |
| XsdauK142 | GTCTCGTTGCAATTGAGGTG | GAGGCGAGCTGGAAATACAA | 7EL | 375 | [13,48] |
| XsdauK144 | GGGCAGGAAAGACAACACTT | TTCCCAGCTTTGCTCTTCAT | 7EL | 440 | [13,48] |
| XsdauK159 | GTCTCGTTGCAATTGAGGTG | GAGGCGAGCTGGAAATACAA | 7EL | 370 | [13,48] |
| XsdauK165 | ACTGCGTCAGATCCGAAGTT | TGCAGCAATGAAGTCAGGAA | 7EL | 1600 | [13,48] |
| XsdauK176 | AGGAGGCCTTCATGCAGATA | TCGCCATGACTTCCTCTTCT | 7EL | 775 | [13,48] |
| XsdauK27 | TAAGGTGGCTCCGTTGTCTC | CCTCCAGAGCCTCTTTCTCC | 7EL | 465 | [13,48] |
| XsdauK32 | AGGCCGGAGTCGATTCTATT | ATCACAGTGCGCTCATCAAA | 7EL | 250 | [13,48] |
| XsdauK336 | TCATGTATTTGGACAATGTTAAACG | AGACAGCTCCATCGGAAAAA | 7EL | 400 | [13,48] |
| XsdauK339 | CAATTTGCTTCAAGGGGCTA | TTGGGCTCTTCTTCCCCTAT | 7EL | 425 | [13,48] |
| XsdauK340 | ACACTCATGGGAGGACGAAG | GCTACTAGCAATGGCGGAAC | 7EL | 357 | [13,48] |
| XsdauK341 | TCAGTCCCGTCACTGTCAAC | GTCGCCTGCAGGTAGGAGTA | 7EL | 250 | [13,48] |
| XsdauK342 | TTTACCAAGTGGGTGGAAGC | TCCATAAAACGGAAGGCAAC | 7EL | 325 | [13,48] |
| XsdauK343 | ACGAGGGTGGGAAGAAAGAT | AGCCAATTTCGCTGCTTAAA | 7EL | 324 | [13,48] |
| XsdauK345 | GAAACTAGGGGGTCGGGTAG | TCAATTTACCAGGGGCATTC | 7EL | 500, 410 | [13,48] |
| XsdauK348 | GCCAGATAAGAACCCCGTTT | GTGGTAATTTTTAGGGAGTGC | 7EL | 280, 145 | [13,48] |
| XsdauK350 | GAAAGGCAACGAAAATGGAA | GGTACATGATTTCCCCATGC | 7EL | 240 | [13,48] |
| XsdauK351 | AGTCACCCACAAGGACCAAC | TGGTGCCCATCTTCTGGTAT | 7EL | 775 | [13,48] |
| XsdauK352 | CTTTCCAACGCCGTATCATT | ATTGCGCTGACTGTTGTTTG | 7EL | 440 | [13,48] |
| XsdauK353 | GGCAGGCACCATATTGAAGT | TTGCCAACCACGTAGTGAAA | 7EL | 272 | [13,48] |
| XsdauK355 | GCGAGAACAATAGCCTGATTT | TTTTAATGCATGTTCAGCATTTTT | 7EL | 400 | [13,48] |
| XsdauK356 | ATAGGTTTCTCCCGGCTTTG | AAATCTTCACGAATTCAAAACA | 7EL | 140 | [13,48] |
| XsdauK36 | CCCTGCATGAGTTGTCAATG | CATCCTCCAAGGTCATCACA | 7EL | 286 | [13,48] |
| XsdauK38 | GAAGATGGCGAAGGTGTAGC | TTACACTGTCATCGCCGTCT | 7EL | 345 | [13,48] |
| XsdauK4 | CATCAAGTACGGCGTCGAG | ATCGGTCACAAGGCTATTCG | 7EL | 745, 640 | [13,48] |
| XsdauK45 | GGGTTCCTGCTCAAGTTCCT | GGCCTCCTTATGACCACAAA | 7EL | 860 | [13,48] |
| XsdauK6 | CGCGCTCTTCTACTGGTTCT | GTAGAAATCGTGGCCCTTGA | 7EL | 555 | [13,48] |
| XsdauK60 | TGGCGAAGATTTCTTCAAGTC | CTGATCGCACGCTATCATGG | 7EL | 430 | [13,48] |
| XsdauK66 | ATTTGGCTGCGATCGATAAG | CCATCTTGTTGTTGCCCTCT | 7EL | 330 | [13,48] |
| XsdauK71 | GGAGCCAAATCATCCTTGAA | TGTGCTTCAGTTTCGACAGG | 7EL | 440 | [13,48] |
| XsdauK75 | TCTGCACCCTAGTGTTGTGG | GATCAGGATCCAATGCAGGT | 7EL | 520 | [13,48] |
| XsdauK8 | CGAAGCAGAAGCACCTGAAG | CGCTGAATCGATTACGGAAC | 7EL | 985 | [13,48] |
| XsdauK99 | GTGATGGGCAACATGTTCTG | CCGGTAATGGAAACCAAGAA | 7EL | 700, 660 | [13,48] |
| Xsews19 | AAGGCAATGGAGATGTTCG | CCCGTTGATGACCGTGTT | 7EL | 275, 195 | [49] |
| Xwmc273 | AGTTATGTATTCTCTCGAGCCTG | GGTAACCACTAGAGTATGTCCTT | 7EL | 200, 150 | [49] |
| Xwmc702 | GAATCACATCGAATGGATCTCA | GAGGCCTTTTTCGATATTCTGC | 7EL | 194 | [49] |
| Xwmc606 | CCGATGAACAGACTCGACAAGG | GGCTTCGGCCAGTAGTACAGGA | 7EL/7ES | 175 | [49] |
| Xbarc154 | GTAATTCCGGTTCCACTTGACATT | GGATGGGCAGCTTCAAGGTATGTT | 7ES | 520 | [49] |
| Xbarc70 | GCGAAAAACGATGCGACTCAAAG | GCGCCATATAATTCAGACCCACAAAA | 7ES | 100 | [49] |
| Xcfa2049 | TAATTTGATTGGGTCGGAGC | CGTGTCGATGGTCTCCTTG | 7ES | 257 | [49] |
| Xcfa2174 | ACGGCATCACAGGTTAAAGG | GGTCTTTGCACTGCTAGCCT | 7ES | 240 | [49] |
| Xcfd14 | CCACCGGCCAGAGTAGTATT | TCCTGGTCTAACAACGAGAAGA | 7ES | 103 | [49] |
| Xcfd21 | CCTCCATGTAGGCGGAAATA | TGTGTCCCATTCACTAACCG | 7ES | 260 | [49] |
| Xcfd31 | GCACCAACCTTGATAGGGAA | GTGCCTGATGATTTTACCCG | 7ES | 340 | [49] |
| Xcfd66 | AGGTCTTGGTGGTTTTGGTG | TTTTCACATGCCCACAGTTG | 7ES | 119 | [49] |
| Xedm109 | ATGAGGCACAAGTGGATGGT | CTCCTGAAGGGAAAAGCTCA | 7ES | 446 | [39] |
| Xedm16 | TCACCTAACAGCACCACGAG | GCCGAGTACCAGCAGTACCA | 7ES | 195 | [39] |
| Xedm34 | TTGTTTCTTGGCTGGGTTTC | GATCTGCCAAAGCCTTCATAA | 7ES | 150 | [39] |
| Xgwm154 | TCACAGAGAGAGAGGGAGGG | ATGTGTACATGTTGCCTGCA | 7ES | 325 | [49] |
| Xgwm156 | CCAACCGTGCTATTAGTCATTC | CAATGCAGGCCCTCCTAAC | 7ES | 158 | [39] |
| Xgwm350 | ACCTCATCCACATGTTCTACG | GCATGGATAGGACGCCC | 7ES | 160 | [49] |
| Xgwm44 | GTTGAGCTTTTCAGTTCGGC | ACTGGCATCCACTGAGCTG | 7ES | 140 | [49] |
| Xgwm471 | CGGCCCTATCATGGCTG | GCTTGCAAGTTCCATTTTGC | 7ES | 141 | [15] |
| Xgwm473 | TCATACGGGTATGGTTGGAC | CACCCCCTTGTTGGTCAC | 7ES | 230, 200 | [49] |
| Xmag2934 | TGTTCACAAGCGATTCAAGG | AATGCAAGATGGTGGCTTCT | 7ES | 280 | [13,43-44] |
| Xwmc83 | TGGAGGAAACACAATGGATGCC | GAGTATCGCCGACGAAAGGGAA | 7ES | 153 | [49] |
| Xwmc653 | AGTGTTTTAGGGGTGGAAGGGA | CGGAACCCTAAACCCTAGTCG | 7ES | 190 | [49] |
| Xwmc809 | CAGGTCGTAGTTGGTACCCTGAA | TGAACACGGCTGGATGTGA | 7ES | 360 | [49] |
新窗口打开
The authors have declared that no competing interests exist.
