0 引言
【研究意义】糜子(Panicum miliaceum L.)又叫黍稷,为旱地农业重要经济作物。糜子在亚洲中部和东部、印度、非洲、中东、东欧(俄罗斯和乌克兰)和北美均有分布[1]。糜子脱壳后称黄米,糯质黄米可用于蒸糕、炸油糕、包粽子和酿酒,粳质黄米多用于制作凉粉、煎饼、黄米酸饭和炒米[2]。糜子在欧美主要用于饲鸟和禽畜育肥。糜子作为C4植物,蒸腾速率低,在干旱、高温等恶劣条件下快速成熟,是干旱、半干旱地区的稳产作物。中国是糜子起源地,资源丰富,明确不同种质的遗传背景,准确评估糜子遗传多样性,有利于糜子作物的合理开发和高效利用。【前人研究进展】分子标记是评估作物遗传差异和鉴定群体间亲缘关系远近的有效手段。由于糜子是异源四倍体,基因组复杂,序列信息缺乏,分子标记的开发相对欠缺。1994—2006年相继开发的适用于糜子多样性分析的分子标记(RAPD、AFLP、RFLP、ISSR等)非常有限[3-6],尤其是变异丰富、多态性高、重复性好、检测能力强[7]的SSR标记更少。2008—2014年,HU等[8]和RAJPUT等[9]分别开发了46个和254个来自小麦、水稻、燕麦和柳枝稷的糜子非特异性微卫星标记,然而种间差异的存在影响遗传多样性评估准确性。2010年,CHO等[10]首次开发了糜子特异性微卫星标记25个,随后利用上述标记评估糜子遗传资源差异的研究渐次进行。2011年,HUNT等[11]利用其中16对引物分析了欧亚大陆的98份糜子资源,检测到78个等位基因,每个位点检测到等位变异为2—15个(平均4.9个),基因多样性指数和多态性信息含量(polymorphism information content,PIC)分别为0.391和0.360;2015年,连帅等[12]利用5对SSR引物分析来源于5个糜子生态区的40份资源的多样性,检测到15个等位变异,平均为3个,遗传多样性指数和PIC值分别为0.76和0.48;董俊丽等[13]利用其中19对引物检测96份糜子种质,发现112个等位变异,基因多样性指数和PIC值分别为0.4097和0.392。2016年,刘笑瑜等[14]选用其中6对SSR引物分析中国不同省份的40份糜子资源,发现20个等位变异,遗传多样性指数和PIC值分别为0.5426和0.3403;2017年,王瑞云等[15]用15对引物,评估不同生态区132份糜子的遗传多样性,共检测到107个等位变异,每个位点鉴定到的2—14个(平均7个),基因多样性指数和PIC值分别为0.5298和0.4864。近年来,随着高通量测序技术的广泛运用,全基因组SSR标记批量挖掘取得长足进展。2016年,RAJPUT等[16]用100个SSR(80个来自柳枝稷、7个来自水稻、6个为糜子、5个来自小麦、2个来自燕麦)评估了欧亚和北美等25个国家的90份糜子资源,检测到1 287个等位变异(长度为40—1 500 bp),每个位点检测到等位变异1—29个(平均4.9个),每份材料检测到等位变异41—317。同年,LIU等[17]和连帅等[18]用糜子高通量测序开发的130条SSR引物分析了280份国内外的糜子栽培品种和野生资源。【本研究切入点】糜子基因组序列未知,可供遗传分析的SSR标记数量有限,且以往研究所用标记多为低基元(二、三核苷酸)序列重复。【拟解决的关键问题】基于RNA-Seq手段筛选到一批高基元(四、五、六核苷酸)序列重复SSR,并用以评估糜子遗传多样性,以期为糜子连锁图谱构建及进化研究提供更多分子检测工具,批量筛选高基元微卫星标记,明确引物分辨率特征。1 材料与方法
1.1 供试材料
试验材料包括96份种质资源,来源于国家资源库的地方品种(有国家种质资源库统一编号)76份、笔者实验室征集的地方品种(标注为农家种,无国家种质资源库统一编号)18份和育成品种2份(附表1),分布于6个糜子生态栽培区(表1)。Table 1
表1
表1糜子资源生态区分布
Table 1Ecotope of common millet in this study
| 生态区 Ecotope | 来源 Origin | 数量 Accession number |
|---|---|---|
| 南方秋冬糜子区 Southern autumn & winter-sowing ecotope (SAW) | 海南 Hainan | 1 |
| 西北春夏糜子区 Northwest spring & summer-sowing ecotope (NWSS) | 新疆 Xinjiang | 2 |
| 北方春糜子区 Northern spring-sowing ecotope (NSP) | 青海 Qinghai | 13 |
| 甘肃 Gansu | 10 | |
| 内蒙古 Inner Mongolia | 10 | |
| 山西 Shanxi | 6 | |
| 黄土高原春夏糜子区 Loess Plateau spring & summer-sowing ecotope (LPSS) | 山西 Shanxi | 14 |
| 陕西 Shaanxi | 8 | |
| 宁夏 Ningxia | 10 | |
| 东北春糜子区 Northeast spring-sowing ecotope (NES) | 黑龙江 Heilongjiang | 5 |
| 吉林 Jilin | 3 | |
| 辽宁 Liaoning | 1 | |
| 华北夏糜子区 Northern summer-sowing ecotope (NSU) | 河北 Hebei | 9 |
| 山东 Shandong | 2 | |
| 江苏 Jiangsu | 1 | |
| 河南 Henan | 1 | |
| 合计 Total | 96 |
新窗口打开
1.2 表型性状测量
96份糜子资源分别于2015年5月12日和2016年5月14日种植在山西农业大学农作站,分5行点播,每行10株,株距5 cm,行距8 cm,小区间距50 cm。参照王星玉等[19]的方法,成熟期测量表型性状(株高、穗长、分蘖数、主茎节数、千粒重、叶长、叶宽)。1.3 基因组DNA提取
取15—20 d糜子幼苗(1株)叶片约0.3 g,用改良CTAB法[20]提取基因组DNA。利用1%琼脂糖凝胶电泳检测DNA质量,用紫外微量核酸仪(NanoDropND-1000,Eppendorf)测定DNA纯度和浓度。
1.4 SSR分析
用地理来源差异显著的8份糜子材料对前期抗旱和抗盐相关转录组测序获得的151个高基元SSR引物进行初步筛选(附表2和附表3)。85个标记可以扩增出清晰度高且重复性好的多态性片段(附表4,由上海生工生物工程技术服务有限公司合成),用于评估96份糜子资源遗传多样性。PCR反应体系(20 μL)包括10×buffer(Mg2+含量25 mmol·L-1)2 μL、10 mmol·L-1 dNTP 1.8 μL,5 U·L-1 Taq聚合酶0.4 μL、1 mmol·L-1前后引物各0.6 μL、ddH2O 13.6 μL和30 ng·μL-1 DNA模板1 μL。PCR扩增在LifeEco基因扩增仪(TC-96/G/H(b)c,杭州博日科技有限公司)上进行,程序为94℃ 5 min;94℃ 45 s,不同Tm退火50 s,72℃ 1 min,38个循环;72℃ 10 min。用8%聚丙烯酰胺凝胶电泳检测PCR扩增产物,硝酸银染色显影。等位变异长度用50 bp DNA Marker(北京庄盟国际生物基因科技有限公司)比对条带。
1.5 数据分析
用SAS软件对各性状进行主成分和相关性分析。利用模糊隶属函数法计算各性状的隶属函数值以进行综合评价及Shannon-wiener遗传多样性指数评价,方法参考董俊丽等[13]。
凝胶条带表示标记(>50 bp)大小,相邻条带大小相差25 bp。SSR标记如果在至少2份材料扩增出不同的DNA条带,则具有多态性。DNA条带代表等位变异,相同条带表示相同等位变异。引物分辨率(resolving power,Rp)计算按照PREVOST等[21]方法,Rp = ∑ Ib,其中,Ib=1-(2×︱0.5 - p︱),Ib为某个等位基因信息量,p为某个等位基因在6份材料中出现的频率。用PowerMarker 3.25[22]计算每对引物的多样性参数,包括等位基因数(Na)、等位基因频率、基因多样性指数(H)、多态性信息含量指数(PIC)等。用PopGen1.32[23]进行群体间聚类分析。用MEGA 5.0[24]构建Neighbour-Joining聚类图。用Structure 2.2[25]分析群体遗传结构。
2 结果
2.1 糜子表型遗传多样性分析
对96份糜子资源的7个表型性状进行了表型多样性分析(表2),7项指标变异系数为18.21%—42.19%,分蘖数最大,主茎节数和株高次之,叶长最小,说明糜子不同种质分蘖数相差较大。糜子7项指标遗传多样性指数为1.78—2.08,株高最大,说明株高变异丰富。Table 2
表2
表2糜子表型性状多样性分析
Table 2Diversity analysis of phenotypic traits in common millet
| 性状 Trait | 范围 Range | 平均值±标准差 Mean±SD | 变异系数 CV (%) | Shannon 指数 Shannon index (H') |
|---|---|---|---|---|
| 株高 Plant height (PH, cm) | 73.23—257.70 | 178.63±42.14 | 23.59 | 2.08 |
| 穗长 Panicle length (PL, cm) | 14.67—60.17 | 43.10±9.06 | 21.01 | 1.91 |
| 分蘖数 Tiller number (TN) | 0.67—11.67 | 3.30±1.39 | 42.19 | 1.83 |
| 主茎节数 Nodes per main stem (NMS) | 3.33—11.33 | 7.31±1.84 | 25.12 | 1.85 |
| 千粒重 Thousand of kernel weight (TKW, g) | 2.15—7.01 | 5.22±1.06 | 20.27 | 1.81 |
| 叶长 Leaf blade length (LBL, cm) | 15.50—52.50 | 40.19±7.32 | 18.21 | 1.78 |
| 叶宽 Leaf blade width (LBW, cm) | 1.20—3.70 | 2.48±0.54 | 21.63 | 1.89 |
新窗口打开
2.2 糜子表型性状综合分析
对96份糜子资源的表型性状进行相关性分析(表3)。21对相关性中,13对(61.9%)呈正相关,8对呈负相关,7对(33.3%)极显著相关,1对(千粒重和分蘖数)显著相关。其中,株高与主茎节数相关系数最大(0.601),其次为叶长与叶宽(0.561),说明株高越高,主茎节数越多,叶长越长,叶宽也越大。分蘖数和主茎节数极显著负相关(-0.216),说明主茎节数越少,分蘖数越多。对96份糜子资源进行主成分分析(表4)。结果表明,前5个主成分累积贡献率为91.51%,包含表型特征多数信息,可用以综合分析糜子资源。其中,第一主成分贡献率33.29%,株高载荷值最大,第一主成分值大的资源,株高较高。第二主成分和第五主成分均为分蘖数载荷值最大,累积贡献率29.36%,说明第二和第五主成分值大的资源分蘖数多;同时第五主成分穗长和千粒重均为负值,且绝对值较大,育种中选择第五主成分小的种质,分蘖数少,穗长和千粒重大。第三主成分千粒重、株高和穗长载荷值均较大,为产量相关性状,在育种中可选择第三主成分值大的种质。第四主成分千粒重、叶长和叶宽贡献率均较大,株高、穗长为负值,绝对值较大,说明第四主成分值高的种质,千粒重大、叶片长而宽,但植株低、穗短,可用于矮杆、高产育种的选育。
Table 3
表3
表3糜子表型性状间的相关性分析
Table 3Correlation of phenotypic traits in common millet
| 性状 Trait | 株高 PH | 穗长 PL | 分蘖数 TN | 主茎节数 NMS | 千粒重 TKW | 叶长 LBL | 叶宽 LBW |
|---|---|---|---|---|---|---|---|
| 株高 PH | |||||||
| 穗长 PL | 0.453** | ||||||
| 分蘖数 TN | -0.076 | 0.064 | |||||
| 主茎节数 NMS | 0.601** | 0.026 | -0.216** | ||||
| 千粒重 TKW | -0.082 | -0.041 | 0.086* | -0.006 | |||
| 叶长 LBL | 0.244** | 0.395** | 0.008 | 0.141 | -0.132 | ||
| 叶宽 LBW | 0.333** | 0.086 | -0.100 | 0.310 | -0.119 | 0.561** |
新窗口打开
Table 4
表4
表4表型性状5个主成分的特征向量、主成分特征值、贡献率及累积贡献率
Table 4Power vector (PV), eigen values (E), contribution rate (CR) and cumulative contribution (CCR) of the first five principal components based on 7 phenotypic traits
| 项目 Item | PV1 | PV2 | PV3 | PV4 | PV5 |
|---|---|---|---|---|---|
| 株高 PH | 0.5164 | -0.1315 | 0.3797 | -0.2433 | 0.1716 |
| 穗长 PL | 0.3446 | 0.4744 | 0.3192 | -0.4103 | -0.3879 |
| 分蘖数 TN | -0.1212 | 0.5817 | 0.2831 | 0.1683 | 0.7299 |
| 主茎节数 NMS | 0.4144 | -0.5133 | 0.2486 | 0.0601 | 0.3039 |
| 千粒重 TKW | -0.1377 | -0.0345 | 0.6397 | 0.6461 | -0.3854 |
| 叶长 LBL | 0.4468 | 0.3928 | -0.2821 | 0.2796 | -0.1953 |
| 叶宽 LBW | 0.4578 | 0.0162 | -0.3510 | 0.4949 | 0.0913 |
| 主成分特征值 E | 2.3305 | 1.2571 | 1.0908 | 0.9299 | 0.7981 |
| 贡献率 CR (%) | 33.29 | 17.96 | 15.58 | 13.28 | 11.40 |
| 累积贡献率 CCR (%) | 33.29 | 51.25 | 66.83 | 80.12 | 91.52 |
新窗口打开
分析糜子资源表型性状隶属值分布(图1)。株高主要集中在4—9级,占材料的84.38%;穗长主要集中在5—8级,占材料的77.08%;分蘖数主要集中在2—4级,占材料的84.38%;主茎节数主要集中在3—8级,占材料的82.29%;千粒重主要集中在5、6、8和10级,占材料的68.75%,叶长主要集中在6—10级,占材料的80.12%,叶宽主要集中在4—8级,占材料的76.04%。综合评价各试验材料隶属函数平均值,发现甘肃省的榆中黄糜(00002787)的值最高(0.738),综合评价最好;山西孝义黄糜子值最低(0.231),综合评价最低。
2.3 高基元SSR引物筛选
山西农业大学农学院糜子分子育种课题组前期基于RNA-Seq筛选到151对高基元SSR引物,用这些引物扩增6份糜子材料。发现25对扩增不出DNA条带,126对(83.4%)有扩增条带。126对引物中,41对扩增的条带呈单态性,85对(附表4)呈多态性,占151对引物的56.3%。其中四、五和六碱基重复引物分别为71对(83.5%)、10对(11.8%)和4对(4.7%)。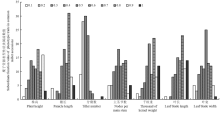 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1糜子资源表型性状隶属值分布
-->Fig. 1Distribution diagram of subordinate function values of phenotypic traits
-->
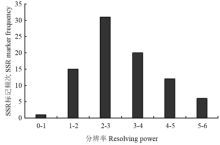
引物分辨率(Rp值)是衡量引物对不同基因型辨别能力的指标。85对高基元SSR的引物特征(等位基因大小和Rp值)见附表5和图2。从附表5可以看出,85个SSR的等位基因为100—450 bp;85个SSR的Rp值为1.00—5.75(平均3.15),标记RYW42的Rp值最小,标记RYW47的Rp值最大;四、五和六碱基重复SSR的平均Rp值分别为3.15、2.8和4.0,六碱基重复SSR数量最少(4个),但Rp值较高,可能与其重复单元差异较大有关。基于Rp值分析85个SSR的分布频次(图2),发现频次分布区间为0—1、1—2、2—3、3—4、4—5和5—6,分别包含1(1.18%)、15(17.65%)、31(36.47%)、20(23.53%)、12(14.12%)和6(7.06%)个标记,60%(51个)的标记分布在区间2—3和3—4。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图285个糜子多态性SSR标记的Rp值
-->Fig. 2Resolving power values of 85 polymorphic SSR markers in common millet
-->
2.4 基于高基元SSR分析糜子资源遗传多样性
用85对SSR引物扩增96份试材,分析糜子资源遗传多样性(附表6)。从附表6可以看出,85个标记在96份试材中共检出232个等位变异,每个位点检测到等位变异2—3个,平均为2.7294个;62个位点产生3个变异,23个SSR位点产生和2个变异。85个位点多样性指数为0.2842(RYW75)—1.0633(RYW37),平均为0.7708。85个位点PIC值为0.1133(RYW46)—0.7281(RYW8),平均为0.4723。分析不同生态区糜子资源的遗传多样性(表5),结果发现,北方春糜子区PIC值、多样性指数及观测等位基因数等遗传多样性衡量指标最高,黄土高原春夏糜子区次之,南方秋冬糜子区均最低,说明北方春糜子区和黄土高原春夏糜子区遗传多样性比较丰富。
2.5 不同生态区糜子种质间遗传相似性分析
利用PopGen 1.32计算不同生态区糜子种质间的遗传相似性(表6)。结果表明,遗传距离为0.0093—0.5052(平均0.1798)。遗传一致度为0.6034—0.9907(平均0.8485)。南方秋冬糜子区与西北春夏糜子区遗传距离最大,遗传一致度最低,这与2个生态区地理分布较远、气候环境差别大、亲缘关系较远有关;北方春糜子区和黄土高原春夏糜子区遗传距离最小,遗传一致度最高,这与2个生态区相邻、气候差异小、亲缘关系较近有关。说明遗传距离越大,亲缘关系越远,遗传相似性越低。Table 5
表5
表5不同生态区糜子的遗传多样性分析
Table 5Evaluation of genetic diversity of different ecotopes of common millet
| 来源地 Origin | 资源数 Accessions | 观测等位基因 Observed allele number | 有效等位基因 Effective allele number | 多样性指数 Gene diversity | 观测杂合度 Observed heterozygosity | 期望杂合度 Expected heterozygosity | 多态性信息含量 PIC |
|---|---|---|---|---|---|---|---|
| 南方秋冬糜子区 SAW | 1 | 1.6235±0.4874 | 1.6235±0.4875 | 0.4322±0.3378 | 0.3765±0.4874 | 0.6235±0.4874 | 0.3047 |
| 西北春夏糜子区 NWSS | 2 | 1.8824±0.3241 | 1.7647±0.3326 | 0.5731±0.2184 | 0.3000±0.3716 | 0.5392±0.2115 | 0.3219 |
| 北方春糜子区 NSP | 39 | 2.6118± 0.4902 | 2.0201±0.2796 | 0.7605±0.1334 | 0.3391±0.1504 | 0.5018 ±0.0732 | 0.4573 |
| 黄土高原春夏糜子区 LPSS | 32 | 2.5176± 0.5258 | 1.9950±0.3158 | 0.7415±0.1618 | 0.3893±0.1955 | 0.4924±0.0961 | 0.4327 |
| 东北春糜子区 NES | 9 | 2.2235±0.4191 | 1.9423±0.3040 | 0.6950±0.1585 | 0.3634±0.2702 | 0.4984±0.1050 | 0.3491 |
| 华北夏糜子区 NSU | 13 | 2.4824±0.5027 | 2.0336±0.4014 | 0.7562±0.1807 | 0.3982±0.2314 | 0.5082±0.1089 | 0.4002 |
新窗口打开
Table 6
表6
表6不同生态区糜子资源的遗传距离与遗传一致度
Table 6Genetic distance and genetic identity of common millet resources with different ecotopes
| 生态区 Ecotope | 南方秋冬糜子区 SAW | 北方春糜子区 NSP | 西北春夏糜子区 NWSS | 黄土高原春夏糜子区 LPSS | 东北春糜子区 NES | 华北夏糜子区 NSU |
|---|---|---|---|---|---|---|
| 南方秋冬糜子区 SAW | 0.9278 | 0.6034 | 0.9197 | 0.8846 | 0.9017 | |
| 北方春糜子区 NSP | 0.075 | 0.6687 | 0.9907 | 0.9739 | 0.9608 | |
| 西北春夏糜子区 NWSS | 0.5052 | 0.4024 | 0.6701 | 0.6471 | 0.6692 | |
| 黄土高原春夏糜子区 LPSS | 0.0837 | 0.0093 | 0.4004 | 0.977 | 0.9599 | |
| 东北春糜子区 NES | 0.1227 | 0.0265 | 0.4352 | 0.0233 | 0.9735 | |
| 华北夏糜子区 NSU | 0.1035 | 0.04 | 0.4017 | 0.041 | 0.0269 |
新窗口打开
2.6 基于UPGMA的糜子资源聚类分析
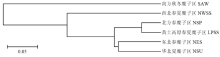
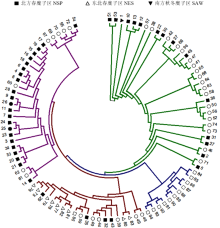
基于UPGMA对不同生态区糜子进行聚类(图3),发现6个生态区资源在遗传距离0.1827处划分为4个类群(类群Ⅰ、Ⅱ、Ⅲ和Ⅳ)。类群Ⅰ将南方秋冬糜子区归为一类,试材来自海南。类群Ⅱ将西北春夏糜子区归为一类,试材来自新疆。类群Ⅲ将北方春糜子区和黄土高原春夏糜子区归为一类,试材来自青海、甘肃、内蒙古、山西、陕西和宁夏。类群Ⅳ将东北春糜子区和华北夏糜子区归为一类,试材来自黑龙江、吉林、辽宁、河北、山东、江苏和河南。基于UPGMA对96个糜子基因型进行聚类(图4),发现96份试材归为4个群组,第一群组30份,包括山西2份、陕西3份、宁夏4份、内蒙古3份、青海7份,甘肃9份、新疆2份,大部分基因型属于北方春糜子区和黄土高原春夏糜子区;第二群组20份,其中内蒙古3份、山西6份、黑龙江5份、吉林3份及陕西、辽宁和江苏各1份,主要属于东北春糜子区;第三群组11份,包括河北9份、山东2份,属于华北夏糜子区;第四群组35份,包括青海6份、甘肃1份、内蒙古4份、山西12份、陕西4份、宁夏6份、海南和河南各1份,主要属于黄土高原春夏糜子区和北方春糜子区。各群组分布与地理来源基本相符。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3基于遗传距离的不同生态区糜子资源聚类图
-->Fig. 3Cluster diagram of common millet with different ecotopes based on genetic distance
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4基于遗传距离的96份糜子种质资源聚类图
-->Fig. 4Cluster diagram of 96 accessions of common millet based on genetic distance
-->
2.7 基于模型的群体结构分析
对96份试材进行基于模型的群体结构分析(图5)。从图5可以看出,等位变异频率特征数(遗传群体数)在K=2、K=4和K=9处峰值明显,基于K=4和K=9峰值基本相同,且Delta K越大且K值越小模拟结果越可靠,仅对K=2和K=4进行分析,分别将试材划分为2和4个群组(图6)。由图6可知,K=2时,划分为2个类群,类群Ⅰ为红色(60份),主要来自于北方春糜子区和黄土高原春夏糜子区;类群Ⅱ为绿色(36份),主要来自于北方春糜子区和黄土高原春夏糜子区;K=4时,划分为4个类群,类群Ⅰ为红色(19份),代表黄土高原基因库,多样性指数和PIC值均最高;类群Ⅱ为绿色(22份),代表东北和华北基因库;类群Ⅲ为蓝色(20份),代表黄土高原基因库和北方基因库;类群Ⅳ为黄色(35份),代表黄土高原基因库和北方基因库。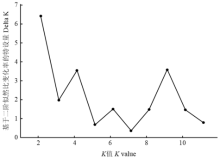 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5运用 Structure对糜子基因库数目建模
Delta K根据EVANNO等[
-->Fig. 5Modelling of number of gene pools in common millet using Structure
Delta K was calculated by the method of EVANNO et al.[
-->
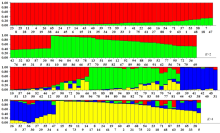 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6糜子种质资源群体遗传结构图
横坐标的数字代表糜子材料序号
-->Fig. 6Population genetic structure graph of common millet germplasm resources
Numbers in the horizontal axis represent serial number of accession
-->
K=2红色类群分化为红、绿、蓝和黄色(K=4),14份山西糜子中1份(7.14%)为红色,蓝色和黄色分别为10份(71.43%)和3份(21.43%)。10份青海糜子中1份(10%)属于蓝色,9份(90%)属于黄色。
K=2绿色类群半数以上材料没分化,其余分化为红色和蓝色(K=4),6份山西材料为红色,8份河北材料中,绿色和红色分别为7份(87.5%)和1份(12.5%)。
分析K=2和K=4遗传结构图各分类群的遗传多样性参数(表7),结果发现,K=2时,红色类群和绿色类群分别占62.5%和37.5%;就多样性指数和PIC值而言,绿色类群(分别为0.7726和0.4845)高于红色类群(分别为0.7499和0.4379)。绿色类群资源主要来自于河北、宁夏和山西,地区间相距较远,气候条件差异大,资源间遗传多样性丰富;红色类群糜子主要来自于山西、甘肃和内蒙古,这些地区彼此接壤,气候相似,资源间亲缘关系近,遗传一致性程度高。K=4时,黄色类群资源最多(53.03%);红色类群的PIC值和多样性指数均为最大(分别为0.4868和0.7627)。
Table 7
表7
表7遗传结构图中各分类群的多样性统计
Table 7Diversity statistics by structure clusters
| 群组 Cluster | 资源份数 Sample size | 观测等位基因 Observed allele number | 有效等位基因 Effective allele number | 多样性指数 Gene diversity | 观测杂合度 Observed heterozygosity | 期望杂合度 Expected heterozygosity | 多态性信息含量 PIC |
|---|---|---|---|---|---|---|---|
| K=2 | |||||||
| 红色 Red | 60 | 2.6235 | 2.0036 | 0.7499 | 0.3314 | 0.4967 | 0.4379 |
| 绿色 Green | 36 | 2.6235 | 2.0492 | 0.7726 | 0.4248 | 0.5038 | 0.4845 |
| K=4 | |||||||
| 红色 Red | 19 | 2.6000 | 2.0184 | 0.7627 | 0.4557 | 0.5009 | 0.4868 |
| 绿色 Green | 22 | 2.4118 | 2.0049 | 0.7388 | 0.3882 | 0.4973 | 0.4283 |
| 蓝色 Blue | 20 | 2.4588 | 1.9904 | 0.7364 | 0.3618 | 0.4939 | 0.4068 |
| 黄色 Yellow | 35 | 2.5059 | 1.9746 | 0.7299 | 0.3069 | 0.4924 | 0.4042 |
新窗口打开
3 讨论
3.1 高基元糜子特异性SSR是分析糜子遗传多样性的有效标记
基于SSR引物初步筛选,983个种间SSR中,209个(38%)扩增不出条带[9];本研究用PCR扩增151个标记,发现25个(16.56%)无结果,这些SSR可能受到基序插入、缺失、点突变、侧翼重复缺失等因素影响,引起DNA电泳条带不出现[27]。用983个水稻等种间SSR扩增118份糜子资源,46个标记(4.7%)扩增出多态性片段[8]。用548个柳枝稷SSR扩增8份糜子材料,254个标记(46.4%)扩增片段具多态性[9]。本研究用151个SSR扩增6份糜子材料,85个标记(56.3%)扩增出多态性片段,明显高于上述研究结果,这可能与引物来自糜子基因组有关,糜子特异性标记筛选效果优于种间标记。
转录组测序是开发SSR标记的有效手段,用糜子高通量测序获得的500对SSR引物扩增8份糜子材料,162个标记(32.4%)扩增出多态性片段[17],低于本研究结果,可能与本研究引物为高基元有关。本研究首次利用RNA-Seq开发了一批高基元糜子微卫星引物,为糜子育种提供了有效分子检测工具。
Rp值(引物分辨率)反映SSR多态性,直接关联标记信息[21],Rp值越高,遗传多样性越丰富。以往研究发现254和100个糜子非特异性SSR(6个为糜子细菌人工染色体克隆除外)的Rp值分别为2.71和5.43[9,16],与本研究结果(3.15)基本一致;同时发现,糜子分子标记的Rp值和苎麻[28]的研究结果(3.22)类似,低于密花石斛(6.15)、柽麻(6.59)和穿心莲(10.8)[29-31]。Rp值与糜子种特异性/非特异性引物的相关性大小有待于进一步研究。本研究首次构建的一批高基元SSR,不仅丰富了糜子作物DNA标记,而且也为糜子连锁图谱构建和基因组功能研究提供了理论依据。
高基元SSR多态性低于低基元多态性,小麦中已有报道[32],糜子中也检测到类似结果。用低基元微卫星标记分析糜子遗传多样性,192、132和118份资源的PIC值分别为0.4855[18]、0.5298[15]和0.793[8],本研究高基元SSR检测结果为0.4723,明显低于上述结果。
迄今,糜子中可用的高级基元SSR极少,仅包括1个糜子种特异性SSR[10,13]和3个种间SSR[9]。其中,糜子特异性标记EF117731由CHO等[10]开发,为四碱基重复“(AGTC)4”。CHO等[10]用该标记检测50份糜子材料,发现期望杂合度为0.04,PIC值为0.04;董俊丽等[13]用该标记检测评估96份糜子的遗传差异,发现基因多样性指数为0.2112,PIC值为0.2035;与上述研究类似,本研究也筛选到一个四碱基重复引物“(AGTT)6”,遗传多样性分析发现期望杂合度(0.4575)、基因多样性指数(0.6500)和PIC值(0.3226)均高于上述研究结果,这可能与本研究所选试材地理来源差异大、资源丰富有关。
3.2 中国糜子资源的遗传多样性和群体结构
用RAPD标记分析5个黍属植物遗传多样性,发现糜子多态性高,且划分类群与地理来源一致[3]。用RFLP标记比较12份栽培和野生糜子资源,发现UPGMA聚类结果与地理来源无关[5]。上述相互冲突的结果可能与材料来源及检测标记不同有关。本研究用85个高基元SSR扩增96份试材,发现多样性指数(0.7708)和PIC值(0.4723)均高于RAPD和低基元SSR的标记鉴定结果(最高分别为0.725和0.3920)[3,10-15,17-18],这可能与本研究试材来源广泛(6个生态区)、多样性丰富有关,也可能与高基元引物有关。基于遗传结构欧亚大陆糜子资源划分群组与地理来源相符[11],基于遗传距离美国糜子资源划分群组与地理来源、系谱发生及农艺特征相符[16],基于遗传结构和遗传距离中国糜子资源聚类群组与地理来源相关[13,15]。本研究基于UPGMA将6个生态区及96个基因型均划分为4个主要类群,分别属于北方、黄土高原、东北和华北糜子区;遗传结构分析将96个基因型划归4个主要的基因库:北方、黄土高原、东北和华北基因库。遗传距离和遗传结构聚类结果基本一致,均与地理起源相关,表明不同生态区内遗传变异广泛,糜子不同群体间遗传关系复杂[17]。本研究西北基因库和南方基因库资源选材偏少、青藏高原基因库资源缺乏,未来研究需丰富材料来源,有利于准
确评价中国糜子资源的遗传多样性。
4 结论
在糜子中开发了85个四、五和六碱基重复微卫星标记,分析这些SSR的引物分辨率(Rp),发现信息量大、多态性高。运用该高基元分子检测系统评估中国糜子资源的遗传差异,发现聚类群组与地理起源相关,其中北方春糜子区和黄土高原春夏糜子区遗传多样性最丰富。The authors have declared that no competing interests exist.
