0 引言
【研究意义】柑橘衰退病毒(Citrus tristeza virus,CTV)引起的柑橘衰退病是具有经济重要性的柑橘病害,在世界各柑橘产区均有危害[1-4]。明确不同寄主中CTV强弱毒株系 p20 基因片段的变异情况,对于了解CTV致病机理以及防治柑橘衰退病均有重要意义。【前人研究进展】对中国11个省(市)柑橘主产区近4 000余份柑橘样品的检测结果表明,近1/3的柚类样品和几乎所有的甜橙、宽皮柑橘和杂柑样品均感染了CTV[5]。CTV主要引起酸橙砧木柑橘的快速死亡或严重衰退,木质部产生茎陷点症状,进而导致树势减弱、果品品质变差,亦可引起酸橙、葡萄柚和柠檬实生苗的苗黄症状[6]。由于致病力和寄主品种的不同,CTV侵染引起的症状差异很大[7]。CTV的基因组为19 226—19 296 nt的正义单链RNA,包装于2 000 nm×11 nm的螺旋对称的线形粒体中,可翻译表达19种蛋白质[8]。CTV可编码3个RNA沉默抑制子,其中p20编码的蛋白为其中之一[9]。对病毒编码RNA沉默抑制子的研究表明,其可通过干扰基因沉默的起始、siRNA的积累或干扰系统性基因沉默等方式抑制寄主的基因沉默,进而影响病毒在植株体内的复制和表达,与病毒致病性密切相关[10-12]。CTV株系对不同柑橘类植株的适应性存在差异,甜橙可能更适宜于CTV的增殖,不同株系之间的序列差异可能与其在不同类柑橘植株上的适应性有关[13-14]。在柚类中检测到的CTV株系相对较单一[15]。将保存在新会橙上的引起甜橙茎陷点症状的CTV强毒株系嫁接接种到风凰柚后发现,某些能在甜橙上增殖的CTV株系在凤凰柚上的增殖受到了抑制,表明不同柑橘类寄主植物与CTV株系间可能存在相互作用[16]。此外,AYLLÓN等[17]的研究也表明,寄主影响CTV种群结构,在CTV进化中扮演了重要的角色。【本研究切入点】作为CTV编码的3个RNA沉默抑制子之一,p20蛋白对其致病性具有重要的作用,但具体分析寄主甜橙和柚中CTV强弱毒株系p20种群变异差异的文献国内外尚不多见。【拟解决的关键问题】建立CTV p20种群并对其种群结构及变异进行分析,明确寄主对CTV不同株系p20种群变异的影响,为深入开展该病毒病害的防控研究提供依据。1 材料与方法
试验于2012—2015年在西南大学植物保护学院植物病毒实验室完成。1.1 材料
1.1.1 试验材料 甜橙品种北碚-447、纽荷尔脐橙、佩拉甜橙和柚品种琯溪蜜柚、三台台北柚、脆香甜柚,共包含7个已经鉴定致病力的CTV株系,甜橙为实生苗,柚分离株保存在强得勒柚为砧木的琯溪蜜柚上。其中CTV强毒株TR-L514(北碚-447)、CT3(三台台北柚)、CT22(脆香甜柚)、CT23(琯溪蜜柚),以及CTV弱毒株CT9(琯溪蜜柚)、CT31(纽荷尔脐橙)由中国农业科学院柑桔研究所脱毒中心提供。弱毒株PeraIAC(佩拉甜橙)由巴西Centro de Citricultrua Sylvio Moreira 研究所Marcos A Machado 博士赠送。1.1.2 试剂、克隆载体及菌株 RNA提取试剂盒购自Tiangen公司,PowerScript ⅡTM反转录酶试剂盒及PrimeStar DNA高保真性聚合酶购自TaKaRa公司,DNA纯化回收试剂盒购自Tiangen公司,PGEM-T Easy Vector SystemⅠ购自Promega公司,Escherichia coli DH5α为西南大学植物病毒实验室保存,BM2000+ DNA Maker、Taq DNA聚合酶购自BioFlux,所用引物由深圳华大基因科技服务有限公司合成。
1.2 方法
1.2.1 RNA的提取与纯化 采用多糖多酚植物RNA提取试剂盒进行柑橘叶片总RNA的提取纯化。1.2.2 p20扩增及回收 以RNA为模板,采用反转录试剂盒进行cDNA合成,以合成的cDNA为模板用特异性引物F 5′-ACAATATGCGAGCTTACTTTA-3′及R 5′-AACCTACACGCAAGATGGA-3′进行PCR扩增[18]。为保证扩增基因片段的高保真性,对反应体系和条件均进行了优化。优化后的反应体系:ddH2O 14.5 μL,5×buffer 5.0 μL,dNTPs 2.0 μL,F/R各1 μL,cDNA 1 μL,以及高保真性的Prime Star DNA聚合酶(2.5 U·μL-1)0.5 μL。优化后的反应条件:94℃预变性3 min;然后20个循环(98℃变性10 s,54℃退火15 s,72℃延伸90 s);最后72℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测,确认目标条带后即纯化回收目标DNA片段。
1.2.3 p20种群建立及变异分析 通过PGEM-T Easy Vector SystemⅠ将目标片段连接到载体后转入E. coli DH5α,菌液PCR筛选阳性克隆,随机选取至少20个阳性克隆测序以构建相应的CTV p20种群,参照任芳等[19]的方法进行种群结构及变异的分析,以每个种群的优势序列作为标准序列,将所得序列与之进行比较,每一个发生改变的核苷酸视为一个单一突变,分别计算所建立种群的突变克隆百分比(突变克隆的总数与总克隆数的比值)和碱基突变频率(突变的碱基总数与测序的碱基总数的比值),以此作为CTV p20种群遗传多样性和种群变异水平的指标。同时,以提取的含p20的质粒为模板进行相同条件的PCR扩增,以此作为对照种群,对Prime Star DNA聚合酶在PCR扩增过程中所产生的错误率进行矫正。
1.2.4 序列处理及分析 序列测定委托深圳华大基因科技服务有限公司进行。借助DNAStar软件进行序列处理,多序列比对采用DNAStar Clustal W方法,序列相似性分析借助DNAStar软件进行,单倍型系统发育树构建采用MEGA6的neighbor-joining(NJ)方法(Bootstrap选择1 000次以评估系统发育树的置信度),单倍型多样性、核苷酸多样性及中性检验借助DnaSP软件完成。
2 结果
2.1 CTV p20种群中的单倍型分析
共建立了7个CTV p20种群,由162条p20序列组成,包含11个单倍型;单个种群内检测到1个或多个单倍型(表1)。序列分析发现,来自不同种群的单倍型对应的原始核苷酸序列一致性为88.2%—100%(表2),其中单倍型PeraIAC-4、CT22和CT9-1对应的原始核苷酸序列一致性为100.0%,为同一单倍型;TR-L514-2和CT3也为同一单倍型。CTV甜橙强毒种群TR-L514中共检测到2个不同的单倍型,CTV甜橙弱毒种群CT31和PeraIAC中共检测到6个不同的单倍型;CTV柚强毒种群CT23、CT3和CT22中共检测到4个不同的单倍型,CTV柚弱毒种群CT9中共检测到2个不同的单倍型(表1)。其中单倍型多样性最丰富的是寄主为甜橙的种群PeraIAC(0.800±0.051),单倍型较单一的是寄主为柚的种群CT3(0.186±0.110)和CT22(0.170±0.102)。核苷酸多样性分析表明,甜橙种群普遍有较丰富的核苷酸多样性,在(0.04208±0.00304)—(0.05677±0.00564);柚中不同株系种群差异较大,其中CT23有丰富的核苷酸多样性,其他3个种群CT22、CT3和CT9的核苷酸多样性相对较低(表1)。Table 1
表1
表1CTV p20种群的单倍型及核苷酸多样性
Table 1The diversity of haplotype and nucleotide detected in CTV p20 populations
| 种群 Population | 寄主 Host | 致病力 Pathogenicity | 单倍型 Haplotypes | 单倍型多样性 Haplotype diversity (Hd±SD) | 核苷酸多样性 Nucleotide diversity (Pi±SD) |
|---|---|---|---|---|---|
| TR-L514 | 甜橙 Sweet orange | 强 Severe | TR-L514-1、TR-L514-2 | 0.772±0.057 | 0.05677±0.00564 |
| CT31 | 甜橙 Sweet orange | 弱 Mild | CT31-1、CT31-2 | 0.513±0.037 | 0.04208±0.00304 |
| PeraIAC | 甜橙 Sweet orange | 弱 Mild | PeraIAC-1、PeraIAC-2、PeraIAC-3、PeraIAC-4 | 0.800±0.051 | 0.04483±0.00939 |
| CT23 | 柚 Pummelo | 强 Severe | CT23-1、CT23-2 | 0.552±0.066 | 0.05919±0.00528 |
| CT3 | 柚 Pummelo | 强 Severe | CT3 | 0.186±0.110 | 0.00035±0.00021 |
| CT22 | 柚 Pummelo | 强 Severe | CT22 | 0.170±0.102 | 0.00032±0.00020 |
| CT9 | 柚 Pummelo | 弱 Mild | CT9-1、CT9-2 | 0.297±0.115 | 0.00305±0.00236 |
新窗口打开
Table 2
表2
表2各单倍型对应的原始核苷酸序列及相应的氨基酸序列的一致性
Table 2Identity of haplotypes based on the original nucleotide sequences and the corresponding amino acid sequences (%)
| 单倍型 Haplotype | TR-L 514-1 | TR-L 514-2 | CT31 -1 | CT31 -2 | PeraI AC-1 | PeraI AC-2 | PeraI AC-3 | PeraI AC-4 | CT23 -1 | CT23 -2 | CT3 | CT22 | CT9 -1 | CT9 -2 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TR-L514-1 | 94.5 | 95.1 | 96.2 | 94.0 | 97.8 | 97.8 | 94.5 | 96.7 | 94.5 | 94.5 | 94.5 | 94.5 | 93.4 | |
| TR-L514-2 | 88.7 | 96.2 | 95.1 | 95.1 | 96.7 | 96.7 | 100.0 | 93.4 | 100.0 | 100.0 | 100.0 | 100.0 | 98.4 | |
| CT31-1 | 92.2 | 89.3 | 95.6 | 98.9 | 97.3 | 97.3 | 96.2 | 94.0 | 96.2 | 96.2 | 96.2 | 96.2 | 95.1 | |
| CT31-2 | 95.1 | 88.2 | 91.8 | 94.5 | 98.4 | 98.4 | 95.1 | 95.1 | 95.1 | 95.1 | 95.1 | 95.1 | 94.0 | |
| PeraIAC-1 | 92.2 | 88.9 | 98.2 | 91.4 | 96.2 | 96.2 | 95.1 | 92.9 | 95.1 | 95.1 | 95.1 | 95.1 | 94.0 | |
| PeraIAC-2 | 97.3 | 90.3 | 93.1 | 96.0 | 93.1 | 100.0 | 96.7 | 96.7 | 96.7 | 96.7 | 96.7 | 96.7 | 95.6 | |
| PeraIAC-3 | 97.4 | 90.5 | 93.3 | 96.2 | 93.3 | 99.5 | 96.7 | 96.7 | 96.7 | 96.7 | 96.7 | 96.7 | 95.6 | |
| PeraIAC-4 | 88.9 | 99.8 | 89.4 | 88.3 | 89.1 | 90.5 | 90.7 | 93.4 | 100.0 | 100.0 | 100.0 | 100.0 | 98.4 | |
| CT23-1 | 98.0 | 88.5 | 91.6 | 94.2 | 91.6 | 96.7 | 96.9 | 88.7 | 93.4 | 93.4 | 93.4 | 93.4 | 92.3 | |
| CT23-2 | 88.7 | 99.6 | 89.3 | 88.2 | 88.9 | 90.3 | 90.5 | 99.8 | 88.5 | 100.0 | 100.0 | 100.0 | 98.4 | |
| CT3 | 88.7 | 100.0 | 89.3 | 88.2 | 88.9 | 90.3 | 90.5 | 99.8 | 88.5 | 99.6 | 100.0 | 100.0 | 98.4 | |
| CT22 | 88.9 | 99.8 | 89.4 | 88.3 | 89.1 | 90.5 | 90.7 | 100.0 | 88.7 | 99.8 | 99.8 | 100.0 | 98.4 | |
| CT9-1 | 88.9 | 99.8 | 89.4 | 88.3 | 89.1 | 90.5 | 90.7 | 100.0 | 88.7 | 99.8 | 99.8 | 100.0 | 98.4 | |
| CT9-2 | 89.8 | 96.5 | 89.6 | 89.6 | 89.3 | 91.6 | 91.8 | 96.7 | 89.6 | 96.5 | 96.5 | 96.7 | 96.7 |
新窗口打开
借助MEGA6构建系统发育树(图1)与CTV标准株系T30、T36、VT和T3比较分析,结果显示来自甜橙的分离株种群结构复杂,与标准株系T30、T36、VT和T3均有相关性。进化树最主要的分支是由单倍型TR-L514-2、CT3、CT23-2、PeraIAC-4、CT22和CT9-1组成的,共有93条序列,占所测克隆序列的57.4%,与标准株系T36亲缘关系较近,这6个单倍型两两间对应的原始核苷酸序列一致性均大于99.5%,仅在CTV甜橙弱毒种群CT31中未检测到。其中PeraIAC-4、CT22和CT9-1共有50条序列,是所测序列的优势单倍型。与标准株系T3相距很近的CT31-2与这3个优势单倍型在系统发育树上距离最远,对应的原始核苷酸序列一致性仅为88.3%。此外,源自同一种群的单倍型在系统发育树上的距离有的很近,如PeraIAC-2与 PeraIAC-3,有的较远,如TR-L514-1与TR-L514-2;源自不同种群的单倍型在系统发育树的距离有的也很近,如TR-L514-2和CT3,但有的也较远,如TR-L514-2和CT31-2。各单倍型对应的氨基酸序列的一致性为92.3%—100%,最低的氨基酸序列一致性发生在CT23-1和CT9-2之间(表2)。
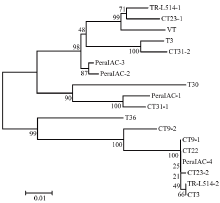 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1基于p20原始核苷酸序列的单倍型系统发育树
-->Fig. 1Phylogenetic tree of haplotypes based on the original nucleotide sequences of p20
-->
2.2 CTV p20的种群结构及变异水平
运用DnaSP软件对CTV p20种群进行了中性检验(表3),结果表明CT9种群的Tajima’s D值、Fu和Li’s D*值及Fu和Li’s F*值均为负值,达到显著水平,说明CT9种群已趋于稳定并处于扩张阶段。CT3和CT22种群的Tajima’s D值、Fu和Li’s D*值及Fu和Li’s F*值尽管都为负数,但并没有达到显著水平,说明CT3和CT22种群可能处于扩张阶段。而TR-514Y、CT31和CT23种群的Tajima’s D值、Fu和Li’s D*值及Fu和Li’s F*值均为正值,达到显著水平,表明这3个种群处于平衡或收缩状态。PeraIAC种群的Tajima’s D值、Fu和Li’s D*值及Fu和Li’s F*值虽均为正值,但Tajima’s D值及Fu和Li’s F*值不显著,表明此种群可能处于平衡或收缩状态。另外,运用DnaSP软件对各种群进行重组分析表明,各种群均未有重组事件发生(结果未显示)。Table 3
表3
表3基于CTV p20的中性检验
Table 3Neutrality test based on the CTV p20
| 种群 Population | Tajima’s D | Fu 和 Li’s D* Fu and Li’s D* | Fu 和 Li’s F* Fu and Li’s F* |
|---|---|---|---|
| TR-514Y | 2.898* | 1.431* | 2.238* |
| CT31 | 3.595* | 1.763* | 2.748* |
| PeraIAC | 0.488 | 1.622* | 1.492 |
| CT23 | 3.323* | 1.665* | 2.535* |
| CT3 | -1.514 | -2.082 | -2.214 |
| CT22 | -1.515 | -2.135 | -2.260 |
| CT9 | -2.473* | -3.927* | -4.072* |
新窗口打开
种群结构分析表明,CTV p20甜橙强毒种群由26个克隆构成,其中有8个克隆在7个位点发生突变,共检测到11个碱基发生突变,其突变克隆百分比达到30.8%,种群突变频率为7.706×10-4(表4)。CTV p20甜橙弱毒种群共获得46个克隆,有2个克隆发生变异,在2个位点检测到了2个碱基发生突变,种群突变克隆百分比为4.3%,种群突变频率为7.920×10-5。甜橙中CTV弱毒种群的突变克隆百分比和突变频率均明显低于强毒种群,种群突变频率相差一个数量级。CTV p20柚强毒种群由65个克隆构成,其中有4个克隆在4个位点发生突变,共发现了4个碱基发生突变,其突变克隆百分比为6.2%,种群突变频率为1.121×10-4(表4)。CTV p20柚弱毒种群共获得25个克隆,有1个克隆发生变异,在1个位点检测到了1个碱基发生突变,种群突变克隆百分比为4.0%,种群突变频率为7.286×10-5。柚中CTV弱毒种群的突变克隆百分比和突变频率略低于强毒种群。对照组的25个克隆序列未检测到碱基突变。
Table 4
表4
表4CTV p20种群遗传结构及变异
Table 4Genetic structure and variation of the CTV p20 populations
| 种群 Population | 寄主 Host | 致病力 Pathogenicity | 突变克隆百分比(突变克隆数/总克隆数) Mutated clones (Mutated number/total number) (%) | 突变频率 Mutation frequency | 总计 Total |
|---|---|---|---|---|---|
| TR-L514 | 甜橙 Sweet orange | 强 Severe | 30.8 (8/26) | 7.706×10-4 | 7.706×10-4 |
| CT31 | 甜橙 Sweet orange | 弱 Mild | 0 (0/25) | 0 | 7.920×10-5 |
| PeraIAC | 甜橙 Sweet orange | 弱 Mild | 9.5 (2/21) | 1.735×10-4 | |
| CT23 | 柚 Pummelo | 强 Severe | 4.8 (1/21) | 8.674×10-5 | 1.121×10-4 |
| CT3 | 柚 Pummelo | 强 Severe | 14.3 (3/21) | 2.602×10-4 | |
| CT22 | 柚 Pummelo | 强 Severe | 0 (0/23) | 0 | |
| CT9 | 柚 Pummelo | 弱 Mild | 4.0 (1/25) | 7.286×10-5 | 7.286×10-5 |
| PCR-control | 0 (0/25) | 0 | 0 |
新窗口打开
2.3 CTV p20种群碱基突变类型
对CTV p20种群中的碱基突变类型进行分析,发现检测到的突变以碱基替代为主,仅在柚强毒种群CT3 的156和157位点间发现插入了一个碱基A,没有发现碱基的缺失(表5)。CTV甜橙强毒种群TR-L514共检测到11个突变,其中在259位点检测到3个A→G突变,在261位点检测到2个C→U突变,在427位点检测到2个G→A突变,在45位点和140位点各检测到1个U→C突变,在136位点检测到1个G→C突变,在222位点检测到1个A→U突变,碱基转换与碱基颠换的比例为9﹕2;弱毒种群PeraIAC共检测到2个突变,其中在139位点检测到1个U→C突变,在537位点检测到1个A→G突变,均为碱基转换。CTV柚强毒种群CT3在76位点检测到1个G→A突变并在98位点检测到1个A→G突变,种群CT23在298位点检测到一个A→G突变,均为碱基转换;CTV柚弱毒种群CT9仅在198位点检测到1个G→U突变,为碱基颠换,种群CT31及CT22均未检测到突变。Table 5
表5
表5CTV p20种群的碱基替代突变类型及位点
Table 5The types and sites of nucleotide substitution of CTV p20 populations
| 碱基替代突变类型 Type of substitution | 寄主甜橙Sweet orange | 寄主柚Pummelo | ||||
|---|---|---|---|---|---|---|
| TR-L514 | PeraIAC | CT23, CT3 | CT9 | |||
| 颠换Transversions | U→G | — | — | — | — | |
| U→A | — | — | — | — | ||
| G→U | — | — | — | (198)1 | ||
| G→C | (136)1 | — | — | — | ||
| C→G | — | — | — | — | ||
| C→A | — | — | — | — | ||
| A→U | (222)1 | — | — | — | ||
| A→C | — | — | — | — | ||
| 转换 Transitions | U→C | (45)1,(140)1 | (139)1 | — | — | |
| G→A | (427)2 | — | (76)1 | — | ||
| C→U | (261)2 | — | — | — | ||
| A→G | (259)3 | (537)1 | (98)1,(298)1 | — | ||
| 总计 Total | 11 | 2 | 3 | 1 | ||
新窗口打开
3 讨论
P20编码的蛋白作为CTV的3个沉默抑制子之一,在病毒克服寄主基因沉默方面发挥着重要作用[9]。有研究表明,寄主对CTV不同株系的抗病水平与CTV p20基因的表达情况息息相关[20]。本研究分析了甜橙和柚中CTV不同株系p20种群结构及变异情况,结果发现在甜橙和柚中,CTV强弱毒p20种群结构差异明显。CTV p20种群共包含11个单倍型,单个种群由1个或多个单倍型组成,与ZABLOCKI等[21]的研究结果一致。系统发育树分析表明,CTV甜橙分离株与CTV各标准株系均有相关性,说明CTV甜橙分离株种群结构复杂。在CTV甜橙弱毒p20种群中检测到最多的单倍型,共6个,而在其余种群中总共才检测到6个不同的单倍型,其中CTV柚强毒种群CT3和CT22均只有1个单倍型,与之前宋震等[15]报道的柚类以受相对单一的CTV株系侵染为主的结果一致;且CTV甜橙弱毒种群中这些单倍型与优势单倍型差异较大,与周彦等[13-14]关于CTV株系对不同柑橘类植株的适应性存在差异的研究结果相吻合。中性检验分析表明,甜橙分离株种群可能处于平衡或收缩状态,柚分离株种群CT23也处于平衡或收缩状态,而柚分离株种群CT9则已形成稳定的种群并处于扩张阶段,CT3、CT22种群也可能处于扩张阶段,这可能与CTV不同株系在寄主甜橙和柚中选择压力不同有关。甜橙和柚中CTV强弱毒p20种群的变异水平分析表明,各种群碱基突变频率在0—7.706×10-4,与前人已报道的RNA病毒编码区的碱基突变频率相吻合[22-23]。本研究显示甜橙种群单倍型多样性和核苷酸多样性更为丰富。SENTANDREU等[24]的研究也表明,寄主可能在CTV进化中扮演着重要角色。本研究发现CTV p20种群在寄主甜橙中有更复杂的种群结构和更高的变异水平,CTV柚强毒种群的突变克隆百分比和碱基突变频率均低于CTV甜橙强毒种群,CTV柚弱毒种群的单倍型多样性低于CTV甜橙弱毒种群,表明寄主甜橙和柚对CTV p20种群结构和变异的影响存在差异。此外寄主甜橙和柚中CTV弱毒种群的突变克隆百分比和碱基突变频率均低于强毒种群,此现象说明寄主甜橙和柚中CTV p20强毒种群较弱毒种群更易发生碱基突变。
对甜橙和柚中CTV强弱毒p20种群碱基突变类型的分析表明,在CTV甜橙强毒种群中碱基突变最易发生,优势突变类型为A→G突变。GE等[25]在研究中国番茄黄化曲叶病毒(Tomato yellow leaf curl China virus,TYLCCNV)种群结构及遗传变异时发现,TYLCCNV子代种群中只有碱基替代突变且易发生G→A突变和C→T突变,而A→G突变和T→C突变很少发生,表明RNA病毒和DNA病毒在碱基突变类型方面可能存在差异。本研究同时分析了甜橙和柚中CTV强弱毒p20种群的碱基突变位点,结果表明CTV甜橙强毒p20种群发现了更多的突变位点,说明寄主甜橙可能对CTV强毒株系p20种群结构及变异有着更大的影响。
本研究综合分析了甜橙和柚中具有不同致病力的CTV株系的p20种群变异情况,讨论了引起p20种群变异水平差异的可能原因。由于本研究建立的CTV种群数量较少,且不同寄主中毒源致病力分布状况欠均衡,今后还需要扩大种群数量来进一步解读CTV强弱毒p20种群变异与寄主之间的相互关系。
4 结论
寄主甜橙和柚中CTV强弱毒p20种群结构及变异情况存在差异,CTV甜橙p20种群具有更复杂的种群结构和更高的种群变异水平。同一寄主中,CTV p20强毒种群变异水平高于弱毒种群。The authors have declared that no competing interests exist.
