0 引言
【研究意义】目前,全球约有1/3的奶牛患有不同类型的乳房炎,乳房炎给奶牛养殖业造成了巨大的经济损失[1-2]。由于乳房炎的遗传力较低[3],直接选择低遗传力的临床乳房炎并不是遗传改良的有效方法,而是通过对其他高遗传力且与乳房炎有高遗传相关的性状进行间接选育。标记辅助选择(marker-assisted selection, MAS)可将乳房炎抗性基因和其它分子标记结合入现有的选育方案进行选育。因此通过全基因组关联分析(genome wide association study, GWAS)寻找到显著的单核苷酸突变(single nucleotide polymorphism, SNP),然后通过MAS进行奶牛乳房炎抗性的选育。【前人研究进展】AXFORD等指出,奶牛生产性能测定(dairy herd improvement, DHI)中的体细胞数(somatic cell count, SCC)是用于提高乳房炎抗性的首选性状[4],RUPP的研究发现,对低SCC的奶牛进行选育后,其乳房炎的发病率降低[5]。WIJGA通过全基因组关联分析报道,与泌乳期平均体细胞评分(lactation-average SCS,LASCS)和测定日记录体细胞评分标准差(test-day SCS standard deviation,SCS-SD)显著相关的SNPs位于4,6和18号染色体上[6]。【本研究切入点】首先对北京地区中国荷斯坦牛的SCC进行对数转化(Log- transformed),获得SCS;再依据LASCS和SCS-SD高低将牛只划分为乳房炎易感牛(case)及抗性牛(control);进一步利用Case-control方法,将乳房炎易感性及抗性性状与Illumina54k芯片的54 001个SNPs进行全基因组关联分析。【拟解决的关键问题】通过全基因组关联分析找到与荷斯坦牛乳房炎易感性及抗性存在显著关联的SNPs,揭示与乳房炎抗性密切相关的基因变异,为发现乳房炎易感性及抗性相关的分子标记及乳房炎分子抗病育种奠定理论依据。1 材料与方法
试验于2014年3—5月在中国农业大学动物科技学院分子数量遗传学实验室进行。1.1 表型和基因型
2 093头中国荷斯坦母牛DHI数据及其54K SNP基因型数据(BovineSNP50, Illumina, USA)。1.2 数据处理
本研究利用SAS(Statistical Analysis System, Cary, North Carolina, USA)9.1.3软件进行数据筛选。原始数据为北京地区36个奶牛场2093头中国荷斯坦母牛的头3个胎次的DHI记录,共计35 522条测定日记录。其中SCC(单位:×103个/mL)的最小值为0,最大值为9 952,平均值为237.8,标准差为550.0。产犊日期和DHI测定时间段为2000年至2008年,测定天数为5—305d,测定间隔为0—215d,所有母牛来自于14头公牛家系。
首先对测定日记录SCC进行对数转化,将测定日记录SCC转化为泌乳期平均SCC评分(Lactation- average SCC,LASCS)和测定日记录SCC评分标准差(Test-day SCC standard deviation,SCS-SD),即:LASCS=log2
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1用于Case-control分析的LASCS和SCS-SD的统计性描述
-->Fig.1Descriptive statistics of LASCS and SCS-SD for case-control association testing
-->
依据LASCS和SCS-SD将泌乳牛划分为Case- control两个子数据集[7]。划分标准为:将LASCS和SCS-SD进行半个标准差(Half of standard deviation, 0.5 SD)和一个标准差(One standard deviation, 1 SD)的划分,高LASCS和高SCS-SD的牛归到易感组(Case),低LASCS和低SCS-SD的牛则归入抗性组(Control)(图1),用于Case-control关联分析方法的LASCS和SCS-SD的统计性描述如表1所示。
Table 1
表1
表1Case-control关联分析中LASCS和SCS-SD划分的描述性统计
Table 1Descriptive statistics of LASCS and SCS-SD for case-control association testing
| 性状 Traits | 母牛数 No. of cows | 最小值 Minimum | 最大值 Maximum | 均值 Mean | 标准差 Standard deviation | 总计 Total |
|---|---|---|---|---|---|---|
| LASCS1 | 652 vs 719 | 7.90 vs 4.48 | 11.01 vs 6.69 | 8.71 vs 6.02 | 0.65 vs 0.47 | 1371 |
| LASCS2 | 343 vs 365 | 8.50 vs 4.48 | 11.01 vs 6.09 | 9.20 vs 5.64 | 0.51 vs 0.35 | 708 |
| SCS-SD1 | 701 vs 684 | 16.44 vs 4.30 | 23.29 vs 12.97 | 18.47 vs 10.71 | 1.47 vs 1.67 | 1385 |
| SCS-SD2 | 358 vs 358 | 18.17 vs 4.30 | 23.29 vs 11.24 | 19.60 vs 9.44 | 1.16 vs 1.32 | 716 |
新窗口打开
1.3 质量控制
将54 001个SNPs进行质控,剔除不符合条件的SNPs。剔除条件:(1)SNPs的call rate<90%;(2)严重偏离哈迪-温伯格平衡(HWE)(P< 10E-6);(3)最小等位基因频率(MAF)<0.03。经过质控之后,共有43781/43671(43817/43704)个SNPs分别可用于LASCS(SCS-SD)半个标准差/一个标准差的关联分析,其所在牛染色体上的分布情况如表2所示。Table 2
表 2
表 2LASCS(SCS-SD)半个标准差/一个标准差质控后SNPs的分布和相邻SNPs的平均距离
Table 2Distribution of SNPs on chromosomes after quality control and the average distances between adjacent SNPs of 0.5/ 1 SD of LASCS(SCS-SD)
| 染色体 Chr. | SNP数 No. of SNPs | 平均距离(kb) Average distance (kb) | 染色体 Chr. | SNP数 No. of SNPs | 平均距离(kb) Average distance (kb) | |
|---|---|---|---|---|---|---|
| 1 | 2735/2729 (2741/2733) | 57.89/58.02 (57.77/57.94) | 17 | 1299/1292 (1301/1294) | 57.86/58.17 (57.77/58.08) | |
| 2 | 2202/2193 (2202/2199) | 62.24/62.50 (62.24/62.33) | 18 | 1109/1108 (1111/1107) | 59.52/59.57 (59.41/59.62) | |
| 3 | 2104/2096 (2106/2104) | 57.71/57.93 (57.66/57.71) | 19 | 1148/1148 (1148/1145) | 55.80/55.80 (55.80/55.95) | |
| 4 | 2041/2035 (2044/2031) | 59.20/59.38 (59.11/59.49) | 20 | 1321/1320 (1324/1317) | 54.54/54.58 (54.41/54.70) | |
| 5 | 1754/1745 (1748/1755) | 69.09/69.45 (69.33/69.05) | 21 | 1125/1124 (1127/1122) | 63.64/63.70 (63.53/63.81) | |
| 6 | 2079/2074 (2078/2077) | 57.46/57.60 (57.49/57.52) | 22 | 1036/1034 (1039/1035) | 59.30/59.42 (59.13/59.36) | |
| 7 | 1821/1818 (1819/1818) | 61.86/61.96 (61.92/61.96) | 23 | 903/900 (904/899) | 58.17/58.37 (58.11/58.43) | |
| 8 | 1939/1937 (1940/1938) | 58.48/58.54 (58.45/58.51) | 24 | 1043/1044 (1045/1041) | 60.13/60.07 (60.01/60.24) | |
| 9 | 1642/1644 (1644/1646) | 64.38/64.30 (64.30/64.22) | 25 | 816/816 (818/816) | 52.58/52.58 (52.45/52.58) | |
| 10 | 1753/1745 (1753/1747) | 59.50/59.77 (59.50/59.71) | 26 | 878/876 (880/877) | 58.86/59.00 (58.73/58.93) | |
| 11 | 1859/1850 (1860/1853) | 57.72/58.01 (57.69/57.91) | 27 | 815/811 (816/809) | 55.72/55.99 (55.65/56.13) | |
| 12 | 1356/1348 (1355/1349) | 67.23/67.63 (67.28/67.58) | 28 | 780/778 (781/779) | 59.38/59.53 (59.30/59.45) | |
| 13 | 1438/1438 (1444/1438) | 58.58/58.58 (58.34/58.58) | 29 | 862/860 (865/860) | 59.75/59.89 (59.54/59.89) | |
| 14 | 1394/1388 (1395/1391) | 60.72/60.99 (60.68/60.85) | X | 554/555 (554/555) | 268.64/268.15 (268.64/268.15) | |
| 15 | 1382/1379 (1379/1379) | 61.72/61.85 (61.85/61.85) | 0* | 1305/1302 (1307/1308) | ||
| 16 | 1288/1284 (1289/1282) | 63.45/63.65 (63.40/63.75) | 总计Total | 43781/43761 (43817/43704) |
新窗口打开
1.4 关联分析
本研究的关联分析方法是基于前期使用的ROADTRIPS软件(版本 1.2)[7-8],该软件可以通过大量基因型信息并结合系谱信息估计出一个可以反应个体间遗传联系的经验协方差矩阵。ROADTRIPS(Robust Association-Detection Test for Related Individuals with Population Substructure)一共包括3种检验:RM(ROADTRIPS-MQLS test)检验、RCHI(ROADTRIPS-χ2 test)检验和RW(ROADTRIPS- WQLS test)检验。该软件通过构建服从自由度为1的卡方分布的统计量,计算出每个检验的SNP的P值。
1.5 关联分析结果的显著性检验
为了降低多重检验带来的假阳性升高问题,采用Bonferroni方法对关联分析结果进行校正。如果设定0.05为显著性水平,则犯I类错误的累积概率就要控制在0.05以内。所以当进行m次显著性水准为α的假设检验时,即有Bonferroni不等式0.05≤mα成立。令各次比较的显著性水准α=0.05/m,并规定P≤0.05/m时拒绝原假设[9]。在本研究中,针对牛的每条染色体分别制定各条染色体的显著水平,以0.05分别除每条染色体上的SNPs数目,作为每条染色体水平的显著性水平[10]。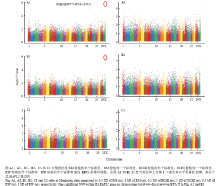 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2基于LASCS的乳房炎抗性检验后的全基因组SNP位点的-log10 (P value)
-->Fig. 2Manhattan plots (-log10 (P value)) of genome-wide SNPs for mastitis resistance based on LASCS
-->
2 结果
2.1 全基因组关联分析
经过对基于LASCS和SCS-SD的乳房炎抗性进行全基因组关联分析,得到了3种检验(RM检验、RW检验和RCHI检验)的全基因组SNPs位点的-log10 (P value)结果,如图2和图3所示。通过对基于半个标准差SCS-SD的乳房炎抗性进行全基因组关联分析发现一个全基因组水平显著的SNP(Hapmap48573-BTA-104531, P=1.11E-06)位于X染色体上(图3-B1)。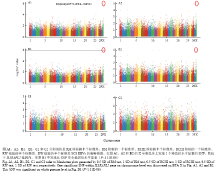 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3基于SCS-SD的乳房炎抗性检验后的全基因组SNP位点的-log10 (P value)
-->Fig.3Manhattan plots (-log10 (P value)) of genome-wide SNPs for mastitis resistance based on SCS-SD
-->
2.2 显著SNPs及相关基因
通过Bonferroni校正得到每条染色体0.05显著性水平的阈值(表3),共发现5个达到染色体水平显著的SNPs。其中3个SNPs定位到X染色体上,其它2个SNPs分别定位到7和28号染色体上。结果发现,X染色体的显著性SNPs(Hapmap48573-BTA-104531和Hapmap54175- rs29021817)位于IL1RAPL2基因内,7号染色体的显著性SNP周围存在与炎症反应相关的基因(ILF3)。Table 3
表3
表3牛基因组每条染色体所对应Bonferroni校正的显著性标准
Table 3Significant P-value of Bonferroni correction on each bovine chromosome
| 染色体 Chr. | 显著水平(0.5/1 SD of LASCS(SCS-SD)) Significant P-values (0.5/1 SD of LASCS (SCS-SD)) | 染色体 Chr. | 显著水平(0.5/1 SD of LASCS(SCS-SD)) Significant P-values (0.5/1 SD of LASCS (SCS-SD)) | |
|---|---|---|---|---|
| 1 | 1.83E-05/1.83E-05 (1.82E-05/1.83E-05) | 17 | 3.85E-05/3.87E-05 (3.84E-05/3.86E-05) | |
| 2 | 2.27E-05/2.28E-05 (2.27E-05/2.27E-05) | 18 | 4.51E-05/4.51E-05 (4.50E-05/4.52E-05) | |
| 3 | 2.38E-05/2.39E-05 (2.37E-05/2.38E-05) | 19 | 4.36E-05/4.36E-05 (4.36E-05/4.37E-05) | |
| 4 | 2.45E-05/2.46E-05 (2.45E-05/2.46E-05) | 20 | 3.79E-05/3.79E-05 (3.78E-05/3.80E-05) | |
| 5 | 2.85E-05/2.87E-05 (2.86E-05/2.85E-05) | 21 | 4.44E-05/4.45E-05 (4.44E-05/4.46E-05) | |
| 6 | 2.40E-05/2.41E-05 (2.41E-05/2.41E-05) | 22 | 4.83E-05/4.84E-05 (4.81E-05/4.83E-05) | |
| 7 | 2.75E-05/2.75E-05 (2.75E-05/2.75E-05) | 23 | 5.54E-05/5.56E-05 (5.53E-05/5.56E-05) | |
| 8 | 2.58E-05/2.58E-05 (2.58E-05/2.58E-05) | 24 | 4.79E-05/4.79E-05 (4.78E-05/4.80E-05) | |
| 9 | 3.05E-05/3.04E-05 (3.04E-05/3.04E-05) | 25 | 6.13E-05/6.13E-05 (6.11E-05/6.13E-05) | |
| 10 | 2.85E-05/2.87E-05 (2.85E-05/2.86E-05) | 26 | 5.69E-05/5.71E-05 (5.68E-05/5.70E-05) | |
| 11 | 2.69E-05/2.70E-05 (2.69E-05/2.70E-05) | 27 | 6.13E-05/6.17E-05 (6.13E-05/6.18E-05) | |
| 12 | 3.69E-05/3.71E-05 (3.69E-05/3.71E-05) | 28 | 6.41E-05/6.43E-05 (6.40E-05/6.42E-05) | |
| 13 | 3.48E-05/3.48E-05 (3.46E-05/3.48E-05) | 29 | 5.80E-05/5.81E-05 (5.78E-05/5.81E-05) | |
| 14 | 3.59E-05/3.60E-05 (3.58E-05/3.59E-05) | X | 9.03E-05/9.01E-05 (9.03E-05/9.01E-05) | |
| 15 | 3.62E-05/3.63E-05 (3.63E-05/3.63E-05) | 0* | 3.83E-05/3.84E-05 (3.83E-05/3.82E-05) | |
| 16 | 3.88E-05/3.89E-05 (3.88E-05/3.90E-05) | 整体Whole | 1.14E-06/1.14E-06 (1.14E-06/1.14E-06) |
新窗口打开
3 讨论
本研究通过全基因组关联分析,获得5个在染色体水平与中国荷斯坦牛乳房炎抗性显著关联的SNPs,其中X染色体上2个显著性SNPs(Hapmap48573- BTA-104531和Hapmap54175-rs29021817)都位于白介素1受体附属蛋白2基因(Interleukin 1 receptor accessory protein-like-2,IL1RAPL2)内。7号染色体上的显著SNP(BTA-78357- no-rs)在100 kp范围内发现了与炎症相关的白介素增强结合因子3基因(Interleukin enhancer binding factor 3,ILF3)。Table 4
表4
表4乳房炎抗性全基因组关联分析显著SNPs和相关基因
Table 4Significant SNPs and the nearest genes of genome-wide association with mastitis resistance
| 染色体 Chr. | 位置 Position (bp) | SNP名 SNP names | 检验 Tests | 表型 Phenotype | 相关基因 Nearest genes | 距离 Distance (bp) | P值 P-value |
|---|---|---|---|---|---|---|---|
| X | 32705282 | Hapmap48573-BTA-104531 | RM/RCHI | LASCS&0.5SD | IL1RAPL2 | within | 1.67E-06/1.64E-06 |
| RM/RCHI | SCS-SD&0.5SD | 1.15E-06/1.11E-06 | |||||
| RM/RCHI | SCS-SD&1SD | 1.04E-05/9.74E-06 | |||||
| 7 | 13510712 | BTA-78357-no-rs | RW | LASCS&0.5SD | ILF3 | 98207 | 6.73E-06 |
| RW | SCS-SD&0.5SD | 1.15E-05 | |||||
| 28 | 35175402 | BTB-00988209 | RW | LASCS&0.5SD | NA | NA | 1.98E-05 |
| X | 33009361 | Hapmap54175-rs29021817 | RM/RCHI | LASCS&1SD | IL1RAPL2 | within | 2.79E-05/2.65E-05 |
| X | 88118444 | BTA-28466-no-rs | RW | SCS-SD&0.5SD | NA | NA | 6.74E-05 |
新窗口打开
IL1RAPL2基因已经被证实在大脑发育过程中有特殊的表达,并在神经功能的发育中起着关键作用。目前的研究尚未发现IL1RAPL2基因与特定奶牛疾病相关,但被认为是神经失调的候选基因[11]。ILF3基因是白介素家族中的一个跟炎症反应相关的因子,其功能与抑制翻译蛋白有关[12]。其他研究还发现,小鼠中的ILF3基因在剧毒的弗朗西斯菌中曝光4h后,其表达量会增加,此过程与NFAT(nuclear factor of activated T cells)信号通路有关。而当感染弗朗西斯菌急性发病时,很多出现表达增加的免疫相关基因参与了淋巴细胞激活过程,其中就包括NFAT信号通路中的基因[13]。目前关于这两个基因的研究较少,尚未发现其与奶牛乳房炎抗性有关,但这两个基因都与白介素有关,而白介素4、5、6、12、13、17、22、23等都参与了不同类型的炎症反应,并发挥了重要的作用[14-16],因此应该进一步对这两个基因进行功能验证,以作为奶牛乳房炎抗性的候选基因。
本研究基于WIJGA的研究[6],构建了中国荷斯坦牛乳房炎抗性的统计量LASCS和SCS-SD。尽管LASCS和SCS-SD为连续性状,但奶牛乳房炎抗性作为一个阈性状,更适合使用Case-control进行全基因组关联分析[17-18]。最近几年里,关联分析研究开始向Case和Control转变,目的是为了增加检验效力[19]。为检测到与奶牛乳房炎症反应功能相关的基因,本研究采用Case-control方法研究奶牛乳房炎易感性及抗性,并对LASCS和SCS-SD进行了乳房炎易感性牛(Case)和抗性牛(Control)的划分。通过全基因组关联分析发现,显著SNPs主要集中于X染色体上,其中包括全基因组水平显著的SNP(Hapmap48573- BTA-104531)。而WIJGA将SCC主要定位到牛的4、6和18号染色体上,其关联分析显著SNP的阈值是7.94E-06[6]。通过对比其它研究发现,仅有Abdel-Shaft在德国荷斯坦牛的研究将与SCS有关显著的SNP(rs41629005, Position: 30639394)定位到X染色体上[20]。虽然其它研究通过常规的单标记回归分析将影响乳房炎抗性的SNPs定位到常染色体上,但结果多种多样[21-23],这也说明奶牛乳房炎抗性受微效多基因控制,常规方法不易定位到效应较大的分子标记。本研究首次通过LASCS和SCS-SD对中国荷斯坦牛乳房炎易感性牛和抗性牛进行划分,然后进行Case-control关联分析。本研究的检验效力要高于前期的研究,找到的相关基因也与炎症反应关系紧密[7]。
本研究还通过线性混合模型(linear mixed model,MMA)对LASCS和SCS-SD进行全基因组关联分析以验证Case-control关联分析的结果,共检测到4个染色体水平显著的SNPs,并都定位到X染色体上。其中BTA-28466- no-rs被MMA和Case-control方法同时定位到(表5)。对比之前的研究发现[7, 24],用两种方法对同一性状进行关联分析,可以定位到相同的位点,但不同性状的结果就各不相同。我们前期对体细胞评分育种值(somatic cell score estimated breeding value, SCSEBV)进行关联分析,两种方法共同定位到14号染色体上的SNP(ARS- BFGL-NGS-100480),其中线性模型检验效力较高(P = 1.24E-10)[24]。然而用Case-control方法对测定日记录[7]、SCSEBV[24]、LASCS和SCS-SD的Case和Control进行关联分析,结果不尽相同,可能与SCC受环境影响较大、通过SCC划分的Case和Control关联分析检验效力较低有关。因此对于奶牛乳房炎抗性性状,还应直接对乳房炎牛和健康牛进行二分类的Case-control关联分析。
Table 5
表5
表5基于线性混合模型的全基因组关联分析显著SNPs
Table 5Significant SNPs of genome-wide association based on MMA method
| 染色体 Chr. | 位置 Position (bp) | SNP名 SNP name | 表型 Phenotype | P值 P-value |
|---|---|---|---|---|
| 12 | 32421528 | ARS-BFGL-NGS-89267 | LASCS&SCS-SD | 7.62E-06&1.14E-05 |
| X | 88118444 | BTA-28466-no-rs | LASCS&SCS-SD | 1.92E-05&7.84E-05 |
| 25 | 11031070 | ARS-BFGL-NGS-30473 | LASCS | 5.56E-05 |
| X | 53530716 | Hapmap39261-BTA-29176 | LASCS | 7.34E-05 |
| X | 33009361 | Hapmap54175-rs29021817 | LASCS | 8.84E-05 |
| 15 | 83722393 | ARS-BFGL-NGS-89374 | SCS-SD | 2.31E-05 |
| 17 | 27258429 | Hapmap51101-BTA-97588 | SCS-SD | 2.63E-05 |
| X | 88118444 | BTA-28466-no-rs | SCS-SD | 7.84E-05 |
新窗口打开
由于乳房炎易感性及抗性性状属于功能性状,遗传力偏低,受环境效应影响较大,而3种原理不同的检验方法可能导致共同定位的SNP的检验效力大小不一。尽管本研究未检出对两种性状(LASCS和SCS-SD)关联分析均达到显著的SNP,但X染色体上的SNP(Hapmap48573-BTA-104531)用3种检测方法均发现其P值较低(表6)。进一步的研究拟增加SNPs标记密度及牛群数量,扩群验证已获得的乳房炎易感性及抗性相关分子标记及基因。此外还需对已发现的显著SNPs及临近基因进行生物学通路分析及功能验证,为揭示奶牛乳房炎易感性及抗性的分子机制提供更多数据。
Table 6
表6
表6对SNP(Hapmap48573-BTA-104531)用3种方法检验后的P值
Table 6The P values of SNP (Hapmap48573-BTA-104531) by three tests
| 检验 Tests | LASCS | SCS-SD | ||
|---|---|---|---|---|
| 0.5 SD | 1 SD | 0.5 SD | 1 SD | |
| RM | 1.67E-06 | 1.09E-04 | 1.15E-06 | 1.04E-05 |
| RCHI | 1.64E-06 | 1.16E-04 | 1.11E-06 | 9.74E-06 |
| RW | 5.50E-03 | 1.38E-02 | 4.55E-03 | 1.34E-03 |
新窗口打开
4 结论
本研究利用Case-control方法对荷斯坦牛乳房炎易感性及抗性进行了全基因组关联分析,共检测5个显著性单核苷酸突变及2个与炎症反应密切相关的基因(IL1RAPL2和ILF3基因),并发现X染色体上的SNP(Hapmap48573-BTA-104531)在全基因组水平显著(P<1.14E-06)。本研究首次通过对泌乳期平均体细胞评分和测定日记录体细胞评分标准差进行Case-control方法关联分析,并找到了与炎症反应相关的基因,为奶牛乳房炎抗性功能相关基因的研究提供数据支持。The authors have declared that no competing interests exist.
