0 引言
【研究意义】亚麻(Linum usitatissimum L.)是亚麻科亚麻属一年生草本植物,其纤维来源于成熟亚麻茎的韧皮纤维。亚麻韧皮纤维细胞起源于单个细胞,其发育在形态学上经历了细胞伸长和次生细胞壁加厚2个不重叠时期,这两个阶段被主茎上的分界点(the snap point,SP)分开。植株进入快速生长期后,SP分界点之上为韧皮细胞伸长阶段,之下为细胞壁加厚阶段[1]。这两个阶段分别决定了成熟亚麻韧皮纤维的长度和厚度,最终影响亚麻纤维的产量和品质。因此,研究与细胞壁形成相关基因在亚麻SP点不同发育阶段细胞中的表达模式对进一步理解亚麻纤维细胞形成、细胞壁加厚的发育规律具有重要意义。【前人研究进展】β-半乳糖苷酶家族(BGALs),纤维素合成酶家族(CESAs)等在植物细胞壁形成和重建过程中起重要作用。BGAL属于糖苷水解酶家族成员(glycosyl hydrolyse family 35,GH35),参与细胞壁纤维素形成[2-3]和细胞壁细胞伸长[4]等过程。拟南芥中有17个β-半乳糖苷酶基因,其中,已证实AtBGAL1、AtBGAL2、AtBGAL5、AtBGAL6、AtBGAL8和AtBGAL12在细胞壁重建和扩展中起作用[5]。β-半乳糖苷酶还与棉花纤维细胞的起始和分化相关[6]。亚麻中预测有35个BGAL,芯片和qRT-PCR方法检测发现部分BGAL在纤维中表达[7]。纤维素是植物细胞壁中最重要的组分,由位于质膜上的纤维素合酶复合体(cellulose synthase complexs,CSCs)合成。拟南芥、杨树、棉花、豌豆、水稻等多种植物中研究发现纤维素合酶是多基因家族,不同组分形成不同CSC复合体,在初生细胞壁和次生细胞壁形成过程中分别起作用,作用模式现已基本研究清楚。拟南芥CESA1、CESA3、CESA6蛋白形成CSC参与初生壁纤维素合成[8],缺失其中任一组分,植株致死;而CESA4、CESA7、CESA8决定次生壁纤维素合成[9],其他CESAs基因在不同生理时期和组织中参与细胞壁形成。棉花中至少有15个CESA基因是纤维素合成所必需的[10],其中CESA1、CESA2、CESA7、CESA8主要参与次生细胞壁的合成,CESA3、CESA6、CESA9、CESA10在棉花初生细胞壁的合成中起作用[11]。已有研究表明杨树中有18个CESA基因,其中5个基因(PtrCesA4、PtrCesA7-A、PtrCesA7-B、PtrCesA8-A、PtrCesA8-B)主要在次生细胞壁合成中发挥作用[12-16],其他CESAs在杨树不同生长发育过程中参与细胞壁构建过程[17-19]。通过病毒介导的基因沉默(VIGS)法,研究者发现CESA1、CESA3、CESA4、CESA6、CESA8在亚麻纤维细胞壁构建中扮演重要角色[20]。CESA7和CESA8在亚麻快速生长期、花期和绿熟期的表达高,推测这两个基因在亚麻次生细胞壁加厚过程中起重要作用[21]。【本研究切入点】尽管已有研究分析了亚麻不同组织器官的基因表达谱,但各个研究者关于韧皮纤维发育的芯片数据结果并不一致,亚麻快速生长期与纤维合成密切相关的BGALs、CESAs等基因的表达模式还不清楚[20, 22-24]。因此,LuBGALs和LuCESAs等基因家族在亚麻韧皮纤维细胞中的表达模式需进一步分析明确。【拟解决的关键问题】本研究通过qRT-PCR方法,拟探讨与细胞壁形成相关的BGALs和CESAs等基因在亚麻快速生长期韧皮纤维细胞伸长和细胞壁加厚过程中的表达模式,为研究纤用亚麻韧皮纤维发育机制奠定理论基础。1 材料与方法
试验于2016年在新疆大学生命科学与技术学院植物培养室完成。1.1 材料
亚麻(Linum usitatissimum L.)品种范妮(FANY)。范妮亚麻种子种植在花土:蛭石=3:1的土壤中,置于光照强度0—10 000 lx,16 h光照/8 h黑暗,(25±2)℃温室中培养45 d。1.2 亚麻总RNA的提取和cDNA的合成
种植后21—50 d为亚麻快速生长期,45 d的亚麻处于快速生长期后期,茎的表皮和木质部较易分离。取生长45 d亚麻茎3段,每段约2 cm。最上端(TOP)距茎尖1—3 cm且位于分界点(SP)上部。第二段(MID)距茎尖4—6 cm。基部(BOT)距茎尖17—19 cm。TOP直接取茎,MID和BOT取茎的表皮(图1-A)。每9棵亚麻茎段为一个池,每个部位设置6个重复,共18个样品。所取亚麻器官还包括生长了45 d的亚麻幼叶、成熟叶、根和基部,每个器官设置4个生物学重复,共16个样品。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1亚麻茎片段和电镜分析
A:不同部位茎的解剖;B:亚麻茎不同部位纤维细胞细胞壁电镜分析。SP:分界点;TOP:分界点上部;MID:分界点下部;BOT:基部;CW:细胞壁;ML:中间层。下同
-->Fig. 1Representative sections of stem segments and electron microscopy analysis
A: The anatomy in different parts of the stem; B: Electron microscope analysis of cell walls in different parts of flax stem. SP: Snap point; TOP: The stem above the snap point; MID: The stem below the snap point; BOT: The base of the stem; CW: Cell wall; ML: Middle lamellae. The same as below
-->
参照TIANGEN RNAprep Pure 植物总RNA提取试剂盒说明书提取总RNA,使用Thermo超微量分光光度计进行RNA定量。参照TaKaRa第一链合成试剂盒说明书合成cDNA。
1.3 引物设计
根据已知拟南芥相关基因序列,通过Phytozome v9.1和Quant Prime qPCR在线工具设计qRT-PCR引物。引物如电子版附表1所示。Table 1
表1
表1亚麻茎在SP点不同区域韧皮纤维厚度
Table 1The thickness of flax bast fibers in different parts of SP
| 部位 Position | |||
|---|---|---|---|
| TOP | MID | BOT | |
| 韧皮纤维厚度 The thickness of bast fiber (µm) | 0.115±0.059Aa | 0.509±0.181Bb | 0.653±0.256Cc |
新窗口打开
1.4 亚麻茎透射电镜TEM观察
亚麻茎TOP、MID、BOT样品经过固定液(2.5%戊二醛-磷酸缓冲液)固定、梯度乙醇脱水、包埋、固化后切片,切片经3%醋酸铀-枸橼酸铅双染色后在透射电镜下观察拍照。每个部位设置3个生物学重复。1.5 实时荧光定量PCR(qRT-PCR)检测基因的相对表达量
以合成的cDNA为模板,LuETIF1(eukaryotic translation initiation factor 1)为内参基因[7],使用ABI PRISM 7500实时荧光定量PCR仪进行qRT-PCR 试验,每个样品设置3个重复。反应体系为20 µL;包括1 µL cDNA、10 µL iTaqTM Universal SYBR® Green Supermix、上下游引物各0.5 µL和8 µL RNase free H2O。PCR反应条件为95℃ 2 min;95℃ 15 s,60℃ 20 s,72℃ 15 s,40个循环。相对表达水平数据采用2-ΔΔCt法进行分析。用GraphPad Prism 5和SPSS 19软件对目的基因的相对表达量进行统计学分析。用△CT法对相对转录水平进行分析,用MeV 4.9.0(Multi Experiment Viewer,http://www.tm4.org/)作图。
2 结果
2.1 亚麻茎不同部位韧皮纤维细胞细胞壁的发育
亚麻进入快速生长期后,株高呈现明显的日变化。为了探讨亚麻茎韧皮纤维细胞细胞壁在快速生长期的发育变化,研究检测了亚麻茎SP点上下不同部位细胞壁发育的情况。结果表明,SP点上部,靠近茎尖生长点,茎部组织分化不明显,茎的表皮与髓部无法分离。SP点下部靠近茎中部区域,细胞壁开始加厚,茎表皮能够与木质部分离,越往茎的基部,亚麻茎表皮越易剥离,韧皮纤维也越厚(图1-A)。透射电镜观察亚麻韧皮纤维在快速生长期细胞壁的厚度变化(图1-B),结果表明,在SP点上部TOP端纤维细胞可见结构疏松的细胞壁物质,壁薄约110 nm;紧邻SP点下部的MID区,细胞壁厚度明显增厚,约500 nm,壁物质密度增大;靠近茎中下部的BOT区,细胞壁厚度增大到约650 nm(表1)。尽管中下部细胞壁厚度增加,从图中可看出,细胞壁质地均一,没有明显的分层现象。2.2 LuBGALs在亚麻快速生长期不同器官和茎韧皮纤维的表达分析
为了获得β-半乳糖苷酶家族基因在亚麻快速生长期细胞壁形成中的作用,研究分析了这些基因在亚麻茎SP点不同部位韧皮纤维细胞和根、茎、叶不同器官中的表达水平(图2)。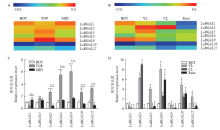 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2LuBGALs的表达分析
A、C:茎不同部位的表达分析;B、D:不同组织器官的表达分析。YL:幼叶;CL:成熟叶;Root:根。*、**和***分别代表在P<0.05、P<0.01和P<0.001水平显著。下同
-->Fig. 2Expression of LuBGALs
A and C: The analysis of different parts of a stem; B and D: The analysis of different tissues and organs. YL: Young leaf; CL: Climax leaf; Root: Root. “*”, “**” and “***” indicate significance at P<0.05, P<0.01 and P<0.001, respectively. The same as below
-->
2.2.1 LuBGALs在亚麻韧皮纤维中的表达分析 图2-A和2-C显示了SP点上下不同部位表达有差异的BGAL。结果表明,LuBGAL1在亚麻茎的3个不同部位的表达量存在极显著差异(P<0.01),TOP区的表达显著低于MID区和BOT区(P<0.01),表明它主要促进韧皮纤维细胞壁加厚过程。而LuBGAL3、LuBGAL6、LuBGAL9在TOP区表达最高,MID区次之,表明此类基因主要参与亚麻韧皮细胞伸长和细胞壁形成过程。LuBGAL5在幼嫩的TOP区表达量高,麻茎较为成熟的MID区较低(P<0.05)说明LuBGAL5主要参与细胞壁细胞伸长过程。LuBGAL20、LuBGAL22在亚麻茎的表达都较低,但在TOP区的表达极显著高于MID和BOT区(P<0.01),说明这两个基因可能在亚麻韧皮纤维细胞伸长过程中起作用。LuBGAL3、LuBGAL5、LuBGAL6、LuBGAL9在茎韧皮纤维中都具有较高的表达水平,说明在细胞壁加厚过程中多个BGAL共同作用。
2.2.2 LuBGALs在亚麻不同器官中的表达分析 为了进一步分析BGALs基因家族的表达模式,研究检测了BGALs基因在亚麻根、茎、叶3个不同器官中的表达情况(图2-B和2-D)。结果显示LuBGAL1和LuBGAL9在幼叶中、成熟叶和根中的表达极显著低于BOT区(P<0.001)。LuBGAL3在幼叶和成熟叶中的表达极显著高于BOT区(P<0.001),但在根中的表达极显著低于BOT区(P<0.001)。LuBGAL5和LuBGAL22具有相似的表达模式,在幼叶中的表达显著高于BOT(P<0.001),根和成熟叶的表达量显著低于BOT(P<0.01)。LuBGAL6和LuBGAL20在幼叶、成熟叶、根中的表达显著高于对照(P<0.05)。
尽管BGALs家族基因在亚麻中预测有30多个成员,但多数在麻茎韧皮纤维的生长发育过程中处于不表达状态(数据未显示)。LuBGAL1在BOT和MID的表达较高,而在根中表达较低;LuBGAL3在幼茎、幼叶和成熟叶中的表达量高,而在根中的表达低;LuBGAL5、LuBGAL6在幼嫩部位(TOP和YL)的表达高;LuBGAL9在所有组织的表达都较高;LuBGAL20和LuBGAL22在所有研究组织中的表达都较低。说明BGALs家族基因的表达具有明显的组织器官差异。
2.3 LuCESAs在亚麻快速生长期不同器官和茎韧皮纤维中的表达分析
拟南芥、杨树、棉花等植物中CESAs基因有多个组合,分别在不同的细胞壁发育阶段起作用[8, 11-19]。研究探讨了该家族基因在亚麻快速生长期细胞壁形成过程中的表达模式(图3)。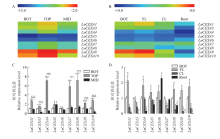 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3LuCESAs的表达分析
-->Fig. 3Expression of LuCESAs
-->
2.3.1 LuCESAs在亚麻韧皮纤维中的表达分析 图3-A和图3-C结果显示在麻茎幼嫩的TOP区纤维细胞中,LuCESA1、LuCESA3、LuCESA7、LuCESA8、LuCESA9和LuCESA10都检测出较高的表达量,且明显高于其在MID区和BOT区的表达(P<0.001)。其中LuCESA3和LuCESA10在MID区的表达显著低于BOT区(P<0.05),其他几个CESAs基因在MID和BOT的表达并无明显差异。上述结果说明亚麻中6个CESA基因(LuCESA1、LuCESA3、LuCESA7、LuCESA8、LuCESA9和LuCESA10)主要促进亚麻韧皮纤维细胞的伸长。LuCESA5在TOP和MID的表达极显著高于BOT区(P<0.01),而在TOP和MID的表达无显著性差异。LuCESA6在茎不同部位的表达无显著性差异。LuCESA9和LuCESA10不仅在亚麻TOP区的表达较高,在BOT和MID区也具有较高的表达,表明这两个基因也参与细胞壁加厚过程。
2.3.2 LuCESAs在亚麻不同器官中的表达分析 图3-B和3-D表明在纤维素合酶基因家族中,除了LuCESA3和LuCESA7,其余的LuCESAs成员都在幼叶中有显著高表达(P<0.01)。LuCESA1、LuCESA3、LuCESA9和LuCESA10等基因在根中的表达较低,达到极显著水平(P<0.001),而LuCESA7在根中的表达与茎BOT区中的表达没有显著差异,而在幼叶和成熟叶中的表达极显著低于根和茎BOT区(P<0.001)。
LuCESA1、LuCESA3、LuCESA9和LuCESA10在茎和叶中都表达,在TOP和YL的表达更高。结合纤维素合成酶复合体的模式,LuCESA1、LuCESA3、LuCESA9和LuCESA10可能参与初生壁的形成。
2.4 LuSuSy和LuXTH4在快速生长期不同组织和茎韧皮纤维中的表达分析
蔗糖合酶(SuSy)和木聚糖水解酶(XTH)在细胞壁形成与加厚过程中起作用。本研究检测了SuSy和XTH4的表达情况(图4)。结果显示LuSuSy在亚麻幼嫩组织中相对表达量较高(图4-A),在茎的TOP区和幼叶中的表达显著高于其他部位。LuXTH4在茎、叶和根中的表达都较低(图4-B),尽管LuXTH4在亚麻麻茎MID区的表达显著高于TOP区(P<0.05,图4-C),但在幼叶和根中LuXTH4明显比在茎中活跃(图4-D)。表明LuSuSy和LuXTH4在亚麻细胞壁细胞形成和增大过程中起作用。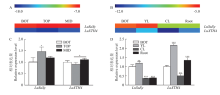 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4 LuSuSy和LuXTH4的表达分析
-->Fig. 4Expression of LuSuSy and LuXTH4
-->
尽管细胞壁形成相关基因LuBGAL20、LuBGAL22、LuCESA4、LuCESA5、LuCESA6和LuXTH4在不同部位和器官中的表达量较低,但其表达具有显著性差异,研究认为其在亚麻纤维发育表达水平上仍有一定的生物学意义。
3 讨论
亚麻是研究韧皮纤维细胞发育理想的模式植物[25]。本研究通过观察亚麻茎纤维细胞细胞壁的发育,发现亚麻茎SP点基部和中部在细胞壁形态上没有明显区别,只有细胞壁的加厚,没有出现明显的分层,说明此时细胞还未进入次生细胞壁加厚阶段。国内研究认为亚麻纤维细胞壁加厚阶段在现蕾期到绿熟期[26-27],而GORSHKOVA等[28]认为快速生长期伴随次生细胞壁加厚过程,本文的结果表明至少在快速生长期后期,仍未出现次生壁加厚情况。β-半乳糖苷酶是糖苷水解酶家族成员,广泛分布于植物组织中,在植物果实生长成熟[29-30]过程中研究较多[31]。BGALs家族基因在亚麻中预测有30多个成员,多数在亚麻细胞壁发育过程中的功能并不清楚[7]。在亚麻茎韧皮纤维细胞中,发现多个BGALs在幼嫩的茎中高表达。结合这些基因在幼叶、成熟叶、根中的表达(图2-D)情况,认为在亚麻快速生长期,LuBGAL1主要在SP点下部表达,LuBGAL9在全株的表达较高,LuBGAL5主要在幼叶和幼茎(SP点上部)起作用,与前人研究结果一致[7]。LuBGAL3不仅在茎中表达,在叶中表达更高,LuBGAL6在幼茎、幼叶和根中有较高的表达量,LuBGAL20和LuBGAL22在开花前各部位表达都较低,与NEIL HOBSON[7]的结果有差异。前人根据BGAL蛋白序列将LuBGAL1、LuBGAL3、LuBGAL5、LuBGAL6和LuBGAL9划分在A1族[7],认为这些半乳糖苷酶在亚麻韧皮纤维发育中有重要作用,本研究证实了这个推论。
纤维素合酶基因在亚麻中的研究主要从2012年亚麻全基因组测序完成后开始,通过拟南芥、杨树等植物中的同源序列分析,推测亚麻中有10—16个CESA基因可能参与不同阶段细胞壁合成过程。本研究结果显示,LuCESA1、LuCESA3、LuCESA9、LuCESA10主要促进韧皮纤维细胞初生细胞壁的形成,与前人报道相同[32]。已有研究报道LuCESA4、LuCESA7和LuCESA8在木质部中参与亚麻次生细胞壁的合成[23,32],亚麻外茎组织中,LuCESA4、LuCESA7和LuCESA8在亚麻韧皮纤维细胞的表达模式与前人结果一致[23]。结合三者的表达情况和茎的横切,认为亚麻快速生长期的后期,尽管细胞壁没有明显分层,但与次生壁相关的基因已经开始表达。LuCESA5、LuCESA6在CESA家族中处于同一亚族,亲缘关系非常近[21, 23, 32],由于在检测的组织中它们的表达量都较低,因此认为LuCESA5、LuCESA6可能在亚麻韧皮纤维细胞发育过程中出现功能冗余。
在植物细胞形成和增大过程中,蔗糖合酶(SuSy)为细胞壁纤维素合成提供底物UDPG,糖苷水解酶家族GH16中的木聚糖水解酶(XTH)[33]催化木葡聚糖分子的断裂和重新连接,参与细胞壁的合成或松弛过程,二者在初生壁和次生壁的合成中起重要作用。LuSuSy在亚麻幼嫩组织中相对表达量较高说明LuSuSy在新生细胞中提供底物;同时,LuSuSy在麻茎中的表达量高于成熟叶和根,说明在亚麻快速生长期,茎是重要的代谢库,需要更多的UDPG参与到韧皮纤维细胞壁加厚的过程。尽管LuXTH4在亚麻麻茎MID区的表达显著高于TOP区(P<0.05),但在幼叶中表达量更高,说明LuXTH4可能主要在幼叶中参与细胞壁的形成。LuSuSy和LuXTH4的表达与茎发育变化呈现一致性。
前人对亚麻韧皮纤维发育的研究,在研究材料、取材时期和取材部位上多有不同[20, 22-23],不同研究者的结果存在差异。研究同一时期,同一植株的不同部位细胞壁形成相关基因的表达,为亚麻快速生长期韧皮纤维细胞的发育提供基本的理论支持。
4 结论
4.1 快速生长期亚麻茎韧皮纤维细胞厚度TOP<MID<BOT区,SP点下部细胞壁没有次生加厚过程。4.2 多个BGALs共同参与亚麻细胞壁的形成,其中LuBGAL3、LuBGAL5、LuBGAL6和LuBGAL9主要促进亚麻细胞壁细胞伸长,LuBGAL1主要参与亚麻细胞壁加厚过程。
4.3 LuCESA1、LuCESA3、LuCESA9、LuCESA10参与初生壁的形成。
4.4 LuSuSy和LuXTH4的表达与茎发育变化呈现一致性。
Tabel 1
附录1
附录1引物序列
Tabel 1Primer sequences
| 基因名称 Genes | 基因代码 The genetic code | 引物序列 Primer sequences |
|---|---|---|
| LuCESA1 | Lus10018902F | GACTAATGGCCAGCAGATTTCGG |
| Lus10018902R | TCGAGATAAGGAGACGACTGCAC | |
| LuCESA3 | Lus10007538F | ATCTCCTGGACGGTACTCGATG |
| Lus10007538R | TCTCCCAGTCTAATGGTTGCTCTG | |
| LuCESA4 | Lus10008225F | TGCAGAATGGACAGAACGAGTGG |
| Lus10008225R | GTTGTCTGGCATCAGCGGTTAAG | |
| LuCESA5 | Lus10003525F | GGCTATGAATGCACTGCGATGG |
| Lus10003525R | TTAATGAGGAGGTCGTGGCTCTGG | |
| LuCESA6 | Lus10002940F | CTGTTCTGCGGGTCTAGGAAGAAC |
| Lus10002940R | TCCAGCCAACCTGTTCAACTTC | |
| LuCESA7 | Lus10007296F | GGTTTGCTTTCTCTTGGGTGCTG |
| Lus10007296R | ACCTGGCCTTTCAAACCTTGC | |
| LuCESA8 | Lus10029245F | GGTTTGCTTTCTCTTGGGTGTTGG |
| Lus10029245R | ACCTGGCCTTTCAAACCTTGC | |
| LuCESA9 | Lus10002939F | CAGAGAGGCCATCCAAGTTATCAG |
| Lus10002939R | CGAACCATATATCCAGCCAACCTC | |
| LuCESA10 | Lus10028597F | TGAAGCAGGAGAAGGGAATGATGC |
| Lus10028597R | GAGCATCGTCAGCCATTTGAAGC | |
| LuSuSy | Lus10030176F | TAGATTTCGGGATGGCCGTTTG |
| Lus10030176R | ACGGATGACAACATTGCATTGCC | |
| LuXTH4 | Lus10011052F | CCACCTCGATAACTACACTGGAAC |
| Lus10011052R | TCCACCAACCATCTTGATGTGC | |
| LuBGAL1 | Lus10008974F | ACTCATCGGTAGCGCTTATGGC |
| Lus10008974R | GAAGTGTAAGCCAACATTCGCAAG | |
| LuBGAL3 | Lus10006009F | TGCATTCCTGGCGAACTACGAC |
| Lus10006009R | GCACTCTCGGAGCAGAAACCTTTG | |
| LuBGAL5 | Lus10000701F | AGCTGTCATGCCTTGCACTCATAC |
| Lus10000701R | ACCAATTCTGTCCAACACAAAGCC | |
| LuBGAL6 | Lus10015625F | GGTGAACATCTGAGCCTTCATTCC |
| Lus10015625R | GCAGGAGCATTGAATGTGGTCTTG | |
| LuBGAL9 | Lus10024292F | TAGCGGTTGGATTGCCGAATG |
| Lus10024292R | ACCATCTGTGGCCTGTCAAGTC | |
| LuBGAL20 | Lus10003343F | AGCTGTTTGCTTCTCAGGGAGAC |
| Lus10003343R | TTGAGCAACGGCCATTTGAGC | |
| LuBGAL22 | Lus10025108F | ACCGAGAATTGGTCTGGATGGTG |
| Lus10025108R | AGTTGACCGGTGAGGATTAGTGG | |
| LuETIF1 | Lus10015124 F | GATCCAGCTTCAAGGTGACCAG |
| Lus10015124 R | TCACAATTCCAGCCTGAACGAG |
新窗口打开
The authors have declared that no competing interests exist.
