0 引言
【研究意义】分子育种技术在家畜育种中的应用,不仅可以增加选择强度、提高选择准确性,同时还可实现早期个体的基因型选择[1-2]。而分子育种技术应用的前提是找到影响家畜经济性状的主效基因,本研究以初步探索ERAP2、Cwc27、PPFIBP2、THADA、ZNF804B和AMHR2 多态性与新疆褐牛体细胞评分及泌乳性状的相关性,以期为新疆褐牛产奶性状的分子标记辅助育种提供理论依据。【前人研究进展】近十年来,遗传标记育种、转基因技术、克隆技术等高新生物技术的研究取得了一定的进展,给奶牛育种工作带来了新机遇。众多的分子标记中,SNP(single nucleotide polymorphism)标记是当前遗传标记研究中最多的,作为目前最具发展潜力的分子标记,因其在基因组中数量多、分布广,已经广泛应用于基因组制图、疾病相关性分析、群体遗传学及药物研究等众多领域[3]。目前,越来越多的泌乳性状QTL和候选基因被相继报道[4-5],如王丽娟等[6]发现bPRL第4外显子C8377G位点与产奶量显著相关,VIILALA等[7]发现GHR上F279Y突变显著影响乳蛋白率和乳脂率,LEONARD等[8]对13 662头牛群体进行OPN多态性研究,发现第8 514位点突变与乳脂率和乳蛋白率性状存在显著关联,王杰等[9]研究发现,中国荷斯坦牛OPN内含子上的多态性与乳脂率和乳蛋白率显著关联,张晓东等[10]研究发现,DGAT1外显子8的多态性与中国荷斯坦牛的乳脂率显著相关,贾晋等[11]研究发现,DGAT1不同基因型之间的305 d产奶量和乳脂率有显著差异外,还发现该基因对乳蛋白率影响显著。季敏等[12]采用直接测序和PCR-RFLP技术研究槟榔江水牛STAT5A变异,结果发现STAT5A多态性与产奶量及乳脂率等产奶性状具有显著相关性。研究表明,利用候选基因策略寻找影响产奶性状的主效基因进而应用于奶牛的遗传改良,从分子遗传角度来说是可行的[13]。【本研究切入点】新疆褐牛为乳肉兼用型品种,其作为新疆地区特有的品种资源,具有高乳蛋白、高乳脂、低体细胞数、抗逆性强、适应性好,乳肉平衡等优秀特质。产奶量和乳成分性状(主要包括乳脂率、乳蛋白率、乳糖率等)作为奶牛生产中的重要经济性状,属典型微效多基因控制的数量性状,不仅对奶业发展有巨大影响[14],而且在新疆褐牛产业发展中也占有重要地位。乳房炎是世界奶业的常见疾病,对牛产奶量和乳品质都会造成较大影响,通常以体细胞数为指示性状,选育提高乳房炎抗性是增加奶牛生产效益的重要措施。基质辅助激光解吸电离飞行时间质谱技术(MALDI-TOF-MS)是近年来应用在生物大分子检测领域的一项新技术,在基因组SNP的分型检测中具有高精准度、灵敏度高、一次可检测多个SNPs且成本低和周期短等优点。该分型技术是在单碱基延伸的基础上与质谱技术相结合从而进行分型,其原理是通过基质辅助激光解吸电离飞行时间质谱技术,检测延伸产物分子量的大小,应用专用的分析软件进行突变位点的分型。目前,国内外已有很多文献报道生物质谱技术在SNP分型方面的应用,研究表明,质谱检测法与其他检测方法相比,准确性更高[15-17]。【拟解决的关键问题】将分子育种技术应用于新疆褐牛育种工作中势在必行,但目前对新疆褐牛泌乳性状相关基因的研究还相对较少,因此,本文根据前期中国荷斯坦牛重测序结果选取了10个SNPs位点,探索这些SNPs的遗传多态性,并分析其与新疆褐牛SCS、305d产奶量、乳脂率、乳蛋白率、乳糖率和总固体数之间的相关性,试图寻找新疆褐牛体细胞评分和泌乳性状的有效分子标记,为今后新疆褐牛乳用性能分子育种奠定基础。1 材料与方法
1.1 材料
1.1.1 实验动物 本研究以新疆地区新疆乌鲁木齐种牛场和新疆天山畜牧生物工程股份有限公司2个牛场169头具有DHI测定记录、年龄及胎次等数据的新疆褐牛母牛作为实验动物,同时采集奶样和血样。奶样自2008年至2015年期间每月采集一次送至新疆维吾尔自治区奶业办公室进行DHI测定,获得乳脂率、乳蛋白率、乳糖率、总固体以及体细胞计数等表型数据;血液为2014年分别在两个牛场采集,EDTA抗凝,用于基因型检测。1.1.2 性状表型记录 产奶性状记录包括305 d产奶量(不足305 d的产奶量根据付雪峰[18]制定的新疆褐牛305 d产奶量校正系数进行校正)、乳脂率、乳蛋白率、乳糖率、总固体和体细胞计数(Somatic cell counts,SCC)。在进行统计分析时,利用公式将体细胞数转换为呈正态分布的体细胞评分(Somatic cell score,SCS):SCS=log2(SCC/100000)+3,并保留2位有效数字。对DHI测定记录的质控遵循以下标准[19]:305 d产奶量为1 000—18 000 kg;乳脂率为2%—7%;乳蛋白率为2%—6%;乳糖率为2%—5.5%;总固体为9%—18%;SCC为1—100 000。
1.1.3 影响因素水平划分 数据来自新疆乌鲁木齐种牛场和新疆天山畜牧生物工程股份有限公司,由于新疆天山畜牧生物工程股份有限公司样本量较少,在分析时不考虑场效应;年份和季节均以自然产犊年季划分,以中国气象数据网公布的新疆地区近30年的平均气温记录,依据候温法将新疆地区的季节划分为春季(4—5月)、夏季(6—8月)、秋季(9月)、冬季(10月至次年3月),各月份、各季节平均气温见表1和表2;胎次按自然胎次划分6个水平(1—6胎及以上);泌乳月划分为12个水平。
Table 1
表1
表11981-2010年各月份平均气温
Table 1Average temperature of each month from 1981 to 2010
| 月份Month | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 温度Temperature (℃) | -14.2 | -10.5 | 0.5 | 12.3 | 19 | 23.8 | 32.6 | 24.1 | 18 | 9 | -4.1 | -11 |
新窗口打开
Table 2
表2
表21981-2010年各季节平均气温
Table 2Average temperature of each season from 1981 to 2010
| 季节 Season | 春节 Spring | 夏季 Summer | 秋季 Autumn | 冬季 Winter |
|---|---|---|---|---|
| 温度Temperature (℃) | 15.6 | 26.8 | 18 | -5.1 |
新窗口打开
1.2 方法
1.2.1 血液基因组DNA提取 利用苯酚-氯仿抽提法进行基因组DNA提取,TE缓冲液溶解后,利用NANODROP2000紫外分光光度计和凝胶电泳检测DNA提取质量,将合格DNA放置于-20°冰箱保存备用。1.2.2 DNA池重测序筛选SNP 课题组前期以15头新疆褐牛为试验对象,运用DNA混池重测序技术筛选到大量SNPs(表3),为保证后续试验的可行性和准确性,将对筛选出的SNPs进行进一步的筛选,筛选条件如下:①变异位点位于外显子区域;②变异类型选取错义突变;③一个基因出现多个位点变异的SNPs;④样本基因型为杂合变异;⑤测序深度大于40,测序深度越深,变异位点准确性越高。
Table 3
表3
表310个SNPs相关信息
Table 3Information of 10 SNPs
| SNP | 染色体 Chromosome | 基因 Gene | 物理位置 Physical location | 突变类 Mutation type |
|---|---|---|---|---|
| SNP1 | 7 | ERAP2 | Chr7:98741711bp | T/C |
| SNP2 | 7 | ERAP2 | Chr7:98736141bp | A/G |
| SNP3 | 20 | Cwc27 | Chr20:14533269bp | T/A |
| SNP4 | 4 | ZNF804B | Chr4:73896754 bp | A/G |
| SNP5 | 5 | AMHR2 | Chr5:26753813 bp | C/T |
| SNP6 | 5 | AMHR2 | Chr5:26758055 bp | C/G |
| SNP7 | 11 | THADA | Chr11:25592180 bp | A/C |
| SNP8 | 11 | THADA | Chr11:25592332 bp | T/C |
| SNP9 | 15 | PPFIBP2 | Chr15:45667492 bp | C/G |
| SNP10 | 4 | ZNF804B | Chr4:73894130 bp | C/T |
新窗口打开
文中选择错义突变SNP的原因:错义突变是编码某种氨基酸的密码子经碱基替换以后,变成编码另一种氨基酸的密码子,从而使多肽链的氨基酸种类和序列发生改变。而氨基酸是构成蛋白质的基本单位,氨基酸的改变可能会改变其编码的蛋白质,而蛋白质对基因的表达有调节作用,蛋白质的改变会引起基因的功能变化[20]。
1.2.3 SNP分型 将上一步确定的SNP利用Illumina公司的MassARRAY Genotype分型技术进行SNP分型。主要步骤如下[21]:(1)引物设计:对目的SNP做PCR多重设计,使每一个SNP具有一对扩增引物及一个延伸引物,以便待分型的多个核苷酸片段能够在相同的反应体系中做多重PCR反应;(2)多重PCR扩增;(3)SAP反应;(4)延伸反应;(5)去盐。将结束反应的板内均匀填充树脂并放置10 min晾干,每个孔加入16 μL DEPC水,并在Dimpe板上缓慢扣放样本板,轻轻敲打使树脂能够落入样本孔中,离心均匀1 h。
根据已知SNP位点序列信息,使用Sequenom MassArray Assay Design软件对待测SNPs进行引物设计,引物设计中包括PCR扩增引物和单碱基延伸引物。引物设计完成后经软件测试可以使用后交由生物公司进行引物合成。共设计10对引物,10个SNPs由DNA混池全基因组重测序结果提供,表4为各位点引物信息表。
Table 4
表4
表4引物信息表
Table 4Primer information
| SNPs | 突变类型 Mutation type | 一级引物 Primary amplification primer | 二级引物 Secondary amplification primer | 延伸引物 Extend amplification primer |
|---|---|---|---|---|
| SNP_1 | T98741711C | ACGTTGGATGTCTCTCCCGTTTAGGATGAC | ACGTTGGATGGCTTCATTGGAGATAGGATG | ccccCTTCAAAACACGTGTTCG |
| SNP_2 | G98736141A | ACGTTGGATGTCAGGCCGAAGGACAGACTA | ACGTTGGATGTCCTGTGAAATCAGTTGGAG | TTACTAGCTGAAACGCAT |
| SNP_3 | T14533269A | ACGTTGGATGGCTTGATAACCGTGCTTACC | ACGTTGGATGAAAGCGGGAACTCTTAGCAG | aggAAGCAGAAAATTCCGCAA |
| SNP_4 | G73896754A | ACGTTGGATGTCCCAGCCACGTTTTAATCC | ACGTTGGATGGGGATGACCTGCTAAGAAAG | GGTGTGCAGGGATGA |
| SNP_5 | C26753813T | ACGTTGGATGAGGACTGCTGGGATGCAGAC | ACGTTGGATGAGGCGCTGCTGTACACACTC | ccttTACACACTCAGCTGTTAGC |
| SNP_6 | C26758055G | ACGTTGGATGCTGAATCTGACTCTGGCTCG | ACGTTGGATGGACCCTAAGGCTGTTCTCTG | ccgGCTGCAGCGGAAGGCCTGCCGAGA |
| SNP_7 | A25592180C | ACGTTGGATGTGGCTCTTCTGGAAGGAAAG | ACGTTGGATGCGTTAACTGATTCGCTGCTG | ggtggTGCTGCAGGAGAATCCA |
| SNP_8 | T25592332C | ACGTTGGATGATGCTCCGTCACCTTCAGAG | ACGTTGGATGAAAAATTCAGCAGTGGGAGG | AGGCAGCACTCTGAAGAGC |
| SNP_9 | C45667492G | ACGTTGGATGATCAAGTGTGCTGAGGCCC | ACGTTGGATGATCGACGGACTACATTTGCC | tgaCCAATGGAGCCCAGCAACA |
| SNP_10 | C73894130T | ACGTTGGATGTGGAATCTCACCCAGACATC | ACGTTGGATGTCACTCATGCCAAGCCAATG | caccAACATTTACAAGCATAGTGATG |
新窗口打开
1.2.4 统计分析 计算基因型频率、等位基因型频率并检测各位点的哈代-温伯格平衡情况。运用SAS 8.1的GLM过程对基因型与SCS和泌乳性状进行关联分析的线性模型为:
Yijklm=μ+Gi+Lj+Sk+Pl+eijklm
式中,Yijklm为产奶性状表型值(305 d 产奶量、乳脂率、乳蛋白率、乳糖率、总固体数和SCS);μ为群体均值;Gi为基因型效应;Lj为年季组合效 应;Sk为胎次效应;Pl为泌乳月效应;eijklm为随机误差。
为保证多重比较过程总的I型错误概率低于0.05和0.01,将显著性水平校正为P<0.005(显著)和P<0.001(极显著)。
2 结果
2.1 产奶性状描述性统计
根据2008年至2015年课题组对两个新疆褐牛牛群进行性能测定数据,统计分析得出SCS和泌乳性状的简单统计量,结果见表5。Table 5
表5
表5泌乳性状及SCS描述性统计量
Table 5Descriptive statistics of milk production traits and SCS
| 性状 Traits | 样本量 N | 最小值 Minimum | 最大值 Maximum | 平均值 Mean | 标准差 Std. Deviation | 变异系数 CV |
|---|---|---|---|---|---|---|
| 305 d产奶量 Milk yield of 305 d (kg) | 386 | 1566.24 | 12823.23 | 4716.33 | 1441.83 | 0.31 |
| 乳脂率 Milk fat percentage (%) | 2125 | 2.00 | 7.00 | 3.91 | 0.85 | 0.22 |
| 乳蛋白率 Milk protein percentage (%) | 2125 | 2.25 | 5.56 | 3.39 | 0.35 | 0.11 |
| 乳糖率 Lactose percentage (%) | 1982 | 2.20 | 5.48 | 4.79 | 0.39 | 0.08 |
| 总固体数 Total solid content (%) | 1982 | 9.06 | 16.35 | 11.94 | 0.96 | 0.80 |
| 体细胞评分 SCS | 1935 | -1.64 | 10.90 | 5.05 | 2.12 | 0.42 |
新窗口打开
2.2 基因型检测
利用MassArray技术对新疆褐牛169个样本10个SNPs进行基因型分型,通过质谱软件统计分析得到了分型结果,10个SNPs均具有多态性,且未识别出的碱基数较少,基因多态性以AG、CT基因型为主,可以进行下一步SNP位点多态性与产奶性状遗传效应的研究。2.3 群体等位基因频率和基因型频率
利用Excel统计上述10个SNPs的等位基因频率和基因型频率,并进行哈代-温伯格平衡检验,结果见表6。χ2检验表明SNP1、SNP2、SNP3、SNP4、SNP6、SNP 7、SNP 8、SNP 9和SNP 10这9个位点均处于Hardy-Weinberg平衡状态(P>0.05)。SNP5、SNP6和SNP9位点的优势等位基因为C,其等位基因频率分别为0.985、0.912、0.736,其中SNP5、SNP6、SNP9位点的优势基因型为CC,其优势基因型频率分别为0.975、0.843、0.534;SNP1位点的优势基因型为CT,优势基因型频率为0.435。SNP2、SNP3、SNP4、SNP7位点的优势等位基因为A,其优势等位基因频率分别为0.607、0.801、0.710、0.984,其中SNP3、SNP4、SNP7位点的优势基因型为AA,其优势等位基因型频率分别为0.641、0.503、0.968;SNP2位点的优势基因型为AG,其基因型频率为0.448。SNP8和SNP10位点的优势等位基因为T,其优势等位基因频率分别为0.984、0.587,其中SNP8位点的优势基因型为TT,其基因型频率为0.968,SNP10的优势基因型为CT,其基因型频率为0.439。SNP5、SNP6、SNP7、SNP8位点处于低度多态(PIC<0.25),其余6个位点均处于中度多态(0.25<PIC<0.5)。Table 6
表6
表6新疆褐牛10个位点的基因型频率和等位基因频率及χ2检验
Table 6Genotype and allelic frequencies and Hardy-Weinberg equilibrium χ2 test of 10 SNPs in Xinjiang Brown Cattle
| SNPs | 基因型(个数) Genotypes(number) | 基因型频率 Frequencies | 等位基因 Allele | 等位基因频率 Frequencies | 多态信息含量PIC | 有效等位基因数 Ne | 哈代-温伯格平衡 H-W equilibrium | |
|---|---|---|---|---|---|---|---|---|
| χ2 | P | |||||||
| SNP_1_ERAP2 | TT(24) | 0.156 | T | 0.373 | 0.36 | 1.879 | 0.760 | 0.383 |
| CT(67) | 0.435 | C | 0.627 | |||||
| CC(63) | 0.409 | |||||||
| SNP_2_ERAP2 | AA(59) | 0.383 | A | 0.607 | 0.36 | 1.912 | 0.569 | 0.451 |
| AG(69) | 0.448 | G | 0.393 | |||||
| GG(26) | 0.169 | |||||||
| SNP_3_CWC27 | AA(150) | 0.641 | A | 0.801 | 0.27 | 1.467 | 0.006 | 0.936 |
| AT(50) | 0.321 | T | 0.199 | |||||
| TT(6) | 0.038 | |||||||
| SNP_4_ZNF804B | AA(79) | 0.503 | A | 0.710 | 0.33 | 1.700 | 0.005 | 0.942 |
| AG(65) | 0.414 | G | 0.290 | |||||
| GG(13) | 0.083 | |||||||
| SNP_5_AMHR2 | CC(159) | 0.975 | C | 0.985 | 0.03 | 1.031 | 24.876 | 0.000 |
| CT(3) | 0.018 | T | 0.015 | |||||
| TT(1) | 0.006 | |||||||
| SNP_6_AMHR2 | CC(134) | 0.843 | C | 0.912 | 0.15 | 1.191 | 3.047 | 0.081 |
| CG(22) | 0.138 | G | 0.088 | |||||
| GG(3) | 0.019 | |||||||
| SNP_7_THADA | AA(153) | 0.968 | A | 0.984 | 0.03 | 1.032 | 0.041 | 0.840 |
| AC(5) | 0.032 | C | 0.016 | |||||
| SNP_8_THADA | TT(153) | 0.968 | T | 0.984 | 0.03 | 1.032 | 0.041 | 0.840 |
| CT(5) | 0.032 | C | 0.016 | |||||
| SNP_9_PPFIBP2 | CC(86) | 0.534 | C | 0.736 | 0.31 | 1.636 | 0.244 | 0.621 |
| CG(65) | 0.404 | G | 0.264 | |||||
| GG(10) | 0.062 | |||||||
| SNP_10_ZNF804B | CC(30) | 0.194 | C | 0.413 | 0.37 | 1.941 | 1.403 | 0.236 |
| CT(68) | 0.439 | T | 0.587 | |||||
| TT(57) | 0.368 | |||||||
新窗口打开
2.4 10个SNPs与泌乳性状及SCS的关联分析
表7显示10个SNPs不同基因型对新疆褐牛305 d产奶量、乳脂率、乳蛋白率、乳糖率、总固体数及SCS的影响。由表可知,SNP1和SNP2位点对新疆褐牛305 d 产奶量有极显著影响(P<0.001),基因型为TT和GG的个体305 d 产奶量最高。SNP3位点对乳脂率和总固体含量有显著影响(P<0.005),对SCS有极显著影响(P<0.001),其中纯合型TT个体乳脂率和总固体均最高,而杂合型AT个体两个指标均较低,AA型个体SCS最高。SNP6对乳蛋白率和总固体性状均有极显著影响(P<0.001),其中GG型个体具有较高的乳蛋白率和总固体含量。SNP9位点对乳糖率有显著影响(P<0.005),其中基因型为GG的个体乳糖率显著高于CC型,而GC型显著高于CC型。Table 7
表7
表710个SNP与泌乳性状及SCS的关联分析(最小二乘均值±标准误)
Table 7Association of 10 SNPs with milk production traits and SCS (LSM± SE)
| SNP | 基因型 Genotype | 305 d产奶量 305 d milk (kg) | 乳脂率 Fat percentage (%) | 乳蛋白率 Protein percentage (%) | 乳糖率 Lactose percentage (%) | 总固体 Total solid (%) | 体细胞评分SCS Somatic cell score |
|---|---|---|---|---|---|---|---|
| SNP1_ ERAP2 | CC | 4151.17±141.60C | 4.01±0.05 | 3.41±0.02 | 4.72±0.03 | 12.12±0.06 | 5.15±0.15 |
| TT | 5124.32±167.13A | 3.94±0.062 | 3.41±0.02 | 4.79±0.03 | 12.13±0.07 | 5.26±0.17 | |
| CT | 4727.86±115.66B | 3.88±0.05 | 3.42±0.02 | 4.77±0.03 | 12.06±0.06 | 4.99±0.16 | |
| P值 | <0.0001** | 0.0474 | 0.9979 | 0.0072 | 0.453 | 0.1674 | |
| SNP2 _ERAP2 | AA | 4151.17±141.60C | 4.01±0.05 | 3.41±0.02 | 4.72±0.03 | 12.11±0.06 | 5.16±0.15 |
| GG | 5124.32±167.13A | 3.96±0.06 | 3.42±0.02 | 4.78±0.03 | 12.14±0.07 | 5.25±0.17 | |
| AG | 4727.86±115.66B | 3.89±0.05 | 3.42±0.02 | 4.78±0.03 | 12.07±0.06 | 4.99±0.15 | |
| P值 | <0.0001** | 0.0821 | 0.9303 | 0.0051 | 0.5703 | 0.1768 | |
| SNP3_ Cwc27 | AA | 4650.79±117.09 | 3.98±0.04b | 3.43±0.02 | 4.76±0.03 | 12.13±0.06b | 5.19±0.14A |
| TT | 4626.74±359.92 | 4.16±0.11a | 3.42±0.04 | 4.75±0.05 | 12.32±0.12a | 3.89±0.28C | |
| AT | 4665.02±125.65 | 3.84±0.06c | 3.33±0.02 | 4.76±0.03 | 11.98±0.07c | 5.08±0.17B | |
| P值 | 0.9923 | 0.0041* | 0.0721 | 0.9772 | 0.0014* | <0.0001** | |
| SNP4_ZNF804B | AA | 4835.48±123.01 | 3.92±0.05 | 3.41±0.02 | 4.77±0.03 | 12.08±0.06 | 5.17±0.15 |
| GG | 4380.02±271.77 | 4.03±0.08 | 3.45±0.03 | 4.71±0.04 | 12.14±0.10 | 4.85±0.23 | |
| GA | 4522.45±118.62 | 3.98±0.05 | 3.41±0.02 | 4.74±0.03 | 12.12±0.07 | 5.12±0.16 | |
| P值 | 0.5575 | 0.2462 | 0.4238 | 0.2159 | 0.6966 | 0.2848 | |
| SNP5 _AMHR2 | CC | 4662.44±95.8 | 3.96±0.04 | 3.42±0.17 | 4.76±0.03 | 12.11±0.06 | 5.15±0.14 |
| TT | 4498.83±502.61 | 3.30±0.40 | 3.42±0.16 | 4.72±0.71 | 11.94±0.16 | 4.22±0.38 | |
| CT | 3735.05±999.44 | 3.79±0.15 | 3.38±0.06 | 4.80±0.28 | 11.84±0.61 | 4.84±1.43 | |
| P值 | 0.6157 | 0.1421 | 0.8081 | 0.8441 | 0.4638 | 0.0302 | |
| SNP6_ AMHR2 | CC | 4680.69±103.63 | 3.97±0.04 | 3.41±0.016B | 4.76±0.027 | 12.13±0.06B | 5.12±0.14 |
| GG | 4182.67±710.82 | 3.99±0.18 | 3.68±0.07A | 4.83±0.08 | 12.56±0.19A | 4.85±0.45 | |
| GC | 4562.74±191.04 | 3.79±0.075 | 3.39±0.029C | 4.67±0.03 | 11.87±0.08C | 5.19±0.20 | |
| P值 | 0.6694 | 0.0373 | 0.0005** | 0.0084 | <0.0001** | 0.7507 | |
| SNP7_THADA | AA | 4676.69±95.13 | 3.94±0.042 | 3.41±0.016 | 4.75±0.02 | 12.09±0.06 | 5.13±0.14 |
| CA | 4171.28±379.06 | 4.06±0.11 | 3.48±0.04 | 4.73±0.05 | 12.26±0.12 | 4.95±0.30 | |
| P值 | 0.1872 | 0.3294 | 0.1046 | 0.6877 | 0.1508 | 0.5081 | |
| SNP8_THADA | CT | 4172.26±380.28 | 4.06±0.11 | 3.48±0.046 | 4.73±0.05 | 12.26±0.12 | 4.96±0.30 |
| TT | 4677.38±95.47 | 3.95±0.04 | 3.41±0.01 | 4.75±0.02 | 12.10±0.06 | 5.13±0.14 | |
| P值 | 0.1889 | 0.3306 | 0.1063 | 0.669 | 0.1535 | 0.5311 | |
| SNP9_ PPFIBP2 | CC | 4503.15±112.21 | 3.96±0.05 | 3.40±0.02 | 4.73±0.03c | 12.07±0.06 | 5.23±0.15 |
| GG | 5091.64±299.97 | 3.96±0.08 | 3.46±0.03 | 4.87±0.04a | 12.24±0.09 | 4.89±0.22 | |
| GC | 4845.89±125.44 | 3.95±0.05 | 3.42±0.02 | 4.76±0.03b | 12.12±0.07 | 5.06±0.15 | |
| P值 | 0.0236 | 0.9885 | 0.1059 | 0.0015* | 0.1054 | 0.1054 | |
| SNP10_ZNF804B | CC | 4490.92±188.42 | 4.05±0.06 | 3.44±0.03 | 4.75±0.03 | 12.19±0.07 | 4.77±0.18 |
| CT | 4621.98±119.11 | 3.96±0.05 | 3.42±0.02 | 4.75±0.03 | 12.12±0.07 | 5.22±0.16 | |
| TT | 4762.23±133.35 | 3.92±0.05 | 3.40±0.02 | 4.77±0.03 | 12.07±0.07 | 5.17±0.15 | |
| P值 | 0.3851 | 0.0999 | 0.2199 | 0.6139 | 0.127 | 0.0073 |
新窗口打开
关联分析结果表明,SNP1和SNP2与新疆褐牛305 d产奶量达到极显著关联(P<0.001);SNP3与乳脂率和总固体达到显著关联(P<0.005),与SCS性状达到极显著关联(P<0.001);SNP6与乳蛋白率和总固体均达到极显著关联(P<0.001);SNP9与乳糖率达到显著关联(P<0.005)。
2.5 单倍型与连锁不平衡分析
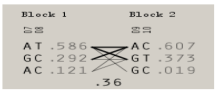
运用Haploview软件对所有SNPs进行了单倍型与连锁不平衡分析,如图1所示,得出10个SNP位点的连锁不平衡检测结果。图的上方为标记在基因中的位置,红色方块表示D’>0.8,方块颜色越浅表示D’值越低,加框的标记处于同一单倍型域中。图2为单倍型组成,连线表示单倍型域可能的单倍型组合,下方数字是单倍型域间主要的单倍型组合的比例。由图2可以看出,本研究群体中,10个SNPs共构成2个单倍型模块,分别命名为Block1、Block2,其中Block2处于强连锁不平衡状态(r2>0.6)。Block1包含SNP4和SNP10,其中D’和r2值为1.000和0.587,因此Block1为连锁不平衡状态,构成3种单倍型,其中TA、CG和CA单倍型频率分别为0.596、0.292、0.121;Block2包含SNP1和SNP2,其中D’和r2值为1.000和0.921,因此Block2为强连锁不平衡状态,构成3种单倍型,其中AC、GT和GC单倍型频率分别为0.607、0.373、0.019,两个单倍型模块之间的连锁不平衡系数D’为0.36(图2)。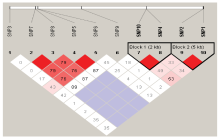 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图110个SNPs连锁不平衡估计
-->Fig. 1The linkage disequilibrium estimated between 10 SNPs
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2单倍型域中的单倍型组成
-->Fig.2Haplotype construction based on the blocks
-->
2.6 单倍型与泌乳性状及SCS关联分析
运用SAS8.1软件进行单倍型与新疆褐牛SCS及泌乳性状的关联分析,结果如表8可知,共存在5种单倍型(本研究群体中未发现GGCC个体),在进行各单倍型与新疆褐牛泌乳性状的关联分析时,均未发现单倍型与泌乳性状的关联性,可能是由于样本量较少,或者是还有其他标记与本研究的SNPs位点紧密连锁影响着新疆褐牛SCS和泌乳性状。Table 8
表8
表8单倍型与泌乳性状和SCS的关联分析(最小二乘均值±标准误)
Table 8The association of haplotype with milk production traits and SCS(LSM±SE)
| 单倍型(个数) Haplotype (number) | 乳脂率 Fat percentage (%) | 乳蛋白率 Protein percenyage (%) | 乳糖率 Lactose percentage (%) | 总固体 Total solid (%) | 体细胞评分SCS Somatic cell score | |
|---|---|---|---|---|---|---|
| Block1 | CCAA(1) | 3.07±0.33 | 3.23±0.13 | 5.08±0.15 | 11.39±0.33 | 3.70±0.77 |
| CCGG(13) | 4.04±0.09 | 3.45±0.03 | 4.73±0.04 | 12.15±0.10 | 4.83±0.23 | |
| TTAA(58) | 3.92±0.05 | 3.40±0.02 | 4.77±0.03 | 12.07±0.07 | 5.16±0.15 | |
| P值P value | 0.0229 | 0.1877 | 0.1845 | 0.0233 | 0.0078 | |
| Block2 | AACC(59) | 4.01±0.05 | 3.41±0.02 | 4.72±0.03 | 12.11±0.07 | 5.16±0.16 |
| GGTT(24) | 3.95±0.06 | 3.42±0.02 | 4.80±0.03 | 12.14±0.08 | 5.26±0.18 | |
| P值P value | 0.0945 | 0.8190 | 0.0207 | 0.5343 | 0.4514 |
新窗口打开
3 讨论
3.1 SNPs遗传多态性分析
采用MassArray技术对运用DNA混池重测序结果筛选的SNPs进行个体基因分型并检测其多态性。结果得知,10个位点均有多态性,卡方检验表明其中9个位点符合哈代-温伯格平衡状态,说明新疆褐牛在人工选育、迁移和遗传漂变等因素作用下,这些位点仍处于动态平衡中,反映了这些位点的选择压力不强;仅SNP5位点不符合哈代-温伯格平衡状态,说明该位点在新疆褐牛群体中具有较大的选择潜力。SNP5、SNP6、SNP7和SNP8位点均处于低度多态,且有效等位基因数目较少,即这4个位点在新疆褐牛群体中遗传变异为低等水平,4个位点位于AMHR2和THADA基因上,推测新疆褐牛在长期的育种选择及进化过程中,两个基因所受的选择压力不大;其余6个位点均处于中度多态,遗传变异较大。因此,综上所述,在新疆褐牛泌乳性状的选育过程中应当加强人工选择的强度。3.2 SNPs与泌乳性状及SCS关联分析
本文涉及的6个基因与牛产奶性状的关联性研究鲜有报道,本研究旨在初步探索这些基因与新疆褐牛SCS和泌乳性状的相关性。其中,SNP1和SNP2位于牛7号染色体ERAP2上(内质网氨基肽酶2),该基因是氨基肽酶M2家族中的一个多功能酶,是参与ER抗原加工的另一个氨基肽酶,具有高度多态性。有研究表明,ERAP2与一种以高血压和蛋白尿为特征的孕期综合征——先兆子痫有关,在可能发生先兆子痫女性的前三个月胎盘中,ERAP2表达常发生改变[22],其次,关于人类鸟枪弹样脉络膜视网膜病变、乳腺癌和妊娠高血压疾病等方面的研究居多[23-25]。本研究初次探索了该基因与新疆褐牛产奶性状的相关性,结果表明SNP1和SNP2均与新疆褐牛305d产奶量达到极显著相关,对于SNP1,TT基因型个体的305d产奶量显著高于CC型个体;对于SNP2,GG基因型个体305d产奶量显著高于AA基因型个体。SNP3位于Cwc27上,Cwc27位于牛20号染色体上,由24个外显子组成。Cwc27蛋白家族与RNA剪接体的功能相关,其表达模式和生物学功能研究至今未见实验性报道。本文初次研究了Cwc27 T14533269A位点与新疆褐牛泌乳性状的关联性,结果表明该位点与乳脂率、总固体和SCS性状达到显著或极显著关联,对于乳脂率和总固体性状,TT基因型个体显著高于AA基因型个体。牛奶中体细胞数与奶牛乳房炎存在显著的相关性,由于SCC分布是非正态的,通常将SCC转化为体细胞评分来研究[26]。奶牛乳房炎是一种常见的奶牛疾病,对世界奶业造成的经济损失极大,目前,已有许多****开展了奶牛分子抗病育种研究工作,并取得了一定的成果。本研究得出T14533269A位点与新疆褐牛SCS性状达到极显著关联,初次探讨了Cwc27与牛体细胞评分的关联性。
SNP4和SNP10均位于ZNF804B上,结果表明两位点与新疆褐牛产奶性状无显著性关联性。ZNF804为锌指蛋白基因,本研究之前还未有该基因与中国荷斯坦牛产奶性状的相关报道,2014年,SANTANA和OLIVEIRA等研究结果表明该基因与肉牛干物质采食量有关[27-28]。JIANG等全基因组关联分析结果表明锌指蛋白ZNF623中的一个位点(rs41627764)对北京地区中国荷斯坦牛乳脂率性状有显著影响[1]。
SNP5和SNP6位于AMHR2上,AMHR2为抗苗勒氏管激素II型受体,也叫苗勒管抑制物,属于转化生长因子βII型受体(TβRII)之一。最初分别由DI CLEMETE和BAARENDS等所在的两个实验室用不同的方法于1994年克隆获得[29-30]。关于AMHR2基因大多是关于女性多囊性卵巢综合征的研究[31-32],而在牛上的研究相对较少,仅有的几篇是关于牛卵巢颗粒细胞发育研究[33-35]。CARTER等在瘤牛上研究了AMHR2 RNA表达与卵母细胞数量相关,且该基因与牛繁殖性能相关 [36-37]。然而,目前该基因与牛产奶性状的关联性研究还未见报道。本研究得出SNP6与新疆褐牛乳蛋白率和总固体性状达到极显著关联,而SNP5与新疆褐牛各产奶性状未达到显著关联。
SNP9与新疆褐牛乳糖率性状达到显著关联。该位点位于牛15号染色体PPFIBP2上,ABAL等[38]研究表明,该基因可作为检测人类子宫内膜癌的分子标记,COLAS等[39]研究结果表明,该基因在人类子宫内膜肿瘤中的差异表达与其在相应子宫液体样品中的表达水平相关。但目前为止,关于该基因与奶牛产奶性状的相关研究还未见报道。研究结果显示,对于遗传力低、只能在一个性别中表达、不能早期测量或测量难度大、成本高的性状,标记辅助选择(MAS)相对于常规选择法有较大优势。奶牛的产奶性状(产奶量、乳脂率、乳蛋白率)具有中等偏下的遗传力,只在母牛表现且表现时间是在一胎产奶结束之后,因此,奶牛育种中实施MAS大有可为[40]。
3.3 单倍型与连锁不平衡分析
有研究表明,与单标记SNP相比,单倍型分析对复杂性状的遗传变异具有更好的效果[41]。随着高通量SNPs检测的出现,发现的SNP量越来越多,需要进行连锁不平衡分析来估计有效群体的大小,长距离连锁不平衡可以用来估计近世代的有效群体大小,短 距离连锁不平衡可以用来估计远世代的有效群体大小[42-43]。本研究群体中,检测的10个SNPs共构成2个单倍型模块,且均处于强连锁不平衡状态。由图2可以看出,单倍型模块与模块之间连锁程度较低,这可能是由于各个位点间物理位置相距较远所致。有研究表明,随着物理距离的增加,LD呈递减趋势,但某些物理位置较远的SNP之间也可能存在强LD,牛基因组不仅存在近距离的连锁不平衡,而且远距离的标记也以一定的模式存在连锁不平衡[44]。3.4 单倍型与泌乳性状及SCS关联分析
本研究未发现单倍型与新疆褐牛SCS和泌乳性状的关联性,可能是由于样本量较少。何峰等研究结果表明单个位点乳脂量的效应不显著,而单倍型组合对乳脂量的效应却达到了显著水平,由此可见,用多个SNPs构建单倍型,进而分析单倍型对产奶性状的效应,为以标记辅助选择为基础的奶牛分子育种带来更多的遗传学信息[45]。4 结论
本研究基于DNA混池重测序结果获得的SNPs数据,筛选6个基因的10个SNP位点在169头新疆褐牛群体中验证其遗传多态性,并分析其对新疆褐牛体细胞评分和泌乳性状的影响。初步发现了Cwc27与新疆褐牛体细胞评分性状有关,ERAP2、PPFIBP2和AMHR2与新疆褐牛305 d产奶量、乳脂率、乳蛋白率、乳糖率和总固体性状有关,其中A145332691T和C26758055G位点与新疆褐牛多个泌乳性状达到显著关联,提示,这些SNPs可为新疆褐牛产奶性状的分子标记辅助选育提供参考依据,但各位点的遗传效应差异及突变对乳汁合成等生理生化反应作用方面值得进一步深入研究。The authors have declared that no competing interests exist.
