0 引言
【研究意义】乳房炎是奶牛最常见的疾病之一,也是造成奶牛业损失最严重的疾病[1-2]。奶牛患乳房炎后导致奶品质降低、产奶量下降,甚至危害到消费者的健康。奶牛生产寿命是指奶牛从第1次产犊到死亡或者淘汰之间的时间,它能够反映奶牛避免被淘汰的能力。奶牛的生产寿命是一个重要的经济指标,它在增加牧场主的收入和育种方面有重要的作用。有研究表明:荷斯坦牛的生产寿命与终生产奶量呈显著正相关[3],奶牛的产奶胎次从第3胎提高到第4胎,其效益将增加11%—13%[4]。奶牛生产寿命与生产性能、繁殖性能及健康状况等紧密相关,其中临床乳房炎是造成奶牛淘汰的主要原因之一。临床乳房炎的遗传力为0.03—0.2[5],奶牛生产寿命的遗传力为0.029—0.072[6]。由于临床乳房炎和生产寿命的遗传力均较低,直接选择很难获得理想的遗传进展。因此,结合分子标记辅助选择技术对乳房炎和生产寿命的研究越来越受到关注。【前人研究进展】 趋化因子受体1(chemokine receptor 1, CXCR1)属于G蛋白偶联受体的家族成员,目前至少有两种不同的类型(CXCR1和CXCR2)[7]。I型受体(CXCR1)特异性地与白细胞介素-8(Interleukin-8,IL8)结合,主要分布于中性粒细胞、单核细胞和G细胞;II型受体(CXCR2)除了与IL8结合外,还可以和生长调节癌基因(Growth-regulated oncogene, GRO)、巨噬细胞炎性蛋白22(Macrophage inflammatory protein-2,MIP-2)和中性粒细胞激活蛋白-2(Neutrophil activating protein-2,NAP-2)等结合,CXCR2主要分布于中性粒细胞和髓样前体细胞系。CXCR1和CXCR2均具有介导活化中性粒细胞的作用,可促进中性粒细胞脱颗粒、释放贮存酶,增强中性粒细胞的吞噬功能,启动超氧离子释放,导致机体局部炎症反应,达到杀灭病原菌的目的[8-9]。大量研究表明:IL8和CXCR1基因及其突变与奶牛乳房均存在一定的关系[10-16]。周雷等[17]通过对中国荷斯坦奶牛CXCR1基因外显子2进行多态性分析发现4个突变位点291 C>T、333 C>T、337 A>G 和365 C>T,并且发现CCCTGGCC基因型组合个体具有低SCS和高产奶量的优势。奶牛的生产寿命与牛场的生产性能、饲养管理、日粮配合、环境、季节、其它疾病等非遗传因素有一定关系[18],同时与遗传也有一定关系[19-20]。近年来,国内外围绕影响奶牛生产寿命的分子标记进行了部分研究,发现了部分有价值的结果。ASHWELL等[21-22]分别在奶牛第2、12、16、21、23号染色体上发现了与其生产寿命相关的微卫星;HEYEN等[23]在BTA21上发现了长度为85 cM的微卫星ILSTS054;KHATIB [19-20]、KOMISAREK等[24]、JOHN等[25]又发现了ATP结合盒子亚家族G成员(ATP-binding cassette sub-family G member 2,ABCG2)、过氧化物酶体增殖物增殖受体γ辅激活因子(Peroxisome proliferators-activeted receptor-γcoactovator 1 A,PPARGC1A)、氧化低密度脂蛋白受体1(Oxidized low-density lipoprotein receptor 1,OLR1)和硬脂酰辅酶A去饱和酶1(stearoyl-CoA desaturase 1, SCD1)与生产寿命有一定程度的相关性。CHEBEL 等[26]发现Leptin 基因R4C位点与奶牛生产寿命相关疾病的发生显著相关,TT基因型个体真胃变位的发生率较高,而CT型各种疾病的发病率最低。【本研究切入点】国内周雷等[17]、徐敏等[27]和官永强等[28]检测了中国荷斯坦牛CXCR1基因部分编码区(coding sequence,CDS)序列SNP多态性,并与泌乳性状进行了关联分析,但尚未见对CXCR1基因整个CDS区SNP检测的报告,也未见CXCR1基因CDS区SNP多态与临床乳房炎和生产寿命相关分析的报道。【拟解决的关键问题】本研究的目的是在检测中国荷斯坦牛CXCR1基因CDS区所有SNP突变基础上,同时分析SNP多态与临床乳房炎和生存寿命的相关性,以期为中国荷斯坦牛临床乳房炎及生产寿命的分子标记辅助选择提供参考。1 材料与方法
1.1 试验材料
2011年7—8月于江苏省某大型奶牛场随机采集中国荷斯坦牛血样共866头。采用尾静脉采血(10 mL/头),ACD抗凝,-20℃冷冻保存备用。牧场基本情况如下:全场共有奶牛10 000余头,其中成年泌乳奶牛近6 000头,散栏饲养,鱼骨式挤奶大厅进行挤奶,日挤奶三次,采用全混合日粮(Total Mixed Ration, TMR)饲喂。1.2 DNA提取及CXCR1基因CDS区SNP突变检测
奶牛血液基因组DNA采用常规酚氯仿提取法,TE溶解,取部分DNA样品稀释至100 ng·µL-1,-20℃保存备用。根据GenBank公布的牛CXCR1基因CDS区序列(登录号:NM_001105038.1),用Primer 5.0软件设计引物1对如下:F:ATGACAATCATCCTGAA AGA,R:TCAGAGGGTAGTAGACGTGT。扩增产物长度为1 082 bp,PCR扩增体系如下:总体积20 μL,其中包括10×buffer 2.0 μL,25 mmol·L-1 Mg2+ 1.5 μL、dNTP(10 mmol·L-1)0.5 μL、Taq DNA聚合酶(5 U·µL-1)0.3 μL,上下游引物各(10 pmol·µL-1)1.0 μL, 模板DNA(100 ng·µL-1)1.0 μL、ddH2O 12.7 μL。扩增程序为:94℃预变性5 min;94℃变性40 s,56℃复性40 s,72℃延伸10 min,30个循环,最后72℃延伸10 min,4℃保存。PCR扩增产物经1%琼脂糖凝胶(0.05% EB)电泳0.5 h后,紫外灯下观察结果,并拍照。
从所采样本中选择20个高SCS样本(5.85±1.22)和20个低SCS样本(1.02±0.05),进行常规PCR扩增,经琼脂糖凝胶电泳检测其扩增效果,后送至上海生物公司正反测序。用DNAMAN软件对测序所得结果进行比对,寻找突变位点,并初步计算各基因各位点连锁程度。
经对40个样本的初步分析,CXCR1基因CDS区共发现13个SNP位点,分为4个连锁群,故从4个连锁群中各选择1个SNP位点进行后续分析,分别是:642 A>G、816 A>C、980 A>G和1068 A>G 4个SNP位点。对大样本CXCR1 SNP突变检测采用飞行时间质谱法(MassARRAY® MALDI-TOF System,Sequenom,Inc.,USA)。同时,为保证SNP分析结果可靠性,其中40个样本重复测定2次(测试员并不知道这个40个是重复样本)。结果表明该方法SNP分型结果准确性为100%。
1.3 临床乳房炎发生次数和生产寿命相关数据的收集
奶牛临床乳房炎以可以看到明显的乳房红肿、触摸有疼痛感和硬块或乳样呈豆腐样等症状为判断条件,以兽医治疗记录为准。如果某奶牛在治愈后10d内又发病,则算一次病例;治愈后10d以上又发病的,则算为新发病例。根据该原则记录每头奶牛从2010年1月到2014年12月共5年内各胎次发生临床乳房炎的次数。根据采样牛只的出生时间、第一胎产犊时间、淘汰时间和胎次等信息,计算出初产月龄、生产月龄(或在群月龄)、离群月龄、离群胎次等信息,作为生产寿命相关指标,进行统计分析。
1.4 统计分析
1.4.1 遗传学分析 用遗传学软件SHEsis进行常规群体遗传学统计分析(包括基因频率、基因型频率、Hardy-Weinberg平衡检测等),同时进行连锁不平衡分析[29]。1.4.2 关联分析 用多因素方差分析模型分析CXCR1基因CDS区各SNP位点不同基因型与奶牛各胎次临床乳房炎次数和生产寿命相关指标的相关性,模型如下:
Y=μ+S+M+G+e
式中:Y为各胎次临床乳房炎次数和生产寿命相关指标的观察值,μ为群体均值,S 为初产季节的效应值,M 为初产月龄的效应值,G的基因型或单倍型的效应值,e为随机误差。根据当地气候特点,初产季节划分如下:3—5月为春季,6—8月为夏季,9—11月为秋季,12月至次年2月为冬季。
由于部分牛只记录不完整,只选择同时具有1—3胎临床乳房炎记录的牛只进入方差分析。经筛选,最终有721头奶牛用于分析。对于生产寿命相关指标,只选择已淘汰,且同时具有出生、产犊等相关信息的牛只进行分析。经筛选,最终有647头奶牛用于分析。
1.4.3 生产寿命的生存分析 用Cox回归模型计算CXCR1基因CDS区各SNP位点不同基因型对生产寿命相关指标的影响,并制作生存曲线,由统计软件SPSS(Ver 16.0)完成,模型如下:
h(t/X)=h0(t) exp (β1 X1 + β2 X2 +β3 X3 + β4X4 )
式中:
h0(t):基准风险函数,即所有变量取零时的t时刻的风险函数
X1、X2 、X2和 X4:各SNP位点变量
β1、β2、β3和β4:各SNP位点变量的回归系数
2 结果
2.1 CXCR1基因CDS区SNP筛查
通过对20头高SCS和低SCS奶牛用所设计的引物PCR扩增并测序,发现CXCR1基因CDS区共发现13个SNP位点,详细见表1。经连锁分析,13个SNP位点分为4个连锁群(图1),故从4个连锁群中各选择1个SNP位点进行后续分析,分别是:642 A>G、816 A>C、980 A>G和1068 A>G 4个SNP位点。另外,由于CXCR1基因SNP的变化均发生在CDS区,因此SNP的变化可能导致所编码氨基酸的变化。根据CXCR1基因CDS区SNP导致氨基酸变化情况,制作各位点氨基酸变化情况如图2 所示。CXCR1基因CDS区共有5个非同义突变,I113V、K327R和H332R 3个非同义突变是完全连锁的,属于如前所述第2个连锁群,V122A和H245Q属于第1连锁群。第3、4两个连锁群均是同义突变,不导致氨基酸的变化。2个非同义突变连锁群可以组成4种氨基酸单倍型。
Table 1
表1
表1CXCR1基因CDS区SNPs
Table 1The SNPs in the CDS of CXCR1 gene
| CDS区位置 CXCR1 SNP position | 碱基变化 Nucleotide change | 氨基酸变化 AA change | 连锁群 Linkage group | 备注 Memo. |
|---|---|---|---|---|
| 291 | C>T | 3 | ||
| 333 | C>G | 2 | 新突变 new mutation | |
| 337 | A>G | I113V | 2 | 新突变 new mutation |
| 365 | T>C | V122A | 1 | |
| 570 | G>A | 1 | ||
| 642 | A>G | 4 | 新突变 new mutation | |
| 735 | C>G | H245Q | 1 | |
| 816 | C>A | 3 | ||
| 819 | G>A | 1 | ||
| 980 | A>G | K327R | 2 | |
| 995 | A>G | H332R | 2 | |
| 1008 | C>T | 3 | ||
| 1068 | A>G | 1 |
新窗口打开
2.2 基因(型)频率分布及连锁不平衡分析
利用飞行质谱法对866头奶牛群体CXCR1基因CDS区4个SNP位点进行分型,所得基因频率、基因型频率分布及Hardy-Weinberg平衡检验结果见表2。CXCR1-642、CXCR1-816、CXCR1-980和CXCR1-1068 4个SNP位点优势基因型分别为GG、CC、GG和GG型,频率分别为0.580、0.466、0.607和0.658,优势基因分别为G、C、G和G,频率分别为0.758、0.688、0.784和0.811。经分析,CXCR1基因4个SNP位点均处于Hardy-Weinberg平衡状态。另外,CXCR1基因4个SNP位点共有9种单倍型(表3),其中单倍型GAGG频率最高(0.3141),而单倍型ACAA频率最低(0.0017)。2.3 CXCR1基因不同SNP位点临床乳房炎发病次数
CXCR1基因不同SNP位点各胎次临床乳房炎发生次数见表4。由表4可知:随着胎次增加,奶牛患临床乳房炎发生次数增加较快:1、2、3胎及1—3胎共计发生临床乳房炎的平均次数分别为0.01±0.01、0.52±0.03、0.91±0.05和1.44±0.07。CXCR1-642与2胎牛、CXCR1-816与3胎牛临床乳房炎发生次数有显著相关(P<0.05),其它SNP位点与各胎次临床乳房炎发生次数均无显著相关(P>0.05)。多重比较表明:2胎牛CXCR1-642 AG基因型个体临床乳房炎发生次数(0.61±0.06)显著高于AA型(0.33±0.09),3胎牛CXCR1-816 AA基因型个体临床乳房炎发生次数(0.55±0.10)显著低于AC和CC型个体(0.99± 0.08和0.90±0.07)。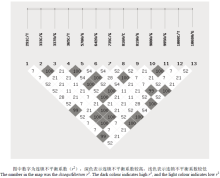 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图140个个体CXCR1基因CDS区SNP位点连锁图
-->Fig. 1The linkage map of SNP in the CDS of CXCR1 gene for 40 Holstein cows
-->
Table 2
表2
表2CXCR1基因 CDS区4个SNP位点等位基因、基因型频率及H-W平衡检验
Table 2The genotypic frequencies, alleles, and values of X2 test significance for 4 SNP in the CDS of CXCR1 gene
| SNPs或单倍型 SNPs or haplotypes | 基因型 Genotype | 基因型频率 Genotypic frequency | 个体数 No. | 等位基因 Alleles | 基因频率 Allelic frequency | 平衡检验X2值 X2 value for H-W test |
|---|---|---|---|---|---|---|
| CXCR1-642 | AA | 0.064 | 55 | A | 0.242 | 0.698 |
| AG | 0.356 | 308 | G | 0.758 | ||
| GG | 0.580 | 502 | ||||
| CXCR1-816 | AA | 0.089 | 77 | A | 0.312 | 1.219 |
| AC | 0.445 | 385 | C | 0.688 | ||
| CC | 0.466 | 403 | ||||
| CXCR1-980 | AA | 0.038 | 33 | A | 0.216 | 2.023 |
| AG | 0.355 | 305 | G | 0.784 | ||
| GG | 0.607 | 521 | ||||
| CXCR1-1068 | AA | 0.036 | 31 | A | 0.189 | 0.001 |
| AG | 0.306 | 265 | G | 0.811 | ||
| GG | 0.658 | 569 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2CXCR1基因CDS区AA变化图
-->Fig. 2The change of AA in the CDS of CXCR1 gene
-->
2.4 CXCR1基因不同SNP位点的生产寿命
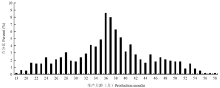
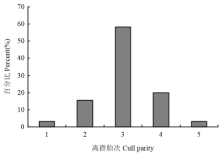
在本研究中,结合奶牛出生、初产季节、初产月龄、淘汰日期等信息,用生产月龄、离群月龄和离群胎次3个指标来度量奶牛的生产寿命。生产月龄从13到58个月不等(图3),其中36个月最多,其次为37个月,平均为36.86±0.33月。离群月龄从26—85不等(图4),其中64最多,其次为65,平均为64.19±8.30。离群胎次从1—5胎不等(图5),其中3胎最多,其次为4胎,平均为3.04±0.78。Table 3
表3
表3CXCR1基因4个SNP位点单倍型频率
Table 3The haplotypes frequencies of 4 SNP, in the CDS of CXCR1 gene
| 单倍型 Haplotypes | CXCR1-642 | CXCR1-816 | CXCR1-980 | CXCR1-1068 | 数量 N | 频率 Frequencies |
|---|---|---|---|---|---|---|
| 1 | G | A | G | G | 544 | 0.3141 |
| 2 | G | C | G | G | 318 | 0.1835 |
| 3 | G | C | A | G | 307 | 0.1773 |
| 4 | A | C | G | A | 188 | 0.1085 |
| 5 | A | C | G | G | 180 | 0.1041 |
| 6 | G | C | G | A | 126 | 0.0728 |
| 7 | A | C | A | G | 41 | 0.0235 |
| 8 | G | A | A | G | 25 | 0.0145 |
| 9 | A | C | A | A | 3 | 0.0017 |
新窗口打开
Table 4
表4
表4CXCR1基因不同SNP位点各胎次临床乳房炎发生次数
Table 4The number of CM for SNPs of CXCR1 of different parities
| SNPs | 基因型 Genotypes | 样本量 N | 1胎 Pairty-1 | 2胎 Pairty-2 | 3胎 Pairty-3 | 1—3胎合计 Parity 1 to 3 |
|---|---|---|---|---|---|---|
| CXCR1-642 | A A | 46 | 0.02±0.02 | 0.33±0.09b | 0.89±0.20 | 1.24±0.24 |
| A G | 251 | 0.02±0.01 | 0.61±0.06a | 0.94±0.09 | 1.57±0.13 | |
| G G | 424 | 0.00±0.00 | 0.48±0.04ab | 0.89±0.06 | 1.38±0.08 | |
| F | 1.203 | 2.991 | 0.089 | 1.014 | ||
| P | 0.301 | 0.050 | 0.915 | 0.363 | ||
| CXCR1-816 | A A | 71 | 0.00±0.00 | 0.45±0.09 | 0.55±0.10b | 1.00±0.15 |
| A C | 332 | 0.01±0.01 | 0.55±0.05 | 0.99±0.08a | 1.55±0.10 | |
| C C | 318 | 0.02±0.01 | 0.50±0.05 | 0.90±0.07a | 1.42±0.09 | |
| F | 0.361 | 0.473 | 3.508 | 1.879 | ||
| P | 0.697 | 0.623 | 0.03 | 0.154 | ||
| CXCR1-980 | AA | 27 | 0.00±0.00 | 0.33±0.11 | 0.96±0.26 | 1.30±0.26 |
| AG | 256 | 0.00±0.00 | 0.46±0.05 | 0.87±0.07 | 1.34±0.10 | |
| GG | 433 | 0.02±0.01 | 0.57±0.05 | 0.92±0.07 | 1.50±0.09 | |
| F | 1.071 | 1.869 | 0.115 | 0.794 | ||
| P | 0.343 | 0.155 | 0.891 | 0.452 | ||
| CXCR1-1068 | A A | 25 | 0.00±0.00 | 0.32±0.14 | 0.92±0.28 | 1.24±0.31 |
| A G | 214 | 0.03±0.02 | 0.62±0.07 | 0.90±0.09 | 1.55±0.13 | |
| G G | 482 | 0.00±0.00 | 0.48±0.04 | 0.91±0.06 | 1.40±0.08 | |
| F | 2.659 | 2.387 | 0.005 | 0.430 | ||
| P | 0.071 | 0.093 | 0.995 | 0.651 | ||
| 合计Total | 721 | 0.01±0.01 | 0.52±0.03 | 0.91±0.05 | 1.44±0.07 |
新窗口打开
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3奶牛生产月龄分布
-->Fig. 3The distribution of production months of cows
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4离群月龄分布
-->Fig. 4The distribution of culling months of cows
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5离群胎次分布
-->Fig. 5The distribution of culling parities of cows
-->
CXCR1基因CDS区不同SNP位点奶牛生产月龄、离群月龄和离群胎次见表5。由表5可知:CXCR1-816基因型与奶牛离群月龄有极显著相关(P<0.01),CXCR1-816基因型与奶牛生产月龄和离群胎次有显著相关(P<0.05),而其它SNP位点与生产月龄、离群月龄和离群胎次均无显著相关(P>0.05)。多重比较表明:CXCR1-816 AA基因型个体生产月龄、离群月龄和离群胎次均显著高于CC基因型个体(P<0.05)。
2.5 生存分析
CXCR1基因CDS区4个SNP位点的奶牛生存分析结果表明:仅有CXCR1-816位点与奶牛生存时间有显著相关(P<0.05,表6)。进一步分析发现:CXCR1-816 CC基因型个体生存时间显著低于AA和AC基因型(表7)。CXCR1-816 不同基因型生存曲线见图6。由图6可知:CXCR1-816 CC基因型个体在各时间段的生存概率均低于AA和AC基因型个体。Table 5
表5
表5CXCR1基因不同SNP位点奶牛生产月龄、离群月龄和离群胎次
Table 5The productive months, culling months and culling parity for SNPs of CXCR1 gene
| SNPs | 基因型 Genotypes | 样本量 N | 生产月龄(月) Productive months | 离群月龄(月) Culling months | 离群胎次 Culling parity |
|---|---|---|---|---|---|
| CXCR1-642 | A A | 36 | 37.56±1.48 | 65.50±8.13 | 3.06±0.92 |
| A G | 231 | 37.56±1.48 | 63.34±9.11 | 3.00±0.76 | |
| G G | 380 | 37.11±0.41 | 64.58±7.76 | 3.07±0.78 | |
| F | 1.295 | 2.241 | 0.438 | ||
| P | 0.275 | 0.107 | 0.645 | ||
| CXCR1-816 | A A | 62 | 38.35±1.17a | 66.19±8.50a | 3.34±0.77a |
| A C | 277 | 37.59±0.47ab | 65.04±7.76ab | 3.09±0.77b | |
| C C | 308 | 35.90±0.49b | 63.03±8.58b | 2.94±0.78b | |
| F | 3.591 | 5.066 | 4.060 | ||
| P | 0.028 | 0.007 | 0.018 | ||
| CXCR1-980 | A A | 25 | 37.96±1.33 | 64.08±7.09 | 3.08±0.70 |
| A G | 224 | 36.60±0.52 | 64.17±7.41 | 3.00±0.72 | |
| G G | 394 | 37.01±0.44 | 64.27±8.86 | 3.07±0.82 | |
| F | 0.358 | 1.166 | 0.926 | ||
| P | 0.699 | 0.322 | 0.428 | ||
| CXCR1-1068 | A A | 24 | 36.77±1.88 | 62.71±10.59 | 2.92±0.78 |
| A G | 195 | 36.35±0.63 | 63.72±9.03 | 2.98±0.80 | |
| G G | 428 | 37.10±0.39 | 64.49±7.80 | 3.07±0.77 | |
| F | 0.297 | 0.612 | 0.176 | ||
| P | 0.743 | 0.543 | 0.838 | ||
| 合计Total | 647 | 36.86±0.32 | 64.19±8.30 | 3.04±0.78 |
新窗口打开
Table 6
表6
表6CXCR1基因CDS区4个SNP位点奶牛的Cox生存分析
Table 6The Cox survival analysis of 4 SNPs of CXCR1 gene for dairy cows
| SNPs | B值 B value | 标准误 SE | Wald X2 值 Wald X2 value | 显著水平 Sig. | 相对危险度 RR | 95%CI上限 Lower of 95% CI for RR | 95%CI下限 Upper of 95% CI for RR |
|---|---|---|---|---|---|---|---|
| CXCR1-642 | 0.046 | 0.074 | 0.389 | 0.533 | 1.047 | 0.905 | 1.212 |
| CXCR1-816 | 0.234 | 0.081 | 8.387 | 0.004 | 1.264 | 1.079 | 1.480 |
| CXCR1-980 | 0.018 | 0.085 | 0.046 | 0.830 | 1.018 | 0.863 | 1.202 |
| CXCR1-1068 | 0.099 | 0.100 | 0.992 | 0.319 | 1.105 | 0.908 | 1.343 |
新窗口打开
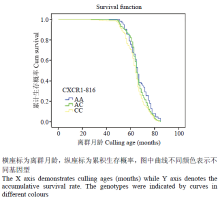 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6奶牛CXCR1-816不同基因型生存曲线
-->Fig. 6The survival curve of cows for different genotypes of CXCR1-816
-->
Table 7
表7
表7CXCR1-816不同基因型生存时间平均数与中数的估计
Table 7The estimation value of mean and median of survival time for different genotypes of CXCR1-816
| CXCR1-816 | 样本量 N | 平均数±标准误 Mean±SE | 中位数±标准误 Median±SE |
|---|---|---|---|
| AA | 62 | 66.19a±1.08 | 65±0.84 |
| AC | 277 | 65.04a±0.47 | 64±0.34 |
| CC | 308 | 63.03b±0.49 | 63±0.42 |
| 合计Overall | 647 | 64.19±0.33 | 64±0.27 |
新窗口打开
3 讨论
3.1 CXCR1基因遗传多态性分析
趋化因子受体和趋化因子在机体炎症发生时起着非常重要的作用,当奶牛发生乳房炎时CXCR1基因表达产物增多[30]。本试验在中国荷斯坦牛第2号染色体CXCR1基因CDS区共发现了13个SNP突变位点。PIGHETTI等[31]报道,CXCR1基因CDS区共有11个SNP位点,也包括4个连锁群。本研究与之相比,333C>G、337A>G和642A>G均为新发现的突变位点,并未发现621A>G位点,也分为4个连锁群,群内完全连锁。由于621A>G 为终止密码子突变,会导致蛋白翻译提前终止,说明本研究所检测的中国荷斯坦牛群体中不存在该位点终止密码子突变。333C>G和337A>G属于连锁群2,642 A>G 为独立的连锁群,与其它位点不连锁。徐敏等[27]分析了荷斯坦牛、西门塔尔牛、通江黄牛3个牛品种共160头个体CXCR1基因编码区多态性,在CDS区643、735、819位发现3个SNP位点。官久强等[28,32]分析了470头中国荷斯坦牛CXCR1基因编码区的多态性,结果发现了6个SNPs:735 C>G 、816 C>A、819 A>G、980 A>G、995 A>G和1008 C>T。本研究与上述研究相比较,多发现了4个SNP位点。3.2 CXCR1基因与临床乳房炎发病次数和生产寿命的关系
本研究所发现的CXCR基因CDS区4个SNP连锁群(642 A>G、816 A>C、980A>G和1068 C>T)内,CXCR1-642位点与2胎奶牛发生乳房炎次数有显著相关(P<0.05)。由于CXCR1-642位点为首次发现的位点,并且CXCR1-642位点与其他胎次临床乳房炎发生次数无显著相关(P>0.05),所以该结果有待进一步探索。CXCR1-980和CXCR1-1068位点与临床乳房炎发病次数无显著相关(P>0.05),同时这两个位点与奶牛生存时间也无显著相关。CXCR1-816位点与3胎临床乳房炎发病次数有显著相关(P<0.05),AA基因型个体临床乳房炎发病次数显著低于AC和CC基因型个体。多因素方差分析和生存分析也表明:CXCR1-816位点与奶牛生产寿命有显著相关,CC型个体生存时间平均为63.68月,极显著低于AA型个体(65.5月),CC型比AA型奶牛提前淘汰2个月左右。可见临床乳房炎发病次数较少的基因型个体存活时间较长。综合以上3方面统计数据所得结果,发现CXCR1-816位点与3胎临床乳房炎发病次数及奶牛生产月龄、离群月龄和离群胎次均有显著相关,推测该位点及其连锁群的突变可能对其基因表达产生影响,从而影响乳房炎发病次数、生产寿命等相关指标,但这一推论有待进一步通过实验进行验证。因此,在种公牛站进行遗传评定工作中,增加CXCR1-816基因型检测,筛选其中AA型个体公牛并留种,逐渐淘汰后代中CC型母牛,可降低奶牛群体中临床乳房炎发病次率,延长生产寿命,从而提高奶牛养殖的经济效益。在本研究中,816 A>C、291 C>T、1008 C>T及3′端792 G>A和1079 C>T处于第三连锁群内[31],虽然CXCR1基因CDS区上述3个位点均为同义突变,没有造成AA改变。而3′端792 G>A和1079C>T 2个突变位点,经RNAhybrid软件进行预测,均没有发现miRNA结合位点。由于本研究并未对CXCR1基因5′端启动子区SNP突变及其与CDS区SNP进行连锁分析,在该基因5′端启动子区是否存在与CDS区816 A>C完全连锁的突变并不得而知,也未见相关报道。因此,816 A>C与奶牛临床乳房炎发病次数及生产寿命显著关联的遗传机制有待进一步深入研究。
另外,本研究也发现同一基因型在不同胎次间表现不一致,如CXCR1-816位点仅与3胎临床乳房炎发病次数有显著相关,而与1胎和2胎奶牛临床乳房炎发病次数无显著相关。造成该结果可能有以下几个原因:一是由于奶牛年青时抗病力较强,临床乳房炎等疾病性状发生率较低,从而造成统计结果不显著;二是临床乳房炎本身的度量问题。在生产中临床乳房炎常以可以看到明显的乳房红肿、触摸有疼痛感和硬块或乳样呈豆腐样等症状为判断条件,并伴随着乳中体细胞数上升和血液中某些免疫因子发生变化。经转换后的体细胞数(也称为体细胞评分)与临床乳房炎有较高的遗传相关[5],且在一个泌乳期中有多次记录,增加了结果判断的准确性。第三个原因是引起乳房炎的病原菌类型较为复杂。不同病原菌导致临床乳房炎的表现可能不一致,其发病机制也有所不同,这无疑增加基因型与病原菌间互作的复杂性。
4 结论
CXCR1-816 A>C突变与中国荷斯坦牛患临床乳房炎次数和生产寿命有显著相关, CXCR1-816 CC型基因型个体有较多的临床乳房炎发病次数和较短的生产寿命,因此被提前淘汰。在进一步验证其功能情况下,该位点可用于中国荷斯坦牛生产寿命的分子标记辅助选择。The authors have declared that no competing interests exist.
