0 引言
【研究意义】榛子为桦木科(Betulaceae)榛属(Corylus)植物,是世界四大坚果之一。全世界榛属植物约有25种[1],主要分布于亚洲、欧洲及北美洲,其中商业化栽培的只有欧洲榛(C. avellana L.)的一些品种[2];中国原产的榛属植物分为8个种和2个变种,广泛分布于中国的20余个省区,栽培种为通过平榛(C. heterophylla Fisch.)和欧洲榛种间杂交获得的平欧杂种榛(C. heterophylla Fisch. × C. avellana L.)的若干品种[3]。上述榛属植物资源丰富,在树体特征、坚果性状、抗逆性,以及自交不亲和特性等方面存在丰富的变异类型,大量特异性状基因亟待挖掘和利用。目前,针对东部榛疫病(Eastern Filbert Blight)的欧洲榛抗性育种[4-5],针对抗寒性的中国平榛与欧洲榛的种间杂交[6-7],以及针对榛属植物孢子体自交不亲和特性开展的S位点基因研究[8-10],是世界榛子育种领域研究的热点问题。随着研究的深入,分子生物学技术将成为榛属植物资源利用和育种创新方面重要的研究手段,将在特异性状基因挖掘和分子辅助育种工作中发挥精准、高效的研究优势,具有重要的研究意义。【前人研究进展】实时荧光定量PCR(quantitative real-time PCR,RT-qPCR)是基因克隆工作的重要组成部分,指在反应体系中引入荧光染料或带荧光标记探针的一种改良型PCR,其具有耗时短、高灵敏度、重复性强、样品需要量少等优点[11-13],被广泛的用于基因表达与转录分析。RT-qPCR是以cDNA为模板,初始的RNA质量、产量、反转录效率和扩增效率等均会影响基因表达分析结果的准确性[14],目前,常用引入内参基因进行标准化处理的方式来减弱误差[15]。理想的内参基因是在各种条件下均能稳定表达[11],因此,在前期研究中一般选取Tubulin(微管蛋白基因),Actin(肌动蛋白基因),GAPDH(3-磷酸甘油醛脱氢酶基因),18S rRNA等管家基因(House-keeping Gene)作为内参基因。越来越多的研究证明[16-20]:管家基因在一般情况下可以稳定表达,但在一些特殊的生命活动中或试验处理条件下其表达出现了不稳定性。因此,筛选出适合的稳定的内参基因,成为了实时荧光定量PCR的关键性问题,常用的内参基因筛选方法有:geNorm[21]、Normfinder[22]、BestKeeper[19]和Delta Ct[23]。【本研究切入点】目前,关于平欧杂种榛的相关分子机理研究日益增多,国内外还没有关于系统性筛选适合平欧杂种榛实时荧光定量PCR内参基因的相关报道,而利用实时荧光定量PCR分析其相关基因的表达时,则是未经实验验证和筛选,直接引用其他物种基因作为内参基因[24]。随着实时荧光定量PCR技术成熟和成本逐渐降低,筛选获取适合平欧杂种榛自身的稳定内参基因变得尤为重要。使用稳定、合适的内参基因,才能够更加准确计算出基因的表达情况。【拟解决的关键问题】运用反转录PCR和实时荧光定量PCR技术,分析12个候选内参基因在平欧杂种榛不同组织器官中的稳定性,并筛选出稳定的内参基因,以期构建其荧光定量PCR内参基因的筛选体系,为后期平欧杂种榛相关的基因表达研究提供基础。1 材料与方法
试验于2015年1月至2016年8月在中国林业科学研究院林木遗传育种国家重点实验室进行。1.1 试验材料
本研究选用平欧杂种榛主栽品种‘达维’(育种代号84-254)作为材料,所有样品采自中国林业科学研究院玉泉山榛子实验基地。根据榛属植物的植物学特性并参阅其他相似研究,试验选用了8个不同的组织部位(表1)。2015年1月,随机采集‘达维’的一年生枝条200条,分成两份(其中一份去雄),置于0—2℃冷库中保湿存放。2015年3月,于25℃温室中隔离水培:去雄的一份,待雌花开放后,收集雌蕊;另一份待雄花序拉长后收集花粉,装在带有棉塞的玻璃瓶中,置于-80℃冰箱中备用。其余组织样品依据榛子的植物学特性,分别在田间取样。所用样品均经液氮速冻后,存放于-80℃冰箱备用。Table 1
表1
表1研究材料
Table 1Samples
| 材料说明 Description of samples | 简称 Abbreviation |
|---|---|
| 盛花期雌蕊 Blooming styles | St |
| 未伸长期雄花序 Catkins before elongation | Ck |
| 幼嫩叶片 Young leaves | Lv |
| 花粉 Pollen | Pn |
| 一年生枝形成层 Cambium of annual branch | Ca |
| 嫩茎 Green stem | Se |
| 根尖 Root tip | Ro |
| 根蘖 Sucker | Su |
新窗口打开
1.2 候选内参基因的选取和引物设计
利用笔者课题组前期对平欧杂种榛不同亲和性授粉、授粉后不同时间雌蕊的转录组测序数据(暂未公布),以FDR值(False Discovery Rate)小于0.01和表达量Log2比率值介于-1和1之间为筛选标准,选出表达稳定的序列作为候选内参基因;此外,通过Blast获得actin、ubiquitin、18S ribosomal RNA、alpha tubulin、beta-tubulin、elongation factor 1-alpha和GAPDH这7种常用传统内参基因的同源序列,通过上述筛选方法获取候选内参基因;根据榛属相关文献[24-26],收集其所使用的内参基因及其引物。利用在线引物设计程序Primer 3.0(http://bioinfo. ut.ee/primer3-0.4.0/)设计RT-qPCR引物,参数设置:引物Tm值为58—62℃,扩增片段长度范围为150—200 bp,引物长度范围为19—21 nt,GC含量范围为45%—55%,其他为默认设置参数;并运用NCBI Blast验证引物的特异性。
1.3 RNA提取和cDNA合成
采用改良后的CTAB法提取总RNA,取部分RNA溶液经过1%的琼脂糖凝胶电泳和微量分光光度计Nanodrop 8000进行检测,分析其完整性与浓度。每个样品取1 μg RNA使用BIO-ROD公司的iScript™ cDNA合成试剂盒进行20 μL体系的反应。反应程序:25℃,5 min→42℃,30 min→85℃,5 min于PCR仪上完成。所得cDNA溶液(20 μL)稀释5倍,保存于-20℃冰箱,用于后续试验。
1.4 反转录PCR
使用2×TSINGKE Master Mix(北京擎科新业生物技术有限公司),于Veriti 96 well Thermal cycler PCR仪进行反应。反应体系见表2;反应程序:94℃预变性3 min;94℃变性30 s,56℃退火30 s,72℃延伸30 s,循环35次;72℃彻底延伸7 min;保存于4℃。取5 μL PCR产物进行1.5%的琼脂糖凝胶电泳检查。1.5 荧光定量PCR
采用SYBR Green染料法,利用iTaqTM Universal SYBR® Green Supermix(BIO-RAD,USA)于C1000 Touch TM Thermal cycler PCR仪上进行反应。每个反应设置3次技术重复且每组引物设置一个阴性对照。反应体系如表3所示;PCR扩增反应程序(两步法扩增): 95℃预变性,3 min→(95℃变性,5 s→ 56℃退火+延伸,30 s)循环40次;溶解曲线反应程序:65℃升温至95℃,以0.5℃递增,恒温5 s。Table 2
表2
表2RT-PCR反应体系
Table 2The reaction system of RT-PCR
| 成分 Components | 体积 Volume (μL) |
|---|---|
| 2×TSINGKE Master Mix | 12.5 |
| 10 μmol·L-1 Primer F | 1 |
| 10 μmol·L-1 Primer R | 1 |
| cDNA | 1 |
| ddH2O | 9.5 |
新窗口打开
Table 3
表3
表3RT-qPCR反应体系
Table 3The reaction system of RT-qPCR
| 成分 Components | 体积Volume (μL) |
|---|---|
| iTaq Universal SYBR Green Supermix (2×) | 10 |
| 10 μmol·L-1Primer F | 0.5 |
| 10 μmol·L-1 Primer R | 0.5 |
| cDNA | 4 |
| ddH2O | 5 |
新窗口打开
1.6 引物评价
取等量不同组织部位样品的cDNA模板,混合后依次稀释5个梯度,每个梯度稀释5倍,其浓度分别表示为55、54、53、52、51、50倍,作为模板,并参照1.5中的方法进行荧光定量PCR试验。利用PCR仪自带的计算软件(CFX Manager 3.1),根据Cq(quantification cycle)值建立线性回归方程,绘制标准曲线,得到斜率k和线性相关系数R2,并求得扩增效率E(E=10-1/K-1);并收集溶解曲线。1.7 数据分析
采用Microsoft Excel 2010对原始Cq值进行统计学分析;使用qBase软件获得候选内参基因的Q值(Q=E Cq min-Cq sample;E代表扩增效率,Cq min代表基因在一组样品中最小的Cq值,Cq sample代表基因在一组样品中各样品的Cq值)。利用程序geNorm、Normfider、Bestkeeper和Delta Ct法分析候选内参基因在8个样品中的稳定性;运用线网站RefFinder(http://fulxie.0fees.us/?type=reference& ckattempt=1&i=1)进行综合评价。根据geNorm、NormFinder、BestKeeper和Delta Ct计算12个候选内参基因的稳定性值,利用SPSS软件获取其皮尔逊相关系数。
利用Microsoft Excel 2010和Origin 9.0软件处理数据,绘制图表。
2 结果
2.1 候选内参基因的确定及其引物特异性和扩增效率分析
在转录组数据中,以FDR值小于0.01和表达量Log2比率值介于-1和1之间为筛选标准,选出稳定表达的3个作为候选内参基因(表4)。7个传统内参基因的同源序列中,18S rRNA和TUB在转录组中同源序列的表达存在差异,说明不适合作为内参基因,最终选出5个作为候选内参基因(表4)。软件设计的8对特异性引物和文献收集4对特异引物(表5),合计12对引物。12对引物的扩增效率E在86.3%—121.6%,而R2值均大于0.98(0.982—0.997),说明其线性回归方程可信[27]。
Table 4
表4
表4候选内参基因
Table 4Description of candidate reference genes
| 序号 No. | 基因ID号 Gene ID | 说明 Description | 基因 Gene |
|---|---|---|---|
| 1 | Unigene27057_All | transcription elongation factor SPT5 | ChaSTP5 |
| 2 | Unigene8224_All | Homeodomain-containing transcription factor | ChaTF |
| 3 | Unigene376_All | ubiquitin C | ChaUBC |
| 4 | Unigene17976_All | Polyubiquitin 14 (拟南芥,Arabidopsis thaliana NM_001125450.1a) | ChaUBQ14 |
| 5 | CL4792.Contig2_All | α-tubulin (白桦,Betula platyphylla, FJ228477.1a) | ChaTUA |
| 6 | CL750.Contig12_All | Actin (白桦,Betula platyphylla, EU588981.1a) | ChaActin |
| 7 | CL828.Contig4_All | EF1-α (拟南芥,Arabidopsis thaliana, NM_001035916.2a) | ChaEF1-α |
| 8 | Unigene22682_All | GAPDH (毛果杨,Populus trichocarpa, FN396887.1a) | ChaGAPDH |
新窗口打开
Table 5
表5
表5候选内参基因引物
Table 5Primers of candidate reference genes
| 基因 Gene | 引物(5′-3′) Primer (5′-3′) | 长度 Length (bp) | 扩增效率 Efficiencies (E%) | R2 | 来源 Reference |
|---|---|---|---|---|---|
| ChaSTP5 | F: ACCATTTCCGGATGTTTGAG | 179 | 109.4 | 0.990 | 本研究 This study |
| R: GTCGCCCTTCTTACAGTTGC | |||||
| ChaTF | F: GTGCCTAGCCATCCTCATGT | 192 | 91.9 | 0.997 | 本研究 This study |
| R: ATCACCCTGACATCCTCGTC | |||||
| ChaUBC | F: CAGGCTCGCCAATCTTACTC | 155 | 107.1 | 0.986 | 本研究 This study |
| R: ACCCCCTTTTTCAGAAGCAT | |||||
| ChaUBQ14 | F: CCTTGCATCTGGTGTTGAGA | 197 | 92.8 | 0.995 | 本研究 This study |
| R: AGTACGCCCATCCTCCAAT | |||||
| ChaTUA | F: TCTCCACAGGTTTCCACCTC | 155 | 97.5 | 0.995 | 本研究 This study |
| R: GTGTAGGTGGGTCGCTCAAT | |||||
| ChaActin | F: GAGCTGAGAGATTCCGTTGC | 179 | 91.7 | 0.993 | 本研究 This study |
| R: AGCAATACCTGGGAACATGG | |||||
| ChaEF1-α | F: TTGCCTTTACCCTTGGTGTC | 175 | 86.3 | 0.995 | 本研究 This study |
| R: TCGAAACCAGAGATGGGAAC | |||||
| ChaGAPDH | F: AGCTCGTCGCTGTTAACGAT | 164 | 88.3 | 0.982 | 本研究 This study |
| R: GTTCCTGAAGCCGAAAACTG | |||||
| Ch18S rRNA | F: AGACACTCGTGCCTTCTTGCC | - | 100.8 | 0.988 | 文献[24] Reference [24] |
| R:CAACGATGCGTGACACCCAG | |||||
| AtActin | F: TGGTCAAGGCTGGGTTTGC | - | 118.1 | 0.984 | 文献[25] Reference [25] |
| R:CTGACCCATCCCAACCATGA | |||||
| VvUBQ | F: TCTGAGGCTTCGTGGTGGTA | - | 93.2 | 0.997 | 文献[26] Reference [26] |
| R:AGGCGTGCATAACATTTGCG | |||||
| VvActin | F: GCCCCTCGTCTGTGACAATG | - | 121.6 | 0.994 | 文献[26] Reference [26] |
| R:CCTTGGCCGACCCACAATA | |||||
| R: CAAGGTTGGTCATCACATGG |
新窗口打开
RT-PCR(reverse transcription PCR,反转录PCR)产物的琼脂糖电泳(图1)表明,12对引物均为单一条带,说明其特异性良好(AtActin电泳图存在一条较低的条带,经阴性对照组比较,发现其应该为引物二聚体,较高条带为特异性扩增的单一条带);而基因ChaSTP5和ChaTF的表达存在明显差异,初步判断这两个基因不适合内参基因。12个候选内参基因引物的RT-qPCR溶解曲线(图2)均为单一峰,也表明12个引物的特异性良好。
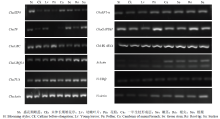 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图112个候选内参基因的RT-PCR琼脂糖电泳图
-->Fig. 1Agarose gel of RT-PCR products of 12 candidate reference genes
-->
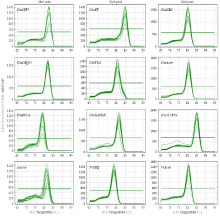 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图212个候选内参基因的溶解曲线
-->Fig. 2The melting curves of 12 candidate reference genes
-->
2.2 候选内参基因表达谱
根据RT-qPCR的结果,分析了12个候选内参基因在8个样品中的Cq值分布情况(图3)。12个候选内参基因的Cq值介于32.09—16.02,波动范围较大。Cq值与其表达量成反比,因此,Ch18S rRNA的表达量最高(16.02—20.55),ChaSTP5表达量最低(25.34—32.09),其余10个候选内参基因均为中等表达量。通过Excel分析每个候选内参基因在所有样品中Cq值的平均数(mean),标准差(standard deviation,SD)和变异系数(co-variance,CV)如表6所示。根据变异系数,可初步判断12个候选内参基因的稳定性:ChaTF(9.92%)稳定性最差,而VvActin(4.31%)和ChaActin(4.66%)则较稳定;其余候选内参基因的稳定性处于中等水平。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图312个候选内参基因在8个样品中的Cq值分布
-->Fig. 3Ct values of 12 candidate reference genes in 8 samples
-->
Table 6
表6
表6候选内参基因Cq值分析
Table 6Analysis of the Cq values of candidate reference genes
| 基因 Gene | 平均数 Mean | 标准差 SD | 变异系数 CV |
|---|---|---|---|
| ChaSTP5 | 26.89 | 2.47 | 8.74% |
| ChaTF | 25.41 | 2.68 | 9.92% |
| ChaUBQc | 25.82 | 1.56 | 6.06% |
| ChaUBQ14 | 21.97 | 1.12 | 5.11% |
| ChaTUA | 22.93 | 1.69 | 7.25% |
| ChaActin | 23.13 | 1.07 | 4.66% |
| ChaEF1-α | 22.19 | 1.86 | 8.35% |
| ChaGAPDH | 24.87 | 1.87 | 7.35% |
| Ch18S rRNA | 18.07 | 1.52 | 8.16% |
| AtActin | 25.50 | 1.92 | 7.35% |
| VvUBQ | 22.36 | 1.39 | 6.42% |
| VvActin | 26.40 | 1.12 | 4.31% |
新窗口打开
2.3 候选内参基因稳定性分析
geNorm程序是根据计算所得的M值来表示候选基因表达稳定性,M值越小的候选基因稳定性越好。本研究中,geNorm程序的分析结果表明,ChaActin的M值最低,其稳定性最好,适合作为内参基因;其次,VvActin、ChaEF1-α和Ch18S rRNA的排名比较靠前,也可作为候选内参基因;而ChaSTP5和ChaTF的M值高排名靠后,不适合作为备选的内参基因(表7)。此外,geNorm程序会根据变异系数(pairwise variation)来评估适合作为标准化处理所需内参基因的数目,默认变异系数(Vn/Vn+1)小于0.15时,所需最优内参基因数即为n个。但本研究的变异系数分析(图4)的值均大于0.15,根据图4中变异系数变化趋势,认为V6/V7(0.189)已经属于较低值,即选取6个内参基因进行精准的数据标准化处理。
NomFinder程序则是根据候选基因的表达稳定值(stability value,SV),选出稳定性最好的基因。SV值越小,候选基因越稳定,反之亦然。本研究NormFinder分析结果表明,ChaActin的SV值最小,最为稳定,这与geNorm中结果一致;其次稳定性较好的为VvActin、ChaTUA、ChaEF1-α和Ch18S rRNA;最不稳定的还是ChaSTP5和ChaTF(表7)。
导入Ct值,BestKeeper程序能计算获得候选内参基因在所有样品中Ct值的标准差(SD),变异系数(CV)和基因间相关系数(r)等值。由于程序限制仅可同时分析10个候选内参基因的稳定性,根据BestKeeper分析结果的SD排名(表7)可知:ChaActin依然是最为稳定的内参基因;其次稳定性较好的为ChaUBQ14、VvActin和Ch18S rRNA;AtActin的稳定性最差,不建议作为内参基因使用。
Delta Ct法,通过Ct值计算候选内参基因的平均标准差(Mean SD),以此评价其稳定性,其值越小内参基因越稳定。本研究中Delta Ct法分析结果表明,稳定性最好的是ChaActin、VvActin和Ch18S rRNA;ChaSTP5和ChaTF的SD最高,不适合作为内参基因(表7)。
2.4 候选内参基因综合评价
RefFinder在线网站包括了2.3中4种内参基因稳定性评价程序,并能对其分析结果进行评价给出综合排名。RefFinder综合排名(表8)表明,ChaActin和VvActin排名靠前,说明Actin适合作为榛属植物不同组织器官分析时的内参基因,ChaActin位列第一,说明根据榛属自身基因设计的引物更为合适;Ch18S rRNA则紧靠Actin之后,也是较为稳定的候选内参基因,而根据2.3可知Ch18S rRNA在4个不同的稳定性程序分析结果中排名均在前五之中;而ChaSTP5和ChaTF基因的综合排名靠后,这与前面稳定性分析结果相一致,说明其最不适合作为内参基因。一般选取双内参基因更能对荧光定量结果进行较为准确的标准化处理,综合分析可知ChaActin和Ch18S rRNA可选取为最适的双内参基因。Table 7
表7
表7候选内参基因稳定性排名
Table 7The stability ranking of candidate reference genes
| geNorm | NormFinder | BestKeeper | Delta Ct | |||||
|---|---|---|---|---|---|---|---|---|
| 基因 Gene | M值 M | 基因 gene | 稳定值 SV | 基因 gene | 标准差 SD | 基因 gene | 标准差 SD | |
| 1 | ChaActin | 1.46 | ChaActin | 0.36 | ChaActin | 0.82 | ChaActin | 1.43 |
| 2 | VvActin | 1.56 | VvActin | 0.39 | ChaUBQ14 | 0.83 | VvActin | 1.48 |
| 3 | ChaEF1-α | 1.65 | ChaEF1-α | 0.54 | VvActin | 0.86 | Ch18S rRNA | 1.67 |
| 4 | Ch18S rRNA | 1.73 | ChaTUA | 0.63 | Ch18S rRNA | 1.11 | ChaEF1-α | 1.70 |
| 5 | ChaTUA | 1.75 | Ch18S rRNA | 0.70 | VvUBQ | 1.13 | VvUBQ | 1.71 |
| 6 | ChaUBQ14 | 1.84 | ChaUBQ14 | 0.75 | ChaUBC | 1.20 | ChaTUA | 1.74 |
| 7 | VvUBQ | 1.91 | VvUBQ | 0.81 | ChaGAPDH | 1.46 | ChaUBQ14 | 1.87 |
| 8 | AtActin | 2.18 | AtActin | 0.97 | ChaTUA | 1.47 | AtActin | 2.01 |
| 9 | ChaUBQc | 2.29 | ChaUBC | 1.17 | ChaEF1-α | 1.48 | ChaUBC | 2.17 |
| 10 | ChaGAPDH | 2.29 | ChaGAPDH | 1.21 | AtActin | 1.64 | ChaSTP5 | 2.32 |
| 11 | ChaSTP5 | 2.43 | ChaTF | 1.41 | ChaGAPDH | 2.40 | ||
| 12 | ChaTF | 2.54 | ChaSTP5 | 1.66 | ChaTF | 2.72 | ||
新窗口打开
Table 8
表8
表8稳定性的综合排名
Table 8The comprehensive ranking of stability
| 基因 Gene | 排名的几何平均数 Geomean of ranking values |
|---|---|
| ChaActin | 1 |
| VvActin | 1.86 |
| Ch18S rRNA | 3.46 |
| VvUBQ | 4.61 |
| ChaUBQ14 | 5.12 |
| ChaEF1-α | 5.48 |
| ChaTUA | 5.83 |
| ChaUBQc | 8.13 |
| AtActin | 8.46 |
| ChaGAPDH | 9.59 |
| ChaSTP5 | 10.49 |
| ChaTF | 12 |
新窗口打开
利用4种程序的排名稳定性值的皮尔逊相关系数(pearson correlation coefficient,r)分析其相关性(表9):4个程序均在0.01水平上显著相关,且相关系数均高于0.9;NormFinder和Delta Ct的相关性最高(r=0.994),其次为geNorm和NormFinder;而geNorm和Delta Ct与BestKeeper的相关性较低。
Table 9
表9
表9程序的相关性分析
Table 9Correlation analysis of program
| 程序 Program | 相关系数 r |
|---|---|
| geNorm VS NormFinder | 0.985** |
| geNorm VS BestKeeper | 0.942** |
| geNorm VS Delta Ct | 0.977** |
| NormFinder VS BestKeeper | 0.950** |
| NormFinder VS Delta Ct | 0.994** |
| BestKeeper VS Delta Ct | 0.935** |
新窗口打开
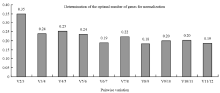 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4变异系数分析
-->Fig .4Pairwise variation (V) calculated by geNorm
-->
3 讨论
筛选合适的稳定内参基因是实现通过实时荧光定量PCR技术分析基因表达情况的先决条件,本研究根据笔者课题组前期转录组测序数据及前人相关研究,筛选出2个最适的稳定内参基因ChaActin和Ch18S rRNA。而戴超等[28]的研究证明,18S rRNA、Actin和Ubiquitin 3个基因适合作为白桦半定量PCR的内参基因;陈新[25]的研究证明,半定量PCR中Actin也适合作为平榛的内参基因,这均与本研究结果相一致,说明Actin在桦木科中表达稳定,适合作为内参基因。此外,这两个基因在其他物种中也是常用的内参基因,如:蒋婷婷等[29]研究中,Actin适合作为换锦花(Lycoris sprengeri)不同组织和不同花期以及石蒜属(Lycoris)不同杂交种鳞茎的内参基因;李冉等[30]研究表明在水稻(Oryza sativa)中稻纵卷叶螟(Cnaphalocrocis medinalis Guenee)处理后Actin的表达最稳定;苏晓娟等[31]认为Actin和18S rRNA等基因适合作为毛果杨(Populus trichocarpa)不同组织以及锌胁迫下的组培苗表达分析的内参基因;张岗等[32]则以EF-1α/18S rRNA为内参基因分析铁皮石斛(Dendrobium officinale)FPS的组织表达情况;KIM等[33]认为水稻逆境胁迫中18S rRNA表达稳定性最高。本研究发现,根据材料本身转录组测序数据设计的基因引物比直接引用其他材料引物的稳定性更好,如ChaActin的稳定性明显优于AtActin,说明根据材料基因序列设计特异引物效果更好。前人研究也证明利用转录组等基因表达数据库可以快速筛选获得理想的内参基因:SANG等[34]利用转录组数据筛选出了适合分析超积累型东南景天(Hyper- accumulating Sedum alfredii Hance)的内参基因UBC9和TUB;朱友银等[35]利用转录组数据筛选出适合作为中国樱桃(Prunus pseudocerasus)低温响应和盐碱胁迫处理的内参基因GAPDH,以及ABA处理和花芽休眠解除过程中的内参基因ACTB和UBCE;而刘洪峰等[36]利用转录组数据筛选出适合牡丹(Paeonia ostii)的新型内参基因PUF1639、MBF1A、PP2CFP和RPS9。综上说明,利用转录组和基因芯片等基因表达数据库能够更加高效、快速的筛选出合适的内参基因。
本研究在构建内参基因筛选体系时发现,通过反转录PCR能够快速剔除在分析材料中表达明显不稳定的基因,实现对候选内参基因的初步筛选,节约试验时间和经费;相关性分析表明4个程序排名显著相关,其中NormFinder和DeltaCt的相关性最高,与GUO等[37]的研究结果相一致。此外,geNorm程序可以根据配对变异(pairwise variation)V值,来确定理想候选内参基因个数[21]。程序默认Vn/Vn+1小于0.15,则没有必要再引入第n+1个基因,反之则需引入新的内参基因。本研究所用的材料为不同组织器官,其跨度本身较大,这可能是导致变异系数分析中其值均高于0.15的原因,而蒋婷婷等[29]也提到可能由于材料跨度过大会导致变异系数分析值均高于0.15。此外,程序本身也说明0.15并不是一个苛刻的限制,根据试验情况,其值可进行相应的调整。但是为了筛选出适合于不同组织材料的内参基因应该选取更多的候选基因进行表达稳定性分析,获取最为合适的内参基因,以达到数据标准化分析的要求。本研究中12个候选内参基因在4种不同程序中的稳定性排名存在一定的差异,这应该是由于程序设计的原理不同而导致,在筛选内参基因时应该用4个程序同时进行分析,从而选出最适的稳定内参基因,这样结果的可靠性更高;而RefFinder在线网站能快速的对原始Ct值进行分析得出综合排名,由于其计算过程中未考虑到引物扩增效率,其分析结果可能存在一定误差,建议与4个程序单独计算的结果进行比较分析。
4 结论
本研究利用geNorm、Normfider、Bestkeeper和Delta Ct法以及在线网站RefFinder,分析了12个候选内参基因在榛属植物8个不同组织器官中的表达稳定性,筛选出2个最适的稳定内参基因ChaActin和Ch18S rRNA;并构建了平欧杂种榛内参基因的筛选体系:反转录PCR初筛引物,荧光定量PCR分析引物特性及基因表达,4个程序(geNorm、Normfider、Bestkeeper、和Delta Ct法)分别评价引物稳定性,RefFinder综合分析选出最适的稳定内参基因。研究结果为榛属植物实时荧光定量PCR内参基因的筛选与分析提供了研究基础,也为其相关基因的组织表达分析提供了可参考的稳定内参基因。The authors have declared that no competing interests exist.
