 ,, 韩露, 谢建国, 潘文婧, 张泽鑫, 辛大伟, 刘春燕, 陈庆山
,, 韩露, 谢建国, 潘文婧, 张泽鑫, 辛大伟, 刘春燕, 陈庆山 ,, 齐照明
,, 齐照明 ,东北农业大学农学院大豆遗传改良实验室,哈尔滨 150030
,东北农业大学农学院大豆遗传改良实验室,哈尔滨 150030Mapping QTL for Soybean Fatty Acid Composition Based on RIL and CSSL Population
QU KeXin ,, HAN Lu, XIE JianGuo, PAN WenJing, ZHANG ZeXin, XIN DaWei, LIU ChunYan, CHEN QingShan
,, HAN Lu, XIE JianGuo, PAN WenJing, ZHANG ZeXin, XIN DaWei, LIU ChunYan, CHEN QingShan ,, QI ZhaoMing
,, QI ZhaoMing ,Soybean Genetic Improvement Laboratory, College of Agriculture, Northeast Agricultural University, Harbin 150030
,Soybean Genetic Improvement Laboratory, College of Agriculture, Northeast Agricultural University, Harbin 150030通讯作者:
责任编辑: 李莉
收稿日期:2021-02-3接受日期:2021-03-22
| 基金资助: |
Received:2021-02-3Accepted:2021-03-22
作者简介 About authors
渠可心,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (1323KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
渠可心, 韩露, 谢建国, 潘文婧, 张泽鑫, 辛大伟, 刘春燕, 陈庆山, 齐照明. 基于RIL和CSSL群体定位大豆脂肪酸组分QTL[J]. 中国农业科学, 2021, 54(15): 3168-3182 doi:10.3864/j.issn.0578-1752.2021.15.003
QU KeXin, HAN Lu, XIE JianGuo, PAN WenJing, ZHANG ZeXin, XIN DaWei, LIU ChunYan, CHEN QingShan, QI ZhaoMing.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】大豆(Glycine max)原产于中国,是一种重要的经济作物,占世界油籽产量的61%,蛋白质食物消费量的68%[1]。大豆是植物油的主要供给原料,油分含量占籽粒干重的18%—23%,主要包含5种重要脂肪酸组分:棕榈酸(palmitic acid)、硬脂酸(stearic acid)、油酸(oleic acid)、亚油酸(linoleic acid)和亚麻酸(linolenic acid)。大豆已经成为人们日常生活中膳食消耗不可缺少的食品,与国民粮食安全的可持续发展紧密相连[2,3,4]。目前,中国处于大豆进口最多的国家,而且高品质的大豆在食品、饲料、纺织品等多种加工工业中的需求量日益剧增,出现了较为明显的供需矛盾,鉴于这一问题,选育高品质大豆新品种已成为育种者和生产者的聚焦问题[5,6]。【前人研究进展】大豆脂肪酸中,亚油酸含量最高,油酸次之,亚麻酸、棕榈酸、硬脂酸含量相对较低[7],其中,油酸已被证明可以降低血液中的胆固醇含量[4],亚油酸已被证明可以防止动脉硬化的作用[8],亚麻酸具有降血压、健脑抗衰老等作用[9]。因此,在衡量大豆油品质的优劣中,脂肪酸各组分的比例是一个非常重要的指标。大豆脂肪酸组分含量的检测方法主要包括气相色谱法(gas chromatography,GC)[10,11]、近红外反射光谱分析法(near infrared reflectance spectrometry,NIRS)[12,13]和高效液相色谱法(high performance liquid chromatography,HPLC)[14,15],而气相色谱法是应用最为广泛的[16]。气相色谱法是色谱法中的一种,主要是利用混合物各物质之间的沸点、极性及吸附性的差异性来实现分离,目前,现代分离分析主要应用该技术[17]。大豆脂肪酸的5种组分是由多基因控制的数量性状,容易受不同环境条件的影响[18]。国内外研究****鉴定影响大豆籽粒贮藏物含量的QTL位点,主要通过分子标记技术结合区间作图(interval mapping,IM)[19]、复合区间作图(composite interval mapping,CIM)[20]、多区间作图(multiple interval mapping,MIM)[21]和完备区间作图(inclusive composite interval mapping,ICIM)[22]等常用的分析方法来进行。QTL定位的实质是分析分子标记与目标性状之间的连锁关系[23],随着分子生物技术的飞速发展,国内外研究****已开发出适应各种情况的QTL定位方法,大大地解决了分子辅助育种过程中所遇到的难题,为分子辅助育种的高效快速发展奠定了坚实基础[24]。目前,国内外的研究****对大豆脂肪酸含量进行了大量的QTL定位研究[25,26]。LI等[27]共定位到26个与脂肪酸含量相关的稳定QTL;XIA等[28]对2个群体在不同环境下进行大豆脂肪酸各组分的QTL定位分析,共定位到16个相关的QTL;FAN等[29]共定位到52个相关的QTL;盛英华等[30]对2个群体的脂肪酸含量进行QTL定位分析,共定位到57个相关的QTL。【本研究切入点】目前,虽然与大豆脂肪酸相关的QTL已被大量报道,但是可以在不同环境或不同遗传背景中被重复检测到的QTL较少[31]。【拟解决的关键问题】本研究于2017年和2018年测定190份染色体段代换系(chromosome segment substitution lines,CSSLs)群体及亲本和147份重组自交系(recombinant inbred lines,RILs)群体及亲本的脂肪酸各组分含量,并对大豆脂肪酸各组分进行QTL定位,以期获得能在不同遗传背景下被重复检测到的脂肪酸QTL。对检测到QTL的定位区段进行候选基因的挖掘,并选择与脂肪酸相关的基因,为后续与脂肪酸各组分相关基因的精细定位及基因功能的研究奠定基础,并对大豆品质改良方面具有一定的指导意义。1 材料与方法
1.1 试验材料
选用东北农业大学农学院大豆遗传改良实验室历经12年构建,以栽培大豆绥农14与野生大豆ZYD00006为亲本,并以绥农14为轮回亲本,采用分子辅助选择段构建的一套全基因组染色体片段代换系(CSSLs)的190份材料。以美国半矮秆大豆品种Charleston(♂,中国农业科学院作物科学研究所提供)和高蛋白大豆品系东农594(♀,东北农业大学大豆研究所提供)亲本构建的重组自交系(RILs)群体的147份材料。2017—2018年播种在具有适当土壤含水量(15%—20%)的中国哈尔滨(45.75°N,126.53°E)的向阳农场试验田,采用完全随机区组设计,行长5 m,株距5 cm,垄距60 cm,3次重复,田间管理遵循正常的生产实践。1.2 脂肪酸的测定方法
采用Agilent7890B气相色谱仪测定大豆脂肪酸的5种组分,每份材料随机选取3粒籽粒,于65℃烘箱中烘干(12—14 h),研磨成粉末,称取5 mg左右样品,置于储存管中。在储存管中分别加入1 mL 2.5%的浓硫酸甲醇溶液、5 μL BHT(2,6-二叔丁基-4-甲基苯酚50 mg·mL-1,甲醇溶解)还原剂和50 μL的内标十七烷酸(10 mg·mL-1,乙酸乙酯溶解),混匀后85℃水浴1.5 h,冷却至室温,加入160 µL 9%的NaCl溶液和700 µL正己烷,涡旋、震荡,4 000 r/min常温离心10 min,吸取上清液400—500 μL于新储存管中,置于通风橱中吹干、过夜。最后加400 μL乙酸乙酯并迅速用气相色谱仪进行脂肪酸的测定,每个样品测定3次,取平均值作为该脂肪酸组分的含量。Agilent7890B气相色谱仪所用色谱柱型号为:30 m×320 µm×0.25 µm。载气:氮气60 mL·min-1,氢气60 mL·min-1,空气450 mL·min-1。进样量:1 µL,分流进样方式,分流比为10﹕1;进样口温度170℃。反应程序为180℃保持1 min,以25℃·min-1的速率上升至250℃保持7 min。
气相色谱分析过程中,首先试样在汽化室内被汽化,然后被惰性气体(即流动相)带入色谱柱,柱内含有固定相,由于试样中各组分之间的气相和固定相之间的分子作用力不同,使得在色谱柱中各个组分的运行速度不同,从而产生了不同的峰值,最后分析色谱图中各种组分的峰值,计算出不同组分的相对含量[21]。
1.3 脂肪酸各组分含量计算
气相色谱仪的峰图是以标准溶液浓度为横坐标,色谱峰面积为纵坐标,从而绘制不同浓度梯度的标准曲线[16]。根据5种脂肪酸标准样品的保留时间和峰面积进行定性定量分析各个脂肪酸组分,以十七烷酸的峰值为内标,计算5种脂肪酸的绝对含量(mg·g-1)和相对含量(%),油分含量为5种脂肪酸的总和。绝对含量计算公式:$\omega i=Ai \times \frac{ms}{As \times m}$,Ai为第i种脂肪酸组分的峰面积,As为内标的峰面积,ms为内标的物质质量,m为样品干重。
相对量计算公式:$w (\%)=(\frac{Ai}{\sum Ai})\times 100\%$。
1.4 脂肪酸含量表型及QTL分析
采用Microsoft Excel 2010软件对2个群体亲本和后代单株脂肪酸各组分进行基本统计量分析,包括最小值、最大值、平均值、变异系数、标准差、变异幅度、偏度和峰度。采用SPSS软件对脂肪酸各组分进行相关性分析。RIL群体的遗传图谱由QI等[32]构建完成,图谱总长度为2 655.68 cm,包括5 308个特异长度扩增片段标记,覆盖20个连锁群(linkage group,LGs),标记间平均距离为0.5 cm。CSSL群体的遗传图谱由XIN等[33]构建完成。使用Win-QTLCart2.5软件通过CIM对RILs群体进行QTL定位,使用1 000次排列检验分析表型数据,并设置显著性P=0.05,取LOD=2.5为阈值判断QTL的存在。采用ICIM Mapping软件中ICIM对CSSLs群体进行与脂肪酸相关的QTL定位分析,使用1 000次排列检验分析表型数据,并设置显著性P=0.05。QTL命名基于前研究者的方法,简而言之,QTL名称的构造如下:q+性状名称+连锁群或连锁群数+“-”+QTL数目[34]。
2 结果
2.1 2个群体的脂肪酸含量和油分总量分布情况
运用气象色谱仪对2017—2018年的RILs和CSSLs群体籽粒中的5种脂肪酸含量和油分总量进行测定,并计算5种脂肪酸和油分含量的频率分布,以分析大豆籽粒在不同年份、不同群体脂肪酸积累的特点。结果表明,2017—2018年2个群体的脂肪酸各组分都呈现连续正态分布(图1和图2),表明脂肪酸是以数量方式遗传的,并适用于脂肪酸各组分含量的QTL定位。油分含量变化范围为19.66%—24.26%,符合正态分布(图3),适用于油分含量的QTL定位,此外,在后代中也观察到明显的超亲分离现象[27],表明父母双方对脂肪酸组成都有贡献。图1
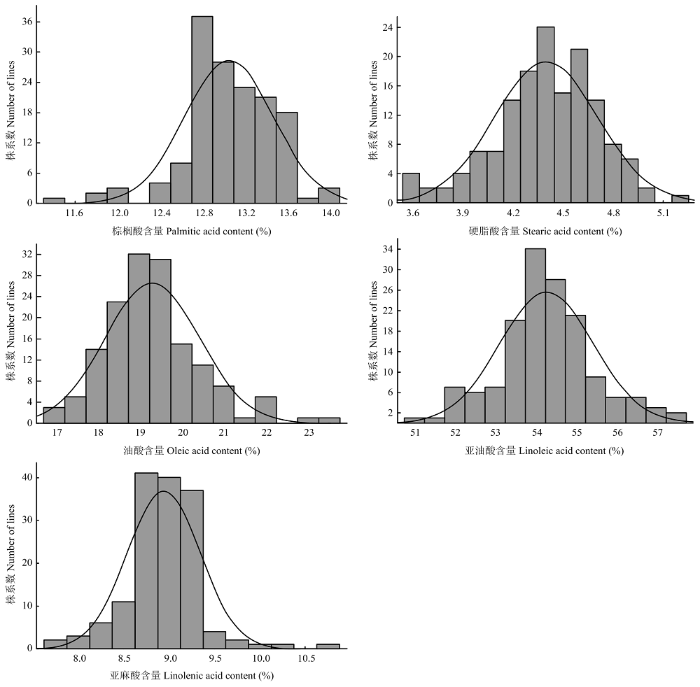 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图12017年RILs群体中5种脂肪酸含量分布直方图
X轴代表脂肪酸各组分所占总油分的百分比,Y轴代表每个脂肪酸各组分所占总油分百分比的株系数,黑色实线表示RILs群体的正常曲线。2018年RILs群体的5种脂肪酸含量频率分布与2017年的结果相同,呈现连续正态分布,所以在这里仅展示了2017年的结果。下同
Fig. 1Distribution histogram of the 5 fatty acids content in the RILs population in 2017
The X-axis represents the percentage of each fatty acid component to the total oil content, the Y-axis represents the plant coefficient of the percentage of each fatty acid component in the total oil content, and the black solid line represents the normal curve of the RILs population. The frequency distribution of the five fatty acid content of the RILs population in 2018 is the same as the 2017 results, showing continuous normal distribution, so only the 2017 results are shown here. The same as below
图2
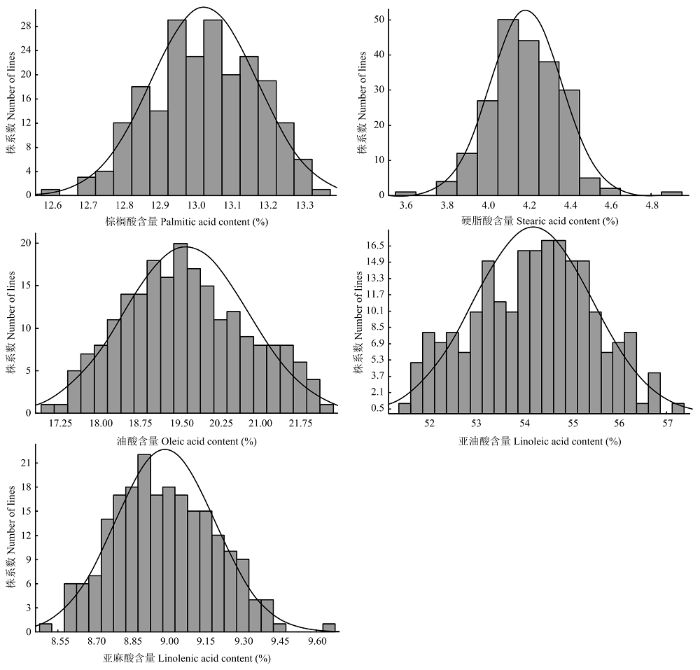 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图22017年CSSLs群体5种脂肪酸含量分布直方图
黑色实线表示CSSLs群体的正常曲线
Fig. 2Distribution histogram of the 5 fatty acids content in the CSSLs population in 2017
Black solid line means normal curve of the CSSLs population
图3
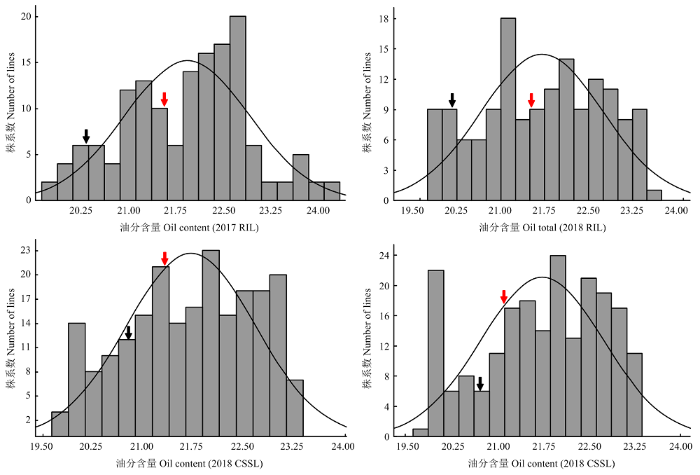 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图32017—2018年RILs和CSSLs群体油分总量分布直方图
黑色箭头代表RILs群体和CSSLs群体的父本Charleston和野生豆(ZYD00006)油分总量所在位置,红色箭头代表RILs群体和CSSLs群体的母本东农594和绥农14油分总量所在位置
Fig. 3Distribution histogram of total oil in the RILs and CSSLs populations in 2017-2018
The black arrow represents the oil content of the male parent Charleston and wild bean (ZYD00006) in the RILs and CSSLs populations, and the red arrow represents the oil content of the female parent Dongnong 594 and Suinong 14 of the RILs and CSSLs groups
2.2 RILs和CSSLs群体脂肪酸含量表型分析
对2017—2018年2个群体亲本及后代单株的脂肪酸各组分含量和油分总量进行基本统计分析(表1)。2年的RILs、CSSLs群体母本的棕榈酸、硬脂酸和油酸含量均低于父本,而亚油酸和亚麻酸含量均高于父本,RILs群体2个亲本的油酸含量差异较大,而CSSLs群体2个亲本的油酸、亚油酸含量差异较大。后代群体的5种脂肪酸含量中除硬脂酸的平均值比亲本高,其余性状的平均值均位于两亲本之间,并且具有双向超亲的现象。这5种脂肪酸在RILs和CSSLs 2个群体中均存在较大变异,变异系数为1.13%—6.99%,其中,亚油酸的变异系数(coefficient of variation,CV)最小,为1.76%—2.31%;硬脂酸的变异系数(CV)最大,为3.91%—6.99%,2017年RILs群体的变异系数达到6.99%;油酸的变异系数仅次于硬脂酸,表明硬脂酸和油酸在后代群体具有较大差异,提高其在大豆5种脂肪酸组分中所占的比例具有一定意义。同一群体中5种脂肪酸各组分最大值和最小值差异较明显,说明在后代分离群体中的表现分离程度较大,且变异连续,是属于多基因控制的数量性状,受基因和环境的共同影响。Table 1
表1
表12个群体亲本和后代单株脂肪酸含量的基本统计
Table 1
| 性状 Trait | 群体 Population | 亲本Parent | 群体Population | |||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 父本 Male | 母本 Female | 最小值 Minimum | 最大值 Maximum | 平均值 Mean | 标准差 SD | 变异系数 CV | 偏度 Skewness | 峰度 Kurtosis | ||
| 棕榈酸 PA (%) | 17-RIL | 12.80 | 13.15 | 11.48 | 13.98 | 13.05 | 0.42 | 3.21 | -0.76 | 1.82 |
| 17-CSSL | 12.75 | 13.17 | 12.62 | 13.33 | 13.03 | 0.15 | 1.13 | -0.18 | -0.61 | |
| 18-RIL | 12.74 | 13.09 | 12.11 | 13.27 | 12.90 | 0.18 | 1.42 | -0.63 | -1.04 | |
| 18-CSSL | 12.81 | 13.12 | 12.70 | 13.60 | 13.05 | 0.15 | 1.16 | -0.09 | -0.05 | |
| 硬脂酸 SA (%) | 17-RIL | 4.09 | 4.28 | 3.59 | 5.19 | 4.40 | 0.31 | 6.99 | -0.43 | 0.15 |
| 17-CSSL | 3.94 | 4.17 | 3.64 | 4.95 | 4.19 | 0.17 | 4.17 | 0.21 | 1.19 | |
| 18-RIL | 4.03 | 4.23 | 3.92 | 4.99 | 4.53 | 0.27 | 5.88 | -0.28 | -0.86 | |
| 18-CSSL | 4.04 | 4.15 | 3.80 | 4.68 | 4.23 | 0.17 | 3.91 | 0.28 | -0.01 | |
| 油酸 OA (%) | 17-RIL | 19.19 | 20.35 | 17.00 | 23.53 | 19.33 | 1.12 | 5.78 | 0.84 | 1.59 |
| 17-CSSL | 19.10 | 20.71 | 17.00 | 22.20 | 19.61 | 1.17 | 5.94 | 0.16 | -0.63 | |
| 18-RIL | 19.13 | 20.06 | 18.09 | 22.25 | 19.53 | 0.83 | 4.25 | 0.64 | 0.59 | |
| 18-CSSL | 19.29 | 20.72 | 17.03 | 22.25 | 19.45 | 0.98 | 5.03 | 0.43 | -0.13 | |
| 亚油酸 LA (%) | 17-RIL | 54.81 | 53.35 | 51.15 | 57.37 | 54.28 | 1.16 | 2.13 | 0.12 | 0.46 |
| 17-CSSL | 55.17 | 53.19 | 51.59 | 57.37 | 54.20 | 1.25 | 2.31 | -0.07 | -0.62 | |
| 18-RIL | 54.97 | 53.76 | 51.47 | 56.12 | 54.10 | 0.95 | 1.76 | -0.14 | -0.06 | |
| 18-CSSL | 54.85 | 53.14 | 51.65 | 57.09 | 54.27 | 1.06 | 1.95 | -0.29 | -0.29 | |
| 亚麻酸 LNA (%) | 17-RIL | 9.11 | 8.87 | 7.75 | 10.87 | 8.94 | 0.40 | 4.51 | 0.47 | 3.93 |
| 17-CSSL | 9.04 | 8.76 | 8.52 | 9.63 | 8.98 | 0.20 | 2.25 | 0.22 | -0.42 | |
| 18-RIL | 9.13 | 8.86 | 8.29 | 9.34 | 8.93 | 0.19 | 2.11 | -0.81 | 0.37 | |
| 18-CSSL | 9.01 | 8.87 | 8.30 | 9.63 | 8.99 | 0.19 | 2.12 | -0.26 | 0.96 | |
| 油分 OIL (%) | 17-RIL | 20.25 | 21.62 | 19.82 | 24.26 | 21.94 | 0.99 | 4.53 | -0.08 | -0.44 |
| 17-CSSL | 20.79 | 21.34 | 19.74 | 23.35 | 21.70 | 0.96 | 4.40 | -0.24 | -0.97 | |
| 18-RIL | 20.11 | 21.46 | 19.66 | 23.38 | 21.58 | 1.04 | 4.83 | -0.11 | -1.04 | |
| 18-CSSL | 20.53 | 21.19 | 19.87 | 23.33 | 21.74 | 1.00 | 4.59 | -0.36 | -0.93 | |
新窗口打开|下载CSV
对2017—2018年RILs和CSSLs群体的5种脂肪酸含量进行相关性分析(表2和表3),表明2个群体中5种脂肪酸含量相关性的趋势一致,其中,油酸与棕榈酸、硬脂酸、亚油酸和亚麻酸呈显著负相关;棕榈酸与硬脂酸、亚油酸和亚麻酸呈显著正相关,亚油酸与亚麻酸呈显著正相关。其中,油酸和亚油酸的相关系数最大,2018年RILs群体中达到-0.939,2017年CSSLS群体中达到-0.991,说明可以通过对油酸或亚油酸的选择,从而达到对另一种成分的选择。
Table 2
表2
表2RILs群体脂肪酸含量的相关分析
Table 2
| 性状 Traits | 棕榈酸PA | 硬脂酸SA | 油酸OA | 亚油酸LA | 亚麻酸LNA |
|---|---|---|---|---|---|
| 棕榈酸PA | 1 | 0.388** | -0.387** | 0.582** | 0.261** |
| 硬脂酸SA | 0.301** | 1 | -0.561** | 0.714** | 0.605** |
| 油酸OA | -0.015 | -0.171* | 1 | -0.939** | -0.790** |
| 亚油酸LA | 0.336** | 0.398** | -0.853** | 1 | 0.717** |
| 亚麻酸LNA | 0.301** | 0.275** | -0.352** | 0.230** | 1 |
新窗口打开|下载CSV
Table 3
表3
表3CSSLs群体脂肪酸含量的相关分析
Table 3
| 性状 Traits | 棕榈酸PA | 硬脂酸SA | 油酸OA | 亚油酸LA | 亚麻酸LNA |
|---|---|---|---|---|---|
| 棕榈酸PA | 1 | 0.649** | -0.846** | 0.873** | 0.855** |
| 硬脂酸SA | 0.815** | 1 | -0.651** | 0.726** | 0.684** |
| 油酸OA | -0.862** | -0.778** | 1 | -0.987** | -0.883** |
| 亚油酸LA | 0.895** | 0.830** | -0.991** | 1 | 0.868** |
| 亚麻酸LNA | 0.843** | 0.771** | -0.881** | 0.864** | 1 |
新窗口打开|下载CSV
2.3 大豆脂肪酸含量QTL定位
基于所构建的大豆遗传连锁图谱,采用Win-QTLCart2.5和ICIM Mapping软件分别对RILs和CSSLs群体进行QTL定位(表4和表5),2017—2018年的2个群体共定位到54个与脂肪酸组分相关的QTL,RILs和CSSLs群体分别定位到34和20个QTL,分布在除B2、C1、G、H、J、M和O以外的13个连锁群上,其中,以N连锁群上最多,有10个QTL;其次是A1连锁群上,有8个QTL;而A2和L连锁群上均只有1个。Table 4
表4
表4RIL群体脂肪酸含量QTL定位
Table 4
| 性状 Trait | 年份 Year | QTL名称 QTL name | 染色体 Chromosome | 起始位置 Start position (Mb) | 终止位置 End position (Mb) | 距离 Size (Mb) | LOD | 贡献率 R2 (%) | 加性效应 Additive effect |
|---|---|---|---|---|---|---|---|---|---|
| 棕榈酸 PA | 2017 | qPA-N-1 | 3 | 14.80 | 18.50 | 3.70 | 4.00 | 9.56 | 0.15 |
| 2017 | qPA-C2-1 | 6 | 20.47 | 21.61 | 1.14 | 4.44 | 10.51 | -0.05 | |
| 2017 | qPA-C2-2 | 6 | 27.37 | 27.84 | 0.46 | 3.74 | 8.95 | -0.04 | |
| 2017 | qPA-E-1 | 15 | 33.29 | 33.49 | 0.21 | 2.64 | 6.66 | -0.06 | |
| 2017 | qPA-E-2 | 15 | 32.53 | 32.61 | 0.07 | 5.60 | 13.50 | -0.09 | |
| 2017 | qPA-E-3 | 15 | 28.34 | 28.72 | 0.37 | 3.84 | 9.05 | 0.07 | |
| 2017 | qPA-E-4 | 15 | 22.73 | 22.77 | 0.04 | 2.70 | 6.47 | 0.05 | |
| 2018 | qPA-D1b-1 | 2 | 42.46 | 42.49 | 0.03 | 3.73 | 8.99 | -0.05 | |
| 2018 | qPA-A1-1 | 5 | 34.54 | 34.55 | 0.01 | 2.89 | 6.96 | -0.04 | |
| 2018 | qPA-F-1 | 13 | 15.74 | 15.83 | 0.09 | 5.32 | 13.16 | 0.09 | |
| 硬脂酸 SA | 2017 | qSA-N-1 | 3 | 40.50 | 44.10 | 3.60 | 2.98 | 7.62 | -0.03 |
| 2018 | qSA-D1b-1 | 2 | 42.61 | 42.62 | 0.01 | 2.84 | 6.90 | -0.03 | |
| 2018 | qSA-F-1 | 13 | 15.74 | 15.83 | 0.09 | 4.51 | 11.11 | 0.06 | |
| 2018 | qSA-D2-1 | 17 | 38.23 | 38.91 | 0.68 | 3.20 | 7.73 | 0.03 | |
| 油酸 OA | 2017 | qOA-D1a-1 | 1 | 13.87 | 14.26 | 0.39 | 3.24 | 7.71 | 0.10 |
| 2017 | qOA-N-1 | 3 | 23.25 | 23.29 | 0.04 | 3.86 | 9.09 | -0.11 | |
| 2017 | qOA-N-2 | 3 | 24.72 | 24.96 | 0.24 | 3.89 | 9.17 | -0.11 | |
| 2017 | qOA-A1-1 | 5 | 38.94 | 39.09 | 0.15 | 2.65 | 5.64 | 0.10 | |
| 2017 | qOA-D2-1 | 17 | 36.95 | 37.10 | 0.16 | 2.94 | 7.40 | 0.10 | |
| 2018 | qOA-A1-2 | 5 | 38.94 | 39.09 | 0.15 | 3.65 | 7.64 | 0.13 | |
| 2018 | qOA-D2-2 | 17 | 36.95 | 37.10 | 0.16 | 3.94 | 9.43 | 0.12 | |
| 亚油酸 LA | 2017 | qLA-N-1 | 3 | 23.05 | 23.11 | 0.06 | 2.76 | 6.99 | -0.24 |
| 2017 | qLA-N-2 | 3 | 28.10 | 28.27 | 0.17 | 3.25 | 8.29 | 0.26 | |
| 2017 | qLA-K-1 | 9 | 33.51 | 33.52 | 0.01 | 3.26 | 8.34 | -0.19 | |
| 2017 | qLA-L-1 | 19 | 42.92 | 43.49 | 0.57 | 2.86 | 7.26 | 0.19 | |
| 2018 | qLA- A1-1 | 5 | 0.03 | 0.80 | 0.77 | 3.10 | 8.03 | 0.17 | |
| 2018 | qLA-I-1 | 20 | 24.43 | 24.44 | 0.02 | 2.54 | 6.40 | -0.16 | |
| 亚麻酸 LNA | 2017 | qLNA-N-1 | 3 | 37.15 | 37.22 | 0.07 | 4.19 | 10.13 | 0.05 |
| 2017 | qLNA-K-1 | 9 | 33.51 | 33.52 | 0.01 | 5.46 | 13.49 | -0.05 | |
| 2017 | qLNA-B1-1 | 11 | 38.31 | 38.71 | 0.39 | 3.25 | 7.74 | -0.04 | |
| 2018 | qLNA -B1-2 | 11 | 38.31 | 38.71 | 0.39 | 4.25 | 9.89 | -0.07 | |
| 2018 | qLNA-I-1 | 20 | 45.55 | 45.74 | 0.19 | 3.20 | 8.16 | 0.03 | |
| 油分 OIL | 2017 | qOIL- D1b-1 | 2 | 6.25 | 6.51 | 0.25 | 2.67 | 6.95 | -0.60 |
| 2018 | qOIL-F-1 | 13 | 15.74 | 15.83 | 0.09 | 3.79 | 10.04 | 0.58 |
新窗口打开|下载CSV
Table 5
表5
表5CSSL群体脂肪酸含量QTL定位
Table 5
| 性状 Trait | 年份 Year | QTL名称 QTL name | 染色体 Chromosome | 起始位置 Start position (Mb) | 终止位置 End position (Mb) | 距离 Size (Mb) | LOD值 LOD | 贡献率 PVE (%) | 加性效应 Additive effect |
|---|---|---|---|---|---|---|---|---|---|
| 棕榈酸 PA | 2017 | qPA-N-2 | 3 | 18.57 | 19.23 | 0.66 | 2.86 | 4.62 | 0.16 |
| 2018 | qPA-N-3 | 3 | 18.57 | 19.23 | 0.66 | 6.87 | 7.75 | 0.16 | |
| 硬脂酸 SA | 2017 | qSA-C2-1 | 6 | 49.19 | 49.24 | 0.05 | 1.78 | 3.21 | 0.41 |
| 2018 | qSA-D1a-1 | 1 | 5.71 | 5.79 | 0.08 | 2.95 | 5.00 | 0.27 | |
| 油酸 OA | 2017 | qOA-F-1 | 13 | 13.72 | 13.81 | 0.09 | 9.60 | 8.80 | 0.30 |
| 2017 | qOA-E-1 | 15 | 40.66 | 40.67 | 0.01 | 3.98 | 5.22 | 0.29 | |
| 2018 | qOA-D2-3 | 17 | 14.17 | 14.25 | 0.09 | 2.75 | 4.49 | 0.22 | |
| 亚油酸 LA | 2017 | qLA-A1-2 | 5 | 0.51 | 0.53 | 0.02 | 11.65 | 22.09 | 0.33 |
| 2018 | qLA-A1-3 | 5 | 0.51 | 0.53 | 0.02 | 2.59 | 4.28 | 1.02 | |
| 亚麻酸 LNA | 2017 | qLNA-A1-1 | 5 | 34.40 | 34.44 | 0.04 | 3.80 | 3.93 | 0.05 |
| 2017 | qLNA-A2-1 | 8 | 2.45 | 2.49 | 0.03 | 4.43 | 4.73 | 0.03 | |
| 2018 | qLNA-K-2 | 9 | 2.35 | 2.40 | 0.05 | 2.31 | 2.92 | 0.23 | |
| 2018 | qLNA-F-1 | 13 | 0.80 | 0.82 | 0.02 | 2.96 | 2.97 | 0.07 | |
| 2018 | qLNA-F-2 | 13 | 11.48 | 11.55 | 0.07 | 5.51 | 5.86 | -0.04 | |
| 2018 | qLNA-E-1 | 15 | 49.40 | 49.44 | 0.03 | 2.97 | 3.01 | 0.04 | |
| 油分 OIL | 2017 | qOIL- D1a-1 | 1 | 5.71 | 5.79 | 0.08 | 3.59 | 7.96 | 0.43 |
| 2017 | qOIL-N-1 | 3 | 18.57 | 19.23 | 0.66 | 3.37 | 7.44 | 0.31 | |
| 2017 | qOIL-C2-1 | 6 | 49.19 | 49.24 | 0.05 | 3.67 | 8.14 | 0.52 | |
| 2018 | qOIL-A1-1 | 5 | 0.51 | 0.53 | 0.02 | 1.84 | 3.02 | 1.43 | |
| 2018 | qOIL-F-2 | 13 | 31.17 | 31.19 | 0.02 | 2.74 | 3.71 | 0.36 |
新窗口打开|下载CSV
检测到与棕榈酸相关的QTL共有12个,RILs群体检测到10个QTL分别位于A1、C2、D1b、E、F和N等6个连锁群上,贡献率为6.47%—13.50%,LOD值为2.64—5.60。其中,qPA-E-2被检测到位于E连锁群上,QTL效应值较大,贡献率为13.50%。CSSLs群体检测到2个QTL都位于N连锁群上,表型贡献率为4.62和7.75,LOD值为2.86和6.87。
检测到6个与硬脂酸相关的QTL,其中RILs群体有4个QTL,分布在D1b、D2、F和N等4个连锁群上,贡献率为6.90%—11.11%,LOD值为2.84—4.51。其中,qSA-F-1被检测到位于F连锁群上,QTL 效应值较大,贡献率为11.11%;CSSLs群体检测到2个QTL分别位于C2和D1b连锁群上,表型贡献率为3.21和5.00,LOD值为1.78和2.95。
检测到与油酸相关的QTL共有10个,其中RILs群体检测到7个QTL分别位于A1、D1a、D2、和N等4个连锁群上,贡献率为5.64%—9.43%,LOD值为2.65—3.94;CSSLs群体检测到3个QTL分别位于D2、E和F连锁群上,表型贡献率为4.49%、5.22%和8.80%,LOD值为2.75、3.98和9.60。
检测到8个与亚油酸相关的QTL,其中RILs群体有6个QTL,分布在A1、I、K、L和N等5个连锁群上,贡献率为6.40%—8.34%,LOD值为2.54—3.26;CSSLs群体检测到2个QTL都位于A1连锁群上,表型贡献率为22.09%和4.28%,LOD值为11.65和2.59。其中,qLA-A1-1被检测到位于A1连锁群上,QTL效应值较大,贡献率为22.09%。
检测到11个与亚麻酸相关的QTL,其中RILs群体有5个QTL,分布在B1、I、K和N等4个连锁群上,贡献率为7.74%—13.49%,LOD值为3.20—5.46,其中qLNA-K-1被检测到位于K连锁群上,QTL效应值较大,贡献率为13.49%;CSSLs群体检测到6个QTL分别位于A1、A2、E、F和K等5个连锁群上,表型贡献率为2.92%—5.86%,LOD值为2.31—5.51。
检测到7个与油分相关的QTL,其中RILs群体有2个QTL,分布在D1b和F等2个连锁群上,贡献率为6.95%和10.04%,LOD值为2.67和3.79,其中qOIL-F-1被检测到位于F连锁群上,QTL效应值较大,贡献率为10.04%;CSSLs群体检测到5个QTL分别位于A1、C2、D1a、F和N等5个连锁群上,表型贡献率为3.02%—8.14%,LOD值为1.84—3.67。
2.4 RIL和CSSL群体QTL分析结果比较
2017—2018年RIL和CSSL群体共定位到54个QTL,分布在13个连锁群上,在同一群体中被重复检测到的QTL有10对,分别位于A1、B1、C2、D1a、D2、F、K和N连锁群上(表4、表5和图4)。在RILs群体中重复检测到的QTL有6对,其中,在A1和D2连锁群上有2对QTL,均连续2年被重复检测到与油酸相关,说明这是2对是油酸相关的稳定QTL;在B1连锁群上的1对QTL同样连续2年被重复检测到与亚麻酸相关,分别解释了7.74%和9.89%的贡献率,说明该QTL是与亚麻酸相关的稳定QTL;另外在F连锁群上定位到的3个QTL,与棕榈酸、硬脂酸和油分均相关,定位在同一区间内,且位置相同,说明为棕榈酸、硬脂酸和油分含量重叠的QTL,可能调控多种脂肪酸的合成;定位在连锁群K上的1对QTL,检测与亚油酸、亚麻酸相关,说明该QTL可能会调控多种脂肪酸的合成;定位在连锁群N上的1对QTL,检测为油酸和亚油酸含量重叠的QTL,说明该QTL可能会调控多种脂肪酸的合成。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图42017—2018年RILs和CSSLs群体QTL在连锁群的分布




Fig. 4The linkage group distribution of QTLs of RILs and CSSLs population in 2017-2018




在CSSLs群体中被重复检测的有4对。定位在N连锁群上的3个QTL,其中qPA-N-1和qPA-N-2连续2年被重复检测到并与棕榈酸相关,说明可能是棕榈酸相关的稳定QTL,同时又与油分相关的qOIL-N-2为同一个QTL,说明其为棕榈酸和油分含量重叠的QTL,并且能够调控多种脂肪酸的合成,值得关注;定位在C2连锁群上的1对QTL和定位在D1a连锁群上的1对QTL,均为硬脂酸和油分含量重叠的QTL,说明可能是调控多种脂肪酸合成的QTL;在A1连锁群上有3个QTL,其中qLA-A1-2和qLA-A1-3连续2年被检测到并与亚油酸相关,并且qLA-A1-1在2017年表型贡献率达到了22.09%,说明可能是亚油酸相关的稳定QTL,同时又与油分相关的qOIL-A1-1为同一个QTL,说明其为亚油酸和油分含量重叠的QTL,可能是控制大豆脂肪酸含量的重要位点。
对不同群体定位到的脂肪酸组分的QTL进行比较发现,N连锁群上检测到的QTL最多,并且5种脂肪酸的QTL均被检测到,说明可能较多的与脂肪酸相关的QTL定位在该连锁群上。RILs群体的qLA-A1-1与CSSLs群体的qLA-A1-2被检测定位在A1连锁群上,且区间较近,可能为同一个QTL,可能是一个在不同群体中能够稳定遗传的QTL,定位结果需进一步验证。
2.5 候选基因挖掘及富集分析
通过对RILs和CSSLs群体的5种脂肪酸和油分总量进行的QTL定位,定位到54个QTL,对QTL定位所获得的区间进行候选基因的挖掘及GO富集数据分析。从基因注释数据集[35]中发现有9个区间不含基因,共筛选出485个候选基因,对具有GO注释的候选基因进行进一步的GO富集数据分析,如表6所示,有15个基因与大豆脂肪酸的形成直接或间接相关。这15个候选基因都在脂肪酸的合成或分解过程中发挥重要作用。其中候选基因Glyma.02G074000与酰基辅酶a/酰基转移酶有关,能够调节脂肪酸链的延伸,影响脂肪酸的合成[36,37];Glyma.06G211300编码植物酰基-酰基载体蛋白(acyl carrier protein,ACP)硫酯酶,ACP的特异性决定了大多数植物组织中饱和脂肪酸的水平[36,38],候选基因Glyma.06G211300对脂肪酸的形成非常重要,这两个基因都与脂肪酸合成的调控有关。候选基因Glyma.17G219100编码磷脂酶D1,已被证实磷脂酶D在甘油磷脂的代谢中起重要作用[39];候选基因Glyma.05G008100与二羟基丙酮激酶/FAD还原酶有关,是甘油醛生产的重要介质,与脂类的甘油代谢过程有关[40],这两个基因主要影响脂质代谢,从而调节植物组织中的脂肪酸含量。候选基因Glyma.02G073800、Glyma.06G211200和Glyma.13G044100都与乙醇乙酰基转移酶有关,是脂肪酸合成途径中的重要催化剂[41]。候选基因Glyma.05G208900编码一种脂肪酸-羟化酶,通过介导长链脂肪酸转化为ω-羟基脂肪酸,影响角质、软木脂和蜡的生物合成[42]。Glyma.05G000700是丙酮酸激酶家族蛋白成员,丙酮酸激酶是糖酵解途径中的一种限速酶,能够催化磷酸烯醇式丙酮酸生成ATP和丙酮酸的相关反应,而丙酮酸又是三羧酸循环的前体物质,参与物质代谢和氨基酸合成等多种生理生化反应[43],有研究发现,丙酮酸激酶有调节与控制光合作用的产物转化为油脂的作用[44]、是催化光合产物生成油脂的关键酶[45]。另外Glyma.17G228400编码脂肪酸去饱和酶,而Glyma.03G070200与酮酸脱羧酶有关,Glyma.05G007700、Glyma.13G044500、Glyma.19G171000和Glyma.20G221200 4个候选基因都与脂肪酶/酰基水解酶有关,这几个基因编码的蛋白质在几个方面影响脂肪酸含量。Table 6
表6
表6基因注释候选基因
Table 6
| 基因 Gene | KO注释 KO annotation | GO注释 GO annotation | 同源基因 Homologous gene | 基因功能 Gene description |
|---|---|---|---|---|
| Glyma.02G073800 | GO:0016747 | Glyma.06G211200 | 乙醇-乙酰基转移酶Alcohol-acetyltransferase | |
| Glyma.13G044100 | ||||
| Glyma.02G074000 | GO:0008080 | 酰基辅酶a/酰基转移酶Acyl-CoA/N-acyltransferases (NAT) | ||
| Glyma.03G070200 | K00167 | GO:0008152 | α-酮酸脱羧酶E1β亚基Alpha-keto acid decarboxylase E1 beta subunit | |
| Glyma.05G007700 | GO:0016788 | Glyma.13G044500 | 脂肪酶/酰基水解酶Lipase/Acyl hydrolase | |
| Glyma.19G171000 | ||||
| Glyma.20G221200 | ||||
| Glyma.05G008100 | K00326 | GO:0016491 | FAD/NAD(P)结合氧化还原酶 FAD/NAD(P)-binding oxidoreductase | |
| Glyma.05G208900 | K15398 | GO:0055114 | 脂肪酸ω-羟化酶(CYP86A4S)Fatty acid omega-hydroxylase (CYP86A4S) | |
| Glyma.05G000700 | K00873 | GO:0030955 | 丙酮酸激酶家族蛋白Pyruvate kinase family protein | |
| Glyma.06G211300 | K10781 | GO:0006633 | 脂肪酰基-ACP硫酯酶B Fatty acyl-ACP thioesterases B | |
| Glyma.17G219100 | 磷脂酶D1 Phospholipase D1 | |||
| Glyma.17G228400 | K13076 | GO:0006629 | 脂肪酸去饱和酶Fatty acid desaturase |
新窗口打开|下载CSV
3 讨论
本研究进行基因精细定位所利用的染色体片段代换系(CSSLs)群体,已成功应用在包括水稻、玉米等作物中[46,47,48]。CIM和ICIM是QTL定位的重要方法,广泛应用于大豆性状的QTL定位中,CIM能够克服区间作图法连锁背景的影响;而ICIM能够解决QTL算法上的缺陷,充分利用所有标记,本研究同时运用了这两种QTL定位方法,以期能获得较为准确的QTL[19,20,21]。对2个群体的5脂肪酸的相关性分析结果:棕榈酸与硬脂酸、亚油酸、亚麻酸呈显著正相关,而油酸与其余4种组分均呈显著负相关;与于福宽[49]研究结果相同,说明5种脂肪酸之间的相关性稳定,可以在以后的大豆品质改良研究中作为参考。本研究于2017—2018年在2个群体中共定位到54个QTL,与目前报道的脂肪酸相关的QTL结果比较,发现2个群体可能共有5个QTL区间与前人结果相近,其中,在D1a连锁群上定位到的区间与盛英华等[30]相近,但表型解释率较低,为6.95%;在D2连锁群上定位到的区间与盛英华等[30]、朱明月[50]的定位结果相近,这个区间存在2个与油酸相关的QTL,其中qOA-D2-2的表型解释率较高,为9.4%,可能是一个稳定的QTL区间且与油酸相关;在E连锁群上定位到的区间与DIERS等[51]的相同,且位置相近可能是同一个QTL,其中qPA-E-2和qPA-E-3的表型解释率较高,分别为13.5%和9.4%,可能是一个稳定的QTL,并且该区间与多种脂肪酸相关;在L连锁群上定位的区间与HYTEN等[52]的相近,但表型解释率较低,为7.26%;在I连锁群上定位到的区间在FAN等[29]定位的区间内,推测有多种脂肪酸相关的QTL分布在这一区间内。本研究定位到的QTL从贡献率上来说,大部分是一些微效的QTL,可能是由于双亲与后代群体的脂肪酸各组分和油分的变异系数较小,最大变异系数仅为6.99%(表1),微效基因较多与双亲和后代群体的表型差异较小有关。除了部分QTL与已有的定位结果相近,同时发现了一些新的QTL区间,并且2个群体中共有10对QTL被多次重复检测到,表现稳定;同时还有部分的QTL区间存在重叠,并与多种脂肪酸组分相关,可能是由于一个基因可以影响生物多种性状的表型,也可能是由于脂肪酸各组分QTL之间的紧密连锁[30];但能在2个群体重复检测到的QTL较少,仅有1对。总之,本研究检测到的部分QTL与前人定位的QTL区间一致,进一步验证了本研究的可靠性;另一方面能在不同年份、不同群体中被重复检测到的QTL区间和新发现的区间均是与大豆脂肪酸含量相关的重要区段,具有更高的利用价值。
进一步对所定位到的这些区间进行候选基因的挖掘及富集分析,一共有15个候选基因与脂肪酸相关,这些候选基因主要分布在第2、3、5、6和17染色体上,其中,基因Glyma.06G211300被许多****研究报道[36],编码植物酰基-酰基载体蛋白(ACP)硫酯酶,说明该基因对脂肪酸的形成非常重要(GO:0006633)[36];Glyma.05G000700是丙酮酸激酶家族蛋白成员,其家族成员已在甘蓝和拟南芥中被报道与脂肪酸代谢相关[53];Glyma.02G074000与酰基辅酶a/酰基转移酶有关,辅酶A已经被多次报道参与饱和脂肪酸的生物合成[36-37,53]。此外,本研究在其他QTL的置信区间内发现许多未知功能基因,可能存在与大豆脂肪酸相关的基因,有待进一步探索。
4 结论
2017—2018年在RILs群体和CSSLs群体中分别定位到34和20个与脂肪酸含量相关的QTL。其中,有10对QTL在不同年份的2个群体中被重复检测到,在RILs群体重复检测到6对,在CSSLs群体中重复检测到4对,并且与多种脂肪酸相关。检测到15个候选基因与脂肪酸相关。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s00122-002-1086-yURL [本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s11746-000-0036-2URL [本文引用: 1]
DOI:10.1007/BF02674138URL [本文引用: 1]
DOI:10.1021/ac60146a047URL [本文引用: 1]
DOI:10.2134/agronj1997.00021962008900040022xURL [本文引用: 1]
DOI:10.1007/BF02660596URL [本文引用: 1]
DOI:10.1016/S0021-9673(01)88338-2URL [本文引用: 1]
DOI:10.1016/S0022-2275(20)38027-5URL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.1534/genetics.106.066811URL [本文引用: 1]
DOI:10.1093/genetics/135.1.205URL [本文引用: 2]
[本文引用: 2]
DOI:10.1093/genetics/134.4.1277URL [本文引用: 3]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
DOI:10.2307/2531212URL [本文引用: 1]
DOI:10.4236/ajps.2014.51021URL [本文引用: 1]
DOI:10.1111/pbr.2018.137.issue-2URL [本文引用: 1]
[本文引用: 2]
DOI:10.1111/pbr.2017.136.issue-3URL [本文引用: 1]
[本文引用: 2]
[本文引用: 4]
[本文引用: 4]
[本文引用: 1]
DOI:10.1371/journal.pone.0104871URL [本文引用: 1]
DOI:10.1371/journal.pone.0149380URL [本文引用: 1]
[本文引用: 1]
DOI:10.3389/fpls.2018.01280URL [本文引用: 1]
DOI:10.1111/pbr.v138.6URL [本文引用: 5]
DOI:10.1046/j.1365-313X.2003.016010.xURL [本文引用: 2]
DOI:10.1007/s00122-016-2725-zURL [本文引用: 1]
DOI:10.1074/jbc.272.28.17354URL [本文引用: 1]
[本文引用: 1]
DOI:10.1002/(ISSN)1097-0010URL [本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
DOI:10.1105/tpc.106.048629URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s10681-018-2319-8URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[本文引用: 1]
DOI:10.1007/s11746-004-1027-zURL [本文引用: 1]
[本文引用: 2]
[本文引用: 2]
