 ,1,2, 吴建祥3, 李小宇2, 张春雨2, 侯吉超1, 周雪平3,4, 王永志
,1,2, 吴建祥3, 李小宇2, 张春雨2, 侯吉超1, 周雪平3,4, 王永志 ,2,4
,2,4Mapping of Epitopes and Establishment of Rapid DAS-ELISA for Potato Virus Y Coat Protein
LIANG YuXin ,1,2, WU JianXiang3, LI XiaoYu2, ZHANG ChunYu2, HOU JiChao1, ZHOU XuePing3,4, WANG YongZhi
,1,2, WU JianXiang3, LI XiaoYu2, ZHANG ChunYu2, HOU JiChao1, ZHOU XuePing3,4, WANG YongZhi ,2,4
,2,4通讯作者:
责任编辑: 岳梅
收稿日期:2020-06-11接受日期:2020-08-14网络出版日期:2021-03-16
| 基金资助: |
Received:2020-06-11Accepted:2020-08-14Online:2021-03-16
作者简介 About authors
梁雨欣,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (500KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
梁雨欣, 吴建祥, 李小宇, 张春雨, 侯吉超, 周雪平, 王永志. 马铃薯Y病毒衣壳蛋白抗原表位分析及其快速ELISA检测方法的建立[J]. 中国农业科学, 2021, 54(6): 1154-1162 doi:10.3864/j.issn.0578-1752.2021.06.007
LIANG YuXin, WU JianXiang, LI XiaoYu, ZHANG ChunYu, HOU JiChao, ZHOU XuePing, WANG YongZhi.
开放科学(资源服务)标识码(OSID):

0 引言
【研究意义】马铃薯是世界第四大粮食作物,2015年中国提出马铃薯主粮化发展战略,目前中国是马铃薯第一大生产国[1,2]。已知能侵染马铃薯的病毒至少有53种,我国已发现10余种[3]。其中,马铃薯Y病毒(potato virus Y,PVY)是侵染范围最广也是造成经济损失最严重的病毒之一[4,5]。PVY于1931年在马铃薯中首次被发现[6],目前在世界各地均有发生[7,8,9,10,11]。在自然条件下PVY可通过蚜虫以非持久性方式传播,同时也可以通过汁液摩擦、嫁接等机械方式传播。PVY单独侵染马铃薯可引起叶面黄化褪绿、皱缩、叶脉坏死等症状,且由于带毒薯块自身无法清除病毒,病毒会逐代传播积累最终导致马铃薯种质退化,若与其他病毒复合侵染可造成马铃薯高达80%的减产[12,13,14,15]。由于尚未发现有效的病毒病防治药剂,高质量的脱毒种薯成为病毒病的重要防治措施,而快速、高效的病毒检测技术是种薯质量控制的关键。【前人研究进展】目前常用的PVY检测方法主要有生物学检测法、电子显微镜检测法、分子生物学检测法和血清学检测法[16]。生物学检测法和电子显微镜检测法都需要培养或制备合适的观察对象,耗时较长且结果判定较为主观,并不适用于快速检测。以高灵敏度为优点的RT-PCR是近年来常用于PVY检测的分子生物学检测法,而CP基因以其高保守性成为RT-PCR扩增的首选基因,其表达的CP蛋白亦是血清学检测法检测的首选蛋白。李玉琦等[17]针对PVY-CP保守区设计一对特异性引物,建立了可以快速、准确地检测马铃薯带病种薯和植株中PVY含量的RT-PCR检测方法。虽然RT-PCR灵敏度高、特异性强,但在实际应用时对RNA的提取、cDNA的反转录要求较高,不适用于高通量样品检测。ELISA(enzyme-linked immunosorbent assay)是目前最常用的血清学检测方法之一,具有高通量、不依赖特定仪器设备,操作简单稳定性好等优点。早在1981年,张鹤龄等[18]利用提取的PVY病毒免疫家兔,制备了PVY多克隆抗体,并检测了烟草中的PVY,然而可检测的感病汁液最高稀释度仅为1﹕100;1993年,SINGH等[19]制备了多株PVY抗体,并以特异性最佳的单克隆抗体为基础建立了dot-ELISA,对马铃薯叶片中PVYN进行检测,最高稀释度为1﹕640;2008年,高方方等[20]通过PVYN株系分离物CP重组蛋白免疫新西兰大白兔制备了PVY兔血清,建立了PVY间接ELISA并对烟草中的PVY进行检测,最高稀释度为1﹕128;2016年,宋革等[21]制备了4株PVY单克隆抗体,以其为核心建立了ACP-ELISA、dot-ELISA、tissue blot-ELISA和IC-RT-PCR 4种血清学检测方法,灵敏度极高但检测步骤繁琐。【本研究切入点】前期虽有部分研究者制备了PVY特异性抗体,但甚少有应用方面的报道。而已建立的血清学检测方法,由于灵敏度较低,试验流程较为繁琐且耗时较长,无法满足快速高通量的检测需求。【拟解决的关键问题】通过PVY-CP的分段表达多肽筛选到一组能进行夹心ELISA的配对单克隆抗体(9G6和3D3),并以这对单抗为基础建立PVY快速DAS-ELISA检测方法。通过优化DAS-ELISA工作条件,将样品和检测抗体同时加入酶标板共同孵育,简化操作步骤并缩短试验时间,从而提高检测效率,为PVY的快速检测、诊断及脱毒种薯的生产提供关键技术。1 材料与方法
试验于2019年在吉林省农业科学院植物保护研究所微生物重点实验室完成。1.1 材料与试剂
PVY病毒、大肠杆菌Rosetta感受态细胞为吉林省农业科学院植物保护研究所微生物实验室保存,PVY单克隆抗体3D3、9G6为吉林省农业科学院植物保护研究所微生物实验室制备,PVY单克隆抗体3E4为浙江大学制备;限制内切酶EcoR I、Xho I,In-Fusion? HD Cloning Kit均购自TaKaRa公司;AxyPrep DNA凝胶回收试剂盒购自爱思进生物技术有限公司;RNeasy Plant Mini Kit购自QIAGEN公司;ReverAid First Strand cDNA Synthesis Kit购自赛默飞世尔科技公司;双组分TMB显色液购自北京索莱宝科技有限公司;脱脂奶粉、辣根过氧化物酶(horseradish peroxidase,HRP)均购自生工生物工程(上海)股份有限公司;HRP标记兔抗小鼠IgG购自Sigma公司。1.2 仪器与设备
洗板机、酶标仪购自美国伯腾仪器有限公司;高速冷冻台式离心机购自日本日立公司;恒温培养振荡器购自上海智城分析仪器制造有限公司。1.3 PVY-CP蛋白抗原表位分析
将PVY-CP基因分段表达为6条多肽:PVY-CP1-180、PVY-CP90-281、PVY-CP1-59、PVY-CP31-89、PVY-CP90-148和PVY-CP120-180,设计特异性引物(表1),进行PCR扩增,EcoR I和Xho I双酶切pGEX-6p-1载体,同时对目的基因PCR扩增产物及酶切载体大片段进行胶回收,通过In-Fusion试剂盒将二者连接,构建重组质粒并转化大肠杆菌Rosetta感受态细胞,表达PVY-CP1-180、PVY-CP90-281、PVY-CP1-59、PVY-CP31-89、PVY-CP90-148和PVY-CP120-180 6条多肽。以表达的多肽进行SDS-PAGE电泳,恒压转膜,MAb(杂交瘤细胞培养上清)为一抗,HRP标记的兔抗小鼠IgG为二抗,通过Western blot分析MAb所识别的抗原表位。Table 1
表1
表1PVY-CP蛋白分段表达引物
Table 1
| 片段 Segment | 引物 Primer | 引物序列Primer sequence (5′-3′) |
|---|---|---|
| PVY-CP1-180 | PVY-AF | GGGATCCCCGGAATTCATGGGAAATGACACAATCGATG |
| PVY-AR | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGCGGTTCCTTTTTGTTGCGC | |
| PVY-CP90-281 | PVY-BF | GGGATCCCCGGAATTCCAGTTTGATACGTGGTATGAAGC |
| PVY-BR | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGTGCGGCCGCAAGCTTGTCGA | |
| PVY-CP1-59 | PVY-CF | GGGATCCCCGGAATTCATGGGAAATGACACAATCGATG |
| PVY-CR | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGCTTAGGCATCCTCATTTTGGAC | |
| PVY-CP31-89 | PVY-DF | GGGATCCCCGGAATTCAAGGAAAAGGACGTGAATGTTGG |
| PVY-DR | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGTGATTGAGTTGCTCGAGTATTTG | |
| PVY-CP90-148 | PVY-EF | GGGATCCCCGGAATTCCAGTTTGATACGTGGTATGAAGC |
| PVY-ER | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGTGGTTTCAGTGGGTATTCGAC | |
| PVY-CP120-180 | PVY-GF | GGGATCCCCGGAATTCTGCATTGAAAATGGAACCTCG |
| PVY-GR | GATGCGGCCGCTCGAGTTAGTGGTGGTGGTGGTGGTGCGGTTCCTTTTTGTTGCGC |
新窗口打开|下载CSV
1.4 MAb HRP标记
采用简易过碘酸钠法对MAb进行HRP标记,详细步骤参照文献[22]。1.5 快速ELISA检测方法的建立
捕获抗体100 μL/孔,包被酶标板;加入5%脱脂奶280 μL/孔,37℃封闭30 min;将待测样品按1﹕10(W/V,g·mL-1)比例加入0.01 mol·L-1 PBS缓冲液研磨后离心取上清,以上清为溶剂稀释检测抗体,将混合液加入酶标板,100 μL/孔,37℃静置孵育;以上每一步结束后TBST洗板3次;TMB显色液100 μL/孔,避光显色5—10 min后加入2 mol·L-1 H2SO4溶液50 μL/孔,终止反应;在450 nm波长读取OD值。以P(待测抗原OD450值)/N(阴性抗原OD450值)≥2.1判定检测结果为阳性。1.6 快速ELISA工作条件的优化
分别以健康马铃薯、感染PVY马铃薯的叶片研磨上清液为阴性和阳性对照,运用常规DAS-ELISA程序进行3株PVY MAb的配对,以P/N最大值对应的抗体组合为工作抗体。采用方阵法确定抗体工作浓度;通过控制变量法分别对捕获抗体包被条件、待测样品与检测抗体混合液孵育时间进行优化,以P/N最大值确定捕获抗体包被条件及待测样品和检测抗体共同孵育时间。采集感染PVY的马铃薯叶片,0.01 mol·L-1 PBS缓冲液为研磨液充分研磨,将叶片按质量体积比从1﹕10(1 g=10 mL)倍比稀释至1﹕5 120,12 000 r/min离心10 min,收集上清液,运用优化后的检测方法对其进行检测,以稀释倍数为横坐标,P/N值为纵坐标,得出叶片最佳检测范围的稀释倍数。
1.7 检测灵敏度分析
以0.01 mol·L-1 PBS缓冲液为稀释液将PVY-CP蛋白从10 000 ng·mL-1稀释至0.05 ng·mL-1,运用优化后的检测方法对其进行检测,每个浓度3个重复,同时设置阴性对照,以蛋白浓度为横坐标,P/N值为纵坐标,绘制灵敏度曲线[23]。1.8 特异性分析
利用优化后的PVY快速DAS-ELISA对PVY马铃薯薯块及叶片、马铃薯S病毒(potato virus S,PVS)、马铃薯M病毒(potato virus M,PVM)、马铃薯卷叶病毒(potato leaf-roll virus,PLRV)叶片进行检测,每份样品3个重复,根据P/N值≥2.1为阳性检验检测方法的特异性。1.9 符合率检测
对吉林省农业科学院试验地随机采集的50份疑似感染PVY的马铃薯样品,运用建立的快速DAS-ELISA及RT-PCR分别进行检测,比较检测结果,对检测方法的符合率进行检验。按照RNeasy Plant Mini Kit试剂盒提取马铃薯叶片的总RNA,反转录试剂盒合成cDNA第一链。根据PVY检疫鉴定方法国家标准(GB/T 36816—2018)合成用于扩增PVY的RT-PCR简并引物,PVY-a1-F420:5′-CGATACAAG ACTGATGYCCAGAT-3′;PVY-a1-R1200:5′-TAYTGT TGRGCACAGGTRGGG-3′。PCR条件:94℃预变性4 min;94℃变性30 s,62℃退火45 s,72℃延伸1 min,共35个循环;最后72℃再延伸3 min。PCR产物经5%琼脂糖凝胶电泳,并送往吉林省库美科技有限公司进行测序验证。1.10 不同株系马铃薯样品检测
运用建立的快速DAS-ELISA对实验室保存的不同株系PVY(PVYN与PVYO)感染的马铃薯样品进行检测。2 结果
2.1 MAb识别抗原表位分析
如图1所示,共表达了6条多肽,首先将PVY-CP蛋白分段表达为有重叠部分的两条肽段:PVY-CP1-180与PVY-CP90-281,通过Western blot分析MAb 3D3、3E4、9G6识别的抗原表位,结果如表2所示,3D3对两条肽段均有识别,即3D3识别的抗原表位位于两条肽段的重叠部分:PVY-CP90-180,同理可知9G6识别的抗原表位亦位于PVY-CP90-180。而3E4仅识别PVY-CP1-180,不识别PVY-CP90-281,即3E4识别的抗原表位位于PVY-CP1-90。由此得知,3株MAb识别的抗原表位均集中在PVY-CP1-180。进一步分析抗原表位,继续将PVY-CP1-180分段表达为PVY-CP1-59、PVY- CP 31-89、PVY-CP90-148、PVY-CP120-180 4条多肽,仍通过Western blot分析MAb 3D3、3E4、9G6识别的抗原表位,结果如表2,最终发现3D3识别的抗原表位位于PVY-CP120-148,3E4识别的抗原表位位于PVY- CP31-59,9G6识别的抗原表位位于PVY-CP90-119,3株MAb均识别不同抗原表位。图1
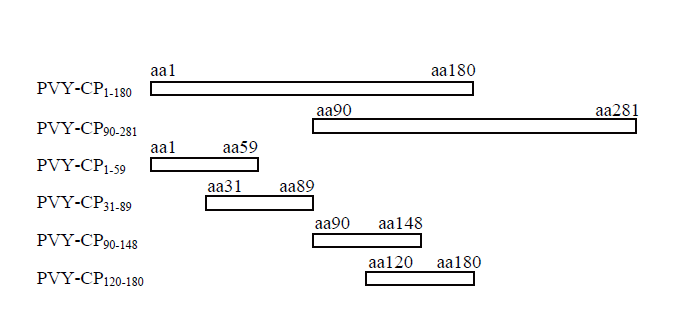 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1PVY-CP蛋白分段表达示意图
Fig. 1Schematic diagram of the segmented expression of PVY-CP protein
Table 2
表2
表23株MAb识别抗原表位分析
Table 2
| 分段表达蛋白 Segmented expression protein | 单克隆抗体Monoclonal antibody | ||
|---|---|---|---|
| 3D3 | 9G6 | 3E4 | |
| PVY-CP1-180 | + | + | + |
| PVY-CP90-281 | + | + | - |
| PVY-CP1-59 | - | - | + |
| PVY-CP31-89 | - | - | + |
| PVY-CP90-148 | + | + | - |
| PVY-CP120-180 | + | - | - |
新窗口打开|下载CSV
2.2 PVY快速DAS-ELISA检测方法的建立及优化
以常规DAS-ELISA检测方法确定捕获抗体为9G6,检测抗体为HRP-3D3。采用方阵法对抗体工作浓度进行优化,当捕获抗体浓度为4 μg·mL-1,检测抗体浓度为2 μg·mL-1时,P/N值最大(表3),检测效果最好,故确定其为抗体工作浓度。由P/N值最大可确定捕获抗体最佳包被条件为4℃过夜(图2-A),包被封闭好的酶标板即可用于PVY检测。图2
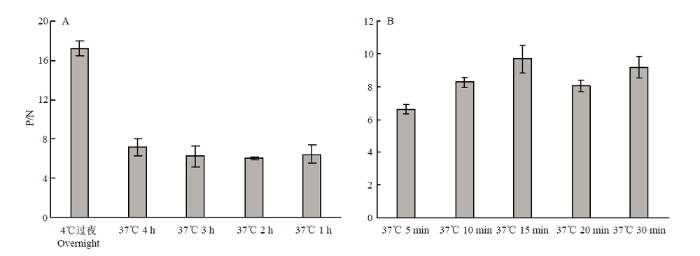 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2快速DAS-ELISA检测条件的优化
A:捕获抗体包被条件对检测结果的影响
B:待测样品与检测抗体混合液孵育条件对检测结果的影响
Fig. 2Optimization of rapid DAS-ELISA detection conditions
The effect of capture antibody incubating condition on the rapid DAS-ELISA;
The effect of incubation condition of mixture of sample and the detection antibody on the rapid DAS-ELISA
Table 3
表3
表3抗体最佳工作浓度确定
Table 3
| 检测抗体浓度 Concentration of detection antibody (μg·mL-1) | 捕获抗体浓度Concentration of capture antibody (μg·mL-1) | |||
|---|---|---|---|---|
| 4 | 2 | 1 | 0.5 | |
| 2 | 12.72±0.95* | 7.56±0.93 | 5.10±0.53 | 4.05±0.13 |
| 1 | 7.44±0.77 | 4.42±0.07 | 2.82±0.12 | 2.46±0.01 |
| 0.5 | 5.17±0.36 | 3.31±0.04 | 2.34±0.19 | 2.03±0.07 |
| 0.25 | 2.87±0.25 | 2.14±0.01 | 1.81±0.18 | 1.59±0.15 |
新窗口打开|下载CSV
检测步骤只需将待测样品与检测抗体混合液100 μL/孔加入酶标板于37℃进行孵育,孵育完成洗涤后加入TMB显色液,显色5—10 min后加入2 mol·L-1 H2SO4终止反应,在450 nm波长读取OD值。如图2-B,37℃孵育5 min时已可对样品进行定性判断,孵育15 min时检测效果可达到最佳。当P/N值≥2.1判定阳性,1.5≤P/N值<2.1判定为疑似阳性,需进一步检测,P/N值<1.5判为阴性结果。全部检测过程可以在30 min内完成。
利用优化后的快速DAS-ELISA对PVY马铃薯叶片进行检测,结果显示,当叶片稀释倍数在10—1 280倍时,P/N值≥2.1,检测结果呈阳性(图3-A),确定该检测方法对PVY马铃薯叶片的检测范围为10—1 280倍稀释。
图3
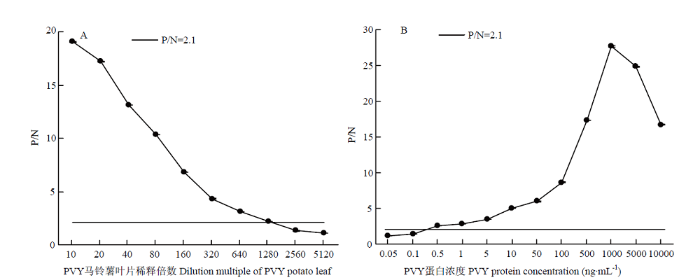 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3快速DAS-ELISA灵敏度曲线
A:PVY马铃薯叶片检测范围
B:PVY蛋白灵敏度曲线
Fig. 3Sensitivity curve of rapid DAS-ELSIA
Detection range of PVY potato leaf;
Sensitivity curve of PVY protein
2.3 检测灵敏度分析
运用建立的快速DAS-ELISA,以PVY-CP蛋白浓度为横坐标,P/N值为纵坐标,建立标准曲线(图3-B),当蛋白浓度为0.5 ng·mL-1时P/N>2.1,检测结果呈阳性;当蛋白浓度为0.1 ng·mL-1时,P/N值<2.1,检测结果呈阴性,故该检测方法对PVY-CP蛋白的检测限为0.5 ng·mL-1。2.4 特异性分析
如图4所示,建立的PVY快速DAS-ELISA检测PVY马铃薯薯块及叶片结果均呈阳性,对PVS、PLRV、PVM马铃薯叶片进行检测,检测结果呈阴性,表明检测方法特异性良好。图4
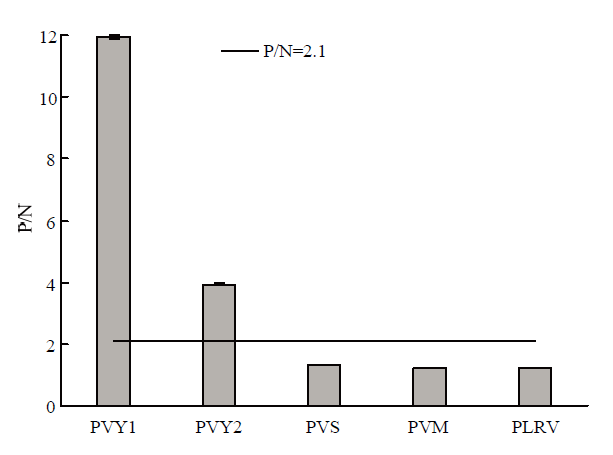 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4快速DAS-ELISA特异性分析
PVY1:感染PVY的马铃薯叶片Potato leaves infected with PVY;PVY2:感染PVY的马铃薯种薯Potato seeds infected with PVY;PVS、PLRV、PVM:分别为感染PVS、PLRV、PVM的马铃薯叶片Potato leaves infected with PVS, PLRV, PVM, respectively
Fig. 4Specificity analysis of rapid DAS-ELISA
2.5 符合率检测
为了对建立的快速DAS-ELISA符合率进行检验,运用快速DAS-ELISA对田间随机采集的50份疑似感染PVY的马铃薯样品进行检测。结果显示,有33份马铃薯样品检出PVY,17份样品未检出。同时,将上述所有样品进行RT-PCR检测,结果表明快速DAS-ELISA检测阳性样品均检测到PVY特异性PCR产物,而在17份DAS-ELISA检测阴性样品中,有2份样品RT-PCR检测结果为阳性,推测是样品中PVY的含量低于本检测方法的最低检测限。本研究建立的快速DAS-ELISA与RT-PCR共有48份样品检测结果一致,符合率达96%。2.6 马铃薯样品检测
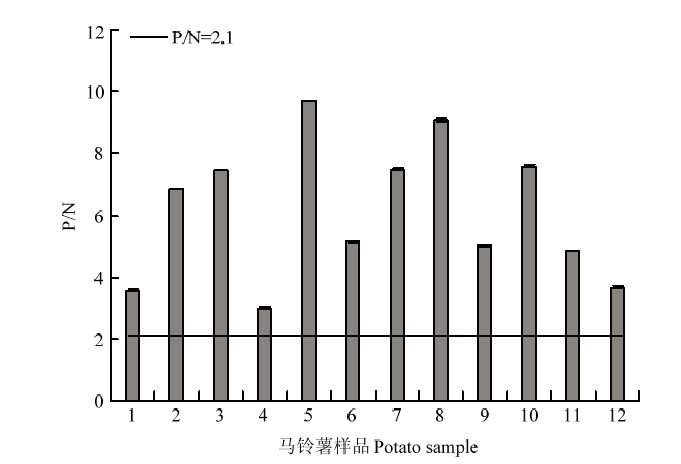
如图5所示,建立的快速DAS-ELISA检测方法对PVYN与PVYO马铃薯样品检测结果均为阳性,可满足田间发生最为广泛的株系检测需求。图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5快速DAS-ELISA检测不同株系马铃薯样品
1—6:PVYN马铃薯样品PVYN potato samples;7—12: PVYO马铃薯样品PVYO potato samples
Fig. 5Detection of different strains of PVY by rapid DAS-ELISA
3 讨论
PVY所在的马铃薯Y病毒属(Potyvirus)是最大的植物病毒属之一,该属基因组包含一个大的开放阅读框,表达时先编码一个多聚蛋白,随后通过自身编码的蛋白酶切割为至少10个功能不同的成熟蛋白[24,25],其中高度保守的CP蛋白在病毒侵染过程中起重要作用,且含有多个抗原决定簇,决定着病毒的抗原性[26,27]。本研究建立快速DAS-ELISA的基础是捕获抗体与检测抗体识别不同的抗原表位,从而避免抗体识别的表位有重叠甚至重合导致抗体竞争性结合抗原,减弱检测信号、降低检测方法灵敏度。目前常用的病毒抗原表位研究方法主要有预测法、抗原肽合成法、噬菌体展示法、核磁共振分析法及表面等离子共振技术等[28],本研究采用重叠多肽合成法将PVY-CP蛋白分段表达为有部分重叠的小段蛋白,通过抗原抗体特异性结合的原理对抗体识别的抗原表位进行鉴定。在本研究中使用的3株MAb,虽然皆识别不同抗原表位,但由于抗体本身的亲和力对抗体的配对效果也有很大影响,故配对效果有一定差异。本研究通过分段表达PVY-CP蛋白,对其抗原表位进行分析,将MAb识别的抗原表位确定在30个氨基酸,筛选出了9G6与3D3两株识别不同抗原表位且配对检测效果最佳的MAb,以此为基础建立了PVY快速DAS-ELISA。病毒病的发生和流行是病毒、宿主、环境等因素共同作用导致的结果。病毒病流行中存在的暴发性、间歇性和迁移性与病毒生态系统中各种生物和非生物因素密切相关,也由于具有“三性”的特点,病毒病的发生变得颇为复杂,使得病毒病的流行预测困难重重[29,30]。因此,高效快速、准确的检测方法对于有效预防和控制病毒病至关重要。DAS- ELISA作为血清学检测技术的代表性检测方法已广泛应用于各类检测,但受抗体本身的特性影响,DAS-ELISA的检测步骤较为固定且检测时间也较长。BIOREBA出产的DAS-ELISA PVY检测试剂盒需至少2 d [31],秦艳红建立的PVY DAS-ELISA为确保试验效果检测时间至少需要5 h[32]。本研究建立的PVY快速DAS-ELISA,经抗原表位分析后确定的捕获抗体与检测抗体识别不同的抗原表位,不存在抗原表位竞争现象,直接以待测样品研磨液为溶剂稀释酶标检测抗体至工作浓度,将抗原抗体混合液直接加入包被封闭好的酶标板孵育5 min,显色5—10 min即可终止,较常规DAS-ELISA简化了试验步骤,缩短了检测时间,30 min即可完成定性检测,检测效率显著提升。
PVY株系分化严重,可分为非重组株系和重组株系。在非重组株系中,发生较为频繁的是PVYN和PVYO株系,而在重组株系中,发生较为频繁的是PVYN×PVYO类型重组株系[33]。在PVY的研究过程中,虽然不断有新的重组株系与非重组株系出现,但在田间实际调查发现,PVYN×PVYO类型重组株系是田间主流株系,占据着非常高的比例[34]。鉴于此,本研究使用建立的快速DAS-ELLSIA检测方法,对实验室保存的PVYN和PVYO的马铃薯样品进行了检测,结果显示两株系样品检测结果均为阳性,表明本研究所建立的检测方法尽管以识别单一位点的单抗为基础,但仍可满足田间生产上的检测需求。TIAN等[35]对3株商品化PVY单抗识别的抗原表位进行分析后发现3株商品化单抗表位保守性相对较好,可检测多种PVY株系,这也证明了单克隆抗体可以用于马铃薯种质监测,满足生产上的检测需求。
4 结论
以高度保守的PVY-CP蛋白作为检测对象,建立了PVY快速DAS-ELISA,该检测方法检测限为0.5 ng·mL-1,仅与感染PVY的马铃薯样品反应呈阳性结果,灵敏度高、特异性强。30 min即可完成检测,操作简便、快捷,检测效率高,有待进一步开发为试剂盒,为PVY的防控提供技术及产品支持。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:30743397 [本文引用: 1]
DOI:10.1007/s00705-007-1059-1URL [本文引用: 1]

Potato virus Y (PVY) strain groups are based on host response and resistance gene interactions. The strain groups PVYO, PVYC and PVYN are well established for the isolates infecting potato in the field. A switch in the emphasis from host response to nucleotide sequence differences in the virus genomes, detection of isolates recombining sequences of different strains, and the need to recognize isolates that cause necrotic symptoms in potato tubers have led to the assignment of new acronyms, especially to isolates of the PVYN strain group. This discussion paper proposes that any newly found isolates should be described within the context of the original strain groups based on the original methods of distinguishing strains (i.e., tobacco and potato assays involving use of ‘differential’ potato cultivars). Additionally, sequence characterization of the complete genomes of isolates is highly recommended. However, it is acceptable to amend the names of PVY isolates with additional, specific codes to show that the isolate differs at the molecular, serological or phenotypic level from the typical strains within a strain group. The new isolates should preferably not be named using geographical, cultivar, or place-association designations. Since many new variants of PVY are being discovered, any new static classification system will be meaningless for the time being. A more systematic investigation and characterization of PVY from potato at the biological and molecular levels should eventually result in a biologically meaningful genetic strain concept.
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:30831903 [本文引用: 1]
DOI:10.1007/s00705-011-1182-xURLPMID:22139355 [本文引用: 1]

Isolates of potato virus Y (PVY) have been divided into several strains. We determined the genomic sequences of PVY isolates AQ4 and FZ10 from tobacco in China. AQ4 and FZ10 had genome of 9700 and 9698 nucleotides, respectively. In phylogenetic analysis of complete genome sequences, AQ4 was clustered with strain N-Wi, and FZ10 with NTN. AQ4 had two recombination sites within the P1 and P3 genes, while FZ10 had three within the P1, P3 and NIa-Pro genes. When compared to typical NTN isolates, FZ10 lacked a recombination site within the CP gene and, thus, represents a novel recombination type of PVY.
URLPMID:30812355 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URLPMID:20438367 [本文引用: 1]
[本文引用: 1]
DOI:10.1126/sciadv.aau4202URLPMID:31392261 [本文引用: 1]

Signaling through the receptor tyrosine kinase RET is essential during normal development. Both gain- and loss-of-function mutations are involved in a variety of diseases, yet the molecular details of receptor activation have remained elusive. We have reconstituted the complete extracellular region of the RET signaling complex together with Neurturin (NRTN) and GFRalpha2 and determined its structure at 5.7-A resolution by cryo-EM. The proteins form an assembly through RET-GFRalpha2 and RET-NRTN interfaces. Two key interaction points required for RET extracellular domain binding were observed: (i) the calcium-binding site in RET that contacts GFRalpha2 domain 3 and (ii) the RET cysteine-rich domain interaction with NRTN. The structure highlights the importance of the RET cysteine-rich domain and allows proposition of a model to explain how complex formation leads to RET receptor dimerization and its activation. This provides a framework for targeting RET activity and for further exploration of mechanisms underlying neurological diseases.
URLPMID:14963126 [本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
[D].
[本文引用: 1]
DOI:10.1111/mpp.2013.14.issue-5URL [本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
DOI:10.1371/journal.pone.0115766URLPMID:25542005 [本文引用: 1]

BACKGROUND: Potato virus Y (PVY, genus Potyvirus) causes substantial economic losses in solanaceous plants. Routine screening for PVY is an essential part of seed potato certification, and serological assays are often used. The commercial, commonly used monoclonal antibodies, MAb1128, MAb1129, and MAb1130, recognize the viral coat protein (CP) of PVY and distinguish PVYN strains from PVYO and PVYC strains, or detect all PVY strains, respectively. However, the minimal epitopes recognized by these antibodies have not been identified. METHODOLOGY/PRINCIPAL FINDINGS: SPOT peptide array was used to map the epitopes in CP recognized by MAb1128, MAb1129, and MAb1130. Then alanine replacement as well as N- and C-terminal deletion analysis of the identified peptide epitopes was done to determine critical amino acids for antibody recognition and the respective minimal epitopes. The epitopes of all antibodies were located within the 30 N-terminal-most residues. The minimal epitope of MAb1128 was 25NLNKEK30. Replacement of 25N or 27N with alanine weakened the recognition by MAb1128, and replacement of 26L, 29E, or 30K nearly precluded recognition. The minimal epitope for MAb1129 was 16RPEQGSIQSNP26 and the most critical residues for recognition were 22I and 23Q. The epitope of MAb1130 was defined by residues 5IDAGGS10. Mutation of residue 6D abrogated and mutation of 9G strongly reduced recognition of the peptide by MAb1130. Amino acid sequence alignment demonstrated that these epitopes are relatively conserved among PVY strains. Finally, recombinant CPs were produced to demonstrate that mutations in the variable positions of the epitope regions can affect detection with the MAbs. CONCLUSIONS/SIGNIFICANCE: The epitope data acquired can be compared with data on PVY CP-encoding sequences produced by laboratories worldwide and utilized to monitor how widely the new variants of PVY can be detected with current seed potato certification schemes or during the inspection of imported seed potatoes as conducted with these MAbs.
