 ,, 李宝聚
,, 李宝聚 ,中国农业科学院蔬菜花卉研究所,北京 100081
,中国农业科学院蔬菜花卉研究所,北京 100081Identification of the Pathogen Causing Cabbage Died in Gansu Province and Detection of Anastomosis Groups
WANG Duo, XIE XueWen, CHAI ALi, SHI YanXia ,, LI BaoJu
,, LI BaoJu ,Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081
,Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081通讯作者:
收稿日期:2019-04-4接受日期:2019-05-17网络出版日期:2019-08-16
| 基金资助: |
Received:2019-04-4Accepted:2019-05-17Online:2019-08-16
作者简介 About authors
王朵,E-mail:15713890570@163.com。

摘要
关键词:
Abstract
Keywords:
PDF (1769KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
王朵, 谢学文, 柴阿丽, 石延霞, 李宝聚. 甘肃省白菜死棵病病原菌鉴定及其融合群检测技术[J]. 中国农业科学, 2019, 52(16): 2787-2799 doi:10.3864/j.issn.0578-1752.2019.16.005
WANG Duo, XIE XueWen, CHAI ALi, SHI YanXia, LI BaoJu.
0 引言
【研究意义】由立枯丝核菌(Rhizoctonia solani)引起的十字花科蔬菜死棵病是一种重要的土传病害。甘肃武威、兰州等地,地势较低,气候冷凉,主要种植喜凉蔬菜如白菜,近年来由于土壤连作以及栽培管理不当等原因,死棵现象频发。由于立枯丝核菌寄主范围广和融合群(anastomosis groups,AGs)间关系的复杂性,在病害早期难以判断和准确量化[1,2],因此明确引起白菜死棵病的主要致病群体,以及建立一种能够准确、灵敏及快速检测田间样品的方法,对于该病害的有效防控具有重要意义。【前人研究进展】基于菌丝融合反应,立枯丝核菌可划分为多个AGs;根据致病力、寄主范围、形态变异和遗传特征等,一些AGs又可划分为多个融合亚群[3,4,5,6]。十字花科蔬菜丝核菌病害主要由AG-2-1引起,导致植株茎部溢缩,直至腐烂、倒伏,最终死亡,造成严重的经济损失[7,8,9];AG-1-IB常引起莴苣叶腐病,同时也对白菜等十字花科蔬菜有致病力[10,11]。前人对融合群的鉴定多基于菌丝融合反应,但2008年SHARON等[12]利用菌丝融合鉴定融合群时,发现该过程易受环境因素等影响,重复性较差,结果不可靠。目前有关融合群的分子生物学检测技术主要有基于SYBR Green I和TaqMan探针的实时荧光定量PCR(real-time PCR)技术,这两种方法均能特异性检测融合群,准确度好、灵敏度高,常用来对引起某种寄主病害的一个或多个融合群进行检测,如利用TaqMan探针检测马铃薯黑痣病菌AG-3[13]以及定量检测其菌核[14]、水稻纹枯病菌AG-1-IA[15]及毒素[16]和油菜根腐病菌AG-2-1[17],以及基于SYBR Green和TaqMan检测小麦根部病菌[18,19]等。【本研究切入点】目前尚未对甘肃省白菜死棵病原菌进行系统报道,笔者项目组于2015—2018年在甘肃地区采集疑似白菜死棵病株作为样本,进行病原菌融合群鉴定和检测技术的建立。【拟解决的关键问题】采用ITS和TEF通用引物对白菜死棵病菌进行PCR扩增和测序分析,明确其具体种类,并建立real-time PCR检测体系,实现田间植株和土壤样品的准确检测,为病害预警和提早防控提供技术支持。1 材料与方法
试验于2018年在中国农业科学院蔬菜花卉研究所完成。1.1 病样的采集与病原菌分离、保存
笔者课题组于2015—2018年共采集白菜死棵病样100份,品种为韩国介实金悦白菜。采用水琼脂分离法[20]进行病原菌快速分离鉴定,挑取菌丝边缘于PDA培养基上,25℃黑暗培养3 d后,记录菌落形态学特征,对病原菌进行显微形态观测、拍照,之后转移至灭菌麦粒上,待菌丝长满麦粒后保存于-20℃冰箱[21],共保存立枯丝核菌86株。1.2 主要试剂及仪器
植物基因组DNA提取试剂盒(天根生化科技有限公司)、胶回收试剂盒(TaKaRa Mini BEST Agarose GEL DNA Extraction Kit)、质粒提取试剂盒(TIANprep Mini Plasmid Kit,天根生化科技有限公司)、pMDTM 19-T Vector Cloning Kit(TaKaRa公司)、感受态细胞(Trans 5α Competent Cells,TaKaRa公司)、DNase/RNase free water(天根生化科技有限公司)、FastDNA Spin Kit for Soil(MP Bio-medical,USA)、实时荧光定量PCR仪(ABI 7500美国ABI公司)。1.3 病原菌的鉴定
1.3.1 病原菌的分子生物学鉴定 采用试剂盒法提取病原菌基因组DNA。采用ITS通用引物对[22]及立枯丝核菌翻译延伸因子引物对RSF/RSR[23]对各菌株进行PCR扩增(表1),反应结束后,取5 μL PCR产物用1.0%的琼脂糖凝胶电泳进行检测。将含有目的条带的PCR产物交由北京博迈德基因技术有限公司分别用各基因相对应的PCR扩增引物进行双向测序,将得到双向序列拼接,并将拼接后得到的序列提交到 GenBank(表2),然后用MEGA 7.0软件根据各菌株ITS和TEF-1α基因序列进行系统发育进化分析。采用最大似然法(maximum likelihood,ML)构建系统发育进化树。同时采用MATSUMOTO[24]根据立枯丝核菌不同融合群的核糖体DNA序列差异设计的两对特异性引物(表1)对分离菌株进行PCR特异性扩增,用于鉴定分离菌株。Table 1
表1
表1本研究中所用融合群鉴定的引物序列及其来源
Table 1
| 基因 Gene | 片段长度 Fragment length (bp) | 引物名称 Primer name | 方向 Direction | 序列 Sequence | 引物来源 Primer source |
|---|---|---|---|---|---|
| ITS | 700 | ITS1 | Forward | TCCGTAGGTGAACCTGCGG | 文献[22] Reference [22] |
| ITS4 | Reverse | TCCTCCGCTTATTGATATGC | |||
| TEF-1α | 680 | RSF | Forward | CGTGAYTTYATCAAGAACATG | 文献[23] Reference [23] |
| RSR | Reverse | GACTTGACTTCAGTGGTCAC | |||
| 28S | F-Common | Forward | CTCAAACAGGCATGCTC | 文献[24] Reference [24] | |
| 300 | R-AG-2-1 | Reverse | AGGCAATAGGTTATTGGACC | ||
| 250 | R-AG-1-IB | Reverse | AAGGTCCTTTGGGGTTGGGG |
新窗口打开|下载CSV
Table 2
表2
表2本研究中所用菌株及序列
Table 2
| 菌株 Strain | 登录号 GenBank accession number | |
|---|---|---|
| ITS | TEF-1α | |
| WWC18081412 | MK336671 | MK391084 |
| WWC18081413 | MK336672 | MK391085 |
| WWC18081623 | MK336664 | MK391077 |
| WWC18081509 | MK336665 | MK391078 |
| WWC18081604 | MK336666 | MK391079 |
| WWC18081506 | MK336696 | MK391109 |
| WWC18081510 | MK336673 | MK391086 |
| WWC18081514 | MK336674 | MK391087 |
| WWC18081527 | MK336675 | MK391088 |
| WWC18081528 | MK336684 | MK391097 |
| WWC18081606 | MK336685 | MK391098 |
| WWC18081618 | MK336686 | MK391099 |
| WWC18081619 | MK336687 | MK391100 |
| WWC18081622 | MK336668 | MK391081 |
| WWC18081625 | MK336667 | MK391080 |
| WWC18081636 | MK336669 | MK391082 |
| WWC18081638 | MK336670 | MK391083 |
| WWC18081640 | MK336676 | MK391089 |
| WWC18081641 | MK336677 | MK391090 |
| WWC18081649 | MK336678 | MK391091 |
| WWC18081651 | MK336679 | MK391092 |
| WWC18081513 | MK336688 | MK391101 |
| WWC18081515 | MK336689 | MK391102 |
| WWC18081653 | MK336690 | MK391103 |
| WWC18081656 | MK336691 | MK391104 |
| WWC1507261101 | MK336682 | MK391095 |
| WWC1507261102 | MK336694 | MK391107 |
| WWC1507261103 | MK336683 | MK391096 |
| WWC1507261104 | MK336695 | MK391108 |
| WWC1507261105 | MK336697 | MK391110 |
| WWC1608082901 | MK336680 | MK391093 |
| WWC1608082902 | MK336692 | MK391105 |
| WWC1608082903 | MK336693 | MK391106 |
| WWC1608082904 | MK336698 | MK391111 |
| WWC1608082905 | MK336681 | MK391094 |
| WWC1608082501 | MK336700 | MK391113 |
| WWC1608082502 | MK336701 | MK391114 |
| WWC1608082503 | MK336702 | MK391115 |
| WWC1608082504 | MK336703 | MK391116 |
| WWC1608082505 | MK336704 | MK391117 |
| WWC1608081601 | MK336705 | MK391118 |
| WWC1608081602 | MK336706 | MK391119 |
| WWC1608081603 | MK336707 | MK391120 |
| WWC1608081604 | MK336708 | MK391121 |
| WWC1608081605 | MK336709 | MK391122 |
| WWC1608090401 | MK336710 | MK391123 |
| WWC1608090402 | MK336711 | MK391124 |
| WWC1608090403 | MK336712 | MK391125 |
| WWC1608090404 | MK336713 | MK391126 |
| WWC1608090405 | MK336699 | MK391112 |
新窗口打开|下载CSV
1.3.2 分离菌株对不同白菜品种致病力测定 为比较不同融合群菌株的致病力差异,选取两个融合群代表菌株WWC18081640(AG-2-1)和WWC18081618(AG-1-IB),在日光温室中对20个白菜品种进行致病力测定。采用叶部喷雾和茎基部灌根法,用无菌纱布过滤菌悬液,将菌丝打碎并用无菌水稀释到一定浓度接种[25]。对照处理喷无菌水,每处理10株苗,重复3次,将各种处理的幼苗置于25℃恒温保湿柜中,每天观察并记录发病情况,参照分级标准[26],记录接种植株的病级数,并计算病情指数(DI)。
1.4 Real-time PCR引物的设计与合成
根据NCBI数据库中立枯丝核菌ITS基因的保守序列,运用Primer 5.0软件设计real-time PCR检测特异性引物,并通过NCBI中的Primer-BLAST进行比对,保证所设计引物的特异性。上游引物F-RS:CAATACAAAGGCAATAGG;下游引物R-RS:GTCCTCAGCGATAGATAA。引物由博迈德基因技术有限公司进行合成。1.5 质粒标准品的制备
以保存的DNA为模板进行PCR扩增,总反应体系为50 μL。PCR产物回收后参考丁天波等[27]的方法对回收产物进行连接、转化,并筛选出阳性质粒,计算质粒浓度拷贝数。1.6 反应条件的优化及标准曲线的建立
质粒标准品按10倍梯度进行稀释,获得终浓度为8.41×109—8.41×103 copies/μL的7个质粒样品作为模板进行检测。反应体系为20 μL:SYBR Green PremixPlus 10 μL,Rox Reference 0.4 μL,上下游引物各0.3 μL,模板1 μL,ddH2O补足。1.7 特异性检测
用优化后的real-time PCR反应条件分别对其他病原菌,如茄镰孢(Fusarium solani)、尖镰孢(F. oxysporum)、芸薹生链格孢(Alternaria brassicicola)、胶孢炭疽菌(Colletotrichum gloeosporioides)、瓜果腐霉(Pythium aphanidermatum)、终极腐霉(P. ultimum)、水稻纹枯病菌(R. solani AG-1-IA)、马铃薯黑痣病菌(R. solani AG-3)、小麦纹枯病菌(R. cerealis)等进行特异性检测。1.8 Real-time PCR的灵敏度检测
将质粒标准品按照10倍梯度稀释成8.41×109—8.41×103 copies/μL的7个样品,并以此作为模板分别进行常规PCR和real-time PCR检测,比较这两种方法的检测灵敏度。1.9 白菜病株及根际土壤中立枯丝核菌的检测
分别对1.3.2中接种后1、5、10、15、20 d的白菜病株及根际土壤取样,提取植物组织和土壤总DNA,通过real-time PCR检测立枯丝核菌(每个样品取3次重复,检测结果取平均值),根据扩增曲线、熔解曲线和Ct值确定其带菌情况。同时对田间采集的17份疑似白菜病株及0—10 cm深度根际土壤[19]进行总DNA提取,通过real-time PCR进行检测,根据扩增曲线、熔解曲线和Ct值确定其带菌情况。2 结果
2.1 序列测定和系统发育分析
采用ITS1/ITS4和RSF/RSR对50个分离菌株进行PCR扩增,均获得了约650 bp的序列(图1),将测序结果提交至GenBank,获得登录号(表2)。将获得的序列进行比较并构建系统发育树,可看出32条序列与立枯丝核菌AG-2-1的参考序列聚在同一分支,且亲缘关系置信度为100%,另外18条与AG-1-IB参考序列聚在另一分支,而外群菌株Athelia rolfsii则以较远的亲缘关系处在系统发育树的外围(图2)。序列测定和系统发育分析结果表明,共有32株菌(占64%)属于立枯丝核菌AG-2-1,另外18株(36%)为AG-1-IB。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1ITS和TEF-1α片段扩增产物电泳图谱
Fig. 1PCR amplification pattern of ITS and TEF-1α
M:DNA分子量标准(5 000 bp)DNA Marker (5 000 bp);1—24:分离菌株经ITS和TEF-1α通用引物扩增后的产物电泳条带 The amplification of R. solani fragment by universal primers of ITS and TEF-1α;25:阴性对照 Negative control
图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2基于rDNA-ITS 和TEF-1α核酸序列的系统发育树
Fig. 2Phylogenetic tree based on sequence of rDNA-ITS and TEF-1α regions
2.2 特异性引物的扩增结果
用F-Common/R-AG-2-1和F-Common/R-AG-1-IB特异性引物分别对代表菌株进行PCR扩增,可分别产生约300和250 bp的扩增片段(图3)。特异性扩增结果表明,该引物对可准确鉴定分离菌株为立枯丝核菌AG-2-1和AG-1-IB。图3
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3特异性引物F-Common/R-AG-2-1和F-Common/R-AG-1-IB对分离菌株的扩增产物电泳图
Fig. 3PCR products of R. solani strains collected from cabbage in Gansu Province using specific primer pairs of F-Common/ R-AG-2-1 and F-Common/R-AG-1-IB
M:DNA分子量标准(5 000 bp) DNA marker (5 000 bp);1—8:F-Common/R-AG-2-1特异性扩增 Amplification by primer F-Common/R-AG-2-1 for AG-2-1;9—17:立枯丝核菌融合群AG-3—AG-11菌株 R. solani AG3 to AG11;18:阴性对照 Negative control;19—23:F-Common/R-AG-1-IB特异性扩增 Amplification by primer F-Common/R-AG-1-IB for AG-1-IB
2.3 分离菌株对白菜品种的致病力测定
致病力试验结果表明,接种后两融合群菌株在20个白菜品种上均出现死棵症状,融合群AG-1-IB致病力稍强于AG-2-1,对叶片及茎基部均有致病力,而AG-2-1对金娃娃及小皇宝两个品种的白菜叶片不具有致病力。从病情指数来看,两融合群在不同白菜品种上存在差异(表3),茎基部病情指数≥50的品种,AG-2-1仅有3个,而AG-1-IB占9个,且均高于AG-2-1。Table 3
表3
表3WWC18081640和WWC18081618对20个白菜品种的致病力测定(病情指数)
Table 3
| 品种 Cultivar | AG-2-1 | AG-1-IB | ||
|---|---|---|---|---|
| 叶片 Leaf | 茎基部 Stem | 叶片 Leaf | 茎基部 Stem | |
| 贝贝皇 Beibeihuang | 30.4 | 45.0 | 39.2 | 61.7 |
| 炎童 Yantong | 11.9 | 36.7 | 54.2 | 63.3 |
| 黄孩 Huanghaier | 3.0 | 11.7 | 42.1 | 58.3 |
| 全新玉皇大帝 Quanxinyuhuangdadi | 2.2 | 6.7 | 33.1 | 65.0 |
| 瑞丽 Ruili | 0.7 | 3.3 | 52.1 | 60.0 |
| 纯皇 Chunhuang | 5.2 | 38.3 | 49.3 | 66.7 |
| 兴苑 Xingyuan | 12.6 | 36.7 | 37.1 | 46.7 |
| 黄金 Huangjin | 30.4 | 50.0 | 59.1 | 60.0 |
| 卓生黄金 Zhuoshenghuangjin | 23.7 | 41.7 | 48.0 | 35.0 |
| 金宝 Jinbao | 38.5 | 20.0 | 62.1 | 46.7 |
| 京春娃2号 Jingchunwa 2 | 0.7 | 6.7 | 55.1 | 53.3 |
| 沃德春娃 Wodechunwa | 3.0 | 50.0 | 63.1 | 66.7 |
| 金娃娃 Jinwawa | 0 | 8.3 | 15.0 | 21.7 |
| 金贝贝 Jinbeibei | 0.7 | 26.7 | 7.0 | 28.3 |
| 小皇宝 Xiaohuangbao | 0 | 5.0 | 6.0 | 25.0 |
| 绿亨 Lvheng | 17.8 | 51.7 | 2.0 | 5.0 |
| 金旺 Jinwang | 21.5 | 41.7 | 2.0 | 3.3 |
| 格莱美 Gelaimei | 18.5 | 36.7 | 4.0 | 13.3 |
| 芭比 Babi | 4.4 | 26.7 | 2.0 | 5.0 |
| 金童秀 Jintongxiu | 12.6 | 18.3 | 26.0 | 26.6 |
新窗口打开|下载CSV
2.4 rDNA-ITS基因片段的克隆与鉴定
以白菜死棵病组织提取总DNA为模板,运用F-RS/R-RS引物进行常规PCR扩增,其中实验室保存的立枯丝核菌DNA作为阳性对照,对照菌基因组DNA及健康白菜样品DNA作阴性对照,ddH2O作为空白对照。PCR扩增产物经1%的琼脂糖凝胶电泳,结果表明(图4),白菜病株样品出现特异单一条带,位置在389 bp,同阳性对照条带大小一致,而阴性对照和空白对照则无特异性条带出现。将目的条带切胶回收,经连接克隆载体至博迈德基因技术有限公司进行测序,将测序结果在NCBI数据库中进行BLASTn比对,结果证实所获得的序列为ITS基因片段。图4
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4常规PCR扩增目的片段
Fig. 4The amplification of R. solani fragment by conventional PCR
M:DNA分子量标准(5 000 bp) DNA marker (5 000 bp);1:阳性对照Positive control;2:感染立枯丝核菌的白菜样品R. solani-infected cabbage sample;3—21:立枯丝核菌融合群AG-3—AG-11菌株、水稻纹枯病菌、双核丝核菌AG-A、小麦纹枯病菌、茄镰孢、尖镰孢、胶孢炭疽菌、瓜果腐霉、终极腐霉、芸薹生链格孢菌和致病疫霉 R. solani AG-3 to AG-11, R. solani AG-1-IA, binulceate Rhizoctonia AG-A, R. cerealis, F. solani, F. oxysporum, C. gloeosporioides, P. aphanidermatum, P. ultimum, A. brassicicola, P. infestans;22:健康白菜样品 Healthy cabbage sample;23:空白对照 Blank control
2.5 标准曲线的建立
以10倍梯度进行稀释的7个质粒样品作为模板,进行real-time PCR,结果表明随着阳性质粒模板浓度逐渐降低,Ct值出现递增趋势,并且在质粒标准品浓度8.41×104—8.41×108 copies/μL范围内,Ct值同质粒标准品浓度之间线性关系良好,标准曲线斜率为-3.546,决定系数R2=0.9983,扩增效率为91%,直线方程为y=-3.546x+ 44.241(y代表循环阈值即 Ct 值,x代表质粒初始浓度)(图5)。该标准曲线将用于立枯丝核菌含量的测定。图5
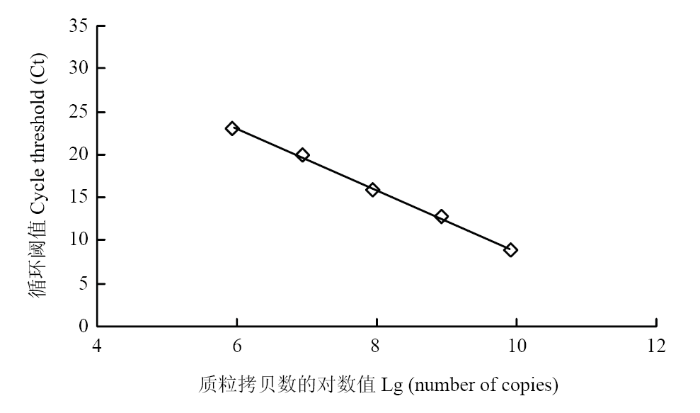 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5Real-time PCR 标准曲线图
Fig. 5The standard curve of real-time PCR for R. solani
2.6 质粒标准品灵敏度检测
Real-time PCR和常规PCR的灵敏度检测比对结果表明,real-time PCR的立枯丝核菌检测方法能够最低检测出8.41×103 copies/μL的样品(图6),而常规PCR仅能够检测到8.41×105 copies/μL的样品(图7),real-time PCR检测立枯丝核菌的灵敏度是常规PCR的100倍。图6
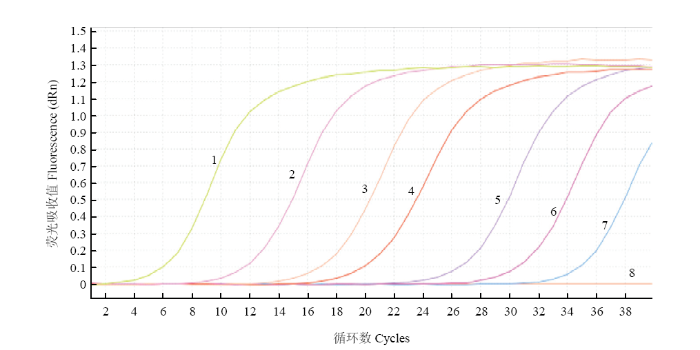 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6Real-time PCR的检测灵敏度
Fig. 6The detection sensitivity of R. solani by real-time PCR
1—7:8.41×109—8.41×103 copies/μL质粒标准品8.41×109-8.41×103 copies/μL standard plasmids;8:空白对照 Blank control
图7
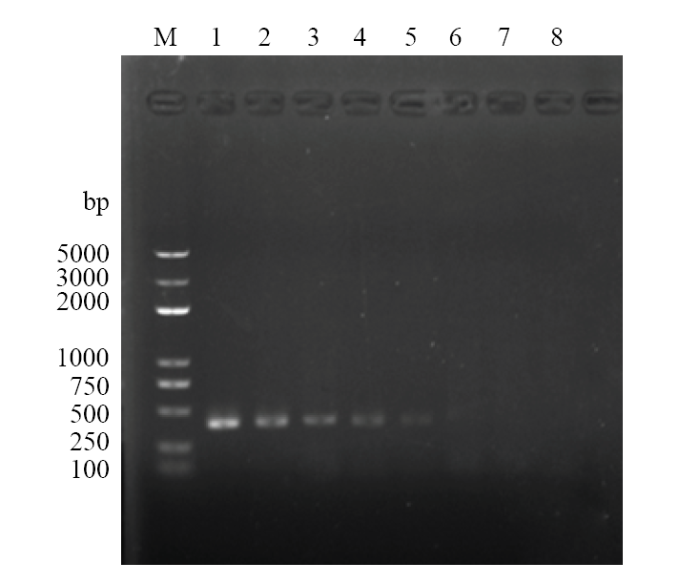 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7常规PCR的检测灵敏度
Fig. 7The detection sensitivity of R. solani by conventional PCR
M:DNA分子量标准(5 000 bp) DNA marker (5 000 bp);1—7:8.41×109—8.41×103 copies/μL质粒标准品8.41×109-8.41×103 copies/μL standard plasmids;8:空白对照 Blank control
2.7 白菜病株及根际土壤样品检测
利用real-time PCR检测技术对人工接种的白菜茎基部和根际土壤进行检测,结果表明人工接种白菜植株和土壤后第5天,立枯丝核菌在白菜植株体内和根际土壤中积累量最大(表4)。对田间采集病株及根际土壤进行real-time PCR检测,除了阴性对照和空白对照,其他样品均可检测到相应的荧光信号值(图8)。此外,对上述样品进行常规PCR检测,发现大多数样品均在相同位置检测到特异性单一条带(图9),但有1个样品(第16孔)未出现明显条带,因此常规PCR的检出率低于real- time PCR。Table 4
表4
表4Real-time PCR检测人工接种植株及根际土壤的带菌量
Table 4
| 接种后天数 Days after inoculation (d) | 平均Ct 值 The average cycle threshold value | DNA平均拷贝数的对数值 Logarithm of average copy number of DNA (lg copies/μL) | ||
|---|---|---|---|---|
| 茎基部 Stem | 根际土壤 Soil | 茎基部 Stem | 根际土壤 Soil | |
| 1 | 23.46 | 31.28 | 5.85 | 3.66 |
| 5 | 11.92 | 18.84 | 9.11 | 7.16 |
| 10 | 20.86 | 23.57 | 6.59 | 5.76 |
| 15 | 28.92 | 26.88 | 4.31 | 4.90 |
| 20 | 30.40 | 32.85 | 3.90 | 3.21 |
新窗口打开|下载CSV
图8
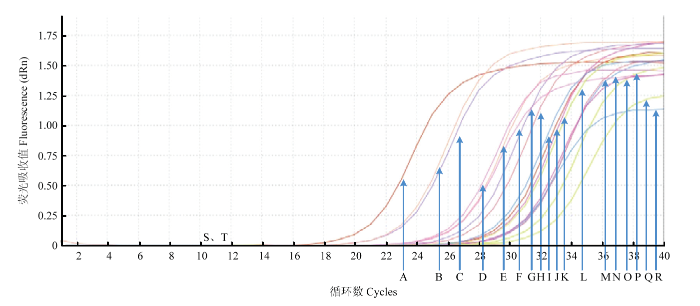 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图8田间样品的real-time PCR检测
Fig. 8The R. solani detection of field samples by real-time PCR
A:阳性对照 Positive control;B—Q:田间样品 Samples in field;S:阴性对照 Negative control;T:空白对照 Blank control
图9
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图9田间样品的常规PCR检测
Fig. 9The R. solani detection of field samples by conventional PCR
M:DNA分子量标准(5 000 bp) DNA marker(5 000 bp);1:阳性对照 Positive control;2—8:田间死棵病株 Died plants in fields;9—18:田间病株根际土壤 Soils on died plants;19:阴性对照Negative control;20:空白对照 Blank control
3 讨论
白菜在我国种植范围广,市场消费较大。甘肃以干燥、冷凉的气候特点,将白菜作为重要的蔬菜品种,其种植面积逐年增加。近年来由于栽培面积扩大,死棵问题不断出现。本研究发现引起白菜死棵的病原菌是立枯丝核菌。立枯丝核菌是一类在自然界中广泛存在的土壤习居真菌,能引起多种植物病害[28,29],根据菌丝融合、培养形态、致病力以及DNA同源性等,被划分为14个不同的AG以及一些融合亚群[30,31,32]。传统分类学基于形态特征、菌丝融合、同工酶、可溶性蛋白等生物学和生化的方法对融合群进行划分,耗时长、信息量少[33],目前越来越多****通过分子学方法进行融合群的分类,其中核糖体DNA(rDNA)内部转录间隔区(ITS)区域已被证明能够准确划分融合群及亚群,但该区域在进行融合群群内各菌株之间遗传差异分析时缺乏多态性[34]。TEF-1α是蛋白翻译及延伸中的重要蛋白质,该基因具有丰富的信息量,比ITS的多态性更高[35],其序列在不同物种中高度相似,已被用于多种病原菌的鉴定及系统发育研究,如GEISER等[36]利用此基因分析镰孢菌系统发育关系及ANEES等[37]用来研究木霉属真菌。本研究结合PCR反应、基于ITS和TEF-1α基因序列的系统发育分析及苗期品种抗病性鉴定,明确了从死棵白菜上分离到的病原菌主要为立枯丝核菌AG-2-1和AG-1-IB,此后还会继续进行病株采集和鉴定。近年来对立枯丝核菌检测的相关报道也逐渐增多,目前使用较为普遍的方法是real-time PCR,其中基于TaqMan探针的real-time PCR越来越多的应用于AG检测,如BUDGE等[38]检测到土壤中11个融合群,WOODHALL等[39]检测到引起马铃薯黑痣的4个融合群,但其检测成本均较高,而基于荧光染料SYBR Green的检测方法能够很好地弥补上述缺陷,并已广泛应用于多种融合群的检测和研究中。本研究选取ITS的保守区域设计出可同时检测AG-2-1和AG-1-IB 的real-time PCR特异性引物F-RS/R-RS,通过常规PCR扩增发现AG-2-1和AG-1-IB样品均在目的大小处条带单一,并且不能扩增出其他AG和对照菌,说明F-RS/R-RS特异性较强,能够满足后续验证试验;real-time PCR检测结果发现F-RS/R-RS扩增曲线良好,有特异性熔解峰,结合本研究摸索的反应体系和条件,能够最大程度保证反应过程中目标基因的有效扩增,增强反应特异性。本研究选用纯化的重组质粒DNA作为标准品,与普通试剂盒提取的DNA相比,具有稳定易保存等特点[40]。以立枯丝核菌重组质粒DNA作为标准品构建的标准曲线,质粒标准品中立枯丝核菌拷贝数的对数值同Ct值之间呈现良好的线性关系,相关系数达0.9983,扩增效率为91%,完全满足样品中立枯丝核菌含量测定的需求。通过比较常规PCR和 real-time PCR的检测灵敏度,发现real-time PCR的检测灵敏度是常规PCR的100倍,因此对田间低丰度样品的准确有效检测成为现实。本研究对人工接种的白菜、根际土壤以及田间采集的死棵病株和土壤进行检测,通过分析扩增曲线、熔解曲线并与阳性、阴性和空白对照进行对比,均检测到了病原菌,证实了该方法的检测适用性。对人工接种的不同天数白菜植株和根际土壤样品进行检测,发现带菌量均在接种后第5天最大,此时植株的发病程度也最大,但土壤带菌量均低于茎基部带菌量,很可能是在土壤DNA提取过程中造成了部分损失[41],需要进一步筛选出更好的提取土壤DNA方法。
4 结论
甘肃省白菜主产区死棵病的病原菌是立枯丝核菌AG-2-1和AG-1-IB。基于SYBR Green I建立的real-time PCR 检测方法特异性强、灵敏度高,能够满足针对田间植物及根际土壤中立枯丝核菌的检测和含菌量测定,可为田间白菜死棵病的预测预报提供技术支持。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[D].
[本文引用: 2]
[D].
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2018.10.020Magsci [本文引用: 1]

<p><b>目的</b> 建立一种实时荧光定量PCR(real-time fluorescent quantitative polymerase chain reaction,RT-qPCR)检测番茄褪绿病毒(<italic>Tomato chlorosis virus</italic>,ToCV)的方法,用于测定田间植株样品及其传毒媒介烟粉虱(<italic>Bemesia tabaci</italic>)中ToCV的含量。<b>方法</b> 首先根据ToCV次要外壳蛋白(minor coat protein,CPm)基因的保守区域序列,运用Primer 3.0在线软件设计ToCV特异性检测引物ToCVqF和ToCVqR,并通过Primer-BLAST进行比对保证检测引物的特异性;其次分别以感染ToCV、番茄黄化曲叶病毒(<italic>Tomato yellow leaf curl virus</italic>, TYLCV)、番茄斑萎病毒(<italic>Tomato spotted wilt virus</italic>, TSWV)的番茄样品为模板,应用该对引物进行ToCV基因片段的克隆鉴定;将阳性克隆样品放入含有氨苄青霉素的LB液体培养基中扩大培养,并提取质粒,获得ToCV质粒标准品,将ToCV质粒标准品按10倍浓度梯度稀释,建立标准曲线;同样以按10倍浓度梯度稀释的ToCV质粒标准品为模板,同时运用RT-qPCR和反转录PCR(reverse transcription-polymerase chain reaction,RT-PCR),比较这两种方法检测ToCV的灵敏度;最后应用该方法对田间采集的8份疑似ToCV感染的番茄样品以及获毒24 h后的单头烟粉虱进行病毒检测,结合扩增曲线、熔解曲线和Ct值对待测样品中的带毒情况和带毒量进行测定。<b>结果</b> 在感染ToCV的番茄样品和阳性对照中,引物ToCVqF和ToCVqR均能够特异扩增出ToCV CPm基因片段,目的片段大小为230 bp,而在感染TYLCV、TSWV的番茄样品、健康番茄样品和空白对照中,该对引物则无法扩增出特异性目的条带,说明该对引物特异性较强;构建ToCV的RT-qPCR标准曲线结果表明,随着ToCV质粒标准品模板浓度逐渐降低,Ct值出现递增趋势,并且在质粒标准品浓度2.7×10<sup>3</sup>—2.7×10<sup>9</sup> copies/μL范围内,ToCV标准曲线Ct值同质粒标准品浓度之间呈现良好的线性关系,扩增效率为100%,决定系数<italic>R<sup>2</sup></italic>=0.9911,直线方程为<italic>y</italic>= -3.32×lg<italic>x</italic>+40.06(<italic>y</italic>代表循环阈值即Ct值,<italic>x</italic>代表质粒初始浓度),该曲线将用于ToCV含量的测定。RT-qPCR灵敏度检测结果表明,该方法能够检测出的ToCV质粒标准品最低浓度为2.7×10<sup>3</sup> copies/μL,而常规RT-PCR仅能够检测出2.7×10<sup>5</sup> copies/μL的病毒样品,通过RT-qPCR法检测ToCV比常规RT-PCR法灵敏100倍。该方法能成功检测出田间植物样品携带ToCV情况,8份疑似ToCV感染的番茄样品中有6份携带ToCV,同常规RT-PCR检测结果一致,结合标准曲线计算出感染ToCV的番茄样品带毒量分别为2.48×10<sup>5</sup>、2.21×10<sup>5</sup>、7.97×10<sup>4</sup>、3.74×10<sup>7</sup>、3.37×10<sup>7</sup>、2.78×10<sup>6</sup> copies/μL;烟粉虱获毒24 h后,携毒率为100%,单头烟粉虱最高带毒量为2.55×10<sup>4</sup> copies/μL,最低为6.46×10<sup>2</sup> copies/μL。<b>结论</b> 建立的RT-qPCR检测ToCV的方法特异性强、灵敏度高,可以实现植物样品和媒介昆虫携带ToCV情况和病毒含量的检测,可为ToCV的准确、有效监测和预警提供技术支持。</p>
DOI:10.3864/j.issn.0578-1752.2018.10.020Magsci [本文引用: 1]

<p><b>目的</b> 建立一种实时荧光定量PCR(real-time fluorescent quantitative polymerase chain reaction,RT-qPCR)检测番茄褪绿病毒(<italic>Tomato chlorosis virus</italic>,ToCV)的方法,用于测定田间植株样品及其传毒媒介烟粉虱(<italic>Bemesia tabaci</italic>)中ToCV的含量。<b>方法</b> 首先根据ToCV次要外壳蛋白(minor coat protein,CPm)基因的保守区域序列,运用Primer 3.0在线软件设计ToCV特异性检测引物ToCVqF和ToCVqR,并通过Primer-BLAST进行比对保证检测引物的特异性;其次分别以感染ToCV、番茄黄化曲叶病毒(<italic>Tomato yellow leaf curl virus</italic>, TYLCV)、番茄斑萎病毒(<italic>Tomato spotted wilt virus</italic>, TSWV)的番茄样品为模板,应用该对引物进行ToCV基因片段的克隆鉴定;将阳性克隆样品放入含有氨苄青霉素的LB液体培养基中扩大培养,并提取质粒,获得ToCV质粒标准品,将ToCV质粒标准品按10倍浓度梯度稀释,建立标准曲线;同样以按10倍浓度梯度稀释的ToCV质粒标准品为模板,同时运用RT-qPCR和反转录PCR(reverse transcription-polymerase chain reaction,RT-PCR),比较这两种方法检测ToCV的灵敏度;最后应用该方法对田间采集的8份疑似ToCV感染的番茄样品以及获毒24 h后的单头烟粉虱进行病毒检测,结合扩增曲线、熔解曲线和Ct值对待测样品中的带毒情况和带毒量进行测定。<b>结果</b> 在感染ToCV的番茄样品和阳性对照中,引物ToCVqF和ToCVqR均能够特异扩增出ToCV CPm基因片段,目的片段大小为230 bp,而在感染TYLCV、TSWV的番茄样品、健康番茄样品和空白对照中,该对引物则无法扩增出特异性目的条带,说明该对引物特异性较强;构建ToCV的RT-qPCR标准曲线结果表明,随着ToCV质粒标准品模板浓度逐渐降低,Ct值出现递增趋势,并且在质粒标准品浓度2.7×10<sup>3</sup>—2.7×10<sup>9</sup> copies/μL范围内,ToCV标准曲线Ct值同质粒标准品浓度之间呈现良好的线性关系,扩增效率为100%,决定系数<italic>R<sup>2</sup></italic>=0.9911,直线方程为<italic>y</italic>= -3.32×lg<italic>x</italic>+40.06(<italic>y</italic>代表循环阈值即Ct值,<italic>x</italic>代表质粒初始浓度),该曲线将用于ToCV含量的测定。RT-qPCR灵敏度检测结果表明,该方法能够检测出的ToCV质粒标准品最低浓度为2.7×10<sup>3</sup> copies/μL,而常规RT-PCR仅能够检测出2.7×10<sup>5</sup> copies/μL的病毒样品,通过RT-qPCR法检测ToCV比常规RT-PCR法灵敏100倍。该方法能成功检测出田间植物样品携带ToCV情况,8份疑似ToCV感染的番茄样品中有6份携带ToCV,同常规RT-PCR检测结果一致,结合标准曲线计算出感染ToCV的番茄样品带毒量分别为2.48×10<sup>5</sup>、2.21×10<sup>5</sup>、7.97×10<sup>4</sup>、3.74×10<sup>7</sup>、3.37×10<sup>7</sup>、2.78×10<sup>6</sup> copies/μL;烟粉虱获毒24 h后,携毒率为100%,单头烟粉虱最高带毒量为2.55×10<sup>4</sup> copies/μL,最低为6.46×10<sup>2</sup> copies/μL。<b>结论</b> 建立的RT-qPCR检测ToCV的方法特异性强、灵敏度高,可以实现植物样品和媒介昆虫携带ToCV情况和病毒含量的检测,可为ToCV的准确、有效监测和预警提供技术支持。</p>
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
Magsci [本文引用: 1]

水稻纹枯病是一种世界性病害,目前该病已成为限制水稻高产、稳产的主要障碍之一。本文简述了引起水稻纹枯病的病原菌立枯丝核菌的形态特征,概述了依据菌丝形态特征和融合群进行分类的概况,阐述了同工酶、RAPD、ISSR和AFLP等分子生物学技术在水稻纹枯病菌遗传多样性研究中的应用。 <BR>
Magsci [本文引用: 1]

水稻纹枯病是一种世界性病害,目前该病已成为限制水稻高产、稳产的主要障碍之一。本文简述了引起水稻纹枯病的病原菌立枯丝核菌的形态特征,概述了依据菌丝形态特征和融合群进行分类的概况,阐述了同工酶、RAPD、ISSR和AFLP等分子生物学技术在水稻纹枯病菌遗传多样性研究中的应用。 <BR>
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
Magsci [本文引用: 1]

菠萝凋萎相关病毒(<em>Pineapple mealybug wilt associated virus-2</em>,PMWaV-2)是引起的菠萝凋萎病(Mealybug wilt of pineapple,MWP)的重要病原。本研究中根据PMWaV-2外壳蛋白基因序列设计特异性引物和探针,建立基于TaqMan探针的实时荧光定量RT-PCR检测方法。该方法能高灵敏检测出阳性样品,对阴性样品及空白对照均无荧光反应;灵敏度比普通PCR高100倍;重复性试验表明批内和批间变异系数均在1.85%以内,表明本方法是一种操作简便、特异性强、灵敏度高、重复性较好的PMWaV-2定量检测方法。
Magsci [本文引用: 1]

菠萝凋萎相关病毒(<em>Pineapple mealybug wilt associated virus-2</em>,PMWaV-2)是引起的菠萝凋萎病(Mealybug wilt of pineapple,MWP)的重要病原。本研究中根据PMWaV-2外壳蛋白基因序列设计特异性引物和探针,建立基于TaqMan探针的实时荧光定量RT-PCR检测方法。该方法能高灵敏检测出阳性样品,对阴性样品及空白对照均无荧光反应;灵敏度比普通PCR高100倍;重复性试验表明批内和批间变异系数均在1.85%以内,表明本方法是一种操作简便、特异性强、灵敏度高、重复性较好的PMWaV-2定量检测方法。
DOI:10.3864/j.issn.0578-1752.2015.01.06Magsci [本文引用: 1]

【目的】纹枯病是小麦生产上重要的土传病害之一,早期准确定量检测病原是病害预测预报及实现有效防控的基础。传统组织分离鉴定方法费时、程序复杂,而且无法做到准确定量。为实现小麦纹枯病早期快速准确定量检测,研究旨在建立小麦纹枯病菌(<em>Rhizoctonia cerealis</em>)的SYBR Green I实时荧光定量PCR(real-time PCR)检测方法。【方法】根据小麦纹枯病菌的<em>β</em><em>-tubilin</em>序列,设计1对特异性引物,建立并优化SYBR Green I real-time PCR反应体系,利用小麦纹枯病菌、立枯丝核菌(<em>R</em>.<em> solani</em>)、双核丝核菌AG-A和AG-F菌株、<span>小麦全蚀病菌(</span><em>Gaeumannomyces graminis</em> var.<em> tritici</em>)<em>、</em>小麦根腐病菌(<em>Bipolaris sorokiniana</em>)、小麦茎基腐病菌假禾谷镰孢(<em>Fusarium pseudograminearum</em>)和禾谷镰孢(<em>F</em>. <em>graminearum</em>)等8种小麦根部或土壤病原物的菌丝DNA进行普通PCR和real-time PCR特异性检测,并对灵敏度、可重复性进行评价。利用该体系分别检测接种后5、10、60 d不同接种浓度的盆栽小麦病株。【结果】研究设计的引物特异性强,普通PCR检测结果仅<span>小麦纹枯病菌</span>DNA有扩增条带。Real-time检测结果表明该对引物对<span>小麦纹枯病菌只有唯一的产物吸收峰,对其余供试菌株均未检测到荧光信号。</span>普通PCR检测的灵敏度为6.5×10<sup>3 </sup>copies/μL, real-time PCR的灵敏度可达到6.5×10<sup>2 </sup>copies/μL。以携带目的基因片段的重组质粒为标准品,构建的real-time PCR标准曲线循环阈值与模板浓度呈良好的线性关系,熔解曲线的吸收峰单一,相关系数为0.997,扩增效率为0.91。对人工接种的盆栽小麦病株进行real-time PCR检测,结果表明与病情指数和接种量分别呈显著正相关。【结论】研究建立的基于real-time PCR技术的小麦纹枯病菌快速检测方法速度快、灵敏度高、特异性强、重复性好。可以用于小麦纹枯病菌检测、指导病害预测和防治。
DOI:10.3864/j.issn.0578-1752.2015.01.06Magsci [本文引用: 1]

【目的】纹枯病是小麦生产上重要的土传病害之一,早期准确定量检测病原是病害预测预报及实现有效防控的基础。传统组织分离鉴定方法费时、程序复杂,而且无法做到准确定量。为实现小麦纹枯病早期快速准确定量检测,研究旨在建立小麦纹枯病菌(<em>Rhizoctonia cerealis</em>)的SYBR Green I实时荧光定量PCR(real-time PCR)检测方法。【方法】根据小麦纹枯病菌的<em>β</em><em>-tubilin</em>序列,设计1对特异性引物,建立并优化SYBR Green I real-time PCR反应体系,利用小麦纹枯病菌、立枯丝核菌(<em>R</em>.<em> solani</em>)、双核丝核菌AG-A和AG-F菌株、<span>小麦全蚀病菌(</span><em>Gaeumannomyces graminis</em> var.<em> tritici</em>)<em>、</em>小麦根腐病菌(<em>Bipolaris sorokiniana</em>)、小麦茎基腐病菌假禾谷镰孢(<em>Fusarium pseudograminearum</em>)和禾谷镰孢(<em>F</em>. <em>graminearum</em>)等8种小麦根部或土壤病原物的菌丝DNA进行普通PCR和real-time PCR特异性检测,并对灵敏度、可重复性进行评价。利用该体系分别检测接种后5、10、60 d不同接种浓度的盆栽小麦病株。【结果】研究设计的引物特异性强,普通PCR检测结果仅<span>小麦纹枯病菌</span>DNA有扩增条带。Real-time检测结果表明该对引物对<span>小麦纹枯病菌只有唯一的产物吸收峰,对其余供试菌株均未检测到荧光信号。</span>普通PCR检测的灵敏度为6.5×10<sup>3 </sup>copies/μL, real-time PCR的灵敏度可达到6.5×10<sup>2 </sup>copies/μL。以携带目的基因片段的重组质粒为标准品,构建的real-time PCR标准曲线循环阈值与模板浓度呈良好的线性关系,熔解曲线的吸收峰单一,相关系数为0.997,扩增效率为0.91。对人工接种的盆栽小麦病株进行real-time PCR检测,结果表明与病情指数和接种量分别呈显著正相关。【结论】研究建立的基于real-time PCR技术的小麦纹枯病菌快速检测方法速度快、灵敏度高、特异性强、重复性好。可以用于小麦纹枯病菌检测、指导病害预测和防治。
