
Genome-wide Association Analysis of Some Phytotoxicity Related Traits at Seedling Stage in Rapeseed under Glufosinate Stress
CHENDong-Liang
通讯作者:
收稿日期:2016-12-15
接受日期:2017-11-21
网络出版日期:2018-01-19
版权声明:2018作物学报编辑部作物学报编辑部
基金资助:
展开
摘要
关键词:
Abstract
Keywords:
-->0
PDF (3439KB)元数据多维度评价相关文章收藏文章
本文引用格式导出EndNoteRisBibtex收藏本文-->
全球农田广泛分布的各种杂草危害着农业生产, 严重影响作物的产量、品质和经济效益。油菜是世界重要的经济作物之一, 随着对其经济价值和食用价值的不断开发, 油菜的种植面积逐年上升, 随之而来的草害问题也越来越突出, 化学除草是解决这一问题的主要的手段。由于草甘膦(glyphosate)抗性杂草数量的逐渐增多和抗性的不断增强, 草甘膦市场日渐低迷。百草枯(paraquat)药害作用严重, 也于2016年7月1日正式被禁用[1]。草铵膦这一具有杀草谱广、快速、高效、低毒等特点的触杀型除草剂取而代之的可能性愈发凸显。草铵膦是世界上广泛使用的除草剂, 是L-谷氨酸的衍生物, 强烈抑制植物谷氨酰胺合成酶(GS)的活性[2,3]。植物通过叶片或其他绿色部分快速吸收草铵膦, GS活性立即受到抑制, 继而导致细胞内氨的含量迅速积累。而氨的积累则直接抑制光系统和光系统反应的正常有序进行, 随之叶绿体结构解体, 最后引起整个植物体死亡[4]。鲁军雄[5]从油菜单株鲜重、株高、单株叶面积、净光合速率、SPAD值、全株角果数、每角粒数、千粒重等方面, 研究了草铵膦对抗草铵膦转基因杂交油菜生长、产量及产量构成、品质的影响, 认为油菜单株叶面积对除草剂最敏感, 其余依次为株高、SPAD值、单株鲜重、净光合速率, 因此单株叶面积比平均株高能更好地评价作物对除草剂的耐受性。信晓阳等[6]认为叶片药害指数、叶片数减少率、叶夹角减少率与植株死亡率间均存在显著或极显著相关, 说明这3个指标可作为早期鉴定油菜对苯磺隆耐药性的指标。全基因组关联分析(genome-wide association studies, GWAS)[7], 可以在全基因组水平上对复杂性状的遗传变异进行关联分析[8], 对于剖析复杂的代谢途径是有效的。吴金峰[9]基于SSR和SNP标记对油菜的耐旱性进行了全基因组关联分析。针对杂草除草剂抗性研究近年来相继报道, Murcray等[10]采用随机扩增多态性(RAPD)揭示感抗三嗪型除草剂的龙葵(Solanum nigrum)的遗传差异与系统发育的关系。Cha等[11]采用RAPD标记剖析牛筋草(Eleusine indica L. Gaertn)的草甘膦抗性, 并发现了2个与抗性相关的遗传位点。王园园[12]对202份半野生棉材料进行莽草酸含量和地上部分相对干重2个草甘膦抗性相关表型的测定, 结合覆盖棉花全基因组的182对SSR多态性标记对202份半野生棉的草甘膦抗药性进行全基因组关联分析, 共检测到72个与草甘膦抗性相关的位点, 并找到了87个与草甘膦灭生机制相关的候选基因。但是, 对于油菜在草铵膦逆境胁迫下的生理、生化及形态变化进行全基因组关联分析的报道较少。虽然草铵膦的灭生机制已明确, 但作物对草铵膦的抗性机制也在不断被发现, 多机制共同作用已经成为普遍的抗性机制。油菜属于常异花授粉作物, 基因组内存在丰富的SNP变异位点。本研究以具有不同遗传背景和广泛地理来源的506份油菜种质为研究群体, 在适宜浓度的草铵膦胁迫下, 结合覆盖油菜全基因组的SNP变异位点分别对胁迫后的单位叶面积干重耐除草剂系数(CLW)、叶绿素含量耐除草剂系数(CCC)和综合药害指数(CPC) 3个耐药性性状进行全基因组关联分析, 扫描与对应性状相关位点, 并进行候选基因预测, 从生物信息学和基因组学层面探究油菜在草铵膦逆境胁迫下的生理形态反应的基因调控机制, 为油菜的草铵膦耐药性研究提供参考。
1 材料与方法
1.1 材料与药剂
材料包括国内外油菜主产区的506份具有代表性的油菜种质资源[13,14]。其中, 国内材料475份, 主要来自重庆、湖北、湖南、陕西、江苏等地, 国外材料31份, 主要来自德国、加拿大、丹麦、瑞典等国。所有材料均由重庆市油菜工程技术研究中心提供。处理药剂为20%永农百速顿牌草铵膦水剂, 由浙江永农生物科学有限公司生产。
1.2 田间试验
于2015年9月上旬, 随机选取100份油菜材料播于西南大学歇马实验基地(29º45'39.99"N, 106º22'38.47"E, 海拔238.57 m), 进行预实验。随机区组设计, 穴播, 每个品系一行, 每行10窝, 每窝8~10粒, 行距40 cm, 窝距20 cm, 待3~5叶期分别用100 mg L-1和200 mg L-1草铵膦定向喷雾, 施药浓度均为有效成分含量, 每公顷施药量600 kg (下同)。以每个浓度梯度处理3窝, 对照不作处理。30 d后调查各浓度处理下油菜苗期的形态变化, 根据叶片形态变化初步确定不同株系在不同浓度处理下对草铵膦的药效表现。根据初选结果, 分别取敏感、中等敏感和耐性的20份品种(系), 将其播于直径25 cm, 深17 cm的花盆中, 每盆定苗5株。3次重复, 适时适量补充水分, 待三至五叶期分别用100、200、400和800 mg L-1的草铵膦喷施, 对照不作处理。30 d后调查药害恢复程度, 统计存活率。将极少数材料苗期表现出一定程度药害, 但后期恢复, 跟对照无差别; 极少数材料苗期死亡; 大部分材料苗期表现出一定药害, 后期恢复但与对照相比差别较大的喷施浓度作为适宜的草铵膦胁迫浓度。2015年9月下旬将506份群体材料播于西南大学实验温室。采取完全随机设计, 起垄, 垄长约100 cm, 宽约40 cm, 高约15 cm, 垄边距约15 cm, 每个品系一垄, 每垄80~100粒, 定苗每垄约60株。苗期不进行人工除草, 田间管理按照常规进行。待3~5叶期用已确定的适宜筛选浓度的草铵膦定向喷雾, 对照不做处理。
1.3 性状调查
参考油菜耐湿性所采用的方法[15]和药害程度目测分级的方法[16], 仅在已选定的适宜胁迫浓度下, 用叶绿素含量系数、单位叶面积干重系数[17]和综合药害指数[6]来评价各品系在草铵膦胁迫下的生理形态变化。采用Microsoft Excel 2007软件初步整理表型数据, 后采用SPSS 17.0软件统计分析。1.3.1 单位叶面积干重系数 施药后15 d, 于各小区对角5点取样10株, 选取叶龄一致的20个叶片, 用直径3 cm的打孔器取样, 杀青, 烘干称重。
单位叶面积干重系数(%) = 处理组单位叶面积干重/对照组单位叶面积干重×100[15,17]。
1.3.2 叶绿素含量系数 于施药后第7天, 利用SPAD-502叶绿素测定仪分别测定对照组和处理组的叶绿素含量。各小区5点对角选取10株, 测3次, 每次选取叶龄一致的30个叶片, 测定30个数据, 3次测量求取平均值。
叶绿素含量系数(%)=处理组叶绿素含量/对照组叶绿素含量×100 [15,17]。
1.3.3 综合药害指数 以各品系处理小区内的幼苗为评估对象, 于施药后第15天, 按照药害程度由小到大划分6级。0级: 叶片平展, 呈绿色; 1级: 心叶下卷, 第2叶平展, 两叶呈绿色; 2级: 心叶、第2叶均下卷, 两叶程均匀黄绿色; 3级: 心叶、第2叶均下卷, 两叶呈较深黄绿色; 4级: 心叶、第2叶均下卷, 第2叶边缘枯干; 5级: 心叶边缘枯干, 下卷, 第2叶完全枯干; 6级: 心叶, 第2叶均枯干, 组织器官坏死。统计处理总苗数和各受害级别苗数, 计算综合药害指数。
综合药害指数(%) = Σ[(每垄各受害级别株数×级别)/(每垄株数×最高级别)]×100 [6,16]。
1.4 基因型测定与分析
利用芸薹属60k Illumina Infinium SNP芯片, 严格按照试验操作说明对506份甘蓝型油菜群体材料进行SNP基因型分析[18]。采用Genome Studio (Illumina公司)软件分析SNP基因型, 排除最小基因型频率(minor allele frequency, MAF)低于0.05和SNP得率(call frequency)小于80%的标记, 最终获得31 839个在油菜基因组上具有唯一位置的高质量SNP标记用于后续分析。1.5 群体结构分析与全基因组关联分析
根据SNP标记在甘蓝型油菜19条染色体上的位置, 挑取11 268个均匀分布于染色体上的SNP, 以Structure 2.3.4[19]进行群体结构分析获得Q矩阵, 不作数迭代(length of burn-in period)设置为10 000, MCMC重复为100 000, 组群数(K)设定为1~10, 进行5次模拟运算。利用STRUCTURE HARVESTER (http://taylor0.biology.ucla.edu/structureHarvester/)[20]选择合适的K值作为最适亚群数。将基因型数据导入Tassel 5.1.0[21]后进行亲缘关系分析(K矩阵)和LD (linkage disequilibrium, LD)分析。然后以Q和K矩阵作协变量, 采用基于混合线性模型(mixed linear model, MLM)的Q+K模型, 结合筛选出的31 839个高质量SNP标记, 进行全基因组关联分析, 显著关联SNP阈值设为1/31 839 (3.14×10-5)[22]。利用Manhattan Plot绘制曼哈顿图[23]。利用Tassel 5.1.0分析LD在甘蓝型油菜各染色体上的分布, 绘制各染色体的LD衰减图, LD类型参数设置为Full Matrix。以决定系数R2=0.2为衰减阈值, 计算显著关联SNP所在染色体的LD衰减距离。用于候选基因预测和功能注释分析。
1.6 候选基因分析
根据LD区间在油菜基因组中的位置, 以法国公布的甘蓝型油菜品种“Darmor-Bzh”的基因组[23]注释信息(http://www.genoscope.cns.fr/brassicanapus)分析LD区间内的基因数目和基因编码蛋白序列。在拟南芥信息资源网(The Arabidopsis Information Resource, https://www.arabidopsis.org/)对拟南芥同源基因的基因表达模式、可能的共表达基因和互作蛋白进行了分析, E-value阈值为1×10-10, 以同源性最高的拟南芥基因注释候选基因功能。2 结果与分析
2.1 适宜草铵膦胁迫浓度的确定
选取在200 mg L-1浓度下表现为极敏感、中度敏感、和耐性材料的共20份品种(系), 进行100、200、400和800 mg L-1的浓度梯度筛选。结果表明(图1), 在100 mg L-1的浓度下, 除2个品系表现出严重药害, 部分组织器官坏死, 后期恢复之外, 其他品系药害程度较轻, 后期恢复正常, 形态差异不明显; 在200 mg L-1的浓度下, 5%的品系存活率在75%以上, 且后期恢复生长, 表现出一定的耐药性, 40%的品系前期表现出严重药害, 叶片发黄, 后期全部枯死, 55%的品系的存活率在25%~50%之间, 前期表现出一定药害, 叶片皱缩, 发黄, 后期部分恢复; 在400 mg L-1的浓度下, 65%的品系前期药害程度较重, 后期全部死亡, 35%的品系前期药害较严重, 组织器官坏死, 后期部分恢复; 在800 mg L-1的浓度下, 仅有1个品系存活率为25%, 其他材料药害程度较严重, 后期全部枯死。从几个浓度比较来看, 200 mg L-1的喷施浓度下各品系之间的形态变化差异明显, 可以作为草胺磷胁迫浓度。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1不同浓度草铵膦胁迫下油菜苗期的生理形态差异
-->Fig. 1Physiological and morphological differences under glufosinate in different concentration on seedling
-->
2.2 表型统计分析
对506份自然群体材料苗期对草铵膦胁迫的叶片部分相关性状统计分析(图2和表1)。结果表明, 3个目标性状在品种间的差异性均达到极显著水平, 各目标性状均表现出广泛的表型变异, 如单位叶面积干重系数(CLW)的平均值为37.3%, 变幅为21.1%~68.8%; 综合药害指数(CPC)的平均值为58.934%, 变幅为0.490%~95.000%, 其变异系数高达23.9%。对3个性状用SPSS 17.0进行相关性分析, 结果如表2所示, 3个耐药性性状之间均存在极显著相关性。综合药害指数与单位叶面积干重系数、叶绿素含量系数存在显著的负相关, 相关系数分别为-0.134和-0.314, 而单位叶面积干重系数与叶绿素含量系数之间存在显著的正相关, 相关系数为0.185。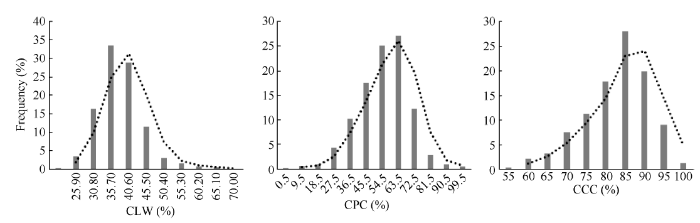 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2相关性状频率分布图
CLW: 单位叶面积干重系数; CCC: 叶绿素含量系数; CPC: 综合药害指数。
-->Fig. 2Phenotype frequency distribution of CLW, CCC, and CPC
CLW: coefficient of dry weight per unit leaf area; CCC: coefficient of chlorophyll content; CPC: comprehensive phytotoxicity coefficient.
-->
Table 1
表1
表1相关性状的表型统计
Table 1Phenotypic statistics of related traits
| 性状 Trait | 均值±标准差 Mean±SD | 变幅范围 Range (%) | 变异系数 CV (%) |
|---|---|---|---|
| 单位叶面积干重系数CLW | 37.30±6.210** | 21.1-68.8 | 16.649 |
| 叶绿素含量系数CCC | 80.24±8.323** | 54.2-100.0 | 10.373 |
| 综合药害指数CPC | 58.93±14.144** | 0.490-95.000 | 24.001 |
新窗口打开
Table 2
表2
表23个性状的相关性分析
Table 2Correlation analysis of CLW, CCC, and CPC
| 测定指标Index | 单位叶面积干重系数CLW | 综合药害指数CPC | 叶绿素含量系数CCC |
|---|---|---|---|
| 单位叶面积干重系数CLW | 1.000 | ||
| 综合药害指数CPC | -0.134** | 1.000 | |
| 叶绿素含量系数CCC | 0.185** | -0.314** | 1.000 |
新窗口打开
2.3 群体结构与亲缘关系分析
本研究利用11 268个均匀分布于染色体上的SNP标记对含506份油菜种质进行了群体结构分析。根据软件的输出结果, 结合分析后验概率ln P(K)以及2个连续的后验概率值的变化速率∆K, 发现当组群数K为2时, ∆K有最大值[24](图3)。经分析506份自然群体材料可以划分为P1和P2两个亚群, 其中P1亚群包括425份材料, 主要是我国的半冬性油菜栽培种; P2亚群包括81份, 绝大多数源自于国内的春性油菜(甘肃和青海)与少量的半冬性材料及从国外引进的种质资源材料。基于贝叶斯数学模型对本研究的自然群体的类群划分, 基本上和油菜的地理栽培属性一致。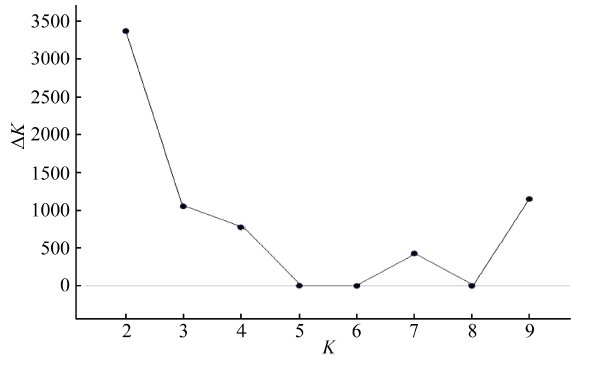 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3自然群体的∆K值
-->Fig. 3Estimation of the ∆K value in the natural population
-->
通过Tassel 5.1.0软件对506份材料构成的自然群体在11 268个SNP标记位点上进行亲缘关系(relative kinship)分析。对计算任意2个材料之间的亲缘关系值所得的结果作频率分布直方图(图4)。发现55.37%的品种(系)亲缘关系值等于0, 约有77.60%的亲缘关系小于0.05, 表明整个群体的材料之间的亲缘关系较弱, 对关联分析产生的影响较小。
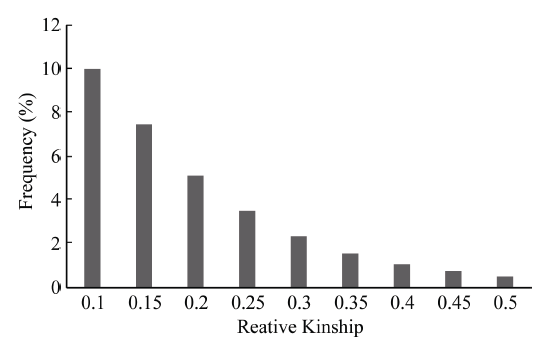 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4自然群体内不同油菜品系两两之间的亲缘关系分布
-->Fig. 4Distribution of pairwise kinship of different B. napus lines in natural population
-->
2.4 LD分析
利用Tassel 5.1.0软件计算两标记间的r2值, 通过按照标记的物理距离来划分区段的方法来估算LD在甘蓝型油菜AC基因组中的衰减(图5)。结果显示, AC基因组的LD均随着物理距离的增加而下降。在R2的阈值为0.2标准下, A基因组的平均衰减距离为600 kb, C基因组的平均衰减距离为1200 kb, A基因组的衰减距离整体比C基因组的衰减距离小得多。这可能与中国半冬性甘蓝型油菜 A 基因组在育种中发生大规模重组, 打破连锁不平衡有关。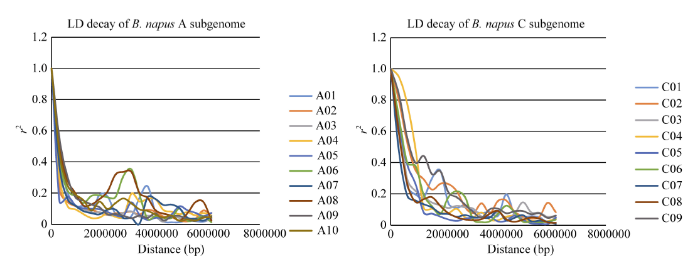 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5甘蓝型油菜A和C基因组不同染色体的LD衰减
-->Fig. 5LD decay of different chromosomes for A and C subgenomes in B. napus
-->
2.5 全基因组关联分析
利用Tassel 5.1.0软件的MLM模型程序, 分别对506份材料在苗期的各耐药性性状的表型值进行全基因组关联分析(图6)。分别统计3个指标检测到的SNP位点(表3)。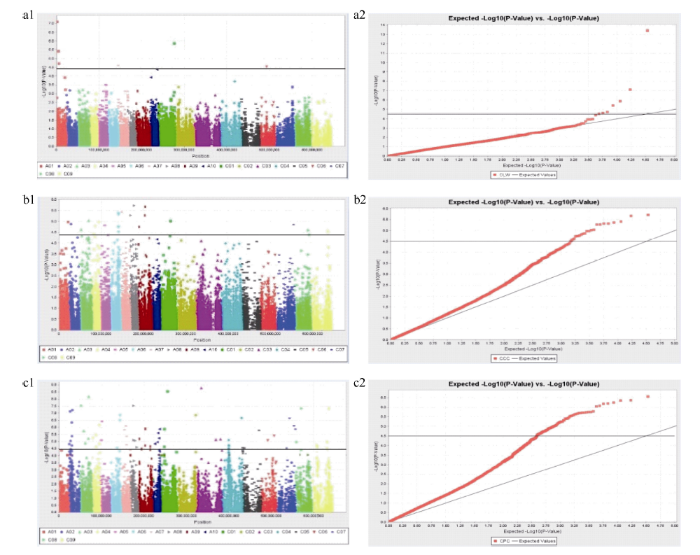 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6Q+K模型下甘蓝型油菜各耐药性性状曼哈顿图
a1、a2分别为单位叶面积干重系数的曼哈顿图和QQ图; b1、b2分别为叶绿素含量系数的曼哈顿图和QQ图; c1、c2分别为综合药害指数的曼哈顿图和QQ图。
-->Fig. 6Manhattan plots of tolerance traits of Brassica napus on seedling growth by Q+K model
a1, a2: Manhattan plot and QQ plot of CLW; b1, b2: Manhattan plot and QQ plot of CCC; c1,c2: Manhattan plot and QQ plot of CPC.
-->
Table 3
表3
表3相关性状的关联位点
Table 3Associations of SNP sites
| 性状 Trait | 位点名称 SNP | 染色体 Chr. | P值 P-value | 贡献率 R2 (%) | 位点名称 SNP | 染色体 Chr. | P 值 P-value | 贡献率 R2 (%) |
|---|---|---|---|---|---|---|---|---|
| 单位叶面积干重系数 CLW | Bn-A01-p1704123 | A01 | 2.36×10-6 | 5.362 | Bn-A07-p593034 | A07 | 2.45×10-5 | 6.526 |
| Bn-A01-p4549945 | A01 | 3.86×10-6 | 7.723 | Bn-C01-p38105098 | C01 | 1.35×10-6 | 8.348 | |
| Bn-A01-p5849310 | A01 | 1.94×10-5 | 6.766 | Bn-C06-p17529141 | C06 | 2.90×10-5 | 6.526 | |
| 叶绿素含量系数 CCC | Bn-A01-p21360797 | A01 | 4.25×10-6 | 5.418 | Bn-A06-p20421942 | A06 | 1.83×10-5 | 5.179 |
| Bn-A02-p4940608 | A02 | 1.35×10-5 | 5.313 | Bn-A07-p3477622 | A07 | 3.81×10-6 | 5.883 | |
| Bn-A03-p21755979 | A03 | 9.06×10-6 | 5.493 | Bn-A08-p9184409 | A08 | 1.90×10-6 | 6.195 | |
| Bn-A03-p4171667 | A03 | 2.37×10-5 | 5.063 | Bn-A08-p984273 | A08 | 5.34×10-6 | 5.730 | |
| Bn-A04-p16495266 | A04 | 1.06×10-5 | 5.423 | Bn-A09-p16498800 | A09 | 2.14×10-6 | 6.142 | |
| Bn-A04-p7873782 | A04 | 1.64×10-5 | 5.228 | Bn-A09-p15859343 | A09 | 5.27×10-6 | 5.737 | |
| Bn-A05-p10742077 | A05 | 1.52×10-5 | 5.261 | Bn-A09-p4171667 | A09 | 1.77×10-5 | 5.193 | |
| Bn-A06-p20575568 | A06 | 4.27×10-6 | 5.831 | Bn-C01-p25343230 | C01 | 9.59×10-6 | 5.468 | |
| Bn-A06-p20526477 | A06 | 4.78×10-6 | 5.780 | Bn-C07-p41693853 | C07 | 1.41×10-5 | 5.294 | |
| Bn-A06-p20546803 | A06 | 4.83×10-6 | 5.775 | Bn-C08-p32299051 | C08 | 2.55×10-5 | 5.030 | |
| Bn-A06-p20602779 | A06 | 1.79×10-5 | 5.187 | Bn-C09-p40117533 | C09 | 2.89×10-5 | 4.974 | |
| 综合药 害指数 CPC | Bn-A01-p986711 | A01 | 2.30×10-5 | 9.031 | Bn-A09-p6931495 | A09 | 1.41×10-7 | 7.465 |
| Bn-A01-p994322 | A01 | 3.35×10-5 | 4.360 | Bn-A10-p12019156 | A10 | 2.21×10-6 | 7.783 | |
| Bn-A02-p6931495 | A02 | 2.75×10-5 | 12.645 | Bn-A10-p19586200 | A10 | 6.82×10-6 | 4.996 | |
| Bn-A02-p6901239 | A02 | 1.11×10-6 | 5.712 | Bn-A10-p10124750 | A10 | 2.88×10-6 | 5.236 | |
| Bn-A02-p6976201 | A02 | 2.36×10-7 | 18.659 | Bn-A10-p14852876 | A10 | 1.28×10-7 | 5.632 | |
| Bn-A02-p6371001 | A02 | 8.58×10-6 | 4.896 | Bn-A10-p15935269 | A10 | 3.45×10-5 | 7.852 | |
| Bn-A02-p8005692 | A02 | 1.39×10-9 | 10.455 | Bn-A10-p15524190 | A10 | 1.86×10-5 | 4.891 | |
| Bn-A02-p8945120 | A02 | 4.27×10-9 | 7.960 | Bn-C01-p9201350 | C01 | 1.84×10-7 | 8.123 | |
| Bn-A02-p145473 | A02 | 1.75×10-5 | 4.513 | Bn-C01-p9611053 | C01 | 9.24×10-6 | 4.866 | |
| Bn-A02-p121255 | A02 | 1.01×10-5 | 4.838 | Bn-C01-p9965323 | C01 | 2.01×10-6 | 5.624 | |
| Bn-A02-p9652140 | A02 | 4.23×10-6 | 4.020 | Bn-C01-p9604036 | C01 | 5.12×10-6 | 5.108 | |
| Bn-A02-p9570320 | A02 | 3.69×10-5 | 4.181 | Bn-C01-p32514001 | C01 | 2.32×10-10 | 8.583 | |
| Bn-A02-p965241 | A02 | 2.06×10-5 | 5.342 | Bn-C01-p33876059 | C01 | 9.97×10-7 | 5.798 | |
| Bn-A02-p974395 | A02 | 1.02×10-6 | 5.745 | Bn-C02-p41927269 | C02 | 8.48×10-5 | 4.122 | |
| Bn-A02-p3483764 | A02 | 1.87×10-5 | 4.337 | Bn-C02-p41639953 | C02 | 9.25×10-9 | 8.390 | |
| Bn-A02-p3546981 | A02 | 3.69×10-5 | 4.321 | Bn-C03-p55465412 | C03 | 5.56×10-5 | 4.391 | |
| Bn-A02-p6627159 | A02 | 5.02×10-7 | 12.607 | Bn-C03-p57991205 | C03 | 4.04×10-6 | 5.938 | |
| Bn-A03-p9845120 | A03 | 1.96×10-8 | 16.363 | Bn-C03-p57106187 | C03 | 2.87×10-9 | 8.559 | |
| Bn-A03-p9468697 | A03 | 1.39×10-5 | 4.706 | Bn-C03-p57326280 | C03 | 1.52×10-8 | 7.918 | |
| Bn-A03-p9999846 | A03 | 4.75×10-7 | 4.842 | Bn-C04-p6292605 | C04 | 2.30×10-6 | 5.720 | |
| Bn-A03-p9451200 | A03 | 2.96×10-5 | 3.958 | Bn-C04-p6200125 | C04 | 5.97×10-6 | 5.387 | |
| Bn-A03-p21867237 | A03 | 8.88×10-6 | 4.472 | Bn-C04-p6312067 | C04 | 5.11×10-6 | 5.460 | |
| Bn-A04-p16524169 | A04 | 2.94×10-5 | 4.735 | Bn-C04-p621851 | C04 | 5.98×10-6 | 5.426 | |
| Bn-A04-p16714914 | A04 | 3.05×10-8 | 7.548 | Bn-C04-p6313229 | C04 | 5.51×10-5 | 4.388 | |
| Bn-A04-p16732201 | A04 | 4.77×10-6 | 5.505 | Bn-C04-p6985469 | C04 | 4.38×10-6 | 5.512 | |
| Bn-A04-p1983502 | A04 | 3.09×10-8 | 7.544 | Bn-C04-p20152051 | C04 | 3.48×10-5 | 4.623 | |
| Bn-A04-p1945120 | A04 | 1.24×10-6 | 6.115 | Bn-C04-p9140899 | C04 | 2.70×10-5 | 4.689 | |
| Bn-A04-p9002613 | A04 | 1.03×10-8 | 8.011 | Bn-C05-p7747904 | C05 | 8.26×10-7 | 6.148 | |
| 性状 Trait | 位点名称 SNP | 染色体 Chr. | P值 P-value | 贡献率 R2 (%) | 位点名称 SNP | 染色体 Chr. | P 值 P-value | 贡献率 R2 (%) |
| 综合药 害指数 CPC | Bn-A05-p19765662 | A05 | 2.77×10-5 | 4.675 | Bn-C05-p7765985 | C05 | 1.89×10-10 | 9.835 |
| Bn-A05-p19693221 | A05 | 7.18×10-5 | 4.364 | Bn-C05-p3235412 | C05 | 8.74×10-5 | 4.207 | |
| Bn-A06-p15660438 | A06 | 3.14×10-5 | 4.626 | Bn-C05-p3698257 | C05 | 3.25×10-5 | 4.656 | |
| Bn-A06-p14512365 | A06 | 5.24×10-6 | 5.467 | Bn-C05-p36474843 | C05 | 5.81×10-5 | 4.140 | |
| Bn-A06-p15261863 | A06 | 2.47×10-5 | 4.741 | Bn-C05-p21774776 | C05 | 2.47×10-6 | 18.623 | |
| Bn-A06-p15365248 | A06 | 4.82×10-7 | 6.452 | Bn-C05-p2564789 | C05 | 6.21×10-5 | 4.119 | |
| Bn-A07-p23001203 | A07 | 1.00×10-8 | 8.026 | Bn-C06-p24235721 | C06 | 7.16×10-5 | 4.061 | |
| Bn-A07-p23418663 | A07 | 2.23×10-5 | 4.812 | Bn-C06-p32728327 | C06 | 4.25×10-5 | 4.069 | |
| Bn-A07-p4480260 | A07 | 2.25×10-6 | 4.260 | Bn-C07-p22140411 | C07 | 5.55×10-7 | 10.771 | |
| Bn-A07-p20935217 | A07 | 2.44×10-6 | 5.263 | Bn-C07-p12256348 | C07 | 2.45×10-5 | 4.487 | |
| Bn-A07-p23340612 | A07 | 7.48×10-5 | 4.260 | Bn-C07-p22118892 | C07 | 2.87×10-7 | 6.745 | |
| Bn-A07-p28542122 | A07 | 2.77×10-5 | 4.865 | Bn-C07-p20147854 | C07 | 1.33×10-5 | 4.756 | |
| Bn-A08-p10182282 | A08 | 1.81×10-5 | 4.869 | Bn-C08-p13638153 | C08 | 2.39×10-9 | 5.369 | |
| Bn-A08-p13250140 | A08 | 7.39×10-5 | 4.362 | Bn-C08-p12356824 | C08 | 8.26×10-6 | 4.912 | |
| Bn-A08-p7652410 | A08 | 2.12×10-7 | 6.728 | Bn-C08-p19139658 | C08 | 3.25×10-8 | 6.952 | |
| Bn-A08-p7505807 | A08 | 7.75×10-8 | 7.289 | Bn-C08-p19568928 | C08 | 1.37×10-6 | 5.627 | |
| Bn-A09-p2201470 | A09 | 1.12×10-7 | 7.154 | Bn-C09-p40117533 | C09 | 4.16×10-7 | 6.356 | |
| Bn-A09-p2176198 | A09 | 9.27×10-6 | 5.182 | Bn-C09-p46985366 | C09 | 6.42×10-5 | 5.632 | |
| Bn-A09-p16521452 | A09 | 7.81×10-6 | 5.211 | Bn-C09-p12891995 | C09 | 2.99×10-5 | 3.265 | |
| Bn-A09-p15882405 | A09 | 2.91×10-5 | 4.721 | Bn-C09-p12356923 | C09 | 3.54×10-5 | 4.631 | |
| Bn-A09-p6598420 | A09 | 1.90×10-5 | 5.304 | Bn-C09-p13003243 | C09 | 1.67×10-5 | 4.365 |
新窗口打开
与单位叶面积干重系数(CLW)关联的位点共6个, 其中A01染色体上有3个, A07染色体上有1个, C01染色体上有1个, C06染色体上有1个。可解释的表型贡献率范围为6.53%~10.04%。与叶绿素含量系数(CCC)相关联的SNP位点较多, 共22个。检测到的位点遍布A01~A09染色体, 可解释的表型贡献率范围为5.06%~6.20%, 在C01、C07、C08和C09染色体上均检测到与叶绿素含量系数相关的SNP位点, 对表型的贡献率范围为4.97%~5.47%。其中A06染色体上检测到的5个位点的遗传距离较近。与综合药害指数(CPC)相关的SNP位点最多, 共98个, 基本覆盖了AC基因组, 表型贡献率为3.25%~ 18.66%。
2.6 候选基因预测
根据关联的SNP位点所在染色体的不同, 提取与各性状显著关联的SNP位点, 确定SNP位点在甘蓝型油菜基因组染色体上的LD置信区间, 在决定系数R2=0.2对应的LD区域寻找与草铵膦抗性有关的候选基因(表4)。Table 4
表4
表4耐药性相关性状的候选基因
Table 4A summary of candidate genes associated with herbicide tolerance related traits
| 性状 Trait | 位点 Site | 染色体 Chr. | 物理位置 Physical interval (bp) | 拟南芥基因 Arabidopisis genes | 候选基因 Candidate genes in the LD interval |
|---|---|---|---|---|---|
| 单位叶面积干重系数 | Bn-A01-p1704123 | A01 | 1633033-1633658 | AT4G33467 | BnaA01g03470D |
| CWL | Bn-A01-p4549945 | A01 | 4548199-4552433 | AT4G18490 | BnaA01g09330D |
| 叶绿素含量系数 | Bn-A01-p21360797 | A01 | 21153560-21155842 | AT3G11290 | BnaA01g31070D |
| CCC | Bn-A02-p4940608 | A02 | 4607681-4610486 | AT5G55050 | BnaA02g09320D |
| Bn-A02-p4940608 | A02 | 459901-4602406 | AT5G55070 | BnaA02g09300D | |
| Bn-A02-p4940608 | A02 | 5204578-5206797 | AT5G53470 | ACBP1 | |
| Bn-A03-p21755979 | A03 | 21582364-21584595 | AT4G17250 | BnaA03g42990D | |
| Bn-A03-p21755979 | A03 | 21958105-21965340 | AT2G07680 | ATABCC13 | |
| Bn-A03-p21755979 | A03 | 21953245-21955187 | AT4G18070 | BnaA03g43580D | |
| Bn-A03-p4171667 | A03 | 4122660-4124952 | AT5G59960 | BnaA03g09150D | |
| Bn-A04-p16495266 | A04 | 16368845-16371398 | AT2G37500 | BnaA04g21470D | |
| Bn-A04-p16495266 | A04 | 16372246-16376604 | AT2G37520 | BnaA04g21480D | |
| Bn-A04-p16495266 | A04 | 16498173-16499643 | AT2G20625 | BnaA04g21710D | |
| Bn-A04-p16495266 | A04 | 16508877-16511523 | AT2G37960 | BnaA04g21730D | |
| Bn-A04-p7873782 | A04 | 7808158-7809504 | AT5G67150 | BnaA04g08790D | |
| Bn-A05-p10742077 | A05 | 10774154-10779061 | LACS2 | BnaA05g16170D | |
| Bn-A07-p3477622 | A07 | 3235720-3238222 | AT2G16270 | BnaA07g03580D | |
| Bn-A09-p16498800 | A09 | 16425283-16427170 | AT1G33270 | BnaA09g23800D | |
| Bn-C07-p41693853 | C07 | 41701416-41703232 | AT2G25150 | BnaC07g42100D | |
| Bn-C07-p41693853 | C07 | 41797435-41799523 | AT4G30060 | BnaC07g42290D | |
| 综合药害指数 | Bn-A02-p6901239 | A02 | 6771193-6774102 | AT1G66860 | BnaA02g12600D |
| CPC | Bn-A04-p1983502 | A04 | 2101543-2102589 | AT2G23060 | BnaA04g03220D |
| Bn-A04-p1983502 | A04 | 2110543-2111592 | AT3G57100 | BnaA04g03240D | |
| Bn-A07-p4480260 | A07 | 4562018-4563915 | AT2G06025 | BnaA07g04340D | |
| Bn-A08-p10182282 | A08 | 10187701-10189221 | AT4G34520 | BnaA08g11130D | |
| Bn-A08-p13250140 | A08 | 13047661-13049407 | AT4G37580 | BnaA08g15700D | |
| Bn-C07-p22140411 | C07 | 22179972-22180303 | AT5G67160 | EPS1 | |
| Bn-C07-p22140411 | C07 | 22181878-22183299 | AT5G67150 | BnaC07g16220D |
新窗口打开
对单位叶面积干重系数(CLW)的全基因组关联分析, 在A01染色体的LD区域的基因BnaA01g03470D和BnaA01g09330D分别与功能未知的拟南芥基因AT4G33467和AT4G18490匹配。
扫描与叶绿素含量系数(CCC)显著关联SNP标记的LD区域, 找到11个与草铵膦灭生机制有关的候选基因。在A02染色体上的基因BnaA02g09320D和BnaA02g09300D分别与拟南芥基因AT5G55050和AT5G55070同源, 均参与酰基转移酶活性的调控, 基因ACBP1与拟南芥基因AT5G53470同源, 与酰基CoA结合蛋白活性有关。A03染色体上的基因ATABCC13, 在拟南芥中的同源基因AT2G07680是拟南芥耐药相关蛋白MRP/ABCC亚科的成员。位于A04染色体上的基因BnaA04g21470D在拟南芥中的同源基因AT2G37500与谷氨酸乙酰转移酶活性有关, 与该基因相距约3.4 kb处的基因BnaA04g21480D在拟南芥中的同源基因AT2G37520编码乙酰CoA转移酶; A04染色体上的基因BnaA04g08790D和C07染色体上的基因BnaC07g42100D分别与拟南芥基因AT5G67150和AT2G25150同源, 编码一种HXXXD型酰基转移酶家族蛋白, 调控酰基转移酶活性。在A05染色体上发现的基因BnaA05g16170D与拟南芥基因LACS2同源, 编码酰基CoA合成酶。A09染色体上的基因BnaA09g23800D在拟南芥中的同源基因AT1G33270与乙酰转移酶活性有关。在C07染色体上找到的基因BnaC07g42290D在拟南芥中的同源基因AT4G30060与β-1,6-N-二乙酰氨基转移酶活性有关。
在与综合药害指数(CPC)关联的SNP位点的LD区域找到了7个与Bar基因草铵膦抗性机制有关的候选基因。在A02染色体上找到的基因BnaA02g12600D在拟南芥中的同源基因AT1G66860与谷氨酰胺转移酶活性有关, 参与谷氨酰胺代谢。A04染色体上的基因BnaA04g03220D和A07染色体上的基因BnaA07g04340D分别与拟南芥基因AT2G23060和AT2G06025同源, 编码的蛋白为酰基CoA的酰基转移酶(NAT)家族蛋白, 具有调节乙酰转移酶活性的功能。A08染色体上的基因BnaA08g11130D在拟南芥中的同源基因AT4G34520与乙酰CoA合成酶有关, 基因BnaA08g15700D与拟南芥基因AT4G37580同源, 与乙酰转移酶活性有关。C07染色体上的基因EPS1编码BAHD酰基转移酶。基因BnaC07g16220D编码的蛋白为HXXXD型酰基转移酶家族蛋白, 与酰基转移酶活性有关。
3 讨论
3.1 草铵膦灭生机制及其抗性遗传研究
草铵膦是L-谷氨酸的膦酸衍生物, 以植物绿色茎叶接触吸收为主, 喷施草铵膦3~5 d后, 植株固定CO2的速率下降, 植物叶片出现枯黄和坏死[25]。为了减轻草铵膦对作物的危害, 培育抗草铵膦品种就成为育种的主要目标之一。目前发现的草铵膦抗性基因主要有bar、pat等基因, 都从链霉菌中克隆[26,27], 并已经成功转入各种作物[28,29,30,31], 获得极好的抗草铵膦效果, 因此多认为, 草铵膦抗性受到主基因控制。我们在喷施草铵膦进行油菜田化学除草的过程中发现, 在一定浓度下, 部分油菜苗期虽然受到草铵膦的毒害作用(不足以致死), 但是随着草铵膦药害解除, 在后期能够恢复正常生长, 可能是因为草铵膦是触杀型除草剂, 在体内传导性差, 另外也说明不同油菜品种(系)对草铵膦的耐性具有一定的差异, 抗(耐)草铵膦特性可以作为质量-数量性状遗传进行研究。本实验选用不同浓度的草铵膦处理油菜, 结果表明在喷施低浓度(200 mg L-1)时, 部分材料虽然受到影响, 但能恢复, 该浓度可作为耐受性的适宜筛选浓度。
3.2 候选基因
作物对除草剂的选择性与除草剂的代谢密切相关, 抗性植物能够迅速代谢除草剂而使其失去活性。本研究表明, 综合药害指数与单位叶面积干重系数、叶绿素含量系数存在显著的负相关, 而单位叶面积干重系数与叶绿素含量系数之间存在显著的正相关。这是因为叶片吸收草铵膦后, 叶绿素被破坏, 光合作用受到抑制, 进而有机物合成减弱, 同化作用小于异化作用[2,3], 导致单位叶面积干重下降。这与信晓阳等[6]、王仕林等[17]研究相吻合。同时, 前人在非胁迫条件下对油菜的叶绿素含量的遗传规律研究表明, 叶绿素含量除受核基因控制外, 还受母体质基因影响[32]。本研究共得到18个候选基因。这些基因有的与酰基转移酶活性有关, 有的编码乙酰CoA结合蛋白, 有的调控乙酰CoA酰基转移酶活性。已有的研究结果表明, 草铵膦可以强烈抑制植物谷氨酰氨合成酶(GS)的活性[2,3], 继而导致细胞内氨的含量迅速积累。而氨的积累则直接抑制光系统和光系统反应的正常有序进行, 导致跨膜PH梯度减少, 使光合磷酸化解偶联, 随之叶绿体结构解体, 最后引起整个植物体死亡[4]。位于A04染色体上的基因BnaA04g21470D和A02染色体上找到的基因BnaA02g12600D与谷氨酸乙酰转移酶活性有关, 在距标记Bn-A02-p4940608约332 kb处的基因BnaA03g43580D也参与酰基转移酶活性的调控, 进而催化蛋白质的酰基化和去酰基化[27], 由此影响蛋白质的生物活性和生理过程。在拟南芥中, 基因BnaA02g09300D可参与支链氨基酸降解对细胞产生的毒害的调控过程[28]。这些已知功能的部分基因进一步说明了草铵膦胁迫下油菜的应激反应。此外, 本研究在与3个性状相关联的SNP的LD区域找到了12个拟南芥功能未知的的基因, 这些基因具有重要的挖掘价值, 将成为下一步研究的重要内容。4 结论
共扫描得到与单位叶面积干重耐除草剂系数(CLW)显著相关的SNP位点共6个, 与叶绿素含量耐除草剂系数(CCC)相关的SNP位点共22个, 与综合药害指数(CPC)相关的SNP位点共监测到98个, 覆盖了A、C基因组。共找到BnaA02g09320D、BnaA04g21730D、BnaC07g42290D、BnaA07g04340D等18个与草铵膦的灭生机制相关的候选基因, 这些候选基因可能通过酰基转移酶或者谷氨酰氨合成酶(GS)的活性的调控导致油菜对草铵膦的耐药性的差异。The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
| [1] | |
| [2] | . . |
| [3] | . . |
| [4] | . |
| [5] | . . |
| [6] | . . |
| [7] | . |
| [8] | . |
| [9] | . . |
| [10] | . |
| [11] | . |
| [12] | . . |
| [13] | . . |
| [14] | . |
| [15] | . . |
| [16] | . . |
| [17] | . . |
| [18] | . |
| [19] | . |
| [20] | . |
| [21] | . |
| [22] | . . |
| [23] | . . |
| [24] | . |
| [25] | . |
| [26] | . |
| [27] | . |
| [28] | . |
| [29] | . |
| [30] | . |
| [31] | . |
| [32] | . . |
