 ,, 潘鑫峰, 毛志君, 叶方婷, 李兆伟, 林伟伟, 林文雄
,, 潘鑫峰, 毛志君, 叶方婷, 李兆伟, 林伟伟, 林文雄 ,*福建农林大学农学院作物遗传育种与综合利用教育部重点实验室, 福建福州 350002
,*福建农林大学农学院作物遗传育种与综合利用教育部重点实验室, 福建福州 350002Identification and analysis of sHSP gene family in Gossypioides kirkii
FAN Kai ,, PAN Xin-Feng, MAO Zhi-Jun, YE Fang-Ting, LI Zhao-Wei, LIN Wei-Wei, LIN Wen-Xiong
,, PAN Xin-Feng, MAO Zhi-Jun, YE Fang-Ting, LI Zhao-Wei, LIN Wei-Wei, LIN Wen-Xiong ,*Key Laboratory of Ministry of Education for Genetics, Breeding and Multiple Utilization of Crops, College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fuzhou, China
,*Key Laboratory of Ministry of Education for Genetics, Breeding and Multiple Utilization of Crops, College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fuzhou, China通讯作者: *林文雄, E-mail:lwx@fafu.edu.cn
收稿日期:2020-10-29接受日期:2021-03-19网络出版日期:2021-03-31
| 基金资助: |
Corresponding authors: *E-mail:lwx@fafu.edu.cn
Received:2020-10-29Accepted:2021-03-19Published online:2021-03-31
| Fund supported: |
作者简介 About authors
E-mail:fankai@fafu.edu.cn

摘要
植物小分子热激蛋白(small heat shock protein, sHSP)是植物热激蛋白中一类分子量最小的基因家族, 该家族具有保守的α-晶体结构域, 在响应外界环境胁迫过程中具有重要的作用。叉柱棉(Gossypioides kirkii)有关sHSP家族的鉴定与特征分析还未见相关报道。本研究在叉柱棉中共鉴定了39个GksHSP成员, 并且可以将GksHSP成员进一步分为10个亚家族。在GksHSP家族中发现了7个基因复制事件, 并且所有的基因复制事件都是片段复制事件。棉属特有的全基因组复制事件主要导致了GksHSP成员的扩增, 其扩增还与蛋白激酶家族、线粒体载体蛋白家族以及植物生长素响应蛋白家族有关。此外, GksHSP家族可能与ABA和茉莉酸甲酯调控的胁迫响应相关, 并且GksHSP26成员及其在陆地棉中的同源基因可能在胁迫响应中具有关键的作用。本研究的结果可以进一步为今后叉柱棉和棉花的抗逆育种研究提供一定的理论基础。
关键词:
Abstract
Small heat shock protein (sHSP) has the lowest molecular weight in heat shock protein and sHSP has the highly conserved α-crystallin domain. The sHSP family is very important in response to various stresses, but the identification and analysis of sHSP gene family in Gossypioides kirkii have not been reported. In this study, we identified 39 GksHSPs with 10 subfamilies in Gossypioides kirkii. There were seven gene duplication events in GksHSP family and all of duplicated gene pairs were involved in segmental duplication events. The cotton-specific whole genome duplication event primarily resulted in the GksHSP expansion. The GksHSP expansion was also related to the protein kinase, mitochondrial carrier protein, and auxin responsive protein. Besides, GksHSP members might respond to various stresses by ABA/MeJA-mediated pathways, and GksHSP26 and its corresponding orthologous genes in Gossypium hirsutum were very important in stress responses. These results could provide the theoretical basis of the breeding of stress tolerance in Gossypioides kirkii and cotton.
Keywords:
PDF (1335KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
范凯, 潘鑫峰, 毛志君, 叶方婷, 李兆伟, 林伟伟, 林文雄. 叉柱棉sHSP基因家族的鉴定与特征分析[J]. 作物学报, 2021, 47(10): 1913-1926 DOI:10.3724/SP.J.1006.2021.04235
FAN Kai, PAN Xin-Feng, MAO Zhi-Jun, YE Fang-Ting, LI Zhao-Wei, LIN Wei-Wei, LIN Wen-Xiong.
在自然环境中, 植物会频繁受到诸如干旱、盐渍、高温等胁迫的影响, 这些胁迫严重制约着植物的生长和发育过程, 进而影响植物的产量和品质。目前已经发现有许多基因家族可以响应外界的环境胁迫, 如WRKY家族[1]、PP2C家族[2]、NAC家族[3]等, 其中植物热激蛋白(heat shock protein, HSP)是一类重要的响应外界环境胁迫的基因家族[4]。
植物热激蛋白依据分子量的大小可以进一步分为HSP100家族、HSP90家族、HSP70家族、HSP60家族和HSP20家族, 其中HSP20家族是分子量最小的一类植物热激蛋白[5]。HSP20成员的分子量集中在15~42 kD之间, 因此又称为植物小分子热激蛋白(small heat shock protein, sHSP)[5]。sHSP成员在C端具有长度大约为80~100个氨基酸组成的保守的α-晶体结构域, 该结构域主要由β-sandwich结构组成, 其N端具有相对多样化的结构[6]。sHSP家族不仅存在于酵母、动物等中[7,8], 而且在拟南芥[9]、水稻[10]、番茄[11]、马铃薯[12]、棉花[13]、银杏[14]、簸箕柳[15]等植物中也识别出许多成员。在植物中, sHSP家族主要以分子伴侣形式保护其他蛋白质免受多种胁迫的伤害, 已有大量的研究表明sHSP成员可以响应多种外界胁迫[6]。过表达水稻的sHSP成员OsHSP16.9可以提高水稻植株的抗盐和抗旱能力[16]。过表达苜蓿的sHSP成员MsHSP17.7可以提高拟南芥植株的抗热、抗旱以及抗氧化胁迫的能力[17]。过表达拟南芥的sHSP成员AtHSP17.6A可以提高拟南芥植株的抗盐和抗旱能力[18]。
叉柱棉(Gossypioides kirkii)主要分布在莫桑比克、肯尼亚、坦桑尼亚以及南非等地区, 其可以产生棕色的纤维, 种子呈椭圆状且无短绒毛。叉柱棉属于锦葵科棉族叉柱棉属, 是常见的重要经济作物棉花的近缘物种, 是研究棉花进化趋势的关键物种。叉柱棉常年生长在高温地区, 对其高温相关基因的研究有助于揭示其耐高温调控机制并对棉花的耐高温育种具有重要的意义。叉柱棉全基因组测序的完成为研究叉柱棉乃至棉花的耐高温机制提供了便利的条件[19]。目前有关叉柱棉的研究主要集中在揭示其染色体数目(n=12)比棉花染色体数目(n=13)少一条的起因上[19,20,21], 而叉柱棉基因家族的研究还未见相关报道, 其相关的逆境胁迫研究还处于空白状态。本研究主要对叉柱棉sHSP家族的鉴定、结构特征、系统发育关系、保守结构域、基因结构、染色体定位、基因复制事件、启动子中的顺式调控元件分布等方面进行综合分析, 旨在全面解析叉柱棉sHSP家族的进化历史, 为今后叉柱棉和棉花的抗逆育种研究提供一定的理论基础。
1 材料与方法
1.1 材料
从Cottongen(1.2 sHSP成员的鉴定以及序列分析
从pfam数据库中下载(通过MEME程序(
1.3 系统发育分析
所有的sHSP成员通过MAFFT软件进行序列联配分析。本研究使用最大似然法利用IQ-tree软件构建系统发育进化树, 其中最合适的模型使用ModelFinder程序进行评估, bootstrap值设定为1000。进化树使用MEGA软件进行可视化展示。1.4 基因复制事件分析
GksHSP成员的基因复制事件主要通过MCScan (Python version)软件进行识别, 每个复制框中至少包括5个基因复制事件, 其余均为默认参数, 并且用Circos软件对基因复制关系进行可视化展示。此外, 通过MCScan (Python version)软件对与GksHSP成员复制事件相关的微共线关系进行可视化分析, 利用NCBI中CDD程序(1.5 启动子区的顺式调控元件分析
从CottonGen网站中提取GksHSP成员启动子区域2000 bp的序列, 通过PlantCARE数据库(2 结果与分析
2.1 叉柱棉sHSP成员的识别和特征分析
本研究通过HMMER 3.0程序在叉柱棉基因组中共鉴定出39个叉柱棉sHSP成员(GksHSP), 并且每一个GksHSP成员均包含sHSP家族保守的PF00011结构域(表1)。依据其在染色体上的位置, 将鉴定出的GksHSP成员命名为GksHSP01~GksHSP39。GksHSP成员编码氨基酸的长度在93~327 aa之间, 其分子量介于10.5039~36.8318 kD之间。GksHSP32编码长度最短(93 aa)和最小分子量(10.5039 kD)的氨基酸, 而GksHSP36编码长度最长(327 aa)和最大分子量(36.83181 kD)的氨基酸。GksHSP成员理论pI为4.37~9.22, 最低的pI为4.37 (GksHSP11), GksHSP22有最高的pI (9.22)。所有的GksHSP成员均具有典型的4种蛋白质二级结构(α-螺旋、延伸链、β-折叠和无规则卷曲)。在蛋白质二级结构中, 除了GksHSP31和GksHSP18包含最高比例的α-螺旋结构外, 其余GksHSP成员包含最高比例的无规则卷曲结构, 而所有的GksHSP成员均包含最低比例的β-折叠结构。
2.2 叉柱棉sHSP成员的系统发育分析
本研究利用IQ-tree软件对拟南芥和叉柱棉中sHSP成员进行系统发育分析(图1)。使用ModelFinder模式计算发现, JTT+I+G4模型为构建该系统发育关系中最合适的氨基酸替换模型。按照之前对sHSP亚家族的分类标准[6], 本研究可以将GksHSP成员分为10个亚家族, 并且每个亚家族包含不同数目的GksHSP成员。CI亚家族有18个GksHSP成员, CII亚家族有5个GksHSP成员, CP亚家族有3个GksHSP成员, CIV亚家族、CV亚家族、ER亚家族、MTI/CP亚家族和PX亚家族分别有2个GksHSP成员, CIII亚家族和MTII亚家族分别有1个GksHSP成员, 另外还有1个GksHSP成员不属于任何亚家族。图1
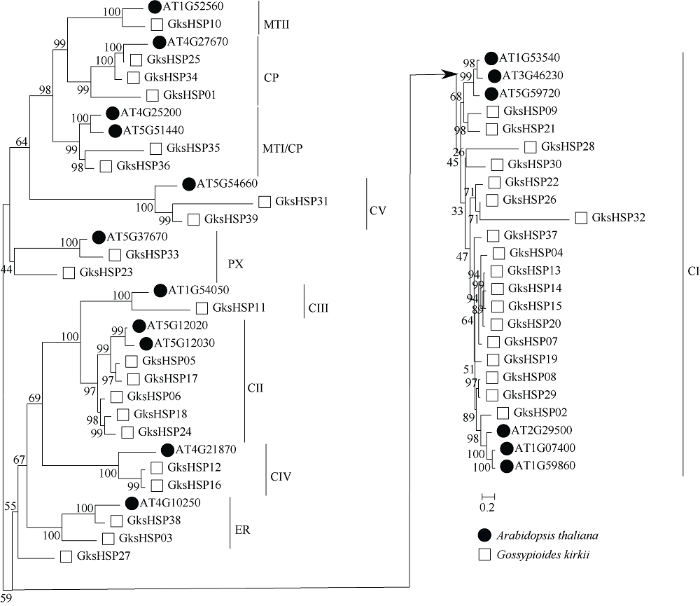 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1拟南芥和叉柱棉中sHSP成员的系统发育分析
Fig. 1Phylogenetic analysis of sHSP members in Arabidopsis thaliana and Gossypioides kirkii
Table 1
表1
表1本研究中GksHSP成员的鉴定以及结构分析
Table 1
| 名称 Name | 位点名称 Locus name | 亚家族 Subfamily | 蛋白质长度 Protein length (aa) | 分子量 Molecular weight (kD) | 理论pI Theoretical pI | α-螺旋 α-helix | 延伸链 Extended strand | β-折叠 β-turn | 无规则卷曲 Random coil |
|---|---|---|---|---|---|---|---|---|---|
| GksHSP01 | Kirkii_Version3_Juiced.00g028900 | CP | 144 | 16.32340 | 4.95 | 23 | 30 | 6 | 85 |
| GksHSP02 | Kirkii_Version3_Juiced.00g047410 | CI | 159 | 18.32872 | 5.98 | 28 | 29 | 11 | 91 |
| GksHSP03 | Kirkii_Version3_Juiced.00g056780 | ER | 187 | 21.44579 | 6.77 | 64 | 31 | 8 | 84 |
| GksHSP04 | Kirkii_Version3_Juiced.00g059950 | CI | 153 | 17.69095 | 5.35 | 34 | 28 | 7 | 84 |
| GksHSP05 | Kirkii_Version3_Juiced.00g085690 | CII | 159 | 18.01872 | 6.64 | 52 | 34 | 8 | 65 |
| GksHSP06 | Kirkii_Version3_Juiced.00g085700 | CII | 156 | 17.66154 | 6.62 | 53 | 38 | 11 | 54 |
| GksHSP07 | Kirkii_Version3_Juiced.00g091700 | CI | 156 | 17.91837 | 5.79 | 38 | 30 | 10 | 78 |
| GksHSP08 | Kirkii_Version3_Juiced.00g091710 | CI | 156 | 17.80026 | 6.18 | 36 | 29 | 9 | 82 |
| GksHSP09 | Kirkii_Version3_Juiced.00g092160 | CI | 133 | 14.79902 | 9.00 | 40 | 22 | 5 | 66 |
| GksHSP10 | Kirkii_Version3_Juiced.00g096000 | MTII | 246 | 27.95450 | 7.80 | 82 | 46 | 13 | 105 |
| GksHSP11 | Kirkii_Version3_Juiced.00g097580 | CIII | 102 | 11.37751 | 4.37 | 35 | 16 | 5 | 46 |
| GksHSP12 | Kirkii_Version3_Juiced.00g101120 | CIV | 139 | 15.76492 | 5.34 | 28 | 29 | 10 | 72 |
| GksHSP13 | Kirkii_Version3_Juiced.00g120690 | CI | 159 | 18.25460 | 6.18 | 20 | 34 | 10 | 95 |
| GksHSP14 | Kirkii_Version3_Juiced.00g120700 | CI | 159 | 18.19460 | 6.18 | 28 | 31 | 10 | 90 |
| GksHSP15 | Kirkii_Version3_Juiced.00g120710 | CI | 159 | 18.27965 | 6.64 | 29 | 32 | 8 | 90 |
| GksHSP16 | Kirkii_Version3_Juiced.00g134580 | CIV | 139 | 15.85984 | 4.61 | 30 | 34 | 6 | 69 |
| GksHSP17 | Kirkii_Version3_Juiced.00g136170 | CII | 159 | 18.18400 | 6.19 | 49 | 31 | 7 | 72 |
| GksHSP18 | Kirkii_Version3_Juiced.00g136180 | CII | 153 | 17.05883 | 8.59 | 53 | 40 | 9 | 51 |
| GksHSP19 | Kirkii_Version3_Juiced.00g184450 | CI | 156 | 17.82725 | 5.77 | 32 | 31 | 10 | 83 |
| GksHSP20 | Kirkii_Version3_Juiced.00g184460 | CI | 160 | 18.36475 | 6.18 | 39 | 33 | 8 | 80 |
| GksHSP21 | Kirkii_Version3_Juiced.00g184750 | CI | 135 | 14.96507 | 5.91 | 38 | 33 | 4 | 60 |
| GksHSP22 | Kirkii_Version3_Juiced.00g188220 | CI | 143 | 16.28479 | 9.22 | 25 | 28 | 7 | 83 |
| GksHSP23 | Kirkii_Version3_Juiced.00g193540 | PX | 165 | 18.81639 | 5.98 | 18 | 40 | 8 | 99 |
| GksHSP24 | Kirkii_Version3_Juiced.00g194110 | CII | 158 | 17.75257 | 5.97 | 51 | 34 | 11 | 62 |
| GksHSP25 | Kirkii_Version3_Juiced.00g209640 | CP | 232 | 25.77139 | 6.84 | 39 | 43 | 8 | 142 |
| GksHSP26 | Kirkii_Version3_Juiced.00g215570 | CI | 159 | 18.38593 | 5.83 | 30 | 30 | 10 | 89 |
| GksHSP27 | Kirkii_Version3_Juiced.00g230720 | SINGLE | 140 | 16.28668 | 5.39 | 28 | 26 | 10 | 76 |
| GksHSP28 | Kirkii_Version3_Juiced.00g244800 | CI | 163 | 18.52218 | 8.83 | 33 | 36 | 10 | 84 |
| GksHSP29 | Kirkii_Version3_Juiced.00g246020 | CI | 156 | 17.87539 | 6.76 | 27 | 39 | 10 | 80 |
| GksHSP30 | Kirkii_Version3_Juiced.00g256930 | CI | 144 | 16.53776 | 5.58 | 26 | 34 | 8 | 76 |
| GksHSP31 | Kirkii_Version3_Juiced.00g267060 | CV | 166 | 19.59569 | 6.33 | 57 | 39 | 17 | 53 |
| GksHSP32 | Kirkii_Version3_Juiced.00g273810 | CI | 93 | 10.50390 | 4.61 | 11 | 32 | 9 | 41 |
| GksHSP33 | Kirkii_Version3_Juiced.00g301400 | PX | 141 | 15.98226 | 6.92 | 24 | 36 | 10 | 71 |
| GksHSP34 | Kirkii_Version3_Juiced.00g331020 | CP | 230 | 25.96835 | 5.86 | 40 | 38 | 9 | 143 |
| GksHSP35 | Kirkii_Version3_Juiced.00g336540 | MTI/CP | 181 | 20.93121 | 6.23 | 61 | 35 | 10 | 75 |
| GksHSP36 | Kirkii_Version3_Juiced.00g336590 | MTI/CP | 327 | 36.83181 | 5.49 | 103 | 48 | 22 | 154 |
| GksHSP37 | Kirkii_Version3_Juiced.00g343620 | CI | 149 | 17.03446 | 7.83 | 30 | 35 | 9 | 75 |
| GksHSP38 | Kirkii_Version3_Juiced.00g375140 | ER | 184 | 20.66178 | 5.76 | 57 | 33 | 8 | 86 |
| GksHSP39 | Kirkii_Version3_Juiced.00g377690 | CV | 196 | 22.48230 | 5.45 | 44 | 40 | 17 | 95 |
新窗口打开|下载CSV
2.3 叉柱棉sHSP成员的保守基序分析
在GksHSP家族中, 通过MEME程序识别出20个潜在的保守基序(图2)。利用CDD程序对这20个保守基序进行注释发现, 只有3个基序(motif 1、motif 2和motif 3)注释为sHSP保守结构域, 其他的基序暂时没有发现任何注释信息(表2)。基于这20个保守基序的分布, 可以进一步将GksHSP家族分成类似于系统发育研究中10个亚家族。motif 1、motif 2和motif 3存在于大部分的GksHSP成员中, 而其他的基序存在于特定的GksHSP亚家族。例如motif 9和motif 12只存在来自CII亚家族的GksHSP成员, motif 10和motif 18只存在来自CIV亚家族的GksHSP成员, motif 11和motif 19只存在来自CV亚家族的GksHSP成员。图2
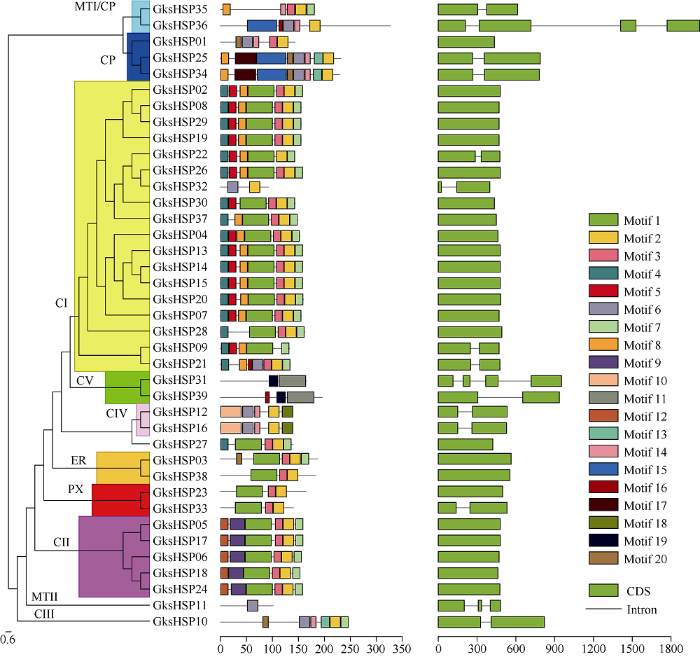 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2GksHSP成员的系统发育(左)、保守基序(中)以及基因结构(右)分析
Fig. 2Phylogenetic relationship (left), conserved motif (middle), and gene structure (right) of GkHSPs
Table 2
表2
表2通过MEME程序识别有关GksHSP成员的基序信息
Table 2
| 基序ID Motif ID | 基序序列 Motif sequence | 基序长度 Motif length (aa) |
|---|---|---|
| MEME-1 | RIDWKETPEAHVFKADVPGLKKEEVKVEVEDDRVLQISGERNVEKEDKNDT | 51 |
| MEME-2 | NAKMDQIKASMEBGVLTVTVP | 21 |
| MEME-3 | ERSSGKFMRRFRLPE | 15 |
| MEME-4 | MAMIPSFFGNRRSSI | 15 |
| MEME-5 | FDPFSLDVWDPFKDF | 15 |
| MEME-6 | HVFKADLPGLKKEEVKVEVED | 21 |
| MEME-7 | KLEVKKPDVKAIEIS | 15 |
| MEME-8 | SLSTRSPETSAFVNA | 15 |
| MEME-9 | MLDIPDETEKSPNAPSRAYVRDAKAMAAT | 29 |
| MEME-10 | MEFPFPSDQHSPLYHYLLPSPPLFSNQLLPENHLNWTQTP | 40 |
| MEME-11 | QQREGKKKDWRSCNWWEYGYVRRLELPZDADWRKIEAFLSNDVVLEIRIPRN | 52 |
| MEME-12 | MDFRIMGFDSPLLHT | 15 |
| MEME-13 | WTNRSYSSYBTSLQLPD | 17 |
| MEME-14 | VLVIKGERKEE | 11 |
| MEME-15 | QERAVEKRPKRLAMDVSPFGLLDPLSPMRSMRQMLDTMDRIFEDAMIFPGSNRRQGG | 57 |
| MEME-16 | DWKETPEA | 8 |
| MEME-17 | PFSVSFPSKNPCNSRLSVVRAZAAGDNNKDTSVDVHVNKDN | 41 |
| MEME-18 | GSFYIDPADVPDRVEVLARAA | 21 |
| MEME-19 | NIQIHVEKGKIMEIFGQ | 17 |
| MEME-20 | RAPWDIKDGEH | 11 |
新窗口打开|下载CSV
2.4 叉柱棉sHSP成员的基因结构分析
本研究对GksHSP成员的基因结构进行分析, 根据外显子和内含子的数量, 可以将GksHSP成员的基因结构分为4种不同的类型(图2)。第1种类型包括24个GksHSP成员, 它们均只含有1个外显子, 没有内含子。第2种类型包括2个外显子和1个内含子, 有12个GksHSP成员属于这种类型。除此以外, GksHSP11包括3个外显子和2个内含子, GksHSP31和GksHSP36包括4个外显子和3个内含子。不同亚家族的基因结构具有一定的保守性, 如CII亚家族和ER亚家族的GksHSP成员只含有第1种类型的基因结构, CIV亚家族和MTII亚家族的GksHSP成员只含有第2种类型的基因结构。2.5 叉柱棉sHSP成员的染色体定位分析
本研究鉴定的39个GksHSP成员定位在叉柱棉的11条染色体上, 但在叉柱棉的KI_01染色体上不存在任何GksHSP成员(图3)。39个GksHSP成员并非平均分布在这11条染色体上。其中KI_05染色上包含最多的GksHSP成员(11个), 其次是KI_07染色体(6个)以及KI_12染色体(4个)。KI_06、KI_09和KI_10染色体上各包含3个GksHSP成员, KI_2_4、KI_03、KI_08和KI_13染色体上各包含2个GksHSP成员, 而KI_11染色体上仅包括1个GksHSP成员。另外, 一些GksHSP成员在叉柱棉的染色体上集中存在, 例如KI_05染色体的上半部和下半部以及KI_07染色体的下半部。图3
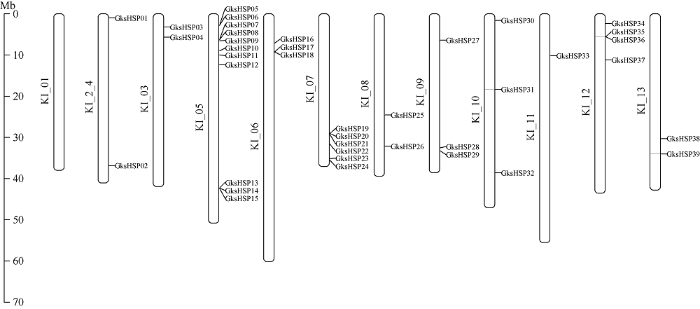 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图3GksHSP成员在叉柱棉12条染色体上的分布
Fig. 3Chromosomal locations of GkHSPs on Gossypioides kirkii 12 chromosomes
2.6 叉柱棉sHSP成员的基因复制事件分析
在叉柱棉中通过MCScan软件共识别出7个与sHSP成员有关的基因复制事件(表3), 复制的GksHSP成员主要位于KI_05、KI_06、KI_07、KI_08以及KI_12染色体上(图4-A)。鉴于相同复制事件中复制GksHSP基因位于不同的染色体上, 所以在叉柱棉中所有的与sHSP成员有关的复制事件均为片段复制事件, 而不是串联复制事件。在GksHSP家族中, 7个片段复制事件主要分布在CI亚家族、CII亚家族和CP亚家族中, 其中CII亚家族包含4个基因复制事件, CI亚家族包含2个基因复制事件, CP亚家族包含1个基因复制事件(图4-B)。本研究对这些基因复制事件的Ks值分布进行计算发现, 大部分的基因复制事件的Ks值集中分布在0.731左右, 仅有1对复制基因(GksHSP06/GksHSP24)的Ks值为1.04 (图4-C)。同时, 本研究对这些基因复制事件的Ka/Ks比值进行计算发现, 所有的基因复制事件的Ka/Ks比值均小于0.25。Table 3
表3
表3叉柱棉中复制的sHSP成员的Ka和Ks分析
Table 3
| 复制基因1 Duplicated gene 1 | 复制基因2 Duplicated gene 2 | 亚家族 Subfamily | 非同义 替换率 Ka | 同义替换率 Ks | 非同义替换率/同义替换率 Ka/Ks | 纯化选择 Purifying selection | 复制类型 Duplicated type |
|---|---|---|---|---|---|---|---|
| GksHSP05 | GksHSP17 | CII | 0.063959 | 0.809673 | 0.078994 | 是Yes | 片段复制事件 Segmental |
| GksHSP06 | GksHSP18 | CII | 0.090699 | 0.742610 | 0.122136 | 是Yes | 片段复制事件Segmental |
| GksHSP07 | GksHSP20 | CI | 0.045458 | 0.629437 | 0.072220 | 是Yes | 片段复制事件Segmental |
| GksHSP09 | GksHSP21 | CI | 0.159061 | 0.715073 | 0.222441 | 是Yes | 片段复制事件Segmental |
| GksHSP06 | GksHSP24 | CII | 0.113536 | 1.039721 | 0.109199 | 是Yes | 片段复制事件Segmental |
| GksHSP18 | GksHSP24 | CII | 0.103983 | 0.730373 | 0.142370 | 是Yes | 片段复制事件Segmental |
| GksHSP25 | GksHSP34 | CP | 0.125830 | 0.606857 | 0.207347 | 是Yes | 片段复制事件Segmental |
新窗口打开|下载CSV
图4
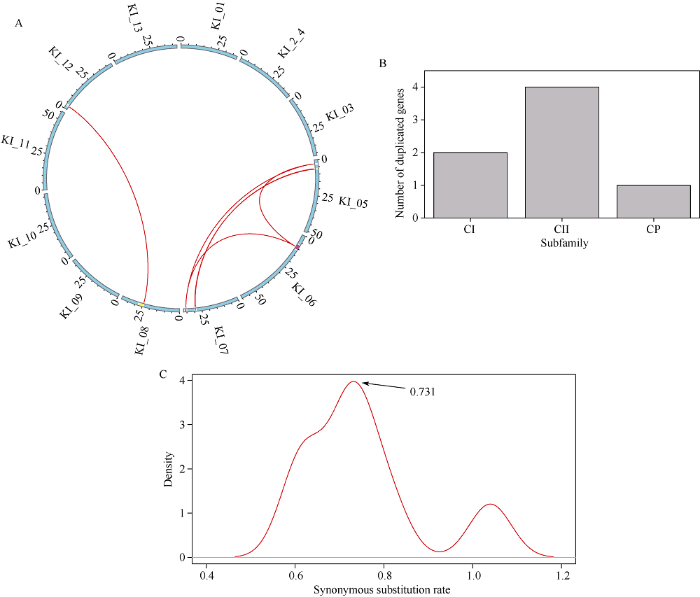 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图4GksHSP成员的基因复制事件分析
A: 复制的GksHSP成员分析, 与GksHSP家族有关的复制框使用不同的颜色在染色体上展示。B: 复制事件在不同亚家族的分布。C: 复制的GksHSP同义替换率分布。
Fig. 4Gene duplication analysis of GkHSP members
A: duplicated relationship of GkHSP members, the duplicated blocks were shown by the colors. B: the distribution of the duplication events in different subfamilies. C: the distribution of synonymous substation rate about duplicated GksHSPs.
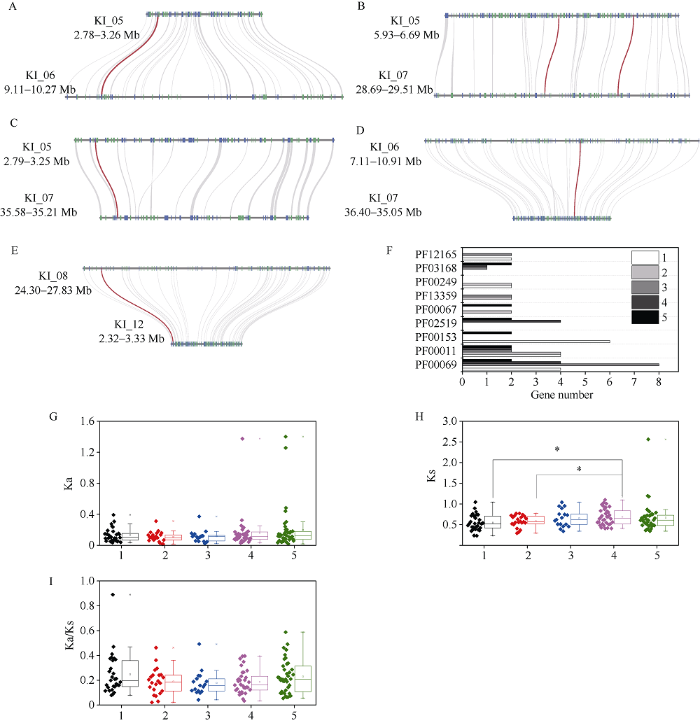
在叉柱棉中, 7个GksHSP基因复制事件主要与5个复制框(复制框1~复制框5)有关, 其中复制框1和2均包括2个基因复制事件, 另外3个复制框只包括1个基因复制事件(图5-A~E)。随后, 对这些复制框中的基因进行注释发现, 复制框中的基因除了包含11个sHSP成员(PF00011)外, 还主要包含15个蛋白激酶成员(PF00069)、8个线粒体载体蛋白(PF00153)以及6个植物生长素响应蛋白(PF02519) (图5-F)。其中复制框1主要包括6个线粒体载体蛋白、4个sHSP成员和4个蛋白激酶成员, 复制框2主要包括4个sHSP成员, 复制框3主要包括8个蛋白激酶成员和2个sHSP成员, 复制框4主要包括4个蛋白激酶成员、4个植物生长素响应蛋白和2个sHSP成员, 复制框5主要包括2个蛋白激酶成员、2个线粒体载体蛋白、2个sHSP成员和2个蛋白激酶成员。此外, 对这5个复制框的Ka值、Ks值和Ka/Ks比值进行计算发现, 这5个复制框的Ka值和Ka/Ks比值彼此相似, 但对于Ks值而言, 复制框1和复制框2的Ks值显著低于复制框4的Ks值, 其他复制框的Ks值类似(图5-G~I)。
图5
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图5与GksHSP家族有关的复制框分析
A~E: 与GksHSP家族有关的复制框1 (A)、复制框2 (B)、复制框3 (C)、复制框4 (D)和复制框5 (E)的微共线分析。F: 与GksHSP家族有关的5个复制框的基因注释分布。G~I: 与GksHSP家族有关的5个复制框的Ka(G)、Ks(H)和Ka/Ks(I)分析。*表示在P < 0.05水平上差异显著。
Fig. 5Duplicated block analysis of the GkHSP family
A-E: microsyntenic analysis of duplicated block 1 (A), duplicated block 2 (B), duplicated block 3 (C), duplicated block 4 (D), and duplicated block 5 (E). F: the distribution of gene annotation about the five duplicated blocks in the GkHSP family. G-I: Ka (G), Ks (H), and Ka/Ks (I) analysis of the five duplicated blocks in the GkHSP family. * represents significant difference at P < 0.05.
2.7 叉柱棉sHSP成员启动子区的顺式调控元件分析
本研究利用PlantCARE数据库对GksHSP成员启动子区的顺式调控元件进行分析发现, 所有的GksHSP成员除了包含真核生物共有的顺式调控元件(如TATA框和CAAT框)外, 还包括一些与胁迫响应相关的顺式调控元件(图6)。所有的GksHSP成员均包含至少一种与胁迫响应相关的顺式调控元件, 其中GksHSP26包含最多的与胁迫响应相关的顺式调控元件(13个), 其次为GksHSP16 (11个)、GksHSP06 (10个)、GksHSP10 (10个)和GksHSP35 (10个)。在GksHSP家族中, 最多的与胁迫响应相关的顺式调控元件为ABA响应元件(64个), 其次为茉莉酸甲酯响应元件(47个)、赤霉素响应元件(36个)、低温响应元件(27个)、水杨酸响应元件(24个)、MYB结合位点(22个)和生长素响应元件(21个), 最少的与胁迫响应相关的顺式调控元件为防御/胁迫响应元件(15个)。此外, 来自相同亚家族的GksHSP成员的启动子区通常包含类似的顺式调控元件。例如大部分CI亚家族的GksHSP成员的启动子区包含ABA响应元件和茉莉酸甲酯响应元件, 大部分CII亚家族的GksHSP成员的启动子区包含ABA响应元件, CP亚家族的GksHSP成员的启动子区均包含低温响应元件。图6
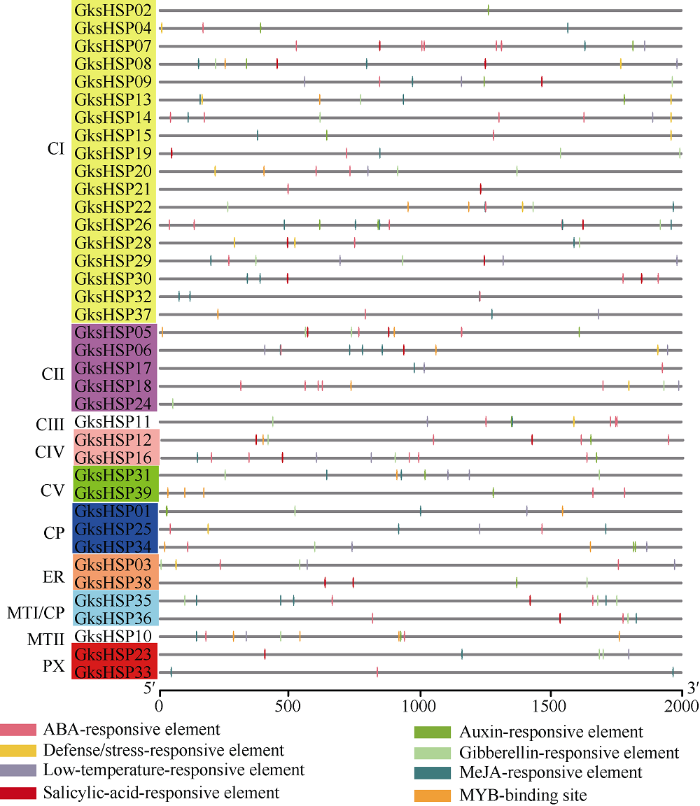 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图6GksHSP成员的启动子区中与胁迫响应相关的顺式调控元件分析
GksHSP亚家族用不用的颜色和字母表示。每一个颜色的方框代表一种顺式调控元件。
Fig. 6Stress-related regulatory elements in the promoter regions of GksHSPs
The GksHSP subfamilies were denoted by using different colors and letters. Each colored box represents a cis-regulatory element.
2.8 陆地棉中GksHSP26的同源基因在高温、干旱和盐胁迫下的表达分析
本研究利用CottonFGD数据库对启动子中包含最多的与胁迫响应相关的顺式调控元件的GksHSP26在陆地棉中的同源基因进行识别发现, 在陆地棉中Gh_D08G181200和Gh_A08G183800是叉柱棉GksHSP26的同源基因(相似度均大于94%) (图7-A)。随后在高温、干旱和盐胁迫条件下对Gh_D08G181200和Gh_A08G183800的表达量进行分析发现, 在高温和盐胁迫下Gh_D08G181200和Gh_A08G183800的表达量均显著上调, 尤其是高温处理6 h和盐胁迫处理1 h其表达量达到最大值, 但对干旱胁迫响应并不明显(图7-B, C)。图7
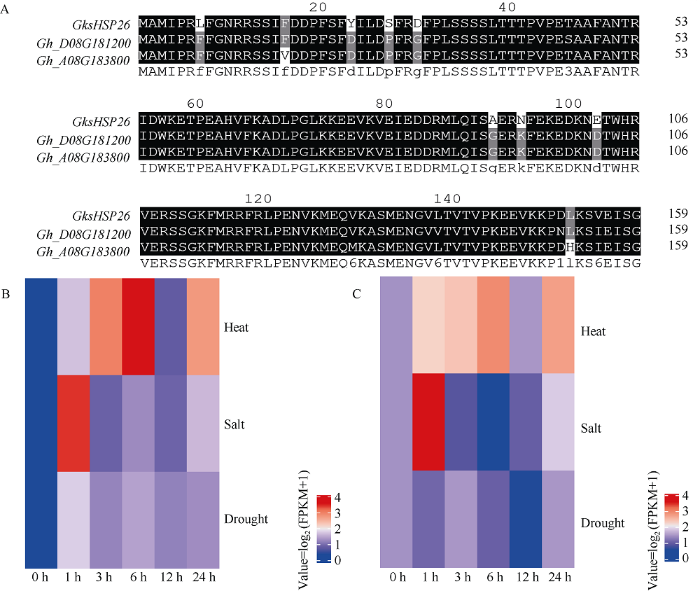 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图7在陆地棉中GksHSP26同源基因的识别和表达分析
A: GksHSP26与其在陆地棉中的同源基因Gh_D08G181200和Gh_A08G183800的氨基酸序列分析。B~C: 在高温、干旱和盐胁迫下Gh_D08G181200 (B)和Gh_A08G183800 (C)的表达分析。
Fig. 7Identification and expression levels of the orthologous genes of GksHSP26 in Gossypium hirsutum
A: alignment analysis of GksHSP26 and its corresponding orthologous genes in Gossypium hirsutum (Gh_D08G181200 and Gh_A08G183800). B-C: heat map representation of Gh_D08G181200 (B) and Gh_A08G183800 (C) expression levels under heat, salt, and drought stresses.
3 讨论
本研究从叉柱棉中识别出39个GksHSP成员(表1), 低于许多双子叶植物中sHSP成员数量, 如番茄(43个)[11]、马铃薯(48个)[12]、棉花(94个)[13]以及小麦(109个)[24]等。叉柱棉中识别出较少的sHSP成员可能与该物种本身基因组(544 Mb)较小有关[19]。本研究识别的每一个GksHSP成员都具有sHSP家族保守的α-晶体结构域、分子量介于10.5039 kD到36.83181 kD之间、基因结构比较简单(大部分外显子数量少于2个)等特征(表1和图2), 这些特征也出现在其他植物的sHSP成员中[5,6]。GksHSP家族通过系统发育分析可以进一步分为10个亚家族(图1), 不同的亚家族含有不同数量的GksHSP成员, 其中CI亚家族和CII亚家族含有最多的GksHSP成员, CIII亚家族和MTII亚家族含有最少的GksHSP成员。在sHSP家族中, 相似的亚家族分布也出现在拟南芥[9]、水稻[10]、番茄[11]等植物中。在GksHSP家族中, 其保守基序、基因结构和启动子区的顺式调控元件等的分布也与系统发育分析中的分布相一致(图2和图6)。另外, GksHSP成员在KI_05染色体的上半部和下半部以及KI_07染色体的下半部中集中存在, 表明这些染色体区段可能是与GksHSP家族有关的热点区域(图3), 相似的现象也出现在玉米NAC家族[25]、棉花LTP家族[26]、木薯GRAS家族[27]等中。此外, 本研究在GksHSP家族中共发现7个基因复制事件, 而这些基因复制事件并非平均分布在整个亚家族中(表3和图4-B), 其中CII亚家族(4个)和CI亚家族(2个)包括大部分的基因复制事件。CI亚家族和CⅡ亚家族具有较多的基因复制事件, 这是导致这2个亚家族含有更多GksHSP成员的一个原因。类似的基因复制事件导致不同亚家族含有不同成员的现象也出现在叉柱棉的近缘物种棉花的NAC家族[28]、KUP家族[22]、PEL家族[29]等基因家族中。在GksHSP家族中, 所有的复制事件均与片段复制事件有关, 并没有发生任何串联复制事件(表3、图3和图4-A), 这可能与叉柱棉中由转座子活性等导致基因组的片段化、基因丢失等现象有关[30,31,32]。本研究发现的7个片段复制事件均位于5个复制框中(图4-A和图5-A~E), 表明GksHSP家族的扩增主要与叉柱棉的全基因组复制事件有关[19]。通过Ks计算发现, 大部分的片段复制事件的Ks值集中位于0.731左右(表3和图4-C), 这个时间正好对应于棉属特有的全基因组复制事件所发生的时间[33]。因此, GksHSP成员的扩增主要发生在棉属特有的全基因组复制事件中。与此同时, 本研究对与GksHSP家族扩增有关的复制框区域进行详细的分析发现, 这5个复制框中含有较多的蛋白激酶成员、线粒体载体蛋白以及植物生长素响应蛋白(图5-F)。目前很多研究已发现蛋白激酶、线粒体载体蛋白以及植物生长素响应蛋白均与胁迫响应相关[34,35,36]。例如拟南芥的蛋白激酶CDPK27成员与植株的抗盐性有关[37], 南美杉的线粒体载体蛋白PUMP成员可以响应植株的冷害胁迫[38], 过表达小麦的生长素响应蛋白TaSAUR75可以提高拟南芥植株的干旱和盐渍抗性[39]。因此, 在叉柱棉中, GksHSP家族的进化可能与蛋白激酶家族、线粒体载体蛋白家族以及植物生长素响应蛋白家族有关。此外, 本研究发现, 复制框1和复制框2的Ks值显著低于复制框4的Ks值(图5-G~I), 并且不同的复制框中所包含的成员种类也是不一样的(图5-F), 表明在叉柱棉中与GksHSP家族有关的不同复制框的进化速率显著不同。
启动子区的顺式调控元件在生物学功能及其调控过程中具有非常关键的作用[40]。本研究发现, 在叉柱棉中GksHSP成员的启动子区包含大量的与胁迫响应相关顺式调控元件, 这表明GksHSP成员可能与植物的胁迫响应相关(图6)。在所有与胁迫响应相关的顺式调控元件中, 最多的顺式调控元件为ABA响应元件(64个), 其次为茉莉酸甲酯响应元件(47个), 表明大部分GksHSP成员可能与ABA和茉莉酸甲酯调控的胁迫响应相关, 目前已经在马铃薯[12]、小麦[12]、簸箕柳[15]等植物的sHSP成员的启动子区发现类似的顺式调控元件分布。此外, 在GksHSP家族中, 来自CⅠ亚家族的GksHSP26包含最多的与胁迫响应相关的顺式调控元件, 并且在陆地棉中识别了GksHSP26的同源基因Gh_D08G181200和Gh_ A08G183800, 发现在高温和盐胁迫下这2个同源基因的表达量显著上调(图7), 其在拟南芥中的同源基因AtHSP17.4[41]和在水稻中的同源基因OsHsp17.0[42]也已经报道了可以响应多种胁迫。
4 结论
在叉柱棉中共鉴定了39个GksHSP成员, 并且可以将GksHSP成员根据系统发育关系进一步分为10个亚家族, 每个亚家族都具有保守的基序、类似的基因结构以及相似的启动子区的顺式调控元件分布。在叉柱棉中, 棉属特有的全基因组复制事件主要导致了GksHSP成员的扩增, 与此同时其扩增还与蛋白激酶家族、线粒体载体蛋白家族以及植物生长素响应蛋白家族有关。此外, GksHSP家族可能与ABA和茉莉酸甲酯调控的胁迫响应相关, 尤其是GksHSP26可以作为叉柱棉抗逆育种的一个重要候选基因。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
PMID [本文引用: 1]

The WRKY proteins are a superfamily of transcription factors with up to 100 representatives in Arabidopsis. Family members appear to be involved in the regulation of various physio-logical programs that are unique to plants, including pathogen defense, senescence and trichome development. In spite of the strong conservation of their DNA-binding domain, the overall structures of WRKY proteins are highly divergent and can be categorized into distinct groups, which might reflect their different functions.
PMID [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
URL [本文引用: 3]
URL [本文引用: 3]
DOIURL [本文引用: 4]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 2]
DOIURL [本文引用: 2]
[本文引用: 3]
[本文引用: 3]
DOIURL [本文引用: 4]
DOIURL [本文引用: 2]
URL [本文引用: 1]
[本文引用: 1]
DOIURL [本文引用: 2]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
PMID [本文引用: 1]

Owing to their sessile lifestyle, it is crucial for plants to acquire stress tolerance. The function of heat-shock proteins, including small heat-shock proteins (smHSPs), in stress tolerance is not fully explored. To gain further knowledge about the smHSPs, the gene that encoded the cytosolic class II smHSP in Arabidopsis thaliana (At-HSP17.6A) was characterized. The At-HSP17.6A expression was induced by heat and osmotic stress, as well as during seed development. Accumulation of At-HSP17.6A proteins could be detected with heat and at a late stage of seed development, but not with osmotic stress, suggesting stress-induced post-transcriptional regulation of At-HSP17.6A expression. Overproduction of At-HSP17.6A could increase salt and drought tolerance in Arabidopsis. The chaperone activity of At-HSP17.6A was demonstrated in vitro.
DOIURL [本文引用: 4]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 3]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIPMID [本文引用: 1]

The recent increase in genomic data is revealing an unexpected perspective of gene loss as a pervasive source of genetic variation that can cause adaptive phenotypic diversity. This novel perspective of gene loss is raising new fundamental questions. How relevant has gene loss been in the divergence of phyla? How do genes change from being essential to dispensable and finally to being lost? Is gene loss mostly neutral, or can it be an effective way of adaptation? These questions are addressed, and insights are discussed from genomic studies of gene loss in populations and their relevance in evolutionary biology and biomedicine.
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
DOIPMID [本文引用: 1]

Mitochondria play a key role in respiration and energy production and are involved in multiple eukaryotic but also in several plant specific metabolic pathways. Solute carriers in the inner mitochondrial membrane connect the internal metabolism with that of the surrounding cell. Because of their common basic structure, these transport proteins affiliate to the mitochondrial carrier family (MCF). Generally, MCF proteins consist of six membrane spanning helices, exhibit typical conserved domains and appear as homodimers in the native membrane. Although structurally related, MCF proteins catalyze the specific transport of various substrates, such as nucleotides, amino acids, dicarboxylates, cofactors, phosphate or H+. Recent investigations identified MCF proteins also in several other cellular compartments and therefore their localization and physiological function is not only restricted to mitochondria. MCF proteins are a characteristic feature of eukaryotes and bacterial genomes lack corresponding sequences. Therefore, the evolutionary origin of MCF proteins is most likely associated with the establishment of mitochondria. It is not clear whether the host cell, the symbiont, or the chimerical organism invented the ancient MCF sequence. Here, we try to explain the establishment of different MCF proteins and focus on the characteristics of members from plants, in particular from Arabidopsis thaliana.
URL [本文引用: 1]
URL [本文引用: 1]
DOIPMID [本文引用: 1]

Ca(2+)-dependent protein kinases (CDPKs) play vital roles in plant adaptations to environmental challenges. The precise regulatory mechanism of CDPKs in mediating salt stress still remains unclear, although several CDPK members have been identified to be involved in salt stress accumulation in various plants, such as Arabidopsis thaliana and Oryza sativa. Here, we investigated the function of an Arabidopsis CDPK, CPK27, in salt stress-signaling. CPK27 is a membrane-localized protein kinase; its expression was induced by NaCl. cpk27-1, a T-DNA insertion mutant of CPK27, was much more sensitive to salt stress than wild-type plants in terms of seed germination and post-germination seedling growth. In ion-flux assay, cpk27-1 mutants exhibited a lower capacity than wild-type plants to extrude Na(+) and import H(+) after a long-term salt treatment (110mM NaCl for 10days). Moreover, the content of Na(+) was higher and K(+) was lower in cpk27-1 mutants than in wild-type plants under salt stress. In addition, the level of salt-elicited H2O2 production was higher in cpk27-1 mutants than in wild-type plants Col after a short-term NaCl shock and long-term salt treatment. Collectively, our results suggest that CPK27 is required for plant adaptation to salt stress.Copyright © 2015 Elsevier B.V. All rights reserved.
DOIURL [本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]
URL [本文引用: 1]
DOIURL [本文引用: 1]
DOIURL [本文引用: 1]
