 ,1,*, 张耀元1,*, 张俊杰1, 胡卫国3, 李俊4, 王长有1, 张宏1, 陈春环11.
,1,*, 张耀元1,*, 张俊杰1, 胡卫国3, 李俊4, 王长有1, 张宏1, 陈春环11. 2.
3.
4.
Genetic effects of important yield traits analysed by mixture model of major gene plus polygene in wheat
XIE Song-Feng1,2,*, JI Wan-Quan ,1,*, ZHANG Yao-Yuan1,*, ZHANG Jun-Jie1, HU Wei-Guo3, LI Jun4, WANG Chang-You1, ZHANG Hong1, CHEN Chun-Huan11.
,1,*, ZHANG Yao-Yuan1,*, ZHANG Jun-Jie1, HU Wei-Guo3, LI Jun4, WANG Chang-You1, ZHANG Hong1, CHEN Chun-Huan11. 2.
3.
4.
通讯作者:
收稿日期:2019-07-1接受日期:2019-09-26网络出版日期:2020-03-12
| 基金资助: |
Received:2019-07-1Accepted:2019-09-26Online:2020-03-12
| Fund supported: |
作者简介 About authors
解松峰,E-mail:

摘要
关键词:
Abstract
Keywords:
PDF (2720KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
解松峰, 吉万全, 张耀元, 张俊杰, 胡卫国, 李俊, 王长有, 张宏, 陈春环. 小麦重要产量性状的主基因+多基因混合遗传分析[J]. 作物学报, 2020, 46(3): 365-384. doi:10.3724/SP.J.1006.2020.91044
XIE Song-Feng, JI Wan-Quan, ZHANG Yao-Yuan, ZHANG Jun-Jie, HU Wei-Guo, LI Jun, WANG Chang-You, ZHANG Hong, CHEN Chun-Huan.
小麦是世界上最重要的三大主粮作物之一, 其17%的种植面积比例提供的粮食超过了30%的全球人口供给及20%的能量消耗(
小麦单株产量、株高、千粒重、分蘖数等重要产量性状一直是小麦育种的重要目标性状。多年以来, 以重要产量性状为代表的复杂数量性状一直受到育种家的重视, 但这些性状又容易受到环境条件影响, 即使相同的遗传材料在多个环境下作遗传分析得出的结果也不尽相同。因此, 应用永久群体材料进行有重复的比较试验显得尤为关键, 尤其是以重组自交系群体(RIL)为代表, 比较适合对环境影响较大的复杂性状作遗传研究。数量遗传学与植物育种相结合, 对数量性状遗传体系的认识也提高到了 新的阶段。盖钧镒[3]提出了泛主基因加多基因理论即控制数量性状的基因数目有多有少, 各对基因效应大小不同且容易受环境影响。
近年来, 数量性状主基因-多基因遗传分离分析体系[3,4,5,6,7]在各类植物育种研究上得到了广泛的应用, 如在水稻稻曲病抗性及穗部性状[8,9]、油菜农艺性状及耐湿性[10,11,12,13,14]、甜荞株高和茎粗[15]、黄瓜节间长和叶面积[16,17]、辣椒花期及花节位[18]、大白菜的产量性状[19]等方面, 初步得到各类植物育种性状的遗传组成, 为粮油作物和园艺作物等植物提取了有意义的遗传信息。但迄今, 对多个环境下小麦重要产量性状的主基因加多基因遗传分析的研究较少。本课题研究组在以品冬34为母本和BARRAN为父本构建重组自交系基础上, 利用2年4个环境下P1、P2、F7:8和F8:9重组自交系, 共476份衍生的家系, 初步分析了各性状的遗传相关性, 并通过运用主基因+多基因混合遗传模型的方法, 研究重要产量性状相关的6个性状的遗传方差和基因效应。以期揭示重要产量性状的遗传机制, 为下一步研究利用及制订相应的育种策略, 揭示重要产量性状遗传组成及遗传机制奠定基础并提供理论依据。
1 材料与方法
1.1 试验材料
以品冬34为母本(P1)和BARRAN为父本(P2)配制杂交组合, 品冬34由中国农业科学院作物品种资源研究所选育, 主要特性为矮秆、千粒重高、籽粒大。BARRAN是引进由国外的种质, 粒小、千粒重较低。课题组从2007年秋开始在西北农林科技大学小麦试验基地种植亲本, 2008年春人工去雄授粉杂交得到F1 (P1×P2)籽粒。同年, 将亲本种在基地内, 以此类推, 采用单籽粒传递方法, 于2016年和2017年构建了476份衍生的F7:8、F8:9重组自交系。1.2 田间试验
2016年9月, 将亲本和RIL群体籽粒同期播种在陕西杨凌西北农林科技大学农学试验基地(环境E1: 2016SY), 2017年9月, 再将亲本和RIL群体分别播种在陕西杨凌西北农林科技大学节水农业试验农场(环境E2: 2017SY)、河南省农业科学院原阳现代农业示范基地(环境E3: 2017HY)、四川省农业科学院广汉小麦育种基地(环境E4: 2017SG), 行距24 cm, 株距10 cm, 每行20株, 试验地前茬为空闲地, 田间管理和普通大田生产基本相同。种植P1、P2各6行, RIL衍生群体家系各2行。1.3 重要产量性状调查
2017年至2018年5月中旬避开行边株分单株收获群体考察表型, 参考李立会等[20]的方法, 对每个家系调查10株, 株高(PH)为成熟收割前田间测量从地面至穗顶(不包括芒)的高度。主穗基部小穗节至穗下第一节间的长度为穗下节间长(SIL); 旗叶基部至主穗基部的长度为旗叶上节间长(FIL); 小麦成熟时单株成穗数为有效分蘖数(NTP); 单株总实粒(含标准含水量)的重量为单株产量(GYP); 千粒重(TGW)为随机取2份1000粒重量的平均值晒干(含水量不超过13%)、去除杂质籽粒。1.4 重要数据统计分析
利用SPSS Statistics 17.0软件(SPSS, Chicago, IL, USA)进行表型数据、偏度及峰度系数估计值基本统计。RIL群体中各基因位点的遗传方差等于加性方差, 其广义遗传力等于狭义遗传力, 公式为hB2 = Va/(Va+Ve); Va为加性方差, Ve为环境方差。根据Choo和Reinbergs[21]提出的基因间互作方式分析原理, 由各性状偏度系数(g1)、峰度系数(g2)的正负号及其相对大小可以估计基因的上位性互作及其互作方式; 利用R软件包绘制频率分布直方图、邦弗朗尼(Bonferroni)相关性及线性拟合线。邦弗朗尼校正(Bonferroni correction)的概念重点指的是给出一个在统计上可行的方法来避免在搜索数据时出现的大部分“臆造”正响应。使用邦弗朗尼事后检验法分析数据显著性时, 当纠正P值小于等于0.05时, 表示差异显著; P值小于等于0.01表示差异极显著。利用章元明教授团队最新开发的主基因+多基因混合遗传分析R软件包SEA (https://cran.r-Project. org/web/Packages/SEA/index.html)分析P1、P2和RIL群体在每一环境下的数量性状表型观察值, 获得7类38个遗传模型[2, 5]的极大似然函数值(maximum likelihood method, MLV)和AI (Akaike’s information criterion, AIC)值; 选出AIC值最小或接近最小的几个备选遗传模型, 进行群体次数分布与模型理论分布的均匀性检验(U12、U22和U32)、Smirnov检验(nW2)和Kolmogorov检验(Dn), 根据这些检验结果与AIC值选出最优遗传模型; 通过SEA软件包计算出最优遗传模型的遗传效应等一阶遗传参数、遗传方差和遗传率等二阶遗传参数, 其中σ2P、σ2mg和σ2Pg分别是群体表型方差、主基因和多基因遗传方差; h2mg和h2Pg分别是主基因和多基因遗传率。
2 结果与分析
2.1 重要产量性状表型数据分析
由表1可知, 在4个环境下, 亲本(P1和P2)的单株产量分别是28.04 g和13.01 g、千粒重分别是60.63 g和35.74 g、株高分别是88.73 cm和94.46 cm、分蘖数分别是19.19和21.67、穗下节间长分别是33.38和33.49、旗叶上节间长分别是11.31和18.29。群体平均值依次是单株产量22.54 g 株-1, 介于双亲之间, 高于低值亲本; 千粒重44.08 g, 介于双亲中间, 略高于低值亲本; 株高103.10, 高于高值亲本; 分蘖20.22, 介于双亲之间, 略高于低值亲本, 略低于高值亲本; 穗下节间长38.82, 高于高值亲本; 旗叶上节间长18.53, 略高于高值亲本。重要产量性状的变异系数(CV)揭示了群体的数量性状之间的很大分离(15.75%~59.26%)分别为: 4个环境下各性状平均变化极值范围株高为38.43~156.01、穗下节间长为16.42~62.15、旗叶上节间长为2.33~38.30、有效分蘖数为5.46~53.25、单株产量为1.79~68.48和千粒重为19.55~65.89, 表型变异幅度大, 适合遗传分析, 此外, 所有平均变异系数均大于15%。单株产量的变异系数(48.11%)大于千粒重的变异系数(39.99%), 表明单株产量在形态变异上比在千粒重性状更丰富。平均变异系数从高到低为单株产量(48.11%)、旗叶上节间长(31.27%)、有效分蘖数(28.37%)、千粒重(39.99%)、穗下节间长(20.83%)、株高(19.58%)和千粒重(17.10%)。这些结果表明, 单株产量的变异在形态上比在农艺学表型中更为突出; 同时, 在2年4个生态环境条件下, 小麦RIL群体重要产量性状偏度系数在0.43~1.94之间, 峰度系数在-0.39~4.63之间。其中, 偏度和峰度位于0~1间, 表型近似正态分布的有E3环境下的单株产量、E3环境下的有效分蘖数、E1和E2环境下的旗叶上节间长。在4个环境下各性状的遗传力在0.33~0.94之间, 各性状的平均遗传力较高, 株高的遗传力最高, 达0.92, 其次是穗下节间长遗传力0.88, 旗叶上节间长遗传力 0.81, 单株产量遗传力0.71, 分蘖数遗传力是0.60, 千粒重遗传力是0.52; E1、E2和E3环境下的千粒重, E1、E2、E3和E4环境下的株高, E1、E2和E4环境下的穗下节间长, E4环境下的旗叶上节间长这些性状的偏度系数和峰度系数都无显著差异, 表明基因间无互作; E1、E2、E3和E4 4个环境下的单株产量、有效分蘖数及E1、E2环境下的旗叶上节间长, 这些性状的偏度和峰度都显著大于0, 表明基因间存在互作。Table 1
表1
表1重组自交系及其亲本穗部性状最佳线性无偏预测描述性分析
Table 1
| 环境Environ. | 亲本Parent | 重组自交系群体 RIL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 品东34 Pindong 34 | Warran MY11847 | 最小值 Min. | 最大值 Max. | 平均数 Average | 标准差 SD | 变异系数CV (%) | 遗传力 Heritability | 偏度系数 Skew. | 峰度系数 Kurt. | ||
| 单株产量GYP (g) | |||||||||||
| E1 | 36.45 | 5.53 | 1.18 | 89.83 | 24.32 | 14.41 | 0.59 | 0.86 | 1.02** | 1.55** | |
| E2 | 18.66 | 6.24 | 2.03 | 65.55 | 23.31 | 9.93 | 0.43 | 0.47 | 0.66** | 1.11** | |
| E3 | 28.50 | 15.89 | 2.85 | 71.32 | 25.56 | 11.03 | 0.43 | 0.77 | 0.66** | 0.82** | |
| E4 | 28.56 | 24.38 | 1.10 | 47.20 | 16.97 | 8.00 | 0.47 | 0.75 | 0.48** | 0.23** | |
| Average | 28.04 | 13.01 | 1.79 | 68.48 | 22.54 | 10.84 | 0.48 | 0.71 | 0.705** | 0.9275** | |
| 千粒重TGW (g) | |||||||||||
| E1 | 55.85 | 26.00 | 23.25 | 66.50 | 45.12 | 7.43 | 0.16 | 0.33 | 0.09 | -0.29 | |
| E2 | 58.40 | 24.65 | 24.18 | 66.17 | 45.01 | 7.09 | 0.16 | 0.37 | -0.08 | -0.26 | |
| E3 | 57.08 | 24.15 | 18.90 | 59.70 | 38.30 | 7.37 | 0.19 | 0.76 | -0.10 | -0.20 | |
| E4 | 71.20 | 68.15 | 11.88 | 71.20 | 47.89 | 8.27 | 0.17 | 0.63 | -0.25 | 0.35** | |
| Average | 60.63 | 35.74 | 19.55 | 65.89 | 44.08 | 7.54 | 0.17 | 0.52 | -0.08 | -0.10 | |
| 株高 PH (cm) | |||||||||||
| E1 | 88.00 | 86.00 | 30.00 | 158.00 | 96.51 | 19.09 | 0.20 | 0.91 | -0.21 | -0.07 | |
| E2 | 86.50 | 90.50 | 50.80 | 161.80 | 108.26 | 22.05 | 0.20 | 0.92 | -0.18 | -0.39 | |
| E3 | 85.75 | 99.33 | 39.25 | 149.25 | 102.88 | 18.74 | 0.18 | 0.94 | -0.43 | 0.02 | |
| E4 | 94.67 | 102.00 | 33.67 | 155.00 | 104.76 | 20.89 | 0.20 | 0.92 | -0.35 | -0.24 | |
| Average | 88.73 | 94.46 | 38.43 | 156.01 | 103.10 | 20.19 | 0.20 | 0.92 | -0.29 | -0.17 | |
| 分蘖数NTP | |||||||||||
| E1 | 28.00 | 16.00 | 3.00 | 79.00 | 20.48 | 7.33 | 0.36 | 0.57 | 1.94** | 2.54** | |
| E2 | 15.75 | 18.25 | 6.50 | 54.00 | 22.02 | 5.63 | 0.26 | 0.42 | 1.09** | 3.30** | |
| E3 | 23.00 | 35.50 | 9.00 | 59.00 | 28.60 | 7.43 | 0.26 | 0.90 | 0.58** | 0.56** | |
| E4 | 10.00 | 17.00 | 3.33 | 21.00 | 9.76 | 2.55 | 0.26 | 0.44 | 0.96** | 2.10** | |
| Average | 19.19 | 21.69 | 5.46 | 53.25 | 20.22 | 5.74 | 0.28 | 0.60 | 1.14** | 4.63** | |
| 穗下节间长SIL (cm) | |||||||||||
| E1 | 36.00 | 34.00 | 16.00 | 66.00 | 40.40 | 9.12 | 0.23 | 0.89 | 0.00 | -0.17 | |
| E2 | 33.25 | 33.75 | 18.60 | 66.00 | 40.45 | 8.54 | 0.21 | 0.88 | -0.11 | -0.20 | |
| E3 | 31.45 | 32.70 | 14.23 | 54.10 | 35.31 | 6.42 | 0.18 | 0.86 | -0.32 | 0.20* | |
| E4 | 32.83 | 33.50 | 16.83 | 62.50 | 39.13 | 8.27 | 0.21 | 0.91 | -0.23 | -0.24 | |
| Average | 33.38 | 33.49 | 16.42 | 62.15 | 38.82 | 8.09 | 0.21 | 0.88 | -0.16 | -0.10 | |
| 旗叶上节间长FIL (cm) | |||||||||||
| E1 | 13.00 | 21.00 | 3.00 | 46.00 | 20.19 | 6.94 | 0.34 | 0.87 | 0.27** | 0.18* | |
| E2 | 11.75 | 20.00 | 3.80 | 42.20 | 20.16 | 6.35 | 0.32 | 0.89 | 0.15* | 0.14** | |
| E3 | 9.50 | 14.65 | 0.00 | 30.00 | 15.39 | 4.31 | 0.28 | 0.59 | 0.01 | 0.51** | |
| E4 | 11.00 | 17.50 | 2.50 | 35.00 | 18.40 | 5.58 | 0.30 | 0.89 | 0.06 | -0.02 | |
| Average | 11.31 | 18.29 | 2.33 | 38.30 | 18.53 | 5.80 | 0.31 | 0.81 | 0.12* | 0.21* | |
新窗口打开|下载CSV
图1显示了4个环境(2016SY、2017SY、2017HY和2017SG)下的重要产量性状的频率分布, 表明分离群体呈现出多个不同的混合组成分布, 图中的柱形表示频率分布, 实线表示理论拟合分布, 虚线表示成分分布。其中, 具有单峰分布特征的有E1环境下的穗下节间长及分蘖数, E2环境下的单株产量、千粒重和旗叶上节间长, E3环境下的单株产量, E4环境下的千粒重和分蘖数。单峰态表明可能为多基因控制; 其余各生态环境下性状分布具有明显连续多峰分布特点, 揭示可能存在主基因。该群体重要产量性状具有明显的以数量性状为主的遗传特性, 多数表现连续性分布, 具有较广泛的遗传变异。同时表明该群体重要产量性状可能受到主基因和多基因遗传效应的影响。
2.2 重要产量性状间的遗传相关性分析
对RILs群体进行邦弗朗尼(Bonferroni)相关性及线性拟合线的分析, 建立了小麦重组自交系(RILs)的线性拟合散点图矩阵(图2-A, B, C, D)。分析了4个环境中群体重要产量性状的频率分布, 每个变量的分布显示在对角线上。相关和拟合曲线分析表明, 所研究的性状大多是相互关联的, 部分性状之间存在较强的相关性。图2中A显示, 穗下节间长与旗叶上节间长相关系数最高(r = 0.90, P≤0.001)。各性状中单株产量与穗下节间长相关系数最高(r = 0.30, P≤0.001), 千粒重与株高的相关系数最高(r = 0.36, P≤0.001), 株高与穗下节间长相关系数最高(r = 0.76, P≤0.001), 其次是旗叶上节间长(r = 0.73, P≤0.001), 分蘖数与株高相关系数最高(r = 0.25, P≤0.001)。由图A左下角可知, 相关性越强, 正态分布越明显, 拟合曲线越好; 图2中B显示, 穗下节间长与旗叶上节间长相关系数最高(r = 0.94, P≤0.001), 其次是株高与穗下节间长(r = 0.90, P≤0.001), 株高与旗叶上节间长(r = 0.85, P≤0.001), 单株产量与分蘖(r = 0.55, P≤0.001), 单株产量与千粒重(r = 0.0.43, P≤0.001), 千粒重与穗下节间长(r = 0.34, P≤0.001), 单株产量与穗下节间长(r = 0.32, P≤0.001)。由图B左下角可知, 相关系数越高, 相关性越强, 两两性状对原始数据的拟合程度越高, 拟合效果越好; 图2中C显示, 穗下节间长与旗叶上节间长相关系数最高(r = 0.87, P≤0.001), 其次是株高与穗下节间长(r = 0.84, P≤0.001), 株高与旗叶上节间长(r = 0.74, P≤0.001), 单株产量与千粒重(r = 0.55, P≤0.001), 单株产量与有效分蘖数(r = 0.0.52, P≤0.001), 千粒重与株高(r = 0.50, P≤0.001), 千粒重与穗下节间长(r = 0.45, P≤0.001) , 单株产量与株高(r = 0.43, P≤0.001)。由图B左下角可知, 两性状相关系数越高, 拟合优度越好; 图2中D显示, 穗下节间长与旗叶上节间长相关系数最高(r = 0.92, P≤0.001), 其次是株高与穗下节间长(r = 0.86, P≤0.001), 株高与旗叶上节间长(r = 0.73, P≤0.001), 单株产量与千粒重(r = 0.50, P≤0.001), 单株产量与有效分蘖数(r = 0.0.50, P≤0.001), 千粒重与株高(r = 0.49, P≤0.001), 千粒重与穗下节间长(r = 0.49, P≤0.001), 单株产量分别与株高和穗下节间长(r = 0.44, P≤0.001)。由图B左下角可知, 两性状相关系数越高, 拟合优度越好。因此, 这也进一步验证了, 两性状间线性相关系数|r|越大, 各观测点在拟合直线周围聚集的紧密程度越高, 两个变量的线性相关性越强, 拟合的效果越好。田间重要产量性状的变异系数显著性越大, 变异越强, 尤其是穗下节间长和旗叶上节间长以及与其显著相关的性状, 具有一定的生物学意义。同时也说明, 穗下节间长与旗叶上节间长可以做为判断单株产量、千粒重及株高等的间接指标, 和育种实践紧密结合, 从而指导新品系的选育。
图1
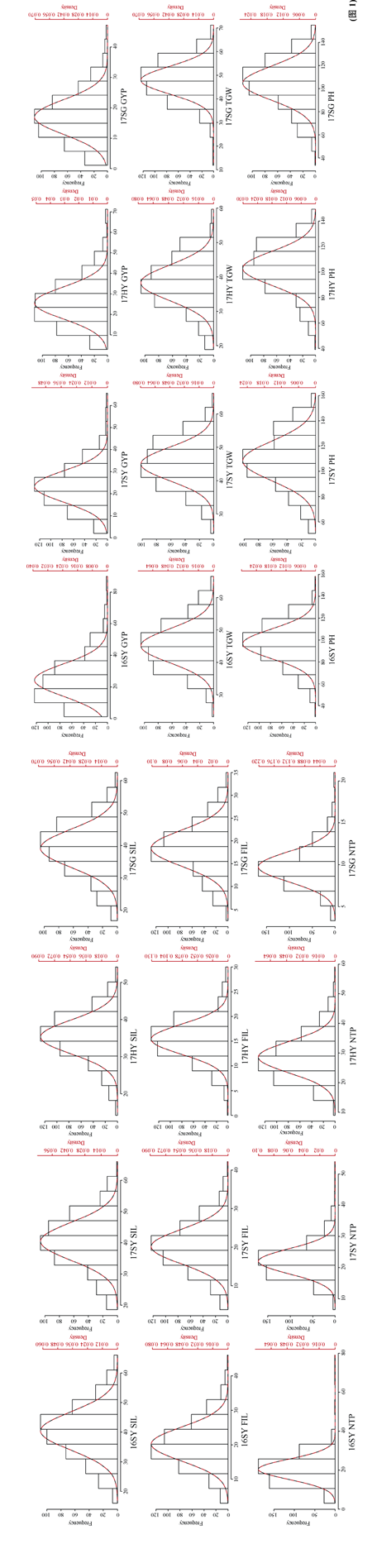 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1RIL 群体重要产量性状的次数分布(柱形)、拟混合分布(实线)与成分(虚线)分布
Fig.1Frequent(column),mixed(solid line,theoretical), and component(dotted line) distributions for wheat panicle-related traits in RILs
2.3 重要产量性状主基因+多基因混合遗传分析
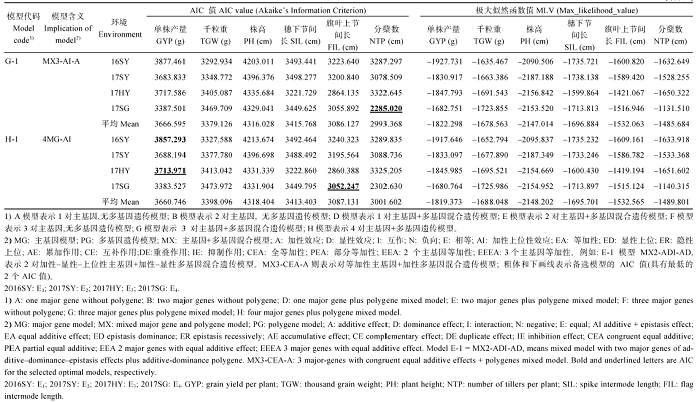
2.3.1 遗传模型的选择 利用植物数量性状主基因+多基因混合遗传模型的单世代(P1、P2、RIL)分析。根据遗传模型选取的AIC值最小准则, 选取AIC值最小的一组和接近最小AIC的一组遗传模型作为备选模型(表2)。结果表明38种遗传模型中, 不同生态环境下, 小麦单株产量的B-2-1(PG-AI)、E-2-9(MX2- IE-A)和H-1(4MG-AI)模型的AIC值较低, 分别为3378.430、3857.293和3713.971, 可以作为备选模型。千粒重的B-2-1(PG-AI)、B-2-1(PG-AI)、B-2-2 (PG-A)、E-2-7(MX2-CE-A)和E-2-8(MX2-DE-A)模型的AIC值较低, 分别为3359.526、3453.097、3334.821、3282.456和3282.456, 可以作为备选模型。株高的E-2-4(MX2-ED-A)、E-2-6(MX2-AE-A)、E-2-7(MX2- CE-A)、E-2-8(MX2-DE-A)和G-0(MX3-AI-AI)模型的AIC值较低, 分别为4392.348、4191.116、4321.722、4321.722和4325.307, 可以作为备选模型。有效分蘖数模型E-2-5(MX2-ER-A), F-1(3MG- AI), G-0(MX3-AI-AI)和G-1(MX3-AI-A)的AIC值较低, 分别为3318.093、3074.965、3271.55和2285.020, 可以作为备选模型。穗下节间长B-2-1(PG-AI), E-2-0(MX2-AI-AI)、E-2-8(MX2-DE-A)和E-2-9(MX2- IE-A)的遗传模型AIC值较低, 分别为3490.122、3439.562、3213.584和3486.552, 可以作为备选模型。旗叶上节间长B-2-1(PG-AI)和H-1(4MG-AI)的模型AIC值较低, 分别为3219.065、3192.343、2854.608和3052.247, 可以作为备选模型。结果显示估算获得38个遗传模型并分为7种类型, 即1对主基因(模型A), 2对主基因(模型B), 1对主基因加多基因(模型D), 2对连锁主基因加多基因(模型E), 3对主基因(模型F), 3对主基因加多基因(模型G), 和4对主基因加多基因(模型H)。模型的极大似然函数值AIC值见表2。图2
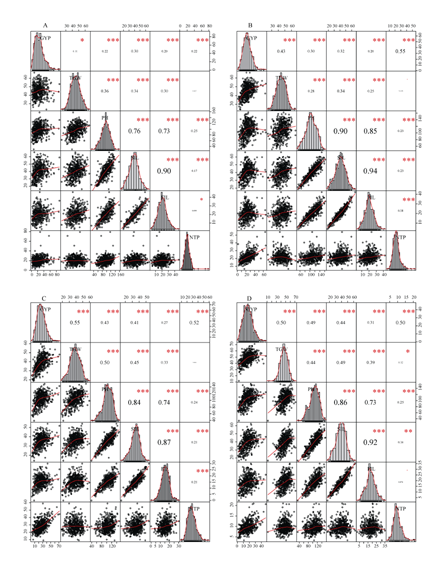 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图2A?B?C?D分别表示小麦重组自交系群体在2015-2016年陕西杨凌(E1: 2016SY)?2016-2017年陕西杨凌(E2: 2017SY)?2016-2017河南原阳(E3: 2017HY)?2016-2017四川广元(E4: 2017SG) 4个环境下重要产量性状的频率分布?相关性和拟合曲线
上三角形面板(对角线的右上方)显示了2个变量属性之间的相关系数以及显著性水平, 相关系数越大字号越大(星号越多表明越显著); 下三角形面板(对角线的左下方)为2个变量属性的散点图,显示的是具有拟合线的双变量散点图?对角线是各个变量的直方图, 每个显着性水平由一个符号表示, 对应关系依次为: 无符号 P < 0.1, “*” P < 0.05, “**”P < 0.01, “***”P < 0.001, “****”P < 0.0001?
Fig. 2Bonferroni-corrected genetic correlations for field yield traits with model parameters and a scatter plot matrix of linear fit lines in wheat-recombinant inbred lines (RILs). A, B, C, and D indicate the frequency distribution, correlation and fit curve of yield traits of RILs group in the four environments of Shaanxi Yangling in 2015-2016, Yangling in Shaanxi in 2016-2017, Yuanyang in Henan in 2016-2017, and Guangyuan in 2016-2017, respectively
The distribution of each variable is shown on the diagonal. On the bottom of the diagonal, the bivariate scatter plots with a fitted line are displayed. On the top of the diagonal, the value of the correlation plus the significance level are shown as stars. Each significance level is associated with a symbol: no symbol P < 0.1, “*”P < 0.05, “**” P < 0.01, “***” P < 0.001, “****” P < 0.0001.
Table 2
Table 2Akaike information criterion(AIC) and Maximum likelihood values(MLV) of the genetic models from 'Pindong 34' and 'Barran'
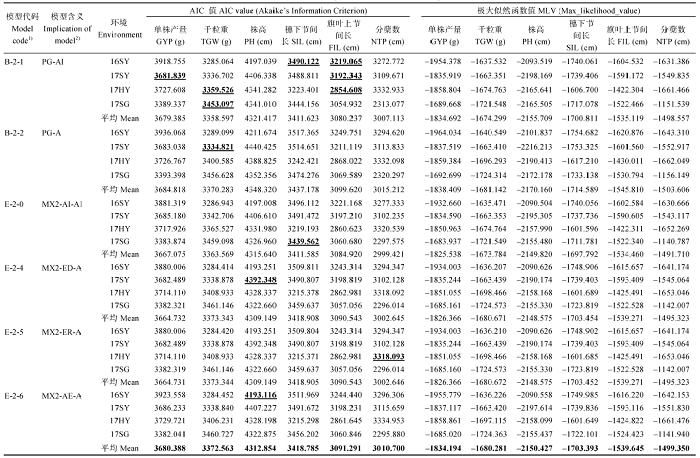 |
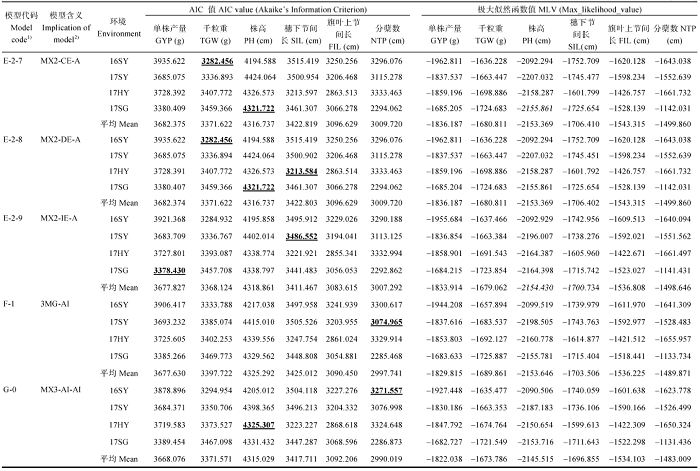 |
 |
新窗口打开|下载CSV
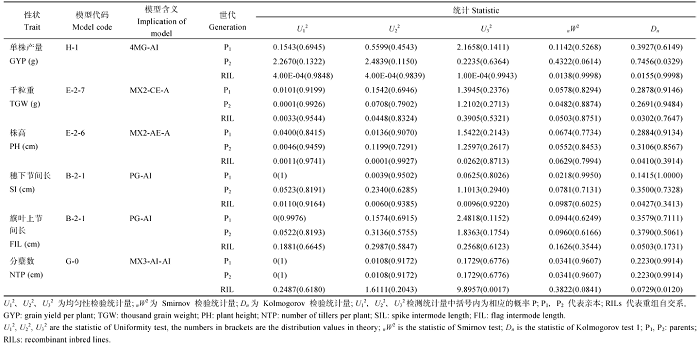
2.3.2 候选模型的适合性检测与最优遗传模型估算
表3显示了各个环境下的最适遗传模, 同时对备选模型进行一组(U12、U22、U32、nW2和Dn)适合性检验, 选择AIC值最小且统计量达到显著水平个数较少的模型作为最优模型。结果表明, 2年4个环境下, 株高的E-2-6 (E1)模型有最少的统计量, 差异达到显著水平(P<0.05)。穗下节间长的B-2-1(PG-AI)(E1)模型有最少的统计量, 差异达到显著水平(P<0.05)。旗叶下节间长的B-2-1(PG-AI)(E1)模型有最少的统计量, 差异达到显著水平(P<0.05)。有效分蘖数的G-0 (MX3-AI-AI) (E1)模型有最少的统计量, 差异达到显著水平(P<0.05)。单株产量的H-1(4MG-AI) (E3) 模型有最少的统计量, 差异达到显著水平(P<0.05)。千粒重的E-2-7(MX2-CE-A) (E1和E2)模型有最少的统计量, 差异达到显著水平(P<0.05)。因此, 株高的最优遗传模型是E-2-6(MX2-AE-A)(E1)。说明该性状的遗传受2对连锁主基因, 累加作用多基因混合遗传模型控制, 2对主基因加多基因连锁, 具有累积作用, 表示2对增效主基因在一起时, 株高有较大幅度的提高; 穗下节间长的最优遗传模型是B-2-1(PG-AI)(E1), 说明该性状受2对连锁主基因, 加性-上位性多基因遗传模型控制, 多基因遗传模型具有明显的加性上位性作用; 旗叶上节间长的最优遗传模型是B-2-1(PG-AI)(E2), 即2对连锁主基因, 加性-上位性多基因遗传模型; 分蘖数的最优遗传模型是G-0(MX3-AI-AI)(E1)即加性-上位性多基因混合遗传模型, 说明该性状的3对主基因具有明显的加性上位性作用, 多基因同样也有加性上位性作用; 单株产量的最优遗传模型是H-1(4MG-AI)(E1)即主基因加性上位性模型, 说明控制该性状的4对主基因具有明显的加性上位性作用; 千粒重的最优遗传模型是E-2-7(MX2-CE-A)(E1)即2对连锁主基因, 互补作用多基因混合遗传模型, 说明该性状受多基因控制, 2对主基因具有明显的互补作用, 多基因具有加性效应。
Table3
Table3Fitness trsts of selected models in ‘Pindong 34’ and ‘Barran’
 |
新窗口打开|下载CSV
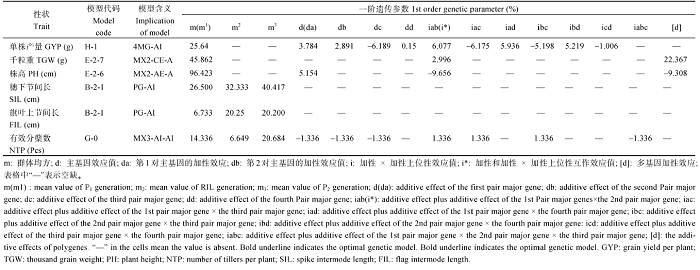
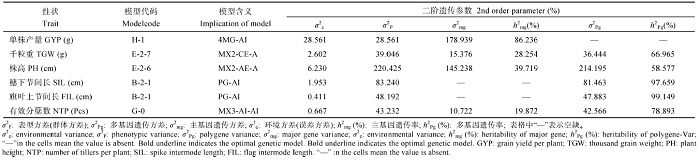
2.3.3 遗传参数估计 根据2年4个环境下入选的最适遗传模型估算重要产量性状的一阶遗传参数和二阶遗传参数(表4和表5), 控制株高的第1对主基因的加性效应值是5.154(da), 为正向遗传效应, 加性和加性×加性上位性互作效应值为(iab) -9.656, 为负向遗传效应。多基因的加性效应值[d]为-9.308, 主基因的遗传率为58.57%, 多基因的遗传率为39.71%, 说明该群体控制株高性状的多基因具有明显的累加负效应; 控制穗下节间长和旗叶上节间长的最佳遗传模型相同, 都是由2对连锁主基因, 加性-上位性多基因遗传模型控制, 多基因遗传模型具有明显的加性上位性作用, 其中穗下节间长主基因遗传率为97.65%, 旗叶上节间长的主基因遗传率为99.14%; 控制分蘖数的第1对主基因的加性效应值(da)是-1.336, 具有负向效应。第2对主基因的加性效应值(db)和第3对主基因的加性效应值(dc)数值相等, 是-1.33, 且为负向效应, 说明控制该性状的3对主基因加多基因模型有明显的负向加性上位性作用, 加性和加性×加性上位性互作效应值(iab)、加性和第1对主基因加性×第3对主基因加性上位性互作效应值(iac)、加性和第2对主基因加性×第3对主基因加性上位性互作效应值(ibc)三者相同, 都是1.336, 表现正向效应。加性效应和第1对主基因×第2对主基因×第3对主基因的加性效应值(iabc)是-1.33, 3个基因相关作用表现为负向效应。主基因遗传率为78.89%, 多基因遗传率为19.87%; 控制单株产量的第1至4对主基因加性效应值(da、db、dc、dd)分别为3. 784、2.891、-6.180和0.150, 第3对主基因加性效应值最高且为负向遗传效应。第2对主基因加性效应稍大于第2对, 表现为正向效应。第4对主基因加性效应值最小。加性和加性×加性上位性互作效应值(iab)是6.007, 表现为正向的遗传效应。加性和第1对主基因加性×第3对主基因加性上位性互作效应值(iac)是-6.17, 表现为负向的遗传效应, iab和iac两者效应值相似, 单表现效应相反。加性和第1对主基因加性×第4对主基因加性上位性互作效应值(iad)是5.93, 表现正向效应。加性和第2对主基因加性×第3对主基因加性上位性互作效应值(ibc)是-5.19, 表现负向效应。加性和第2对主基因加性×第4对主基因加性上位性互作效应值(ibd)是5.21, ibd和iad二者效应值相近, 同为正向效应, 这两者与ibc绝对值相近但效应相反, 加性和第3对主基因加性×第4对主基因加性上位性互作效应值(icd)是-1, 表现负向遗传效应。控制单株产量的多基因遗传率为86.23%, 总的来看控制单株产量性状的4对主基因的加性上位性作用明显, 累加一起时主基因表现很弱, 结果表现多基因遗传, 多基因起着主导作用; 控制千粒重性状的加性和加性×加性上位性互作效应值(iab)是2.996, 表现为正向的遗传效应。多基因的加性效应值[d]是22.367, 表现为很高的正向遗传效应。主基因遗传率为66.96%, 多基因遗传率为28.25%, 说明控制该性状的2对主基因加多基因具有明显的互补作用, 表现加性效应的多基因混合遗传。
Table 4
Table 4Estimates of 1st order genetic parameters of important yield traits for population from Pindong 34 $\times$ Barran
 |
新窗口打开|下载CSV
Table 5
Table 5Estimates of 2nd order genetic parameters of important yeild traits for population from Pindong 34 $\times$ Barran
 |
新窗口打开|下载CSV
3 讨论
3.1 小麦重要产量性状的最佳遗传模型分析
小麦各个性状对产量均产生直接或间接的影响, 这不仅是因为环境效应的影响, 更重要的是产量各性状的综合表现受遗传控制。本研究表明, 对4个环境遗传模型进行筛选后确定最优遗传模型是E1环境下的H-1(4MG-AI)模型即主基因加性上位性模型。说明控制该性状的4对主基因具有明显的加性上位性作用。王翠玲等[47]利用25个F1杂交组合研究发现单株产量的遗传受加性和非加性基因共同控制。本研究中单株产量的遗传也具有明显的加性效应, 这和王翠玲等的研究观点相似。只是本研究得到的遗传参数更为详细具体, 该性状受到4对具有加性上位性的主基因模型控制。本研究中在不同环境下相关分析表明, 单株产量与株高、千粒重、分蘖数和穗下节间长等均极显著或显著相关, 这说明单株产量性状的复杂性, 是一个综合性状。这与丁安明等[48]、Jamali等[49]和Kato等[50]的研究观点相似, 丁安明[48]利用重组自交系群体研究认为单株产量与株高的相关性达到显著水平。当株高在80 cm以上时, 伴随植株增高的增产效果不显著; 本文对4个环境遗传模型筛选后千粒重的最优遗传模型是E-2-7 (MX2-CE-A)(E1)即2对连锁主基因, 互补作用多基因混合遗传模型。说明该性状受多基因控制, 2对主基因具有明显的互补作用, 多基因具有加性效应。王培等[30]利用F2:3家系群体发现千粒重由多基因控制符合无主基因模型(A-0模型)。杜希朋等[31]利用F2群体研究发现千粒重受微效多基因控制, 无主基因存在。朱欣果等[32]利用小麦重组自交系研究发现千粒重最优模型是4MG-AI即主基因加性上位性模型。本研究发现在4个环境下千粒重均符合多基因模型特征, 这与王培等[30]、杜希朋等[31]研究结果相似, 与朱欣果等[32]研究结果不同, 不同的原因可能是因为人工合成小麦材料的特殊性, 具有祖先明显野生性的结果, 这种遗传背景的差异加上环境影响导致了结果不一致; 4个环境下株高的最优遗传模型是E-2-6(MX2-AE-A)(E1)。说明该性状的遗传受2对连锁主基因, 累加作用多基因混合遗传模型控制, 2对主基因加多基因连锁, 具有累积作用。姚金保等[23]通过对7个亲本双列杂交的F1杂交组合研究认为小麦株高的遗传可能受3~4对主效基因控制, 表现为加性效应和显性效应的共同作用, 以加性效应为主, 显性程度表现为部分显性, 遗传力高。国外有研究[33,34]表明株高的遗传模型以加性效应或以显现效应为主的加性-显性模型, 也有研究认为株高是以上位性效应为主的加性-显性-上位性模型[36]。李斯深等[35]利用RIL群体研究表明株高是由2对连锁的主基因加微效基因共同控制的。杜希朋等[32]利用F2群体研究认为株高是受多基因控制的数量性状, 无主基因影响。李法计等[36]利用小麦重组自交系研究发现株高符合2对主基因+多基因遗传, 主基因遗传率为82.32%。毕晓静等[37]利用F2, F3群体研究得出株高性状符合D-2模型即一对加显性主基因+加性-显性多基因混合遗传模型。陈树林[38]利用苏麦3号矮杆密穗材料研究发现小麦株高受一对不完全显性基因控制。朱欣果等[32]利用人工合成小麦重组自交系得出小麦株高的最优模型是MX3-AI-A即加性-上位性多基因混合遗传模型。本研究中株高最优遗传模型是E1环境下的多基因混合遗传模型即E-2-6 (MX2-AE-A)模型, 说明在该群体中E1环境下控制株高的2对主基因具有明显的累加效应, 加多基因具有明显的加性效应。这与杜希朋等[31]、朱欣果等[32]、李斯深等[35]和李法计等[36]的研究结果相似, 与姚金保等[22]、Khan等[33]、Dere等[34]、毕晓静等[37]和陈树林等[38]的研究结果不同, 可能是因为本研究使用的材料是高代重组自交系, 这与F2等杂交群体不同, 没有显性及其相互作用的效应, 只有一些可以固定的效应值, 如加性与加性和加性间的相互作用效应值。与F2等材料相比较, 重组自交系(RILs)群体可以作为永久性群体使用, 可以设置多年多点试验, 这和DH群体一样。因此, 可能比分离世代更能减少环境影响, 进而获得较准确的参数估值。对4个环境遗传模型筛选后穗下节间长最优遗传模型是B-2-1(PG-AI)(E1), 说明该性状受2对连锁主基因, 加性-上位性多基因遗传模型控制, 多基因遗传模型具有明显的加性上位性作用。姚金保等[23]通过小麦双列杂交研究发现, 穗下节间长可能受1-3对主效基因控制。李法计等[38]通过对小麦重组自交系研究认为穗下茎长性状符合3对主基因+多基因遗传。毕晓静等[37]研究发现穗下节间距的遗传符合D-3模型, 即一对完全显性主基因+加性-显性多基因混合遗传模型。本研究中, 在多个环境下都符合多基因遗传模型, 最优模型是B-2-1(PG-AI)(E1)即加性上位性多基因遗传模型, 表明控制该性状的遗传模型有2对连锁主基因且多基因具有加性上位性效应。这与李法计研究结果近似, 与姚金保等[23]和毕晓静等[37]研究结果不同, 可能是因为使用不同的材料。本研究中穗下节间长度和株高、分蘖等具有显著或极显著的相关性, 在小麦育种过程中, 充分考虑穗下节间长对株高、单株产量等的影响会对选择理想株型起到积极作用; 本研究中旗叶上节间长最优遗传模型是B-2-1(PG-AI)即2对连锁主基因, 加性-上位性多基因遗传模型。姚金保等[23]利用8个小麦品种双列杂交的F1及其亲本研究发现, 小麦穗颈长的遗传符合加性-显性模型, 基因作用以加性效应为主。范平等[39]研究认为穗颈长的遗传受加性-显性-上位性模型控制。蒲定福等[40]研究认为穗颈长不符合加性-显性模型。卫云宗等[41]研究认为穗颈长受到加性基因效应影响。本研究中旗叶上节间长(穗颈长)的最适遗传模型与范平等[39]、卫云宗等[43]研究中的加性, 上位性效应影响观点相似, 所不同的是没有显性效应影响, 这可能是因为实验材料、实验环境等的不同。本研究中旗叶上节间长(穗颈长)与穗下节间长、单株产量、千粒重、有效分蘖数等性状显著或极显著相关。穗颈长对群体产量有着较大正向效应影响[42], 说明小麦新品系选育中要充分考虑选择适宜长的穗颈长, 且与株高协同考虑, 才能取得较好效果; 本研究中在多环境下有效分蘖最适模型是G-0 (MX3-AI-AI)(E1)即加性-上位性多基因混合遗传模型, 说明该性状的3对主基因具有明显的加性上位性作用, 多基因同样也有加性上位性作用。张倩辉等[43]利用普通小麦-冰草衍生F2代群体研究发现有效分蘖数的最佳遗传模型是B-2, 受2对主基因控制, 并且主基因表现为加性-显性效应。王培等[30]利用F2:3家系为材料, 发现小麦单株穗数由一对加性和部分显性主基因(A-1模型)。毕晓静等[37]研究发现有效分蘖数符合D-4模型, 一对负向完全显性主基因+加性-显性多基因模型。闫林[44]研究得出有效分蘖数符合2对加性-显性-上位性主基因+加性-显性多基因遗传模型。谢玥等[45]利用杂交F2-3及BC1-2群体研究发现有效分蘖可能受2对主效核基因和一些微效基因的共同控制, 其中1对基因对另1对基因有抑制作用。杜希鹏等[31]研究认为有效分蘖受1对主基因+多基因控制, 主基因遗传率为55.7%, 主基因表现为负完全显性。朱欣果等[32]利用小麦重组自交系研究发现有效分蘖数的最优模型是4MG-AI即主基因加性上位性模型。本研究群体有效分蘖数的分离呈现连续的偏态分布, 表明有效分蘖数具有数量性状的遗传特点, 除了受到微效多基因控制外还可能有主效基因的影响, 这与之前的研究结果不一样[30-31,37,43-45], 可能是因为选用试验材料类型不同, 重组自交系较纯合, 只能显示加性效应, 同时生态环境条件的影响导致了结果不一致。这也进一步验证了单株分蘖数受数量性状位点控制的观点。有效分蘖数是小麦产量构成的重要农艺性状之一, 不仅是复杂的发育性状, 更容易受到生产条件、栽培及环境因素的影响。只有生产上保证适宜的有效穗数才有利于形成高产稳产[46]。
3.2 研究最佳遗传模型对小麦育种起着重要推动作用
本研究通过邦弗朗尼(Bonferroni)相关性及线性拟合线的分析发现, 各性状遗传相关性较显著。穗下节间与旗叶上节间平均相关系数更是达到0.91 (P≤0.001)。同时二者和株高、单株产量、分蘖数、千粒重均表现了较为明显且显著的相关性, 说明穗下节间长与旗叶上节间长可以作为判断株高、单株产量、千粒重等的间接指标, 可通过选择穗下节间长和旗叶上节间长进而控制植株高度, 从而指导新品系的选育。因此, 具有一定的生物学意义。姚金保等[23]通过对小麦株高及其构成因素的相关性分析表明株高与穗下节间长等构成因素有显著的遗传正相关, 这与本文相关分析结果相似。卢娟等[24]利用小麦亚群为材料, 研究发现分蘖数、千粒重等性状与产量表型显著或极显著水平相关。杨德龙等[25]利用小麦RIL群体研究发现, 在干旱胁迫条件下, 不同发育时期株高与千粒质量之间显著正相关。王慧如等[26]以小麦回交导入系(IL)群体为材料研发发现千粒质量、株高等性状与小区产量有着较高的相关性。Kato等[27]研究发现, 株高和产量的显著相关。Law等[28]研究表明株高和产量呈显著正相关, Wu等[29]研究表明穗长与粒重显著正相关。本研究的6个重要产量性状间存在显著或极显著正相关, 这与之前的研究观点相似。所不同的是在多环境下进行, 得出的结果更具有代表性。同时穗下节间长和旗叶上节间长作为株高的重要组成部分, 可作为间接指标来选育优良单株, 深入研究二者与株高和产量的关系, 可以获得高产潜力的株型, 这对获得以高产为目标的株型育种具有重要意义。本研究选取的单株产量、千粒重、株高和有效分蘖数等性状, 是小麦育种中重要的目标经济性状, 且多为数量性状。在小麦重要产量性状的遗传中, 环境方差在表型方差中占据着较高的比例, 表明在重要产量因素的遗传中环境和基因型间有着很强的互作效应。因此, 在小重要产量性状的育种过程中, 要重视生态环境条件对提高单产的限制, 降低环境效应对小麦产量因素的影响, 增加选育优良新品种的生态适应性及广适性。如何识别数量性状的主要基因一直也是数量遗传学领域的主要研究热点。重组自交系(RILS)是由连续自交产生的一系列不杂交直到纯合的品系, 能够准确地识别每一品系的表型值, 减少环境误差。因此, RILS群可以用于多年多点检验, 比分离世代更准确地确定各品系的表型值, 减少环境影响, 获得更准确的参数估计。由于每个株系均为纯合子, 不存在显性和显性互作, 群体可用于精确估计主基因的加性效应值和加性互作效应值[35]。与传统的遗传分析方法相比, 植物数量性状主基因和多基因混合遗传模型可以从当前的研究中获得更客观的信息。通过标准曲线与频率分布的比较, 检测主基因的存在, 估计主基因和多基因的遗传力, 包括遗传模型和遗传参数。主基因+多基因遗传体系的分离分析方法与分子标记的QTL定位方法的区别在于, 前者只需要数量性状表型数据, 后者需要分子数据才能定位到控制数量性状的基因位点[3]。在实践中可以用前者校对后者, 以便于育种工作者快速判断基于表型得出对育种性状遗传组成结果的准确性, 也可以用后者校正前者, 以便于进一步掌握QTL定位所反映性状遗传组成的结果。本研究的多个重要产量性状在多个环境下主要受主基因+多基因混合遗传控制。同时, 该方法的大量应用提供了各性状的遗传组成、遗传规律特性及遗传参数, 为科研工作提供了有意义的遗传信息, 为下一步通过高通量测序开发SNP标记、QTL初步定位及精细定位、阐明各性状的多基因遗传调控网络及其间的互作调控关系, 以及小麦分子设计育种提供重要理论依据。
4 结论
2年4个环境下最优遗传模型, 对单株产量的是4对加性上位性主基因+多基因遗传模型。对千粒重是2对互补作用主基因+加性效应多基因混合遗传模型。对株高是2对累积作用主基因+加性作用多基因混合遗传模型。对穗下节间长和旗叶上节间长是加性-上位性多基因遗传模型。对分蘖数是加性-上位性多基因混合遗传模型。穗下节间长与旗叶上节间长可以做为判断单株产量、千粒重及株高等的间接指标, 这些性状在多个环境下主要受主基因+多基因混合遗传控制。在选育优良品系的过程中, 和育种实践紧密结合, 要兼顾适应生态环境条件的重要表现, 进一步为筛选与目标性状紧密连锁标记及分子标记辅助选择提供理论依据。参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 5]
[本文引用: 5]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 4]
[本文引用: 4]
[本文引用: 5]
[本文引用: 5]
[本文引用: 6]
[本文引用: 6]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
[本文引用: 3]
[本文引用: 6]
[本文引用: 6]
[本文引用: 3]
[本文引用: 3]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 1]
[本文引用: 1]
