 ,1,*, 郑军
,1,*, 郑军 ,1,*
,1,*Genetic Analysis of Haplotype-blocks from Wheat Founder Parent Linfen 5064
QIAO Ling1,**, LIU Cheng2,**, ZHENG Xing-Wei1, ZHAO Jia-Jia1, SHANG Bao-Hua1, MA Xiao-Fei1, QIAO Lin-Yi1, GE Hong-Mei3, JI Hu-Tai1, LIU Jian-Jun2, ZHANG Jian-Cheng ,1,*, ZHENG Jun
,1,*, ZHENG Jun ,1,*
,1,*通讯作者:
第一联系人:
收稿日期:2017-11-13接受日期:2018-03-25网络出版日期:2018-06-12
| 基金资助: |
Received:2017-11-13Accepted:2018-03-25Online:2018-06-12
| Fund supported: |

摘要
关键词:
Abstract
Keywords:
PDF (1162KB)元数据多维度评价相关文章导出EndNote|Ris|Bibtex收藏本文
本文引用格式
乔玲, 刘成, 郑兴卫, 赵佳佳, 尚保华, 马小飞, 乔麟轶, 盖红梅, 姬虎太, 刘建军, 张建诚, 郑军. 小麦骨干亲本临汾5064单元型区段的遗传解析[J]. 作物学报, 2018, 44(6): 931-937. doi:10.3724/SP.J.1006.2018.00931
QIAO Ling, LIU Cheng, ZHENG Xing-Wei, ZHAO Jia-Jia, SHANG Bao-Hua, MA Xiao-Fei, QIAO Lin-Yi, GE Hong-Mei, JI Hu-Tai, LIU Jian-Jun, ZHANG Jian-Cheng, ZHENG Jun.
我国小麦品质育种起步较晚, 长期忽视品质改良造成了推广品种品质普遍较差。新世纪以来, 我国小麦品质育种快速发展, 建立了品种品质评价体系与分子改良技术, 并且育成和推广了一批优质品种[1]。产量和品质同步提高是今后育种的主要目标, 而优异种质资源的创新和利用是实现这一目标的关键。因此, 深入挖掘和利用我国优质小麦种质资源对今后小麦遗传改良具有重要意义。临汾5064、小偃6号和中作8131-1并称为我国小麦三大强筋优质源[2,3], 据统计, 临汾5064作为亲本选育出衍生品种(系)80多个, 已成为我国小麦优质骨干亲本。后代品种中济南17和济麦19成为制粉业替代进口的优质品牌, 得到大面积推广和产业化[4]; 新麦26是我国加工品质最好的品种之一, 也是当前黄淮南片主推品种[5]; 此外, 农大152、农大135、农大1189和农大1195在北部冬麦区生产和育种中也得到应用[6]。这些临汾5064的衍生品种不仅生产上表现突出, 也是当前品质育种的主要亲本, 是我国小麦品质改良的基础。
结合系谱分析, 在全基因组水平上分析和认识骨干亲本对育种工作, 特别是组合选配具有重要参考价值。张学勇等[7]利用SSR 标记扫描我国小麦核心种质后, 发现大面积推广品种多为骨干亲本的后代, 老的骨干亲本往往是新骨干亲本的奠基者。盖红梅等[8]证明了鲁麦14对山东新选育小麦品种遗传贡献较大, 且大片段遗传在品种间呈现明显的偏分布。对碧蚂1号、碧蚂4号、欧柔、燕大1817、阿夫等骨干亲本的系统研究, 也发现重要染色体区域在我国品种改良中发挥了重要作用[9,10,11,12]。此外, 繁6、周8425B、科农9204等骨干亲本的研究也进一步证明了优异单元型区段在亲本和品种选育过程中得到定向选择[13,14,15]。骨干亲本在产量和抗病性等方面已有较为深入的研究, 为今后小麦设计育种和亲本预测提供了很多有价值的信息, 而关于品质和适应性等性状的研究亟待开展。
临汾5064于1988年出圃, 具有优质强筋、大粒、早熟、矮秆和综合抗性好的特点, 产量4500~6750 kg hm-2, 由于主茎穗与分蘖穗相差5~10 cm, 导致整齐度较差, 未能通过审定。临汾5064具有较强配合力, 遂向各育种单位推荐, 并育成大量品种(系谱见附图1), 而关于其内在本质、演变规律等问题缺乏深入分析。本文根据衍生系谱信息, 通过SSR标记研究了临汾5064及衍生品种(系)的遗传多样性, 结合前期关联分析寻找重要单元型区段遗传规律, 以期为亲本选配和重要品质资源的利用提供依据。
附图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT附图1临汾5064及其衍生品种系谱图
Supplementary Fig. 1Derivatives of Linfen 5064 and their pedigrees
1 材料与方法
1.1 植物材料
临汾5064及21个衍生品种(系), 其中衍生一代8份, 衍生二代13份(附表1)。山东省审定品种由山东省农业科学院作物研究所提供, 北京市和河北省审定品种由北京市农林科学院杂交小麦中心提供, 其余品种(系)由山西省农业科学院小麦研究所保存。Supplementary Table.1
附表1
附表1临汾5064及其衍生品种(系)信息
Supplementary Table.1
| 品种 | 世代 | 系谱 | 审定年份 | 审定类别 |
|---|---|---|---|---|
| 临汾5064 | 亲本 | 临汾5694/沙瑞克//临汾5054 | 1988 | - |
| 济麦19 | 子一代 | 鲁麦13/临汾 | 2001 | 山东省 |
| 济南17 | 子一代 | 临汾5064/鲁麦13 | 1999 | 山东省 |
| 临优145 | 子一代 | 临汾5064/陕优225 | 2003 | 山西省 |
| 临汾8050 | 子一代 | 临汾5064/陕7622 | 2007 | 山西省 |
| 临优2018 | 子一代 | 农科2号/临汾5064 | 2005 | 山西省 |
| 临汾138 | 子一代 | 临汾5064/烟农15 | 2003 | 山西省 |
| 农大152 | 子一代 | 陕优225/临汾5064 | - | 北京市 |
| 临汾139 | 子一代 | 太原136/临汾5064 | 2006 | 陕西省 |
| 晋麦83 | 子二代 | 临汾5064//烟农15/临辐8001 | 2007 | 山西省 |
| 中麦155 | 子二代 | 济麦19/鲁麦21 | 2012 | 河北省 |
| 婴泊700 | 子二代 | 太谷核不育/济麦19 | 2012 | 河北省 |
| 临优2069 | 子二代 | 临优145/5445 | 2005 | 山西省 |
| 晋麦94 | 子二代 | 临优2018航天育种 | 2014 | 山西省 |
| 鲁原502 | 子二代 | 9940168/济麦19 | 2011 | 国家黄淮冬麦区 |
| 晋麦82 | 子二代 | 临汾138/陕88316 | 2007 | 山西省 |
| 荷麦19 | 子二代 | 烟农19/临汾139 | - | - |
| 新麦26 | 子二代 | 新麦18/济南17 | 2010 | 国家黄淮冬麦区 |
| 中洛08-2 | 子二代 | 偃展1号/济南17 | 2011 | 河南省 |
| 山农14 | 子二代 | G91605/济南17 | 2006 | 山东省 |
| 山农15 | 子二代 | 济南17/济核916 | 2006 | 山东省 |
| 郑麦0943 | 子二代 | 郑97199/济麦19 | 2014 | 河南省 |
新窗口打开|下载CSV
1.2 品质分析方法
委托农业部谷物及制品质量监督检验测试中心(哈尔滨)检测。参考何中虎等[16]的方法, 用分子标记检测籽粒硬度、低分子量谷蛋白和醇溶蛋白等指标。采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)方法[17,18]分析高分子量谷蛋白亚基(HWM-GS)组成。1.3 分子标记分析
按照改良CTAB法提取全基因组DNA, 稀释备用[19]。挑选构建中国小麦核心种质的SSR引物490对[20], 参考郝晨阳等[20] PCR扩增、银染及统计方法。根据GrainGenes (https://wheat.pw.usda.gov/GG3/)的引物信息, 去掉非单一位点和扩增质量较差的引物95对, 使用395对引物完成实验, 其中A基因组132对、B基因组107对, D基因组156对。采用PowerMarker 3.25软件计算等位变异数、Nei’s基因多样性指数及多态性信息含量(PIC)、遗传距离。利用Mega4.0软件按Neighbor-Joining (NJ)法聚类; 用百分比表示衍生品种遗传贡献率和共有位点数。
1.4 遗传图谱的绘制
参考日本Kumogi遗传图谱(http://www.shigen.nig.ac. jp/)估计SSR位点遗传距离, 用软件MapInspect绘制遗传连锁图谱, 通过共有标记标注染色体区段的QTL和关联位点[21,22,23]。2 结果与分析
2.1 临汾5064及衍生后代的品质特性
临汾5064的粗蛋白质含量(干基) 14.84%, 容重790 g L-1, 沉降值33.2 mL, 稳定时间16.3 min, 湿面筋含量40.5%, 吸水率61.4%, 形成时间约为10.3 min, 面包重量148.1 g, 面包体积882 cm3。分子标记检测表明, 临汾5064籽粒硬度基因型为pinb-D1b, 高分子量谷蛋白亚基组成为1、7+8和2+12; 低分子量谷蛋白亚基为GIu-A3d、GIu-B3f和GIu-D3f, 含醇溶蛋白CE-3亚基。尽管不含5+10亚基, 但面筋强度大、烘烤品质优良。临汾5064衍生后代品种的高分子量麦谷蛋白亚基组成见表1。Glu-A1位点有两种组合形式(1和null), Glu-B1位点有4种组合形式(7+8、7+9、14+15和17+18), Glu-D1位点有3种组合形式(2+12、5+10和5+12)。可见, 临汾5064的高分子量麦谷蛋白亚基并未在衍生品种中稳定遗传。
Table 1
表1
表1临汾5064及衍生后代品种的HMW-GS组成
Table 1
| 类别 Type | 品种 Variety | 亚基组成 HMW-GS |
|---|---|---|
| 亲本 Parent | 临汾5064 Linfen 5064 | 1, 7+8, 2+12 |
| 衍生后代 Derived varieties or lines | 济麦19 Jimai 19 | 1, 7+8, 2+12 |
| 济南17 Jinan 17 | 1, 7+8, 5+12 | |
| 临优145 Linyou 145 | 1, 7+8, 2+12 | |
| 临汾8050 Linfen 8050 | 1, 7+8, 2+12 | |
| 临优2018 Linyou 2018 | 1, 7+8, 5+10 | |
| 临汾138 Linfen 138 | 1, 7+9, 2+12 | |
| 农大152 Nongda 152 | 1, 7+8, 2+12 | |
| 临汾139 Linfen 139 | null, 7+8, 2+12 | |
| 晋麦83 Jinmai 83 | 1, 14+15, 2+12 | |
| 中麦155 Zhongmai 155 | null, 7+8, 5+12 | |
| 婴泊700 Yingbo 700 | 1, 7+9, 5+10 | |
| 临优2069 Linyou 2069 | 1, 7+8, 5+12 | |
| 晋麦94 Jinmai 94 | 1, 7+8, 5+10 | |
| 鲁原502 Luyuan 502 | 1, 7+8, 5+12 | |
| 晋麦82 Jinmai 82 | 1, 7+9, 2+12 | |
| 荷麦19 Hemai 19 | null, 17+18, 5+10 | |
| 新麦26 Xinmai 26 | 1, 7+9, 5+10 | |
| 中洛08-2 Zhongluo 08-2 | 1, 14+15, 5+12 | |
| 山农14 Shannong 14 | 1, 7+8, 5+12 | |
| 山农15 Shannong 15 | 1, 7+8, 5+12 | |
| 郑麦0943 Zhengmai 0943 | 1, 7+9, 5+12 |
新窗口打开|下载CSV
2.2 临汾5064及衍生品种(系)的遗传关系
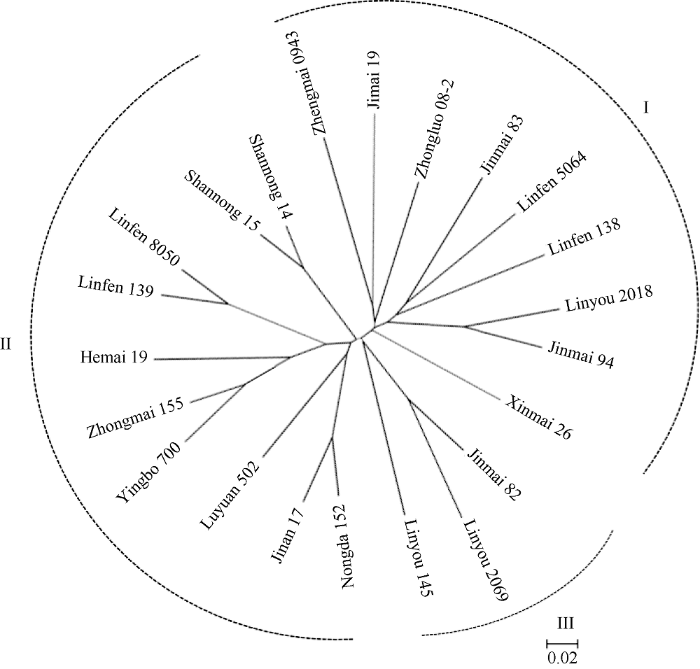
用395对SSR引物在22份材料中共检测到895个等位变异, 单个标记检测出等位变异1~8个, 平均2.27个, 其中Xcfd39等位变异数为8个; A、B和D基因组中分别检测到292、249和354个等位变异; 平均等位变异数分别为2.21、2.31和2.27。等位变异频率为0.20~1.00, 平均为0.78; PIC值为0~0.83, 平均为0.25。将临汾5064及衍生后代分为3个类群(图1), 与系谱来源基本一致。第I类群包括临汾5064、济麦19及两者衍生后代郑麦0943、晋麦94和临优2018等, 第II类群包括临汾5064衍生子一代品种如济南17、临汾139、临汾8050和农大152及部分子二代品种, 第Ⅲ类群为临优145、晋麦82和临优2069。图1
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT图1临汾5064及衍生品种聚类分析(NJ法)
Fig. 1Cluster analysis of Linfen 5064 and its derivatives (NJ method)
2.3 临汾5064对衍生后代品种(系)的遗传贡献
利用基因组平均分布的395对引物分析临汾5064对其衍生后代遗传贡献率表明, 临汾5064和衍生后代中位点一致性超过80%的有176个, 其中完全相同的位点有146个。临汾5064对后代品种(系)的平均遗传贡献率为64.77%, 对子一代、子二代的平均贡献率分别为65.30%和64.24%。在不同基因组中, 临汾5064对子一代A、B和D基因组贡献率分别为64.01%、66.82%和65.07%, 对子二代3个基因组的贡献率分别为63.93%、65.89%和62.89%, 其中2A的遗传贡献率最小, 分别为41.50%和42.76%; 6B的遗传贡献率最高, 分别为75.00%和79.49% (表2)。整体上看, 临汾5064对子代遗传贡献率较高, 且并未随着世代增加而明显下降。Table 2
表2
表2临汾5064对衍生品种(系)的遗传贡献分析
Table 2
| 基因组 Genome | 标记/衍生世代 Marker/derived generation | 染色体 Chromosome | 平均 Average | ||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | |||
| A | 标记数量 No. of markers | 16 | 25 | 21 | 18 | 21 | 14 | 17 | 132 |
| 子一代 First generation | 68.75 | 41.50 | 63.10 | 61.81 | 70.83 | 65.18 | 76.92 | 64.10 | |
| 子二代 Second generation | 71.63 | 42.76 | 65.00 | 58.12 | 67.03 | 69.23 | 73.76 | 63.93 | |
| B | 标记数量 No. of markers | 16 | 19 | 17 | 12 | 23 | 9 | 11 | 107 |
| 子一代 First generation | 49.22 | 69.74 | 63.24 | 68.75 | 67.93 | 75.00 | 73.86 | 66.82 | |
| 子二代 Second generation | 47.11 | 68.82 | 62.44 | 67.95 | 65.55 | 79.49 | 69.93 | 65.89 | |
| D | 标记数量 No. of markers | 22 | 21 | 16 | 16 | 38 | 18 | 25 | 156 |
| 子一代 First generation | 57.95 | 60.11 | 73.44 | 67.97 | 65.46 | 68.06 | 62.50 | 65.07 | |
| 子二代 Second generation | 58.04 | 60.80 | 75.48 | 61.06 | 65.09 | 61.96 | 56.92 | 62.89 | |
新窗口打开|下载CSV
2.4 临汾5064对后代衍生品种(系)贡献率高的单元型区段分析
临汾5064与衍生后代中完全相同的单元型区段共16个, 贡献率大于80%的单元型区段主要集中在1BL、2AS、2BS、2DS、3AS、3DL、4AS、5BL、6DL和7AL等染色体上(附图2)。这些单元型区段在子一代和子二代中基本上完全保留了临汾5064的单元型, 这些单元型区段在育种中被连续保留, 是骨干亲本的遗传基础。例如Xbarc302-Xgwm124-Xgwm153-Xpsp3100 (1BL)、Xbarc168- Xwmc470-Xcfd255-Xcfd116-Xcfd160-Xcfd56-Xbarc228 (2DS)、Xwmc50-Xbarc179-Xgwm32-Xgwm5-Xbarc19 (3AS)、Xgwm4- Xgwm165 (4AS)、Xwmc110-Xgwm126-Xwmc577-Xwmc524- Xwmc727 (5AL)、Xgdm127-Xbarc202-Xbarc123-Xcfd47- Xbarc1121-Xcfd38 (6DL)和Xgwm332-Xwmc17-Xcfa2240- Xgwm573-Xpsp3050-Xwmc422-Xgwm282 (7AL)等区段。有些位点或单元型区段在子一代品种中共有, 而在子二代中出现选择交换的现象, 例如Xcfd83、Xgwm512- Xpsp3153、Xgwm389、Xwmc216、Xcfd67、Xbarc44、Xwmc357和Xbarc278, 这些位点或区段可能在育种选择过程不断优化。此外, 有些位点为临汾5064所特有, 而在后代中未被检测到, 如Xgwm294、Xwmc170、Xgwm102、Xcfd50和Xcfd39, 推测这些位点或区段含有负遗传效应或与不良性状连锁, 在育种过程中发生了替换和重组。附图2
 新窗口打开|下载原图ZIP|生成PPT
新窗口打开|下载原图ZIP|生成PPT附图2临汾5064衍生后代染色体单元型分析
红色区为后代品种(系)与亲本相同的单元型区段,绿色区域为子一代与亲本相同的单元型区段,黑色横线为临汾5064所特有等位变异;染色体右边的竖线代表贡献率超过80%的单元型区段
Supplementary Fig.2Haplotype-blocks shared between core parent lines and Linfen5064 in 21 linkage groups
The identical haplotype-blocks between Linfen 5064 and its derivatives are shown in red. The identical haplotype-blocks between Linfen 5064 and its first derivatives are shown in green. Black line indicates the unique alleles in Linfen 5064. The vertical line on the right of chromosome represents the haplotype-blocks with the genetic contribution ratios higher than 80%
2.5 临汾5064被选择区段的典型特征及遗传效应分析
结合课题组利用481个SSR标记对微核心种质及大面积推广品种全基因组关联分析的结果[21,22,23], 发现这些单元型区段基本上都与重要农艺性状关联, 有些呈一因多效或基因簇分布(附图2)。1B、2A、2B、3A、3D、4D、5B、5D、6B、7A和7B染色体上共有单元型区段都与品质显著相关, 如2AS的Xwmc109、3AL的Xgwm391与蛋白质含量显著关联, 2BL的Xbarc200与蛋白质和淀粉含量显著关联; 1B的Xwmc52-Xgwm11、Xbarc302-Xgwm124- Xgwm153-Xpsp3100、2AL的Xgwm512-Xpsp3153、2DS的Xbarc168-Xwmc470-Xcfd255-Xcfd116-Xcfd160-Xcfd56-Xbarc228、3DL的Xgwm456-Xgwm52、5AL的Xwmc110- Xwmc524、5BL的Xwmc99-Xpsp3065、7AL的Xgwm332- Xgwm282和7DS的Xwmc221-Xgwm437的单元型区段与蛋白质含量、淀粉含量显著相关。在重要农艺性状方面, 每条染色体上的共有单元型区段与产量、株型等性状显著关联, 如1BL的Xbarc302-Xgwm124-Xgwm153-Xpsp3100区段与粒重、穗粒数、穗下节长和有效分蘖数显著关联; 2DS的Xbarc168-Xwmc470-Xcfd255-Xcfd116-Xcfd160-Xcfd56- Xbarc228区段与粒厚相关外, 还与收获指数、穗长、小穗数和穗粒数显著关联; 5AL的Xwmc110-Xgwm126-Xwmc577- Xwmc524-Xwmc727区段则与穗长、穗密度、穗下节长、成熟期、抽穗期和容重等性状显著相关; 6B的Xpsp3009-Xgwm70-Xgwm626区段存在千粒重、穗粒数、穗长和穗密度显著关联遗传位点。此外, 在1A、1B、1D、2B、2D、3B、3D、4B、5A、5B、5D、6B、6D和7D上都有与千粒重相关的位点存在。这些与重要农艺性状相关的单元型区段是育种家选择的主要靶点, 且在衍生后代中优先传递, 可能是临汾5064作为直接和间接亲本发挥作用的根本所在。3 讨论
庄巧生[6]对我国小麦品种系谱分析后, 发现有些亲本品种在育种过程中起着骨干作用, 衍生的推广品种较多。因此, 提出了“骨干亲本”的概念, 即直接用来培育出一批大面积推广品种, 或由其衍生出许多具有应用价值的亲本材料[6]。临汾5064为我国小麦育种作出了巨大贡献, 作为亲本育成30多个优质强筋衍生品种, 这些后代材料也是当前育种的主要亲本。如临汾5064与鲁麦13杂交育成优质面包品种济南17, 累计推广面积373.16万公顷; 济麦19累计推广面积413.27万公顷[24]。临汾5064解决了强筋小麦育种3大问题。(1)品质和粒重负相关。优质强筋品种千粒重低, 籽粒商品性差, PH82-2-2、烟农15、烟农19、山农12、藁城8901、藁优2018、师滦02-1、西农979、泛麦5号、美国硬红冬等强筋小麦的千粒重一般都低于40 g[25], 而临汾5064的千粒重常年稳定在44 g以上, 用其作亲本选育的品种(系)多数千粒重较高, 如济南17千粒重为42 g左右, 济麦19为45 g左右。(2)矮秆和早熟相结合。当前品种大多以Rht1、Rht2、Rht8和Rht9等为矮源[26], 临汾5064株高为75 cm左右、早熟, 克服了矮秆常与晚熟、早衰、多病和高产性能差相关联/连锁的难题[27], 可能与5AL单元型区段中含有Rht12有关。(3)不含5+10亚基但品质优良。临汾5064高分子量麦谷蛋白亚基为(1, 7+8, 2+12), GluB1的7+8亚基、GluD1的2+12均不是高效应优质亚基, 但临汾5064加工品质优良, 且受环境效应小[28], 推测临汾5064的低分子量麦谷蛋白、醇溶蛋白以及蛋白质含量对品质具有补偿或拉动作用。临汾5064优质特性来源于国际玉米小麦改良中心引进春麦SARICF74, 使用冬性偏强的临汾5694与SARICF74杂交后, 又以早熟、综合农艺性状好的临汾5054作为父本复交选育而成。临汾5064具有配合力好、遗传传递力强、含有利基因多等良好的遗传特点, 是种内具有突出优点“偏材”和“良材”相结合的典型。育种家根据育种目标对性状选择, 如抗病、丰产、优质和广适等性状的定向选择, 使具有目标性状的材料在选择中被保留下来, 同时控制目标性状的染色体位点或区段经历了很强的选择[21], 如鲁麦14对子一代贡献率为60.0%~70.6%; 周8425B与其衍生品种间遗传相似性平均为74.6%, 对子一代、子二代和子三代遗传贡献率分别67.7%、63.6%和58.8%[14]。遗传分析发现临汾5064对子一代和子二代的平均遗传贡献率为65.30%和64.24%, 且世代遗传率基本未发生明显变化, 存在很多被育种家选择的单元型区段。结合关联分析和QTL定位研究相关片段/位点遗传效应后发现, 共有单元型区段和位点与重要农艺性状显著相关(附图2), 是一因多效或重要基因(QTL)的富集区[29,30,31,32,33,34,35]。例如, 1BL的Xbarc302-Xgwm124-Xgwm 153-Xpsp3100区段与穗粒数、穗下节长和有效分蘖数性状关联外, 还存在SDS沉降值、面粉灰分和千粒重QTL[29]; 2DS的Xbarc168-Xbarc228基因区段除与粒厚、容重显著相关外, 还有控制产量、收获指数、穗长、小穗数和穗粒数的QTL[30]; 6B的psp3009-gwm70-gwm626存在控制籽粒蛋白含量、株高、穗颈长和穗下节长相关QTL[33]。肖永贵等[14]发现骨干亲本周8425B的衍生品种携带的4个抗条锈病基因均来自周8425B; 繁6的成株抗性及遗传位点在其衍生后代品种选育过程中得到了很强的定向选择[13]。除抗病性外, 周8425B和繁6还含有不良性状连锁少、符合育种目标且配合力高的重要性状(片段)。结合本文研究结果可知, 抗病和优质骨干亲本之所以能培育出很多优良品种, 除本身含有抗病和优质遗传特性外, 还具有很好的产量和其他重要性状相关的遗传背景, 这也是临汾5064作为亲本能在不同生态区选育出较多品种的原因所在。
随着9K、90K和660K芯片的开发, 使小麦遗传图谱的密度进一步增加, 极大地促进了小麦遗传育种的研究[36,37]。与SNP相比, SSR标记为共显性, 具有多态性高, 检测快速, 操作简便的优点。此外, SSR变异常与临近基因的交换重组关系密切, 在基因组中分布密度与基因岛和重组热点高度相关, 对驯化和育种高压选择的位点的检测能力强[38,39]。在标记辅助选择的可行性方面, SSR标记依然具有较大优势。本文在明确单元型区段遗传规律的基础上, 筛选到与品质和产量性状相关的分子标记, 为临汾5064及衍生后代的亲本选配和标记辅助选择提供了依据, 但关键区段的组成及其对产量和品质等农艺性状的遗传效应仍需深入研究。
参考文献 原文顺序
文献年度倒序
文中引用次数倒序
被引期刊影响因子
[本文引用: 1]
[本文引用: 1]
DOI:10.3864/j.issn.0578-1752.2013.20.002URL [本文引用: 1]

小麦是中国第三大粮食作物,在农业生产和食品工业中占有重要地 位。随着中国经济和社会的发展,消费者的生活需求正在发生变化,工业化和城市化使食品工业,特别是传统食品工业化加速发展。与此同时,食品工业对食品原料 的专用化和规模化,即品种的质量、规格和数量,提出了更高的要求。如何研究和解决这一矛盾,已成为小麦产业和食品工业可持续发展的新问题。本文以国内发表 的论著为基础,整理、分析了小麦品质改良历史、研究进展、存在问题;讨论了小麦产业链和食品加工业的可持续发展对优质小麦的需求;结合农业生产和食品工业 的发展趋势提出建议,供小麦育种、生产、收贮、食品加工等部门和研究人员参考。
DOI:10.3864/j.issn.0578-1752.2013.20.002URL [本文引用: 1]

小麦是中国第三大粮食作物,在农业生产和食品工业中占有重要地 位。随着中国经济和社会的发展,消费者的生活需求正在发生变化,工业化和城市化使食品工业,特别是传统食品工业化加速发展。与此同时,食品工业对食品原料 的专用化和规模化,即品种的质量、规格和数量,提出了更高的要求。如何研究和解决这一矛盾,已成为小麦产业和食品工业可持续发展的新问题。本文以国内发表 的论著为基础,整理、分析了小麦品质改良历史、研究进展、存在问题;讨论了小麦产业链和食品加工业的可持续发展对优质小麦的需求;结合农业生产和食品工业 的发展趋势提出建议,供小麦育种、生产、收贮、食品加工等部门和研究人员参考。
DOI:10.3969/j.issn.1008-0864.2010.02.01URL [本文引用: 1]

介绍了我国小麦生产发展概况和我国小麦育种的发展历史,包括以抗病稳产为主的育种阶段(20世纪50~60年代)、以矮化高产为主的育种阶段(20世纪70~80年代)和高产与优质育种并进阶段(20世纪90年代~21世纪初);阐述了当前我国对粮食生产需求的新变化及其对今后小麦育种工作的要求和方向.
DOI:10.3969/j.issn.1008-0864.2010.02.01URL [本文引用: 1]

介绍了我国小麦生产发展概况和我国小麦育种的发展历史,包括以抗病稳产为主的育种阶段(20世纪50~60年代)、以矮化高产为主的育种阶段(20世纪70~80年代)和高产与优质育种并进阶段(20世纪90年代~21世纪初);阐述了当前我国对粮食生产需求的新变化及其对今后小麦育种工作的要求和方向.
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]

为给强筋小麦高产优质栽培中的氮肥合理运筹提供依据,以强筋小麦新品种‘新麦26’为材料,在施氮量为240 kg/hm^2的前提下,研究了分别在起身期、拔节期、孕穗期和开花期4个时期追施氮肥对优质强筋小麦产量及面粉品质的影响。结果表明,在施氮量一致的基础上,从起身期到开花期随着氮肥追施时期的推迟,‘新麦26’籽粒产量呈先增加后降低的趋势,其中孕穗期追施氮肥小麦籽粒产量显著高于其他处理,穗数最多,穗粒数仅次于拔节期追施氮肥,千粒重最高。拔节追施氮处理面团稳定时间最长,粉质评价值最高,籽粒产量次于孕穗氮处理。开花氮处理籽粒产量和粉质评价值最低。‘新麦26’面团吸水率、稳定时间在不同处理间差异较小。建议在生产中将强筋小麦的氮肥追施时期安排在孕穗开始时,既可获得较高的籽粒产量又能保证面粉品质。
URL [本文引用: 1]

为给强筋小麦高产优质栽培中的氮肥合理运筹提供依据,以强筋小麦新品种‘新麦26’为材料,在施氮量为240 kg/hm^2的前提下,研究了分别在起身期、拔节期、孕穗期和开花期4个时期追施氮肥对优质强筋小麦产量及面粉品质的影响。结果表明,在施氮量一致的基础上,从起身期到开花期随着氮肥追施时期的推迟,‘新麦26’籽粒产量呈先增加后降低的趋势,其中孕穗期追施氮肥小麦籽粒产量显著高于其他处理,穗数最多,穗粒数仅次于拔节期追施氮肥,千粒重最高。拔节追施氮处理面团稳定时间最长,粉质评价值最高,籽粒产量次于孕穗氮处理。开花氮处理籽粒产量和粉质评价值最低。‘新麦26’面团吸水率、稳定时间在不同处理间差异较小。建议在生产中将强筋小麦的氮肥追施时期安排在孕穗开始时,既可获得较高的籽粒产量又能保证面粉品质。
[本文引用: 3]
[本文引用: 3]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]

利用十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)技 术,对小麦骨干亲本碧蚂4号80个衍生品种、系谱当中涉及的18个中间亲本、碧蚂4号6个姊妹系品种及其亲本共106个品种(系)的HMW-GS组成进行 分析.结果表明,碧蚂4号HMW-GS组成为Null、7+8、2+12.在Glu-B1位点,碧蚂4号的7+8亚基与一代衍生品种的3个亲本均不同,其 在衍生一代中的遗传受到了显著的选择,遗传频率达到84.6%;6个(54.5%)二代衍生品种的亲本含有与碧蚂4号相同亚基,该代7+8亚基的遗传频率 仍最高,达到64.1%;三代和四代衍生品种的5个亲本中只有北京6号和矮秆早含有7+8亚基,这两个世代7+8亚基遗传频率明显降低,7+9成为主要类 型,频率分别达到了69.2%和64.3%.在Glu-A1和Glu-D1位点,各有11个(64.7%)中间亲本具有与碧蚂4号相同的亚基,它们分布于 不同子代涉及的中间亲本中,Null和2+12亚基在其衍生的4个世代品种的遗传频率均不小于76.9%.系谱分析发现,在三代和四代,中间亲本HMW- GS组成遗传频率显著增加,其中以洛夫林10的Null、7+9、2+12亚基为主.由于HMW-GS组成不是碧蚂4号系谱品种育成时期的选择目标,对骨 干亲本碧蚂4号和洛走林10号被选择的Glu-B1位点进一步研究有助于解析骨干亲本的形成原因.
URL [本文引用: 1]

利用十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)技 术,对小麦骨干亲本碧蚂4号80个衍生品种、系谱当中涉及的18个中间亲本、碧蚂4号6个姊妹系品种及其亲本共106个品种(系)的HMW-GS组成进行 分析.结果表明,碧蚂4号HMW-GS组成为Null、7+8、2+12.在Glu-B1位点,碧蚂4号的7+8亚基与一代衍生品种的3个亲本均不同,其 在衍生一代中的遗传受到了显著的选择,遗传频率达到84.6%;6个(54.5%)二代衍生品种的亲本含有与碧蚂4号相同亚基,该代7+8亚基的遗传频率 仍最高,达到64.1%;三代和四代衍生品种的5个亲本中只有北京6号和矮秆早含有7+8亚基,这两个世代7+8亚基遗传频率明显降低,7+9成为主要类 型,频率分别达到了69.2%和64.3%.在Glu-A1和Glu-D1位点,各有11个(64.7%)中间亲本具有与碧蚂4号相同的亚基,它们分布于 不同子代涉及的中间亲本中,Null和2+12亚基在其衍生的4个世代品种的遗传频率均不小于76.9%.系谱分析发现,在三代和四代,中间亲本HMW- GS组成遗传频率显著增加,其中以洛夫林10的Null、7+9、2+12亚基为主.由于HMW-GS组成不是碧蚂4号系谱品种育成时期的选择目标,对骨 干亲本碧蚂4号和洛走林10号被选择的Glu-B1位点进一步研究有助于解析骨干亲本的形成原因.
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]

阿夫是我国小麦育种的骨干亲本之一,由其衍生的品种(系)有171个.为了了解阿夫对衍生品种在品质育种上的贡献,本文利用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳技术(SDS-PAGE)对阿夫及其衍生品种(系)的高分子量麦谷蛋白亚基(HMW-GS)的组成与演变进行了分析.结果表明,阿夫HMW-GS组成为1/14+15/2+12,品质得分8.5分;而在衍生品种(系)中,携带与阿夫HMW-GS组成相同的品种占7.01%,平均品质得分7.68分,低于亲本阿夫.其中,由阿夫衍生的子一代到子四代品种中,携带与阿夫相同的14+15优质亚基的品种分别占28.30%、19.74%、9.09%和7.69%,平均品质得分分别是7.74分、7.84分、7.56分和7.23分,均低于亲本阿夫.结果说明,骨干亲本阿夫对其衍生后代在品质改良上的贡献很小,而主要贡献可能是当时育种目标需求的抗条锈病和丰产性等性状.
URL [本文引用: 1]

阿夫是我国小麦育种的骨干亲本之一,由其衍生的品种(系)有171个.为了了解阿夫对衍生品种在品质育种上的贡献,本文利用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳技术(SDS-PAGE)对阿夫及其衍生品种(系)的高分子量麦谷蛋白亚基(HMW-GS)的组成与演变进行了分析.结果表明,阿夫HMW-GS组成为1/14+15/2+12,品质得分8.5分;而在衍生品种(系)中,携带与阿夫HMW-GS组成相同的品种占7.01%,平均品质得分7.68分,低于亲本阿夫.其中,由阿夫衍生的子一代到子四代品种中,携带与阿夫相同的14+15优质亚基的品种分别占28.30%、19.74%、9.09%和7.69%,平均品质得分分别是7.74分、7.84分、7.56分和7.23分,均低于亲本阿夫.结果说明,骨干亲本阿夫对其衍生后代在品质改良上的贡献很小,而主要贡献可能是当时育种目标需求的抗条锈病和丰产性等性状.
[本文引用: 1]
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
DOI:10.3724/SP.J.1006.2015.00574URL [本文引用: 1]

科农9204是一个兼具高产和氮高效的候选小麦骨干亲本,其遗传背景复杂,携带冀麦38、小偃5号、绵阳75-18、小偃693和矮丰3号的遗传成分。利用221个PCR标记和89个DAr T标记,绘制了科农9204的全基因组基因型图谱。在2DL上,Xmag3596–Xmag4089区段与增加千粒重和籽粒含氮量的QTL紧密连锁;在4BL上,Xcnl10与增加穗粒数、降低株高和穗茎长的QTL紧密连锁;在6BS上,Xcnl113和Xwmc756均与降低株高、穗茎长和穗下节间长的QTL紧密连锁。这些标记在科农9204衍生后代的传递率均为100.0%。利用已报道的关联性标记检测科农9204基因型在衍生后代的传递情况,与增加穗粒数相关的1个优异等位基因位点在衍生后代中的传递率为71.6%;与增加千粒重相关的4个优异等位基因位点的传递率均为100.0%;与根部性状相关的4个基因位点中,3个传递率为100.0%。这些与重要农艺性状相关位点,科农9204基因型在其衍生后代中有很高的传递率,在很大程度上与其对应的优异的农艺性状密不可分。科农9204染色体区段上存在的重要QTL可能是其成为候选骨干亲本的遗传基础。
DOI:10.3724/SP.J.1006.2015.00574URL [本文引用: 1]

科农9204是一个兼具高产和氮高效的候选小麦骨干亲本,其遗传背景复杂,携带冀麦38、小偃5号、绵阳75-18、小偃693和矮丰3号的遗传成分。利用221个PCR标记和89个DAr T标记,绘制了科农9204的全基因组基因型图谱。在2DL上,Xmag3596–Xmag4089区段与增加千粒重和籽粒含氮量的QTL紧密连锁;在4BL上,Xcnl10与增加穗粒数、降低株高和穗茎长的QTL紧密连锁;在6BS上,Xcnl113和Xwmc756均与降低株高、穗茎长和穗下节间长的QTL紧密连锁。这些标记在科农9204衍生后代的传递率均为100.0%。利用已报道的关联性标记检测科农9204基因型在衍生后代的传递情况,与增加穗粒数相关的1个优异等位基因位点在衍生后代中的传递率为71.6%;与增加千粒重相关的4个优异等位基因位点的传递率均为100.0%;与根部性状相关的4个基因位点中,3个传递率为100.0%。这些与重要农艺性状相关位点,科农9204基因型在其衍生后代中有很高的传递率,在很大程度上与其对应的优异的农艺性状密不可分。科农9204染色体区段上存在的重要QTL可能是其成为候选骨干亲本的遗传基础。
[本文引用: 1]
[本文引用: 1]
DOI:10.1038/227680a0URL [本文引用: 1]
URL [本文引用: 1]

The alleles for the gene loci, Glu-A1, Glu-B1 and Glu-D1 are formally defined and cross referenced to the lettering and numbering systems used by the authors and by other research groups to describe the gene products, the high-molecular-weight subunits of glutenin. It is recommended that these alleles be referred to in future by all research groups, in addition to using their own subunit nomenclature, so enabling the work of different laboratories to be compared without difficulty.
[本文引用: 1]
[本文引用: 2]
[本文引用: 2]
[本文引用: 3]
[本文引用: 3]
DOI:10.2135/cropsci2010.12.0680URL [本文引用: 2]
DOI:10.1371/journal.pone.0029432URLPMID:3273457 [本文引用: 2]

Chinese wheat mini core collection (262 accessions) was genotyped at 531 microsatellite loci representing a mean marker density of 5.1 cM. One-thousand-kernel weights (TKW) of lines were measured in five trials (three environments in four growing seasons). Structure analysis based on 42 unlinked SSR loci indicated that the materials formed two sub-populations, viz., landraces and modern varieties. A large difference in TKW (7.08 g, P<0.001) was found between the two sub-groups. Therefore, TKW is a major yield component that was improved in the past 6 decades; it increased from a mean 31.5 g in the 1940s to 44.64 g in the 2000s, representing a 2.19 g increase in each decade. Analyses based on a mixed linear model (MLM), population structure (Q) and relative kinship (K) revealed 22 SSR loci that were significantly associated with mean TKW (MTKW) of the five trials estimated by the best linear unbiased predictor (BLUP) method. They were mainly distributed on chromosomes of homoeologous groups 1, 2, 3, 5 and 7. Six loci, cfa2234-3A, gwm156-3B, barc56-5A, gwm234-5B, wmc17-7A and cfa2257-7A individually explained more than 11.84% of the total phenotypic variation. Favored alleles for breeding at the 22 loci were inferred according to their estimated effects on MTKW based on mean difference of varieties grouped by genotypes. Statistical simulation showed that these favored alleles have additive genetic effects. Frequency changes of alleles at loci associated with TKW are much more dramatic than those at neutral loci between the sub-groups. The numbers of favored alleles in modern varieties indicate there is still considerable genetic potential for their use as markers for genome selection of TKW in wheat breeding. Alleles that can be used globally to increase TKW were inferred according to their distribution by latitude and frequency of changes between landraces and the modern varieties.
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]

本研究利用RIL群体(山农01-35×藁城9411 F8:9)173个家系进行了小麦DArt和SNP(90K)基因芯片分析,构建了1张高密度遗传图谱;在4年5个环境下进行了小麦籽粒特性、面团流变学特性、淀粉糊化特性、馒头和面包加工等特性的基因定位,开发了4个粒重分子标记的,主要取得了如下结果:1.高密度遗传图谱的构建:构建了覆盖小麦21条染色体的高密度遗传图谱,该图谱共含有6 244个多态性标记,其中SNP标记6 001个
.
URL [本文引用: 1]

本研究利用RIL群体(山农01-35×藁城9411 F8:9)173个家系进行了小麦DArt和SNP(90K)基因芯片分析,构建了1张高密度遗传图谱;在4年5个环境下进行了小麦籽粒特性、面团流变学特性、淀粉糊化特性、馒头和面包加工等特性的基因定位,开发了4个粒重分子标记的,主要取得了如下结果:1.高密度遗传图谱的构建:构建了覆盖小麦21条染色体的高密度遗传图谱,该图谱共含有6 244个多态性标记,其中SNP标记6 001个
[本文引用: 1]
[本文引用: 1]
URL [本文引用: 1]

以8个亲本为材料进行了完全双列杂交,对冬小麦的返青至拔节、拔节至挑旗、挑旗至抽穗、抽穗至扬花、扬花至成熟等5个生育阶段持续期进行了遗传分析。结果表明,5个生育阶段都是遗传性状,h_B~2为85.74%~92.54%,h_(?)~2为62.2%~84.13%,5个性状均为部分显性,平均显性度0.2330~0.6699。除了扬花至成熟阶段系晚熟显性之外,其它4个阶段均为早熟显性。本研究还测算了8个亲本的 GCA 和 SCA 效应。
URL [本文引用: 1]

以8个亲本为材料进行了完全双列杂交,对冬小麦的返青至拔节、拔节至挑旗、挑旗至抽穗、抽穗至扬花、扬花至成熟等5个生育阶段持续期进行了遗传分析。结果表明,5个生育阶段都是遗传性状,h_B~2为85.74%~92.54%,h_(?)~2为62.2%~84.13%,5个性状均为部分显性,平均显性度0.2330~0.6699。除了扬花至成熟阶段系晚熟显性之外,其它4个阶段均为早熟显性。本研究还测算了8个亲本的 GCA 和 SCA 效应。
URL [本文引用: 1]

利用12个小麦品种在5个地点的试验结果,分析了品种、环境以及品种与环境的互作对小麦烘烤品质的影响,结果表明:品种、环境以及品种与环境的互作对小麦烘烤品质都有显著影响,品种和地点的互作效应在同一品种不同地点间是不同的,在较差的环境下,也有表现好的品种。不同环境下,烘烤品质指标的变化大于籽粒蛋白质含量的变化,小麦品种品质的评价,不仅应考虑其蛋白质含量在不同地点之间的变化,更重要的是研究最终加工品质的变化规律。
URL [本文引用: 1]

利用12个小麦品种在5个地点的试验结果,分析了品种、环境以及品种与环境的互作对小麦烘烤品质的影响,结果表明:品种、环境以及品种与环境的互作对小麦烘烤品质都有显著影响,品种和地点的互作效应在同一品种不同地点间是不同的,在较差的环境下,也有表现好的品种。不同环境下,烘烤品质指标的变化大于籽粒蛋白质含量的变化,小麦品种品质的评价,不仅应考虑其蛋白质含量在不同地点之间的变化,更重要的是研究最终加工品质的变化规律。
DOI:10.1007/s00122-008-0869-1URL [本文引用: 2]
DOI:10.1007/s11032-006-9056-8URL [本文引用: 2]

In bread wheat, single-locus and two-locus QTL analyses were conducted for seven yield and yield contributing traits using two different mapping populations (P I and P II). Single-locus QTL analyses involved composite interval mapping (CIM) for individual traits and multiple-trait composite interval mapping (MCIM) for correlated yield traits to detect the pleiotropic QTLs. Two-locus analyses were conducted to detect main effect QTLs (M-QTLs), epistatic QTLs (E-QTLs) and QTL x environment interactions (QE and QQE). Only a solitary QTL for spikelets per spike was common between the above two populations. HomoeoQTLs were also detected, suggesting the presence of triplicate QTLs in bread wheat. Relatively fewer QTLs were detected in P I than in P II. This may be partly due to low density of marker loci on P I framework map (173) than in P II (521) and partly due to more divergent parents used for developing P II. Six QTLs were important which were pleiotropic/coincident involving more than one trait and were also consistent over environments. These QTLs could be utilized efficiently for marker assisted selection (MAS).
[本文引用: 1]
DOI:10.1007/s00122-002-1114-yURL [本文引用: 1]
DOI:10.1186/1471-2156-15-57URLPMID:24885313 [本文引用: 2]

Background Common wheat (Triticum aestivum L.) is one of the most important food crops worldwide. Wheat varieties that maintain yield (YD) under moderate or even intense nitrogen (N) deficiency can adapt to low input management systems. A detailed genetic map is necessary for both wheat molecular breeding and genomics research. In this study, an F6:7 recombinant inbred line population comprising 188 lines was used to construct a novel genetic map and subsequently to detect quantitative trait loci (QTL) for YD and response to N stress. Results A genetic map consisting of 591 loci distributed across 21 wheat chromosomes was constructed. The map spanned 3930.7 cM, with one marker per 6.7 cM on average. Genomic simple sequence repeat (g-SSR), expressed sequence tag-derived microsatellite (e-SSR), diversity arrays technology (DArT), sequence-tagged sites (STS), sequence-related amplified polymorphism (SRAP), and inter-simple sequence repeat (ISSR) molecular markers were included in the map. The linear relationships between loci found in the present map and in previously compiled physical maps were presented, which were generally in accordance. Information on the genetic and physical positions and allele sizes (when possible) of 17 DArT, 50 e-SSR, 44 SRAP, five ISSR, and two morphological markers is reported here for the first time. Seven segregation distortion regions (SDR) were identified on chromosomes 1B, 3BL, 4AL, 6AS, 6AL, 6BL, and 7B. A total of 22 and 12 QTLs for YD and yield difference between the value (YDDV) under HN and the value under LN were identified, respectively. Of these, QYd-4B-2 and QYddv-4B, two major stable QTL, shared support interval with alleles from KN9204 increasing YD in LN and decreasing YDDV. We probe into the use of these QTLs in wheat breeding programs. Moreover, factors affecting the SDR and total map length are discussed in depth. Conclusions This novel map may facilitate the use of novel markers in wheat molecular breeding programs and genomics research. Moreover, QTLs for YD and YDDV provide useful markers for wheat molecular breeding programs designed to increase yield potential under N stress.
DOI:10.1007/s00122-006-0346-7URLPMID:16838135 [本文引用: 1]

Development of high-yielding wheat varieties with good end-use quality has always been a major concern for wheat breeders. To genetically dissect quantitative trait loci (QTLs) for yield-related traits such as grain yield, plant height, maturity, lodging, test weight and thousand-grain weight, and for quality traits such as grain and flour protein content, gluten strength as evaluated by mixograph and SDS sedimentation volume, an F 1 -derived doubled haploid (DH) population of 185 individuals was developed from a cross between a Canadian wheat variety C Karma and a breeding line 87E03-S2B1. A genetic map was constructed based on 167 marker loci, consisting of 160 microsatellite loci, three HMW glutenin subunit loci: Glu-A1 , Glu-B1 and Glu-D1 , and four STS-PCR markers. Data for investigated traits were collected from three to four environments in Manitoba, Canada. QTL analyses were performed using composite interval mapping. A total of 50 QTLs were detected, 24 for agronomic traits and 26 for quality-related traits. Many QTLs for correlated traits were mapped in the same genomic regions forming QTL clusters. The largest QTL clusters, consisting of up to nine QTLs, were found on chromosomes 1D and 4D. HMW glutenin subunits at Glu-1 loci had the largest effect on breadmaking quality; however, other genomic regions also contributed genetically to breadmaking quality. QTLs detected in the present study are compared with other QTL analyses in wheat.
DOI:10.3864/j.issn.0578-1752.2008.02.003URL [本文引用: 1]

【目的】发掘重要性状的QTL及其分子标记进行小麦品质分子改良。【方法】采用PH82-2/内乡188杂交后代240个F5:6家系,按照Latinizedα-lattice设计,2004~2005年度分别种植在河南焦作、安阳和山东泰安。对籽粒蛋白质含量、Zeleny沉降值、和面时间、8分钟带宽、峰值粘度和稀懈值进行测定,利用188个SSR标记和4个蛋白标记构建遗传连锁图谱,采用复合区间作图法(CIM)对上述6个品质性状进行QTL定位。【结果】籽粒蛋白质含量检测出3个QTL,分布在3A、3B染色体上。在1B、1D和3B染色体上检测到3个控制Zeleny沉降值的QTL,其中位于1B和1D染色体上的QTL在3个地点均检测到,可解释5.5%~17.6%表型变异。发现3个控制和面时间的QTL,分布在1B和1D染色体上,在3个地点均能检测到,贡献率为7.9%~55.3%;检测出8分钟带宽的QTL5个,其中1B和1D染色体上的QTL在3种环境下均能检测到,贡献率为11.7%~33.9%。发现峰值粘度QTL4个,分布在1A、1B、3A和7B染色体上;检测出稀懈值QTL5个,位于1B、4A、5B、6B和7A染色体上。1B染色体上存在同时控制Zeleny沉降值、和面时间、8分钟带宽、峰值粘度和稀懈值的QTL,与最近标记Glu-B3j连锁距离为0.1~0.8cM,说明1BL/1RS易位对这些性状有重要影响;1D染色体上存在同时控制Zeleny沉降值、和面时间和8分钟带宽的QTL,与最近的标记Dx5+Dy10连锁距离为2.5~3.3cM,表明Dx5+Dy10高分子量谷蛋白亚基对这3个性状影响很大。和面时间和8分钟带宽位于1B和1D染色体的QTL以及稀懈值位于1B染色体上的QTL在3个地点均能检测到,具有环境稳定性。【结论】本研究定位的品质性状的标记可作为小麦品质分子育种的工具。
DOI:10.3864/j.issn.0578-1752.2008.02.003URL [本文引用: 1]

【目的】发掘重要性状的QTL及其分子标记进行小麦品质分子改良。【方法】采用PH82-2/内乡188杂交后代240个F5:6家系,按照Latinizedα-lattice设计,2004~2005年度分别种植在河南焦作、安阳和山东泰安。对籽粒蛋白质含量、Zeleny沉降值、和面时间、8分钟带宽、峰值粘度和稀懈值进行测定,利用188个SSR标记和4个蛋白标记构建遗传连锁图谱,采用复合区间作图法(CIM)对上述6个品质性状进行QTL定位。【结果】籽粒蛋白质含量检测出3个QTL,分布在3A、3B染色体上。在1B、1D和3B染色体上检测到3个控制Zeleny沉降值的QTL,其中位于1B和1D染色体上的QTL在3个地点均检测到,可解释5.5%~17.6%表型变异。发现3个控制和面时间的QTL,分布在1B和1D染色体上,在3个地点均能检测到,贡献率为7.9%~55.3%;检测出8分钟带宽的QTL5个,其中1B和1D染色体上的QTL在3种环境下均能检测到,贡献率为11.7%~33.9%。发现峰值粘度QTL4个,分布在1A、1B、3A和7B染色体上;检测出稀懈值QTL5个,位于1B、4A、5B、6B和7A染色体上。1B染色体上存在同时控制Zeleny沉降值、和面时间、8分钟带宽、峰值粘度和稀懈值的QTL,与最近标记Glu-B3j连锁距离为0.1~0.8cM,说明1BL/1RS易位对这些性状有重要影响;1D染色体上存在同时控制Zeleny沉降值、和面时间和8分钟带宽的QTL,与最近的标记Dx5+Dy10连锁距离为2.5~3.3cM,表明Dx5+Dy10高分子量谷蛋白亚基对这3个性状影响很大。和面时间和8分钟带宽位于1B和1D染色体的QTL以及稀懈值位于1B染色体上的QTL在3个地点均能检测到,具有环境稳定性。【结论】本研究定位的品质性状的标记可作为小麦品质分子育种的工具。
DOI:10.1038/s41598-017-04028-6URLPMID:28630475 [本文引用: 1]

In crop plants, a high-density genetic linkage map is essential for both genetic and genomic researches. The complexity and the large size of wheat genome have hampered the acquisition of a high-resolution genetic map. In this study, we report a high-density genetic map based on an individual mapping population using the Affymetrix Wheat660K single-nucleotide polymorphism (SNP) array as a probe in hexaploid wheat. The resultant genetic map consisted of 119 566 loci spanning 4424.4 cM, and 119 001 of those loci were SNP markers. This genetic map showed good collinearity with the 90 K and 820 K consensus genetic maps and was also in accordance with the recently released wheat whole genome assembly. The high-density wheat genetic map will provide a major resource for future genetic and genomic research in wheat. Moreover, a comparative genomics analysis among gramineous plant genomes was conducted based on the high-density wheat genetic map, providing an overview of the structural relationships among theses gramineous plant genomes. A major stable quantitative trait locus (QTL) for kernel number per spike was characterized, providing a solid foundation for the future high-resolution mapping and map-based cloning of the targeted QTL.
DOI:10.1038/srep41247URLPMID:28134278 [本文引用: 1]

A Chinese wheat mini core collection was genotyped using the wheat 965K iSelect SNP array.
DOI:10.1371/journal.pone.0022306URL [本文引用: 1]
[本文引用: 1]
